Introduzione
Nei primi embrioni, i morfogeni secreti che regolano un numero limitato di percorsi di segnalazione hanno il compito di istruire la differenziazione cellulare dinamica e coordinata attraverso il tessuto in via di sviluppo. Nel giorno 6,5 embrione di topo, una gerarchia di segnalazione mediata da BMP, WNT, e NODAL porta le informazioni di posizione nell’epiblasto che porta alla formazione dell’asse e alla specifica dello strato germinale (Arnold e Robertson, 2009). Ognuno di questi morfogeni induce l’espressione del ligando successivo (la segnalazione BMP induce l’espressione WNT, e la segnalazione WNT induce l’espressione NODAL), e tutti inducono l’espressione dei propri inibitori. In che misura questa cascata di segnalazione è coinvolta nella gastrulazione umana può ora essere studiata utilizzando modelli in vitro di embrioni umani precoci derivati da cellule staminali embrionali umane (hESC).
Abbiamo già dimostrato che il primo passo di questa gerarchia di segnalazione è conservato negli esseri umani e che in risposta alle BMP4, gli hESC cresciuti in colonie geometricamente confinate, si auto-organizzano per indurre e modellare strati embrionali ed extra-embrionici di germi. Ectoderma è stato specificato al centro di colonie circolari, tessuto extra-embrionico al bordo e mesendoderma in mezzo. Firme molecolari di gastrulazione, come l’induzione di SNAIL e l’attivazione di pERK potrebbe anche essere osservato (Warmflash et al., 2014). Coerentemente con la conservazione del secondo passo della gerarchia di segnalazione, BMP4, che segnala attraverso SMAD1/5/8, è stato dimostrato che induce l’espressione dei ligandi WNT, che possono anche indurre l’emergere di una vena primitiva e l’auto-organizzazione degli strati germinali embrionali in questo sistema (Etoc et al., 2016; Martyn et al., 2018). Noi, quindi, abbiamo definito questo sistema un gastruloide umano.
La terza via, SMAD2/3, che segnala per conto dei ligandi ACTIVIN e NODAL, ha un ruolo intrigante. Da un lato, la segnalazione SMAD2/3 è necessaria per il mantenimento della pluripotenza dell’hESC (James et al., 2005; Vallier et al., 2005). Dall’altro lato, il percorso ACTIVIN/NODAL è necessario per la specificazione del destino, agendo come morfogeno per modellare la blastula degli embrioni vertebrati dall’anfibio al topo (McDowell e Gurdon, 1999; Robertson, 2014). Il percorso ACTIVIN/NODAL è anche necessario per l’auto-organizzazione dei gastruloidi umani, in quanto l’inibizione della SMAD2/3 blocca la formazione di strisce primitive e l’induzione del mesendoderma da parte di BMP4 ed elimina i destini del mesendoderma anteriore indotti da WNT3A (Warmflash et al., 2014; Martyn et al., 2018). Inoltre, la co-presentazione di ACTIVIN con WNT3A a colonie di HESC micropatterned hESC porta all’induzione del marcatore specifico dell’organizzatore GOOSECOID, e ad una vena primitiva umana funzionale, che quando innestata in embrioni di pulcino induce la formazione di un asse secondario (Martyn et al., 2018).
Nonostante questi risultati, il meccanismo con cui la segnalazione SMAD2/3 può specificare sia la pluripotenza che la differenziazione dell’hESC rimane perplesso, e una serie di domande chiave rimangono senza risposta. Tra queste: come può un singolo percorso di segnalazione svolgere queste due funzioni opposte prima e dopo l’inizio della gastrulazione? In che misura la dinamica della segnalazione SMAD influisce su queste letture? Infine, in che misura le cellule hanno una memoria della segnalazione passata?
In questo studio abbiamo voluto fornire risposte altamente quantitative a queste domande seguendo la dinamica della segnalazione del TGFβ durante l’auto-organizzazione dei gastruloidi umani. L’imaging delle linee di reporter hESC ingegnerizzato da CRISPR/Cas9-mediata tagging di SMAD1, SMAD2, o SMAD4 ha dimostrato che ogni ramo del percorso ha una dinamica di segnalazione distinta. In risposta ad ACTIVIN, SMAD2 ha mostrato una drammatica traslocazione nucleare transitoria, in netto contrasto con la risposta stabile di SMAD1 indotta da BMP4. La stimolazione ACTIVIN ha indotto la trascrizione del gene mesendodermico transitorio, che è correlato con la dinamica SMAD2. Questa induzione, tuttavia, non è stato sostenuto e le cellule tornato a pluripotenza in tempi successivi. È interessante notare che, pre-presentazione di WNT3A alle cellule, pur non cambiando la dinamica SMAD2 o l’espressione dei marcatori di pluripotenza, stabilizzato la risposta trascrizionale successiva trascrizione di ACTIVIN per mantenere destini mesendodermici. Ciò implica una capacità inaspettata delle cellule staminali embrionali umane di registrare la loro storia di segnalazione senza cambiamenti evidenti nel destino.
Risultati
I gastruloidi rispondono alla stimolazione ACTIVIN al confine della colonia
Un percorso funzionale SMAD2/3 è necessario sia per il mantenimento della pluripotenza, così come la differenziazione e l’auto-organizzazione dei gastruloidi umani a valle di BMP4 e WNT3A (Warmflash et al., 2014; Martyn et al., 2018). Nel contesto della differenziazione indotta da BMP4, il trattamento con un piccolo inibitore molecolare della segnalazione SMAD2/3, SB431542 (SB), ha eliminato tutti i destini mesendodermici, come indicato dalla perdita di cellule positive BRA e SOX17, lasciando solo i destini extra-embrionici ed ectodermici presunti, contrassegnati rispettivamente dall’espressione CDX2 e SOX2 (Figura 1A) (Warmflash et al., 2014). Presentazione del morfogeno a valle WNT3A indotto formazione di striscia primitiva al confine della colonia, e l’aggiunta di SB in questo contesto rimosso la popolazione mesendodermica positiva SOX17 (Figura 1A) (Martyn et al., 2018). Al fine di chiedersi se l’auto-organizzazione gastruloide può essere avviata al punto ACTIVIN/NODAL nella gerarchia di segnalazione, abbiamo stimolato colonie micropatterned cresciuto in media condizionati con alte concentrazioni di ACTIVIN A (indicato come ACTIVIN in tutto questo lavoro). In contrasto con la stimolazione con BMP4 e WNT3A, nessuna differenziazione è stata osservata dopo 48 ore con la sola ACTIVIN (Figura 1B). Sorprendentemente, la mancanza di differenziazione in risposta all’ACTIVIN non era dovuta ad una mancanza di rilevamento del segnale, in quanto un aumento di SMAD2/3 nucleare è stato rilevato da immunofluorescenza al confine della colonia dopo un’ora di stimolazione (Figura 1C-D). Concludiamo che, mentre la segnalazione SMAD2/3 è necessaria per l’induzione del mesendoderma a valle del BMP4 e WNT3A nei gastruloidi umani, non è sufficiente per indurla.
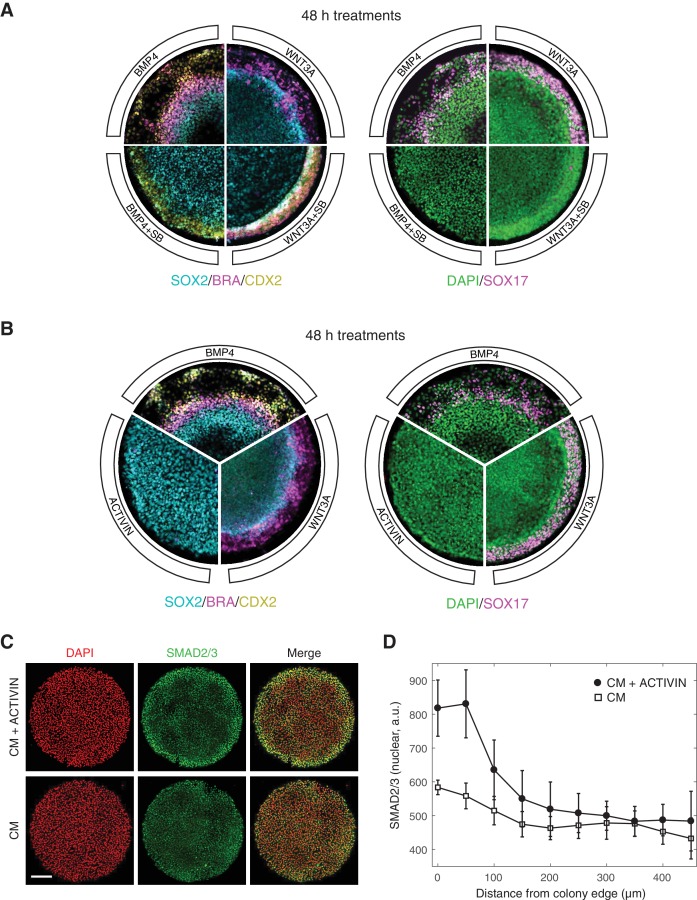
Figura 1.L’ATTIVINA modifica la differenziazione dei gastruloidi, ma non può indurla.(A) Colonie micropatterned in mezzi condizionati trattati per con BMP4 (50 ng/mL), BMP4 (50 ng/mL) +SB (10 µM), WNT3A (100 ng/mL), o WNT3A (100 ng/mL) +SB (10 µM) per 48 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. A sinistra: SOX2 (ciano), BRA (magenta), CDX2 (giallo). A destra: DAPI (verde), SOX17 (magenta). (B) Colonie micropatterned in mezzi condizionati trattati con BMP4 (50 ng/mL), WNT3A (100 ng/mL), o ACTIVIN (100 ng/mL) per 48 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. A sinistra: SOX2 (ciano), BRA (magenta), CDX2 (giallo). A destra: DAPI (verde), SOX17 (magenta). (C-D) Colonie micropatterned in mezzi condizionati (CM) trattate con ACTIVIN (100 ng/mL) per 1 ora o lasciate non trattate. Le colonie sono state fissate e analizzate per immunofluorescenza. (C) Immagini: DAPI (rosso), SMAD2/3 (verde). Barra di scala, 200 µm. (D) Quantificazione della fluorescenza nucleare media SMAD2/3 in funzione della posizione radiale dal bordo della colonia: CM + ACTIVIN (cerchi pieni), CM (quadrati aperti). Le barre di errore rappresentano la deviazione standard tra n = 5 (CM) e n = 6 (CM + ACTIVIN) colonie da un esperimento. Tutti gli esperimenti di coltura micropatterned sono stati eseguiti in almeno tre occasioni separate con risultati simili.

Figura 1.ACTIVIN modifica la differenziazione dei gastruloidi ma non può indurla.(A) Colonie micropatterned in mezzi condizionati trattati per con BMP4 (50 ng/mL), BMP4 (50 ng/mL) +SB (10 µM), WNT3A (100 ng/mL), o WNT3A (100 ng/mL) +SB (10 µM) per 48 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. A sinistra: SOX2 (ciano), BRA (magenta), CDX2 (giallo). A destra: DAPI (verde), SOX17 (magenta). (B) Colonie micropatterned in mezzi condizionati trattati con BMP4 (50 ng/mL), WNT3A (100 ng/mL), o ACTIVIN (100 ng/mL) per 48 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. A sinistra: SOX2 (ciano), BRA (magenta), CDX2 (giallo). A destra: DAPI (verde), SOX17 (magenta). (C-D) Colonie micropatterned in mezzi condizionati (CM) trattate con ACTIVIN (100 ng/mL) per 1 ora o lasciate non trattate. Le colonie sono state fissate e analizzate per immunofluorescenza. (C) Immagini: DAPI (rosso), SMAD2/3 (verde). Barra di scala, 200 µm. (D) Quantificazione della fluorescenza nucleare media SMAD2/3 in funzione della posizione radiale dal bordo della colonia: CM + ACTIVIN (cerchi pieni), CM (quadrati aperti). Le barre di errore rappresentano la deviazione standard tra n = 5 (CM) e n = 6 (CM + ACTIVIN) colonie da un esperimento. Tutti gli esperimenti di coltura micropatterned sono stati eseguiti in almeno tre occasioni separate con risultati simili.
Due rami del percorso TGFβ mostrano diverse dinamiche di segnalazione
L’incapacità di ACTIVIN/SMAD2 di indurre la differenziazione era in netto contrasto con la capacità di BMP/SMAD1 di indurre l’auto-organizzazione dei gastruloidi, compreso il patterning degli strati di germi mesodermi ed endodermi. Questo contrasto nell’attività dei due rami del percorso TGFβ ci ha spinto a valutare possibili differenze nella loro dinamica di segnalazione. Abbiamo usato l’ingegneria del genoma CRISPR/Cas9 su RUES2 per etichettare fluorescentemente il N-termino del recettore endogeno associato, R-SMAD1, con tagRFP (RUES2-RFP-SMAD1) e R-SMAD2 con mCitrino (RUES2-mCit-SMAD2) (Figura 2A e Figura 2-figure supplement 1). Poiché l’attivazione del percorso porta al legame di SMAD1 e 2 al co-SMAD, SMAD4, prima della traslocazione nucleare e della regolazione dell’espressione genica, è stata inclusa anche una linea SMAD4 con GFP (RUES2-GFP-SMAD4) (Figura 2A) (Nemashkalo et al., 2017). Ogni linea è stata anche trasfettata con elementi trasponibili ePiggyBac che portano un marcatore nucleare (H2B-mCitrina o H2B-mCherry) al fine di analizzare la risposta delle singole cellule (Figura 2-figure supplement 2A). Le proteine di fusione SMAD N-terminali hanno dimostrato di funzionare in modo simile alle proteine endogene in saggi biochimici e cellulari (Schmiererer e Hill, 2005). Inoltre, la dinamica di risposta SMAD misurata con le nostre linee di reporter, corrispondeva al comportamento delle proteine endogene misurate per immunofluorescenza e western blotting, come discusso di seguito.
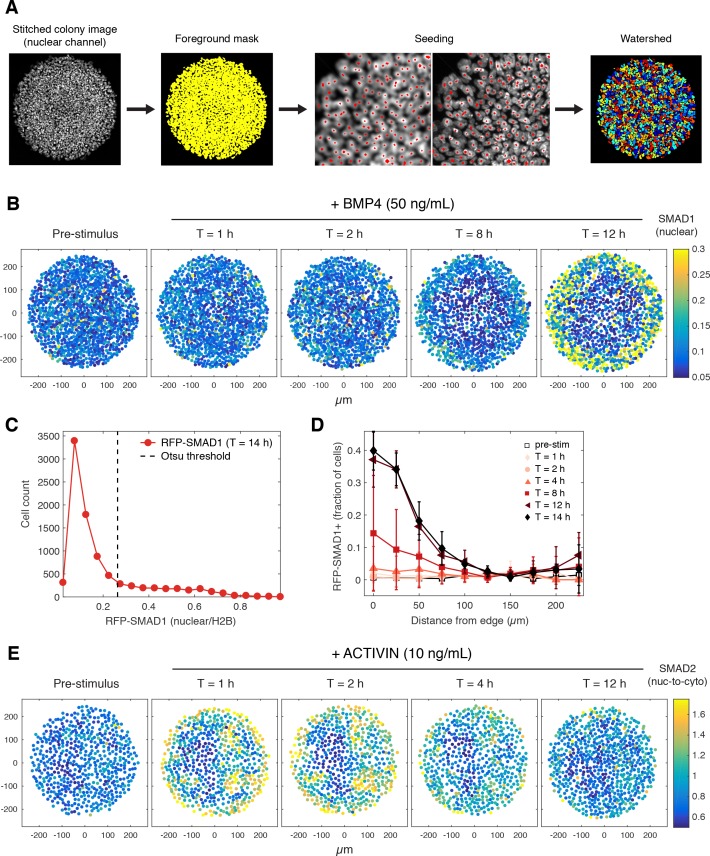
Figura 2-video 3.Figura 2—video 3. Due rami del percorso TGFβ mostrano diverse dinamiche di segnalazione.R-SMAD reporter line generation.Quantificazione della dinamica R-SMAD a livello di singola cellula in colonie micropatterned.RUES2-RFP-SMAD1 colonia micropatterned stimolata con BMP4.RUES2-RFP-SMAD1 colonia micropatterned unstimulated.RUES2-mCit-SMAD2 colonia micropatterned stimolata con ACTIVIN.(A) La segnalazione BMP e la segnalazione ACTIVIN rappresentano i due rami del percorso TGFβ. Segnali BMP attraverso R-SMAD1 e segnali ACTIVIN attraverso R-SMAD2. R-SMAD formano un complesso con co-SMAD4 e il complesso regola l’espressione del gene target. (B-C) RUES2-RFP-SMAD1 cresciuto su colonie micropatterned in E7 e stimolato con BMP4 (50 ng/mL). La risposta delle cellule al bordo della colonia (B) e vicino al centro della colonia (C) in funzione del tempo dopo la presentazione BMP4. L’intervallo di intensità è stato regolato agli stessi valori minimi e massimi in tutte le immagini sia in (B) che in (C). Barre di scala, 25 µm. (D) Segnale nucleare medio RFP-SMAD1 in funzione della posizione radiale all’interno della colonia in diversi punti temporali dopo il trattamento BMP4. L’intensità nucleare monocellulare RFP-SMAD1 è stata quantificata e normalizzata al segnale monocellulare H2B-mCitrino. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento. (E-F) RUES2-mCit-SMAD2 cresciuto su colonie micropatterned in E7 e stimolati con ACTIVIN (10 ng/mL). La risposta delle cellule al bordo della colonia (E) e vicino al centro della colonia (F) in funzione del tempo dopo la presentazione ACTIVIN. L’intervallo di intensità è stato regolato agli stessi valori minimi e massimi in tutte le immagini sia in (E) che in (F). Le barre di scala rappresentano 25 µm. (G) Media mCitrine-SMAD2 segnale nucleare-citrino-SMAD2 in funzione della posizione radiale all’interno della colonia in diversi punti temporali dopo il trattamento con ACTIVIN. L’intensità di mCitrino nucleare monocellulare è stata quantificata e normalizzata al segnale citrino monocellulare citrino. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento.(A) Schema della modifica del locus SMAD1 endogeno tramite ingegneria del genoma CRISPR/Cas9. Un gene di resistenza alla blasticidina (BsdR) è separato dalla fusione tagRFP-SMAD1 da un peptide auto-cleante T2A. La sequenza di codifica SMAD1 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (B) L’analisi della sequenza utilizzando primer specifici per loci (frecce magenta in A) ha dimostrato che entrambi gli alleli SMAD1 sono stati modificati. La traccia mostra la sequenza di alleli modificata. (C) Le cellule RUES2-RFP-SMAD1 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (D) RUES2-RFP-SMAD1 ha un cariotipo XX normale. (E) Schema della modifica del locus SMAD2 endogeno tramite ingegneria del genoma CRISPR/Cas9. Un gene di resistenza alla puromicina (PuroR) è separato dalla fusione mCitrine-SMAD2 da un peptide auto-cleante T2A. La sequenza di codifica SMAD2 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (F) L’analisi della sequenza con l’uso di primer specifici per loci (frecce magenta in E) ha dimostrato che solo uno degli alleli SMAD2 era stato preso di mira. La traccia mostra la sequenza di alleli modificata. (G) Le cellule RUES2-mCit-SMAD2 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (H) RUES2-mCit-SMAD2 ha un cariotipo XX normale.(A) Schema della pipeline di analisi delle immagini per esperimenti di coltura a micropatterned. L’immagine del canale nucleare, cucita e corretta in background, è stata fissata come soglia per generare una maschera in primo piano. L’immagine originale è stata poi filtrata e i massimi locali, che rappresentano i singoli nuclei, sono stati rilevati e utilizzati come semi per la segmentazione dello spartiacque. (B) RUES2-RFP-SMAD1 cresciuto su colonie micropatterned in E7 e stimolato con BMP4 (50 ng/mL). Il segnale nucleare RFP-SMAD1 (normalizzato a H2B) è stato quantificato in singole cellule all’interno della colonia micropatterned. (C-D) Analisi binaria dei dati di risposta SMAD1 mostrati in Figura 2D. (C) L’istogramma della risposta RFP-SMAD1 a T = 14 ore dopo la stimolazione con BMP4 è stato soglia utilizzando il metodo di Otsu (linea tratteggiata). (D) La soglia è stata applicata in ogni punto temporale per determinare la fazione di SMAD1 +nuclei in funzione della posizione radiale all’interno della colonia. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento. (E) RUES2-mCit-SMAD2 cresciuto su colonie micropatterned in E7 stimolati e con ACTIVIN (10 ng/mL). Il mCitrine-SMAD2 rapporto nucleare a citoplasma è stato quantificato in singole cellule all’interno della colonia micropatterned.BMP4 (50 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. La marca temporale mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.ACTIVIN (10 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCherry e mCitrine-SMAD2. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.
Abbiamo iniziato i nostri studi dinamici dei due rami della segnalazione del TGFβ in cellule RUES2 cresciute su colonie micropatterned in un mezzo chimicamente definito (TeSR-E7), che è una versione del mezzo E8 privo di siero che può mantenere gli HESC e che manca di qualsiasi legante TGFβ (Chen et al., 2011). Pertanto, in E7 i livelli esogeni di TGFβ potrebbero essere controllati con precisione. In risposta a BMP4, abbiamo rilevato un aumento del segnale nucleare SMAD1 che era stabile oltre 12 ore (Figura 2B-D, Figura 2-figure supplement 2B, Figura 2-videos 1 e 2). La risposta SMAD1 è stata osservata al margine della colonia, in linea con i nostri precedenti risultati di immunofluorescenza e con la nostra scoperta che i recettori TGFβ sono localizzati sulla superficie apicale solo al confine della colonia (Warmflash et al., 2014; Etoc et al., 2016). Nel nostro lavoro precedente abbiamo quantificato la risposta SMAD1 rilevata dall’immunofluorescenza come il numero di nuclei positivi all’interno della colonia (Etoc et al., 2016). Anche se la nostra linea di reporter ci permette di tracciare il livello di SMAD1 nucleare nelle singole cellule, in contrapposizione ad una quantificazione binaria on/off, otteniamo risultati simili a quelli mostrati nella Figura 2D con un’analisi binaria della linea di reporter RFP-SMAD1 (Figura 2-figure supplement 2C-D). In risposta ad ACTIVIN, SMAD2 d’altra parte, ha risposto solo transitoriamente: un impulso di traslocazione nucleare durante le prime 1-2 ore è stato seguito da una graduale diminuzione durante le successive 6 ore (Figura 2E-G, Figura 2-figure supplement 2E, e Figura 2-video 3). È interessante notare che il livello nucleare SMAD2 a lungo termine non è tornato completamente al livello di pre-stimolo. Come nel caso di SMAD1, la risposta SMAD2 è stata più alta al margine della colonia, anche in questo caso coerente con la localizzazione apicale dei recettori (Etoc et al., 2016).

Figura 2-video 3.Due rami del percorso TGFβ visualizzare diverse dinamiche di segnalazione.R-SMAD reporter line generation.Quantificazione della dinamica R-SMAD a livello di singola cellula in colonie micropatterned.RUES2-RFP-SMAD1 colonia micropatterned stimolata con BMP4.RUES2-RFP-SMAD1 colonia micropatterned non stimolata.RUES2-mCit-SMAD2 colonia micropatterned stimolata con ACTIVIN.(A) La segnalazione BMP e ACTIVIN rappresentano i due rami del percorso TGFβ. Segnali BMP attraverso R-SMAD1 e segnali ACTIVIN attraverso R-SMAD2. R-SMAD formano un complesso con co-SMAD4 e il complesso regola l’espressione del gene target. (B-C) RUES2-RFP-SMAD1 cresciuto su colonie micropatterned in E7 e stimolato con BMP4 (50 ng/mL). La risposta delle cellule al bordo della colonia (B) e vicino al centro della colonia (C) in funzione del tempo dopo la presentazione BMP4. L’intervallo di intensità è stato regolato agli stessi valori minimi e massimi in tutte le immagini sia in (B) che in (C). Barre di scala, 25 µm. (D) Segnale nucleare medio RFP-SMAD1 in funzione della posizione radiale all’interno della colonia in diversi punti temporali dopo il trattamento BMP4. L’intensità nucleare monocellulare RFP-SMAD1 è stata quantificata e normalizzata al segnale monocellulare H2B-mCitrino. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento. (E-F) RUES2-mCit-SMAD2 cresciuto su colonie micropatterned in E7 e stimolati con ACTIVIN (10 ng/mL). La risposta delle cellule al bordo della colonia (E) e vicino al centro della colonia (F) in funzione del tempo dopo la presentazione ACTIVIN. L’intervallo di intensità è stato regolato agli stessi valori minimi e massimi in tutte le immagini sia in (E) che in (F). Le barre di scala rappresentano 25 µm. (G) Media mCitrine-SMAD2 segnale nucleare-citrino-SMAD2 in funzione della posizione radiale all’interno della colonia in diversi punti temporali dopo il trattamento con ACTIVIN. L’intensità di mCitrino nucleare monocellulare è stata quantificata e normalizzata al segnale citrino monocellulare citrino. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento.(A) Schema della modifica del locus SMAD1 endogeno via CRISPR / Cas9 ingegneria del genoma. Un gene di resistenza alla blasticidina (BsdR) è separato dalla fusione tagRFP-SMAD1 da un peptide auto-cleante T2A. La sequenza di codifica SMAD1 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (B) L’analisi della sequenza utilizzando primer specifici per loci (frecce magenta in A) ha dimostrato che entrambi gli alleli SMAD1 sono stati modificati. La traccia mostra la sequenza di alleli modificata. (C) Le cellule RUES2-RFP-SMAD1 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (D) RUES2-RFP-SMAD1 ha un cariotipo XX normale. (E) Schema della modifica del locus SMAD2 endogeno tramite ingegneria del genoma CRISPR/Cas9. Un gene di resistenza alla puromicina (PuroR) è separato dalla fusione mCitrine-SMAD2 da un peptide auto-cleante T2A. La sequenza di codifica SMAD2 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (F) L’analisi della sequenza con l’uso di primer specifici per loci (frecce magenta in E) ha dimostrato che solo uno degli alleli SMAD2 era stato preso di mira. La traccia mostra la sequenza di alleli modificata. (G) Le cellule RUES2-mCit-SMAD2 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (H) RUES2-mCit-SMAD2 ha un cariotipo XX normale.(A) Schema della pipeline di analisi delle immagini per esperimenti di coltura micropatterned. L’immagine del canale nucleare suturato e corretto sfondo è stato soglia per generare una maschera in primo piano. L’immagine originale è stata poi filtrata e i massimi locali, che rappresentano i singoli nuclei, sono stati rilevati e utilizzati come semi per la segmentazione dello spartiacque. (B) RUES2-RFP-SMAD1 cresciuto su colonie micropatterned in E7 e stimolato con BMP4 (50 ng/mL). Il segnale nucleare RFP-SMAD1 (normalizzato a H2B) è stato quantificato in singole cellule all’interno della colonia micropatterned. (C-D) Analisi binaria dei dati di risposta SMAD1 mostrati in Figura 2D. (C) L’istogramma della risposta RFP-SMAD1 a T = 14 ore dopo la stimolazione con BMP4 è stato soglia utilizzando il metodo di Otsu (linea tratteggiata). (D) La soglia è stata applicata in ogni punto temporale per determinare la fazione di SMAD1 +nuclei in funzione della posizione radiale all’interno della colonia. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento. (E) RUES2-mCit-SMAD2 cresciuto su colonie micropatterned in E7 stimolati e con ACTIVIN (10 ng/mL). Il mCitrine-SMAD2 rapporto nucleare a citoplasma è stato quantificato in singole cellule all’interno della colonia micropatterned.BMP4 (50 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. La marca temporale mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.ACTIVIN (10 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCherry e mCitrine-SMAD2. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.
Figura 2-figure supplemento 1.R-SMAD generazione linea reporter.(A) Schema della modifica del locus SMAD1 endogeno via CRISPR / Cas9 ingegneria del genoma. Un gene di resistenza alla blasticidina (BsdR) è separato dalla fusione tagRFP-SMAD1 da un peptide auto-cleante T2A. La sequenza di codifica SMAD1 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (B) L’analisi della sequenza utilizzando primer specifici per loci (frecce magenta in A) ha dimostrato che entrambi gli alleli SMAD1 sono stati modificati. La traccia mostra la sequenza di alleli modificata. (C) Le cellule RUES2-RFP-SMAD1 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (D) RUES2-RFP-SMAD1 ha un cariotipo XX normale. (E) Schema della modifica del locus SMAD2 endogeno tramite ingegneria del genoma CRISPR/Cas9. Un gene di resistenza alla puromicina (PuroR) è separato dalla fusione mCitrine-SMAD2 da un peptide auto-cleante T2A. La sequenza di codifica SMAD2 (e1) e le regioni non tradotte a monte non sono interessate dalla modifica. (F) L’analisi della sequenza con l’uso di primer specifici per loci (frecce magenta in E) ha dimostrato che solo uno degli alleli SMAD2 era stato preso di mira. La traccia mostra la sequenza di alleli modificata. (G) Le cellule RUES2-mCit-SMAD2 mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Campioni separati sono stati preparati per ogni marcatore e la colorazione di immunofluorescenza è stata effettuata utilizzando Alexa Fluor 647 anticorpi secondari coniugati per evitare il segnale dai costrutti reporter: NANOG, OCT4, e SOX2 (verde) e DAPI (rosso). Barra di scala, 50 µM. (H) RUES2-mCit-SMAD2 ha un cariotipo XX normale.

Figura 2-figure supplement 2.Quantificazione della dinamica R-SMAD a livello di singola cellula in colonie micropatterned.(A) Schema della pipeline di analisi delle immagini per esperimenti di coltura a micropatterned. L’immagine del canale nucleare cucita e corretta in background è stata fissata come soglia per generare una maschera in primo piano. L’immagine originale è stata poi filtrata e i massimi locali, che rappresentano i singoli nuclei, sono stati rilevati e utilizzati come semi per la segmentazione dello spartiacque. (B) RUES2-RFP-SMAD1 cresciuto su colonie micropatterned in E7 e stimolato con BMP4 (50 ng/mL). Il segnale nucleare RFP-SMAD1 (normalizzato a H2B) è stato quantificato in singole cellule all’interno della colonia micropatterned. (C-D) Analisi binaria dei dati di risposta SMAD1 mostrati in Figura 2D. (C) L’istogramma della risposta RFP-SMAD1 a T = 14 ore dopo la stimolazione con BMP4 è stato soglia utilizzando il metodo di Otsu (linea tratteggiata). (D) La soglia è stata applicata in ogni punto temporale per determinare la fazione di SMAD1 +nuclei in funzione della posizione radiale all’interno della colonia. Le barre di errore rappresentano la deviazione standard su n = 5 colonie da un esperimento. (E) RUES2-mCit-SMAD2 cresciuto su colonie micropatterned in E7 stimolati e con ACTIVIN (10 ng/mL). Il mCitrine-SMAD2 rapporto nucleare a citoplasma è stato quantificato in singole cellule all’interno della colonia micropatterned.

Figura 2-video 1.RUES2-RFP-SMAD1 colonia micropatterned stimolata con BMP4.BMP4 (50 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.

Figura 2-video 2.RUES2-RFP-SMAD1 micropatterned colony unstimulated.Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCitrino e RFP-SMAD1. L’orario mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.

Figura 2-video 3.Colonia micropatterned RUES2-mCit-SMAD2 stimolata con ACTIVIN.ACTIVIN (10 ng/mL, aggiunto a T = 0 ore). Le immagini sono state acquisite ogni 10 minuti in due canali corrispondenti a H2B-mCherry e mCitrine-SMAD2. La marcatura oraria mostra ore e minuti (hh:mm). Raggio della colonia, 500 µm.
La dinamica di risposta monocellulare riflette il comportamento ai margini dei gastruloidi
Al fine di studiare le dinamiche di segnalazione a livello di singola cellula e di eliminare le influenze del modificatore su entrambi i rami SMAD provenienti da cellule vicine all’interno della colonia micropatterned, abbiamo eseguito lo stesso esperimento su cellule dissociate cresciute in condizioni di coltura regolare (Figura 3A). La risposta SMAD1 a BMP era stabile e il livello di RFP-SMAD1 nucleare dipendeva dalla concentrazione di ligandi BMP4 in accordo con le precedenti misurazioni di immunofluorescenza monocellulare (Figura 3B-C) (Etoc et al., 2016). L’aumento del segnale nucleare medio in funzione del tempo è risultato da quasi tutte le cellule che hanno risposto al BMP4 aggiunto, che è evidente nello spostamento dell’istogramma dell’intensità nucleare RFP-SMAD1 (Figura 3-figure supplement 1A). La risposta SMAD2 ad ACTIVIN era ancora una volta transitoria con un segnale nucleare che è rimasto elevato in tempi più lunghi, come dimostrato dalla nostra linea reporter e dall’analisi di immunofluorescenza e western blot della linea parentale RUES2 (Figura 3D-E, Figura 3-figure supplement 1B-C). Questo è coerente con altri dati basati sull’immunofluorescenza (Heemskerk et al., 2017). Anche se la risposta adattiva è un po’ più pronunciata nella nostra configurazione sperimentale. Come per la risposta SMAD1, la risposta media SMAD2 è il risultato di quasi tutte le cellule che rispondono con dinamiche simili, che è evidente nello spostamento su e giù nell’istogramma dell’intensità nucleare SMAD2 (Figura 3-figure supplement 1D). La risposta di picco SMAD2 ha mostrato una forte dipendenza sigmoidale dalla concentrazione di ACTIVIN (Figura 3-figure supplement 1E). Tuttavia, la linea di base post-stimolazione, definita come il rapporto medio SMAD2 nucleare-citoplasmatico a T > 8 ore dopo l’aggiunta di ACTIVIN, così come la scala temporale della risposta transitoria, non dipendeva dalla dose di ACTIVIN superiore a 0,5 ng/mL (Figura 3-figure supplement 1E-F). Questo comportamento transitorio SMAD2 segnalazione non è risultato da esaurimento di ACTIVIN dal mezzo, come mezzo di coltura recuperato da cellule che sono state incubate con ACTIVIN per 12 ore, ancora indotto una risposta SMAD2 quando presentato alle cellule non stimolate (Figura 3-figure supplemento 1G). SMAD4 ha seguito la dinamica delle R-SMADs pertinenti dopo la presentazione di BMP4 o ACTIVIN (Figura 3F-G, Figura 3-figure supplemento 1H). Le risposte stabili e adattive di SMAD4 a BMP e ACTIVIN, rispettivamente, sono coerenti con i recenti esperimenti che hanno utilizzato anche questa linea di reporter (Nemashkalo et al., 2017; Heemskerk et al., 2017). Così, in risposta ai loro ligandi attivatori, i due rami del percorso TGFβ mostrano dinamiche distinte di trasduzione del segnale sia per l’R-SMAD che per il co-SMAD. Questo dimostra anche che la modifica dei segnali non influenza la dinamica della risposta ai margini delle colonie micropatterned.
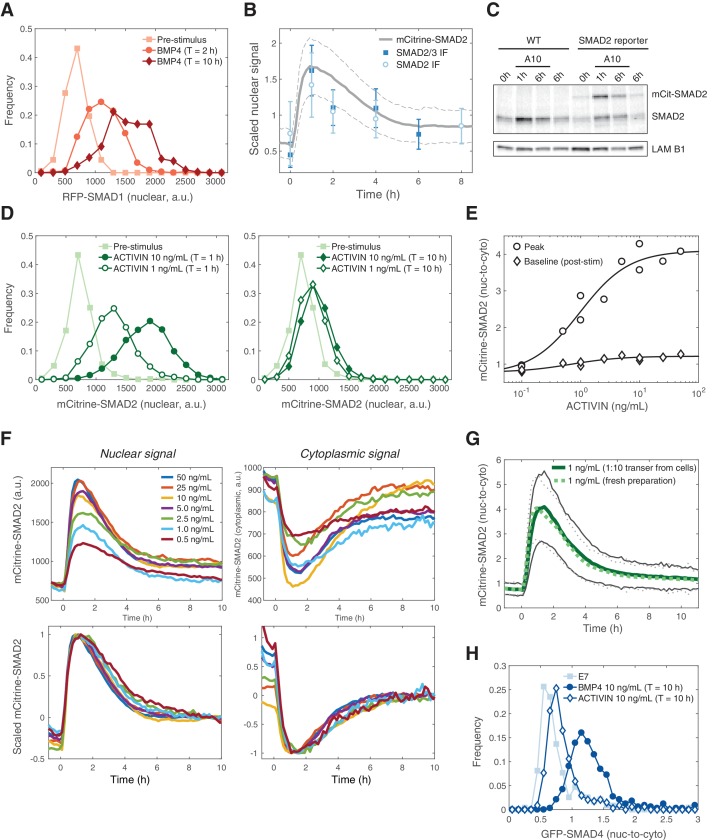
Figura 3-figure supplement 1.BMP e ACTIVIN generano diverse dinamiche di risposta SMAD a cella singola.BMP e ACTIVIN generano diverse dinamiche di risposta SMAD a cella singola.(A) Schema che descrive il protocollo sperimentale monocellulare. (B) RFP-SMAD1 risposta in singole cellule a BMP4 (5 ng/mL, aggiunto a T = 0 ore). (C) Quantificazione del segnale nucleare RFP-SMAD1 in funzione del tempo e della concentrazione BMP4. Le immagini sono state acquisite ogni 10 min. Le linee continue rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Le linee tratteggiate rappresentano la deviazione standard della popolazione per le condizioni BMP4 (rosso chiaro) e BMP4 (5 ng/mL, rosso scuro). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (D) mCitrine-SMAD2 risposta in singole cellule in ACTIVIN (10 ng/mL, aggiunto a T = 0 ore). (E) Quantificazione del mCitrine-SMAD2 rapporto nucleare-citrino-SMAD2 in funzione del tempo e della concentrazione di ACTIVIN. Le immagini sono state acquisite ogni 10 min. Le linee solide rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Le linee tratteggiate rappresentano la deviazione standard della popolazione per le condizioni -ACTIVINA (verde più chiaro) e ACTIVINA (10 ng/mL, verde più scuro). I dati sono stati ottenuti collettivamente da tre esperimenti indipendenti. (F) GFP-SMAD4 risposta in singole cellule in ACTIVIN (10 ng/mL) o BMP4 (10 ng/mL) o cellule che sono stati lasciati non trattati (E7). Ligandi sono stati aggiunti a T = 0 ore. (G) Quantificazione del rapporto nucleare GFP-SMAD4 in funzione del tempo in E7 (blu), E7 +BMP4 (10 ng/mL, rosso), o E7 +ACTIVIN (10 ng/mL, verde). Le immagini sono state acquisite ogni 10 min. Le linee continue rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. Barre di scala, 25 µm.(A) Istogrammi del segnale nucleare monocellulare RFP-SMAD1 in diversi punti temporali dopo la stimolazione con BMP4 (5 ng/mL). (B) Confronto tra la risposta nucleare media mCitrine-SMAD2 della linea reporter (linea grigia solida) e la risposta della linea parentale non modificata mediante immunofluorescenza con anticorpi contro SMAD2/3 (quadrati blu) o SMAD2 (cerchi aperti) in singole cellule trattate con ACTIVIN (10 ng/mL). Al fine di confrontare le variazioni relative dell’intensità della fluorescenza, i segnali sono stati scalati in base al loro valore medio in tutti i punti temporali. Le linee grigie tratteggiate e le barre di errore rappresentano la deviazione standard in ogni punto temporale in mCit-SMAD2 (n > 200 celle per punto temporale) e le misure di immunofluorescenza (n > 3.000 celle per punto temporale), rispettivamente. I dati sono stati raccolti da tre esperimenti indipendenti. (C) Western blot analisi Western blot di SMAD2 in frazioni nucleari raccolti da RUES2 o RUES2-mCit-SMAD2 cellule dopo la presentazione di ACTIVIN (10 ng/mL). I campioni sono stati raccolti prima della presentazione di ACTIVIN (T = 0 ore), in due punti di tempo dopo il trattamento con ACTIVIN (T = 1 e 6 ore), e da un campione non trattato incubato in E7 (T = 6 ore). (D) Istogrammi del segnale nucleare mCitrine-SMAD2 monocellulare a seguito di stimolazione con ACTIVIN (10 ng/mL) o ACTIVIN (1 ng/mL) a T = 1 hr (sinistra) e T = 10 hr (destra). (E) Picco SMAD2 rapporto nucleare-citoplasmatico (cerchi) e rapporto di base post-stimolazione (diamanti) in funzione della concentrazione di ACTIVIN. Il rapporto di base è stato calcolato come rapporto medio per T > 8 ore. Le linee solide mostrano un adattamento Hill della formaR=a×LnLnLn+Kn+b, dove L è la concentrazione del ligando, n è il coefficiente Hill, K è il punto di inflessione, e a e b sono costanti. Per l’adattamento alla risposta al picco: n = 1,05, K = 0,97, a = 3,37, e b = 0,73. Per l’adattamento alla risposta di base post-stimolazione: n = 1,02, K = 0,68, a = 0,44, e b = 0,78. (F) Il segnale nucleare medio mCitrine-SMAD2 (in alto a sinistra) e il segnale citoplasmatico (in alto a destra) in funzione del tempo e della concentrazione di ACTIVIN. I segnali scalati (in basso a sinistra e a destra) sono stati normalizzati sottraendo il segnale di fluorescenza SMAD2 di coda (risposta media a T > 8 ore) e dividendo per il segnale di picco, che è il massimo nel caso del segnale nucleare e il minimo nel caso del segnale citoplasmatico. La scalatura fa collassare le curve indicando che la scala temporale della risposta ai transienti è simile ad ogni concentrazione. (G) mCitrine-SMAD2 risposta all’ACTIVINA (1 ng/mL, linea verde solida) diluita 1:10 da cellule incubate per 12 ore con ACTIVINA (10 ng/mL) o ACTIVINA appena preparata (1 ng/mL, linea verde tratteggiata). Le immagini sono state acquisite ogni 10 min. Le linee nere piene rappresentano la deviazione standard per la risposta di trasferimento 1:10 e le linee grigie tratteggiate rappresentano la deviazione standard per la risposta alla preparazione fresca (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (H) Istogrammi del GFP-SMAD4 monocellulare rapporto nucleare-citoplasmatico in E7, E7 +BMP4 (10 ng/mL), e E7 +ACTIVIN (10 ng/mL) a T = 10 ore dopo l’aggiunta di ligando.

Figura 3-figure supplement 1.BMP e ACTIVIN generano diverse dinamiche di risposta SMAD a cella singola.BMP e ACTIVIN generano diverse dinamiche di risposta SMAD a cella singola.(A) Schema del protocollo sperimentale monocellulare. (B) RFP-SMAD1 risposta in singole cellule a BMP4 (5 ng/mL, aggiunto a T = 0 ore). (C) Quantificazione del segnale nucleare RFP-SMAD1 in funzione del tempo e della concentrazione BMP4. Le immagini sono state acquisite ogni 10 min. Le linee continue rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Le linee tratteggiate rappresentano la deviazione standard della popolazione per le condizioni BMP4 (rosso chiaro) e BMP4 (5 ng/mL, rosso scuro). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (D) mCitrine-SMAD2 risposta in singole cellule in ACTIVIN (10 ng/mL, aggiunto a T = 0 ore). (E) Quantificazione del mCitrine-SMAD2 rapporto nucleare-citrino-SMAD2 in funzione del tempo e della concentrazione di ACTIVIN. Le immagini sono state acquisite ogni 10 min. Le linee solide rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Le linee tratteggiate rappresentano la deviazione standard della popolazione per le condizioni -ACTIVINA (verde più chiaro) e ACTIVINA (10 ng/mL, verde più scuro). I dati sono stati ottenuti collettivamente da tre esperimenti indipendenti. (F) GFP-SMAD4 risposta in singole cellule in ACTIVIN (10 ng/mL) o BMP4 (10 ng/mL) o cellule che sono stati lasciati non trattati (E7). Ligandi sono stati aggiunti a T = 0 ore. (G) Quantificazione del rapporto nucleare GFP-SMAD4 in funzione del tempo in E7 (blu), E7 +BMP4 (10 ng/mL, rosso), o E7 +ACTIVIN (10 ng/mL, verde). Le immagini sono state acquisite ogni 10 min. Le linee continue rappresentano la risposta media in ogni punto temporale (n > 200 celle per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. Barre di scala, 25 µm.(A) Istogrammi del segnale nucleare monocellulare RFP-SMAD1 in diversi punti temporali in seguito alla stimolazione con BMP4 (5 ng/mL). (B) Confronto tra la risposta nucleare media mCitrine-SMAD2 della linea reporter (linea grigia solida) e la risposta della linea parentale non modificata mediante immunofluorescenza con anticorpi contro SMAD2/3 (quadrati blu) o SMAD2 (cerchi aperti) in singole cellule trattate con ACTIVIN (10 ng/mL). Al fine di confrontare le variazioni relative dell’intensità della fluorescenza, i segnali sono stati scalati in base al loro valore medio in tutti i punti temporali. Le linee grigie tratteggiate e le barre di errore rappresentano la deviazione standard in ogni punto temporale in mCit-SMAD2 (n > 200 celle per punto temporale) e le misure di immunofluorescenza (n > 3.000 celle per punto temporale), rispettivamente. I dati sono stati raccolti da tre esperimenti indipendenti. (C) Western blot analisi Western blot di SMAD2 in frazioni nucleari raccolti da RUES2 o RUES2-mCit-SMAD2 cellule dopo la presentazione di ACTIVIN (10 ng/mL). I campioni sono stati raccolti prima della presentazione di ACTIVIN (T = 0 ore), in due punti di tempo dopo il trattamento con ACTIVIN (T = 1 e 6 ore), e da un campione non trattato incubato in E7 (T = 6 ore). (D) Istogrammi del segnale nucleare mCitrine-SMAD2 monocellulare a seguito di stimolazione con ACTIVIN (10 ng/mL) o ACTIVIN (1 ng/mL) a T = 1 hr (sinistra) e T = 10 hr (destra). (E) Picco SMAD2 rapporto nucleare-citoplasmatico (cerchi) e rapporto di base post-stimolazione (diamanti) in funzione della concentrazione di ACTIVIN. Il rapporto di base è stato calcolato come rapporto medio per T > 8 ore. Le linee solide mostrano un adattamento Hill della formaR=a×LnLnLn+Kn+b, dove L è la concentrazione del ligando, n è il coefficiente Hill, K è il punto di inflessione, e a e b sono costanti. Per l’adattamento alla risposta al picco: n = 1,05, K = 0,97, a = 3,37, e b = 0,73. Per l’adattamento alla risposta di base post-stimolazione: n = 1,02, K = 0,68, a = 0,44, e b = 0,78. (F) Il segnale nucleare medio mCitrine-SMAD2 (in alto a sinistra) e il segnale citoplasmatico (in alto a destra) in funzione del tempo e della concentrazione di ACTIVIN. I segnali scalati (in basso a sinistra e a destra) sono stati normalizzati sottraendo il segnale di fluorescenza SMAD2 di coda (risposta media a T > 8 ore) e dividendo per il segnale di picco, che è il massimo nel caso del segnale nucleare e il minimo nel caso del segnale citoplasmatico. La scalatura fa collassare le curve indicando che la scala temporale della risposta ai transienti è simile ad ogni concentrazione. (G) mCitrine-SMAD2 risposta all’ACTIVINA (1 ng/mL, linea verde solida) diluita 1:10 da cellule incubate per 12 ore con ACTIVINA (10 ng/mL) o ACTIVINA appena preparata (1 ng/mL, linea verde tratteggiata). Le immagini sono state acquisite ogni 10 min. Le linee nere piene rappresentano la deviazione standard per la risposta di trasferimento 1:10 e le linee grigie tratteggiate rappresentano la deviazione standard per la risposta alla preparazione fresca (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (H) Istogrammi del GFP-SMAD4 monocellulare rapporto nucleare-citoplasmatico in E7, E7 +BMP4 (10 ng/mL), e E7 +ACTIVIN (10 ng/mL) a T = 10 ore dopo l’aggiunta di ligando.

Figura 3-figure supplement 1.BMP e ACTIVIN suscitano diverse dinamiche di risposta SMAD monocellulare.(A) Istogrammi del segnale nucleare monocellulare RFP-SMAD1 in diversi punti temporali dopo la stimolazione con BMP4 (5 ng/mL). (B) Confronto tra la risposta nucleare media mCitrine-SMAD2 della linea reporter (linea grigia solida) e la risposta della linea parentale non modificata mediante immunofluorescenza con anticorpi contro SMAD2/3 (quadrati blu) o SMAD2 (cerchi aperti) in singole cellule trattate con ACTIVIN (10 ng/mL). Al fine di confrontare le variazioni relative dell’intensità della fluorescenza, i segnali sono stati scalati in base al loro valore medio in tutti i punti temporali. Le linee grigie tratteggiate e le barre di errore rappresentano la deviazione standard in ogni punto temporale in mCit-SMAD2 (n > 200 celle per punto temporale) e le misure di immunofluorescenza (n > 3.000 celle per punto temporale), rispettivamente. I dati sono stati raccolti da tre esperimenti indipendenti. (C) Western blot analisi Western blot di SMAD2 in frazioni nucleari raccolti da RUES2 o RUES2-mCit-SMAD2 cellule dopo la presentazione di ACTIVIN (10 ng/mL). I campioni sono stati raccolti prima della presentazione di ACTIVIN (T = 0 ore), in due punti di tempo dopo il trattamento con ACTIVIN (T = 1 e 6 ore), e da un campione non trattato incubato in E7 (T = 6 ore). (D) Istogrammi del segnale nucleare mCitrine-SMAD2 monocellulare a seguito di stimolazione con ACTIVIN (10 ng/mL) o ACTIVIN (1 ng/mL) a T = 1 hr (sinistra) e T = 10 hr (destra). (E) Picco SMAD2 rapporto nucleare-citoplasmatico (cerchi) e rapporto di base post-stimolazione (diamanti) in funzione della concentrazione di ACTIVIN. Il rapporto di base è stato calcolato come rapporto medio per T > 8 ore. Le linee solide mostrano un adattamento Hill della formaR=a×LnLnLn+Kn+b, dove L è la concentrazione del ligando, n è il coefficiente Hill, K è il punto di inflessione, e a e b sono costanti. Per l’adattamento alla risposta al picco: n = 1,05, K = 0,97, a = 3,37, e b = 0,73. Per l’adattamento alla risposta di base post-stimolazione: n = 1,02, K = 0,68, a = 0,44, e b = 0,78. (F) Il segnale nucleare medio mCitrine-SMAD2 (in alto a sinistra) e il segnale citoplasmatico (in alto a destra) in funzione del tempo e della concentrazione di ACTIVIN. I segnali scalati (in basso a sinistra e a destra) sono stati normalizzati sottraendo il segnale di fluorescenza SMAD2 di coda (risposta media a T > 8 ore) e dividendo per il segnale di picco, che è il massimo nel caso del segnale nucleare e il minimo nel caso del segnale citoplasmatico. La scalatura fa collassare le curve indicando che la scala temporale della risposta ai transienti è simile ad ogni concentrazione. (G) mCitrine-SMAD2 risposta all’ACTIVINA (1 ng/mL, linea verde solida) diluita 1:10 da cellule incubate per 12 ore con ACTIVINA (10 ng/mL) o ACTIVINA appena preparata (1 ng/mL, linea verde tratteggiata). Le immagini sono state acquisite ogni 10 min. Le linee nere piene rappresentano la deviazione standard per la risposta di trasferimento 1:10 e le linee grigie tratteggiate rappresentano la deviazione standard per la risposta alla preparazione fresca (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (H) Istogrammi del GFP-SMAD4 monocellulare rapporto nucleare-citoplasmatico in E7, E7 +BMP4 (10 ng/mL), e E7 +ACTIVIN (10 ng/mL) a T = 10 ore dopo l’aggiunta di ligando.
ACTIVIN ottiene una risposta trascrizionale transitoria e stabile
Abbiamo già dimostrato che la segnalazione BMP4 induce una risposta trascrizionale sostenuta che porta alla differenziazione dei gastruloidi (Warmflash et al., 2014; Etoc et al., 2016). Ciò è coerente con la natura stabile della segnalazione SMAD1 presentata sopra. Il comportamento adattivo della segnalazione SMAD2 ci ha spinto a chiederci se il breve picco di segnalazione SMAD2 era sufficiente a suscitare una risposta trascrizionale e il destino cambia nelle cellule RUES2 esposte all’ACTIVIN. RNA-seq analisi è stata eseguita su cellule dissociate coltivate in E7 e E7 + ATTIVINA a 1, 2,5, 4, 8 e 12 ore dopo la stimolazione. 3529 geni 3529 ha mostrato un cambiamento nel livello di espressione di almeno due volte durante il corso del tempo sperimentale. Sono caduti in tre gruppi distinti. Il primo, che consisteva della maggior parte delle trascrizioni (2.956), ha raggiunto il picco a 2,5 ore e diminuito in punti successivi (Figura 4A, scatola magenta). Questo gruppo corrispondeva alla tempistica della risposta SMAD2 transitoria e comprendeva i regolatori chiave della differenziazione mesendodermica, come EOMES, HHEX, GATA2 e GATA3 (Figura 4A, dati fonte 1) (Teo et al., 2011; Loh et al., 2014). Il secondo gruppo, che consisteva di 452 trascrizioni, mostrava un’induzione stabile (Figura 4A, scatola arancione). Questo gruppo comprendeva geni espressi durante la pluripotenza, come NANOG, NODAL, LEFTY1, LEFTY2 e SMAD7 (Figura 4A, dati fonte 2) (Sato et al., 2003). Infine, il terzo gruppo, che consisteva di 121 trascrizioni, rappresentava geni che erano stabilmente o transitoriamente giù regolati su presentazione ACTIVIN e comprendeva geni che sono coinvolti in percorsi di segnalazione non precedentemente associati con la pluripotenza o differenziazione, come la segnalazione di insulina e la risposta cAMP (Figura 4A, scatola grigia e Figura 4 dati fonte 3). Questi risultati suggeriscono che le cellule attivano transitoriamente la differenziazione in risposta all’ACTIVIN.
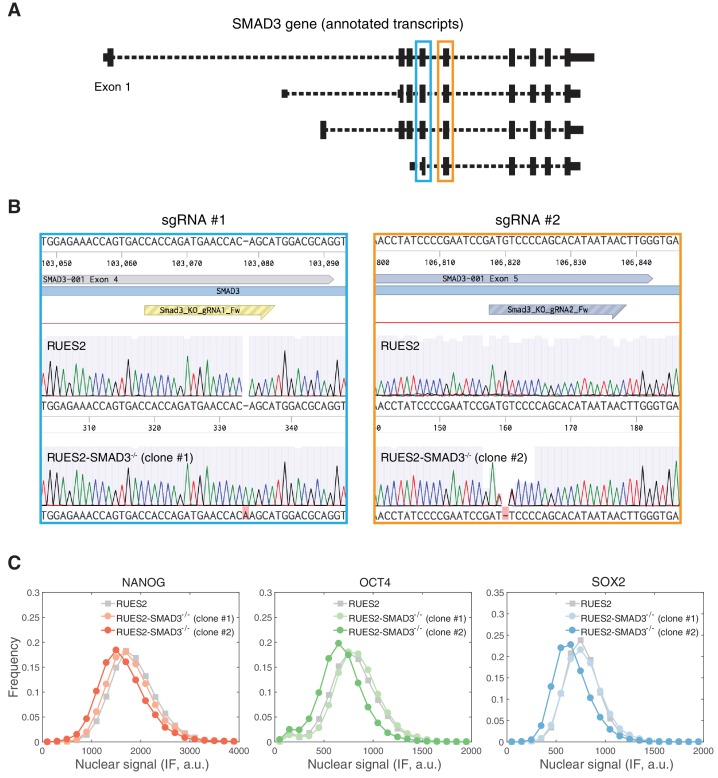
Figura 4-figure supplement 2.Figura 4—supplemento figura 2. ACTIVIN/SMAD2 risultati di segnalazione in risposte trascrizionali stabili e transitorie.clustering gerarchico gruppo uno geni (z-score).clustering gerarchico gruppo due geni (z-score).clustering gerarchico gruppo tre geni (z-score).analisi dell’arricchimento del motivo per i gruppi di geni 1-3.analisi dell’arricchimento del gene per i gruppi di geni 1-3.analisi dell’arricchimento del gene per i gruppi di geni 1-3.Generi sovrapposti ottenuti dall’analisi dell’arricchimento del gene.Gruppo di clustering gerarchico gruppo uno geni (z-score).clustering gerarchico gruppo due geni (z-score).clustering gerarchico gruppo tre geni (z-score).analisi dell’arricchimento del motivo per i gruppi di geni 1-3.Analisi dell’arricchimento del gene per i gruppi di geni 1-3.Analisi dell’arricchimento del gene per i gruppi di geni 1-3.Generi sovrapposti ottenuti dall’analisi dell’arricchimento del gene.analisi del motivo e dell’arricchimento del gene per i gruppi di geni a risposta attiva.RUES2-SMAD3-/- generazione di linee.(A) clustering gerarchico di RNA-seq dati del corso del tempo per i geni che mostrano un fold-change >2 in risposta a ACTIVIN (10 ng / ml) rispetto al livello di pre-stimolo (T = 0 ore). Il gene z-score in ogni punto temporale è stato calcolato sottraendo la media e dividendo per la deviazione standard dei conteggi normalizzati di lettura in tutti i punti temporali. (B-C) RT-PCR analisi RT-PCR di (B) pluripotenza- e (C) geni associati mesendoderm dopo la presentazione di ACTIVIN (10 ng/mL) nella linea parentale RUES2 (barre nere) e le linee RUES2-SMAD3-/- (barre grigie e bianche). È stato raccolto un ulteriore campione che è stato lasciato non trattato per il corso di 12 ore (untr). L’espressione in ogni campione è stata normalizzata a GAPDH e poi al livello di pre-stimolo (T = 0 hr). I dati rappresentano la media ±S.D. per n = 3 repliche tecniche. Risultati simili sono stati ottenuti in tre esperimenti indipendenti utilizzando la linea parentale RUES2.10.7554/eLife.38279.015Cifra dati a 4 fonti 1.Gruppo gerarchico di clustering di un gene (z-score).10.7554/eLife.38279.016Cifra 4—dati fonte 2.Gruppo di clustering gerarchico due geni (z-score).10.7554/eLife.38279.017Cifra dati a 4 fonti 3.Gruppo gerarchico di raggruppamento tre geni (z-score).10.7554/eLife.38279.018Cifra 4—dati fonte 4.Motif analisi di arricchimento dei gruppi di geni 1-3.10.7554/eLife.38279.019Cifra 4—dati fonte 5.Analisi dell’arricchimento genico per i gruppi di geni 1-3.10.7554/eLife.38279.020Cifra 4—dati fonte 6.Generi sovrapposti ottenuti dall’analisi dell’arricchimento genico.(A) L’analisi dell’arricchimento del motivo dei gruppi di geni ottenuti dal raggruppamento gerarchico mostrato in Figura 4A. Le linee continue rappresentano tutti i motivi analizzati e ordinati in base al valore p del loro arricchimento corretto per test multipli. I simboli indicano i motivi selezionati che sono associati con la striscia primitiva e la differenziazione mesendodermica: corretto p-valore<10-1 (simboli pieni) e corretto p-valore>10-1 (simboli vuoti). (B) Confronto dei geni in ciascuno dei gruppi identificati nella figura 4A a geni specificamente arricchiti in endoderma isolato, mesoderma, ed ectoderma / tessuto epiblasto da embrioni di topo E7,5. La dimensione dei cerchi indica il -log10 (p-valore) dell’arricchimento e la scala di colore indica lo z-score del rapporto log2 (osservato / atteso) in ogni gruppo.(A) Strategia ad eliminazione diretta SMAD3 mediata da CRISPR/Cas9. Due diversi sgRNA sono stati progettati per colpire gli esoni codificati (barre nere) condivisi da tutte le isoforme SMAD3 (browser del genoma umano UCSC, http://genome.ucsc.edu/). lo sgRNA #1 colpisce l’esone 4 (scatola blu) e lo sgRNA #2 colpisce l’esone 5 (scatola arancione). (B) Sequenziamento dei cromatogrammi della linea parentale RUES2 e dei cloni RUES2-SMAD3-/- generati da sgRNA #1 e sgRNA #2. Si prevede che il clone #1 abbia un’inserzione omozigote +1, che si traduce in un codone STOP prematuro nell’esone 5, e che il clone #2 abbia una delezione omozigote -1, che si traduce in un codone STOP prematuro nell’esone 6. Le sequenze sono state allineate utilizzando il Benchling (https://benchling.com). (C) RUES2-SMAD3-/- le cellule mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per NANOG, OCT4, e l’espressione SOX2. Gli istogrammi mostrano il segnale nucleare quantificato in singole cellule (n > 10.000 cellule per linea).
L’esame della gerarchia di segnalazione coinvolta nell’auto-organizzazione dei gastruloidi ha rivelato la presenza di loop di feedback a tutti e tre i livelli. ACTIVIN ha indotto l’espressione dei propri ligandi e inibitori, così come quelli del percorso BMP e WNT (Figura 4 dati fonte 1-2). Tuttavia, nonostante l’induzione dell’espressione dei ligandi e degli inibitori, la soglia complessiva di segnalazione non era sufficiente per indurre e mantenere i destini mesendodermici sia del percorso BMP che del percorso WNT.
Abbiamo adottato due approcci aggiuntivi per valutare le risposte trascrizionali transitorie e stabili dopo il trattamento con ACTIVIN. In primo luogo, abbiamo eseguito l’analisi dell’arricchimento dei motivi sui nostri gruppi genici. Abbiamo selezionato 10 fattori di trascrizione che regolano la striscia primitiva e la differenziazione mesendodermica: MIXL1, LEF1, BRACHYURY (BRA), GATA6, FOXH1, FOXA1, FOXA2, GOOSECOID (GSC), SOX17, e EOMES, e abbiamo chiesto se i loro siti di legame sono arricchiti nella regione promotrice dei geni appartenenti a ciascuno dei gruppi dinamici. A sostegno della nostra ipotesi, i motivi sono stati arricchiti in modo significativo solo nei geni espressi in modo transitorio del gruppo uno e non all’interno del gruppo 2 o 3 (Figura 4-figure supplement 1A e Figura 4-source data 4). Questo suggerisce che la rete di regolazione genica attivata durante il picco della segnalazione SMAD2 è associata alla differenziazione mesendodermica. Abbiamo inoltre confrontato i nostri gruppi genici con i geni specifici del tessuto identificati in endoderma isolato, mesoderma ed ectoderma/epiblasto da embrioni di topo E7.5 (Lu et al., 2018). Anche se tutti i gruppi contenevano un arricchimento significativo dei geni da uno o più strati di germi di topo, il gruppo uno ha mostrato l’arricchimento più significativo dei geni endodermici (Figura 4-figure supplement 1B e Figura 4-source data 5). Nel complesso i nostri dati dimostrano che durante il picco di accumulazione nucleare SMAD2, gli hESC sono in viaggio per la differenziazione. Tuttavia, il programma di differenziazione mesendodermica non viene mantenuto e le cellule tornano alla pluripotenza.

Figura 4-figure supplement 2.Figura 4—supplemento alla figura 2. La segnalazione ACTIVIN/SMAD2 risulta in risposte trascrizionali stabili e transitorie.clustering gerarchico gruppo uno geni (z-score).clustering gerarchico gruppo due geni (z-score).clustering gerarchico gruppo tre geni (z-score).analisi dell’arricchimento del motivo per i gruppi di geni 1-3.analisi dell’arricchimento del gene per i gruppi di geni 1-3.analisi dell’arricchimento del gene per i gruppi di geni 1-3.Generi sovrapposti ottenuti dall’analisi dell’arricchimento del gene.Gruppo di clustering gerarchico gruppo uno geni (z-score).clustering gerarchico gruppo due geni (z-score).clustering gerarchico gruppo tre geni (z-score).analisi dell’arricchimento del motivo per i gruppi di geni 1-3.Analisi dell’arricchimento del gene per i gruppi di geni 1-3.Analisi dell’arricchimento del gene per i gruppi di geni 1-3.Generi sovrapposti ottenuti dall’analisi dell’arricchimento del gene.analisi del motivo e dell’arricchimento del gene per i gruppi di geni a risposta attiva.RUES2-SMAD3-/- generazione di linee.(A) Aggregazione gerarchica di RNA-seq dati del corso del tempo per i geni che mostrano un cambiamento di piega >2 in risposta ad ACTIVIN (10 ng/mL) rispetto al livello di pre-stimolo (T = 0 hr). Il gene z-score in ogni punto temporale è stato calcolato sottraendo la media e dividendo per la deviazione standard dei conteggi normalizzati di lettura in tutti i punti temporali. (B-C) RT-PCR analisi RT-PCR di (B) pluripotenza- e (C) geni associati mesendoderm dopo la presentazione di ACTIVIN (10 ng/mL) nella linea parentale RUES2 (barre nere) e le linee RUES2-SMAD3-/- (barre grigie e bianche). È stato raccolto un ulteriore campione che è stato lasciato non trattato per il corso di 12 ore (untr). L’espressione in ogni campione è stata normalizzata a GAPDH e poi al livello di pre-stimolo (T = 0 hr). I dati rappresentano la media ±S.D. per n = 3 repliche tecniche. Risultati simili sono stati ottenuti in tre esperimenti indipendenti utilizzando la linea parentale RUES2.10.7554/eLife.38279.015Figure 4-source data 1.Hierarchical clustering group one genes (z-score).10.7554/eLife.38279.016Cifra dati a 4 fonti 2.Gruppo gerarchico di raggruppamento due geni (z-score).10.7554/eLife.38279.017Cifra 4—dati fonte 3.Gruppo di clustering gerarchico tre geni (z-score).10.7554/eLife.38279.018Cifra 4—dati fonte 4.Motif analisi di arricchimento dei gruppi di geni 1-3.10.7554/eLife.38279.019Cifra 4—dati fonte 5.Analisi dell’arricchimento genico per i gruppi di geni 1-3.10.7554/eLife.38279.020Cifra 4—dati fonte 6.Generi sovrapposti ottenuti dall’analisi dell’arricchimento genico.(A) L’analisi dell’arricchimento del motivo dei gruppi di geni ottenuti dal raggruppamento gerarchico mostrato in Figura 4A. Le linee continue rappresentano tutti i motivi analizzati e ordinati in base al valore p del loro arricchimento corretto per test multipli. I simboli indicano i motivi selezionati che sono associati con la striscia primitiva e la differenziazione mesendodermica: corretto p-valore<10-1 (simboli pieni) e corretto p-valore>10-1 (simboli vuoti). (B) Confronto dei geni in ciascuno dei gruppi identificati nella figura 4A a geni specificamente arricchiti in endoderma isolato, mesoderma, ed ectoderma / tessuto epiblasto da embrioni di topo E7,5. La dimensione dei cerchi indica il -log10 (p-valore) dell’arricchimento e la scala di colore indica lo z-score del rapporto log2 (osservato / atteso) in ogni gruppo.(A) CRISPR/Cas9-mediata SMAD3 strategia knockout. Due diversi sgRNA sono stati progettati per colpire gli esoni di codifica (barre nere) condivisi da tutte le isoforme SMAD3 (browser del genoma umano UCSC, http://genome.ucsc.edu/). sgRNA #1 bersagli esone 4 (scatola blu) e sgRNA #2 bersagli esone 5 (scatola arancione). (B) Sequenziamento dei cromatogrammi della linea parentale RUES2 e dei cloni RUES2-SMAD3-/- generati da sgRNA #1 e sgRNA #2. Si prevede che il clone #1 abbia un’inserzione omozigote +1, che si traduce in un codone STOP prematuro nell’esone 5, e che il clone #2 abbia una delezione omozigote -1, che si traduce in un codone STOP prematuro nell’esone 6. Le sequenze sono state allineate utilizzando il Benchling (https://benchling.com). (C) RUES2-SMAD3-/- le cellule mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per NANOG, OCT4, e l’espressione SOX2. Gli istogrammi mostrano il segnale nucleare quantificato in singole cellule (n > 10.000 cellule per linea).
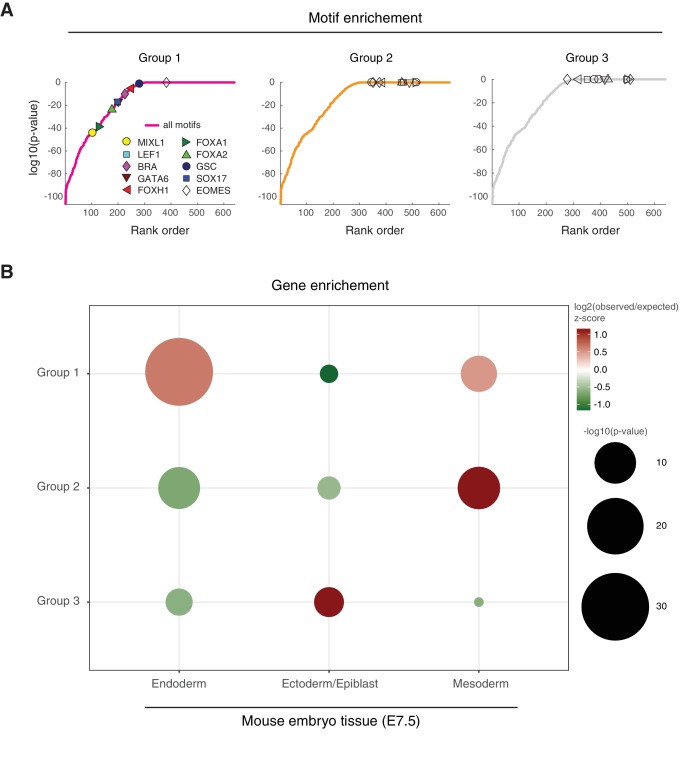
Figura 4-figure supplement 1.Analisi del motivo e dell’arricchimento genetico per gruppi di geni che rispondono alle ACTIVIN.(A) L’analisi dell’arricchimento dei gruppi di geni ottenuti dal clustering gerarchico mostrato in Figura 4A. Le linee continue rappresentano tutti i motivi analizzati e ordinati in base al valore p del loro arricchimento corretto per test multipli. I simboli indicano i motivi selezionati che sono associati con la striscia primitiva e la differenziazione mesendodermica: corretto p-valore<10-1 (simboli pieni) e corretto p-valore>10-1 (simboli vuoti). (B) Confronto dei geni in ciascuno dei gruppi identificati nella figura 4A a geni specificamente arricchiti in endoderma isolato, mesoderma, ed ectoderma / tessuto epiblasto da embrioni di topo E7,5. La dimensione dei cerchi indica il -log10 (p-valore) dell’arricchimento e la scala di colore indica lo z-score del rapporto log2 (osservato / atteso) in ogni gruppo.

Figura 4-figure supplement 2.RUES2-SMAD3-/- generazione di linee.(A) Strategia ad eliminazione diretta SMAD3 mediata da CRISPR/Cas9. Due diversi sgRNA sono stati progettati per colpire gli esoni di codifica (barre nere) condivisi da tutte le isoforme SMAD3 (browser del genoma umano UCSC, http://genome.ucsc.edu/). lo sgRNA #1 colpisce l’esone 4 (scatola blu) e lo sgRNA #2 colpisce l’esone 5 (scatola arancione). (B) Sequenziamento dei cromatogrammi della linea parentale RUES2 e dei cloni RUES2-SMAD3-/- generati da sgRNA #1 e sgRNA #2. Si prevede che il clone #1 abbia un’inserzione omozigote +1, che si traduce in un codone STOP prematuro nell’esone 5, e che il clone #2 abbia una delezione omozigote -1, che si traduce in un codone STOP prematuro nell’esone 6. Le sequenze sono state allineate utilizzando il Benchling (https://benchling.com). (C) RUES2-SMAD3-/- le cellule mantengono l’espressione dei marcatori di pluripotenza in condizioni di crescita standard. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per NANOG, OCT4, e l’espressione SOX2. Gli istogrammi mostrano il segnale nucleare quantificato in singole cellule (n > 10.000 cellule per linea).
SMAD3 è dispensabile per la risposta delle classi di geni adattativi e stabili ad ACTIVIN
Nell’embrione di topo, SMAD3 è dispensabile per lo sviluppo precoce, come dimostrato dal fatto che i topi SMAD3 raggiungono l’età adulta (Zhu et al., 1998; Yang et al., 1999; Datto et al., 1999). In assenza di SMAD2 nell’epiblasto, SMAD3 può mediare una certa induzione mesodermica durante la gastrulazione. Tuttavia, un numero maggiore di lignaggi mesendodermici anteriori sono completamente eliminati e gli embrioni falliscono alla gastrulazione, suggerendo un ruolo critico per SMAD2 in questo processo (Vincent et al., 2003; Dunn et al., 2004). Al fine di decifrare se la risposta SMAD2 transitoria è sufficiente a guidare il programma trascrizionale a valle della presentazione ACTIVIN in hESC, abbiamo generato due linee di knockout indipendenti RUES2 SMAD3 (RUES2-SMAD3-/-) utilizzando l’editing del genoma mediato da CRISPR-Cas9 (Figura 4-figure supplement 2A-B). Queste linee hanno mantenuto l’espressione dei marcatori di pluripotenza NANOG, OCT4 e SOX2, il che è coerente con la precedente scoperta che SMAD2, ma non SMAD3, regola l’espressione NANOG per promuovere la pluripotenza nelle cellule staminali di epiblasto di topo (Figura 4-figure supplement 1C) (Sakaki-Yumoto et al., 2013). In risposta ad ACTIVIN, le cellule RUES2-SMAD3-/- hanno mostrato una risposta trascrizionale identica alla linea parentale RUES2 per entrambi i geni target ACTIVIN associati a pluripotenza e mesendoderm (Figura 4B-C). Anche se i nostri risultati non possono escludere una possibile ridondanza tra SMAD2 e SMAD3, concludiamo che SMAD3 non è necessario per il mantenimento della pluripotenza o la dinamica di risposta trascrizionale dopo la presentazione ACTIVIN.
La linea di base SMAD2 elevata a lungo termine mantiene la pluripotenza
Abbiamo dimostrato che i livelli nucleari SMAD2 sono transitori a seguito di un ingresso a gradino di ACTIVIN, con una linea di base a lungo termine che rimane elevata rispetto al livello di pre-stimolo. Abbiamo poi chiesto se l’aumento della baseline SMAD2 dopo il picco di risposta è necessario per il mantenimento della pluripotenza. Al fine di affrontare questa domanda, RUES2-mCit-SMAD2 cellule sono state trattate con SB al fine di inibire la segnalazione di ACTIVIN, 8 ore dopo la stimolazione, e analizzati per la loro capacità di mantenere la pluripotenza. Il trattamento SB ha portato ad una diminuzione dei livelli di SMAD2 nucleare-citoplasmatici di nuovo alla linea di base non stimolata (Figura 5A). Come osservato in precedenza, la presentazione di SB ha portato ad una perdita di pluripotenza, come indicato dalla perdita di espressione NANOG in RUES2-mCit-SMAD2 (Figura 5B) (James et al., 2005; Vallier et al., 2005; Xu et al., 2008). L’analisi della linea parentale RUES2 ha confermato la perdita di pluripotenza nelle stesse condizioni sperimentali (Figura 5-figure supplement 1A). Concludiamo che la linea di base elevata alla coda della risposta SMAD2 è ligando dipendente e responsabile del mantenimento del programma di pluripotenza a lungo termine.
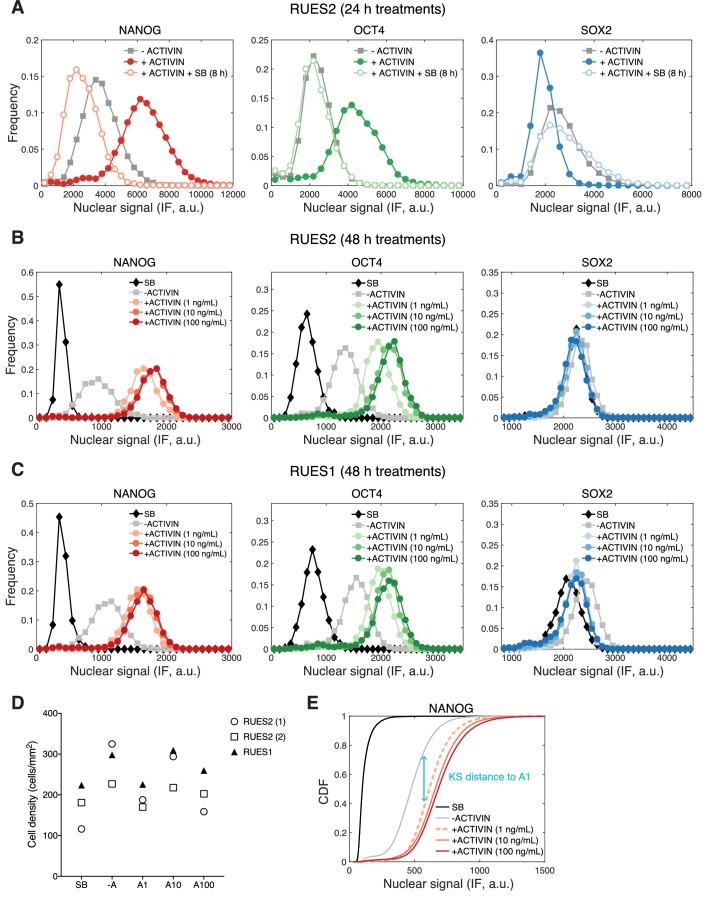
Figura 5-figure supplemento 1.Figura 5—figura 1. A lungo termine, la linea di base SMAD2 elevata mantiene pluripotenza.a lungo termine, la linea di base SMAD2 elevata mantiene pluripotenza.(A) Quantificazione del rapporto mCitrine-SMAD2 nucleare-citrino-SMAD2 dopo il trattamento con ACTIVIN (10 ng/mL aggiunti a T = 0 ore, linea verde solida). In seguito alla risposta transitoria SMAD2, SB (10 µM) è stato aggiunto a uno dei campioni (linea verde punteggiata, aggiunta a T = 8 ore). Un terzo campione è stato lasciato non trattato in E7 (-ACTIVIN) per tutta la durata dell’esperimento (linea nera solida). Le immagini sono state acquisite ogni 10 min. Le linee rappresentano la risposta media in ogni punto temporale (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (B) I campioni in A sono stati fissati 24 ore dopo l’aggiunta di ACTIVIN e analizzati per l’espressione NANOG da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Barra di scala, 50 µM. (C-D) Le cellule RUES2 sono state coltivate in E7 con diversi livelli di Activin (0, 1, 10, 100 ng/mL) o SB (10 µM) per 2 giorni. Le cellule sono state fissate e analizzate per immunofluorescenza (IF). (C) Immagini: NANOG (rosso), OCT4 (verde), SOX2 (ciano), DAPI (grigio). Barra di scala, 50 µM. (D) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). Risultati simili a quelli mostrati in C e D sono stati ottenuti in due esperimenti indipendenti in RUES2 e in un ulteriore linea hESC, RUES1 (vedi Figura 5-figure supplement 1B-C). (E) Kolmogorov-Smirnov (KS) distanza della distribuzione di probabilità cumulativa (CDF) di ogni marcatore alla CDF di riferimento (1 ng/mL condizione ACTIVIN) per esperimenti indipendenti in RUES2 e in RUES1. n.s., non significativo, **p<0.01, ***p<0.001, ****p<0.0001, ANOVA.(A) Analisi della linea RUES2 parentale nelle stesse condizioni sperimentali mostrate in Figura 5A. Le cellule RUES2 sono state coltivate in E7 e trattate con ACTIVIN (10 ng/mL). 8 ore dopo l’aggiunta di ACTIVIN, SB (10 µM) è stato aggiunto ad uno dei campioni. Un terzo campione è stato lasciato non trattato in E7 (-Attivina) per tutta la durata dell’esperimento. I campioni sono stati fissati a 24 ore dopo ACTIVIN e analizzati con immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B-C) Ripetizioni indipendenti dell’esperimento mostrato in Figura 5C-D effettuata in (B) RUES2 e (C) RUES1. Le cellule sono state coltivate in E7 con diversi livelli di ACTIVIN (0, 1, 10, 100 ng/mL) o SB (10 µM) per 2 giorni. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (D) Quantificazione della densità cellulare finale negli esperimenti mostrati in Figura 5E. Non vi è alcuna differenza significativa tra le densità sotto trattamento SB o diversi livelli di ACTIVIN (ANOVA). (E) Schema che mostra la distanza Kolmogorov-Smirnov (KS) tra la distribuzione di probabilità cumulativa (CDF) dei livelli di NANOG misurati sotto la condizione -AtTIVINA e la condizione di riferimento 1 ng/mL ACTIVINA (A1). I CDF sono calcolati dagli istogrammi di intensità di fluorescenza, come quelli mostrati in B e C, e la metrica della distanza KS viene confrontata tra i replicati biologici.
Il fatto che la linea di base SMAD2 post-stimolazione è la stessa indipendentemente dalla concentrazione di ACTIVIN superiore a 0,5 ng/mL, suggerisce che la pluripotenza è insensibile ai livelli di ligandi graduati al di sopra di questa soglia (Figura 3E). Per testare questa ipotesi, abbiamo trattato singole cellule con diversi livelli di ATTIVINA e confrontato l’espressione di NANOG, OCT4, e SOX2 dopo 2 giorni di stimolazione. L’espressione di tutti e tre i marcatori era simile a tre diverse concentrazioni di ACTIVIN aggiunto (1, 10, e 100 ng / ml) e l’espressione di NANOG e OCT4 è stata elevata rispetto alla condizione -ACTIVIN (Figura 5C-D e Figura 5-figure supplemento 1B; risultati da ulteriori RUES1 linea hESC mostrato in Figura 5-figure supplemento 1C). Per verificare l’assenza di input ACTIVIN/SMAD2 abbiamo presentato SB per lo stesso periodo di tempo. La pluripotenza non è stata mantenuta in queste condizioni, come dimostrato dal down-regolamentazione di NANOG e OCT4 al di sotto della condizione -ACTIVIN (Figura 5C-D e Figura 5-figure supplement 1B-C). La tossicità come risultato del trattamento SB o l’aumento dei livelli di ACTIVIN non è stata osservata (Figura 5-figure supplement 1D). Ciò conferma l’insensibilità dello stato pluripotente all’ACTIVIN classificato. Anche alle più alte concentrazioni di ligandi pluripotenza è stata mantenuta e non è stata osservata alcuna differenziazione.
Al fine di confrontare i dati di immunofluorescenza tra i replicati biologici, che possono differire in valori di intensità assoluta, abbiamo calcolato la distanza Kolmogorov-Smirnov (KS) tra la funzione di distribuzione cumulativa di ciascun marcatore (CDF) misurata in condizioni diverse a un CDF di riferimento, in questo caso il CDF misurato sotto la condizione 1 ng/mL ACTIVIN (Figura 5-figure supplement 1E). Nei replicati biologici che utilizzano RUES2 o RUES1 le distribuzioni dei livelli NANOG e OCT nelle condizioni 100 o 10 ng/mL ACTIVIN sono riproducibili vicino alle distribuzioni misurate nella condizione 1 ng/mL ACTIVIN (piccola distanza KS, Figura 5E). Mentre le distribuzioni misurate nelle condizioni -ATTIVINA e SB sono riproducibilmente lontane dalle distribuzioni misurate nella condizione 1 ng/mL di ATTIVINA (grande distanza KS, Figura 5E). Questa analisi dimostra la riproducibilità dei nostri risultati che lo stato pluripotente è insensibile ai livelli di ACTIVINA graduata.

Figura 5-figure supplement 1.A lungo termine, la linea di base SMAD2 elevata mantiene la pluripotenza.a lungo termine, la linea di base SMAD2 elevata mantiene la pluripotenza.(A) Quantificazione del rapporto mCitrine-SMAD2 nucleare/citoplasmatico dopo il trattamento con ACTIVIN (10 ng/mL aggiunti a T = 0 ore, linea verde solida). In seguito alla risposta transitoria SMAD2, SB (10 µM) è stato aggiunto a uno dei campioni (linea verde punteggiata, aggiunta a T = 8 ore). Un terzo campione è stato lasciato non trattato in E7 (-ACTIVIN) per tutta la durata dell’esperimento (linea nera solida). Le immagini sono state acquisite ogni 10 min. Le linee rappresentano la risposta media in ogni punto temporale (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (B) I campioni in A sono stati fissati 24 ore dopo l’aggiunta di ACTIVIN e analizzati per l’espressione NANOG da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Barra di scala, 50 µM. (C-D) Le cellule RUES2 sono state coltivate in E7 con diversi livelli di Activin (0, 1, 10, 100 ng/mL) o SB (10 µM) per 2 giorni. Le cellule sono state fissate e analizzate per immunofluorescenza (IF). (C) Immagini: NANOG (rosso), OCT4 (verde), SOX2 (ciano), DAPI (grigio). Barra di scala, 50 µM. (D) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). Risultati simili a quelli mostrati in C e D sono stati ottenuti in due esperimenti indipendenti in RUES2 e in un ulteriore linea hESC, RUES1 (vedi Figura 5-figure supplement 1B-C). (E) Kolmogorov-Smirnov (KS) distanza della distribuzione di probabilità cumulativa (CDF) di ogni marcatore alla CDF di riferimento (1 ng/mL condizione ACTIVIN) per esperimenti indipendenti in RUES2 e in RUES1. n.s., non significativo, **p<0.01, ***p<0.001, ****p<0.0001, ANOVA.(A) Analisi della linea parentale RUES2 nelle stesse condizioni sperimentali mostrate in Figura 5A. Le cellule RUES2 sono state coltivate in E7 e trattate con ACTIVIN (10 ng/mL). 8 ore dopo l’aggiunta di ACTIVIN, SB (10 µM) è stato aggiunto ad uno dei campioni. Un terzo campione è stato lasciato non trattato in E7 (-Attivina) per tutta la durata dell’esperimento. I campioni sono stati fissati a 24 ore dopo ACTIVIN e analizzati con immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B-C) Ripetizioni indipendenti dell’esperimento mostrato in Figura 5C-D effettuata in (B) RUES2 e (C) RUES1. Le cellule sono state coltivate in E7 con diversi livelli di ACTIVIN (0, 1, 10, 100 ng/mL) o SB (10 µM) per 2 giorni. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (D) Quantificazione della densità cellulare finale negli esperimenti mostrati in Figura 5E. Non vi è alcuna differenza significativa tra le densità sotto trattamento SB o diversi livelli di ACTIVIN (ANOVA). (E) Schema che mostra la distanza Kolmogorov-Smirnov (KS) tra la distribuzione di probabilità cumulativa (CDF) dei livelli di NANOG misurati sotto la condizione -AtTIVINA e la condizione di riferimento 1 ng/mL ACTIVINA (A1). I CDF sono calcolati dagli istogrammi di intensità di fluorescenza, come quelli mostrati in B e C, e la metrica della distanza KS viene confrontata tra i replicati biologici.

Figura 5-figure supplement 1.La linea di base SMAD2 a lungo termine, elevata, mantiene una pluripotenza.(A) Analisi della linea parentale RUES2 nelle stesse condizioni sperimentali mostrate nella Figura 5A. Le cellule RUES2 sono state coltivate in E7 e trattate con ACTIVIN (10 ng/mL). 8 ore dopo l’aggiunta di ACTIVIN, SB (10 µM) è stato aggiunto ad uno dei campioni. Un terzo campione è stato lasciato non trattato in E7 (-Attivina) per tutta la durata dell’esperimento. I campioni sono stati fissati a 24 ore dopo ACTIVIN e analizzati con immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B-C) Ripetizioni indipendenti dell’esperimento mostrato in Figura 5C-D effettuata in (B) RUES2 e (C) RUES1. Le cellule sono state coltivate in E7 con diversi livelli di ACTIVIN (0, 1, 10, 100 ng/mL) o SB (10 µM) per 2 giorni. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (D) Quantificazione della densità cellulare finale negli esperimenti mostrati in Figura 5E. Non vi è alcuna differenza significativa tra le densità sotto trattamento SB o diversi livelli di ACTIVIN (ANOVA). (E) Schema che mostra la distanza Kolmogorov-Smirnov (KS) tra la distribuzione di probabilità cumulativa (CDF) dei livelli di NANOG misurati sotto la condizione -AtTIVINA e la condizione di riferimento 1 ng/mL ACTIVINA (A1). I CDF sono calcolati dagli istogrammi di intensità di fluorescenza, come quelli mostrati in B e C, e la metrica della distanza KS viene confrontata tra i replicati biologici.
Il priming WNT rivela la differenziazione del mesendoderma dipendente da ACTIVIN
Abbiamo dimostrato che l’ACTIVIN da solo non può guidare la differenziazione stabile dei gastruloidi umani. Tuttavia, WNT può portare alla differenziazione e all‘auto-organizzazione dei gastruloidi in modo dipendente da SMAD2/3 (Martyn et al., 2018). Poiché WNT sta operando a monte di ACTIVIN/NODAL nella gerarchia di segnalazione proposta, abbiamo chiesto se le cellule con una storia di segnalazione WNT potrebbero rispondere in modo diverso al trattamento con ACTIVIN. Per affrontare questo, le cellule sono state trattate con WNT per 24 ore, lavate per rimuovere WNT, e poi coltivate con o senza ACTIVIN per un ulteriore 24 ore (Figura 6A). Sorprendentemente, quando le cellule sono state trattate con WNT solo in assenza di ACTIVINA nessuna differenziazione mesendodermica è stata osservata e le cellule sono rimaste pluripotenti (Figura 6B-C). Tuttavia, se le cellule sono state esposte a WNT prima di destini mesendodermici stimolazione ACTIVIN erano robustamente indotti (Figura 6D-E). KS analisi della distanza utilizzando la condizione di pluripotenza (-/ACT) come condizione di riferimento ha dimostrato la riproducibilità di queste osservazioni in RUES2 e RUES1 (Figura 6F-G). Dopo il trattamento WNT e ACTIVIN, l’espressione del marcatore mesendoderm è stata osservata anche nella riga RUES2-SMAD3-/-, suggerendo che SMAD3 non è necessario per l’induzione del mesendoderm dipendente da ACTIVIN (Figura 6-figure supplement 1A). La differenziazione era dipendente dalla concentrazione di ACTIVIN, come dimostrato dall’induzione di BRA a bassa ACTIVIN e l’induzione di BRA, EOMES e GSC ad alte concentrazioni di ACTIVIN (Figura 6-figure supplement 1B-D). Pertanto, dopo il priming WNT ACTIVIN funziona come morfogeno per modellare i destini mesendodermici e SMAD3 è di nuovo dispensabile per questo processo.
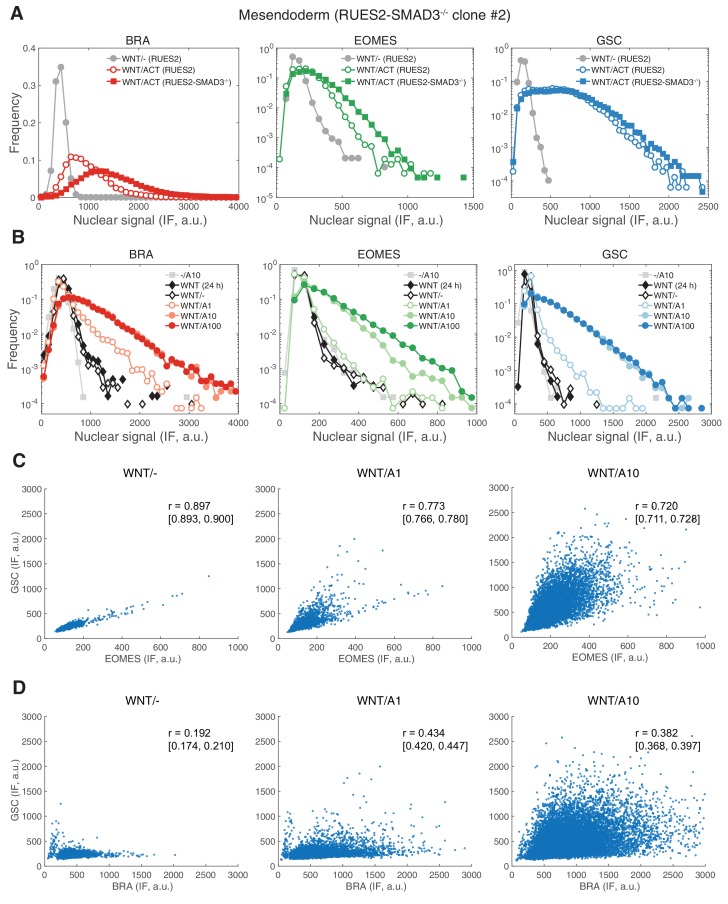
Figura 6-figure supplemento 1.WNT priming rivela la differenziazione mesendoderm dipendente da ACTIVIN.ACTIVIN differenziazione mesendoderm dose-dipendente.(A) Schema che delinea il protocollo sperimentale di 2 giorni. (B-C) Le cellule sono state coltivate per un giorno con o senza WNT3A (100 ng/mL, barra superiore). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con o senza ACTIVIN (10 ng/mL, barra inferiore). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). (B) Immagini: NANOG (rosso), OCT4 (verde), SOX2 (ciano), DAPI (grigio). Barra di scala, 50 µM. (C) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). (D-E) Le cellule sono state coltivate per un giorno con o senza WNT3A (100 ng/mL, barra superiore). Il secondo giorno sono state lavate per rimuovere il WNT e trattate con o senza ACTIVIN (10 ng/mL, barra inferiore). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). (D) Immagini: BRA (rosso), EOMES (verde), GSC (ciano), DAPI (grigio). Barra di scala, 50 µM. (E) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). (F-G) Kolmogorov-Smirnov (KS) distanza della distribuzione di probabilità cumulativa (CDF) di ogni marker al CDF di riferimento (-/ACT) per esperimenti indipendenti in RUES2 e in RUES1 per i set di marker (F) pluripotenza e (G) mesendoderm. n.s. (o confronto non mostrato), non significativo, *p<0.05, **p<0.01, ***p<0.001, ANOVA.(A) Le cellule RUES2 e RUES2-SMAD3-/- sono state coltivate per un giorno con l’adescamento WNT. Il secondo giorno sono stati lavati per rimuovere WNT e trattati con o senza ACTIVIN (10 ng/mL) per un ulteriore giorno. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per BRA, EOMES, e l’espressione GSC. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B) Istogrammi che mostrano i dati di immunofluorescenza da campioni trattati con diverse dosi di ACTIVIN (1, 10, 100 ng/mL) per 12 ore dopo il priming WNT. I dati per WNT (24 ore) mostrano che tutti i marcatori sono stati scarsamente espressi prima dell’aggiunta di ACTIVIN e che le loro distribuzioni sono identiche alle cellule in condizioni di pluripotenza (-/ACT10, 36 ore) o cellule che sono state lasciate non trattate con ACTIVIN per le 12 ore aggiuntive (WNT/-). Quantificazione per n > 5.000 cellule per condizione. (C) Trama di dispersione del segnale nucleare monocellulare EOMES e GSC dai dati mostrati in B. EOMES e l’espressione GSC aumenta in condizione WNT/ACT10 e l’espressione è positivamente correlata. (D) Il diagramma di dispersione del segnale nucleare monocellulare BRA e GSC dai dati mostrati in B. L’espressione non è ben correlata. Un numero simile di celle è stato analizzato in tutte le condizioni in C e D, ma i punti di dati si raggruppano uno sopra l’altro nelle condizioni WNT/- e WNT/ACT1. r, coefficiente di correlazione Pearson con l’intervallo di confidenza del 95% indicato tra parentesi.

Figura 6-figure supplemento 1.WNT priming rivela la differenziazione mesendoderm dipendente da ACTIVIN.ACTIVIN differenziazione mesendoderm dose-dipendente.(A) Schema che delinea il protocollo sperimentale di 2 giorni. (B-C) Le cellule sono state coltivate per un giorno con o senza WNT3A (100 ng/mL, barra superiore). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con o senza ACTIVIN (10 ng/mL, barra inferiore). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). (B) Immagini: NANOG (rosso), OCT4 (verde), SOX2 (ciano), DAPI (grigio). Barra di scala, 50 µM. (C) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). (D-E) Le cellule sono state coltivate per un giorno con o senza WNT3A (100 ng/mL, barra superiore). Il secondo giorno sono state lavate per rimuovere il WNT e trattate con o senza ACTIVIN (10 ng/mL, barra inferiore). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). (D) Immagini: BRA (rosso), EOMES (verde), GSC (ciano), DAPI (grigio). Barra di scala, 50 µM. (E) Istogrammi che mostrano il segnale di IF nucleare quantificato in singole celle (n > 5.000 celle per condizione). (F-G) Kolmogorov-Smirnov (KS) distanza della distribuzione di probabilità cumulativa (CDF) di ogni marker al CDF di riferimento (-/ACT) per esperimenti indipendenti in RUES2 e in RUES1 per i set di marker (F) pluripotenza e (G) mesendoderm. n.s. (o confronto non mostrato), non significativo, *p<0.05, **p<0.01, ***p<0.001, ANOVA.(A) Le cellule RUES2 e RUES2-SMAD3-/- sono state coltivate per un giorno con l’adescamento WNT. Il secondo giorno sono stati lavati per rimuovere WNT e trattati con o senza ACTIVIN (10 ng/mL) per un ulteriore giorno. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per BRA, EOMES, e l’espressione GSC. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B) Istogrammi che mostrano i dati di immunofluorescenza da campioni trattati con diverse dosi di ACTIVIN (1, 10, 100 ng/mL) per 12 ore dopo il priming WNT. I dati per WNT (24 ore) mostrano che tutti i marcatori sono stati scarsamente espressi prima dell’aggiunta di ACTIVIN e che le loro distribuzioni sono identiche alle cellule in condizioni di pluripotenza (-/ACT10, 36 ore) o cellule che sono state lasciate non trattate con ACTIVIN per le 12 ore aggiuntive (WNT/-). Quantificazione per n > 5.000 cellule per condizione. (C) Trama di dispersione del segnale nucleare monocellulare EOMES e GSC dai dati mostrati in B. EOMES e l’espressione GSC aumenta in condizione WNT/ACT10 e l’espressione è positivamente correlata. (D) Il diagramma di dispersione del segnale nucleare monocellulare BRA e GSC dai dati mostrati in B. L’espressione non è ben correlata. Un numero simile di celle è stato analizzato in tutte le condizioni in C e D, ma i punti di dati si raggruppano uno sopra l’altro nelle condizioni WNT/- e WNT/ACT1. r, coefficiente di correlazione Pearson con l’intervallo di confidenza del 95% indicato tra parentesi.

Figura 6-figure supplement 1.ACTIVIN differenziazione mesendoderm dose-dipendente.(A) Le cellule RUES2 e RUES2-SMAD3-/- sono state coltivate per un giorno con il priming WNT. Il secondo giorno sono stati lavati per rimuovere WNT e trattati con o senza ACTIVIN (10 ng/mL) per un giorno in più. Le cellule sono stati fissati e analizzati da immunofluorescenza (IF) per BRA, EOMES, e l’espressione GSC. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). (B) Istogrammi che mostrano i dati di immunofluorescenza da campioni trattati con diverse dosi di ACTIVIN (1, 10, 100 ng/mL) per 12 ore dopo il priming WNT. I dati per WNT (24 ore) mostrano che tutti i marcatori sono stati scarsamente espressi prima dell’aggiunta di ACTIVIN e che le loro distribuzioni sono identiche alle cellule in condizioni di pluripotenza (-/ACT10, 36 ore) o cellule che sono state lasciate non trattate con ACTIVIN per le 12 ore aggiuntive (WNT/-). Quantificazione per n > 5.000 cellule per condizione. (C) Trama di dispersione del segnale nucleare monocellulare EOMES e GSC dai dati mostrati in B. EOMES e l’espressione GSC aumenta in condizione WNT/ACT10 e l’espressione è positivamente correlata. (D) Il diagramma di dispersione del segnale nucleare monocellulare BRA e GSC dai dati mostrati in B. L’espressione non è ben correlata. Un numero simile di celle è stato analizzato in tutte le condizioni in C e D, ma i punti di dati si raggruppano uno sopra l’altro nelle condizioni WNT/- e WNT/ACT1. r, coefficiente di correlazione Pearson con l’intervallo di confidenza del 95% indicato tra parentesi.
La memoria WNT stabilizza la risposta trascrizionale mesendodermica ad ACTIVIN
Al fine di affrontare il meccanismo del priming WNT, abbiamo chiesto per prima cosa se l’effettore trascrizionale della segnalazione canonica WNT, la β-catenina, è coinvolto in questo processo. Abbiamo quindi trattato le cellule con l’inibitore delle piccole molecole endo-IWR-1, che blocca la funzione β-catenina attraverso la stabilizzazione del suo complesso di distruzione. endo-IWR-1 è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. In presenza di endo-IWR-1 è stata eliminata l’espressione del mesendoderm maker e l’espressione del marcatore di pluripotenza è stata mantenuta, indicando un requisito per la β-catenina nel processo di differenziazione (Figura 7A e Figura 7-figure supplement 1A). Abbiamo poi chiesto se il WNT endogeno, indotto da un possibile ciclo di feedback positivo, è necessario per l’induzione stabile del mesendoderma, in particolare durante la fase di trattamento ACTIVIN quando il WNT esogeno è stato rimosso. Per affrontare questo abbiamo bloccato la secrezione di WNT endogeno utilizzando l’inibitore delle piccole molecole IWP-2, che è stato aggiunto con WNT il primo giorno e di nuovo con l’ACTIVIN il secondo giorno. Troviamo che l’aggiunta di IWP-2 non influisce sulla differenziazione mesendoderm dipendente dall’ACTIVIN e sulla perdita di pluripotenza (Figura 7A e Figura 7-figure supplement 1A). Tuttavia, IWP-2 alla stessa concentrazione blocca la differenziazione mesendoderm dipendente da WNT a valle di BMP4 (Figura 7-figure supplement 1B) (Martyn et al., 2018). La tossicità come risultato del trattamento endo-IWR-1 o IWP-2 non è stata osservata (Figura 7B). Insieme, questi risultati dimostrano la presenza di una memoria di segnalazione WNT inaspettata nelle cellule che è mediata tramite β-catenina ed è stabilita prima ed è necessaria per l’attività morfogena di ACTIVIN.
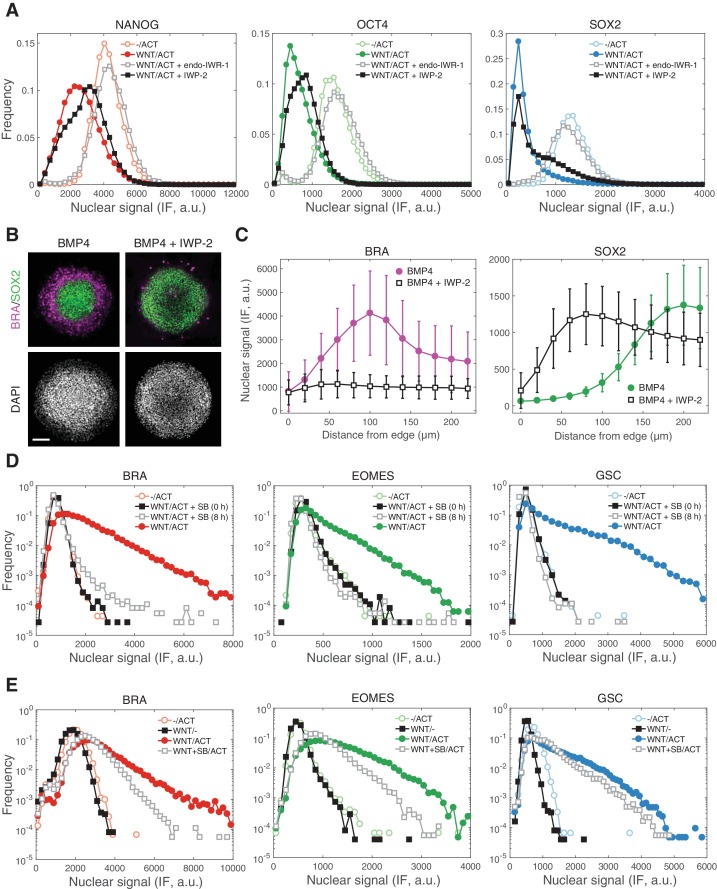
Figura 7-figure supplemento 1.Il priming WNT stabilizza la trascrizione del gene mesendoderm senza modificare le dinamiche di risposta SMAD2 e SMAD4. La segnalazione SMAD2 in corso è necessaria per la differenziazione del mesendoderma ma è dispensabile per il priming WNT.(A) Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono state lavate per rimuovere il WNT e trattate con ACTIVIN (10 ng/mL). Al fine di bloccare la segnalazione WNT attraverso le cellule β-catenina sono stati trattati con endo-IWR-1 (1 µM), che è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. Al fine di bloccare la secrezione di WNT, le cellule sono state trattate in modo simile con IWP-2 (1 µM). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT, corrispondenti alla condizione di pluripotenza (-/ACT, cerchi aperti), rappresentano i livelli del segnale di fondo. (B) Quantificazione della densità finale delle cellule nell’esperimento mostrato in A e in ulteriori repliche. Non vi è alcuna differenza significativa tra le densità in condizioni diverse (ANOVA). (C) mCitrine-SMAD2 (a sinistra) e GFP-SMAD4 (a destra) risposta in singole celle ad ACTIVIN (10 ng/mL) con o senza priming WNT (linee nere solide e tratteggiate, rispettivamente). ACTIVIN è stato aggiunto a T = 0 ore, che corrisponde a 24 ore di stimolazione WNT. WNT è stato rimosso con il lavaggio prima dell’aggiunta di ACTIVIN. Le immagini sono state acquisite ogni 10 min. Le linee nere solide e tratteggiate rappresentano la risposta media in ogni punto temporale e le linee tratteggiate grigie rappresentano la deviazione standard della popolazione della risposta SMAD2 senza adescamento WNT (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (D) La risposta trascrizionale dei geni mesendoderm ad ACTIVIN con o senza priming WNT. Un ulteriore campione è stato raccolto che è stato trattato con WNT e lasciato non trattato con ACTIVIN (-ACT). L’espressione in ogni campione è stata normalizzata a GAPDH e poi al livello prima dell’aggiunta di ACTIVIN (T = 0 ore). I dati rappresentano la media ±S.D. per n = 3 repliche tecniche. n.d., non rilevato. Risultati simili sono stati ottenuti in due esperimenti indipendenti. (E) Modello per il priming WNT come requisito per il funzionamento dell’ACTIVIN come morfogeno. Dopo l’impianto ACTIVIN funziona in modo indipendente dalla dose per mantenere la pluripotenza dell’epiblasto. All’inizio della gastrulazione, le cellule primarie di segnalazione WNT rispondono alla segnalazione graduata ACTIVIN/SMAD2, che porta al modello anteriore-posteriore delle cellule che emergono dalla striscia primitiva (PS).(A) Pluripotenza maker analisi per lo stesso esperimento mostrato in Figura 7A. Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN (10 ng/mL). Al fine di bloccare la segnalazione WNT attraverso le cellule β-catenina sono stati trattati con endo-IWR-1 (1 µM), che è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. Al fine di bloccare la secrezione di WNT, le cellule sono state trattate in modo simile con IWP-2 (1 µM). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano livelli di pluripotenza. (B-C) Colonie micropatterned in mezzi condizionati trattati con BMP4 (50 ng/mL) o BMP4 (50 ng/mL)+IWP-2 (1 µM) per 42 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. (B) Immagini: BRA (magenta), SOX2 (verde), DAPI (grigio). Barra di scala, 100 µM. (C) Quantificazione della fluorescenza nucleare media in funzione della posizione radiale dal bordo della colonia. Le barre di errore rappresentano la deviazione standard attraverso n = 17 (BMP4, cerchi pieni) e n = 12 (BMP4 +IWP-2, quadrati aperti) colonie da un esperimento. (D) Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN o ACTIVIN seguito da SB (10 µM) aggiunta ai tempi indicati. Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’espressione del marcatore viene eliminata quando l’SB viene aggiunto a T = 0 (quadrati neri) o T = 8 ore (quadrati grigi aperti) dopo l’aggiunta di ACTIVIN. (E) Le celle sono state coltivate per un giorno con WNT3A (100 ng/mL) da sole o in combinazione con SB (10 µM) per bloccare la segnalazione ACTIVIN durante l’adescamento WNT. Il secondo giorno le cellule sono state lavate per rimuovere WNT e trattate con ACTIVIN (10 ng/mL). Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’aggiunta di SB durante l’adescamento WNT (quadrati grigi aperti) riduce solo moderatamente l’espressione del marcatore rispetto al trattamento standard (WNT/ACT, cerchi pieni).
Poiché abbiamo dimostrato che la dinamica di risposta SMAD può essere stabile o transitoria a seconda del ramo attivato, abbiamo poi chiesto se la memoria WNT influisce sulla dinamica della trasduzione del segnale SMAD2. La risposta transitoria di SMAD2 e SMAD4 e l’elevazione nella post-stimolazione della linea di base era la stessa sia che le cellule fossero o meno precedentemente esposte a WNT (Figura 7C). Tuttavia, la trascrizione dei geni mesendodermici è stato stabilizzato in risposta alla ACTIVIN dopo l’adescamento WNT (Figura 7D). Come nel caso di pluripotenza, l’acquisizione del destino mesendodermico stabile richiesto in corso SMAD2 segnalazione in corso nella linea di base elevata come trattamento con SB, 8 ore dopo la stimolazione ACTIVIN, ha eliminato la differenziazione mesendodermica a 24 ore dopo ACTIVIN (Figura 7-figure supplemento 1D). Al contrario, e sostenendo ulteriormente per un meccanismo di adescamento WNT che è temporalmente a monte di ACTIVIN, l’aggiunta di SB durante l’adescamento WNT non ha bloccato la differenziazione mesendoderm (Figura 7-figure supplement 1E). Nel complesso, si conclude che le cellule mantengono una memoria dell’esposizione al WNT, che le rende competenti a differenziarsi in risposta all’ATTIVINA (Figura 7E).

Figura 7-figure supplement 1.Il priming WNT stabilizza la trascrizione del gene mesendoderm senza modificare le dinamiche di risposta SMAD2 e SMAD4. La segnalazione SMAD2 è necessaria per la differenziazione del mesendoderma ma è dispensabile per il priming WNT.(A) Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono state lavate per rimuovere il WNT e trattate con ACTIVIN (10 ng/mL). Al fine di bloccare la segnalazione WNT attraverso le cellule β-catenina sono stati trattati con endo-IWR-1 (1 µM), che è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. Al fine di bloccare la secrezione di WNT, le cellule sono state trattate in modo simile con IWP-2 (1 µM). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT, corrispondenti alla condizione di pluripotenza (-/ACT, cerchi aperti), rappresentano i livelli del segnale di fondo. (B) Quantificazione della densità finale delle cellule nell’esperimento mostrato in A e in ulteriori repliche. Non vi è alcuna differenza significativa tra le densità in condizioni diverse (ANOVA). (C) mCitrine-SMAD2 (a sinistra) e GFP-SMAD4 (a destra) risposta in singole celle ad ACTIVIN (10 ng/mL) con o senza priming WNT (linee nere solide e tratteggiate, rispettivamente). ACTIVIN è stato aggiunto a T = 0 ore, che corrisponde a 24 ore di stimolazione WNT. WNT è stato rimosso con il lavaggio prima dell’aggiunta di ACTIVIN. Le immagini sono state acquisite ogni 10 min. Le linee nere solide e tratteggiate rappresentano la risposta media in ogni punto temporale e le linee tratteggiate grigie rappresentano la deviazione standard della popolazione della risposta SMAD2 senza adescamento WNT (n > 200 cellule per punto temporale). Risultati simili sono stati ottenuti in due esperimenti indipendenti. (D) La risposta trascrizionale dei geni mesendoderm ad ACTIVIN con o senza priming WNT. Un ulteriore campione è stato raccolto che è stato trattato con WNT e lasciato non trattato con ACTIVIN (-ACT). L’espressione in ogni campione è stata normalizzata a GAPDH e poi al livello prima dell’aggiunta di ACTIVIN (T = 0 ore). I dati rappresentano la media ±S.D. per n = 3 repliche tecniche. n.d., non rilevato. Risultati simili sono stati ottenuti in due esperimenti indipendenti. (E) Modello per il priming WNT come requisito per il funzionamento dell’ACTIVIN come morfogeno. Dopo l’impianto ACTIVIN funziona in modo indipendente dalla dose per mantenere la pluripotenza dell’epiblasto. All’inizio della gastrulazione, le cellule primarie di segnalazione WNT rispondono alla segnalazione graduata ACTIVIN/SMAD2, che porta al modello anteriore-posteriore delle cellule che emergono dalla striscia primitiva (PS).(A) Analisi del produttore di Pluripotenza per lo stesso esperimento mostrato in Figura 7A. Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN (10 ng/mL). Al fine di bloccare la segnalazione WNT attraverso le cellule β-catenina sono stati trattati con endo-IWR-1 (1 µM), che è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. Al fine di bloccare la secrezione di WNT, le cellule sono state trattate in modo simile con IWP-2 (1 µM). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano livelli di pluripotenza. (B-C) Colonie micropatterned in mezzi condizionati trattati con BMP4 (50 ng/mL) o BMP4 (50 ng/mL)+IWP-2 (1 µM) per 42 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. (B) Immagini: BRA (magenta), SOX2 (verde), DAPI (grigio). Barra di scala, 100 µM. (C) Quantificazione della fluorescenza nucleare media in funzione della posizione radiale dal bordo della colonia. Le barre di errore rappresentano la deviazione standard attraverso n = 17 (BMP4, cerchi pieni) e n = 12 (BMP4 +IWP-2, quadrati aperti) colonie da un esperimento. (D) Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN o ACTIVIN seguito da SB (10 µM) aggiunta ai tempi indicati. Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’espressione del marcatore viene eliminata quando l’SB viene aggiunto a T = 0 (quadrati neri) o T = 8 ore (quadrati grigi aperti) dopo l’aggiunta di ACTIVIN. (E) Le celle sono state coltivate per un giorno con WNT3A (100 ng/mL) da sole o in combinazione con SB (10 µM) per bloccare la segnalazione ACTIVIN durante l’adescamento WNT. Il secondo giorno le cellule sono state lavate per rimuovere WNT e trattate con ACTIVIN (10 ng/mL). Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’aggiunta di SB durante l’adescamento WNT (quadrati grigi aperti) riduce solo moderatamente l’espressione del marcatore rispetto al trattamento standard (WNT/ACT, cerchi pieni).

Figura 7-figure supplemento 1.La segnalazione SMAD2 in corso è necessaria per la differenziazione del mesendoderma, ma è dispensabile per il priming WNT.(A) Pluripotenza maker analisi per lo stesso esperimento mostrato in Figura 7A. Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN (10 ng/mL). Al fine di bloccare la segnalazione WNT attraverso le cellule β-catenina sono stati trattati con endo-IWR-1 (1 µM), che è stato aggiunto con WNT il primo giorno e di nuovo con ACTIVIN il giorno 2. Al fine di bloccare la secrezione di WNT, le cellule sono state trattate in modo simile con IWP-2 (1 µM). Dopo il secondo giorno le cellule sono state fissate e analizzate con immunofluorescenza. Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano livelli di pluripotenza. (B-C) Colonie micropatterned in mezzi condizionati trattati con BMP4 (50 ng/mL) o BMP4 (50 ng/mL)+IWP-2 (1 µM) per 42 ore. Le colonie sono state fissate e analizzate per immunofluorescenza. (B) Immagini: BRA (magenta), SOX2 (verde), DAPI (grigio). Barra di scala, 100 µM. (C) Quantificazione della fluorescenza nucleare media in funzione della posizione radiale dal bordo della colonia. Le barre di errore rappresentano la deviazione standard attraverso n = 17 (BMP4, cerchi pieni) e n = 12 (BMP4 +IWP-2, quadrati aperti) colonie da un esperimento. (D) Le cellule sono state coltivate per un giorno con WNT3A (100 ng/mL). Il secondo giorno sono stati lavati per rimuovere WNT e trattati con ACTIVIN o ACTIVIN seguito da SB (10 µM) aggiunta ai tempi indicati. Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’espressione del marcatore viene eliminata quando l’SB viene aggiunto a T = 0 (quadrati neri) o T = 8 ore (quadrati grigi aperti) dopo l’aggiunta di ACTIVIN. (E) Le celle sono state coltivate per un giorno con WNT3A (100 ng/mL) da sole o in combinazione con SB (10 µM) per bloccare la segnalazione ACTIVIN durante l’adescamento WNT. Il secondo giorno le cellule sono state lavate per rimuovere WNT e trattate con ACTIVIN (10 ng/mL). Le cellule sono stati fissati a 24 ore dopo il trattamento con ACTIVIN e analizzati per BRA, EOMES, e l’espressione GSC da immunofluorescenza (IF). Gli istogrammi mostrano il segnale nucleare IF quantificato in singole cellule (n > 5.000 cellule per condizione). Le cellule coltivate senza WNT (-/ACT, cerchi aperti) rappresentano i livelli del segnale di fondo. L’aggiunta di SB durante l’adescamento WNT (quadrati grigi aperti) riduce solo moderatamente l’espressione del marcatore rispetto al trattamento standard (WNT/ACT, cerchi pieni).
Discussione
Seguendo la tradizione dell’embriologia sperimentale, alla fine degli anni Sessanta, Nieuwkoop iniziò una serie di esperimenti di espianto e trapianto nella blastula anfibia che portò alla scoperta dell’ACTIVIN come induttore di mesodermi negli anni Ottanta (Nieuwkoop, 1969; McDowell e Gurdon, 1999). Quando è stato presentato agli espianti isolati di cappello animale che normalmente danno origine solo all’ectoderma, l’ACTIVIN è stato sufficiente per indurre diversi tipi di cellule mesendodermiche in base alla sua concentrazione, ed è stato quindi qualificato in linea di principio come morfogeno (Green e Smith, 1990; Wilson e Melton, 1994). Quando i recettori ACTIVIN e le vie SMAD sono stati caratterizzati nel 1990 s, è stato dimostrato che la micro-iniezione di diverse quantità di mRNA sintetici che codificano SMAD2 in capsule animali Xenopus, ricapitola anche gli effetti mesendodermici di una presentazione morfogena A ACTIVIN, confermando in modo indipendente che l’attivazione di diverse soglie del percorso era sufficiente per l’induzione del mesoderma (Shimizu e Gurdon, 1999). L’inibizione della via per microiniezione di un recettore dominante negativo di tipo II ACTIVIN nello Xenopus blastula ha portato alla perdita completa dei derivati mesendodermici, dimostrando che la segnalazione ACTIVIN/SMAD2 era necessaria per l’induzione del mesoderma nell’embrione anfibio (Hemmati-Brivanlou e Melton, 1992).
L’analisi della perdita di funzionalità nel topo ha successivamente confermato le conclusioni della rana sul percorso, poiché i topi SMAD2/3 a doppio knockout non sono riusciti a indurre correttamente il mesendoderma e a gastruire (Dunn et al., 2004). Tuttavia un altro ligando TGFβ, NODAL, piuttosto che ACTIVIN è stato dimostrato essere l’induttore nell’embrione di mammifero (Conlon et al., 1994). Eleganti esperimenti genetici eseguiti anche nel topo ha posto la segnalazione NODAL in un loop di feedback positivo che guida la gastrulazione. Il loop viene avviato quando la segnalazione NODAL induce l’espressione BMP4 nell’ectoderma extra-embrionico. La segnalazione BMP induce successivamente l’espressione WNT3, che a sua volta induce alti livelli di NODAL nella parte prossimale-posteriore dell’embrione, che segna il sito di iniziazione della striscia primitiva (Arnold e Robertson, 2009).
Utilizzando i reporter in diretta di SMAD1, SMAD2 e SMAD4, troviamo che la segnalazione BMP/SMAD1 è stabile, la segnalazione ACTIVIN/SMAD2 è transitoria (o adattiva), e che co-SMAD4 segue la dinamica del recettore associato SMADs. In risposta ad ACTIVIN, gli hESCs inducono transitoriamente l’espressione del gene mesendodermico, senza generare destini mesendodermici, e le cellule ritornano allo stato di pluripotenza. Tuttavia, l’espressione degli inibitori di ACTIVIN/NODAL è rimasta elevata e plausibilmente buffer l’ACTIVIN aggiuntivo, come previsto dalla teoria precedente (François e Siggia, 2008). La pre-presentazione di WNT, tuttavia, stabilizza la trascrizione e l’acquisizione del destino senza modificare le dinamiche SMAD2. Così, una risposta di segnalazione transitoria è compatibile con un cambiamento stabile del destino delle cellule, come mostrato in precedenza in una linea cellulare mioblastica murina (Sorre et al., 2014). Sia il mantenimento della pluripotenza che l’acquisizione del destino mesendodermico non sono influenzati dalla perdita di SMAD3. I nostri dati forniscono la prova di un livello di integrazione del segnale non rilevato in precedenza che implica la presenza di una memoria cellulare. Questa memoria opera non a livello della dinamica di segnalazione SMAD2, ma con qualche altro mezzo per modificare la risposta delle cellule ad ACTIVIN. Si è tentati di ipotizzare che possa essere mediata attraverso meccanismi epigenetici, in modo simile a quanto descritto negli esperimenti di Xenopus dove la β-catenina recluta PrMT2 per indurre destini dorsali (Blythe et al., 2010).
Il percorso ACTIVIN/SMAD2 si è dimostrato necessario per il mantenimento della pluripotenza, ma non sufficiente a indurre il mesendoderma negli hESC, tranne quando è influenzato da modificatori di altri percorsi di segnalazione (D’Amour et al., 2005; McLean et al., 2007; Singh et al., 2012; Funa et al., 2015). Il nostro studio mostra che il semplice pretrattamento degli hESC con WNT, conferisce loro una memoria che permette una risposta graduata all’ACTIVIN successivamente applicato; non è richiesta l’esposizione simultanea a WNT e ACTIVIN. Inoltre, il trattamento WNT da solo non è sufficiente a provocare un cambiamento di destino stabile in quanto in media minimi, senza l’ACTIVIN, le cellule ritornano alla pluripotenza. La capacità delle cellule embrionali di registrare i segnali WNT può essere un aspetto ampiamente conservato e fondamentale dello sviluppo animale, come dimostrato da eleganti esperimenti in Drosophilia dove gli autori dimostrano un effetto morfogeno per Dpp (un omologo BMP) dopo che le cellule hanno perso il contatto con la fonte di WNT (Alexandre et al., 2014).
Nel contesto dello sviluppo dei vertebrati, le nostre scoperte costringono a rivalutare la letteratura tradizionale per quanto riguarda la sufficienza del percorso ACTIVIN/SMAD2 per l’induzione mesendodermica e il patterning. Una revisione delle prove nei sistemi modello per l’effetto morfogeno mediato da ACTIVIN / NODAL rivela che in tutte le impostazioni sperimentali, almeno alcune delle cellule erano ancora sotto l’influenza, o erano stati precedentemente esposti alla segnalazione WNT prima o durante la segnalazione ACTIVIN / NODAL. Ad esempio, le cellule del tappo animale Xenopus derivato dalla blastula embrione stadio stadio sono stati sotto l’influenza materna WNT già in fase di due cellule, ore prima che l’ACTIVIN è presentato agli espianti. Questa influenza è la conseguenza della rotazione corticale che si verifica dopo l’ingresso dello sperma, e attiva il percorso WNT sul lato dorsale dell’embrione, come evidenziato dalla localizzazione nucleare dorsale specifica della β-catenina (Schneider et al., 1996; Larabell et al., 1997; Rowning et al., 1997). È chiaro dall’allungamento asimmetrico degli espianti del cappello animale in risposta ad ACTIVIN che l’esposizione WNT precedente stabilisce un pre-pattern dorsale-ventrale che influisce sulla risposta. È anche chiaro che le regioni dorsale e ventrale del cappello animale quando sono separate rispondono in modo diverso all’ACTIVIN (Sokol e Melton, 1991; Bolce et al., 1992). Anche se alcuni mesodermi sono indotti nelle calotte ventrali, solo le cellule della calotta dorsale che hanno visto i segnali WNT subiscono l’induzione mediata da ACTIVIN di GSC + mesendoderm. Questi esperimenti dimostrano che entrambi i percorsi sono necessari per il disegno mesendoderm completo senza distinguere esplicitamente il loro rapporto temporale.
Il nostro studio rivede questo punto di vista portando un ordine temporale. Suggeriamo che solo le cellule dotate di una memoria WNT possono rispondere ad ACTIVIN per specificare l’intera gamma di destini mesendodermici. Anche nell’embrione di Xenopus ampiamente studiato, si discute ancora se nella zona marginale dello stadio tardivo della blastula, prima dell’inizio della gastrulazione, un gradiente ACTIVIN/NODAL definisce già i destini mesendodermici da mediale a laterale o se c’è semplicemente una divisione bipartita in organizzatore dorsale e mesoderma ventrale (Harland and Gerhart, 1997; Smith, 2009). Questo dibattito si traduce anche nel fatto se ACTIVIN/NODAL sia sufficiente a pre-disegnare il mesendoderma o se il disegno completo richieda una precedente esposizione al WNT presente solo nella parte dorsale dell’embrione. Le controversie persistono, dal momento che l’embrione si sta sviluppando rapidamente, non ci sono segnalazioni in diretta, e i destini sono spesso saggiate nella gastrulazione precoce piuttosto che nella blastula tardiva. Nel topo la prima manifestazione della striscia è l’espressione prossimale-posteriore di WNT, che si estende distalmente. A metà della fase di striatura, la segnalazione NODAL è più alta nel Nodo (Tam e Loebel, 2007). Così, le cellule che lasciano la striscia in varie posizioni prossimale-distale e livelli NODAL, manifestamente sono stati esposti a WNT prima (Tam e Behringer, 1997). Questa interpretazione, tuttavia, non elimina il co-richiesta di ACTIVIN e WNT, ma suggerisce piuttosto che il WNT istruttivo, necessario per la differenziazione mesendodermica, si verifica temporalmente a monte della segnalazione ACTIVIN/SMAD2.
In conclusione, una memoria cellulare di segnalazione WNT, conferisce alle cellule embrionali, compresi gli HESC, la competenza di mantenere ACTIVIN/SMAD2-mediata mesendoderm specifica del destino mesendoderm e patterning.
Materiali e metodi
| Tipo di reagente (specie) o risorsa | Designazione | Fonte o riferimento | Identificatori | Ulteriori informazioni |
|---|---|---|---|---|
| Linea cellulare (Homo sapiens, XX) | RUES2 | US National Institutes of Health, registro delle CSE umane | numero di registro del CES umano 0013; RRID:CVCL_VM29 | Linea di cellule staminali embrionali umane |
| Linea cellulare (Homo sapiens, XX) | RUES2-RFP-SMAD1 | questo documento | CRISPR / Cas9-ha modificato la linea di cellule staminali embrionali umane | |
| Linea cellulare (Homo sapiens, XX) | RUES2-mCit-SMAD2 | questo documento | CRISPR / Cas9-ha modificato la linea di cellule staminali embrionali umane | |
| Linea cellulare (Homo sapiens, XX) | RUES2-GFP-SMAD4 | PMID: 28760810 | CRISPR / Cas9-ha modificato la linea di cellule staminali embrionali umane | |
| Linea cellulare (Homo sapiens, XX) | RUES2-SMAD3-/- clone #1 | questo documento | CRISPR / Cas9-ha modificato la linea di cellule staminali embrionali umane | |
| Linea cellulare (Homo sapiens, XX) | RUES2-SMAD3-/- clone #2 | questo documento | CRISPR/Cas9-ha modificato la linea di cellule staminali embrionali umane | |
| Linea cellulare (Homo sapiens, XY) | CORRISPETTI1 | US National Institutes of Health, registro ESC umano | numero di registro del CES umano 0012; RRID:CVCL_B809 | Linea di cellule staminali embrionali umane |
| Anticorpo | anti-Brachyury (poliAb di capra) | Sistemi R e D | Cat. #: AF2085; RRID:AB_2200235 | IF (1:300) |
| Anticorpo | anti-Brachiria (coniglio mAb) | Sistemi R e D | 3. Cat. #: MAB20851 | IF (1:200) |
| Anticorpo | anti-Cdx2 (mAb del mouse) | Abcam | Cat. #: ab15258; RRID:AB_2077042 | IF (1:50) |
| Anticorpo | anti-Eome (mAb del mouse) | Sistemi R e D | Cat. #: MAB6166; RRID:AB_10919889 | IF (1:200) |
| Anticorpo | anti-Lamina B1 (coniglio polyAb) | Proteintech | Cat. #: 12987-1-AP; RRID:AB_2136290 | WB (1:2000) |
| Anticorpo | anti-Goosecoide (poliAb di capra) | Sistemi R e D | Cat. #: AF4086; RRID:AB_2114650 | IF (1:100) |
| Anticorpo | anti-Nanog (poliAb di capra) | Sistemi R e D | Cat. #: AF1997; RRID:AB_355097 | IF (1:200) |
| Anticorpo | anti-Oct3/4 (mAb del mouse) | BD Bioscienze | Cat. #: 611203; RRID:AB_398737 | IF (1:400) |
| Anticorpo | anti-Smad2 (coniglio mAb) | Segnalazione cellulare | Cat. #: 3122; RRID:AB_10697649 | SE (1:200), WB (1:1000) |
| Anticorpo | anti-Smad2/3 (mAb del mouse) | BD Bioscienze | Cat. #: 610842; RRID:AB_398161 | IF (1:100) |
| Anticorpo | anti-Sox17 (poliAb di capra) | Sistemi R e D | Cat. #: AF1924; RRID:AB_355060 | IF (1:200) |
| Anticorpo | anti-Sox2 (coniglio mAb) | Segnalazione cellulare | Cat. #: 3579; RRID:AB_2195767 | IF (1:200) |
| Peptide, proteina ricombinante | ricombinante umano / topo / ratto Attivare A | Sistemi R e D | Cat. #: 338-AC/CF | |
| Peptide, proteina ricombinante | BMP-4 umano ricombinante | Sistemi R e D | Cat. #: 314 BP | |
| Peptide, proteina ricombinante | mouse ricombinante Wnt-3a | Sistemi R e D | Cat. #: 1324-WN | |
| Peptide, proteina ricombinante | Laminina umana ricombinante-521 | BioLamina | ||
| Composto chimico, farmaco | endo-IWR-1 | Tocris | Cat. #: 3532 | |
| Composto chimico, farmaco | IWP-2 | Stemgent | Cat. #: 04–0034 | |
| Composto chimico, farmaco | SB431542 | Stemgent | Cat. #: 04–0010 | |
| Composto chimico, farmaco | Y-27632 | Abcam | Cat. #: Ab120129 | |
| Software, algoritmo | MATLAB | MathWorks | RRID:SCR_001622 | |
| Software, algoritmo | StemCellTracker | https://github.com/ChristophKirst/StemCellTracker | Segmentazione delle immagini e analisi del segnale | |
| Software, algoritmo | R | https://www.r-project.org/ | ||
| Software, algoritmo | DESeq2 Pacchetto bioconduttore | PMID: 25516281 | Analisi dell’espressione genica differenziale | |
| Software, algoritmo | Pacchetto PWMEnrich Bioconductor | https://bioconductor.org/packages/release/bioc/html/PWMEnrich.html | Analisi dell’arricchimento del motivo | |
| Software, algoritmo | Pacchetto GOseq Bioconductor | PMID: 20132535 | Analisi statistica delle categorie sovra/sotto rappresentate | |
| Software, algoritmo | AME | PMID: 25953851 | Analisi dell’arricchimento del motivo | |
| Software, algoritmo | Marea | PMID: 25300484 | Sequenza di decomposizione traccia | |
| Software, algoritmo | Cluster 3.0 | PMID: 14871861 | Clustering | |
| Software, algoritmo | Java TreeView | PMID: 15180930 | Visualizzazione dei dati |
Coltura di cellule staminali embrionali umane
Sono stati effettuati esperimenti con la linea RUES2 hESC (XX femmina; US National Institutes of Health, US National Institutes of Health, human ESC registry no. 0013) o CRISPR/Cas9 linee cellulari modificate sulla base di RUES2. Gli esperimenti chiave sono stati ripetuti con la linea RUES1 hESC (XY maschio; US National Institutes of Health, registro ESC umano n. 0012). RUES1 e RUES2 sono stati autenticati con il profilo STR ed entrambi sono risultati negativi alla contaminazione da micoplasmi. Gli hESC sono stati coltivati in un terreno HUESM condizionato da fibroblasti embrionali di topo e integrato con 20 ng/mL bFGF (MEF-CM). Le cellule sono state coltivate su piatti di coltura dei tessuti (BD Biosciences, San Jose, CA) per il mantenimento e l’espansione a 37°C e 5% di CO2. I piatti sono stati rivestiti durante la notte a 4 ° C con Geltrex (Thermo Fisher Scientific, Waltham, MA) diluito 1:40 in DMEM/F12 e poi incubato a 37 ° C per almeno 20 minuti prima di passare. Le cellule sono state passate come aggregati utilizzando Gentle Cell Dissociation Reagent (STEMCELL Technologies, Vancouver, Canada).
Coltura cellulare micropatterned
I singoli coprioggetti micropatterned (CYTOO, Grenoble, Francia) sono stati lavati una volta con acqua per 5 minuti a temperatura ambiente (RT) per attivare la superficie secondo le raccomandazioni del produttore. I coprioggetti sono stati poi rivestiti a 37°C per 2 ore con 20 µg/mL Lamininin-521 (BioLamina, Sundbyberg, Svezia) in 0,5 mL PBS + Mg/+Ca. La laminina è stata poi rimossa con diluizioni seriali senza lasciare asciugare il coprioggetti (diluizione 1:4 in PBS -Mg/-Ca, sei volte). I chip sono stati seminati immediatamente o conservati per una notte a 4°C in 2 mL di PBS -/- e seminati il giorno successivo.
La semina in cella è stata eseguita come segue. Le cellule che crescono in MEF-CM sono state lavate una volta con PBS +/+ e dissociate a singole cellule con Accutase (STEMCELL Technologies). Le cellule sono state centrifugate e 600.000 cellule sono state risospese in 2 mL di MEF-CM integrate con 10 µM Rock-inhibitor (Y-27632, Abcam, Cambridge, MA). La sospensione cellulare è stato poi posto sopra il coprioggetto in un piatto 35 mm di coltura tissutale. Il campione è stato lasciato imperturbato per 10 minuti a RT al fine di ottenere la semina omogenea delle cellule in tutto il chip prima di essere spostato in incubatrice. Dopo 2 ore, il mezzo è stato sostituito con MEF-CM senza Rock-inibitore e le cellule sono state incubate durante la notte. Il giorno seguente, il mezzo è stato cambiato in MEF-CM con ligandi aggiuntivi, BMP4, WNT3A, o ACTIVIN A (R e D Systems, Minneapolis, MN) con o senza 10 µM SB431542 (Stemgent, Lexington, MA). Abbiamo stimolato le cellule con ACTIVINA A piuttosto che NODAL, perché anche se entrambi i ligandi differenziano gli HESC verso il lignaggio mesendoderm, NODAL richiede concentrazioni molto più elevate per essere efficace (McLean et al., 2007). Per gli esperimenti in TeSR-E7 (STEMCELL Technologies), le cellule sono state spostate al mezzo E7 il giorno dopo la semina e incubate per altre 24 ore prima di aggiungere i ligandi. L’imaging dal vivo è stato effettuato in mezzo di imaging E7, che è stato preparato con FluoroBrite DMEM (Thermo Fisher Scientific) secondo il protocollo pubblicato per E8 (Beers et al., 2012).
Cultura monocellulare
I piatti per coltura di tessuti plastici di qualità ottica (ibidi, Martinsried, Germania) sono stati rivestiti con 10 µg/mL di Lamininin-521 in PBS +/+ per 2 ore a 37°C o per una notte a 4°C. Singole cellule sono state raccolte come descritto per la coltura cellulare micropatterned e piatti sono stati seminati con 50.000 singole cellule risospese in 2 mL E7 integrato con 10 µM Rock-inibitore. I campioni sono stati incubati durante la notte. Il giorno seguente, il mezzo è stato cambiato in E7 integrato con 10 µM Rock-inibitore e ligandi o piccole molecole. Per il protocollo di 2 giorni in cui le cellule sono state commutate da E7 ±100 ng/mL WNT3A a E7 ±10 ng/mL ACTIVIN A, i campioni sono stati lavati con PBS + / + + prima di aggiungere terreno fresco. L’imaging dal vivo è stato effettuato in mezzo di imaging E7. Piccole molecole sono state utilizzate alle seguenti concentrazioni e sono state sostituite ogni 24 ore: 10 µM SB431542 (Stemgent), 1 µM IWP-2 (Stemgent), e 1 µM endo-IWR-1 (Tocris).
Immunofluorescenza e western blotting
Le celle su piatti o coprivetrini sono state risciacquate una volta con PBS -/-, fissate con paraformaldeide al 4% (Alfa Aesar, Thermo Fisher Scientific, Tewksbury, MA) per 20 minuti a RT, e poi risciacquate due volte e conservate in PBS -/-. Le cellule sono state bloccate e permeabilizzate con tampone di blocco (2% siero albumina bovina e 0,1% Triton X-100 in PBS -/-) per 30 minuti a RT. Le cellule sono state incubate con anticorpi primari in tampone di blocco durante la notte a 4 ° C e poi lavate tre volte con 0,1% Tween-20 in PBS -/- (PBST). Le cellule sono state incubate con anticorpi secondari (diluito 1:1000): Alexa Fluor 488, 555, o 647-coniugati da (Invitrogen Molecular Probes, Thermo Fisher Scientific) e colorazione nucleare DAPI (diluizione 1:10.000) in tampone di blocco per 30 minuti a RT, e poi lavate due volte con PBST e una volta con PBS -/-. I piatti sono stati immagazzinati e imagini in PBS -/-. I coprioggetti sono stati montati su slitte utilizzando il mezzo di montaggio Fluoromount-G (SouthernBiotech, Birmingham, AL).
Il blotting occidentale è stato eseguito con tecniche standard. Le frazioni nucleari presentate in Figura 3-figure supplement 1C sono state ottenute utilizzando i reagenti di estrazione nucleare e citoplasmatica NE-PER (Thermo Fisher Scientific) e seguendo il protocollo dei produttori. A seguito di SDS-PAGE e trasferimento occidentale, le membrane sono state colorate con anti-Smad2 Rabbit mAb (Cell Signaling Technology, Danvers, MA) a 1:1000 diluizione. Le membrane sono state spogliate per 15 minuti a RT utilizzando Restore PLUS Western Blot Stripping Buffer (ThermoFisher Scientific) e rimacchiate con anti-Lamin B1 Rabbit polyAb (Proteintech, Rosemont, IL) alla diluizione 1:2000.
Imaging
Le immagini ad ampio campo di campioni fissi sono state acquisite su un microscopio rovesciato Olympus IX-70 con obiettivo ad apertura numerica 10x/0,4. L’acquisizione di immagini piastrellate è stata utilizzata per acquisire immagini di grandi aree di parabole o coprioggetti in quattro canali corrispondenti a DAPI e Alexa Fluor 488, 555 e 647. L’imaging dal vivo è stato eseguito su un microscopio confocale a disco rotante dotato di laser 405, 488 e 561 nm e di una camera ambientale (CellVoyager CV1000, Yokogawa). Le immagini sono state acquisite ogni 10 minuti con un obiettivo ad apertura numerica 20x/0,75. Le cellule sono state mantenute a 37°C e al 5% di CO2 durante l’imaging dal vivo.
Generazione di linee di cellule reporter SMAD1 e SMAD2
Per le cellule reporter tagRFP-SMAD1, l’ingegneria del genoma mediata da CRISPR/Cas9 è stata utilizzata per fondere una cassetta contenente un gene di resistenza alla blasticidina (BsdR), un peptide T2A auto-cleante e una proteina fluorescente tagRFP sul termino N di SMAD1, in modo che il locus produca sia una proteina di fusione tagRFP-SMAD1 che BsdR. Allo stesso modo, per la linea mCitrine-SMAD2 reporter, CRISPR/Cas9 è stato utilizzato per fondere una cassetta contenente un gene di resistenza alla puromicina (PuroR), un peptide T2A auto-cleante e una proteina fluorescente mCitrina sul N-termine di SMAD2, in modo che il locus produca sia una proteina di fusione mCitrine-SMAD2 insieme a PuroR.
RUES2 hESCs sono stati nucleofected con un plasmide pX335 (Cong et al., 2013) che co-esprime la versione nickase di Cas9 e lo sgRNA specifico di interesse (sequenze di protospacer: 5′-GCAGCACTAGACTAGTTATACTCCT-3′ per SMAD1 e 5′-GGACGACGACATGTTTTCTACCAA-3′ per SMAD2), così come l’appropriata omologia del plasmide donatore. La nucleofezione è stata effettuata utilizzando il Kit Nucelofettore Linea Cellulare L (Lonza, Walkersville, MD) e l’impostazione B-016 di uno strumento Nucleofettore II. Le cellule nucleofected sono stati placcati in MEF-CM integrato con 10 µM Rock-inibitore. Dopo 4 giorni, blasticidina o puromicina è stato aggiunto per 7 giorni per selezionare per le cellule che erano state prese di mira. Le cellule che sono sopravvissute alla selezione sono state passate come cellule singole utilizzando Accutase, placcate in MEF-CM integrato con 10 µM Rock-inibitore, e ha permesso di crescere in colonie. Le colonie derivanti da una singola cellula sono state selezionate a mano, espanse, e schermate per il corretto targeting mediante amplificazione PCR della regione genomica e sequenziamento Sanger. I cloni correttamente mirati sono stati successivamente trasfettati con plasmidi ePiggyBac contenenti cassette di H2B-mCitrina o H2B-mCherry per consentire l’etichettatura nucleare per il tracciamento delle cellule (Lacoste et al., 2009). I singoli cloni sono stati nuovamente isolati e controllati per il cariotipo normale (G-banding) e il mantenimento della pluripotenza.
Generazione di linee di cellule SMAD3 knockout
Gli hESC RUES2 sono stati nucleofected con un plasmide pX330 (Cong et al., 2013) che ha coespreso la versione wild-type di Cas9 e uno dei due sgRNA che hanno come obiettivo SMAD3 (protospacer sequences): 5′-CCACCAGATGAACCACACCACAGCA-3′ per sgRNA #1 e 5′-TTATTATGTGTGGGGGGGACAT-3′ per sgRNA #2). Gli sgRNA sono stati progettati per indirizzare il primo o il secondo esone di codifica condiviso da tutte le isoforme SMAD3, una strategia che è stata usata con successo per mettere fuori combattimento SMAD3 nelle linee cellulari primarie umane (Voets et al., 2017). Due diversi sgRNA sono stati utilizzati per controllare gli effetti fuori bersaglio. Abbiamo modificato il plasmide pX330 per esprimere anche una cassetta di puromicina-2A-EGFP per arricchire le cellule che erano state nucleofettate con successo. Nucleofection è stata effettuata come descritto in precedenza e le cellule sono state placcate in MEF-CM integrato con 10 µM Rock-inibitore. Il giorno seguente è stata aggiunta puromicina per 24 ore. Le cellule che sono sopravvissute alla selezione sono state lasciate recuperare per diversi giorni. Le cellule sono state poi passate come cellule singole usando l’Accutase, placcate in MEF-CM integrate con 10 µM Rock-inibitore, e hanno potuto crescere in colonie. Le colonie derivanti da una singola cellula sono state selezionate a mano, espanse, e schermate per il corretto targeting mediante amplificazione PCR della regione genomica e sequenziamento Sanger. I cromatogrammi risultanti sono stati decomposti utilizzando lo strumento Tide web-based (Brinkman et al., 2014).
RT-PCR
Le cellule RUES2 sono state seminate in piastre a 6 pozzetti (1.00.000 singole cellule per pozzetto) in mezzo E7 integrato con 10 µM Rock-inibitore e incubato durante la notte. Il giorno seguente il mezzo è stato cambiato in E7 integrato con 10 µM Rock-inibitore con o senza 10 ng/mL ACTIVIN A. I campioni (tre pozzetti in pool) sono stati raccolti in 1 mL Trizol a 0, 2,5, 4, 6 e 12 ore. Il protocollo di 2 giorni è stato effettuato in modo simile: il mezzo è stato cambiato il giorno dopo la semina a E7 integrato con 10 µM Rock-inibitore con o senza 100 ng/mL WNT3A. Il secondo giorno dopo la semina i pozzi sono stati lavati una volta con PBS e il mezzo è stato cambiato in E7 integrato con 10 µM Rock-inibitore con o senza 10 ng/mL ACTIVIN A. I campioni sono stati raccolti in 1 mL Trizol prima dell’aggiunta di WNT3A, dopo il trattamento WNT3A (indicato come 0 hr), e a 2.5, 4, 8 e 12 ore dopo l’aggiunta di ACTIVINA A. RNA cellulare totale per ogni campione è stato estratto utilizzando il Mini Kit RNeasy (QIAGEN, Germantown, MD) e cDNA è stato sintetizzato utilizzando il Trascriptor First Strand cDNA Synthesis Kit (Roche). La RT-PCR per i geni selezionati è stata eseguita su tre repliche tecniche utilizzando il LightCycler 480 SYBR Green I Master mix in uno strumento LightCycler 480 (Roche, Basilea, Svizzera). I primer sono stati progettati utilizzando Primer-BLAST (Ye et al., 2012) o ottenuti da qPrimerDepot (Cui et al., 2007) o da sequenze precedentemente pubblicate (Mendjan et al., 2014). Le sequenze di primer e la fonte sono elencate nella Tabella 1.
| Simbolo del gene | Avanti primer | Primer invertito | Fonte: |
|---|---|---|---|
| NANOG | TCCAACATCCTATTTGAACCTCAGC | ACCATTGCTATTCTTTTCGGCCA | Primer-BLAST |
| OCT4 | AAACCCACACACTGCAGCAGAT | TGTGCATAGTCGGCTGCTTTTGA | Primer-BLAST |
| SOX2 | TACAGCATGATGATGCAGGACCA | CCGTTCATGTAGGTATGCGA | Primer-BLAST |
| NODALE | AGACATCATCATCCGGCAGCCTAC | CAAAAGCAAACGTCCCCAGTTTTTOO | Primer-BLAST |
| GAPDH | AATCCCATCACCATTTCCA | TGGACTCCACGACGACGTACTCA | Primer-BLAST |
| ■SMAD7 | CCAGGCTCCAGAAGAAGTTG | CCAACTGCAGACTGTCCAGA | qPrimerDepot |
| SINISTRA1 | CTCCATGCCGAACACCAG | GGAAAGGGTTTTCAGCCCCAGAGGGGGGIIIOO DDII GGAAAGGGGGGGGIIIOO | qPrimerDepot |
| SINISTRA2 | TCAATGTAATGTACATTTTTTTTGGCG | CTGGACCTCAGTCAGGGACTATGG | qPrimerDepot |
| BR | CGTTGGCTCACACAGACCACAG | ATGACAATTGGTCCAGTTTOO | qPrimerDepot |
| GSC | GAGGAGAAAGTGGGGAGGTGTCTGGTTTTTT | CTCTGATGATGAGGACCGACCCTTTTTTTOO | Mendjan et al. |
| UOMO | CACATTGTAGTGTGGCAGTGGCAGTGG | CGCCACCAAACTGAGATGATGATAT | Mendjan et al. |
Sequenza RNA totale
Le cellule RUES2 sono state seminate in piastre a 6 pozzetti (200.000 singole cellule per pozzetto) in mezzo E7 integrato con 10 µM Rock-inibitore e incubato durante la notte. Il giorno seguente il mezzo è stato cambiato in E7 integrato con 10 µM Rock-inibitore con o senza 10 ng/mL ACTIVIN A. I campioni (tre pozzetti in pool) sono stati raccolti in 1 mL Trizol a 1, 2,5, 4, 8 e 12 ore per le condizioni trattate con ACTIVIN e dopo 0, 6 e 12 ore per le condizioni no-ACTIVIN (da utilizzare come controlli negativi). RNA cellulare totale per ogni campione è stato estratto utilizzando RNeasy Mini Kit (QIAGEN) e 2 ug di RNA totale è stato utilizzato per preparare ogni singolo RNA-seq biblioteca. La costruzione della biblioteca di RNA-seq è stata condotta con il TruSeq RNA Library Preparation Kit (Illumina, San Diego, CA) secondo le istruzioni del produttore e in sequenza in un apparecchio Illumina HiSeq 2500. Le letture grezze sono state mappate su hg19 utilizzando l’allineatore STAR, e i conteggi delle letture geniche sono stati normalizzati utilizzando il pacchetto DESeq2 Bioconductor (Love et al., 2014). La preparazione della biblioteca, il sequenziamento e la mappatura sono stati eseguiti dal New York Genome Center (New York, NY, USA). Tutti i file di dati grezzi sono disponibili nella banca dati GEO (numero di adesione GSE111717).
Analisi delle immagini
Per le immagini acquisite da esperimenti di coltura cellulare micropatterned, lo stitching e il rilevamento delle colonie sono stati effettuati come descritto in precedenza utilizzando un software personalizzato scritto in MATLAB (Etoc et al., 2016). Per l’analisi delle linee di reporter SMAD su colonie micropatterned è stato analizzato un singolo piano z attraverso il centro della colonia in ogni punto temporale. Lo sfondo in ogni canale è stato rimosso sottraendo un’immagine di minima intensità che è stata generata prendendo il valore minimo ad ogni pixel su tutte le immagini di quel canale nello stesso piano z. La vignettatura è stata corretta dividendo per un’immagine a campo piatto che è stata generata normalizzando l’intensità dell’immagine minima a 1. Questa procedura corregge la caduta di intensità al bordo delle immagini senza alterare ulteriormente l’intensità media dell’immagine.
La segmentazione dei nuclei e la quantificazione del segnale sono state eseguite sulle immagini corrette come segue. L’immagine H2B è stata impostata su una soglia per generare un’immagine binaria che separa il primo piano (nuclei) dallo sfondo. L’immagine H2B originale, corretta, è stata poi filtrata con un filtro mediano e un filtro a sfera con parametri che corrispondono alle dimensioni previste dei singoli nuclei. I massimi locali corrispondenti ai singoli nuclei sono stati rilevati utilizzando la funzione di trasformazione MATLAB extended-maxima transform. I massimi sono stati dilatati per aumentare la probabilità di ottenere un singolo massimo per nucleo. I massimi che rientrano nel primo piano sono stati utilizzati come semi per la segmentazione dello spartiacque, che è stata anch’essa limitata al primo piano ed è stata utilizzata per ottenere un oggetto etichettato corrispondente a ciascun nucleo all’interno dell’immagine. Gli oggetti segmentati sono stati ulteriormente elaborati per eliminare oggetti molto più grandi o più piccoli delle dimensioni previste dei singoli nuclei. I risultati della segmentazione sono stati utilizzati come maschera per ottenere la mediana dell’intensità nucleare per cella in ogni canale. Le immagini acquisite da campioni fissi che sono stati colorati da immunofluorescenza sono stati analogamente segmentati e analizzati. Il canale DAPI è stato utilizzato per eseguire la segmentazione dei nuclei, e gli oggetti segmentati sono stati successivamente utilizzati per ottenere la mediana per cella intensità del segnale in ogni canale.
Per la linea mCitrine-SMAD2 reporter un arricchimento del segnale mCitrino nel citoplasma rispetto al nucleo prima della presentazione ligando potrebbe essere rilevato, che è diminuito in concomitanza con un aumento del segnale nucleare dopo la presentazione ACTIVIN. Pertanto, la risposta mCitrine-SMAD2 è stato quantificato come il rapporto nucleare-citoplasma, che è stato utilizzato in precedenza come lettura per l’attività del percorso TGFβ (Warmflash et al., 2012). Al fine di stimare il segnale citoplasmatico per ogni cellula, una stretta ciambella che circonda la maschera nucleare è stata formata dilatando la maschera nucleare una volta per un raggio interno e una seconda volta per un raggio esterno e sottraendo il primo oggetto dilatato dal secondo. La ciambella, che formava la maschera citoplasmatica, non era limitata ai pixel in primo piano (segnale H2B), ma si evitava la sovrapposizione con le maschere dei nuclei vicini. L’intensità mediana mCitrino all’interno della maschera nucleare è stata divisa per l’intensità mediana all’interno della maschera citoplasmatica su base cellulare. Il comportamento qualitativo della risposta nucleare-citoplasmatica SMAD2 non era sensibile alle dimensioni esatte del raggio interno ed esterno della ciambella. Pertanto, i valori sono stati scelti manualmente e mantenuti fissi durante tutte le analisi. Per la linea reporter RFP-SMAD1 è stato possibile rilevare un debole segnale nucleare prima della presentazione del ligando. Tuttavia, non è stato possibile rilevare alcun segnale citoplasmatico sopra lo sfondo. Pertanto, la risposta RFP-SMAD1 è stata quantificata come il segnale nucleare mediano normalizzato al segnale mediano H2B per normalizzare per le cellule che si muovono dentro e fuori dal piano z. Al fine di analizzare la risposta SMAD in funzione della posizione radiale all’interno delle colonie micropatterned, le cellule all’interno di una singola colonia sono state binned in base alla posizione radiale del loro centro e la risposta media per cella all’interno di ogni bin è stata calcolata. Il profilo radiale per le singole colonie è stato poi mediato su più colonie. L’analisi delle linee di reporter SMAD nella cultura monocellulare è stata effettuata in modo simile. È stata analizzata l’immagine di proiezione di massima intensità, piuttosto che un singolo z-plane. Poiché le cellule sono rimaste piatte in queste condizioni non è stato necessario normalizzare il segnale SMAD1 con H2B.
Analisi RNAseq e clustering
Per ogni gene è stato sottratto dai valori di espressione dei campioni trattati con ACTIVIN un profilo di espressione di base, calcolato utilizzando un’interpolazione lineare tra i campioni di controllo a 0, 6 e 12 ore. La lista dei geni è stata filtrata per contenere solo quei geni che: (1) avevano almeno un punto di tempo con un fold-change assoluto maggiore di 2 (up- o down-regulated) rispetto a 0 hr e (2) avevano almeno un punto di tempo con un conteggio normalizzato di lettura superiore a 100. Ciò ha generato una lista di 3529 geni di interesse, che sono stati poi raggruppati gerarchicamente per i loro valori di espressione z-scored, utilizzando il Cluster 3.0 (de Hoon et al., 2004) con le seguenti opzioni: correlazione centrata come il collegamento metrico di similitudine e collegamento medio come metodo di raggruppamento. L’albero gerarchico risultante è stato visualizzato utilizzando Java TreeView (Saldanha, 2004) per identificare i cluster minimi di interesse.
Analisi del motivo
Per identificare i motivi arricchiti all’interno dei cluster di geni, sono state estratte le sequenze a monte di 2000 bp per tutti i geni utilizzando il pacchetto PWMEnrich Bioconductor. L’arricchimento dei motivi del set di sequenze di ogni cluster è stato eseguito utilizzando AME (Bailey et al., 2015) con il database HOCOMOCOv10 (Kulakovskiy et al., 2016) in background (sequenze a monte di tutti i geni).
Analisi dell’arricchimento genico
Abbiamo valutato l’arricchimento dei geni in ciascuno dei gruppi identificati nella Figura 4A per i set di geni marcatori precedentemente definiti da endoderma, mesoderma ed ectoderma/epiblasto isolati da embrioni di topo E7.5 (Lu et al., 2018) utilizzando il pacchetto GOseq Bioconductor (Young et al., 2010). I geni del topo sono stati mappati su ortologi umani utilizzando i dati scaricati dal database Mouse Genome Informatics (http://www.informatics.jax.org/downloads/reports/HOM_MouseHumanSequence.rpt).
Disponibilità di dati e software
I dati di sequenziamento dell’RNA relativi alla Figura 4 e discussi in questa pubblicazione sono stati depositati nel Gene Expression Omnibus della NCBI (Edgar et al., 2002) e sono accessibili attraverso il numero di adesione alla serie GEO GSE111717 (https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE111717). Il software di analisi delle immagini StemCellTracker (commit n. 1c31fdd) è disponibile tramite GitHub (Kirst, 2014; copia archiviata all’indirizzo https://github.com/elifesciences-publications/StemCellTracker).
References
- Alexandre C, Baena-Lopez A, Vincent JP. Patterning and growth control by membrane-tethered Wingless. Nature. 2014; 505:180-185. DOI | PubMed
- Arnold SJ, Robertson EJ. Making a commitment: cell lineage allocation and axis patterning in the early mouse embryo. Nature Reviews Molecular Cell Biology. 2009; 10:91-103. DOI | PubMed
- Bailey TL, Johnson J, Grant CE, Noble WS. The MEME suite. Nucleic Acids Research. 2015; 43:W39-W49. DOI | PubMed
- Beers J, Gulbranson DR, George N, Siniscalchi LI, Jones J, Thomson JA, Chen G. Passaging and colony expansion of human pluripotent stem cells by enzyme-free dissociation in chemically defined culture conditions. Nature Protocols. 2012; 7:2029-2040. DOI | PubMed
- Blythe SA, Cha SW, Tadjuidje E, Heasman J, Klein PS. beta-Catenin primes organizer gene expression by recruiting a histone H3 arginine 8 methyltransferase, Prmt2. Developmental Cell. 2010; 19:220-231. DOI | PubMed
- Bolce ME, Hemmati-Brivanlou A, Kushner PD, Harland RM. Ventral ectoderm of xenopus forms neural tissue, including hindbrain, in response to activin. Development. 1992; 115:681-688. PubMed
- Brinkman EK, Chen T, Amendola M, van Steensel B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 2014; 42DOI | PubMed
- Chen G, Gulbranson DR, Hou Z, Bolin JM, Ruotti V, Probasco MD, Smuga-Otto K, Howden SE, Diol NR, Propson NE, Wagner R, Lee GO, Antosiewicz-Bourget J, Teng JMC, Thomson JA. Chemically defined conditions for human iPSC derivation and culture. Nature Methods. 2011; 8:424-429. DOI | PubMed
- Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013; 339:819-823. DOI | PubMed
- Conlon FL, Lyons KM, Takaesu N, Barth KS, Kispert A, Herrmann B, Robertson EJ. A primary requirement for nodal in the formation and maintenance of the primitive streak in the mouse. Development. 1994; 120:1919-1928. PubMed
- Cui W, Taub DD, Gardner K. qPrimerDepot: a primer database for quantitative real time PCR. Nucleic Acids Research. 2007; 35:D805-D809. DOI | PubMed
- D’Amour KA, Agulnick AD, Eliazer S, Kelly OG, Kroon E, Baetge EE. Efficient differentiation of human embryonic stem cells to definitive endoderm. Nature Biotechnology. 2005; 23:1534-1541. DOI | PubMed
- Datto MB, Frederick JP, Pan L, Borton AJ, Zhuang Y, Wang XF. Targeted disruption of Smad3 reveals an essential role in transforming growth factor beta-mediated signal transduction. Molecular and Cellular Biology. 1999; 19:2495-2504. DOI | PubMed
- de Hoon MJ, Imoto S, Nolan J, Miyano S. Open source clustering software. Bioinformatics. 2004; 20:1453-1454. DOI | PubMed
- Dunn NR, Vincent SD, Oxburgh L, Robertson EJ, Bikoff EK. Combinatorial activities of Smad2 and Smad3 regulate mesoderm formation and patterning in the mouse embryo. Development. 2004; 131:1717-1728. DOI | PubMed
- Edgar R, Domrachev M, Lash AE. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Research. 2002; 30:207-210. DOI | PubMed
- Etoc F, Metzger J, Ruzo A, Kirst C, Yoney A, Ozair MZ, Brivanlou AH, Siggia ED, Zeeshan Ozair M. A Balance between Secreted Inhibitors and Edge Sensing Controls Gastruloid Self-Organization. Developmental Cell. 2016; 39:302-315. DOI | PubMed
- François P, Siggia ED. A case study of evolutionary computation of biochemical adaptation. Physical Biology. 2008; 5DOI | PubMed
- Funa NS, Schachter KA, Lerdrup M, Ekberg J, Hess K, Dietrich N, Honoré C, Hansen K, Semb H. β-Catenin Regulates Primitive Streak Induction through Collaborative Interactions with SMAD2/SMAD3 and OCT4. Cell Stem Cell. 2015; 16:639-652. DOI | PubMed
- Green JB, Smith JC. Graded changes in dose of a Xenopus activin A homologue elicit stepwise transitions in embryonic cell fate. Nature. 1990; 347:391-394. DOI | PubMed
- Harland R, Gerhart J. Formation and function of Spemann’s organizer. Annual Review of Cell and Developmental Biology. 1997; 13:611-667. DOI | PubMed
- Heemskerk I, Burt K, Miller M, Chabra S, Cecilia Guerra M, Warmflash A. Morphogen dynamics control patterning in a stem cell model of the human embryo. bioRxiv. 2017. DOI
- Hemmati-Brivanlou A, Melton DA. A truncated activin receptor inhibits mesoderm induction and formation of axial structures in xenopus embryos. Nature. 1992; 359:609-614. DOI | PubMed
- James D, Levine AJ, Besser D, Hemmati-Brivanlou A. TGFbeta/activin/nodal signaling is necessary for the maintenance of pluripotency in human embryonic stem cells. Development. 2005; 132:1273-1282. DOI | PubMed
- Kirst C. GitHub. 2014. Publisher Full Text
- Kulakovskiy IV, Vorontsov IE, Yevshin IS, Soboleva AV, Kasianov AS, Ashoor H, Ba-Alawi W, Bajic VB, Medvedeva YA, Kolpakov FA, Makeev VJ. HOCOMOCO: expansion and enhancement of the collection of transcription factor binding sites models. Nucleic Acids Research. 2016; 44:D116-D125. DOI | PubMed
- Lacoste A, Berenshteyn F, Brivanlou AH. An efficient and reversible transposable system for gene delivery and lineage-specific differentiation in human embryonic stem cells. Cell Stem Cell. 2009; 5:332-342. DOI | PubMed
- Larabell CA, Torres M, Rowning BA, Yost C, Miller JR, Wu M, Kimelman D, Moon RT. Establishment of the dorso-ventral axis in Xenopus embryos is presaged by early asymmetries in beta-catenin that are modulated by the Wnt signaling pathway. The Journal of Cell Biology. 1997; 136:1123-1136. DOI | PubMed
- Loh KM, Ang LT, Zhang J, Kumar V, Ang J, Auyeong JQ, Lee KL, Choo SH, Lim CY, Nichane M, Tan J, Noghabi MS, Azzola L, Ng ES, Durruthy-Durruthy J, Sebastiano V, Poellinger L, Elefanty AG, Stanley EG, Chen Q, Prabhakar S, Weissman IL, Lim B. Efficient endoderm induction from human pluripotent stem cells by logically directing signals controlling lineage bifurcations. Cell Stem Cell. 2014; 14:237-252. DOI | PubMed
- Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 2014; 15DOI
- Lu X, Zhao ZA, Wang X, Zhang X, Zhai Y, Deng W, Yi Z, Li L. Whole-transcriptome splicing profiling of E7.5 mouse primary germ layers reveals frequent alternative promoter usage during mouse early embryogenesis. Biology Open. 2018; 7DOI | PubMed
- Martyn I, Kanno TY, Ruzo A, Siggia ED, Brivanlou AH. Self-organization of a human organizer by combined Wnt and Nodal signalling. Nature. 2018; 558:132-135. DOI | PubMed
- McDowell N, Gurdon JB. Activin as a morphogen in Xenopus mesoderm induction. Seminars in Cell & Developmental Biology. 1999; 10:311-317. DOI | PubMed
- McLean AB, D’Amour KA, Jones KL, Krishnamoorthy M, Kulik MJ, Reynolds DM, Sheppard AM, Liu H, Xu Y, Baetge EE, Dalton S, Kevin A. Activin a efficiently specifies definitive endoderm from human embryonic stem cells only when phosphatidylinositol 3-kinase signaling is suppressed. Stem Cells. 2007; 25:29-38. DOI | PubMed
- Mendjan S, Mascetti VL, Ortmann D, Ortiz M, Karjosukarso DW, Ng Y, Moreau T, Pedersen RA. NANOG and CDX2 pattern distinct subtypes of human mesoderm during exit from pluripotency. Cell Stem Cell. 2014; 15:310-325. DOI | PubMed
- Nemashkalo A, Ruzo A, Heemskerk I, Warmflash A. Morphogen and community effects determine cell fates in response to BMP4 signaling in human embryonic stem cells. Development. 2017; 144:3042-3053. DOI | PubMed
- Nieuwkoop PD. The formation of the mesoderm in Urodelean amphibians : ii. the origin of the dorso-ventral polarity of the mesoderm. Wilhelm Roux’ Archiv Fur Entwicklungsmechanik Der Organismen. 1969; 163:298-315. DOI | PubMed
- Robertson EJ. Dose-dependent Nodal/Smad signals pattern the early mouse embryo. Seminars in Cell & Developmental Biology. 2014; 32:73-79. DOI | PubMed
- Rowning BA, Wells J, Wu M, Gerhart JC, Moon RT, Larabell CA. Microtubule-mediated transport of organelles and localization of beta-catenin to the future dorsal side of Xenopus eggs. PNAS. 1997; 94:1224-1229. DOI | PubMed
- Sakaki-Yumoto M, Liu J, Ramalho-Santos M, Yoshida N, Derynck R. Smad2 is essential for maintenance of the human and mouse primed pluripotent stem cell state. Journal of Biological Chemistry. 2013; 288:18546-18560. DOI | PubMed
- Saldanha AJ. Java Treeview–extensible visualization of microarray data. Bioinformatics. 2004; 20:3246-3248. DOI | PubMed
- Sato N, Sanjuan IM, Heke M, Uchida M, Naef F, Brivanlou AH. Molecular signature of human embryonic stem cells and its comparison with the mouse. Developmental Biology. 2003; 260:404-413. DOI | PubMed
- Schmierer B, Hill CS. Kinetic analysis of Smad nucleocytoplasmic shuttling reveals a mechanism for transforming growth factor beta-dependent nuclear accumulation of Smads. Molecular and Cellular Biology. 2005; 25:9845-9858. DOI | PubMed
- Schneider S, Steinbeisser H, Warga RM, Hausen P. Beta-catenin translocation into nuclei demarcates the dorsalizing centers in frog and fish embryos. Mechanisms of Development. 1996; 57:191-198. DOI | PubMed
- Shimizu K, Gurdon JB. A quantitative analysis of signal transduction from activin receptor to nucleus and its relevance to morphogen gradient interpretation. PNAS. 1999; 96:6791-6796. DOI | PubMed
- Singh AM, Reynolds D, Cliff T, Ohtsuka S, Mattheyses AL, Sun Y, Menendez L, Kulik M, Dalton S. Signaling network crosstalk in human pluripotent cells: a Smad2/3-regulated switch that controls the balance between self-renewal and differentiation. Cell Stem Cell. 2012; 10:312-326. DOI | PubMed
- Smith JC. Forming and interpreting gradients in the early xenopus embryo. Cold Spring Harbor Perspectives in Biology. 2009; 1DOI | PubMed
- Sokol S, Melton DA. Pre-existent pattern in xenopus animal pole cells revealed by induction with activin. Nature. 1991; 351:409-411. DOI | PubMed
- Sorre B, Warmflash A, Brivanlou AH, Siggia ED. Encoding of temporal signals by the TGF-β pathway and implications for embryonic patterning. Developmental Cell. 2014; 30:334-342. DOI | PubMed
- Tam PP, Behringer RR. Mouse gastrulation: the formation of a mammalian body plan. Mechanisms of Development. 1997; 68:3-25. DOI | PubMed
- Tam PPL, Loebel DAF. Gene function in mouse embryogenesis: get set for gastrulation. Nature Reviews Genetics. 2007; 8:368-381. DOI
- Teo AK, Arnold SJ, Trotter MW, Brown S, Ang LT, Chng Z, Robertson EJ, Dunn NR, Vallier L, TeoKeong AK, Ray Dunn N. Pluripotency factors regulate definitive endoderm specification through eomesodermin. Genes & Development. 2011; 25:238-250. DOI | PubMed
- Vallier L, Alexander M, Pedersen RA. Activin/Nodal and FGF pathways cooperate to maintain pluripotency of human embryonic stem cells. Journal of Cell Science. 2005; 118:4495-4509. DOI | PubMed
- Vincent SD, Dunn NR, Hayashi S, Norris DP, Robertson EJ. Cell fate decisions within the mouse organizer are governed by graded Nodal signals. Genes & Development. 2003; 17:1646-1662. DOI | PubMed
- Voets O, Tielen F, Elstak E, Benschop J, Grimbergen M, Stallen J, Janssen R, van Marle A, Essrich C. Highly efficient gene inactivation by adenoviral CRISPR/Cas9 in human primary cells. PloS One. 2017; 12DOI | PubMed
- Warmflash A, Zhang Q, Sorre B, Vonica A, Siggia ED, Brivanlou AH. Dynamics of TGF-β signaling reveal adaptive and pulsatile behaviors reflected in the nuclear localization of transcription factor Smad4. PNAS. 2012; 109:E1947-E1956. DOI | PubMed
- Warmflash A, Sorre B, Etoc F, Siggia ED, Brivanlou AH. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nature Methods. 2014; 11:847-854. DOI | PubMed
- Wilson PA, Melton DA. Mesodermal patterning by an inducer gradient depends on secondary cell-cell communication. Current Biology. 1994; 4:676-686. DOI | PubMed
- Xu RH, Sampsell-Barron TL, Gu F, Root S, Peck RM, Pan G, Yu J, Antosiewicz-Bourget J, Tian S, Stewart R, Thomson JA. NANOG is a direct target of TGFbeta/activin-mediated SMAD signaling in human ESCs. Cell Stem Cell. 2008; 3:196-206. DOI | PubMed
- Yang X, Letterio JJ, Lechleider RJ, Chen L, Hayman R, Gu H, Roberts AB, Deng C. Targeted disruption of SMAD3 results in impaired mucosal immunity and diminished T cell responsiveness to TGF-beta. The EMBO Journal. 1999; 18:1280-1291. DOI | PubMed
- Ye J, Coulouris G, Zaretskaya I, Cutcutache I, Rozen S, Madden TL. Primer-BLAST: a tool to design target-specific primers for polymerase chain reaction. BMC Bioinformatics. 2012; 13DOI | PubMed
- Young MD, Wakefield MJ, Smyth GK, Oshlack A. Gene ontology analysis for RNA-seq: accounting for selection bias. Genome Biology. 2010; 11DOI | PubMed
- Zhu Y, Richardson JA, Parada LF, Graff JM. Smad3 mutant mice develop metastatic colorectal Cancer. Cell. 1998; 94:703-714. DOI | PubMed
Fonte
Yoney A, Etoc F, Ruzo A, Carroll T, Metzger JJ, et al. () WNT signaling memory is required for ACTIVIN to function as a morphogen in human gastruloids. eLife 7e38279. https://doi.org/10.7554/eLife.38279





