Introduzione
L’introduzione nel flusso sanguigno di Candida spp. o di altri organismi fungini può essere il risultato dell’introduzione di Candida spp. o di altri organismi fungini, con il potenziale di diventare una malattia debilitante sia per la comunità che per la popolazione ospedaliera. Rispetto alle infezioni localizzate, le infezioni fungine sistemiche presentano spesso una maggiore mortalità dei pazienti, un allungamento dei ricoveri ospedalieri e costi sanitari più elevati (Zaoutis et al., 2010).
Poiché le infezioni batteriche sono più frequenti delle infezioni fungine, il trattamento antimicrobico empirico è spesso diretto a questi microbi, e di solito comprende almeno un antimicrobico mirato contro lo Staphylococcus aureus, insieme ad almeno un antibiotico contro i batteri gram-negativi (Micek et al., 2005). Prima dei risultati positivi delle emocolture fungine, gli antifungini vengono aggiunti al regime solo a discrezione del medico e di solito sono raccomandati solo per i pazienti ad alto rischio (Morrell et al., 2005). Un’analisi retrospettiva di coorte condotta da Morrell et al. nel 2005 ha rilevato che i pazienti che hanno ricevuto un trattamento antimicotico entro le prime 12 ore di infezione avevano un rischio di mortalità ospedaliera inferiore rispetto ai pazienti a cui è stato somministrato il trattamento dopo le 12 ore (Morrell et al., 2005), dimostrando così la necessità di una diagnosi rapida.
L’infezione fungina più importante è quella provocata da Candida albicans. Tuttavia, anche un numero maggiore di infezioni del flusso sanguigno non correlate agli albicani è problematico. Negli Stati Uniti, le specie di Candida si collocano tra i primi cinque patogeni opportunistici per le infezioni nosocomiali, con C. albicans, Candida glabrata, Candida parapsilosis, Candida tropicalis e Candida krusei responsabili del 95% di tutte le infezioni che si verificano (Wisplinghoff et al., 2004; Pappas, 2006; Pfaller e Diekema, 2007; Sievert et al., 2013; Yapar, 2014). Le specie non-albicane sono spesso riscontrate in pazienti con tumori o tumori maligni ematologici e mostrano un livello di virulenza che si traduce in una significativa mortalità (Krcmery e Barnes, 2002).
Sono stati proposti numerosi sistemi per rilevare gli agenti patogeni microbici dal sangue per migliorare il processo diagnostico delle infezioni sistemiche. I test convenzionali includono metodi manuali e semiautomatizzati basati sulla caratterizzazione morfologica e fisiologica (Goodwin et al., 1992). L’analisi delle emocolture seguita da test di suscettibilità antimicrobica è l’approccio più comune per l’identificazione di batteriemie e funghi sistemici (Pardo et al., 2014). Tuttavia, molte preoccupazioni hanno evidenziato limiti nella sensibilità, affidabilità e tempestività dei metodi di emocoltura automatizzati. Mentre le tecnologie più recenti e i kit disponibili in commercio sono stati sviluppati per rispondere meglio alla sfida di una diagnosi precoce, l’emocoltura rimane lo standard e i metodi di rilevamento più frequentemente utilizzati (Morris et al., 1995; Vitale e Nucci, 2014).
Un test del sistema di emocoltura automatizzato BacT/Alert, l’attuale dispositivo gold standard per la diagnostica dei funghi, ha rilevato che la crescita della Candida è stata rilevata nel 74% (479/648) dei flaconi di coltura dei semi, indicando che le infezioni possono non essere rilevate in base alla crescita (Horvath et al., 2007). La sensibilità del test diminuisce con una bassa concentrazione di inoculo [1.000 cellule di lievito in bottiglia (79%); 10 cellule di lievito in bottiglia (70%)], con un tempo medio di rilevamento di C. albicans a 20-30 h, a seconda del tipo di bottiglia e della concentrazione dell’inoculo (Horvath et al., 2007).
In questo rapporto utilizziamo gli elementi morfologici differenziali delle cellule fungine per separare le cellule di Candida dal sangue lisato utilizzando microfluidici. Il processo di isolamento comporta la lisi dei globuli rossi (RBC) per poi far scorrere il fluido attraverso un dispositivo microfluidico inerziale per separare le cellule fungine dai globuli bianchi (WBC), generando un campione di fungo concentrato.
Abbiamo applicato le forze inerziali dinamiche descritte attraverso la microfluidica per separare le cellule fungine, in particolare le specie di Candida, dal sangue lisato, generando un campionamento concentrato che contiene molti degli agenti contaminanti. In questo rapporto, presentiamo i nostri risultati e descriviamo l’efficacia del processo di separazione e di recupero delle cellule fungine. Questa tecnica ha il potenziale per essere un prezioso complemento ai sistemi diagnostici automatizzati, mitigando le esigenze di precedenti metodi di coltura o centrifugazione.
Materiali e metodi
Gli stampi master sono stati prodotti utilizzando la fotolitografia SU-8
Gli stampi sono stati creati seguendo il protocollo descritto da Martel e Toner (2012). Per creare lo stampo master, un wafer di silicio è stato disidratato a 200°C per 20 minuti prima di essere esposto ad un plasma ad ossigeno ad alta potenza per promuovere l’adesione. Il fotoresist negativo a base epossidica SU8-50 (Microchem Corp, Westborough, Massachusetts) è stato filato a 2.650 rpm per raggiungere uno spessore di 50 μm. Due periodi di cottura si sono verificati a 65°C per 10 min e 100°C per 20 min, rispettivamente. Dopo il raffreddamento, SU8-50 è stato esposto alla luce UV attraverso una fotomaschera stampata in emulsione mylar con caratteristiche di canale progettate per dividere il flusso prima di altri due periodi di cottura a 65°C per 10 min e 100°C per 20 min. Il wafer master in silicio è stato sviluppato nel BTS-220 SU8-Developer (J.T. Baker, New Jersey) e misurato tramite profilometraggio per verificare le dimensioni delle caratteristiche in rilievo della SU8.
Realizzazione di microcanali PDMS
Una miscela 10:1 di Sylgard 184 a base di elastomero e indurente (Dow Corning, Midland, Michigan) è stata preparata e versata sopra il wafer di silicio e sottoposta ad essiccazione sotto vuoto per rimuovere eventuali bolle. Per curare il PDMS, lo stampo è stato incubato a 65 ° C per almeno 8 h. Il PDMS polimerizzato è stato poi tagliato lungo il bordo del wafer per rimuovere la lastra che conteneva il design microcanale ed è stato tagliato a dadini in singoli canali o in gruppi di due e le punte degli aghi affilati sono stati inseriti per stabilire le posizioni di entrambi l’ingresso e l’uscita. Per rimuovere il particolato rimanente, i dispositivi sono stati poi puliti con nastro a basso residuo e plasma a ossigeno e PDMS attivato legato a vetrini per microscopio di vetro di 1 mm di spessore. I dispositivi incollati sono stati cotti a 75°C per 10 minuti e i tubi sono stati pressati e incollati alle posizioni di ingresso e di uscita utilizzando Loctite Medical Grade Epoxy 4013 con la considerazione di mantenere la capacità di flusso. Il dispositivo sviluppato è stato designato come focheggiatore di funghi inerziale (IFF).
Ceppi e condizioni di coltura
I ceppi di riferimento di funghi utilizzati negli studi descritti sono elencati nella Tabella 1. I ceppi sono stati conservati a -80°C fino al momento del bisogno. I funghi sono stati coltivati in estratto di lievito, peptone, e destrosio (YPD) media con agitazione a 30 ° C, salvo diversa indicazione.
| Funghi | Ceppo |
|---|---|
| Candida albicans | SC5314 (CAN14) |
| Candida glabrata | ATCC 90030 |
| Candida parapsilosis | ATCC 22019 |
| Candida tropicalis | ATCC 13803 |
Colorazione della cella
Le cellule fungine sono state colorate con isotiocianato di fluoresceina (FITC) per osservare il movimento mentre passavano attraverso l’IFF. Una coltura notturna di cellule di C. albicans ceppo CAN14 è stato lavato due volte con tampone fosfato salino (PBS) e raccolti con la centrifugazione. Le cellule sono state contate con un emocitometro e sospese ad una concentrazione di 106 cellule/mL. FITC (10 mg/mL in DMSO) è stato aggiunto per ottenere una concentrazione finale di 0,1 mg/mL in PBS. Le cellule sono state incubate per 30 minuti a temperatura ambiente al buio. Successivamente, le cellule sono state lavate tre volte in PBS contenente 10 mg/mL di sieroalbumina bovina (BSA). Le cellule sono state poi sottoposte a messa a fuoco con l’IFF.
I WBC sono stati anche colorati per documentare meglio il loro movimento attraverso l’IFF. WBC isolati da sangue intero con una procedura standard di lisi del cloruro di ammonio sono stati colorati utilizzando 1M Calcein AM rosso/arancione (ThermoFisher Scientific, Waltham, Massachusetts) soluzione di riserva ad un rapporto di 1 uL di macchia per ogni 1 mL di sospensione cellulare, poi mescolati costantemente per 15 min. La sospensione è stata lavata con centrifugazione a 50 g per 5 min con 1X PBS contenente il 2% di siero bovino fetale (FBS) e risospesa nello stesso tampone alla concentrazione delle cellule operatorie in base alla misurazione del CBC del sangue intero (1 × 106 cellule/mL).
Migrazione di cellule fungine sospese in PBS attraverso il dispositivo IFF
Una coltura di C. albicans ceppo di riferimento CAN14 è stata coltivata durante la notte con agitazione a 30°C. Le cellule sono state raccolte e lavate con PBS e macchiate con FITC come descritto sopra. Le cellule sono state poi enumerate con l’aiuto di un emocitometro e sospese ad una concentrazione di 1.600 cellule / ml in PBS. Un’aliquota di 20 mL di cellule sono state filtrate (filtri Millex-SV 5,0 μm, ThermoFisher Scientific, Waltham, Massachusetts) e preparate per i dispositivi IFF. I dispositivi sono stati innescati con 5 mL di soluzione di sodio dodecilsolfato di sodio (SDS) allo 0,01% ad una portata di 400 μL/min utilizzando una pompa a siringa Harvard Apparatus prima di 5 mL di cellule sospese in PBS sono stati fatti passare attraverso i dispositivi. Materiale isolato da ciascuna delle tre porte di uscita è stato raccolto e 100 μL da ciascuna porta è stato placcato su YPD contenente 45 μg/mL kanamicina, 100 μg/mL ampicillina, e 100 μg/mL streptomicina. Le piastre YPD sono state incubate a 30°C per 2 giorni prima di contare le unità di formazione delle colonie (CFU).
Lisi di RBC
La soluzione di lisi 10X RBC (Miltenyi Biotec, Auburn, California) è stata diluita a 1X con ddH2O. Diluizioni seriali di cellule CAN14 esposte a 1X RBC lisi soluzione di lisi tra 0 e 24 h è stato avvistato su supporti YPD e cresciuto a 30 ° C per 2 giorni per osservare la crescita.
Migrazione delle cellule fungine attraverso il dispositivo IFF utilizzando sangue lisato
Una coltura di cellule di Candida è stata coltivata durante la notte in YPD con agitazione a 30°C. Le cellule sono state raccolte attraverso la centrifugazione e lavate due volte con PBS. Sono stati poi enumerati con un emocitometro e sospeso ad una concentrazione di 1.600 cellule / ml nel sangue intero. Il campione di sangue con 5 mL di campione di sangue a punta è stato aggiunto a 45 mL di tampone di lisi 1X Red Blood Cell (Miltenyi Biotech, Inc., Auburn, CA). Per rimuovere i detriti creatisi durante l’emolisi, la soluzione del tampone di lisi è stata fatta passare attraverso un filtro da 5,0 μm (Millex, Millipore Sigma, St. Louis, Missouri) per rimuovere le particelle grossolane che potrebbero ostruire l’IFF. I dispositivi sono stati innescati con 5 mL di 0,01% SDS ad una portata di 400 μL/min utilizzando una pompa a siringa Harvard Apparatus prima che i campioni di sangue sono stati testati. La soluzione tampone di sangue è stata poi fatta passare attraverso l’IFF ad una velocità di flusso di 400 μL/min, raccogliendo il materiale in tre porte di uscita progettate. Per determinare la direzione delle cellule fungine, 100 μL da ogni porta è stato placcato su YPD contenente 45 μg/mL kanamicina, 100 μg/mL ampicillina, e 100 μg/mL streptomicina. Le piastre sono state incubate a 30°C per 2 giorni prima di contare le CFU.
Identificazione di C. albicans isolati
Per determinare se il DNA fungino può essere amplificato dal sangue presentato attraverso il dispositivo IFF senza estrazione preventiva di DNA fungino, è stata effettuata la PCR per analizzare campioni di materiale trattato con IFF utilizzando il kit Hemo Klen Taq (New England Biolabs, Ipswich Massachusetts). Per la miscela di reazione da 20 μL, 1 μL di campione trattato con IFF è stato miscelato con 19 μL di master mix contenente 1X PCR buffer, 62,5 μM dNTPs, 0,375 μM di AF114470 in avanti (5′-GGGGAGGTAGTGACAATAAATAAC-3′) e indietro (5′- CGTCCCTATTAATCATTACGAT-3′) primer (Jaeger et al., 2000), Hemo Klen taq polimerasi, e acqua sterile. I parametri del termociclatore comprendono il riscaldamento dei campioni a 95°C per 3 minuti, seguito da 35 cicli di 95°C per 20 s, 50°C per 2 minuti e 68°C per 2 minuti. Un’estensione finale a 68°C è stata applicata per 10 min.
Il prodotto PCR amplificato è stato clonato in un vettore pGEM T-easy (Promega, Madison, WI). Il plasmide generato contenente l’inserto è stato sequenziato per confermare l’rRNA 18S (Genewiz, South Plainfield, NJ).
Risultati
La manipolazione delle cellule utilizzando forze inerziali microfluidiche permette di concentrare diversi tipi di cellule in posizioni di equilibrio fluidico uniche che sono determinate dalle caratteristiche biofisiche dei diversi tipi di cellule. Soprattutto, la dimensione e la forma delle cellule influenzano il movimento in un canale microfluidico; le posizioni di equilibrio sono ulteriormente influenzate dalla curvatura del canale e dalla velocità del fluido. Le cellule fungine nella forma planctonica differiscono per le dimensioni tra le specie, ma sono, in media, più piccole dei WBC che vanno da 7 a 25 μm tra i vari tipi di leucociti. La maggior parte dei WBCs sono apportati da neutrofili che comprendono dal 60 al 75% dei leucociti all’interno del sangue e in media sono 12-15 μm. Le cellule di Candida hanno un diametro massimo compreso tra 2 e 11 micron. Le cellule di Candida planctoniche sono ellissoidali o sferiche, compatibili con i metodi automatizzati per isolare i funghi dalle altre cellule presenti nel sangue.
Design del dispositivo microfluidico
Per il canale di focalizzazione inerziale a spirale è stato utilizzato un modello di progettazione del dispositivo basato sulle dimensioni dei globuli bianchi, che è stato poi modificato per incorporare una sezione di filtro più grande (Martel e Toner, 2012). Il design dell’uscita è stato adattato ai risultati dei test iniziali che mostravano le posizioni di focalizzazione dei globuli bianchi e delle cellule fungine a diverse velocità di flusso. Il progetto finale è mostrato in Figura 1A. Il canale è largo 100 μm, alto 50 μm e lungo ~ 4 cm. I fondamenti della messa a fuoco inerziale sono descritti altrove, ma brevemente, a causa delle elevate velocità del fluido nelle forze idrodinamiche microcanale idrodinamiche sulle cellule causare loro di muoversi in specifiche sezioni trasversali posizioni nel canale dipendente dal design del canale, i parametri di flusso, e le caratteristiche della cella.
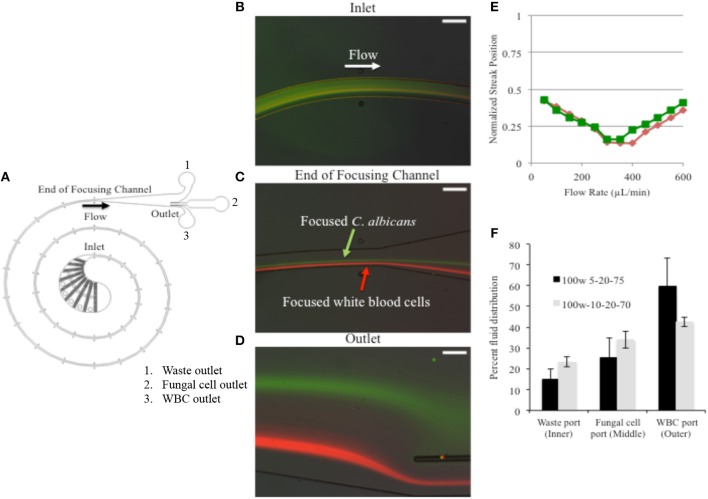
Figura 1.Le cellule fungine sono state colorate con FITC per differenziarsi dai WBC colorati con calceina. (A) Schema del dispositivo. (B) Una miscela di cellule che comprende WBCs colorati e cellule fungine entra nel IFF senza separazione preliminare e sono sottoposti alle forze microfluidiche e la portata specificata. (C) Mentre le cellule viaggiano attraverso il percorso della bobina, ad una specifica portata di 400 μL/min, C. albicans sono focalizzati in un flusso. (D) Nel momento in cui raggiungono l’uscita, la cella forma due flussi distinti. (E) La separazione laterale dei tipi di cellule è stata testata su un intervallo per rilevare la portata ottimale applicata. (F) La distribuzione osservata al dispositivo 100w-5-20-75 è mostrato in nero e la distribuzione per la progettazione del dispositivo IFF 100w-10-20-70 è mostrato in grigio. Le barre di errore indicano la deviazione standard.

Figura 1.Le cellule fungine sono state colorate con FITC per differenziarsi dai WBC colorati con calceina. (A) Schema del dispositivo. (B) Una miscela di cellule che include WBC colorati e cellule fungine entra nell’IFF senza separazione preliminare e sono sottoposti alle forze microfluidiche e alla portata specificata. (C) Mentre le cellule viaggiano attraverso il percorso della bobina, ad una specifica portata di 400 μL/min, C. albicans sono focalizzati in un flusso. (D) Nel momento in cui raggiungono l’uscita, la cella forma due flussi distinti. (E) La separazione laterale dei tipi di cellule è stata testata su un intervallo per rilevare la portata ottimale applicata. (F) La distribuzione osservata al dispositivo 100w-5-20-75 è mostrato in nero e la distribuzione per la progettazione del dispositivo IFF 100w-10-20-70 è mostrato in grigio. Le barre di errore indicano la deviazione standard.
Portata
Siamo stati in grado di identificare 400 μL/min come la portata ottimale per separare le cellule fungine dai WBC in sospensione PBS. Le cellule fungine sono state colorate con FITC, consentendo l’osservazione in tempo reale durante il movimento nel dispositivo IFF. Le cellule fungine sono state osservate per entrare nel dispositivo con una distribuzione casuale, e poi focalizzate in una posizione di equilibrio all’interno del canale durante il flusso (Figure 1B-D). A questa stessa portata, le cellule fungine colorate FITC sono state trovate a separarsi dal WBC (colorate con calceina arancione) per formare flussi separati e focalizzati lungo il canale (Figure 1C,D), mostrando così che C. albicans potrebbe essere focalizzato e separato dal WBC. La portata è stata testata in un intervallo compreso tra 50 e 600 μL/min. Utilizzando immagini fluorescenti per individuare i flussi di WBC e cellule fungine la distanza di separazione laterale è stato trovato per essere massimizzato ad una portata di 400 μL / min. La distanza di separazione è evidenziata nella Figura 1E, dove le posizioni dei picchi di fluorescenza sono tracciate rispetto alla portata.
C. le cellule albicane sono concentrate con il passaggio attraverso l’IFF
Abbiamo testato due diversi design di uscita a microcanali PDMS per determinare il miglior mezzo di distribuzione del fluido con l’obiettivo di ridurre il volume finale di sospensione delle cellule fungine dopo l’elaborazione IFF, concentrando così il materiale fungino. I due design dei dispositivi si distinguono per le relative resistenze idrauliche delle tre uscite destinate a suddividere il flusso nelle diverse porte di raccolta. Il dispositivo 100w-5-20-75 è stato progettato per dirigere il 5% del liquido verso un’uscita interna destinata ai rifiuti, il 20% del liquido verso un’uscita intermedia destinata alle celle fungine isolate e il 75% del liquido verso un’uscita esterna che dovrebbe ricevere i WBC. In alternativa, il dispositivo 100w-10-20-70 è stato progettato per dividere il 10, 20 e 70% del liquido nelle rispettive uscite. Pertanto, il volume finale diretto all’uscita intermedia (l’uscita di raccolta del fungo) sarebbe stato ridotto da un campione iniziale di 5 mL a ~1 mL.
Abbiamo trovato discrepanze tra i rapporti progettati e quelli osservati (Figura 1F). Circa il 40-60% del liquido è stato diretto all’uscita esterna per entrambi i tipi di dispositivi testati, mentre il 15-25% del liquido è stato diretto all’uscita interna. Tuttavia, la quantità di liquido diretto all’uscita centrale è rimasto tra il 25 e il 34% ± 4-9,1%, che corrisponde ai rapporti di progetto più da vicino e conferma che un volume iniziale di 5 mL è stato costantemente ridotto ad un volume finale di 1,3-1,7 mL all’uscita centrale nel tentativo di concentrare le cellule fungine. Dei due progetti, 100W-5-20-75 ha mostrato una maggiore riduzione del volume ed è stato favorito per un ulteriore esame.
Le specie di Candida non albicani sono concentrate con l’IFF
Abbiamo cercato di determinare in che misura il dispositivo IFF è in grado di indirizzare le cellule fungine verso il punto di raccolta appropriato (porta centrale) e di esaminare la conservazione del progetto testando diverse specie di non-albicani per determinare se l’IFF può essere utilizzato per le cellule fungine più in generale. Nel valutare la capacità di separazione di diverse specie di Candida, ci si aspettava che la distribuzione delle unità di formazione delle colonie presentasse una certa variabilità a causa delle differenze di dimensioni tra le specie. La tabella 2 fornisce la percentuale di CFU ottenuta da ogni porto nel corso di una serie di prove che interrogano 10 volte le diluizioni seriali di Candida non alcalina sottoposta a IFF. Iniziando 5 mL di campioni di PBS con cellule fungine a concentrazioni di 1.600, 160 e 16 cellule/mL, la maggior parte delle cellule fungine è stata indirizzata verso l’uscita intermedia appropriata. Per C. albicans, oltre l’80% delle cellule fungine recuperate nelle concentrazioni di 8.000, 800 e 80 cellule per 5 mL sono state indirizzate correttamente. Tuttavia, il numero di CFU indica che c’è stata una perdita di cellule fungine per tutta la durata dello studio. Una parte delle cellule di tutte le specie di Candida testate (C. albicans, C. glabrata, C. parapsilosis e C. tropicalis) è stata effettivamente diretta al porto di raccolta dei funghi. Tra le cellule recuperate, C. albicans è stata indirizzata nel modo più efficiente verso il luogo di raccolta appropriato. Utilizzando lo stesso design IFF, la direzione verso il porto centrale di C. parapsilosis è scesa tra il 69 e il 74% tra le cellule catturate, dimostrando di essere la seconda più adatta alla procedura di separazione.
| Esterno | Medio | Interno | ||
|---|---|---|---|---|
| 1.600 cellule/ml | C. albicans | 8.3 ± 8.7 | 90.0 ± 8.0 | 1.67 ± 2.0 |
| C. glabrata | 25.4 ± 4.7 | 61.7 ± 6.0 | 12.9 ± 1.4 | |
| C. parapsilosi | 5.3 ± 4.8 | 73.6 ± 4.6 | 21.1 ± 0.5 | |
| C. tropicalis | 41.4 ± 1.6 | 52.9 ± 1.6 | 5.70 ± 3.2 | |
| 160 cellule/ml | C. albicans | 11.2 ± 22.4 | 85.6 ± 21.2 | 3.2 ± 6.3 |
| C. glabrata | 34.7 ± 7.8 | 55.9 ± 6.6 | 9.4 ± 2.6 | |
| C. parapsilosi | 5.7 ± 9.9 | 69.7 ± 10.7 | 24.6 ± 15.9 | |
| C. tropicalis | 33.2 ± 13.6 | 62.6 ± 19.6 | 4.2 ± 6.0 | |
| 16 celle/ml | C. albicans | 2.1 ± 4.2 | 91.2 ± 10.3 | 6.8 ± 8.9 |
| C. glabrata | 13.3 ± 5.4 | 67.6 ± 21.9 | 19.0 ± 16.5 | |
| C. parapsilosisi | 7.3 ± 8.5 | 74.1 ± 9.1 | 18.6 ± 9.2 | |
| C. tropicalis | 8.1 ± 11.4 | 85.7 ± 2.6 | 6.3 ± 8.8 |
Per C. glabrata, la percentuale di cellule che sono state dirette allo sbocco intermedio è inferiore, con solo il 55-70% di migrazione corretta delle cellule fungine. Quando testato con campioni di partenza a densità cellulare maggiore, una grande percentuale di cellule fungine C. glabrata flusso di cellule di C. glabrata verso l’uscita inferiore destinato a WBCs. Ciò può essere dovuto al fatto che C. glabrata è notevolmente più piccolo con una forma sferica rispetto alle altre specie di Candida e gli attuali aspetti progettuali potrebbero non essere ottimali per gradi di isolamento più elevati.
Oltre ad osservare la percentuale di distribuzione cellulare a queste concentrazioni, è stato valutato anche il numero di CFU recuperati da ciascun dispositivo. La Figura 2 mostra il numero corrispondente di CFU per ogni concentrazione durante le prove condotte in PBS. Il numero di cellule vitali per tutte le specie è stato notevolmente inferiore alla quantità di picco nel campione di partenza. Per le 8.000 cellule per 5 mL di densità, il 5-10% delle cellule di C. albicans sono state recuperate dalle diverse prove. Anche se si è osservata una variabilità nel numero di CFU tra le diverse densità cellulari, il recupero è sembrato migliorare con la diminuzione della densità del campione. Con un picco di concentrazione di 800 cellule ogni 5 mLs, è stato recuperato in media il 26,5% delle cellule fungine.
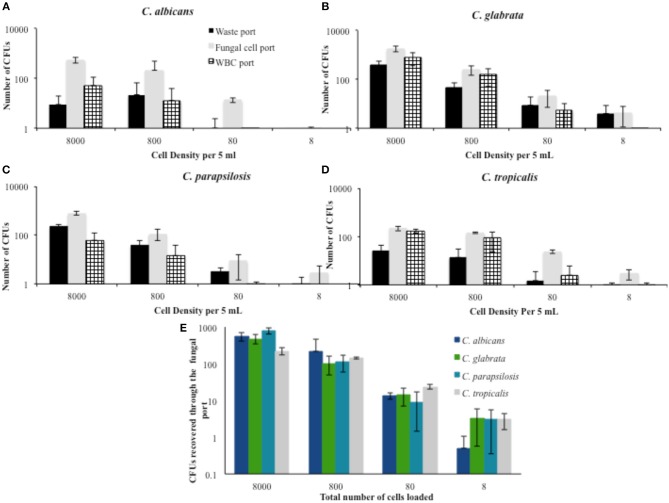
Figura 2.Figura 2. Distribuzione percentuale della migrazione delle cellule fungine a diverse densità cellulari in soluzione di PBS. La porzione di cellule fungine diretta verso il porto dei rifiuti, il porto delle cellule fungine e il porto WBC sono stati determinati per le specie fungine più comunemente incontrate. (A)C. albicans(B)C. glabrata(C)C. parapsilosis, e (D)C. tropicalis. (E) Un riassunto delle cellule recuperate dal porto delle cellule fungine è fornito come confronto tra le barre di errore Candida spp. testate ha indicato la deviazione standard.

Figura 2.Distribuzione percentuale della migrazione delle cellule fungine a diverse densità cellulari in soluzione di PBS. La porzione di cellule fungine diretta al porto di scarico, al porto delle cellule fungine e al porto WBC è stata determinata per le specie fungine più comunemente incontrate. (A)C. albicans(B)C. glabrata(C)C. parapsilosis, e (D)C. tropicalis. (E) Un riassunto delle cellule recuperate dal porto delle cellule fungine è fornito come confronto tra le barre di errore Candida spp. testate ha indicato la deviazione standard.
Rimozione di RBC
Per l’interrogatorio di cellule fungine del sangue puntinato nell’IFF, i globuli rossi devono prima essere impoveriti dalla soluzione. A tal fine, le cellule dei mammiferi devono essere lisate senza danneggiare le cellule fungine. Per realizzare la rimozione degli RBC sfruttiamo le diverse strutture cellulari tra i diversi tipi di cellule; i funghi hanno una parete cellulare che protegge da stress e danni, una struttura che serve come risorsa ambientale e che viene utilizzata nella nostra metodologia di elaborazione. C. albicans sono stati esposti alla soluzione di lisi 1X RBC per un massimo di 24 ore, diluiti e placcati su supporti di lievito per vedere se la soluzione di lisi ha ucciso i funghi o ritardato la crescita. Abbiamo scoperto che l’incubazione per 24 ore in soluzione di lisi 1X RBC lysis non ha avuto alcun effetto sulla crescita di C. albicans (Figura supplementare 1). Così, la soluzione di lisi 1X RBC lysis solution potrebbe essere utilizzata su emocolture contenenti cellule fungine per lisare gli RBC senza ridurre la popolazione di C. albicans all’interno del campione.
C. le cellule di C. albicans sono isolate dal sangue lisato con l’IFF
Una volta ottenuta la segregazione di C. albicans in soluzione PBS, sono state studiate le prestazioni del dispositivo IFF nella concentrazione delle cellule fungine del sangue. Campioni di sangue intero sono stati ottenuti al Rhode Island Hospital (dopo la revisione da parte del Rhode Island Institutional Review Board, dove lo studio è stato determinato per essere esentato) e sono stati trattati con anticoagulante, tenuti su un rotatore, e 5 mL aliquote di sangue sono stati corretti con C. albicans concentrazioni di 1.600 cellule / ml. La lisi di RBC è stata eseguita rapidamente al fine di prevenire la formazione di coaguli che influenzerebbero la velocità di flusso quando presentata attraverso il dispositivo IFF. Il sangue con 5 mL di funghi è stato diluito 10:1 (tampone di lisi RBC: sangue) e filtrato prima di essere sottoposto a IFF.
La figura 3A illustra il numero di unità che formano le colonie e la distribuzione dei funghi in ogni porta. Mentre il numero di unità di formazione delle colonie conferma che la maggior parte delle cellule sono state dirette verso lo sbocco intermedio, la percentuale di distribuzione è stata <50% (44,6% per il dispositivo 5-20-75 IFF), inferiore alla percentuale vista dalle sospensioni di C. albicans in PBS (Figura 3B). Il numero di cellule vitali che sono state recuperate dal campione era simile tra la porta di raccolta dei funghi e la porta di fondo destinata ai WBC. Tuttavia, il porto di raccolta dei rifiuti ha continuato ad avere il numero più basso di CFU. Abbiamo recuperato 265 ± 137 CFU tra gli 8.000 CFU inoculati nel porto centrale per il dispositivo 5-20-75 IFF. Tra tutti i porti, abbiamo raggiunto un tasso di recupero totale dell’8,4% delle cellule fungine che sono state inoculate nel sangue. La viscosità del sangue e la presenza di residui di RBC sono fattori potenziali che spiegano la differenza nell’efficienza di isolamento delle cellule fungine tra i campioni di sangue intero punteggiato e quelli di PBS che utilizzano il dispositivo 5-20-75 IFF.
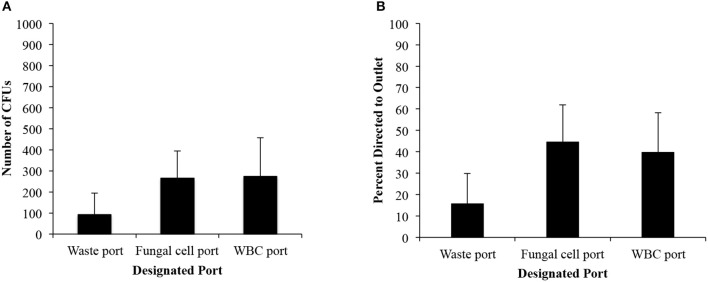
Figura 3.Dati IFF per C. albicans nel sangue lisato. (A) Distribuzione del numero grezzo di CFU. (B) La distribuzione percentuale di C. albicans CFU tra ogni porta da cellule di C. albicans da una concentrazione iniziale di cellule era di 8.000 cellule per 5 mL di campione. Le barre di errore indicavano una deviazione standard.

Figura 3.Dati IFF per C. albicans in sangue lisato. (A) Distribuzione del numero grezzo di CFU. (B) La distribuzione percentuale di C. albicans CFU tra ogni porta da cellule di C. albicans spuntate nel sangue intero da una concentrazione iniziale di cellule era di 8.000 cellule per 5 mL di campione. Le barre di errore indicavano una deviazione standard.
Conferma delle cellule recuperate
Per convalidare le capacità del dispositivo IFF, sono state adottate misure per garantire che le cellule isolate dal sangue erano effettivamente C. albicans e non da altri contaminanti che possono essere presenti nel sangue. Per verificare che la reazione di PCR identificasse correttamente la presenza di C. albicans dal fluido raccolto dalla porta di raccolta dei funghi del dispositivo IFF, i prodotti di PCR sono stati sequenziati. Un’aliquota del materiale raccolto è stata sottoposta a PCR direttamente e il prodotto amplificato (Figura supplementare 2) è stato sequenziato per l’identificazione. Questi risultati hanno suggerito che la Candida poteva essere rilevata dal sangue e dalle cellule fungine concentrate e sottoposta a ulteriori analisi di identificazione per aiutare a migliorare la diagnostica.
Discussione
La diagnosi precoce e l’inizio tempestivo di un’adeguata terapia antimicotica è fondamentale per affrontare la significativa morbilità e mortalità associata alle infezioni fungine invasive e soprattutto alla candidemia (Kourkoumpetis et al., 2012; Arvanitis et al., 2014). Qui presentiamo un mezzo per isolare e concentrare le cellule fungine dal fluido biologico. L’intenzione è quella di mitigare la necessità di una coltura, un passo che richiede tempo. Il dispositivo e la metodologia IFF presentati mostrano anche che le cellule possono essere concentrate in piccoli volumi, a complemento delle nuove tecnologie in via di sviluppo per la prossima generazione di dispositivi di diagnostica fungina che si concentrano su approcci molecolari.
La nostra piattaforma utilizza il fenomeno della focalizzazione inerziale per isolare i contaminanti fungini dai residui di sangue lisato. La focalizzazione inerziale si verifica al numero finito di Reynolds, quando tutti gli oggetti che passano attraverso i microcanali sperimentano sia l’interazione delle pareti, sia le forze di sollevamento del gradiente di taglio. L’assunto fondamentale dietro la forza di interazione della parete è che, a meno che una particella non sia abbastanza piccola da agire come una particella fluida, essa interagirà con le pareti di un microcanale causando la particella a muoversi leggermente più lentamente delle molecole di fluido così come un accumulo di pressione tra la parete e la particella. Questo genera una forza diretta lontano dalle pareti del canale. Questo diventa poi accoppiato alla forza di sollevamento del gradiente di taglio quando una particella sta sperimentando forze di taglio ineguali a causa della natura parabolica del profilo di velocità del fluido nel microcanale, generando una forza verso le pareti del canale dove questa sollecitazione ineguale è ridotta al minimo.
Si possono applicare forze aggiuntive per aiutare la separazione dei componenti all’interno del fluido utilizzando elementi di progettazione del canale. Ad esempio, i canali curvi favoriscono il movimento laterale attraverso l’introduzione di un flusso secondario e la conseguente forza di trascinamento perpendicolare al flusso principale, alterando così le posizioni di equilibrio (Martel e Toner, 2012; Nivedita et al., 2017). I canali a spirale mantengono questo flusso secondario, chiamato flusso Dean, per tutta la lunghezza del canale, creando una posizione di equilibrio più stabile rispetto ai progetti di canali asimmetricamente curvi (Gossett e Di Carlo, 2009). La capacità di modificare la curvatura dei microcanali a spirale offre l’opportunità di un migliore controllo e di un’ulteriore ottimizzazione della portata, che può essere migliorata per la separazione delle particelle ad alto rendimento (Kuntaegowdanahalli et al., 2009).
L’applicazione di queste forze fluide attraverso la microfluidica viene regolarmente esplorata come mezzo per separare le cellule e altre bioparticelle in base alle dimensioni (Martel e Toner, 2014). Una delle applicazioni più comuni è la separazione delle cellule tumorali circolanti (CTC) dalle cellule del sangue a causa della tendenza dei CTC ad essere più grandi. È significativo il fatto che i CTC possono apparire nel flusso sanguigno durante le metastasi a un tasso simile a quello delle cellule fungine durante le candidemie (<100/mL di sangue intero). Mentre le cellule più piccole sono più problematiche da focalizzare a causa della natura delle forze che si scalano fortemente con le dimensioni, le cellule fungine sono state focalizzate in precedenza in canali rettilinei (Masaeli et al., 2012) e i disegni riflessivi hanno utilizzato le dimensioni delle cellule del sangue per focalizzare gli RBC nel tentativo di separare le cellule batteriche (Mach e di Carlo, 2010). La natura ad alto rendimento della focalizzazione inerziale è necessaria per affrontare obiettivi a bassa concentrazione e la rende una tecnica promettente per l’isolamento delle cellule fungine.
L’isolamento dei microrganismi dal sangue differisce dall’attuale convenzione diagnostica della crescita dei microbi da una coltura ematica. L’obiettivo di entrambi è di riconoscere la presenza di microbi. La coltura si basa sulla crescita dei microbi per diventare riconoscibile. Un risultato positivo di un’emocoltura non indica in modo specifico la presenza di batteri e/o funghi, per cui sono necessari tempi supplementari e metodi più specifici. Questo processo che richiede tempo può essere notevolmente ridotto isolando i funghi direttamente dal sangue piuttosto che attendere la crescita. Inoltre, un rapido isolamento può avere un impatto significativo sui pazienti infettati da ceppi resistenti ai farmaci delle specie di Candida o di non-albici che sono meno suscettibili alle terapie antimicotiche comunemente fornite.
I metodi di coltura sono lunghi e richiedono un periodo di incubazione di 5 giorni per garantire un tempo adeguato per la crescita microbica. Mentre gli studi hanno rilevato che i metodi di emocoltura automatizzati indicano una crescita microbica positiva entro le prime 48 ore con un tempo mediano di 12-17 ore, il periodo di tempo per un intervento antimicrobico appropriato è <6 ore dopo l’insorgenza dei sintomi (Jordana-Lluch et al., 2013; Pardo et al., 2014). Infatti, ad una velocità di flusso di 400 μL/min le cellule fungine sono separate da campioni di sangue intero lisato entro 125 minuti utilizzando l’IFF, quindi disponibili per ulteriori metodi di identificazione come la PCR.
Altri hanno dimostrato che i C. albicans possono essere isolati dal sangue utilizzando metodologie microfluidiche alternative o incrementate. Cooper et al. hanno scoperto che il 67% dei funghi seminati nel sangue può essere separato e visualizzato otticamente utilizzando perle rivestite di legante per catturare e isolare le cellule con forza magnetica (Cooper et al., 2014). Una differenza importante rispetto al rapporto attuale è che l’IFF non richiede la manipolazione magnetica per isolare le cellule rare, come sviluppato da Cooper et al. che richiede un eccesso di 1.000 volte di perle magnetiche rispetto all’agente patogeno. Al contrario, sfruttiamo i differenziali dimensionali che si amplificano in microcanali a spirale rispetto al flusso laterale applicato nella procedura di separazione da Cooper et al.
Javanmared et al. riferiscono di aver catturato il 6,4% del totale delle cellule di C. albicans che passano attraverso il loro dispositivo microfluidico (Javanmard et al., 2012). La piattaforma si basa su canali paralleli formattati rivestiti con anticorpi specifici per i funghi. Legando le cellule patogene alla parete del canale, esse creano un’opportunità di imaging che può aiutare a identificare l’agente patogeno quando si verifica uno stato infettivo.
Due aree in cui il trattamento microfluidico ha permesso di ottenere efficienza sono la riduzione del volume e la concentrazione del campione come alternativa alla centrifugazione tradizionale (Martel et al., 2015). I saggi biologici e clinici tradizionalmente realizzano la riduzione di volume attraverso la centrifugazione per purificare e arricchire un campione per ulteriori analisi. I dispositivi microfluidici a flusso continuo non solo offrono una maggiore concentrazione del campione e riduzione del volume, ma evitano anche il potenziale di danneggiare le cellule e di alterare l’espressione genica cellulare che può verificarsi quando le bioparticelle sono esposte ad elevate forze centrifughe (Soto et al., 2007; Peterson et al., 2012). Inoltre, tali dispositivi non richiedono che un grande volume di partenza sia suddiviso in campioni più piccoli per essere trattato, il che aumenta il rischio di contaminare e/o perdere il campione (Martel e Toner, 2014).
Quando si considerano i fattori di influenza, come la dimensione dell’inoculo al momento del rilevamento delle infezioni del flusso sanguigno di Candida, il tempo rappresenta una barriera importante per un rilevamento e un trattamento efficace. Nel sistema BACTEC, le dimensioni dell’inoculo influenzano fortemente il rilevamento della crescita e il tempo di rilevamento per le BSI di Candida, dove inoculi più grandi portano a un rilevamento più veloce e a un minore ritardo di rilevamento dell’inoculo (Jacoby, 2005). Utilizzando l’attuale gold standard, i risultati del sistema di emocoltura BACTEC sono lenti e afflitti da sfide come una scarsa rilevazione in mezzi anaerobici e risultati falsi anche quando si testano inoculi di grandi dimensioni di 100 CFU/mL. Questo è uno svantaggio soprattutto quando si cerca di rilevare la crescita in pazienti con candidemia, dove la concentrazione di cellule fungine in circolazione è <10 CFU/mL. Questa bassa concentrazione di contaminanti potrebbe essere ben indirizzata dall’uso di un trattamento microfluidico. L’isolamento di ogni Candida spp. potrebbe quindi essere determinato in base alle caratteristiche morfologiche che distinguono le cellule fungine dalle cellule dei mammiferi.
In conclusione, si ipotizza che la concentrazione delle cellule fungine direttamente dal campione clinico possa favorire la nascita di una piattaforma diagnostica più rapida. Anche con questa raccolta di differenze tra la popolazione fungina, il design del dispositivo IFF ha dimostrato di essere in grado di ottenere un flusso focalizzato di cellule fungine che può essere diretto per l’isolamento. Sebbene l’IFF non segreghi il 100% delle cellule di Candida, è in grado di ridurre il volume e la concentrazione di soluti per catturare le cellule fungine presenti in piccole quantità ad un ritmo rapido. La tecnologia IFF potrebbe essere incorporata con altri progressi diagnostici che attenuano la necessità di centrifugazione o di incubazione del materiale di partenza. Anche se il dispositivo non quantifica direttamente le cellule, fornisce un mezzo per rilevare qualitativamente la presenza di cellule fungine in modo rapido, un’informazione potenzialmente preziosa per i medici, che ha il potenziale di aiutare nella diagnosi precoce.
Contributi dell’autore
Il design è stato fornito da BF, JM-F, SS e MT. I test dei dispositivi sono stati effettuati da BF, JM-F, SS e SE. L’analisi dei dati è stata effettuata da BF, JM-F, SE, MT e EM. SE e BF hanno scritto la prima bozza del manoscritto e tutti gli autori hanno contribuito alla preparazione finale del manoscritto.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che possano essere interpretate come un potenziale conflitto di interessi.
References
- Arvanitis M., Anagnostou T., Fuchs B. B., Caliendo A. M., Mylonakis E.. Molecular and nonmolecular diagnostic methods for invasive fungal infections. Clin. Microbiol. Rev.. 2014; 27:490-526. DOI | PubMed
- Cooper R. M., Leslie D. C., Domansky K., Jain A., Yung C., Cho M.. A microdevice for rapid optical detection of magnetically captured rare blood pathogens. Lab. Chip.. 2014; 14:182-188. DOI | PubMed
- Goodwin S. D., Fiedler-Kelly J., Grasela T. H., Schell W. A, Perfect J. R.. A nationwide survey of clinical laboratory methodologies for fungal infections. J. Med. Vet. Mycol.. 1992; 30:153-160. DOI | PubMed
- Gossett D. R., Di Carlo D.. Particle focusing mechanisms in curving confined flows. Anal. Chem.. 2009; 81:8459-8465. DOI | PubMed
- Horvath L. L., George B. J., Hospenthal D. R.. Detection of fifteen species of Candida in an automated blood culture system. J. Clin. Microbiol.. 2007; 45:3062-3064. DOI | PubMed
- Jacoby G. A.. Mechanisms of resistance to quinolones. Clin. Infect. Dis.. 2005; 41(Suppl. 2):S120-S126. DOI | PubMed
- Jaeger E. E., Carroll N. M., Choudhury S., Dunlop A. A., Towler H. M., Matheson M. M.. Rapid detection and identification of Candida, Aspergillus, and Fusarium species in ocular samples using nested PCR. J. Clin. Microbiol.. 2000; 38:2902-2908. PubMed
- Javanmard M., Babrzadeh F., Nyren P., Davis R. W.. Improvment in cell capture throughput using parallel bioactive microfluidic channels. Biomed. Microdevices. 2012; 14:625-629. DOI | PubMed
- Jordana-Lluch E., Carolan H. E., Giménez M., Sampath R., Ecker D. J., Quesada M. D.. Rapid diagnosis of bloodstream infections with PCR followed by mass spectrometry. PLoS ONE. 2013; 8:e62108. DOI | PubMed
- Kourkoumpetis T. K., Fuchs B. B., Coleman J. J., Desalermos A., Mylonakis E.. Polymerase chain reaction-based assays for the diagnosis of invasive fungal infections. Clin. Infect. Dis.. 2012; 54:1322-1331. DOI | PubMed
- Krcmery V., Barnes A. J.. Non-albicans Candida spp. causing fungaemia: pathogenicity and antifungal resistance. J. Hosp. Infect.. 2002; 50:243-260. DOI | PubMed
- Kuntaegowdanahalli S. S., Bhagat A. A., Kumar G., Papautsky I.. Inertial microfluidics for continuous particle separation in spiral microchannels. Lab. Chip.. 2009; 9:2973-2980. DOI | PubMed
- Mach A. J., di Carlo D.. Continuous scalable blood filtration device using inertial microfluidics. Biotechnol. Bioeng.. 2010; 107:302-311. DOI | PubMed
- Martel J. M., Smith K. C., Dlamini M., Pletcher K., Yang J., Karabacak M.. Continuous flow microfluidic bioparticle concentrator. Sci. Rep.. 2015; 5:11300. DOI | PubMed
- Martel J. M., Toner M.. Inertial focusing dynamics in spiral microchannels. Phys. Fluids. 2012; 24:32001. DOI | PubMed
- Martel J. M., Toner M.. Inertial focusing in microfluidics. Annu. Rev. Biomed. Eng.. 2014; 16:371-396. DOI | PubMed
- Masaeli M., Sollier E., Amini H., Mao W., Camacho K., Doshi N.. Continuous inertial focusing and separation of particles by shape. Phys. Rev. X. 2012; 2:031017. DOI
- Micek S. T., Lloyd A. E., Ritchie D. J., Richard M., Fraser V. J., Kollef M. H.. Pseudomonas aeruginosa bloodstream infection : importance of appropriate initial antimicrobial treatment. Antimicrob. Agents Chemother.. 2005; 49:1306-1311. DOI | PubMed
- Morrell M., Fraser V. J., Kollef M. H.. Delaying the empiric treatment of Candida bloodstream infection until positive blood culture results are obtained: a potential risk factor for hospital mortality. Antimicrob. Agents Chemother.. 2005; 49:3640-3645. DOI | PubMed
- Morris A. J., Wilson S. J., Marx C. E., Wilson M. L., Mirrett S., Reller L. B.. Clinical impact of bacteria and fungi recovered only from broth cultures. J. Clin. Microbiol.. 1995; 33:161-165. PubMed
- Nivedita N., Ligrani P., Papautsky I.. Dean flow dynamics in low-aspect ratio spiral microchannels. Sci. Rep.. 2017; 7:44072. DOI | PubMed
- Pappas P. G.. Invasive candidiasis. Infect. Dis. Clin. North Am.. 2006; 20:485-506. DOI | PubMed
- Pardo J., Klinker K. P., Borgert S. J., Trikha G., Rand K. H., Ramphal R.. Time to positivity of blood cultures supports antibiotic de-escalation at 48 hours. Ann. Pharmacother.. 2014; 48:33-40. DOI | PubMed
- Peterson B. W., Sharma P. K., van der Mei H. C., Busscher H. J.. Bacterial cell surface damage due to centrifugal compaction. Appl. Environ. Microbiol.. 2012; 78:120-125. DOI | PubMed
- Pfaller M. A., Diekema D. J.. Epidemiology of invasive candidiasis: a persistent public health problem. Clin. Microbiol. Rev.. 2007; 20:133-163. DOI | PubMed
- Sievert D. M., Ricks P., Edwards J. R., Schneider A., Patel J., Srinivasan A.. Antimicrobial-resistant pathogens associated with healthcare-associated infections : summary of data reported to the national healthcare safety network at the centers for disease control and prevention, 2009 – 2010. Infect. Control Hosp. Epidemiol.. 2013; 34:1-14. DOI | PubMed
- Soto T., Núñez A., Madrid M., Vicente J., Gacto M., Cansado J.. Transduction of centrifugation-induced gravity forces through mitogen-activated protein kinase pathways in the fission yeast Schizosaccharomyces pombe. Microbiology. 2007; 153:1519-1529. DOI | PubMed
- Vitale R. G., Nucci M.. Diagnosis of candidemia. Curr. Fungal Infect. Rep.. 2014; 8:90-94. DOI
- Wisplinghoff H., Bischoff T., Tallent S. M., Seifert H., Wenzel R. P., Edmond M. B.. Nosocomial bloodstream infections in US hospitals: analysis of 24,179 cases from a prospective nationwide surveillance study. Clin. Infect. Dis.. 2004; 39:309-317. DOI | PubMed
- Yapar N.. Epidemiology and risk factors for invasive candidiasis. Ther. Clin. Risk Manag.. 2014; 10:95-105. DOI | PubMed
- Zaoutis T., Prasad P., Russell L. A., Coffin S., Bell L. M., Walsh T. J.. Risk factors and predictors for candidemia in pediatric intensive care unit patients: im plications for prevention. Clin. Infect. Dis.. 2010; 51:e38-e45. DOI | PubMed
Fonte
Fuchs BB, Eatemadpour S, Martel-Foley JM, Stott S, Toner M, et al. (2019) Rapid Isolation and Concentration of Pathogenic Fungi Using Inertial Focusing on a Chip-Based Platform. Frontiers in Cellular and Infection Microbiology 927. https://doi.org/10.3389/fcimb.2019.00027





