Introduzione
L’attivazione farmacologica dei meccanismi di longevità per aumentare la resistenza allo stress e migliorare la capacità di proteostasi sembra essere una strategia di trattamento attraente per le malattie degenerative (Balch et al., 2008). Mentre l’esatto meccanismo (o i meccanismi esatti) di morte neuronale nel morbo di Alzheimer, nel morbo di Parkinson e nelle malattie correlate rimangono elusivi, esistono prove genetiche convincenti per un collasso della proteostasi associato all’età che contribuisce all’aggregazione proteica e ai fenotipi degenerativi (Taylor e Dillin, 2011). La maggior parte dei meccanismi di longevità dipendono in modo cruciale dalla capacità di attivare i percorsi di segnalazione dello stress (SSP) e i meccanismi proteostatici (Steinkraus et al., 2008; Tullet et al., 2008; Henis-Korenblit et al., 2010; Lapierre et al., 2013). Ad esempio, uno studio completo in Caenorhabditis elegans di Shore et al. ha dimostrato che l’induzione di SSP è centrale e necessaria per indurre la longevità di 160 RNAis diversi (Shore et al., 2012). Altri hanno dimostrato che con l’invecchiamento degli animali, la loro capacità di rispondere allo stress diminuisce drasticamente (Labbadia e Morimoto, 2015; Dues et al., 2016). Di conseguenza, si può prevedere che i percorsi di longevità i cui meccanismi dipendono dall’attivazione delle SSP diventeranno non reattivi con l’invecchiamento.
Infatti, la maggior parte dei meccanismi di longevità, come la riduzione della segnalazione di insulina/IGF o una riduzione dell’attività della catena di trasporto degli elettroni, non prolungano la durata della vita quando iniziata oltre il quinto giorno dell’età adulta di C. elegans (Dillin et al., 2002a; Dillin et al., 2002b; Rangaraju et al., 2015). Un corretto tempismo è richiesto anche nei topi nani di Ames, dove la carenza di ormone della crescita durante le prime 6 settimane di sviluppo pre-pubertale è fondamentale per la longevità (Panici et al., 2010).
Idealmente si vorrebbe essere in grado di aumentare la capacità di proteostasi e di prolungare la durata della vita e della salute trattando farmacologicamente gli organismi più anziani alla prima comparsa di sintomi neurodegenerativi o di biomarcatori di malattia (ad es. aggregati proteici). Fortunatamente, alcuni meccanismi di longevità come la restrizione alimentare o il trattamento con rapamicina rimangono reattivi anche in età avanzata (Weindruch e Walford, 1982; Harrison et al., 2009). Tuttavia, si sa poco di ciò che distingue i meccanismi di longevità specifici del tempo dai meccanismi di longevità che rimangono reattivi per tutta la vita.
In questo studio abbiamo identificato il minociclinico, un farmaco noto per le sue proprietà neuroprotettive e antinfiammatorie nei mammiferi (Mitra et al., 1975; Choi et al., 2005; Festoff et al., 2006; Seabrook et al., 2006; Choi et al., 2007; Noble et al., 2009; Zheng et al., 2010; Cai et al., 2011; Ferretti et al., 2012; Obici et al., 2012; Cai et al., 2013; Garrido-Mesa et al., 2013), per allungare la durata della vita e ridurre l’aggregazione proteica anche nei vecchi C. elegans che hanno perso la capacità di attivare gli SSP. La successiva chiarificazione del meccanismo d’azione (MOA) mostra che il minociclinico attenua la traduzione citoplasmatica indipendentemente dalla risposta integrata allo stress (ISR), ma ne imita gli effetti benefici. Proponiamo che gli effetti neuroprotettivi e di aggregazione-prevenzione degli effetti della minociclina, osservati nei modelli murini preclinici così come negli studi clinici umani, siano spiegati dalla sua attenuazione della sintesi proteica citoplasmatica. La riduzione della concentrazione di proteine di nuova sintesi, soggette ad aggregazione, allevia le esigenze della rete di proteostasi anche in individui più anziani in cui le SSP e le vie di degradazione delle proteine sono già state compromesse a causa dell’età avanzata.
Risultati
Minociclina estende la durata della vita di C. elegans sia in animali giovani che vecchi
Studi precedenti hanno dimostrato che la capacità di C. elegans di rispondere allo stress diminuisce con l’età (Bansal et al., 2015; Labbadia e Morimoto, 2015). Per ampliare gli studi precedenti e per definire l’età in cui i principali SSP perdono la capacità di rispondere allo stress, abbiamo esposto ceppi trascrizionali GFP-reporter che rispondono allo stress ossidativo (gst-4p::GFP) o proteine dispiegate nel reticolo endoplasmatico (hsp-4p::GFP, UPRER), nei mitocondri (hsp-6p::GFP, UPRmt), o nel citosol (hsp-16.2p::GFP) ai rispettivi fattori di stress -arsenite, tunicamicina, paraquat o calore – all’aumentare dell’età. Il primo giorno come adulti riproduttivi, qui definito come giorno 1, i fattori di stress hanno aumentato l’espressione delle GFP rispetto ai controlli non trattati (Figura 1A,B). L’eccezione è stata hsp-6p::GFP che è stato fortemente attivato allo stadio larvale L2 (Figura 1-figure supplemento 1A), ma ha risposto male il giorno 1. Dal giorno 5 dell’età adulta, l’attività SSP è stata ridotta e dal giorno 8 era quasi impercettibile (Figura 1A,B). Così, 8 giorni dopo aver raggiunto l’età adulta C. elegans non induce più SSP in risposta allo stress, nonostante sia morto per i suoi effetti negativi. Ci riferiremo al giorno 8 come l’età “post-stress-responsive”.
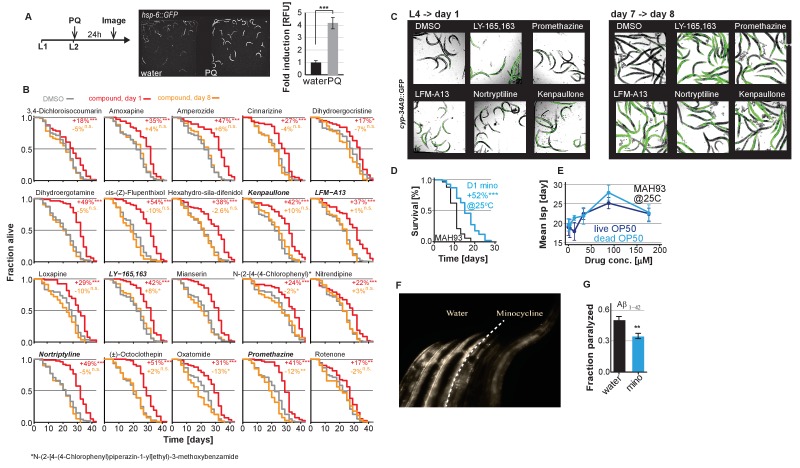
Figura 1-figure supplement 1.Riepilogo dei dati sulla durata della vita di N2, relativi alla Figura 1. Riepilogo dei dati sulla durata della vita di N2, relativi alla Figura 1. Piccole molecole analizzate per la durata della vita post-stress-responsive.(A) I diagrammi di dispersione mostrano l’induzione della fluorescenza GFP indotta dai fattori di stress rispetto agli animali non trattati in gst-4p::GFP (stress ossidativo), hsp-4p::GFP (UPRER), hsp-6p::GFP (UPRmt) e hsp-16.2p::GFP (stress da calore) C. elegans a 1, 5 e 8 giorni di età. Definiamo il primo giorno come il primo giorno dell’età adulta. Le barre di errore mostrano la media ± S.D., ogni punto rappresenta un animale con tutti n > 300 per ogni ceppo e l’età del reporter. Significato determinato da ANOVA seguito dal test di Dunett. Totale di quattro esperimenti indipendenti. (B) Immagini rappresentative al microscopio a fluorescenza di 100 animali selezionati a caso per ogni condizione e ceppo. Stressors indicato a destra di ogni pannello di immagini. Le immagini per ogni ceppo sono state scattate in parallelo lo stesso giorno utilizzando impostazioni identiche. Totale di quattro esperimenti indipendenti. (C) I grafici a barre mostrano la variazione % della durata della vita per 21 piccole molecole quando il trattamento è stato iniziato il giorno 1 di età adulta (grigio) o il giorno 8 di età adulta (arancione). Si noti che alcuni composti che prolungano la durata della vita quando il trattamento è iniziato il primo giorno diventano leggermente tossici in età avanzata. Un esperimento indipendente con almeno 53 animali per condizione. Significato determinato dal log-rank test. (D) Struttura del minociclinico. (E) Curve di sopravvivenza per animali wild-type N2 non trattati (linea nera) e minociclina trattati con minociclina quando il trattamento è stato avviato il giorno 1 (linea blu) o il giorno 8 (linea arancione) dell’età adulta. OP50 sono stati uccisi da γ-irradiazione per separare l’antibiotico dagli effetti della durata della vita. Totale di cinque esperimenti indipendenti. Significato determinato dal log-rank test. (F) Variazione percentuale della durata della vita in funzione della concentrazione di minociclina per N2. OP50 sono stati irradiati con γ. Totale di quattro esperimenti indipendenti. (G) Rappresentante immagini al microscopio a fluorescenza che mostrano le teste di C. elegans all’aumentare delle età che esprimono la α-sinucleina::YFP proteina di fusione YFP. In alto: controllo trattato con acqua, in basso: minociclina trattata (100 μM). Si noti l’aumento della colorazione punteggiata con l’età indicativa dell’aggregazione proteica. (H) Il grafico mostra il numero medio di α-sinucleina::YFP aggregati per verme in funzione dell’età per gli animali trattati con acqua o minociclina. Età quando il trattamento minociclina viene avviato per ogni colore è mostrato a destra del grafico. Le barre di errore indicano S.E.M. Per ogni punto di dati, n > 15. Totale di quattro esperimenti indipendenti. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati di partenza per gli esperimenti sulla durata di vita di N2 sono disponibili nella Figura 1 – dati di partenza 1.10.7554/eLife.40314.004Figure 1-source data 1.Summary of N2 lifespan data, related to Figure 1.(A) Controllo positivo per dimostrare che la mancanza di attivazione di hsp-6p::GFP negli animali adulti (Figura 1A,B) è dovuta all’età e non ad un ceppo difettoso. Timeline sperimentale, immagini fluorescenti e grafici a barre che rappresentano l’espressione indotta dal paraquat (PQ) di hsp-6p::GFP quando aggiunto allo stadio larvale L2. (B) Curve di sopravvivenza per i grafici a barre mostrati in Figura 1C. Piccole molecole schermate per l’estensione della durata della vita quando aggiunte il giorno 1 (linea rossa) o il giorno 8 (linea arancione) rispetto a quelle non trattate (linea grigia). Significato determinato dalla prova log-rank. (C) Conferma dell’assorbimento del farmaco per un sottogruppo di farmaci testati nella Figura 1. Sovrapposizione di campo luminoso e immagini fluorescenti per monitorare il reporter xenobiotico, cyp-34A9p::attivazione GFP. Piccole molecole che attivano cyp-34A9p::Le GFP negli animali del giorno 1 lo fanno anche negli animali del giorno 8 come visualizzato dall’espressione GFP che conferma l’assorbimento dei farmaci ad entrambe le età. (D ed E) rappresentativo di sopravvivenza e curve di risposta della dose che mostra minociclina prolunga la durata della vita di MAH93 glp-1 (ar202), un ceppo che è sterile a 25 ° C, in assenza di FUDR (D) e con o senza γ-irradiato OP50 dose-dipendente (E). (F) Confronto fianco a fianco di 8 giorni NL5901 α-sinucleina::YFP animali trattati il giorno 1 con acqua (a sinistra tre animali) o minociclina (a destra tre animali). (G) Il grafico mostra la frazione di animali adulti paralizzati di 4 giorni C. elegans adulti paralizzati che esprimono Aβ 1-42 dopo uno shock termico di 1 ora a 35 ° C. Il pretrattamento con minociclina (100 μM) riduce la paralisi. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.
Abbiamo poi chiesto se esistano meccanismi di longevità e di proteostasi che rimangano farmacosi negli adulti post-stress-responsabili. A tal fine, abbiamo trattato gli adulti di C. elegans di 8 giorni con 21 molecole diverse che abbiamo precedentemente identificato per prolungare la durata della vita (Ye et al., 2014). Tutte le 21 molecole hanno allungato la durata della vita quando il trattamento è stato iniziato il primo giorno di età adulta. Tuttavia, solo il minocicline ha esteso la durata della vita quando il trattamento è stato avviato il giorno 8 (Figura 1C; Figura 1-figure supplement 1B). Per alcuni dei farmaci inattivi, è stato utilizzato un ceppo xenobiotico-responsivo cyp-34A9p::GFP per confermare l’assorbimento del farmaco nel vecchio C. elegans (Figura 1-figure supplement 1C) (Anbalagan et al., 2012).
Minocycline è un antibiotico tetraciclina approvato dall’agenzia di regolamentazione utilizzato per trattare l’acne vulgaris ed è noto da tempo per ridurre la crescita tumorale, l’infiammazione e l’aggregazione di proteine nei mammiferi da un MOA sconosciuto (Figura 1D) (Garrido-Mesa et al., 2013). Minociclina ancora esteso la durata della vita quando il trattamento è stato avviato il giorno 8, anche se del 22% invece del 48% osservato quando il trattamento è iniziato il giorno 1 (Figura 1E). Successivi esperimenti di controllo hanno dimostrato che l’estensione della durata della vita da minociclina era indipendente dal metodo di sterilizzazione, dalla temperatura di coltivazione (Figura 1-figure supplement 1D,E) e dal fatto che gli animali sono stati alimentati con batteri vivi o morti (OP50). Registrando le curve di risposta della dose abbiamo determinato un EC50 di 22 μM e una concentrazione ottimale di 50 – 100 μM, rispetto alle concentrazioni terapeutiche misurate nel siero del paziente di 5 – 10 μM (Figura 1F; Figura 1-figure supplement 1E) (Agwuh e MacGowan, 2006; Garrido-Mesa et al., 2013). Per studiare il MOA di minociclina indipendentemente dalla sua attività antibiotica, tutti gli esperimenti successivi sono stati condotti utilizzando solo morti, γ-irradiato batteri. Presi insieme, la minociclina prolunga la durata della vita indipendentemente dalla sua attività antibiotica, anche quando il trattamento è iniziato in C. elegans vecchio, post-stress-responsive C. elegans.

Figura 1-figure supplement 1.Riepilogo dei dati sulla durata della vita di N2, relativi alla Figura 1.Riepilogo dei dati sulla durata della vita di N2, relativi alla Figura 1.Piccole molecole schermate per la durata della vita post-stress-responsive.(A) I diagrammi di dispersione mostrano l’induzione della fluorescenza GFP indotta dai fattori di stress rispetto agli animali non trattati in gst-4p::GFP (stress ossidativo), hsp-4p::GFP (UPRER), hsp-6p::GFP (UPRmt) e hsp-16.2p::GFP (stress da calore) C. elegans a 1, 5 e 8 giorni di età. Definiamo il primo giorno come il primo giorno dell’età adulta. Le barre di errore mostrano la media ± S.D., ogni punto rappresenta un animale con tutti n > 300 per ogni ceppo e l’età del reporter. Significato determinato da ANOVA seguito dal test di Dunett. Totale di quattro esperimenti indipendenti. (B) Immagini rappresentative al microscopio a fluorescenza di 100 animali selezionati a caso per ogni condizione e ceppo. Stressors indicato a destra di ogni pannello di immagini. Le immagini per ogni ceppo sono state scattate in parallelo lo stesso giorno utilizzando impostazioni identiche. Totale di quattro esperimenti indipendenti. (C) I grafici a barre mostrano la variazione % della durata della vita per 21 piccole molecole quando il trattamento è stato iniziato il giorno 1 di età adulta (grigio) o il giorno 8 di età adulta (arancione). Si noti che alcuni composti che prolungano la durata della vita quando il trattamento è iniziato il primo giorno diventano leggermente tossici in età avanzata. Un esperimento indipendente con almeno 53 animali per condizione. Significato determinato dal log-rank test. (D) Struttura del minociclinico. (E) Curve di sopravvivenza per animali wild-type N2 non trattati (linea nera) e minociclina trattati con minociclina quando il trattamento è stato avviato il giorno 1 (linea blu) o il giorno 8 (linea arancione) dell’età adulta. OP50 sono stati uccisi da γ-irradiazione per separare l’antibiotico dagli effetti della durata della vita. Totale di cinque esperimenti indipendenti. Significato determinato dal log-rank test. (F) Variazione percentuale della durata della vita in funzione della concentrazione di minociclina per N2. OP50 sono stati irradiati con γ. Totale di quattro esperimenti indipendenti. (G) Rappresentante immagini al microscopio a fluorescenza che mostrano le teste di C. elegans all’aumentare delle età che esprimono la α-sinucleina::YFP proteina di fusione YFP. In alto: controllo trattato con acqua, in basso: minociclina trattata (100 μM). Si noti l’aumento della colorazione punteggiata con l’età indicativa dell’aggregazione proteica. (H) Il grafico mostra il numero medio di α-sinucleina::YFP aggregati per verme in funzione dell’età per gli animali trattati con acqua o minociclina. Età quando il trattamento minociclina viene avviato per ogni colore è mostrato a destra del grafico. Le barre di errore indicano S.E.M. Per ogni punto di dati, n > 15. Totale di quattro esperimenti indipendenti. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati di partenza per gli esperimenti sulla durata di vita di N2 sono disponibili nella Figura 1 – dati di partenza 1.10.7554/eLife.40314.004Figure 1-source data 1.Summary of N2 lifespan data, related to Figure 1.(A) Controllo positivo per dimostrare che la mancanza di hsp-6p::L’attivazione delle GFP negli animali adulti (Figura 1A,B) è dovuta all’età e non a un ceppo difettoso. Timeline sperimentale, immagini fluorescenti e grafici a barre che rappresentano l’espressione indotta dal paraquat (PQ) di hsp-6p::GFP quando aggiunto allo stadio larvale L2. (B) Curve di sopravvivenza per i grafici a barre mostrati in Figura 1C. Piccole molecole schermate per l’estensione della durata della vita quando aggiunte il giorno 1 (linea rossa) o il giorno 8 (linea arancione) rispetto a quelle non trattate (linea grigia). Significato determinato dalla prova log-rank. (C) Conferma dell’assorbimento del farmaco per un sottogruppo di farmaci testati nella Figura 1. Sovrapposizione di campo luminoso e immagini fluorescenti per monitorare il reporter xenobiotico, cyp-34A9p::attivazione GFP. Piccole molecole che attivano cyp-34A9p::Le GFP negli animali del giorno 1 lo fanno anche negli animali del giorno 8, come visualizzato dall’espressione GFP che conferma l’assorbimento dei farmaci ad entrambe le età. (D ed E) rappresentativo di sopravvivenza e curve di risposta della dose che mostra minociclina prolunga la durata della vita di MAH93 glp-1 (ar202), un ceppo che è sterile a 25 ° C, in assenza di FUDR (D) e con o senza γ-irradiato OP50 dose-dipendente (E). (F) Confronto fianco a fianco di 8 giorni NL5901 α-sinucleina::YFP animali trattati il giorno 1 con acqua (a sinistra tre animali) o minociclina (a destra tre animali). (G) Il grafico mostra la frazione di animali adulti paralizzati di 4 giorni C. elegans adulti paralizzati che esprimono Aβ 1-42 dopo uno shock termico di 1 ora a 35 ° C. Il pretrattamento con minociclina (100 μM) riduce la paralisi. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.

Figura 1-figure supplemento 1.Piccole molecole schermate per la durata di vita post-stress-responsive.(A) Controllo positivo per dimostrare che la mancanza di hsp-6p::L’attivazione delle GFP negli animali adulti (Figura 1A,B) è dovuta all’età e non ad un ceppo difettoso. Timeline sperimentale, immagini fluorescenti e grafici a barre che rappresentano l’espressione indotta dal paraquat (PQ) di hsp-6p::GFP quando aggiunto allo stadio larvale L2. (B) Curve di sopravvivenza per i grafici a barre mostrati in Figura 1C. Piccole molecole schermate per l’estensione della durata della vita quando aggiunte il giorno 1 (linea rossa) o il giorno 8 (linea arancione) rispetto a quelle non trattate (linea grigia). Significato determinato dalla prova log-rank. (C) Conferma dell’assorbimento del farmaco per un sottogruppo di farmaci testati nella Figura 1. Sovrapposizione di campo luminoso e immagini fluorescenti per monitorare il reporter xenobiotico, cyp-34A9p::attivazione GFP. Piccole molecole che attivano cyp-34A9p::Le GFP negli animali del giorno 1 lo fanno anche negli animali del giorno 8 come visualizzato dall’espressione GFP che conferma l’assorbimento dei farmaci ad entrambe le età. (D ed E) rappresentativo di sopravvivenza e curve di risposta della dose che mostra minociclina estende la durata della vita di MAH93 glp-1 (ar202), un ceppo che è sterile a 25 ° C, in assenza di FUDR (D) e con o senza γ-irradiato OP50 dose-dipendente (E). (F) Confronto fianco a fianco di 8 giorni NL5901 α-sinucleina::YFP animali trattati il giorno 1 con acqua (a sinistra tre animali) o minociclina (a destra tre animali). (G) Il grafico mostra la frazione di animali adulti paralizzati di 4 giorni C. elegans adulti paralizzati che esprimono Aβ 1-42 dopo uno shock termico di 1 ora a 35 ° C. Il pretrattamento con minociclina (100 μM) riduce la paralisi. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.
Minociclina attenua l’aggregazione proteica sia in giovani e vecchi C. elegans
Abbiamo poi testato il minociclinico per la sua capacità di modulare l’aggregazione delle proteine, una caratteristica molecolare comune a molte malattie neurodegenerative. Abbiamo trattato animali che esprimono α-sinucleina umana fusa a YFP nel muscolo (van Ham et al., 2008) con acqua o minociclina il primo giorno di età adulta e li abbiamo ripresi nei giorni 8, 11, 16 e 19. La minociclina ha soppresso l’aumento dipendente dall’età nell’aggregazione di α-sinucleina. Ha anche ridotto α-sinucleina aggregazione anche quando aggiunto a post-stress-responsive adulti in età avanzata (giorno 8), quando le risposte proteiche unfolded non sono stati più indotti (Figura 1G,H; Figura 1-figure supplemento 1F). Per confermare questo effetto esteso oltre α-sinucleina di aggregazione, abbiamo testato un altro modello di aggregazione proteica e determinato minociclina anche ridotto la paralisi causata dalla temperatura indotta Aβ 1-42 misfolding indotta dalla temperatura che porta all’aggregazione in C. elegans ‘corpo-muscolo parete (Figura 1-figure supplemento 1G) (Jiang et al., 2012; McColl et al., 2012). Così, il trattamento minociclico ha ridotto sia l’aggregazione proteica dipendente dall’età che quella indotta dalla temperatura in C. elegans.
Minociclina estende la durata della vita C. elegans nei mutanti difettosi per l’attivazione SSP o autofagia
Poiché il minociclinico prolunga la durata della vita di C. elegans post-stress-responsive, la sua attività dovrebbe essere indipendente dall’attivazione della SSP. Questa previsione è stata testata in due modi. In primo luogo, abbiamo ripetuto gli esperimenti di attivazione di risposta allo stress basati su GFP reporter, pretrattando gli adulti di 5 giorni per 2 ore con minociclina prima di sottoporli allo stress (Figura 2A). Come previsto, la minociclina da sola non ha indotto alcun segnalatore SSP (Figura 2B,C). Inaspettatamente, il pretrattamento con minociclina ha soppresso l’attivazione indotta da stress o da stress di tutti i segnalatori SSP rispetto al solo stressor. Ciò è stato particolarmente sorprendente in quanto studi precedenti hanno dimostrato che il trattamento delle larve con minociclina attiva l’hsp-6p::GFP reporter (Figura 2-figure supplement 1A,B) (Houtkooper et al., 2013). Al contrario, negli adulti, la minociclina inibisce l’attivazione di tutte le SSP. L’inibizione della minociclina indotta da SSPs non ha comportato un aumento della suscettibilità allo stress. Gli adulti trattati con minociclina sono sopravvissuti allo stress da calore (35°C) molto meglio degli adulti non trattati, con il 60% degli animali trattati con minociclina ancora vivi quando quasi tutti gli animali di controllo erano morti (Figura 2-figure supplement 1C). Inoltre, gli adulti trattati con minociclina erano anche molto più resistenti allo stress ossidativo indotto dal paraquat (Figura 2-figure supplement 1D). Ciò è coerente con l’osservazione che la minociclina protegge anche le cellule del feocromocitoma di ratto di tipo neuronale (PC12) dalla morte cellulare indotta da paraquat (Huang et al., 2012). Così, nonostante l’inibizione dell’attivazione della SSP, la minociclina protegge dallo stress, suggerendo un meccanismo di protezione che bypassa l’attivazione della SSP.
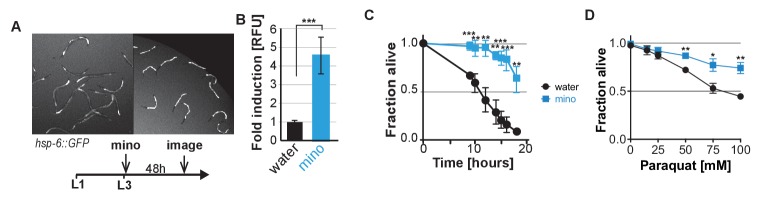
Figura 2-figure supplemento 1.Minociclina sopprime l’attività della via di segnalazione dello stress.riassunto dei dati sulla durata della vita per i ceppi portatori di mutazioni nei regolatori di stress, proteostasi, autofagia e risposte lisosomiali, relativi alla Figura 2.riassunto dei dati sulla durata della vita per i ceppi portatori di mutazioni nei regolatori di stress, proteostasi, autofagia e risposte lisosomiali, relativi alla Figura 2.Nonostante l’inibizione dell’UPRmt e la risposta allo shock termico negli adulti, la minociclina attiva l’UPRmt nelle larve e protegge dallo stress.(A) Timeline sperimentale per monitorare l’attivazione di risposta allo stress di 5 giorni C. elegans adulto adulto C. elegans GFP-reporter ceppi indotti da stressor e / o minociclina. Minociclina (100 μM) è stato aggiunto 2 ore prima di ogni fattore di stress. (B) Trame Spargimento mostra l’induzione piega di induzione di fluorescenza GFP indotta da fattori di stress e / o il trattamento minociclina rispetto agli animali non trattati in 5 giorni di età adulti. Pretrattamento con minociclina sopprime l’attivazione risposta allo stress. Reporter: gst-4p::GFP (stress ossidativo), hsp-4p::GFP (ER UPR), hsp-6p::GFP (UPRmt) e hsp-16.2p::GFP (stress da calore). Le barre di errore mostrano la media ± S.D., ogni punto rappresenta un animale con tutti n > 300. Significato determinato dal t-test di Mann-Whitney. Almeno tre esperimenti indipendenti. (C) Immagini rappresentative microscopia a fluorescenza di 100 animali selezionati a caso per ogni condizione e ceppo. Stressori indicati a destra di ogni pannello. Le immagini per ogni ceppo sono state scattate in parallelo lo stesso giorno utilizzando impostazioni identiche. (D) Curve di sopravvivenza. Minociclina estende significativamente la durata della vita nei ceppi che portano mutazioni nei regolatori di stress e risposte di proteostasi. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 35 e 218. Totale di almeno tre esperimenti indipendenti per ceppo. E) Curve di sopravvivenza. Minociclina prolunga significativamente la durata della vita in due ceppi portatori di mutazioni nei regolatori delle vie lisosomiche e autofagiche. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 56 e 138. Totale di almeno tre esperimenti indipendenti per ceppo. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati fonte per tutti gli esperimenti sulla durata della vita in (D) ed (E) sono disponibili nella Figura 2. Dati fonte 1.10.7554/eLife.40314.007Figure 2-source data 1.Summary of lifespan data for strains carrying mutations in regulators of stress, proteostasis, autofagy e lysosomal responses, related to Figure 2.(A e B) Immagine in fluorescenza e linea temporale sperimentale (A) e quantificazione (B) per hsp-6p::induzione di GFP da minociclina aggiunta alle larve L3. Mentre minociclina soppresso induzione GFP di tutti i giornalisti stress testato negli adulti, si attiva l’hsp-6p::GFP nelle larve. I dati sono rappresentati come media ±S.D. Significato determinato dal t-test di Mann-Whitney. (C) Sopravvivenza in funzione del tempo in ore per l’acqua o 100 μM minociclina trattata con minocicline di tipo selvatico N2 C. elegans sottoposto ad uno shock termico continuo di 35°C. I dati sono rappresentati come media ±S.E.M. Significato determinato dal test t di Mann-Whitney in ogni punto temporale. Totale di tre esperimenti indipendenti. (D) Sopravvivenza di acqua o 100 μM minociclina trattata con minociclina wild-type N2 C. elegans 24 ore dopo l’aggiunta di concentrazioni crescenti di paraquat aggiunto il giorno 5. I dati sono rappresentati come media ±S.E.M. Significato determinato dal Mann-Whitney t-test per ogni concentrazione di paraquat. Totale di tre esperimenti indipendenti. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo.
In secondo luogo, abbiamo misurato la durata della vita di ceppi trattati con acqua e minociclina che portano mutazioni nei geni che codificano i fattori di trascrizione che regolano l’attività SSP, tra cui il fattore di trascrizione FOXO daf-16, l’attivatore trascrizionale UPRER xbp-1, l’attivatore trascrizionale UPRmt atfs-1, il fattore di risposta allo stress ossidativo skn-1, e il fattore di trascrizione dello shock termico universalmente conservato hsf-1. Minociclina esteso durata della vita in tutti i mutanti C. elegans (Figura 2D). Notevolmente, minociclina esteso durata della vita in hsf-1 (sy441) mutanti fino a +159%, un’estensione relativa chiaramente maggiore di quello che è stato osservato in wild-type N2 animali di tipo N2 e quasi raggiungere la durata della vita assoluta di animali trattati con minociclina N2 (Figura 1E; Figura 2 dati fonte 1). Questa estensione migliorata suggerisce che la minociclina non solo prolunga la durata della vita come in N2, ma salva anche alcuni dei difetti associati con hsf-1 mutato (sy441).
Un modo in cui la minociclina potrebbe salvare i difetti associati all’hsf-1 sarebbe quello di eliminare le proteine mal piegate inducendo percorsi autofagici e lisosomiali (Kumsta et al., 2017). Per determinare se l’autofagia è necessaria per l’estensione della durata della vita da minociclina, abbiamo misurato la durata della vita nei ceppi trattati con minociclina che ospitano mutazioni nell’ortologia Atg1/ULK1 unc-51 e nell’ortologia TFEB hlh-30, fattori chiave critici per l’attività autofagica e lisosomiale. In entrambi i ceppi, minociclina significativamente aumentato la durata della vita (Figura 2E). Nonostante i gravi fenotipi comportamentali e morfologici osservati nei mutanti unc-51(e369), la mutazione non ha ridotto la capacità del minocicline di prolungare la durata della vita. Minociclina anche esteso la durata della vita di hlh-30 (tm1978) mutanti, ma meno del 45% osservato in N2. Questi risultati genetici, e la precedente scoperta che l’attivazione dell’autofagia nel già vecchio C. elegans è probabilmente dannosa (Wilhelm et al., 2017), sono coerenti con un meccanismo di minociclina che agisce indipendentemente dall’attivazione dell’autofagia. Tuttavia, poiché unc-51 è essenziale, l’attività residua che rimane nei mutanti unc-51(e369) non ci permette di trarre una conclusione definitiva.

Figura 2-figure supplemento 1.Riassunto dei dati sulla durata della vita per i ceppi portatori di mutazioni nei regolatori di stress, proteostasi, autofagia e risposte lisosomiali, relativi alla Figura 2.Riassunto dei dati sulla durata della vita per i ceppi portatori di mutazioni nei regolatori di stress, proteostasi, autofagia e risposte lisosomiali, relativi alla Figura 2.Nonostante inibisca l’UPRmt e la risposta allo shock termico negli adulti, la minociclina attiva l’UPRmt nelle larve e protegge dallo stress.(A) Timeline sperimentale per monitorare l’attivazione risposta allo stress di 5 giorni di età adulta C. elegans C. elegans GFP-reporter ceppi indotti da stressor e / o minociclina. Minociclina (100 μM) è stato aggiunto 2 ore prima di ogni stressor. (B) Trame Spargimento mostra l’induzione piega di induzione di fluorescenza GFP indotta da fattori di stress e / o il trattamento minociclina rispetto agli animali non trattati in 5 giorni di età adulti. Pretrattamento con minociclina sopprime l’attivazione risposta allo stress. Reporter: gst-4p::GFP (stress ossidativo), hsp-4p::GFP (ER UPR), hsp-6p::GFP (UPRmt) e hsp-16.2p::GFP (stress da calore). Le barre di errore mostrano la media ± S.D., ogni punto rappresenta un animale con tutti n > 300. Significato determinato dal t-test di Mann-Whitney. Almeno tre esperimenti indipendenti. (C) Immagini rappresentative microscopia a fluorescenza di 100 animali selezionati a caso per ogni condizione e ceppo. Stressori indicati a destra di ogni pannello. Le immagini per ogni ceppo sono state scattate in parallelo lo stesso giorno utilizzando impostazioni identiche. (D) Curve di sopravvivenza. Minociclina estende significativamente la durata della vita nei ceppi che portano mutazioni nei regolatori di stress e risposte di proteostasi. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 35 e 218. Totale di almeno tre esperimenti indipendenti per ceppo. E) Curve di sopravvivenza. Minociclina prolunga significativamente la durata della vita in due ceppi portatori di mutazioni nei regolatori delle vie lisosomiche e autofagiche. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 56 e 138. Totale di almeno tre esperimenti indipendenti per ceppo. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati fonte per tutti gli esperimenti sulla durata della vita in (D) ed (E) sono disponibili nella Figura 2. Dati fonte 1.10.7554/eLife.40314.007Figure 2 dati fonte 1.Summary of lifespan data for strains carrying mutations in regulators of stress, proteostasi, autofagia e risposte lisosomiali, relativi alla Figura 2.(A e B) Immagine in fluorescenza e timeline sperimentale (A) e quantificazione (B) per hsp-6p::induzione GFP da minociclina aggiunto a L3 larve L3. Mentre minociclina soppressa induzione GFP di tutti i giornalisti stress testato negli adulti, si attiva l’hsp-6p::GFP nelle larve. I dati sono rappresentati come media ±S.D. Significato determinato dal t-test di Mann-Whitney. (C) Sopravvivenza in funzione del tempo in ore per l’acqua o 100 μM minociclina trattata con minocicline di tipo selvatico N2 C. elegans sottoposto ad uno shock termico continuo di 35°C. I dati sono rappresentati come media ±S.E.M. Significato determinato dal test t di Mann-Whitney in ogni punto temporale. Totale di tre esperimenti indipendenti. (D) Sopravvivenza di acqua o 100 μM minociclina trattata con minociclina wild-type N2 C. elegans 24 ore dopo l’aggiunta di concentrazioni crescenti di paraquat aggiunto il giorno 5. I dati sono rappresentati come media ±S.E.M. Significato determinato dal Mann-Whitney t-test per ogni concentrazione di paraquat. Totale di tre esperimenti indipendenti. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo.

Figura 2-figure supplement 1.Nonostante inibisca l’UPRmt e la risposta allo shock termico negli adulti, la minociclina attiva l’UPRmt nelle larve e protegge dallo stress.(A e B) Immagine in fluorescenza e timeline sperimentale (A) e quantificazione (B) per hsp-6p::induzione GFP da minociclina aggiunto a L3 larve L3. Mentre minociclina soppresso induzione GFP di tutti i giornalisti stress testato negli adulti, si attiva l’hsp-6p::GFP nelle larve. I dati sono rappresentati come media ±S.D. Significato determinato dal t-test di Mann-Whitney. (C) Sopravvivenza in funzione del tempo in ore per l’acqua o 100 μM minociclina trattata con minocicline di tipo selvatico N2 C. elegans sottoposto ad uno shock termico continuo di 35°C. I dati sono rappresentati come media ±S.E.M. Significato determinato dal test t di Mann-Whitney in ogni punto temporale. Totale di tre esperimenti indipendenti. (D) Sopravvivenza di acqua o 100 μM minociclina trattata con minociclina wild-type N2 C. elegans 24 ore dopo l’aggiunta di concentrazioni crescenti di paraquat aggiunto il giorno 5. I dati sono rappresentati come media ±S.E.M. Significato determinato dal Mann-Whitney t-test per ogni concentrazione di paraquat. Totale di tre esperimenti indipendenti. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo.
La minociclina riduce l’etichettatura reattiva della cisteina al ribosoma
In contrasto con il minociclinico, l’estensione della durata della vita indotta in C. elegans dai paradigmi di longevità conservati, tra cui la restrizione alimentare, la ridotta attività mitocondriale, l’ablazione dei germi, la percezione sensoriale, la riduzione della segnalazione dell’insulina o l’inibizione di mTOR dipendono tutti da almeno uno dei fattori sopra testati (Steinkraus et al., 2008; Robida-Stubbs et al., 2012; Houtkooper et al., 2013; Lapierre et al., 2013; Weir et al., 2017). Per ottenere informazioni sul MOA della minociclina, abbiamo condotto il profilo proteico basato sull’attività utilizzando iodoacetamide (IA) come sonda, seguito da proteolisi isotopica tandem ortogonale (isoTOP-ABPP). L’IA si lega specificamente ai residui di cisteina reattiva, compresi i siti catalitici all’interno degli enzimi, i siti di modifica post-traslazionale, i siti di ossidazione della cisteina e altri tipi di domini normativi o funzionali attraverso il proteoma (Roberts et al., 2017). Abbiamo ipotizzato che il trattamento con minociclina avrebbe direttamente, legandosi vicino ad una cisteina reattiva, o indirettamente, modulando l’attività di un percorso, alterato l’etichettatura IA delle modifiche di cisteina reversibili e post-traslazionali.
Per determinare i cambiamenti indotti da minociclina nei modelli di etichettatura cisteina, 5 giorni di età adulta C. elegans sono stati trattati con acqua o minociclina per 3 giorni e il loro proteoma è stato successivamente etichettato con la sonda IA e analizzati dalla spettrometria di massa (Figura 3-figure supplemento 1A). L’analisi dei dati isoTOP-ABPP con il plugin Cytoscape ClueGO (Bindea et al., 2009), che consente la visualizzazione dei termini biologici per grandi gruppi di geni in una rete raggruppata funzionalmente, ha mostrato che il trattamento con minociclina ha ridotto l’etichettatura IA delle proteine coinvolte nella traduzione citoplasmatica, in particolare le proteine coinvolte nell’assemblaggio del ribosoma e nei processi metabolici dei peptidi (Figura 3A). Questi risultati hanno suggerito che la minociclina si rivolge direttamente o indirettamente alla traduzione citoplasmatica, un MOA coerente con l’estensione della durata della vita e che ha spiegato come la minociclina ha impedito l’induzione della SSP (Figura 2B,C) (Vellai et al., 2003; Kapahi et al., 2004; Kaeberlein et al., 2005; Hansen et al., 2007; Pan et al., 2007; Syntichaki et al., 2007; McQuary et al., 2016).
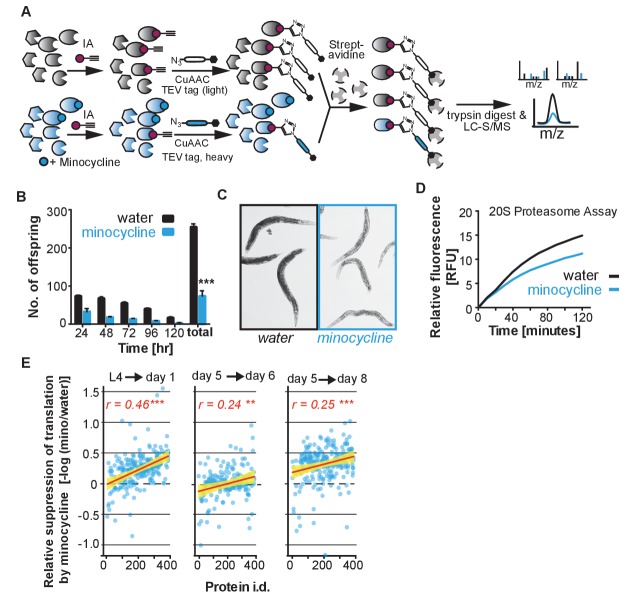
Figura 3-figure supplemento 1.Figura 3—supplemento alla figura 1. Minociclina attenua la traduzione in C. elegans.riepilogo dell’analisi isoTOP-ABPP.riepilogo dell’analisi isoTOP-ABPP.riepilogo dell’analisi isoTOP-ABPP.riepilogo dell’analisi isoTOP-ABPP.gli animali trattati con minociclina presentano fenotipi caratteristici dell’inibizione della traduzione, correlati alla figura 3.(A) Analisi di rete da parte di ClueGO dei risultati di isoTOP-ABPP corrispondenti ai cambiamenti di etichettatura delle proteine che diminuiscono con il trattamento minociclico. La dimensione del cerchio è proporzionale al numero di proteine identificate e il colore rappresenta il significato. Totale di tre esperimenti indipendenti. (B) Il grafico mostra il numero totale di figli per l’acqua o minociclina trattati C. elegans oltre 5 giorni dopo l’aggiunta di minociclina. Significato determinato dal Mann-Whitney t test n = 15. Totale di cinque esperimenti indipendenti. (C) Il grafico mostra la lunghezza di C. elegans in pixel in funzione dell’età in ore dopo l’aggiunta di minocicline. Significato determinato dal test t dello studente di Mann-Whitney n > 50. Totale di cinque esperimenti indipendenti. (D) I grafici mostrano i rapporti di intensità relativa di incorporazione 14N al totale 14N +15N proteine etichettate in acqua (cerchi neri) o minociclina trattati (cerchi blu) animali in funzione dell’abbondanza in animali trattati in acqua in diverse età. Lunghezza ed età dell’etichettatura indicate sopra ogni grafico. Totale di due esperimenti indipendenti. Significato determinato da un test t accoppiato. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati sorgente per gli esperimenti isoTOP-ABPP e gli esperimenti quantitativi di spettrometria di massa sono disponibili nella Figura 3 – dati sorgente 1 e 2, rispettivamente.10.7554/eLife.40314.010Cifre dati a 3 fonti 1.Riepilogo dell’analisi isoTOP-ABPP.10.7554/eLife.40314.011Figure 3-source data 2.Summary of15N-incorporation analysis.(A) Schema della strategia isoTOP-ABPP per rilevare i cambiamenti nelle interazioni minociclina, espressione o modifiche post-traslazionali in C. elegans lysate trattato e non trattato. (B) Stessi dati della Figura 3B, ma con indicazione del numero di figli prodotti al giorno, oltre al totale cumulativo. Significato determinato dal test di Mann-Whitney t. (C) Immagini in campo chiaro di adulti trattati con acqua e minociclina al secondo giorno di età adulta. (D) Quantificazione dell’attività del proteasoma 20S dall’estratto proteico di C. elegans trattato con acqua o minociclina. (E) Stessi dati della Figura 3D, ma analizzati per determinare la riduzione della piega nella traslazione causata da minociclina. I grafici tracciano l’effetto soppressivo del minociclina (-log10 [intensitymino/intensitywater]) in funzione dell’abbondanza di proteine (per rango dalla Figura 3D) con il coefficiente di correlazione r mostrato in alto a destra. Ogni cerchio corrisponde al rapporto di espressione -log10 [intensitymino/intensitywater] per una proteina. L’ombreggiatura gialla indica un intervallo di confidenza del 99%. Poiché la correlazione osservata si basa su un rapporto di espressione della stessa proteina in condizioni diverse, la correlazione non è spiegata da considerazioni tecniche come la maggiore capacità della spettrometria di massa di rilevare proteine più abbondanti. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.

Figura 3-figure supplemento 1.Figura 3—supplemento alla figura 1. La minociclina attenua la traduzione in C. elegans.Riepilogo dell’analisi isoTOP-ABPP.Riepilogo dell’analisi isoTOP-ABPP.Riepilogo dell’analisi isoTOP-ABPP.Riepilogo dell’analisi isoTOP-ABPP.Riepilogo dell’analisi isoTOP-ABPP.Gli animali trattati con minociclina mostrano fenotipi caratteristici dell’inibizione della traduzione, correlati alla figura 3.(A) Analisi di rete da parte di ClueGO dei risultati di isoTOP-ABPP corrispondenti ai cambiamenti di etichettatura delle proteine che diminuiscono con il trattamento con minociclina. La dimensione del cerchio è proporzionale al numero di proteine identificate e il colore rappresenta il significato. Totale di tre esperimenti indipendenti. (B) Il grafico mostra il numero totale di figli per l’acqua o minociclina trattati C. elegans oltre 5 giorni dopo l’aggiunta di minociclina. Significato determinato dal Mann-Whitney t test n = 15. Totale di cinque esperimenti indipendenti. (C) Il grafico mostra la lunghezza di C. elegans in pixel in funzione dell’età in ore dopo l’aggiunta di minocicline. Significato determinato dal test t dello studente di Mann-Whitney n > 50. Totale di cinque esperimenti indipendenti. (D) I grafici mostrano i rapporti di intensità relativa di incorporazione 14N al totale 14N +15N proteine etichettate in acqua (cerchi neri) o minociclina trattati (cerchi blu) animali in funzione dell’abbondanza in animali trattati in acqua in diverse età. Lunghezza ed età dell’etichettatura indicate sopra ogni grafico. Totale di due esperimenti indipendenti. Significato determinato da un test t accoppiato. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati sorgente per gli esperimenti isoTOP-ABPP e gli esperimenti quantitativi di spettrometria di massa sono disponibili nella Figura 3 – dati sorgente 1 e 2, rispettivamente.10.7554/eLife.40314.010Figure 3-source data 1.Summary of isoTOP-ABPP analysis.10.7554/eLife.40314.011Figure 3-source data 2.Summary of15N-incorporazione analisi.(A) Schema della strategia isoTOP-ABPP per rilevare i cambiamenti nelle interazioni minocicliniche, nell’espressione o nelle modifiche post-traslazionali in C. elegans lysate trattato e non trattato. (B) Stessi dati della Figura 3B, ma con indicazione del numero di figli prodotti al giorno, oltre al totale cumulativo. Significato determinato dal test di Mann-Whitney t. (C) Immagini in campo chiaro di adulti trattati con acqua e minociclina al secondo giorno di età adulta. (D) Quantificazione dell’attività del proteasoma 20S dall’estratto proteico di C. elegans trattato con acqua o minociclina. (E) Stessi dati della Figura 3D, ma analizzati per determinare la riduzione della piega nella traslazione causata da minociclina. I grafici tracciano l’effetto soppressivo del minociclina (-log10 [intensitymino/intensitywater]) in funzione dell’abbondanza di proteine (per rango dalla Figura 3D) con il coefficiente di correlazione r mostrato in alto a destra. Ogni cerchio corrisponde al rapporto di espressione -log10 [intensitymino/intensitywater] per una proteina. L’ombreggiatura gialla indica un intervallo di confidenza del 99%. Poiché la correlazione osservata si basa su un rapporto di espressione della stessa proteina in condizioni diverse, la correlazione non è spiegata da considerazioni tecniche come la maggiore capacità della spettrometria di massa di rilevare proteine più abbondanti. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.

Figura 3-figure supplemento 1.Gli animali trattati con minociclina mostrano fenotipi caratteristici dell’inibizione della traduzione, correlati alla Figura 3.(A) Schema della strategia isoTOP-ABPP per rilevare i cambiamenti nelle interazioni minociclina, espressione o modifiche post-traslazionale in C. elegans lisato trattato e non trattato. (B) Stessi dati della Figura 3B, ma con indicazione del numero di figli prodotti al giorno, oltre al totale cumulativo. Significato determinato dal test di Mann-Whitney t. (C) Immagini in campo chiaro di adulti trattati con acqua e minociclina al secondo giorno di età adulta. (D) Quantificazione dell’attività del proteasoma 20S dall’estratto proteico di C. elegans trattato con acqua o minociclina. (E) Stessi dati della Figura 3D, ma analizzati per determinare la riduzione della piega nella traslazione causata da minociclina. I grafici tracciano l’effetto soppressivo del minociclina (-log10 [intensitymino/intensitywater]) in funzione dell’abbondanza di proteine (per rango dalla Figura 3D) con il coefficiente di correlazione r mostrato in alto a destra. Ogni cerchio corrisponde al rapporto di espressione -log10 [intensitymino/intensitywater] per una proteina. L’ombreggiatura gialla indica un intervallo di confidenza del 99%. Poiché la correlazione osservata si basa su un rapporto di espressione della stessa proteina in condizioni diverse, la correlazione non è spiegata da considerazioni tecniche come la maggiore capacità della spettrometria di massa di rilevare proteine più abbondanti. Gli asterischi indicano un significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.
Minociclina attenua la traduzione dell’mRNA citoplasmatico in C. elegans
Il nostro prossimo obiettivo è quello di determinare l’effetto del trattamento minociclico sulla traduzione dell’mRNA citoplasmatico. Coerentemente con la riduzione della traduzione, il trattamento con minociclina ha ridotto la crescita e la riproduzione di C. elegans (Figura 3B,C; Figura 3-figure supplement 1B,C) (Hansen et al., 2007; Pan et al., 2007). Per quantificare la traduzione dell’mRNA in presenza o meno di minociclina, abbiamo misurato l’incorporazione di amminoacidi marcati 14N e 15N nel proteoma mediante spettrometria di massa quantitativa. In una prima serie di esperimenti, popolazioni sincronizzate di animali allo stadio larvale L4 sono stati trattati per 24 ore con acqua o minociclina e nutriti con batteri marcati 14N. Per quantificare le differenze nei livelli di proteine, uno standard 15N è stato aggiunto per determinare un 14N a (14N + 15N) rapporto di intensità. La maggior parte delle proteine (85%) nei campioni trattati con minociclina ha mostrato una significativa riduzione dell’intensità rispetto ai controlli, rivelando che gli animali trattati con minociclina contenevano ~ 24% in meno di proteine (Figura 3D,L4→ giorno 1). Questi effetti sul proteoma non erano probabilmente il risultato di una maggiore degradazione delle proteine, come minociclina ridotto piuttosto che una maggiore attività proteosomica (Figura 3-figure supplemento 1D). Almeno in adulti C. elegans questi dati erano incoerenti con un modello in cui minociclina selettivamente ridotto traduzione, come è stato dimostrato per ifg-1 (Rogers et al., 2011). I pochi fattori che hanno mostrato livelli di espressione più elevati negli animali trattati con minociclina non avevano alcun legame noto con la resistenza allo stress o hanno dimostrato di aumentare la durata della vita quando sono stati abbattuti dall’RNAi.
In una seconda serie di esperimenti, ci siamo avvalsi di una precedente osservazione che mostrava che i tassi di traduzione dell’mRNA in C. elegans diminuiscono con l’età (Gomez-Amaro et al., 2015). Quindi, se il minocicline ha agito attenuando la traduzione, il suo effetto sull’incorporazione del 14N nel proteoma dovrebbe dipendere dal tasso di traduzione e dovrebbe essere più piccolo negli animali adulti di 5 giorni rispetto alle giovani larve L4. Per testare questa previsione, abbiamo iniziato il trattamento con minociclina il 5° giorno di età adulta e raccolto la proteina 24 ore e 72 ore dopo. Come previsto, l’effetto della minociclina sulla traduzione dell’mRNA dipendeva dall’età degli animali. Mentre un trattamento minociclina 24 ore iniziato in L4 larve L4 ha ridotto la sintesi proteica del ~ 24% (Figura 3D, L4 → giorno 1), la stessa durata del trattamento iniziato in adulti di 5 giorni, ha mostrato solo una riduzione del ~ 11% sulla traduzione dell’mRNA (Figura 3D, giorno 5 → giorno 6). Tuttavia, un trattamento minociclico di 72 ore iniziato il 5° giorno ha permesso una traslazione dell’mRNA sufficiente per osservare una soppressione significativa del ~24% (Figura 3D, giorno 5 → giorno 8). Inoltre, abbiamo osservato l’effetto soppressivo di minociclina per correlare con l’abbondanza di proteine, sopprimendo la produzione di proteine altamente espresse in misura maggiore rispetto alle proteine poco espresse (Figura 3-figure supplemento 1E). Presi insieme, questi risultati mostrano che gli animali trattati con minociclina contengono meno proteine, coerente con una attenuazione della traduzione dell’mRNA.
Minociclina attenua la traduzione di mRNA nelle cellule umane
Per determinare se il trattamento con minociclina può anche ridurre la traduzione dell’mRNA nelle cellule umane, abbiamo misurato la traduzione basale dell’mRNA monitorando l’incorporazione di [35S]-metionina / cisteina nelle cellule HeLa non trattate e trattate con minociclina. Un pretrattamento di 6 ore con minociclina ha ridotto la traduzione di mRNA di ~ 30% a 100 μM come monitorato da [35S]-metionina / cisteina incorporazione di [35S]-metionina / cisteina (Figura 4A; Figura 4-figure supplemento 1A). Questo esperimento è stato ripetuto in murino NIH 3T3 cellule murine con risultati simili (Figura 4-figure supplemento 1B). L’entità dell’effetto è stato simile a quello che abbiamo osservato in C. elegans (Figura 3D), ma meno dell’effetto del cicloesimmide (Figura 4A). Poiché la biodisponibilità del minocicloro è vicina al 100%, le concentrazioni in vivo e in vitro dovrebbero essere simili (Garrido-Mesa et al., 2013).
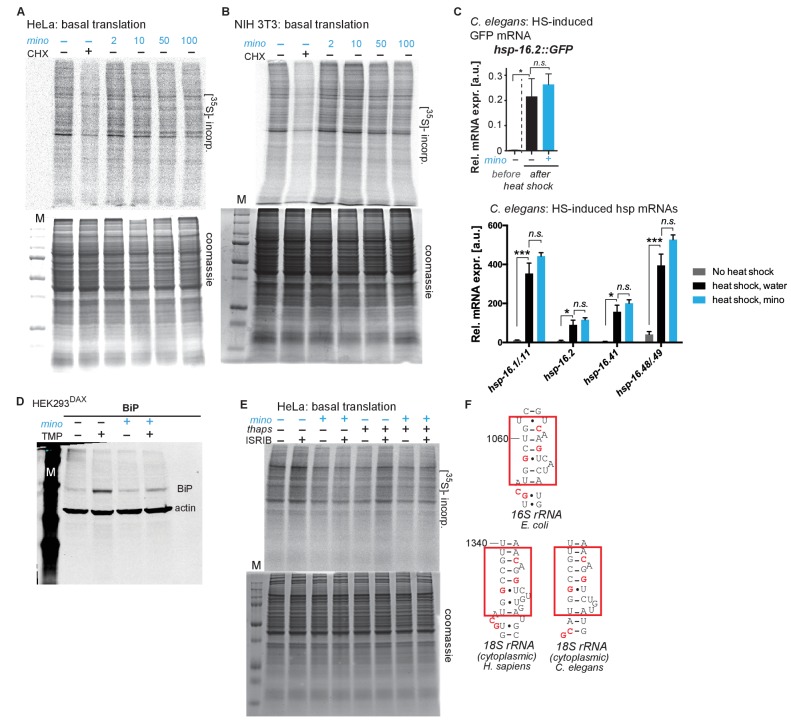
Figura 4-figure supplement 1.Minociclina attenua la traduzione e riduce il carico ribosomiale di mRNA altamente tradotti.Minociclina sopprime l’incorporazione 35S in paradigmi multipli.(A) Autoradiografo (in alto) il monitoraggio dell’incorporazione 35S su 1 ora di cellule HeLa trattate per 6 ore con cicloeossimide o concentrazioni crescenti di minociclina. Coomassie gel (in basso) è mostrato come un controllo di carico. Totale di quattro esperimenti indipendenti. (B) Analisi del profilo del polisoma di 12 ore di acqua (nero) o minociclina trattata (blu) cellule HeLa. Polisomi ad alto peso molecolare (P) e monosomi 80S (M) regioni sono ombreggiate in grigio. Un aumento dei monosomi e una diminuzione dei polisomi ad alto peso molecolare sono indicativi dell’attenuazione della traduzione. Totale di tre esperimenti indipendenti. (C) Immunoblots sondaggio per HSP60 e HSP70 espressione di HSP60 e HSP70 di acqua o minociclina trattati (100 μM) cellule HeLa dopo un 1 ora, 43 ° C shock termico. Totale di tre esperimenti indipendenti. (D) qRT-PCR analisi qRT-PCR di HSP60 e HSP70 espressione mRNA in cellule HeLa trattati con o senza minociclina (100 μM) prima e dopo un 1 ora, 43 ° C shock termico. Il trattamento con minociclina è stato avviato 12 ore prima dello shock termico. I dati sono rappresentati come media ± S.E.M. Significato determinato dal t-test di Mann-Whitney. Totale di tre esperimenti indipendenti. (E) Immunoblots sondaggio per l’espressione BiP di acqua o minociclina trattata (100 μM) cellule HEK293DAX progettato per consentire l’attivazione UPRER da trattamento trimethoprim (TMP). Acqua o 100 μM minociclina è stato aggiunto da solo o in combinazione con 20 uM trimetoprim per 12 ore prima della lisi delle cellule. I campioni sono stati normalizzati dalla concentrazione di RNA. Totale di tre esperimenti indipendenti. (F) Il grafico a barre mostra il cambiamento nella formazione di aggregati proteici, come misurato dal saggio ProteoStat, prima e dopo un 1 ora, 43 ° C shock termico in controllo (nero) e minociclina trattati (blu) cellule HeLa. I dati sono rappresentati come media ± S.E.M. Significato determinato dal t-test di Mann-Whitney. Totale di quattro esperimenti indipendenti. (G) Autoradiografo (in alto) il monitoraggio 35S incorporazione 35S di cellule HeLa trattate con l’inibitore ISR ISRIB (200 nM), minociclina (100 μM), thapsigargin (thaps, 200 nM) e una combinazione dei tre. Coomassie gel (in basso) è mostrato come un controllo di carico. Totale di due esperimenti indipendenti. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. Figura 5. Figura 5. Estensione della durata della vita per minociclina dipende dalla traduzione citoplasmatica.(A) I gel completi sono mostrati dalla Figura 4A. Autoradiografo (in alto) il monitoraggio 35S incorporazione 35S di cellule HeLa trattati con cicloesossimide o aumentando le concentrazioni di minociclina. Coomassie gel (in basso) mostrato come controllo del carico. (B) Autoradiografo (in alto) monitoraggio 35S incorporazione 35S di cellule NIH 3T3 trattate con cicloeossimide o concentrazioni crescenti di minociclina. Coomassie gel (in basso) è mostrato come controllo di carico. Totale di 3 esperimenti indipendenti. (C) qRT-PCR quantificazione qRT-PCR di mRNA GFP relativi mRNA (in alto) o mRNA hsp (in basso) espressione da non trattati o trattati con minociclina (100 μM) hsp-16.2p::GFP C. elegans prima e dopo un 1,5 ore, 35 ° C shock termico. Mentre lo shock termico non riesce a indurre la fluorescenza GFP in hsp-16.2p::GFP reporter animali in presenza di minociclina (Figura 2B,C), induce ancora l’mRNA GFP, rivelando l’effetto di minociclina sulla traduzione. Significato determinato dal test t dello studente di Mann-Whitney. n = 1500 animali per condizione. Totale di tre esperimenti indipendenti. (D) Gel completo mostrato dalla Figura 4E. Immunoblots sondaggio per l’espressione BiP di controllo o minociclina trattata (100 μM) cellule HEK293DAX progettato per consentire l’attivazione UPRER da trattamento trimethoprim (TMP). (E) Gel completo mostrato dalla Figura 4G. Autoradiografo (in alto) il monitoraggio 35S incorporazione di cellule HeLa trattati con l’inibitore ISRIB ISRIB, minociclina, thapsigargin (thaps) e una combinazione dei tre. Coomassie gel (in basso) è mostrato come un controllo di carico. (F) Confronto del noto sito di legame tetraciclina in elica 34 (h34) dell’E. coli 16S rRNA con l’rRNA 18S di H. sapiens e C. elegans. I quadrati rossi indicano la regione di legame della tetraciclina con i residui interagenti evidenziati in rosso. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.
Per ottenere ulteriori informazioni su come minociclina riduce la traduzione di mRNA, abbiamo registrato profili polisomici da cellule HeLa non trattate e minocicline trattate. Il trattamento con minociclina ha causato un aumento pronunciato del picco del monosoma 80S (M), indicativo dell’inibizione della traduzione in stallo (Figura 4B). Inoltre, ha mostrato una riduzione sproporzionatamente forte della frazione pesante del polisoma (P), suggerendo che il minociclinico ha ridotto il carico ribosomiale, il numero di ribosomi per mRNA, di mRNA altamente tradotti. Così, la riduzione osservata del carico ribosomiale da parte del minociclinico si traduce in una sostanziale attenuazione degli mRNA altamente tradotti. L’effetto preferenziale sulla frazione polisomica pesante (P) ha fornito una potenziale spiegazione per il maggiore effetto inibitorio della minociclina sulle proteine altamente espresse che abbiamo osservato nell’esperimento di etichettatura metabolica C. elegans (Figura 3-figure supplement 1E).
In caso di shock termico, la traduzione mediata dal polisoma permette alle cellule di sintetizzare rapidamente grandi quantità di chaperon come HSP60 e HSP70. Se il minocicline attenua la traduzione polisomo-mediata dovrebbe sopprimere sostanzialmente l’espressione indotta da shock termico di HSP60 e HSP70 alla proteina ma non a livello di mRNA. Per testare questo, abbiamo pretrattato le cellule HeLa con acqua o minociclina e le abbiamo sottoposte a shock termico per misurare i livelli di espressione della proteina HSP60 e HSP70 da western blot. Come previsto, le cellule HeLa hanno fortemente indotto l’espressione di HSP60 e HSP70 in risposta ad uno shock termico, mentre il pretrattamento con minociclina lo ha ridotto drasticamente (Figura 4C). Tuttavia, minociclina solo ridotto l’espressione di HSP60 e HSP70 a livello proteico e non ha avuto alcun effetto sull’induzione dei loro mRNA (Figura 4D). Allo stesso modo, abbiamo rivisitato C. elegans e confermato che minociclina soppresso l’attivazione di hsp-16.2p::GFP a livello proteico, ma non a livello di mRNA (Figura 2B; Figura 4-figure supplemento 1C). Così, le cellule trattate con minociclina attivano le risposte di stress a livello di mRNA, ma non sono in grado di tradurle rapidamente in proteine, poiché la minociclina attenua fortemente la traduzione mediata dal polisoma.
Per estendere questi risultati ad altre risposte di stress come la UPRER, abbiamo ripetuto questi esperimenti utilizzando cellule HEK293DAX. HEK293DAX cellule HEK293DAX consentono l’attivazione ligand-indotta della UPRER in assenza di stress, in quanto esprimono l’attivatore trascrizionale UPRER ATF6 fuso a mutante destabilizzato diidrofolato reduttasi (DHFR). Poiché il dominio DHFR è in gran parte dispiegato, la proteina di fusione ATF6-DHFR è continuamente degradata dal proteasoma. L’aggiunta di trimetoprim (TMP) stabilizza la fusione ATF6-DHFR, portando ad una espressione dose-dipendente del chaperone BiP (Shoulders et al., 2013). Come previsto, il trattamento TMP ha indotto drammaticamente l’espressione BiP che è stata completamente soppressa dal co-trattamento minociclina (Figura 4E; Figura 4-figure supplement 1D). Questi risultati mostrano che la minociclina sopprime fortemente l’attivazione di risposte multiple di stress a livello di traduzione sia in C. elegans che nelle cellule umane, indipendentemente dal fatto che l’espressione sia stata indotta da stress o da un ligando artificiale come la TMP.
Come abbiamo fatto prima in C. elegans, abbiamo chiesto se la soppressione delle risposte di stress da minociclina ha compromesso la capacità di una cellula di affrontare lo stress da calore e l’aggregazione delle proteine (Figura 1-figure supplement 1G; Figura 2-figure supplement 1C). Utilizzando il colorante specifico per l’aggregazione ProteoStat, abbiamo misurato l’aggregazione delle proteine nelle cellule HeLa prima e dopo uno shock termico di 1 ora. La fluorescenza indotta dall’aggregazione proteica è aumentata solo nelle cellule di controllo in seguito a shock termico (~ 12%), ma non nelle cellule pretrattate con minociclina (Figura 4F). Così, come già osservato in C. elegans, il trattamento con minociclina protegge dall’aggregazione proteica indotta da shock termico nonostante la soppressione dell’attivazione della risposta allo shock termico (Figura 1-figure supplement 1G; Figura 2-figure supplement 1C).

Figura 4-figure supplemento 1.Minociclina attenua la traduzione e riduce il carico ribosomiale di mRNA altamente tradotti.Minociclina sopprime l’incorporazione 35S in paradigmi multipli.(A) Autoradiografo (in alto) il monitoraggio dell’incorporazione 35S su 1 ora di cellule HeLa trattate per 6 ore con cicloeossimide o concentrazioni crescenti di minociclina. Coomassie gel (in basso) è mostrato come un controllo di carico. Totale di quattro esperimenti indipendenti. (B) Analisi del profilo del polisoma di 12 ore di acqua (nero) o minociclina trattata (blu) cellule HeLa. Polisomi ad alto peso molecolare (P) e monosomi 80S (M) regioni sono ombreggiate in grigio. Un aumento dei monosomi e una diminuzione dei polisomi ad alto peso molecolare sono indicativi dell’attenuazione della traduzione. Totale di tre esperimenti indipendenti. (C) Immunoblots sondaggio per HSP60 e HSP70 espressione di HSP60 e HSP70 di acqua o minociclina trattati (100 μM) cellule HeLa dopo un 1 ora, 43 ° C shock termico. Totale di tre esperimenti indipendenti. (D) qRT-PCR analisi qRT-PCR di HSP60 e HSP70 espressione mRNA in cellule HeLa trattati con o senza minociclina (100 μM) prima e dopo un 1 ora, 43 ° C shock termico. Il trattamento con minociclina è stato avviato 12 ore prima dello shock termico. I dati sono rappresentati come media ± S.E.M. Significato determinato dal t-test di Mann-Whitney. Totale di tre esperimenti indipendenti. (E) Immunoblots sondaggio per l’espressione BiP di acqua o minociclina trattata (100 μM) cellule HEK293DAX progettato per consentire l’attivazione UPRER da trattamento trimethoprim (TMP). Acqua o 100 μM minociclina è stato aggiunto da solo o in combinazione con 20 uM trimetoprim per 12 ore prima della lisi delle cellule. I campioni sono stati normalizzati dalla concentrazione di RNA. Totale di tre esperimenti indipendenti. (F) Il grafico a barre mostra il cambiamento nella formazione di aggregati proteici, come misurato dal saggio ProteoStat, prima e dopo un 1 ora, 43 ° C shock termico in controllo (nero) e minociclina trattati (blu) cellule HeLa. I dati sono rappresentati come media ± S.E.M. Significato determinato dal t-test di Mann-Whitney. Totale di quattro esperimenti indipendenti. (G) Autoradiografo (in alto) il monitoraggio 35S incorporazione 35S di cellule HeLa trattate con l’inibitore ISR ISRIB (200 nM), minociclina (100 μM), thapsigargin (thaps, 200 nM) e una combinazione dei tre. Coomassie gel (in basso) è mostrato come un controllo di carico. Totale di due esperimenti indipendenti. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo. Figura 5. Figura 5. Estensione della durata della vita per minociclina dipende dalla traduzione citoplasmatica.(A) Gel completo mostrato dalla Figura 4A. Autoradiografo (in alto) il monitoraggio 35S incorporazione 35S di cellule HeLa trattate con cicloeossimide o aumentando le concentrazioni di minociclina. Coomassie gel (in basso) mostrato come controllo del carico. (B) Autoradiografo (in alto) monitoraggio 35S incorporazione 35S di cellule NIH 3T3 trattate con cicloeossimide o concentrazioni crescenti di minociclina. Coomassie gel (in basso) è mostrato come controllo di carico. Totale di 3 esperimenti indipendenti. (C) qRT-PCR quantificazione qRT-PCR di mRNA GFP relativi mRNA (in alto) o mRNA hsp (in basso) espressione da non trattati o trattati con minociclina (100 μM) hsp-16.2p::GFP C. elegans prima e dopo un 1,5 ore, 35 ° C shock termico. Mentre lo shock termico non riesce a indurre la fluorescenza GFP in hsp-16.2p::GFP reporter animali in presenza di minociclina (Figura 2B,C), induce ancora l’mRNA GFP, rivelando l’effetto di minociclina sulla traduzione. Significato determinato dal test t dello studente di Mann-Whitney. n = 1500 animali per condizione. Totale di tre esperimenti indipendenti. (D) Gel completo mostrato dalla Figura 4E. Immunoblots sondaggio per l’espressione BiP di controllo o minociclina trattata (100 μM) cellule HEK293DAX progettato per consentire l’attivazione UPRER da trattamento trimethoprim (TMP). (E) Gel completo mostrato dalla Figura 4G. Autoradiografo (in alto) il monitoraggio 35S incorporazione di cellule HeLa trattati con l’inibitore ISRIB ISRIB, minociclina, thapsigargin (thaps) e una combinazione dei tre. Coomassie gel (in basso) è mostrato come un controllo di carico. (F) Confronto del noto sito di legame tetraciclina in elica 34 (h34) dell’E. coli 16S rRNA con l’rRNA 18S di H. sapiens e C. elegans. I quadrati rossi indicano la regione di legame della tetraciclina con i residui interagenti evidenziati in rosso. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.

Figura 4-figure supplement 1.Il minociclinico sopprime l’incorporazione del 35S in più paradigmi.(A) Gel completo mostrato dalla Figura 4A. Autoradiografo (in alto) il monitoraggio 35S incorporazione di cellule HeLa trattati con cicloesimide o aumentando le concentrazioni di minociclina. Coomassie gel (in basso) mostrato come controllo del carico. (B) Autoradiografo (in alto) monitoraggio 35S incorporazione 35S di cellule NIH 3T3 trattate con cicloeossimide o concentrazioni crescenti di minociclina. Coomassie gel (in basso) è mostrato come controllo di carico. Totale di 3 esperimenti indipendenti. (C) qRT-PCR quantificazione qRT-PCR di mRNA GFP relativi mRNA (in alto) o mRNA hsp (in basso) espressione da non trattati o trattati con minociclina (100 μM) hsp-16.2p::GFP C. elegans prima e dopo un 1,5 ore, 35 ° C shock termico. Mentre lo shock termico non riesce a indurre la fluorescenza GFP in hsp-16.2p::GFP reporter animali in presenza di minociclina (Figura 2B,C), induce ancora l’mRNA GFP, rivelando l’effetto di minociclina sulla traduzione. Significato determinato dal test t dello studente di Mann-Whitney. n = 1500 animali per condizione. Totale di tre esperimenti indipendenti. (D) Gel completo mostrato dalla Figura 4E. Immunoblots sondaggio per l’espressione BiP di controllo o minociclina trattata (100 μM) cellule HEK293DAX progettato per consentire l’attivazione UPRER da trattamento trimethoprim (TMP). (E) Gel completo mostrato dalla Figura 4G. Autoradiografo (in alto) il monitoraggio 35S incorporazione di cellule HeLa trattati con l’inibitore ISRIB ISRIB, minociclina, thapsigargin (thaps) e una combinazione dei tre. Coomassie gel (in basso) è mostrato come un controllo di carico. (F) Confronto del noto sito di legame tetraciclina in elica 34 (h34) dell’E. coli 16S rRNA con l’rRNA 18S di H. sapiens e C. elegans. I quadrati rossi indicano la regione di legame della tetraciclina con i residui interagenti evidenziati in rosso. Gli asterischi indicano il significato *<0.05, **<0.01, ***<0.001, n.s. non significativo.
Minociclina attenua la traduzione indipendentemente dalla PVR
L’attivazione SSP porta alla fosforilazione del fattore di iniziazione della traduzione eucariotica eIF2α, inibendo globalmente l’iniziazione della traduzione di tutti gli mRNA tranne pochi mRNA, generalmente chiamata risposta integrata allo stress (ISR) (Pakos-Zebrucka et al., 2016). Il minociclinico potrebbe attenuare la traduzione attraverso l’attivazione dell’ISR o mirando direttamente al ribosoma citoplasmatico per interferire con la traduzione. Per distinguere tra queste possibilità, abbiamo fatto uso dell’inibitore della PVR ISRIB che impedisce l’inibizione traslazionale mediata dalla fosforilazione eIF2α (Sidrauski et al., 2015). Se la minociclina attiva l’ISR per attenuare la traduzione, il co-trattamento con ISRIB dovrebbe ripristinare la normale traduzione anche in presenza di minociclina. Le cellule HeLa sono state trattate con ISRIB, minociclina o entrambi gli agenti. Il monitoraggio della traduzione con [35S]-metionina/cisteina incorporata ha mostrato che il co-trattamento con ISRIB non ha compromesso la capacità della minociclina di attenuare la traduzione (Figura 4G; Figura 4-figure supplement 1E). Al contrario, l’attenuazione della traduzione da parte di thapsigargin, un induttore noto dell’ISR, è stata ripristinata dal co-trattamento con ISRIB (Sidrauski et al., 2015). Così, la minociclina attenua la traduzione con un meccanismo indipendente dalla PVR. Confrontando l’rRNA 16S batterico con l’rRNA 18S citoplasmatico di C. elegans e H. sapiens ha rivelato un sito di legame tetraciclina conservato in elica 34 (h34) (Figura 4-figure supplement 1F) (Brodersen et al., 2000). Un recente studio del gruppo Meyers ha identificato la doxiciciclina per legarsi direttamente alle sottostrutture chiave 18S rRNA del ribosoma citoplasmatico e ha mostrato diverse tetracicline per discriminare tra i diversi siti di legame 18S rRNA (Caballero et al., 2011; Mortison et al., 2018). Così, abbiamo concluso che la minociclina prende di mira direttamente l’rRNA 18S, portando ai ribosomi osservati in stallo (picco 80S) e all’attenuazione della traduzione (Figura 4A,B).
Il minociclinico prolunga la durata della vita attenuando la traslazione
La traduzione di mRNA è essenziale e i mutanti “nulli” privi di rRNA 18S non sono fattibili, impedendoci di testare direttamente la necessità della traduzione di mRNA per la longevità indotta dai minociclinici. In contrasto con i nostri studi su C. elegans e cellule umane, studi che indagano gli effetti degli antibiotici tetraciclici in S. cerevisiae hanno dimostrato che essi attenuano solo la traduzione mitocondriale ma non quella citoplasmatica. Coerentemente con un modello in cui la minociclina deve attenuare la traduzione citoplasmatica per prolungare la durata della vita, la minociclina non ha prolungato la durata della vita replicativa in S. cerevisiae, e a concentrazioni più elevate l’ha ridotta (Figura 5A) (Clark-Walker e Linnane, 1966; Caballero et al., 2011; McCormick et al., 2015).
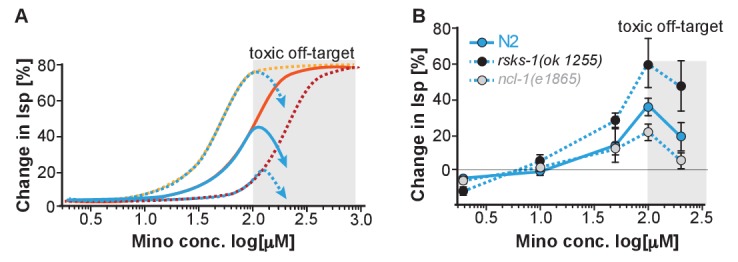
Figura 5-figure supplement 1.(A) Durata della vita replicante di S. cerevisae trattati con concentrazioni crescenti di minociclina.riepilogo dei dati sulla durata della vita di rsks-1 e ncl-1, relativi alla Figura 5 e Figura 5-figure supplement 1.Summary of rsks-1 and ncl-1 lifespan data, relativi alla Figura 5 e Figura 5-figure supplement 1.Evidence for a toxic off-target at higher minocycline concentrations.I dati sono stati analizzati utilizzando il test Wilcoxon rank-sum. (B) Rappresenta gli spostamenti della curva di risposta della dose prevista per i mutanti con tassi di traslazione inferiori (curva nera) o superiori (curva grigia) trattati con minociclina rispetto a quelli del tipo wild-type (curva blu). Poiché la traduzione è essenziale, i mutanti di traduzione devono mantenere una certa attività di traduzione, anche se inferiore a quella di tipo selvaggio. Quindi, nei mutanti con una traduzione ridotta come rsks-1, dovrebbe essere necessaria meno minociclina per abbassare in modo ottimale la traduzione e per aumentare la durata della vita con conseguente curva dose-risposta spostata a sinistra. Se la minociclina punta all’rRNA 18S, un eccesso di rRNA e un aumento della traduzione come nei mutanti ncl-1 dovrebbe risultare in una curva dose-risposta spostata a destra. (C) Le curve di risposta della dose mostrano la variazione % della durata della vita in funzione dell’aumento delle concentrazioni di minociclina per i mutanti N2, rsks-1(ok1255) e ncl-1(e1865). Totale di quattro esperimenti indipendenti eseguiti. I dati sono rappresentati come media ± S.E.M. (D) Curve di sopravvivenza per i mutanti rsks-1(ok1255) trattati con acqua o minociclina (100 μM) C. elegans. Almeno quattro esperimenti indipendenti eseguiti. (E) Curve di sopravvivenza per acqua o minociclina (100 μM) trattati ncl-1(e1865) C. elegans mutanti. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 42 e 87, per un totale di quattro esperimenti indipendenti. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati fonte per gli esperimenti di durata della vita di N2, rsks-1 e ncl-1 sono disponibili nella Figura 5 – dati fonte 1.10.7554/eLife.40314.016Figure 5-source data 1.Summary of rsks-1 and ncl-1 lifespan data, related to Figure 5 and Figure 5-figure supplement 1.(A) Rappresenta l’effetto previsto sulla variazione della durata della vita di un fuori obiettivo tossico indipendente dalla traslazione. Un’eccessiva attenuazione della traslazione da parte di un eccesso di minociclina è probabilmente dannosa. Tuttavia, la concentrazione di minociclina per raggiungere questo effetto dannoso dovrebbe dipendere dal tasso di traslazione endogena di ogni ceppo e dovrebbe verificarsi a concentrazioni più basse nei mutanti rsks-1 e a concentrazioni più alte nei mutanti ncl-1. L’effetto nocivo osservato, tuttavia, si verifica in tutti i ceppi, N2, ncl-1, rsks-1 alla stessa concentrazione di minociclina, indipendentemente dal loro tasso di traduzione endogena. È quindi più coerente con l’impegno di un fuori bersaglio tossico non correlato alla traduzione. (B) Come nella figura 5C, che mostra lo spostamento delle curve dose-risposta e l’effetto tossico off-target tra 100 e 200 μM minociclina (grigio sfumato). Si noti inoltre che 200 μM minociclina M non causano un calo di estensione della durata della vita nei mutanti skn-1 (Figura 2), ma estendere ulteriormente la durata della vita suggerendo che l’espressione della tossicità off-target può essere regolata da skn-1.
Per collegare più direttamente l’inibizione della traslazione da parte della minociclina al suo effetto sulla longevità, abbiamo ragionato che se la minociclina estende la durata della vita riducendo la traslazione direttamente al ribosoma, la sua curva dose-risposta dovrebbe essere spostata a sinistra nei mutanti con traslazione già ridotta e spostata a destra nei mutanti con traslazione aumentata (Figura 5B).
Per un mutante con traslazione ridotta, abbiamo usato il ceppo rsks-1(ok1255) (Hansen et al., 2007; Pan et al., 2007). Il ceppo rsks-1(ok1255) porta una cancellazione nell’omologo della chinasi S6, un regolatore dell’inizio della traduzione. I dati genetici hanno dimostrato che rsks-1 prolunga la durata della vita di un meccanismo dipendente da hlh-30, e quindi diverso dai meccanismi alla base della minociclina (Lapierre et al., 2013). Misurando le concentrazioni di proteine con il saggio di Bradford nei giovani adulti, prima della generazione delle uova, abbiamo ulteriormente confermato l’indipendenza dei due meccanismi. Nei mutanti rsks-1, la concentrazione di proteine è stata ridotta del 18% (±8%) rispetto a N2. Il trattamento dei mutanti rsks-1 (ok1255) con minociclina ha avuto un effetto additivo e ha ulteriormente ridotto la concentrazione proteica di un ulteriore 26%, con conseguente riduzione totale del 44% (±8%, p=0,0018, Dunett) rispetto a N2. Così, minociclina e la mutazione rsks-1 mutazioni ulteriormente ridotto le concentrazioni proteiche. Per un mutante con una maggiore traduzione, abbiamo usato il ceppo ncl-1 (e1865) (Frank e Roth, 1998). I ceppi portatori di mutazioni in ncl-1 esprimono il doppio del 18S rRNA e producono il 22% di proteine in più rispetto agli animali selvatici. Come il nostro e i dati di Mortison (Mortison et al., 2018) suggeriscono che la minociclina si lega direttamente al C. elegans 18S rRNA, un aumento di due volte nel livello 18S rRNA e l’aumento associato di traduzione dovrebbe spostare la curva dose-risposta a destra e diminuire l’effetto della minociclina sulla durata della vita (Figura 5B).
Come illustrato nella Figura 5C, l’estensione della durata della vita da parte del minociclinico è stata ridotta nei mutanti ncl-1(e1865) con una curva dose-risposta spostata a destra (Figura 5C,E). Al contrario, nei mutanti rsks-1(ok1255) a lunga vita, abbiamo osservato una curva dose-risposta spostata a sinistra (Figura 5C,D). Quindi, nei mutanti rsks-1(ok1255) è stato richiesto meno minociclina per raggiungere lo stesso livello di estensione della durata della vita rispetto agli animali wild-type, in quanto i livelli di traslazione sono già ridotti dalla mutazione. Tuttavia, due aspetti delle curve dose-risposta erano inattesi. In primo luogo, il minociclinico ha aumentato la durata della vita di una media di oltre il 60% e fino al 98% nel già longevo rsks-1 mutante. In secondo luogo, le curve dose-risposta per tutti e tre i ceppi hanno iniziato a diminuire tra 100 e 200 μM (Figura 5-figure supplement 1). In teoria, questo declino potrebbe essere causato da un’eccessiva inibizione della traduzione, che a un certo punto diventa dannosa. Tuttavia, poiché tutti i ceppi, indipendentemente dal fatto che traducano a tassi wild-type (N2), tassi più bassi (rsks-1) o tassi più alti (ncl-1) mostrano questo calo alla stessa concentrazione, questo effetto è improbabile il risultato di una troppa inibizione della traduzione, ma più probabilmente il risultato di una tossicità fuori obiettivo (Figura 5-figure supplement 1).

Figura 5-figure supplemento 1.(A) Durata della vita replicante di S. cerevisae trattati con concentrazioni crescenti di minociclina.riepilogo dei dati di durata della vita rsks-1 e ncl-1, relativi alla Figura 5 e Figura 5-figure supplement 1.Summary dei dati di durata della vita rsks-1 e ncl-1, relativi alla Figura 5 e Figura 5-figure supplement 1.Evidence for a toxic off-target a più alte concentrazioni di minociclina.I dati sono stati analizzati utilizzando il test Wilcoxon rank-sum. (B) Rappresenta gli spostamenti della curva di risposta della dose prevista per i mutanti con tassi di traslazione più bassi (curva nera) o più alti (curva grigia) trattati con minociclina rispetto al tipo wild-type (curva blu). Poiché la traduzione è essenziale, i mutanti di traduzione devono mantenere una certa attività di traduzione, anche se inferiore a quella di tipo selvaggio. Quindi, nei mutanti con una traduzione ridotta come rsks-1, dovrebbe essere necessaria meno minociclina per abbassare in modo ottimale la traduzione e per aumentare la durata della vita con conseguente curva dose-risposta spostata a sinistra. Se la minociclina punta all’rRNA 18S, un eccesso di rRNA e un aumento della traduzione come nei mutanti ncl-1 dovrebbe risultare in una curva dose-risposta spostata a destra. (C) Le curve di risposta della dose mostrano la variazione % della durata della vita in funzione dell’aumento delle concentrazioni di minociclina per i mutanti N2, rsks-1(ok1255) e ncl-1(e1865). Totale di quattro esperimenti indipendenti eseguiti. I dati sono rappresentati come media ± S.E.M. (D) Curve di sopravvivenza per i mutanti rsks-1(ok1255) trattati con acqua o minociclina (100 μM) C. elegans. Almeno quattro esperimenti indipendenti eseguiti. (E) Curve di sopravvivenza per acqua o minociclina (100 μM) trattati ncl-1(e1865) C. elegans mutanti. Significato statistico determinato dal test log-rank. Numero di animali n compreso tra 42 e 87, per un totale di quattro esperimenti indipendenti. Gli asterischi indicano la significatività *<0.05, **<0.01, ***<0.001, n.s. non significativo. I dati fonte per gli esperimenti di durata della vita di N2, rsks-1 e ncl-1 sono disponibili nella Figura 5 – dati fonte 1.10.7554/eLife.40314.016Figure 5-source data 1.Summary of rsks-1 and ncl-1 lifespan data, related to Figure 5 and Figure 5-figure supplement 1.(A) Rappresenta l’effetto previsto sulla variazione della durata della vita di un fuori obiettivo tossico indipendente dalla traslazione. Un’eccessiva attenuazione della traslazione da parte di un eccesso di minociclina è probabilmente dannosa. Tuttavia, la concentrazione di minociclina per raggiungere questo effetto dannoso dovrebbe dipendere dal tasso di traslazione endogena di ogni ceppo e dovrebbe verificarsi a concentrazioni più basse nei mutanti rsks-1 e a concentrazioni più alte nei mutanti ncl-1. L’effetto nocivo osservato, tuttavia, si verifica in tutti i ceppi, N2, ncl-1, rsks-1 alla stessa concentrazione di minociclina, indipendentemente dal loro tasso di traduzione endogena. È quindi più coerente con l’impegno di un fuori bersaglio tossico non correlato alla traduzione. (B) Come nella figura 5C, che mostra lo spostamento delle curve dose-risposta e l’effetto tossico off-target tra 100 e 200 μM minociclina (grigio sfumato). Si noti inoltre che 200 μM minociclina M non causano un calo di estensione della durata della vita nei mutanti skn-1 (Figura 2), ma estendere ulteriormente la durata della vita suggerendo che l’espressione della tossicità off-target può essere regolata da skn-1.

Figura 5-figure supplement 1.Prove di un fuori bersaglio tossico a concentrazioni di minocicline più elevate.(A) Rappresenta l’effetto previsto sul cambiamento della durata della vita di un off-target tossico indipendente dalla traslazione. Un’eccessiva attenuazione della traslazione da parte di troppa minociclina è probabilmente dannosa. Tuttavia, la concentrazione di minociclina per raggiungere questo effetto dannoso dovrebbe dipendere dal tasso di traslazione endogena di ogni ceppo e dovrebbe verificarsi a concentrazioni più basse nei mutanti rsks-1 e a concentrazioni più alte nei mutanti ncl-1. L’effetto nocivo osservato, tuttavia, si verifica in tutti i ceppi, N2, ncl-1, rsks-1 alla stessa concentrazione di minociclina, indipendentemente dal loro tasso di traduzione endogena. È quindi più coerente con l’impegno di un fuori bersaglio tossico non correlato alla traduzione. (B) Come nella figura 5C, che mostra lo spostamento delle curve dose-risposta e l’effetto tossico off-target tra 100 e 200 μM minociclina (grigio sfumato). Si noti inoltre che 200 μM minociclina M non causano un calo di estensione della durata della vita nei mutanti skn-1 (Figura 2), ma estendere ulteriormente la durata della vita suggerendo che l’espressione della tossicità off-target può essere regolata da skn-1.
Discussione
Il miglioramento dell’aggregazione delle proteine patologiche richiede meccanismi farmacologici che migliorino la proteostasi, anche in circostanze in cui le SSP sono compromesse, come nell’invecchiamento. Per identificare tali meccanismi, abbiamo cercato un composto geroprotettivo in grado di prolungare la durata della vita e di migliorare la proteostasi in C. elegans post-stress-responsive. Questi sforzi hanno identificato il minociclinico, un antibiotico approvato dall’agenzia di regolamentazione, noto anche per ridurre l’infiammazione e l’aggregazione delle proteine nei topi e negli esseri umani da un MOA finora sconosciuto (File supplementare 1; File supplementare 2). Utilizzando un approccio chemio-proteomico imparziale, abbiamo identificato il MOA con cui la minociclina prolunga la durata della vita e migliora la proteostasi. I nostri dati ABPP mostrano che il minociclinico prende di mira il ribosoma (Figura 3A) per attenuare la traslazione (Figura 3D; Figura 4A), riducendo preferibilmente la formazione del polisoma (Figura 4B), imitando gli effetti protettivi della PVR senza lavorare attraverso questo percorso (Figura 4G; Figura 6). In particolare, questo MOA fornisce anche una spiegazione unificante per i molti altri effetti apparentemente non correlati della minociclina osservati in studi preclinici e clinici, tra cui la sua capacità di ridurre la crescita tumorale, l’infiammazione e migliorare i sintomi della fragile X. Infatti, Mortison et al. sono arrivati allo stesso MOA studiando gli effetti della doxiciclina sull’infiammazione e la crescita tumorale (Garrido-Mesa et al., 2013; Mortison et al., 2018).
Figura 6.Modello su come il minociclinico riequilibra il carico proteostatico con la ridotta capacità di piegamento negli organismi più vecchi.In alto a sinistra: Nei giovani animali le risposte allo stress e la PVR agiscono come meccanismi di feedback per controllare il carico proteostatico guidato dalla traslazione dell’mRNA. La traslazione è impedita dal sovraccarico del macchinario di piegatura in quanto l’accumulo di proteine dispiegate porta all’induzione di risposte di stress, innescando l’attenuazione della traslazione attraverso la fosforilazione eIF2α, agendo come controllo di feedback per ridurre il carico protesostatico. In alto a destra: Il declino della risposta di segnalazione dello stress associato all’età compromette l’attenuazione del feedback traslazionale, consentendo un relativo eccesso di traduzione dell’mRNA rispetto alla diminuzione della capacità di piegamento negli adulti più anziani. Così, nei vecchi organismi i segnali cellulari (cioè i segnali infiammatori) che inducono cambiamenti diffusi nell’espressione genica e nella sintesi proteica possono portare ad un carico proteostatico eccessivo che non può più essere gestito dalla capacità di ripiegamento esistente. In basso: Il minociclinico prende di mira il ribosoma per attenuare la traslazione, allineando così il carico proteostatico con la capacità di piegamento, imitando l’effetto della PVR. Lo spessore delle frecce di “traslazione” e di “piegatura” sono disegnate per indicare la capacità relativa di ogni sistema.
L’abbassamento diretto della traduzione attraverso mutazioni, agenti farmacologici o RNAi è un meccanismo geroprotettivo che estende la durata della vita in taxa (Kapahi et al., 2004; Kaeberlein et al., 2005; Hansen et al., 2007; Harrison et al., 2009; Selman et al., 2009; Han et al., 2017). Il nostro studio rivela che i numerosi effetti benefici della minociclina negli eucarioti sono ottenuti attenuando direttamente la traduzione e che questo meccanismo è in grado di ridurre l’aggregazione proteica anche se avviato in vecchi C. elegans con deficit di SSP (Figura 1G,H). Mentre l’effetto sulla longevità nei vecchi organismi è simile tra la minociclina e la rapamicina, la nostra analisi dell’epistasi mostra che la minociclina differisce per quanto riguarda alcuni requisiti genetici. L’inibizione della segnalazione mTOR o della fosforilazione S6 non prolunga la durata della vita dei mutanti TFEB/hlh-30 e richiede la risposta allo shock termico mediata da hsf-1 per prolungare la durata della vita e per prevenire l’aggregazione delle proteine (Robida-Stubbs et al., 2012; Lapierre et al., 2013; Seo et al., 2013). Al contrario, minociclina estende la durata della vita di hlh-30 (tm1978) e di hsf-1 (sy441) mutanti (Figura 2) e impedisce l’aggregazione di proteine in C. elegans e cellule umane, nonostante inibire l’attivazione della risposta allo shock termico da hsf-1 (Figura 2B-D; Figura 4C-F) (Prahlad et al., 2008; Steinkraus et al., 2008). Queste differenze sollevano la questione di come l’attenuazione della traduzione da parte del minociclinico estende la longevità indipendentemente dagli SSP che sono richiesti per altri meccanismi legati alla traduzione. Una probabile spiegazione deriva dal modo in cui il minocicline attenua la traduzione. Qualsiasi meccanismo che abbia come obiettivo l’rRNA 18S, il sito catalitico del ribosoma, deve attenuare la traduzione in modo non selettivo. Al contrario, molti altri interventi ben studiati mirano all’avvio della traduzione, che in linea di principio permette di attenuare selettivamente la traduzione di sottogruppi di mRNA. Ad esempio, è stato dimostrato che l’abbattimento di eIF4G/ifg-1 porta a un’attenuazione generale della traduzione, ma anche a un aumento selettivo della traduzione di mRNA relativi alla SSP, che hanno un ruolo nell’effetto protettivo (Rogers et al., 2011). Pertanto, il fatto che gli interventi riducano o meno la traduzione in modo selettivo o non selettivo e quindi coinvolgano gli SSP dipende probabilmente dal fatto che essi mirino o meno all’avvio della traduzione o ad aspetti della formazione di legami peptidici. Studi precedenti hanno usato il cicloesossimide per dimostrare che il blocco della traduzione interferendo con la fase di traslocazione protegge dall’aggregazione delle proteine, poiché le proteine di nuova sintesi sono le principali specie proteiche suscettibili di danni e di misfolding collaterale sotto stress. Un’ovvia previsione testabile dei nostri dati minociclinici è che l’effetto protettivo del cicloesossimide dovrebbe essere indipendente da qualsiasi SSP (Medicherla e Goldberg, 2008; Zhou et al., 2014; Xu et al., 2016). Se il minociclito agisce in modo non selettivo, in che modo il suo effetto è maggiore sui polisomi rispetto ai monosomi? Qualsiasi singolo ribosoma rallentato dal minociclinico influenzerà negativamente tutti i ribosomi successivi sullo stesso mRNA, amplificando così l’effetto in modo dipendente dal numero di ribosomi. Per chiarire con precisione questi dettagli saranno necessari studi futuri.
Simile al minocicline, Smith et al. hanno dimostrato che la privazione batterica iniziata nel giorno 8, C. elegans, estende anche la durata della vita, anche tanto quanto quando la si inizia al giorno 4 (Smith et al., 2008). Anche se non misurata direttamente in questo studio, la deprivazione batterica è anche suscettibile di ridurre drasticamente la traduzione citoplasmatica, come suggerito da Steinkraus et al. (2008). Da un punto di vista terapeutico, i meccanismi che impediscono l’aggregazione delle proteine indipendentemente dall’attivazione della rete HSF1 sono particolarmente interessanti in quanto la presenza di aggregati proteici patologici durante la malattia suggerisce già un macchinario per la piegatura delle proteine sovraccaricato (Balch et al., 2008).
L’attenuazione della traduzione è un meccanismo di protezione cellulare fondamentale. In risposta allo stress, gli SSP attivano l’ISR, che, attraverso la fosforilazione di eIF2α, attenua selettivamente la traduzione (Novoa et al., 2003; Martin et al., 2014; Pakos-Zebrucka et al., 2016). L’attenuazione della traduzione fornisce alla cellula un periodo di recupero per ripristinare la proteostasi prima che la traduzione riprenda dopo la deposforilazione dell’eIF2α. Il recupero dopo lo stress è segnato da un’intensa traduzione di mRNA chaperone come HSP70 o BiP che comporta la formazione di polisomi, tassando la rete di proteostasi preesistente (Novoa et al., 2003; Das et al., 2015). L’avvio della traduzione dopo un evento stressante, prima del ripristino della proteostasi, porta alla produzione di ROS e di apoptosi mediata dalla caspase-3 (Han et al., 2013). Attenuando la traduzione, il minociclinico prolunga il periodo di recupero della proteostasi in modo simile a quanto osservato per l’inibitore PPP1R15A Sephin1 (Das et al., 2015). Riducendo di preferenza la formazione del polisoma (Figura 4B), il minocicline riduce generalmente il carico di picco sulla rete di proteostasi, pur consentendo una traduzione sufficiente a mantenere la funzione cellulare. La nostra scoperta del MOA della minociclina conferma le precedenti previsioni di Han et al. (Han et al., 2013) che la limitazione della sintesi proteica dovrebbe proteggere dall’aggregazione proteica (Choe et al., 2016).
Il MOA da noi proposto spiega anche perché l’effetto geroprotettivo della minociclina rimane inducibile in età avanzata rispetto a molti altri meccanismi. Meccanismi di longevità come la segnalazione di insulina ridotta/IGF provocano effetti geroprotettivi attraverso l’attivazione di SSP. Poiché le SSP non sono più inducibili negli adulti post-stress-responsive, i loro effetti geroprotettivi non possono più essere attivati negli animali più anziani, escludendo la possibilità che la minociclina prolunghi la durata della vita quando viene avviata negli adulti post-stress-responsive inibendo direttamente la traduzione mitocondriale e la successiva attivazione dell’UPR mitocondriale (Dillin et al., 2002a; Dillin et al., 2002b; Baker et al., 2012; Labbadia e Morimoto, 2015; Rangaraju et al., 2015). La minociclina bypassa l’attivazione delle SSP o della PVR (Figura 2; Figura 4) per attenuare la traslazione, suscitando effetti geroprotettivi abbassando la concentrazione di proteine di nuova sintesi, soggette ad aggregazione, allineando il carico proteostatico con la minore capacità della rete di proteostasi negli adulti più anziani (Figura 6) (Balch et al., 2008; Plate et al., 2016).
Mentre non è noto se la minociclina prolunga la durata della vita nei mammiferi, i suoi effetti geroprotettivi riducono l’aggregazione delle proteine associate all’età e l’infiammazione, come dimostrato da numerosi studi preclinici e clinici (file supplementare 1; file supplementare 2). La moltitudine di effetti apparentemente non correlati è stata utilizzata per sostenere che le tetracicline agiscono da un’ampia varietà di MOA distinti. L’entusiasmo per diversi risultati clinicamente rilevanti è stato smorzato dalla mancanza di un chiaro MOA necessario per progettare derivati specifici dell’eucariota di questi farmaci (Metz et al., 2004; Lampl et al., 2007). I nostri risultati gettano nuova luce su queste osservazioni. L’attenuazione della traduzione riducendo il carico ribosomiale come MOA fornisce una spiegazione semplice e convincente per questi effetti benefici apparentemente non correlati.
Ad esempio, la minociclina riduce l’infiammazione nei tessuti periferici e nel sistema nervoso centrale (Ferretti et al., 2012). L’inizio della cascata infiammatoria è simile all’inizio di altre risposte di stress e porta all’attivazione dei fattori di trascrizione NF-κB e AP1 che inducono l’espressione di citochine pro-infiammatorie come interleuchina-1 (IL-1), interleuchina-6 (IL-6), TNF-α o ossido nitrico sintasi (NOS). Mentre è stato precedentemente suggerito che gli effetti antinfiammatori della minociclina sono ottenuti dall’inibizione di MMP9, l’attenuazione traslazionale dei fattori pro-infiammatori fornisce un’alternativa convincente. Allo stesso modo, gli effetti benefici della minociclina nel trattamento della sindrome X fragile potrebbero anche essere spiegati dall’attenuazione traslazionale, in quanto la fragile X è causata dalla mancata corretta espressione del repressore traslazionale FMRP (Li et al., 2001).
Tuttavia, è anche importante notare che la minociclina ha mostrato alcuni effetti dannosi in uno studio sulla sclerosi laterale amiotrofica (SLA) (Gordon et al., 2007). Come ci si aspettava da un antibiotico, i pazienti trattati con minociclina hanno mostrato effetti gastrointestinali più negativi come nausea, diarrea e costipazione. Il fatto che la minociclina fosse o meno dannosa per i pazienti affetti da SLA è stato successivamente messo in discussione sulla base del fatto che il dosaggio utilizzato era troppo elevato, aggravando gli effetti collaterali negativi (Leigh et al., 2008). Dato che i pazienti affetti da SLA mostrano un fenotipo ipermetabolico, gli effetti collaterali negativi gastrointestinali possono avere effetti benefici mascherati (Jésus et al., 2018), rendendo altamente desiderabile un composto simile alla minociclina che manca dell’attività antibiotica.
Il riutilizzo di farmaci approvati dalla FDA, come la minociclina, mediante l’uso di schermi fenotipici, rivela effetti promettenti al di fuori dell’indicazione primaria (antibiotico) della minociclina e porta inevitabilmente a promettenti nuovi target di farmaci e MOA. I nostri dati suggeriscono che l’attività antibiotica della minociclina costringe una campagna di relazione struttura-attività della minociclina (SAR) per migliorare l’attenuazione traslazionale eucariotica eliminando l’antibiotico e altre attività. In sintesi, i nostri studi sulla minociclina fanno luce sulla plasticità dei meccanismi di longevità all’invecchiamento e rivelano un MOA per la minociclina che ne spiega gli effetti geroprotettivi.

Figura 6.Figura 6. Modello su come il minocicline riequilibra il carico proteostatico con la ridotta capacità di piegamento negli organismi più vecchi.In alto a sinistra: Nei giovani animali le risposte allo stress e l’ISR agiscono come meccanismi di feedback per controllare il carico proteostatico guidato dalla traduzione dell’mRNA. La traslazione è impedita dal sovraccarico della macchina piegatrice poiché l’accumulo di proteine dispiegate porta all’induzione di risposte di stress, innescando l’attenuazione della traslazione attraverso la fosforilazione eIF2α, agendo come controllo di feedback per ridurre il carico protesostatico. In alto a destra: Il declino della risposta di segnalazione dello stress associato all’età compromette l’attenuazione del feedback traslazionale, consentendo un relativo eccesso di traduzione dell’mRNA rispetto alla diminuzione della capacità di piegamento negli adulti più anziani. Così, nei vecchi organismi i segnali cellulari (cioè i segnali infiammatori) che inducono cambiamenti diffusi nell’espressione genica e nella sintesi proteica possono portare ad un carico proteostatico eccessivo che non può più essere gestito dalla capacità di ripiegamento esistente. In basso: Il minociclinico prende di mira il ribosoma per attenuare la traslazione, allineando così il carico proteostatico con la capacità di piegamento, imitando l’effetto della PVR. Lo spessore delle frecce di “traslazione” e di “piegatura” sono disegnate per indicare la capacità relativa di ogni sistema.
Materiali e metodi
| Tipo di reagente (specie) o risorsa | Designazione | Fonte o riferimento | Identificatori | Ulteriori informazioni |
|---|---|---|---|---|
| Ceppo, fondo di ceppo (Caenorhabditiselegans) | N2 | Centro di Genetica di Caenorhabditis | RRID:WB-STRAIN:N2_(ancestrale) | wild-type (Bristol) |
| Ceppo, fondo di ceppo (C. elegans) | CL2166 | CGC | RRID:WB-STRAIN:CL2166 | dvIs19 [(pAF15)gst-4p::GFP::NLS] III |
| Ceppo, fondo di ceppo (C. elegans) | SJ4005 | CGC | RRID:WB-STRAIN:SJ4005 | zcIs4 [hsp-4::PFP] V |
| Ceppo, fondo di ceppo (C. elegans) | SJ4100 | CGC | RRID:WB-STRAIN:SJ4100 | zcIs13 [hsp-6::PFP] |
| Ceppo, fondo di ceppo (C. elegans) | CL2070 | CGC | RRID:WB-STRAIN:CL2070 | dvIs70 [hsp-16.2p::GFP + rol-6(su1006) |
| Ceppo, fondo di ceppo (C. elegans) | SJ17 | CGC | RRID:WB-STRAIN:SJ17 | xbp-1(zc12) III; zcIs4 V |
| Ceppo, fondo di ceppo (C. elegans) | QV225 | CGC | RRID:WB-STRAIN:QV225 | skn-1(zj15) IV |
| Ceppo, fondo di ceppo (C. elegans) | PS3551 | CGC | RRID:WB-STRAIN:PS3551 | hsf-1(sy441) I |
| Ceppo, fondo di ceppo (C. elegans) | CF1038 | CGC | RRID:WB-STRAIN:CF1038 | daf-16(mu86) I |
| Ceppo, fondo di ceppo (C. elegans) | CB369 | CGC | RRID:WB-STRAIN:CB369 | unc-51(e369) V |
| Ceppo, fondo di ceppo (C. elegans) | RB1206 | CGC | RRID:WB-STRAIN:RB1206 | rsks-1(ok1255) III |
| Ceppo, fondo di ceppo (C. elegans) | CB3388 | CGC | RRID:WB-STRAIN:CB3388 | ncl-1(e1865) III |
| Ceppo, fondo di ceppo (C. elegans) | CL2006 | CGC | RRID:WB-STRAIN:CL2006 | dvIs2(pCL12(unc-54:hu-Aβ 1–42)+pRF4) |
| Ceppo, fondo di ceppo (C. elegans) | NL5901 | CGC | RRID:WB-STRAIN:NL5901 | pkIs2386 [α-sinucleina::YFP unc-119(+)] |
| Ceppo, fondo di ceppo (C. elegans) | MAH93 | Altro | glp-1(ar202), unc-119(ed3), ItIs38[pAAA1; pie-1/GFP::PH(PLCdelta1); unc-119 (+)] III; ItIs37[pAAA64; pie-1/mCHERRY::his-58, unc-119 (+)]IV); dono di M. Hansen | |
| Ceppo, fondo di ceppo (C. elegans) | MAH686 | Altro | hlh-30(tm1978) IV; dono di M. Hansen | |
| Ceppo, fondo di ceppo (C. elegans) | CMH5 | DOI: 10.1371/ diario.pone.0159989 | atfs-1(tm4525) V | |
| Ceppo, fondo di ceppo (C. elegans) | BC20306 | Progetto Baillie Genome GFP, Simon Fraser University | RRID:WB:-STRAIN:BC20306 | cyp-34A9::Polvere da sparo |
| Linea cellulare (Homo sapiens) | HeLa | ATCC | Cat # CCL-2, RRID:CVCL_0030 | |
| Linea cellulare (Mus muscolo muscoloso) | NIH 3T3 | ATCC | Cat # CRL-1658, RRID:CVCL_0594 | |
| Linea cellulare (H. sapiens) | HEK293DAX | DOI: 10.1016/j.celrep.2013.03.024 | ||
| Anticorpo | anti-actina (monoclonale del mouse) | MP Biomedicale | Cat # 08691001, RRID:AB_2335127 | (1:500) |
| Anticorpo | anti-Hsp60 (mouse monoclonale) | ThermoFisher | Cat # MA3-012, RRID:AB_2121466 | (1:250) |
| Anticorpo | anti-Hsp70/72 (mouse monoclonale) | Enzo | Cat # ADI-SPA-810, RRID:AB_10616513 | (1:1000) |
| Anticorpo | anti-GRP78 (BiP) (coniglio policlonale) | Abcam | Cat # ab21685, RRID:AB_2119834 | (1:1000, da 1 mg/ml) |
| Anticorpo | IRDye 800CW (secondario) | Li-Cor | Cat # 926-32210, RRID:AB_621842 | (1:10000) |
| Anticorpo | IRDye 800CW (secondario) | Li-Cor | Cat # 926-32211, RRID:AB_621843 | (1:10000) |
| Reagente a base di sequenza | HSP60 forward primer, 5′ – GCAGAGTTCCTCAGAAGTTGGG-3′. | DOI: 10.1186/ s12974-016-0486-x | qRT-PCR | |
| Reagente a base di sequenza | HSP60 primer invertito, 5′ – GCATCCAGTAAGGCAGTTTTCTC-3′. | DOI: 10.1186/ s12974-016-0486-x | qRT-PCR | |
| Reagente a base di sequenza | HSPA1A (HSP70)primer in avanti, 5′- GGAGGCGGGAGAAGTACA-3′. | DOI: 10.1021/ cb500062n | qRT-PCR | |
| Reagente a base di sequenza | HSPA1A (HSP70)primer invertito, 5′- GCTGATGATGATG GGGTTAACA-3′. | DOI: 10.1021/cb500062n | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.1/.11 primer in avanti, 5′- ACCACTATTATTTC CGTCCAGCT-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.1/.11 primer invertito, 5′- TGACGTTCCATCTGAGCCAT-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.2 forward primer, 5′- TCGATTGAAGCGCCAAAAGAA-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.2 primer invertito, 5′- TCTCTTCGACGACGATTGCCTGT-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.41 forward primer, 5′- TCTTGGGACGAACTCACTGGA-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.41 primer invertito, 5′- AGAGACATCGAGTTGAACCGAACCGA-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.48/.49forward primer, 5′- CTCATGCTCCGTTTTCCATT-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | hsp-16.48/.49 primer invertito, 5′- GAGTTGTGTGATCAGCATTTCCA-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | GFP forward primer, 5′- GGTCCTTTTTGAGTTTGTAAC-3′. | DOI: 10.1074/ jbc.C100556200 | qRT-PCR | |
| Reagente a base di sequenza | Primer invertito GFP, 5′- CTCCACTGACA GAAAATTTG-3”. | DOI: 10.1074/jbc.C100556200 | qRT-PCR | |
| Reagente a base di sequenza | SDHA forward primer, 5′- TGGGTGCTGGGTTGTTCTCATTA-3′. | DOI: 10.1134/ S0003683813090032 | qRT-PCR | |
| Reagente a base di sequenza | SDHA primer invertito, 5′ – ACCTTTCGCCCCCCTTGACTGTT-3′. | DOI: 10.1134/ S0003683813090032 | qRT-PCR | |
| Reagente a base di sequenza | HSPC3 forward primer, 5′- ATGGAAGAGAGA GCAAGGCAAA-3′. | DOI: 10.1134/ S0003683813090032 | qRT-PCR | |
| Reagente a base di sequenza | HSPC3 primer invertito, 5′- AATGCAGCAGCAAG GTGAAGACA-3′. | DOI: 10.1134/ S0003683813090032 | qRT-PCR | |
| Reagente a base di sequenza | crn-3 forward primer, 5′- GAATGCACCACTCAT GAACAAAGAAGTC-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | crn-3 primer invertito, 5′- TAATGTTCGGACT GATGAACCG-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | xpg-1 primer in avanti, 5′- ATTGGAGAACAG GATCATGAGG-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | xpg-1 primer invertito, 5′-ACTAGCA ACTCGTTTATCATCC-3′. | DOI: 10.7554/ eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | rpl-6 primer in avanti, 5′-ACTAGCAACTCGTTTATCATCC-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Reagente a base di sequenza | rpl-6 primer invertito, 5′- GACAGTCTTGGAATGTCCTCCGA-3′. | DOI: 10.7554/eLife.08833 | qRT-PCR | |
| Saggio commerciale o kit | 20S Proteasome Activity Assay Kit di analisi delle attività | Chemicon International | Gatto # APT280 | |
| Saggio commerciale o kit | RNeasy Mini Kit | Qiagen | Gatto # 74104 | |
| Saggio commerciale o kit | iScript RT-Supermix | Bio-Rad | Gatto # 170-8841 | |
| Saggio commerciale o kit | SsoAdvanced SYBR Green Supermix | Bio-Rad | Gatto # 172-5264 | |
| Saggio commerciale o kit | Saggio di aggregazione PROTEOSTAT Prot. | Enzo | Cat # ENZ-51023 | |
| Composto chimico, farmaco | minociclina | Mp Biomedicali | Gatto # 0215571891 | |
| Composto chimico, farmaco | metil viologen idrato (paraquat) | Acros Organics | Cat # 227320010 | |
| Composto chimico, farmaco | tunicamicina | Laboratori LKT | Gatto # T8153 | |
| Composto chimico, farmaco | thapsigargin | Cayman Chemical | Gatto # 10522 | |
| Composto chimico, farmaco | ISRIB | Sistemi R e D | Gatto # 5284/10 | |
| Composto chimico, farmaco | levamisolo | Mp Biomedicali | Cat # 0215522810 | |
| Composto chimico, farmaco | arsenito di sodio | Spettro Chimico | Gatto # S1135 | |
| Composto chimico, farmaco | cicloesimmide | Alfa Aesar | Gatto # J66901-03 | |
| Software, algoritmo | CellProfiler | Ampio Istituto | RRID:SCR_007358 | α-sinucleina::Analisi del numero aggregato YFP e della dimensione del worm |
| Software, algoritmo | Bio-Rad CFX Manager | Bio-Rad | qRT-PCR analisi | |
| Software, algoritmo | ImageQuant | GE Healthcare Life Sciences | RRID:SCR_014246 | 35S analisi di incorporazione |
C. ceppi elegans
Il ceppo di Bristol (N2) è stato usato come ceppo wild-type. I seguenti ceppi di vermi utilizzati in questo studio sono stati ottenuti dal Caenorhabditis Genetics Center (CGC; Minneapolis, MN), salvo diversa indicazione. CL2166 [dvIs19 [(pAF15)gst-4p::GFP::NLS]], SJ4005 [zcIs4 [hsp-4p::GFP]], SJ4100 [zcIs13 [hsp-6p::GFP]], CL2070 [dvIs70 [hsp-16.2p::GFP + rol-6(su1006)]], SJ17 [xbp-1(zc12); zcIs4], QV225 [skn-1(zj15)], PS3551 [hsf-1(sy441)], CF1038 [daf-16(mu86)], CB369 [unc-51(e369)], RB1206 [rsks-1(ok1255)], CB3388 [ncl-1(e1865)], CL2006 [dvIs2(pCL12(unc-54:hu-Aβ 1–42)+pRF4)], NL5901 [pkIs2386 [α-sinucleina::YFP unc-119(+)]]. I ceppi sono stati incrociati almeno tre volte prima dell’analisi sperimentale. MAH93 [glp-1(ar202), unc-119(ed3), ItIs38[pAAA1; pie 1/GFP::PH(PLCdelta1); unc-119 (+)]; ItIs37[pAAA64; pie-1/mCHERRY::his-58, unc-119 (+)]] e MAH686 [hlh-30(tm1978)] erano regali di Malene Hansen e Caroline Kumsta e atfs-1(tm4525) V era un regalo di Cole Haynes (Nargund et al., 2012). BC20306 [cyp-34A9p::GFP] è stato ricevuto da Baillie Genome GFP Project (Simon Fraser University, Burnaby, Vancouver, Canada).
Immagini di stress da verme
1000 – 2000 animali reporter L1 GFP di età sincronizzata sono stati placcati in piastre di coltura da 6 cm con terreno liquido (S-complete medium con 50 µg/ml di carbenicillina e 0,1 μg/ml di fungizone [Amfotericina B]) contenente 6 mg/ml di Escherichia coli OP50 (1.5 × 108 unità formanti colonie [ufc]/ml, resistenti alla carbenicillina per escludere la crescita di altri batteri), preparate di recente con 4 giorni di anticipo, come descritto in precedenza (Solis e Petrascheck, 2011), e sono state mantenute a 20°C. Il volume finale in ogni piastra era di 7 ml. Per evitare l’autofecondazione, 5-fluoro-2′-deossiuridina (FUDR, 0,12 mM finale) (Sigma-Aldrich, gatto # 856657) è stato aggiunto 42 – 45 ore dopo la semina. Il primo giorno è stato il primo giorno di giovane età adulta (il primo giorno dopo la fase L4), vari fattori di stress sono stati aggiunti ad ogni ceppo in diversi punti di tempo: 0,5 mM arsenite al giorno 1/giorno 5/giorno 8 CL2166 gst-4p::GFP e imaged 5 ore dopo, 0,5 mM paraquat alla fine L4/giorno 4/giorno 7 stadio di SJ4100 hsp-6p::GFP e imaged 24 ore dopo, 5 μg/ml tunicamicina al giorno 1/giorno 5/giorno 8 SJ4005 hsp-4p::GFP e imaged 8 ore dopo e un 1.5 hr shock termico a 35°C seguito da recupero a 20°C al giorno 1/giorno 5/giorno 8 CL2070 hsp-16.2p::PFL e ripresa 8 hr dopo. L’intensità della GFP dei vermi è stata quantificata utilizzando un COPAS FP BIOSORT (Union Biometrica) e gli animali sono stati ordinati in piastre a 96 pozzetti e ripresi utilizzando un sistema di screening ad alto contenuto di ImageXpress Micro XL High-Content (Dispositivi Molecolari) con un obiettivo 2x. Per determinare l’effetto della minociclina sull’espressione delle GFP, sono stati aggiunti 100 μM di minociclina 2 ore prima di ogni fattore di stress al giorno 4/giorno 5.
Durata della vita
I C. elegans sincronizzati in base all’età sono stati preparati in mezzo liquido (S-complete medium con 50 µg/ml di carbenicillina e 0,1 μg/ml di fungizone) in piastre a fondo piatto, otticamente trasparenti a 96 pozzetti (Corning, cat # 351172) contenenti 150 μl di volume totale per pozzetto, come descritto in precedenza (Solis e Petrascheck, 2011). Piastre contenenti ~10 animali per pozzetto in 6 mg/ml OP50. Tutti gli esperimenti con minociclina sono stati preparati con γ-irradiato OP50. Gli animali sincronizzati in base all’età sono stati seminati come larve L1 e cresciuti a 20 ° C. Piatti sono stati coperti con sigillanti per prevenire l’evaporazione. Per prevenire l’autofecondazione, FUDR (0,12 mM finale) è stato aggiunto 42 – 45 ore dopo la semina. I farmaci sono stati aggiunti nei giorni indicati e la sopravvivenza è stata segnata manualmente visualizzando il movimento dei vermi con un microscopio invertito 3x/settimana. Quando è stato utilizzato, il DMSO è stato mantenuto ad una concentrazione finale dello 0,33% v/v. L’analisi statistica è stata effettuata utilizzando la versione Mantel-Haenszel del log-rank test come descritto in Petrascheck e Miller (Petrascheck e Miller, 2017).
α-sinucleina::YFP imaging
NL5901 (pkIs2386 [α-sinucleina::YFP unc-119(+)]) gli animali sincronizzati in base all’età sono stati trattati con acqua il giorno 1 o 100 μM minociclina il giorno 1, 5 o 8. Nei giorni 8, 11, 16 o 19, 10 – 15 animali trattati con acqua e minociclina di ogni fase di trattamento sono stati trasferiti in vetrini di vetro contenenti il 3% di agarosio pad e paralizzato con l’aggiunta di una goccia di una soluzione di levamisolo 1 mM levamisolo disciolto in tampone M9. Le immagini in campo chiaro e fluorescenza sono state scattate con un obiettivo 20x utilizzando l’ImageXpress Micro XL. Il numero di aggregati di α-sinucleina nella regione faringea di ogni verme (25% della lunghezza totale del corpo) sono stati determinati analizzando le immagini utilizzando una pipeline personalizzata creata nel software CellProfiler.
Paralisi dell’abeta CL2006
Giorno 1 CL2006 (dvIs2(pCL12(unc-54:hu-Aβ 1–42)+pRF4))) gli animali sincronizzati sono stati trattati con acqua o 100 μM minociclina. Il giorno 4, 50 – 100 animali ciascuno sono stati trasferiti su tre piastre NGM e posizionati a 37°C per 2 ore. Dopo lo shock termico, gli animali sono stati segnati per determinare il numero di animali paralizzati. Gli animali sono stati contrassegnati come paralizzati se non mostravano un movimento a metà del corpo al tocco leggero alla faringe con un piccone verme.
Termotolleranza
Gli animali N2 sincronizzati in base all’età sono stati preparati in piastre di coltura da 6 cm e trattati con acqua o minociclina da 100 μM il primo giorno. La sera del giorno 4, 30 – 50 animali sono stati trasferiti in piastre NGM da 6 cm in triplice copia per ogni condizione e lo stress da calore è stato indotto a 35 ° C. La prima misurazione della sopravvivenza è stata effettuata 8 ore dopo toccando leggermente gli animali con un verme e segnando il movimento. Le piastre sono state mantenute a 35°C e le misurazioni della sopravvivenza sono state effettuate ogni 2 ore fino a quando quasi tutti gli N2 trattati con acqua sono morti.
Saggio di resistenza allo stress da paraquat
La resistenza allo stress ossidativo è stata determinata misurando la sopravvivenza di animali non trattati e trattati con minociclina dopo un’esposizione di 24 ore al ROS-generatore paraquat (metil viologen idrato; Acros Organics, cat # 227320010). Sperimentale C. elegans culture sono stati istituiti come descritto in Lifespan Assay. Acqua o 100 μM minociclina è stato aggiunto il giorno 1. Il Paraquat è stato aggiunto ad una concentrazione finale di 0, 15, 25, 50, 75 e 100 mM il giorno 5 dell’età adulta e la sopravvivenza degli animali è stata valutata 24 ore dopo l’aggiunta del paraquat. È importante notare che abbiamo visto una protezione dalla morte indotta dal paraquat solo quando abbiamo usato OP50 morto, γ-irradiato e non abbiamo visto una protezione quando abbiamo usato OP50 vivo.
Iodoacetamide isoTOP-ABPP
20.000 N2 C. elegans sincronizzati in base all’età sono stati coltivati in coltura liquida in piastre di coltura di 15 cm (Corning, gatto # 351058), due piastre per condizione. Gli animali sono stati trattati con acqua o 100 μM minociclina il giorno 5. Gli animali sono stati raccolti il giorno 8, lavato 3x con DPBS freddo (Gibco, gatto # 14190 – 136) e flash-frozen in azoto liquido. 50 microlitri 1,4 mm di perle di ossido di zirconio (Precellys, gatto # 03961-1-103) e 50 microlitri 0,5 mm di perle di vetro (Precellys, gatto # 03961-1-104) sono stati aggiunti ad ogni campione e gli animali sono stati lisati con un omogeneizzatore di tessuti Precellys 24. Bradford test (Bio-rad, gatto # 5000002) è stato utilizzato per determinare la concentrazione di proteine per ogni campione per la normalizzazione. Proteoma campione (1 mg) è stato trattato con 100 mM IA-alkyne sonda aggiungendo 5 ml di uno stock di 10 mM sonda (in DMSO). Le reazioni di etichettatura sono stati incubati a temperatura ambiente per 1 ora, dopo di che i campioni sono stati coniugati per isotopicamente etichettati, TEV-etichette cancellabili (TEV tags) da rame-catalizzata azide-alchina ciclocarico (CuACC o ‘chimica click’). Pesante miscela di reazione chimica a scatto (60 ml) è stato aggiunto al campione di controllo trattato con acqua, e la miscela di reazione leggera (60 ml) è stato aggiunto al campione trattato con minociclina. La miscela di reazione a scatto consisteva di tag TEV [10 ml di un campione di 5 mM, leggero (trattato con minociclina) o pesante (trattato con acqua)], CuSO4 (10 ml di un campione di 50 mM in acqua), e tris(benzyltriazolylmethyl) ammina (30 ml di un campione di 1,7 mM in 4:1 tBuOH/DMSO), a cui è stato aggiunto tris(2- carbossietil)fosfina (10 ml di un campione di 50 mM). La reazione è stata eseguita per 1 ora a temperatura ambiente. I campioni leggeri e pesanti sono stati poi centrifugati a 16.000 x g per 5 minuti a 4°C per raccogliere le proteine precipitate. I pellet risultanti sono stati risospesi in 500 ml di metanolo freddo per sonicazione, e i campioni pesanti e leggeri sono stati combinati a coppie. I pellet combinati sono stati poi lavati con metanolo a freddo, dopo di che il pellet è stato solubilizzato per sonicazione in DPBS con 1,2% SDS. I campioni sono stati riscaldati a 90 ° C per 5 minuti e sottoposti a arricchimento con streptavidina di proteine marcate con sonda, sequenziale su tripsina e digestione TEV, e cromatografia liquida in tandem spettrometria di massa.
RAW Xtractor (versione 1.9.9.2; disponibile su http://fields.scripps.edu/ downloads.php) è stato utilizzato per estrarre i dati degli spettri MS2 dai file raw (i dati degli spettri MS2 corrispondono ai frammenti analizzati durante il secondo stadio della spettrometria di massa). I dati MS2 sono stati confrontati con una variante non ridondante e concatenata al contrario del database C. elegans UniProt (release, 2017_09) con l’algoritmo ProLuCID (disponibile pubblicamente sul sito http://fields.scripps.edu/downloads.php). I residui di cisteina sono stati ricercati con una modifica statica per la carbossiamidometilazione (+57.02146) e fino ad una modifica differenziale per i tag TEV leggeri o pesanti (+464.28595 o +470.29976, rispettivamente). Ai peptidi era richiesto di avere almeno un terminale triptico e di contenere la modifica TEV. I dati ProLuCID sono stati filtrati attraverso DTASelect (versione 2.0) per ottenere un tasso di falsi positivi peptidici inferiore all’1%.
La quantificazione dei rapporti luce/peso (rapporti isoTOP-ABPP, valori R) è stata eseguita dal software CIMAGE interno utilizzando i parametri di default (tre MS1 per picco e una soglia segnale-rumore di 2,5). Si veda la Figura 3-dati sorgente 1 per ulteriori parametri di filtraggio.
Saggio della prole
Gli animali N2 sono stati sincronizzati in base all’età e hanno potuto crescere in mezzo liquido in un piatto di 6 cm. Con la fase L4, un singolo verme è stato trasferito in ogni pozzo nella prima colonna di una piastra a 96 pozzetti (otto animali) contenente terreno liquido senza FUDR, due piastre per condizione. Il giorno 1, gli animali sono stati trattati con acqua o 100 μM minociclina. Ogni 24 ore, gli animali adulti sono stati trasferiti al pozzo nella colonna successiva e la progenie è stato segnato dal pozzo precedente utilizzando un microscopio invertito. Questo processo è stato continuato fino al quinto giorno dell’età adulta.
Saggio della dimensione del verme
Gli animali preparati come nel test della durata della vita e trattati con acqua o minociclina da 100 μM il giorno 1 sono stati ripresi per 4 giorni utilizzando un obiettivo 10x con ImageXpress Micro XL e la lunghezza in pixel di ogni verme misurata è stata determinata utilizzando il software di analisi CellProfiler.
Spettrometria di massa metabolica
Animali e batteri sono stati metabolicamente etichettati con l’isotopo di azoto desiderato (14N o 15N) come descritto in precedenza (Gomez-Amaro et al., 2015). Dopo un minimo di tre generazioni, ~ 20.000 larve sincronizzate in base all’età per condizione sono state trasferite in piatti di coltura di 15 cm e coltivate in mezzo liquido più metabolicamente etichettati 14N (sperimentale) o 15N (standard di popolazione mista) batteri (6 mg/ml). Acqua o 100 μM minociclina sono stati aggiunti e gli animali sono stati raccolti in diversi punti di tempo (L4 → giorno 1, giorno 5 → giorno 6, giorno 5 → giorno 8). Per controllare le differenze di contenuto proteico dovute al trattamento con minociclina, i campioni sono stati normalizzati per concentrazione di RNA.
Gli animali lisati sono stati preparati per la spettrometria di massa mediante precipitazione in acido tricloroacetico al 13% (TCA) durante la notte a 4°C. Il pellet proteico è stato raccolto per centrifugazione a velocità massima per 20 min. Il pellet è stato lavato con il 10% di TCA e filata di nuovo per 10 min a 4°. Il pellet è stato poi lavato con acetone freddo ghiaccio e filata per altri 10 minuti a 4 °. Il pellet proteico è stato risospeso in 100 – 200 microlitri di 100 mM di bicarbonato di ammonio con il 5% (v / v) acetonitrile. Poi il 10% (in volume) di 50 mM DTT è stato aggiunto e il campione è stato incubato per 10 minuti a 65°. Questa incubazione è stata seguita dall’aggiunta del 10% (in volume) di 1 mM di acido iodoacetico (IAA) e un’incubazione di 30 min a 30° al buio. Un totale di 5 μg di tripsina è stato aggiunto ad ogni campione e incubato durante la notte a 37°. I campioni tripsinizzati sono stati poi puliti per la spettrometria di massa utilizzando colonne PepClean (Pierce) seguendo le indicazioni del produttore. Campioni puliti sono stati essiccati in un vuoto di velocità e poi risospesi in 10 microlitri di acetonitrile 5% con 0,1% (v / v) di acido formico. I campioni sono stati filati ad alta velocità per rimuovere il particolato prima di mettere in provette di spettrometria di massa per l’analisi.
I campioni sono stati analizzati su uno spettrometro di massa ABSCIEX 5600 Triple time of flight (TOF) accoppiato ad un Eksigent nano-LC Ultra dotato di un sistema cHiPLC nanoflex. Le condizioni sull’ABSCIEX 5600 erano le seguenti: Le condizioni del gas sorgente sono state ottimizzate per ogni esperimento e sono state generalmente impostate su GS1 = 8-12, GS2 = 0 e Curtain Gas = 25. La temperatura della sorgente è stata impostata a 150°. Gli esperimenti di acquisizione dipendenti dalle informazioni sono stati eseguiti con un tempo di ciclo di 2 s, con un tempo di accumulo di 0,5 ms nella scansione MS1 e fino a 20 ioni candidati per periodo di tempo MS/MS. La gamma m/z nelle scansioni MS1 era di 400 – 1250 Da, e 100 – 1800 Da per le scansioni MS/MS. Gli ioni target sono stati esclusi dopo due eventi per 12 s per aumentare la copertura del sequenziamento. Gli ioni target con carica da +2 a +5 sono stati selezionati per il sequenziamento una volta raggiunta la soglia di 100 conteggi al secondo.
Le condizioni per il nano-HPLC Eksigent erano le seguenti: Gradienti sono stati eseguiti con un set-up trappola e l’arresto con acqua più 0,1% (v / v) acido formico come la fase mobile A e acetonitrile con lo 0,1% (v / v) acido formico come fase mobile B. I campioni sono stati caricati su un 200 μm × 0,5 mm ChromXP C18-CL 3 μm 120 Å Trappola colonna a 2 μl / min. I gradienti sono stati eseguiti dal 5% di fase mobile A al 40% di fase mobile B su una colonna analitica di 75 μm × 15 cm ChromXP C-18-CL 3 μm 120 Å. Questo è stato seguito da un rapido salto all’80% B per 10 minuti per pulire la colonna e un riequilibrio di 20 minuti al 95% A. I campioni grezzi di acqua sono stati eseguiti tra i campioni per liberare la colonna da eventuali residui di peptidi interferenti, tra cui un breve gradiente, seguito dal riequilibrio dell’80% B e 20 minuti con il 5% A.
I dati sono stati convertiti utilizzando il software di conversione ABSCIEX nel formato mgf e nel formato MZML. La lista dei picchi è stata generata cercando in una banca dati SwissProt con MASCOT, con la tassonomia impostata contemporaneamente su C. elegans ed E. coli (Matrix Science). Una ricerca MS/MS Ion è stata effettuata utilizzando la quantificazione 15N, con una tolleranza di massa peptidica di ±0,1 Da e una tolleranza di massa del frammento di ±0,1 Da. Il numero massimo di scissioni mancanti è stato impostato a 2. Le scansioni MS1 per i peptidi identificati erano adatte a tre distribuzioni isotopiche utilizzando ISODIST.
20S test di proteasoma
2500 animali sincronizzati in base all’età sono stati coltivati su piastre di coltura di 10 cm (Corning, gatto # 351029) per condizione in mezzo liquido. Acqua o 100 μM minociclina sono stati aggiunti il giorno 1. Il giorno 5, gli animali sono stati raccolti e lavati 3x con DPBS freddo, poi una volta con 1x tampone di saggio (Chemicon International, gatto # APT280). Gli animali sono stati lisati con Precellys omogeneizzatore e filata giù a 12.000 x g per 5 min. Le concentrazioni di proteine sono state misurate e normalizzate con il saggio Bradford. 20S proteasoma attività è stata misurata per ogni lisato secondo il protocollo (Chemicon International, gatto # APT280). 200 μg di campioni sono stati caricati con la miscela di saggio in ogni pozzetto di una piastra UV-trasparente a 96 pozzetti e incubati per 1 ora a 37 ° C. La fluorescenza è stata misurata sul Tecan Safire II con un set di filtri da 380/460 nm.
L’autenticazione della coltura cellulare e test
Le celle HeLa (ATCC CCL-2) o NIH 3T3 (ATCC CRL-1658) sono state ottenute direttamente da ATCC. Il test del micoplasma è stato fatto ogni 6 mesi attraverso il TSRI. L’identità delle cellule HEK293DAX è stata verificata mediante l’induzione di ATF6 attraverso TMP.
35S Incorporazione
Le cellule HeLa (ATCC CCL-2) o NIH 3T3 (ATCC CRL-1658) sono state coltivate a 37°C e placcate su una piastra a sei pozzetti (Corning, cat # 353046) in DMEM (Life Technologies, cat # 11995073) integrata con 10% FBS (ATCC, cat # 30).- 2021) e penicillina-streptomicina e le concentrazioni indicate di cicloeossimide o minociclina sono state aggiunte al 70% di cellule confluenti e incubate per 6 ore. Media di coltura DMEM è stato rimosso e pozzi sono stati lavati 2x con DPBS caldo. 2 ml di una soluzione DMEM senza metionina o cisteina (Life Technologies, gatto # 21013024) integrata con il 10% di FBS dializzato (VWR Intl., gatto # 101301 – 494) e [35S]-metionina / cisteina (PerkinElmer, gatto # NEG772002MC) è stato aggiunto a ciascun pozzetto per 1 ora. I supporti sono stati rimossi e i pozzetti sono stati lavati 2 volte con DPBS freddo e 200 μl di buffer RIPA freddo (Fisher Scientific, gatto # 507513771) con inibitore della proteasi (cOmplete mini EDTA-free, Sigma-Aldrich, gatto # 11836170001) è stato aggiunto ad ogni pozzetto per 15 minuti in ghiaccio. I lisati sono stati raccolti e filati a 5000 g per 10 min. I campioni normalizzati dalla concentrazione di proteine, determinata dal saggio di Bradford, sono stati eseguiti su un gel SDS-PAGE. Il gel è stato colorato con Coomassie Brilliant Blue G-250 (MP Biomedicals, cat # 0219034325), ripreso per il controllo del carico, essiccato ed esposto durante la notte ad uno schermo al fosforo e ripreso utilizzando un riproduttore d’immagini Typhoon 9400. Le corsie sono state quantificate utilizzando il software ImageQuant (GE Healthcare Life Sciences).
Profilo del polisoma
Le cellule HeLa sono state coltivate su piastre di coltura di 10 cm (Olympus Plastics, # 25 – 202) a 37 ° C in media di coltura DMEM integrato con il 10% di FBS e penicillina-streptomicina e acqua o 100 microclino M minociclina è stato aggiunto al 70% di cellule confluenti per 12 ore. 100 microgrammi / ml cicloesimero è stato aggiunto ad ogni piastra per 10 minuti. Su ghiaccio, mezzo è stato rimosso, le piastre sono state lavate 2x con tampone di lavaggio a freddo (DPBS integrato con 100 μg / ml di cicloesossimide) e le cellule sono state rimosse in 1 ml di tampone di lavaggio utilizzando un raschietto cella. Le cellule sono state trasferite in tubi di eppendorf e filate per 5 minuti a 2300 g a 4°C. Supernatante è stato rimosso e 300 microlitri di tampone ipotonico freddo (1,5 mM KCl, 10 mM MgCl2, 5 mM Tris-HCl, pH 7,4, 100 μg / ml cicloesossimide) è stato aggiunto e delicatamente mescolato. 300 μl tampone di lisi ipotonica fredda (2% sodio desossicolato, 2% Triton X-100, 2,5 mM DTT, 100 μg / ml cicloesossimide, e 10 unità di RNAsin / ml in tampone ipotonico) è stato aggiunto e le cellule sono state omogeneizzate utilizzando 10 colpi con un omogeneizzatore dounce vetro su ghiaccio. I campioni sono stati filati giù a 2300 x g per 10 minuti a 4 ° C. Supernatanti sono stati trasferiti in tubi di eppendorf fresco e la concentrazione di RNA è stata determinata misurando il A260. 2 mg di RNA sono stati caricati su gradienti di saccarosio 17 – 51% preparati con un creatore di gradienti in tubi SW41 (Denville, gatto # U5030). I tubi sono stati centrifugati a 40.000 giri al minuto per 2,5 ore a 4°C utilizzando un rotore SW41. OD260 è stato misurato per ogni campione utilizzando un monitor di assorbanza/fluorescenza online Isco Modello UA-5.
HSP western blot
Le cellule HeLa sono state coltivate su piastre di coltura di 10 cm a 37 ° C in DMEM integrato con il 10% FBS e penicillina-streptomicina e acqua o 100 microclino μM minociclina è stato aggiunto per 12 ore al 70% di cellule confluenti. Le cellule sono state trasferite a 43°C per uno shock termico di 1 ora, poi sono tornate a 37°C per un periodo di recupero di 6 ore. Piastre sono stati lavati 2x con DPBS freddo, poi 500 microlitri di buffer RIPA freddo contenente inibitore della proteasi è stato aggiunto per 15 minuti in ghiaccio. I lisati sono stati raccolti, normalizzati dalla concentrazione di RNA, eseguito su un gel SDS-PAGE e trasferiti per il rilevamento di immunoblot. Anti-actina (MP Biomedicals, gatto # 08691001), anti-Hsp60 (ThermoFisher, gatto # MA3-012) e anti-Hsp70/72 (Enzo, gatto # ADI-SPA-810) anticorpi primari e anticorpo secondario IRDye 800CW (Li-Cor, gatto # 926 – 32210) sono stati utilizzati per la rilevazione.
PCR quantitativa in tempo reale (qRT-PCR) e analisi dei dati
Tutti gli esperimenti qRT-PCR sono stati condotti secondo le linee guida MIQE (Bustin et al., 2009), tranne che i campioni non sono stati testati in un bio-analizzatore, ma fotometricamente quantificati utilizzando un Nanodrop. Le cellule HeLa sono state coltivate come sopra descritto al 70% di confluenza e trattate con o senza minociclina da 100 μM, due piastre da 10 cm per condizione. 12 ore dopo il trattamento, una ciascuna delle piastre non trattate e minociclina trattati sono stati sottoposti ad uno shock termico di 1 ora a 43 ° C e restituito a 37 ° C per un recupero di 3 ore. Le cellule sono state raccolte e l’RNA è stato estratto come descritto nel protocollo del mini kit Qiagen RNeasy (Qiagen, gatto # 74104). Piastre di coltura cellulare sono stati lavati due volte con DPBS caldo, seguito da aggiunta di Buffer RLT. Le cellule sono stati rimossi dalle piastre con un raschietto cella e raccolti in tubi eppendorf. RNeasy colonne di rotazione sono stati utilizzati per purificare l’RNA e sulla colonna DNasi digestione DNase è stata eseguita. Per i campioni di C. elegans, ~ 2000 giorni di età-sincronizzato 1 CL2070 hsp-16.2p::animali GFP coltivate in piastre di 10 cm sono stati trattati con acqua o 100 microclino μM minociclina per 2 ore prima di indurre lo shock termico a 35 ° C per 1,5 ore, e raccolto subito dopo. Gli animali raccolti sono stati lavati tre volte in DPBS freddo ghiaccio e congelati in azoto liquido. Per estrarre l’RNA, gli animali congelati sono stati ri-sospesi in Trizolo ghiacciato (Qiagen, gatto # 79306), perle di zirconio, e perle di vetro nel rapporto di 5:1:1:1 rispettivamente, e perturbato in Precellys sistema lisante (6500 rpm, 3 × 10 s cicli) seguita da estrazione cloroformio. RNA è stato precipitato utilizzando isopropanolo e lavato una volta con il 75% di etanolo seguito da DNAse (Sigma-Aldrich, gatto # AMPD1-1KT) trattamento.
Per tutti i campioni di RNA, la trascrizione inversa è stata effettuata utilizzando iScript RT-Supermix (Bio-Rad, cat # 170 – 8841) a 42°C per 30 min. Le reazioni di PCR quantitativa sono state impostate in piastre a 384 pozzetti (Bio-Rad, cat # HSP3901), che includevano 2,5 µl di Bio-Rad SsoAdvanced SYBR Green Supermix (cat # 172 – 5264), 1 µl di template cDNA (2.5 ng/µl, a finale di 0,5 ng/µl in 5 µl di reazione PCR), 1 µl di acqua, e 0,5 µl di primer forward e reverse (concentrazione finale 150 nM). La PCR quantitativa è stata effettuata utilizzando un termociclatore in tempo reale Bio-Rad CFX384 (95°C, 3 min; 40 cicli di 95°C 10 s, 60°C 30 s; curva di fusione: 95°C 5 s, 60-95°C con incremento di 0,5°C, 10 s). L’espressione genica è stata normalizzata su due geni di riferimento per i campioni di cellule HeLa, SDHA e HSPC3, e tre geni di riferimento per i campioni C. elegans, crn-3, xpg-1 e rpl-6, utilizzando il software Bio-Rad CFX Manager. La significatività statistica è stata determinata utilizzando il t-test dello studente
HEK293DAXwestern blot
Le cellule HEK293DAX sono state coltivate su piastre a 6 pozzetti a 37°C in DMEM integrate con il 10% di FBS e penicillina-streptomicina. Acqua o 100 microclino μM minociclina è stato aggiunto con o senza 20 uM trimetoprim per 12 ore al 70% di cellule confluenti. Piastre sono stati lavati 2x con DPBS freddo, poi 200 microlitri di tampone RIPA freddo contenente inibitore della proteasi è stato aggiunto per 15 minuti in ghiaccio. I lisati sono stati raccolti, normalizzati dalla concentrazione di RNA, eseguito su un gel SDS-PAGE e trasferiti per il rilevamento immunoblot. Per la rilevazione sono stati utilizzati anticorpi primari BiP (Abcam, gatto # ab21685) e anticorpi secondari IRDye 800CW (Li-Cor, anti-topo, gatto # 926 – 32210 e anti-coniglio, gatto # 926 – 32211).
Saggio di aggregazione ProteoStat
~70% di cellule HeLa confluenti placcate su due piastre a 6 pozzetti sono state trattate per 6 ore con acqua o 100 μM minociclina, tre pozzetti per trattamento. Una piastra è stata sottoposta a shock termico per 1 ora a 43°C seguita da un periodo di recupero di 1 ora a 37°C. I pozzi sono stati lavati due volte con DPBS freddo e poi incubati su ghiaccio per 15 minuti con l’inibitore della RIPA +proteasi freddo. Il lisato per ogni pozzo è stato raccolto e centrifugato a 5000 x g per 10 min. La concentrazione di proteine è stata misurata per ogni campione utilizzato e tutti i campioni sono stati normalizzati. L’aggregazione è stata misurata utilizzando il reagente di rilevazione PROTEOSTAT (Enzo, gatto # ENZ-51023) per 10 μg di proteine di ogni campione. La fluorescenza è stata misurata con eccitazione di 550 nm e l’emissione a 600 nm su un lettore di due piastre Tecan Sapphire su un periodo di 30 minuti, con una lettura al minuto. I valori di fluorescenza per ogni campione sono stati mediati su un periodo di 30 minuti per determinare un valore aggregato corrispondente normalizzato ai campioni trattati con acqua.
Traduzione ISRIB
Le cellule HeLa sono state placcate su una piastra a 6 pozzetti in media di coltura DMEM integrata con 10% FBS e penicillina-streptomicina e ISRIB (200 nM) (sistemi R e D, cat # 5284/10). Minociclina (100 μM), thapsigargin (200 nM) (Cayman Chemical, cat # 10522) o diverse combinazioni dei tre sono stati aggiunti al 70% di cellule confluenti e incubati per 3 ore (ISRIB e / o thapsigargin-trattati campioni) o 6 ore (campioni trattati con minociclina). L’incorporazione 35S è stata misurata utilizzando il protocollo descritto sopra (incorporazione 35S).
Saggio della durata di vita del lievito
I saggi RLS del lievito sono stati eseguiti isolando le cellule figlie vergini del ceppo BY4741 e poi hanno permesso di crescere in cellule madri mentre le loro figlie corrispondenti sono state microdissezionate e contate, fino a quando la cellula madre non ha più potuto dividersi. I dati sono stati analizzati utilizzando il test Wilcoxon rank-sum.
References
- Agwuh KN, MacGowan A. Pharmacokinetics and pharmacodynamics of the tetracyclines including glycylcyclines. Journal of Antimicrobial Chemotherapy. 2006; 58:256-265. DOI | PubMed
- Anbalagan C, Lafayette I, Antoniou-Kourounioti M, Haque M, King J, Johnsen B, Baillie D, Gutierrez C, Martin JA, de Pomerai D. Transgenic nematodes as biosensors for metal stress in soil pore water samples. Ecotoxicology. 2012; 21:439-455. DOI | PubMed
- Baker BM, Nargund AM, Sun T, Haynes CM. Protective coupling of mitochondrial function and protein synthesis via the eIF2α kinase GCN-2. PLoS Genetics. 2012; 8DOI | PubMed
- Balch WE, Morimoto RI, Dillin A, Kelly JW. Adapting proteostasis for disease intervention. Science. 2008; 319:916-919. DOI | PubMed
- Bansal A, Zhu LJ, Yen K, Tissenbaum HA. Uncoupling lifespan and healthspan in Caenorhabditis elegans longevity mutants. PNAS. 2015; 112:E277-E286. DOI | PubMed
- Bindea G, Mlecnik B, Hackl H, Charoentong P, Tosolini M, Kirilovsky A, Fridman WH, Pagès F, Trajanoski Z, Galon J. ClueGO: a Cytoscape plug-in to decipher functionally grouped gene ontology and pathway annotation networks. Bioinformatics. 2009; 25:1091-1093. DOI | PubMed
- Brodersen DE, Clemons WM, Carter AP, Morgan-Warren RJ, Wimberly BT, Ramakrishnan V. The structural basis for the action of the antibiotics tetracycline, pactamycin, and hygromycin B on the 30S ribosomal subunit. Cell. 2000; 103:1143-1154. DOI | PubMed
- Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL, Vandesompele J, Wittwer CT. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 2009; 55:611-622. DOI | PubMed
- Caballero A, Ugidos A, Liu B, Öling D, Kvint K, Hao X, Mignat C, Nachin L, Molin M, Nyström T. Absence of mitochondrial translation control proteins extends life span by activating sirtuin-dependent silencing. Molecular Cell. 2011; 42:390-400. DOI | PubMed
- Cai Z, Zhao Y, Yao S, Bin Zhao B. Increases in β-amyloid protein in the hippocampus caused by diabetic metabolic disorder are blocked by minocycline through inhibition of NF-κB pathway activation. Pharmacological Reports. 2011; 63:381-391. DOI | PubMed
- Cai Z, Yan Y, Wang Y. Minocycline alleviates beta-amyloid protein and tau pathology via restraining neuroinflammation induced by diabetic metabolic disorder. Clinical Interventions in Aging. 2013; 8:1089-1095. DOI | PubMed
- Choe YJ, Park SH, Hassemer T, Körner R, Vincenz-Donnelly L, Hayer-Hartl M, Hartl FU. Failure of RQC machinery causes protein aggregation and proteotoxic stress. Nature. 2016; 531:191-195. DOI | PubMed
- Choi SH, Lee DY, Chung ES, Hong YB, Kim SU, Jin BK. Inhibition of thrombin-induced microglial activation and NADPH oxidase by minocycline protects dopaminergic neurons in the substantia nigra in vivo. Journal of Neurochemistry. 2005; 95:1755-1765. DOI | PubMed
- Choi Y, Kim HS, Shin KY, Kim EM, Kim M, Kim HS, Park CH, Jeong YH, Yoo J, Lee JP, Chang KA, Kim S, Suh YH. Minocycline attenuates neuronal cell death and improves cognitive impairment in Alzheimer’s disease models. Neuropsychopharmacology. 2007; 32:2393-2404. DOI | PubMed
- Clark-Walker GD, Linnane AW. In vivo differentiation of yeast cytoplasmic and mitochondrial protein synthesis with antibiotics. Biochemical and Biophysical Research Communications. 1966; 25:8-13. DOI | PubMed
- Das I, Krzyzosiak A, Schneider K, Wrabetz L, D’Antonio M, Barry N, Sigurdardottir A, Bertolotti A. Preventing proteostasis diseases by selective inhibition of a phosphatase regulatory subunit. Science. 2015; 348:239-242. DOI | PubMed
- Dillin A, Crawford DK, Kenyon C. Timing requirements for insulin/IGF-1 signaling in C. elegans. Science. 2002a; 298:830-834. DOI | PubMed
- Dillin A, Hsu AL, Arantes-Oliveira N, Lehrer-Graiwer J, Hsin H, Fraser AG, Kamath RS, Ahringer J, Kenyon C. Rates of behavior and aging specified by mitochondrial function during development. Science. 2002b; 298:2398-2401. DOI | PubMed
- Dues DJ, Andrews EK, Schaar CE, Bergsma AL, Senchuk MM, Van Raamsdonk JM. Aging causes decreased resistance to multiple stresses and a failure to activate specific stress response pathways. Aging. 2016; 8:777-795. DOI | PubMed
- Ferretti MT, Allard S, Partridge V, Ducatenzeiler A, Cuello AC. Minocycline corrects early, pre-plaque neuroinflammation and inhibits BACE-1 in a transgenic model of Alzheimer’s disease-like amyloid pathology. Journal of Neuroinflammation. 2012; 9DOI | PubMed
- Festoff BW, Ameenuddin S, Arnold PM, Wong A, Santacruz KS, Citron BA. Minocycline neuroprotects, reduces microgliosis, and inhibits caspase protease expression early after spinal cord injury. Journal of Neurochemistry. 2006; 97:1314-1326. DOI | PubMed
- Frank DJ, Roth MB. ncl-1 is required for the regulation of cell size and ribosomal RNA synthesis in Caenorhabditis elegans. The Journal of Cell Biology. 1998; 140:1321-1329. DOI | PubMed
- Garrido-Mesa N, Zarzuelo A, Gálvez J. Minocycline: far beyond an antibiotic. British Journal of Pharmacology. 2013; 169:337-352. DOI | PubMed
- Gomez-Amaro RL, Valentine ER, Carretero M, LeBoeuf SE, Rangaraju S, Broaddus CD, Solis GM, Williamson JR, Petrascheck M. Measuring Food Intake and Nutrient Absorption in Caenorhabditis elegans. Genetics. 2015; 200:443-454. DOI | PubMed
- Gordon PH, Moore DH, Miller RG, Florence JM, Verheijde JL, Doorish C, Hilton JF, Spitalny GM, MacArthur RB, Mitsumoto H, Neville HE, Boylan K, Mozaffar T, Belsh JM, Ravits J, Bedlack RS, Graves MC, McCluskey LF, Barohn RJ, Tandan R, Western ALS Study Group. Efficacy of minocycline in patients with amyotrophic lateral sclerosis: a phase III randomised trial. The Lancet Neurology. 2007; 6:1045-1053. DOI | PubMed
- Han J, Back SH, Hur J, Lin YH, Gildersleeve R, Shan J, Yuan CL, Krokowski D, Wang S, Hatzoglou M, Kilberg MS, Sartor MA, Kaufman RJ. ER-stress-induced transcriptional regulation increases protein synthesis leading to cell death. Nature Cell Biology. 2013; 15:481-490. DOI | PubMed
- Han S, Schroeder EA, Silva-García CG, Hebestreit K, Mair WB, Brunet A. Mono-unsaturated fatty acids link H3K4me3 modifiers to C. elegans lifespan. Nature. 2017; 544:185-190. DOI | PubMed
- Hansen M, Taubert S, Crawford D, Libina N, Lee SJ, Kenyon C. Lifespan extension by conditions that inhibit translation in Caenorhabditis elegans. Aging Cell. 2007; 6:95-110. DOI | PubMed
- Harrison DE, Strong R, Sharp ZD, Nelson JF, Astle CM, Flurkey K, Nadon NL, Wilkinson JE, Frenkel K, Carter CS, Pahor M, Javors MA, Fernandez E, Miller RA. Rapamycin fed late in life extends lifespan in genetically heterogeneous mice. Nature. 2009; 460:392-395. DOI | PubMed
- Henis-Korenblit S, Zhang P, Hansen M, McCormick M, Lee SJ, Cary M, Kenyon C. Insulin/IGF-1 signaling mutants reprogram ER stress response regulators to promote longevity. PNAS. 2010; 107:9730-9735. DOI | PubMed
- Houtkooper RH, Mouchiroud L, Ryu D, Moullan N, Katsyuba E, Knott G, Williams RW, Auwerx J. Mitonuclear protein imbalance as a conserved longevity mechanism. Nature. 2013; 497:451-457. DOI | PubMed
- Huang CL, Lee YC, Yang YC, Kuo TY, Huang NK. Minocycline prevents paraquat-induced cell death through attenuating endoplasmic reticulum stress and mitochondrial dysfunction. Toxicology Letters. 2012; 209:203-210. DOI | PubMed
- Jésus P, Fayemendy P, Nicol M, Lautrette G, Sourisseau H, Preux PM, Desport JC, Marin B, Couratier P. Hypermetabolism is a deleterious prognostic factor in patients with amyotrophic lateral sclerosis. European Journal of Neurology. 2018; 25:97-104. DOI | PubMed
- Jiang D, Rauda I, Han S, Chen S, Zhou F. Aggregation pathways of the amyloid β(1-42) peptide depend on its colloidal stability and ordered β-sheet stacking. Langmuir. 2012; 28:12711-12721. DOI | PubMed
- Kaeberlein M, Powers RW, Steffen KK, Westman EA, Hu D, Dang N, Kerr EO, Kirkland KT, Fields S, Kennedy BK. Regulation of yeast replicative life span by TOR and Sch9 in response to nutrients. Science. 2005; 310:1193-1196. DOI | PubMed
- Kapahi P, Zid BM, Harper T, Koslover D, Sapin V, Benzer S. Regulation of lifespan in Drosophila by modulation of genes in the TOR signaling pathway. Current Biology. 2004; 14:885-890. DOI | PubMed
- Kumsta C, Chang JT, Schmalz J, Hansen M. Hormetic heat stress and HSF-1 induce autophagy to improve survival and proteostasis in C. elegans. Nature Communications. 2017; 8DOI | PubMed
- Labbadia J, Morimoto RI. Repression of the Heat Shock Response Is a Programmed Event at the Onset of Reproduction. Molecular Cell. 2015; 59:639-650. DOI | PubMed
- Lampl Y, Boaz M, Gilad R, Lorberboym M, Dabby R, Rapoport A, Anca-Hershkowitz M, Sadeh M. Minocycline treatment in acute stroke: an open-label, evaluator-blinded study. Neurology. 2007; 69:1404-1410. DOI | PubMed
- Lapierre LR, De Magalhaes Filho CD, McQuary PR, Chu CC, Visvikis O, Chang JT, Gelino S, Ong B, Davis AE, Irazoqui JE, Dillin A, Hansen M. The TFEB orthologue HLH-30 regulates autophagy and modulates longevity in Caenorhabditis elegans. Nature Communications. 2013; 4DOI | PubMed
- Leigh PN, Meininger V, Bensimon G, Cudkowicz M, Robberecht W. Minocycline for patients with ALS. The Lancet Neurology. 2008; 7:119-120. DOI | PubMed
- Li Z, Zhang Y, Ku L, Wilkinson KD, Warren ST, Feng Y. The fragile X mental retardation protein inhibits translation via interacting with mRNA. Nucleic Acids Research. 2001; 29:2276-2283. DOI | PubMed
- Martin I, Kim JW, Lee BD, Kang HC, Xu JC, Jia H, Stankowski J, Kim MS, Zhong J, Kumar M, Andrabi SA, Xiong Y, Dickson DW, Wszolek ZK, Pandey A, Dawson TM, Dawson VL. Ribosomal protein s15 phosphorylation mediates LRRK2 neurodegeneration in Parkinson’s disease. Cell. 2014; 157:472-485. DOI | PubMed
- McColl G, Roberts BR, Pukala TL, Kenche VB, Roberts CM, Link CD, Ryan TM, Masters CL, Barnham KJ, Bush AI, Cherny RA. Utility of an improved model of amyloid-beta (Aβ₁₋₄₂) toxicity in Caenorhabditis elegans for drug screening for Alzheimer’s disease. Molecular Neurodegeneration. 2012; 7DOI | PubMed
- McCormick MA, Delaney JR, Tsuchiya M, Tsuchiyama S, Shemorry A, Sim S, Chou AC, Ahmed U, Carr D, Murakami CJ, Schleit J, Sutphin GL, Wasko BM, Bennett CF, Wang AM, Olsen B, Beyer RP, Bammler TK, Prunkard D, Johnson SC, Pennypacker JK, An E, Anies A, Castanza AS, Choi E, Dang N, Enerio S, Fletcher M, Fox L, Goswami S, Higgins SA, Holmberg MA, Hu D, Hui J, Jelic M, Jeong KS, Johnston E, Kerr EO, Kim J, Kim D, Kirkland K, Klum S, Kotireddy S, Liao E, Lim M, Lin MS, Lo WC, Lockshon D, Miller HA, Moller RM, Muller B, Oakes J, Pak DN, Peng ZJ, Pham KM, Pollard TG, Pradeep P, Pruett D, Rai D, Robison B, Rodriguez AA, Ros B, Sage M, Singh MK, Smith ED, Snead K, Solanky A, Spector BL, Steffen KK, Tchao BN, Ting MK, Vander Wende H, Wang D, Welton KL, Westman EA, Brem RB, Liu XG, Suh Y, Zhou Z, Kaeberlein M, Kennedy BK. A Comprehensive Analysis of Replicative Lifespan in 4,698 Single-Gene Deletion Strains Uncovers Conserved Mechanisms of Aging. Cell Metabolism. 2015; 22:895-906. DOI | PubMed
- McQuary PR, Liao CY, Chang JT, Kumsta C, She X, Davis A, Chu CC, Gelino S, Gomez-Amaro RL, Petrascheck M, Brill LM, Ladiges WC, Kennedy BK, Hansen M. C. elegans S6K Mutants Require a Creatine-Kinase-like Effector for Lifespan Extension. Cell Reports. 2016; 14:2059-2067. DOI | PubMed
- Medicherla B, Goldberg AL. Heat shock and oxygen radicals stimulate ubiquitin-dependent degradation mainly of newly synthesized proteins. The Journal of Cell Biology. 2008; 182:663-673. DOI | PubMed
- Metz LM, Zhang Y, Yeung M, Patry DG, Bell RB, Stoian CA, Yong VW, Patten SB, Duquette P, Antel JP, Mitchell JR. Minocycline reduces gadolinium-enhancing magnetic resonance imaging lesions in multiple sclerosis. Annals of Neurology. 2004; 55DOI | PubMed
- Mitra S, Zubay G, Landy A. Evidence for the preferential binding of the catabolite gene activator protein (CAP) to DNA containing the lac promoter. Biochemical and Biophysical Research Communications. 1975; 67:857-863. DOI | PubMed
- Mortison JD, Schenone M, Myers JA, Zhang Z, Chen L, Ciarlo C, Comer E, Natchiar SK, Carr SA, Klaholz BP, Myers AG. Tetracyclines Modify Translation by Targeting Key Human rRNA Substructures. Cell Chemical Biology. 2018. DOI | PubMed
- Nargund AM, Pellegrino MW, Fiorese CJ, Baker BM, Haynes CM. Mitochondrial import efficiency of ATFS-1 regulates mitochondrial UPR activation. Science. 2012; 337:587-590. DOI | PubMed
- Noble W, Garwood C, Stephenson J, Kinsey AM, Hanger DP, Anderton BH. Minocycline reduces the development of abnormal tau species in models of Alzheimer’s disease. The FASEB Journal. 2009; 23:739-750. DOI | PubMed
- Novoa I, Zhang Y, Zeng H, Jungreis R, Harding HP, Ron D. Stress-induced gene expression requires programmed recovery from translational repression. The EMBO Journal. 2003; 22:1180-1187. DOI | PubMed
- Obici L, Cortese A, Lozza A, Lucchetti J, Gobbi M, Palladini G, Perlini S, Saraiva MJ, Merlini G. Doxycycline plus tauroursodeoxycholic acid for transthyretin amyloidosis: a phase II study. Amyloid. 2012; 19:34-36. DOI | PubMed
- Pakos-Zebrucka K, Koryga I, Mnich K, Ljujic M, Samali A, Gorman AM. The integrated stress response. EMBO Reports. 2016; 17:1374-1395. DOI | PubMed
- Pan KZ, Palter JE, Rogers AN, Olsen A, Chen D, Lithgow GJ, Kapahi P. Inhibition of mRNA translation extends lifespan in Caenorhabditis elegans. Aging Cell. 2007; 6:111-119. DOI | PubMed
- Panici JA, Harper JM, Miller RA, Bartke A, Spong A, Masternak MM. Early life growth hormone treatment shortens longevity and decreases cellular stress resistance in long-lived mutant mice. The FASEB Journal. 2010; 24:5073-5079. DOI | PubMed
- Petrascheck M, Miller DL. Computational Analysis of Lifespan Experiment Reproducibility. Frontiers in Genetics. 2017; 8DOI | PubMed
- Plate L, Paxman RJ, Wiseman RL, Kelly JW. Modulating protein quality control. eLife. 2016; 5DOI | PubMed
- Prahlad V, Cornelius T, Morimoto RI. Regulation of the cellular heat shock response in Caenorhabditis elegans by thermosensory neurons. Science. 2008; 320:811-814. DOI | PubMed
- Rangaraju S, Solis GM, Thompson RC, Gomez-Amaro RL, Kurian L, Encalada SE, Niculescu AB, Salomon DR, Petrascheck M. Suppression of transcriptional drift extends C. elegans lifespan by postponing the onset of mortality. eLife. 2015; 4DOI | PubMed
- Roberts AM, Ward CC, Nomura DK. Activity-based protein profiling for mapping and pharmacologically interrogating proteome-wide ligandable hotspots. Current Opinion in Biotechnology. 2017; 43:25-33. DOI | PubMed
- Robida-Stubbs S, Glover-Cutter K, Lamming DW, Mizunuma M, Narasimhan SD, Neumann-Haefelin E, Sabatini DM, Blackwell TK. TOR signaling and rapamycin influence longevity by regulating SKN-1/Nrf and DAF-16/FoxO. Cell Metabolism. 2012; 15:713-724. DOI | PubMed
- Rogers AN, Chen D, McColl G, Czerwieniec G, Felkey K, Gibson BW, Hubbard A, Melov S, Lithgow GJ, Kapahi P. Life span extension via eIF4G inhibition is mediated by posttranscriptional remodeling of stress response gene expression in C. elegans. Cell Metabolism. 2011; 14:55-66. DOI | PubMed
- Seabrook TJ, Jiang L, Maier M, Lemere CA. Minocycline affects microglia activation, Abeta deposition, and behavior in APP-tg mice. Glia. 2006; 53:776-782. DOI | PubMed
- Selman C, Tullet JM, Wieser D, Irvine E, Lingard SJ, Choudhury AI, Claret M, Al-Qassab H, Carmignac D, Ramadani F, Woods A, Robinson IC, Schuster E, Batterham RL, Kozma SC, Thomas G, Carling D, Okkenhaug K, Thornton JM, Partridge L, Gems D, Withers DJ. Ribosomal protein S6 kinase 1 signaling regulates mammalian life span. Science. 2009; 326:140-144. DOI | PubMed
- Seo K, Choi E, Lee D, Jeong DE, Jang SK, Lee SJ. Heat shock factor 1 mediates the longevity conferred by inhibition of TOR and insulin/IGF-1 signaling pathways in C. elegans. Aging Cell. 2013; 12:1073-1081. DOI | PubMed
- Shore DE, Carr CE, Ruvkun G. Induction of cytoprotective pathways is central to the extension of lifespan conferred by multiple longevity pathways. PLoS Genetics. 2012; 8DOI | PubMed
- Shoulders MD, Ryno LM, Genereux JC, Moresco JJ, Tu PG, Wu C, Yates JR, Su AI, Kelly JW, Wiseman RL. Stress-independent activation of XBP1s and/or ATF6 reveals three functionally diverse ER proteostasis environments. Cell Reports. 2013; 3:1279-1292. DOI | PubMed
- Sidrauski C, McGeachy AM, Ingolia NT, Walter P. The small molecule ISRIB reverses the effects of eIF2α phosphorylation on translation and stress granule assembly. eLife. 2015; 4DOI
- Smith ED, Kaeberlein TL, Lydum BT, Sager J, Welton KL, Kennedy BK, Kaeberlein M. Age- and calorie-independent life span extension from dietary restriction by bacterial deprivation in Caenorhabditis elegans. BMC Developmental Biology. 2008; 8DOI | PubMed
- Solis GM, Petrascheck M. Measuring Caenorhabditis elegans life span in 96 well microtiter plates. Journal of Visualized Experiments. 2011. DOI | PubMed
- Steinkraus KA, Smith ED, Davis C, Carr D, Pendergrass WR, Sutphin GL, Kennedy BK, Kaeberlein M. Dietary restriction suppresses proteotoxicity and enhances longevity by an hsf-1-dependent mechanism in Caenorhabditis elegans. Aging Cell. 2008; 7:394-404. DOI | PubMed
- Syntichaki P, Troulinaki K, Tavernarakis N. eIF4E function in somatic cells modulates ageing in Caenorhabditis elegans. Nature. 2007; 445:922-926. DOI | PubMed
- Taylor RC, Dillin A. Aging as an event of proteostasis collapse. Cold Spring Harbor Perspectives in Biology. 2011; 3DOI | PubMed
- Tullet JM, Hertweck M, An JH, Baker J, Hwang JY, Liu S, Oliveira RP, Baumeister R, Blackwell TK. Direct inhibition of the longevity-promoting factor SKN-1 by insulin-like signaling in C. elegans. Cell. 2008; 132:1025-1038. DOI | PubMed
- van Ham TJ, Thijssen KL, Breitling R, Hofstra RM, Plasterk RH, Nollen EA. C. elegans model identifies genetic modifiers of alpha-synuclein inclusion formation during aging. PLoS Genetics. 2008; 4DOI | PubMed
- Vellai T, Takacs-Vellai K, Zhang Y, Kovacs AL, Orosz L, Müller F. Genetics: influence of TOR kinase on lifespan in C. elegans. Nature. 2003; 426DOI | PubMed
- Weindruch R, Walford RL. Dietary restriction in mice beginning at 1 year of age: effect on life-span and spontaneous cancer incidence. Science. 1982; 215:1415-1418. DOI | PubMed
- Weir HJ, Yao P, Huynh FK, Escoubas CC, Goncalves RL, Burkewitz K, Laboy R, Hirschey MD, Mair WB. Dietary Restriction and AMPK Increase Lifespan via Mitochondrial Network and Peroxisome Remodeling. Cell Metabolism. 2017; 26:884-896. DOI | PubMed
- Wilhelm T, Byrne J, Medina R, Kolundžić E, Geisinger J, Hajduskova M, Tursun B, Richly H. Neuronal inhibition of the autophagy nucleation complex extends life span in post-reproductive C. elegans. Genes & Development. 2017; 31:1561-1572. DOI | PubMed
- Xu G, Pattamatta A, Hildago R, Pace MC, Brown H, Borchelt DR. Vulnerability of newly synthesized proteins to proteostasis stress. Journal of Cell Science. 2016; 129:1892-1901. DOI | PubMed
- Ye X, Linton JM, Schork NJ, Buck LB, Petrascheck M. A pharmacological network for lifespan extension in Caenorhabditis elegans. Aging Cell. 2014; 13:206-215. DOI | PubMed
- Zheng H, Tang M, Zheng Q, Kumarapeli AR, Horak KM, Tian Z, Wang X. Doxycycline attenuates protein aggregation in cardiomyocytes and improves survival of a mouse model of cardiac proteinopathy. Journal of the American College of Cardiology. 2010; 56:1418-1426. DOI | PubMed
- Zhou C, Slaughter BD, Unruh JR, Guo F, Yu Z, Mickey K, Narkar A, Ross RT, McClain M, Li R. Organelle-based aggregation and retention of damaged proteins in asymmetrically dividing cells. Cell. 2014; 159:530-542. DOI | PubMed
Fonte
Solis GM, Kardakaris R, Valentine ER, Bar-Peled L, Chen AL, et al. () Translation attenuation by minocycline enhances longevity and proteostasis in old post-stress-responsive organisms. eLife 7e40314. https://doi.org/10.7554/eLife.40314





