Abstract
Introduzione
Le comunità microbiche delle budella dei mammiferi sono probabilmente strutturate da fattori sia ospitanti che microbici. Finora, un’ampia ricerca si è concentrata su come fattori ospiti come la dieta(David et al., 2014; Claesson et al., 2012), la genetica(Goodrich et al., 2014), la geografia (De Filippo et al., 2010; Yatsunenko et al., 2012; Arumugam et al., 2011) e lo stato immunitario(Hooper et al., 2012) modellano il microbiota intestinale. Tuttavia, il lavoro nei sistemi microbici a vita libera rivela che i batteri svolgono tipicamente un ruolo attivo nel plasmare il proprio ambiente(Shi e Norton, 2000; Gobbetti, 1998; Goddard, 2008; Osono, 2005; Rui et al., 2009; Gerbersdorf et al., 2009). L’identificazione di come questi driver interagiscono è necessaria sia per una più completa comprensione del microbiota intestinale, sia per sviluppare interventi razionali. Se le forze ecologiche si rivelano importanti, allora i cambiamenti compositivi saranno probabilmente il risultato di feedback tra i microbi e il loro ambiente.
Il potenziale redox, una metrica della capacità ambientale di ridurre le reazioni chimiche(cioè quelle che comportano il guadagno di elettroni) che si verificano, è una misura composita di vari fattori che influenzano la struttura del microbiota intestinale(Cowley et al., 2015; Friedman et al., 2017; Dhall et al., 2014). Gran parte delle nostre conoscenze su come vengono determinati i potenziali redox dell’intestino coinvolgono percorsi associati all’ospite(Spees et al., 2013; Rivera-Chávez et al., 2016). La diffusione passiva dell’ossigeno dall’epitelio aumenta il potenziale redox e stimola la crescita dei microbi aerobici(Espey, 2013; Albenberg et al., 2014). La secrezione di molecole immunitarie reattive reattive come le specie reattive dell’ossigeno o i nitrati è nota per essere una caratteristica dell’infiammazione che impone uno stress ossidativo ai microbi commensali e può essere sfruttata da agenti patogeni selezionati per colonizzare l’intestino(Winter et al., 2010; 2013; Faber et al., 2016; Spees et al., 2013; Rivera-Chávez et al., 2016; Kelly et al., 2015; David et al., 2015 ). Il mantenimento dell’omeostasi redox nel tessuto ospite può anche avere effetti di ricaduta sullo stato redox luminale(Circu e Aw, 2011).
Tuttavia, è probabile che il potenziale redox sia anche modellato dal metabolismo microbico. Nelle comunità microbiche a vita libera, la variazione degli accettatori di elettroni disponibili determina dove i microbi che utilizzano la respirazione possono prosperare; il metabolismo microbico differenziale che segue può produrre ulteriori cambiamenti nella disponibilità di accettatori di elettroni(Morris e Schmidt, 2013; Chen et al., 2017; Noll et al., 2005; Orcutt et al., 2011). Inoltre, il metabolismo microbico è stato proposto come meccanismo per gli stati a basso potenziale redox nel polmone dei pazienti affetti da fibrosi cistica(Cowley et al., 2015).
In questa sede, abbiamo studiato la natura delle dinamiche del potenziale redox sotto trattamento antibiotico per valutare l’importanza dei processi dell’ospite e dei processi microbici nella strutturazione delle comunità batteriche intestinali. Gli antibiotici disturbano direttamente i microbioti, ma ci si aspetta anche che alterino la biologia dell’ospite in relazione al potenziale redox. In particolare, gli antibiotici sono stati trovati per aumentare l’ossigenazione dell’epitelio intestinale come risultato dell’alterazione della composizione microbica e della segnalazione metabolica all’ospite(Kelly et al., 2015; Rivera-Chávez et al., 2016). Gli aumenti dell’ossigeno luminale dovuti alla diffusione dalla microvasculatura che fornisce l’epitelio porterebbero ad un maggiore potenziale redox in trattamento antibiotico. Inoltre, è stato dimostrato che le risposte dell’infiammazione dell’ospite al trattamento antibiotico e alla colonizzazione di agenti patogeni associati agli antibiotici producono accettatori di elettroni e altre molecole attive redox che causano stress ossidativo(Faber et al.,2016; Winter et al., 2010; 2013; Spees et al., 2013). Mentre questi percorsi sono stati dimostrati in precedenza, il loro impatto complessivo sul potenziale redox non è stato misurato. Inoltre, il contributo del metabolismo microbico al potenziale redox intestinale non è stato testato, anche se è noto che un’ampia gamma di batteri intestinali residenti può respirare sia aerobicamente che anaerobicamente(Ravcheev e Thiele, 2014). L’inibizione batterica guidata da antibiotici potrebbe aumentare la disponibilità di accettatori di elettroni e quindi servire come un altro meccanismo per i cambiamenti indotti da antibiotici nel potenziale redox intestinale. D’altra parte, l’inibizione antibiotica potrebbe limitare la produzione batterica di agenti ossidanti, determinando così una diminuzione complessiva del potenziale redox sotto antibiotici.
I cambiamenti del potenziale redox sotto antibiotici dovrebbero a loro volta fornire una visione delle forze che strutturano la composizione e la funzione del microbiota. L’aumento del potenziale redox dovuto a una risposta immunitaria dell’ospite potrebbe limitare il microbiota oltre la mortalità diretta da antibiotici, poiché l’infiammazione introdurrebbe un ulteriore stress ossidativo negli ecosistemi microbici. Al contrario, un potenziale redox elevato dovuto all’accumulo di ossigeno o di accettatori di elettroni anaerobici favorirebbe la crescita dei batteri che respirano. Il disturbo antibiotico produce una successione riproducibile della comunità nell’intestino in seguito al trattamento – in particolare, una fioritura transitoria nelle Enterobacteriaceae (Antonopoulos et al., 2009; David et al., 2015; Young e Schmidt, 2004; Theriot et al., 2014; Peterfreund et al., 2012; Dethlefsen et al., 2008; Jakobsson et al., 2010; Looft e Allen, 2012), che è stata attribuita a un aumento della disponibilità di ossigeno (David et al., 2015). Ma, il ruolo del potenziale redox durante questo processo di successione non è stato studiato in precedenza. Se il potenziale redox cambiasse a causa del trattamento antibiotico, prevediamo che il recupero dello stato redox sarebbe necessario per la resilienza della comunità, cioè per il recupero compositivo fino a uno stato di pre-disturbo (Shadeet al., 2012). Inoltre, ci aspettiamo che il feedback tra la comunità(cioè la componente biotica del sistema) e il loro ambiente(cioè la componente abiotica) guidi ulteriori cambiamenti potenziali di redox durante il periodo di successione. Un tale modello evidenzierebbe il potenziale di manipolazione del potenziale redox per alterare le dinamiche della comunità dopo il disturbo.
In questo caso, abbiamo combinato studi antibiotici in vivo ed ex vivo per isolare gli effetti dei percorsi dell’ospite e dei microbi sullo stato redox nell’intestino del mammifero. Abbiamo confermato che il potenziale redox è aumentato durante il trattamento antibiotico in associazione con alcuni cambiamenti nello stato immunitario dell’ospite. Tuttavia, linee di prova multiple in entrambi i topi e un modello di intestino umano artificiale hanno suggerito che i cambiamenti indotti dagli antibiotici nel metabolismo microbico erano sufficienti a causare un aumento del potenziale redox. Dopo la fine dei trattamenti antibiotici, abbiamo osservato il recupero del redox entro una settimana. Un ritorno consecutivo alla composizione convenzionale della comunità si è verificato, tuttavia, solo quando i topi sono stati coabitati e hanno condiviso i microbioti dell’intestino, indicando che la dispersione microbica è necessaria al di là del recupero ambientale per il ritorno della normale struttura della comunità di microbioti.
Risultati
Gli antibiotici hanno causato un significativo aumento del potenziale di redox fecale del topo entro un giorno dal trattamento
Per cinque giorni, abbiamo fatto un gavage orale a una coorte di topi convenzionali con un cocktail di antibiotici (ampicillina, vancomicina, metronidazolo e neomicina[Reikvam et al., 2011]) per inibire ampiamente i batteri intestinali. Abbiamo misurato il potenziale redox nelle feci appena svuotati con un microelettrodo accoppiato con un elettrodo di riferimento al giorno. Entro sedici ore dopo la prima dose di antibiotici, il potenziale redox è aumentato significativamente da 37 ± 164 mV al basale a 227 ± 45 mV (p=0,04 Bonferroni-corretto Mann-Whitney U test; Figura 1A). Durante il trattamento, il potenziale redox differisce nel complesso tra i topi trattati e quelli di controllo (p=0,005 test di probabilità del modello di effetti misti lineari).
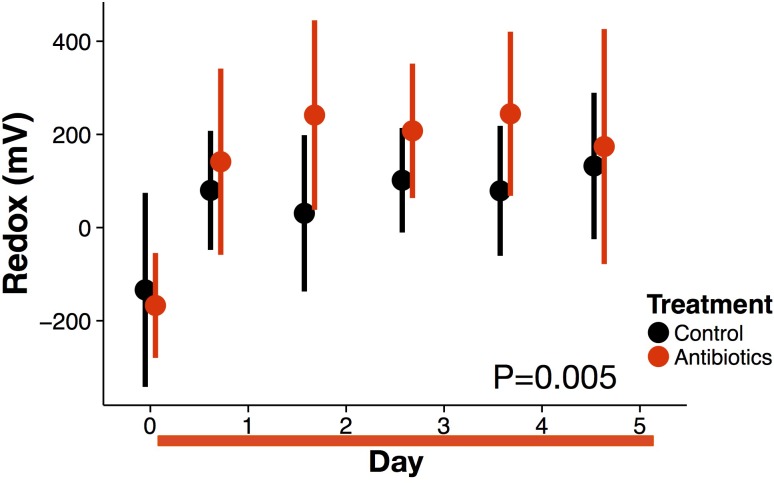
Figura 1-figura supplemento 2.Figura 1—figura 2. Effetti del trattamento antibiotico sullo stato redox dell’intestino e sull’infiammazione dell’ospite.(A) Lo stato di Redox misurato in feci fresche di topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) è diverso (p=0,005, test di probabilità del modello di effetti lineari misti). I dati replicati sono presentati nella figura 1-figure supplement 2.(B) L’espressione genica per quattro geni associati all’infiammazione misurata con RT-qPCR di RNA isolato dalle feci nell’ultimo giorno di trattamento antibiotico (n = 6-7) differisce dai livelli di controllo (p<0,05, Bonferroni-corretto un-campione t-test).(C) L’infiammazione intestinale misurata come concentrazione fecale del biomarcatore lipocalina-2 (n = 9-10 per trattamento) differiva tra i topi trattati e quelli di controllo (p<0,001 linear mixed effects model likelihood tests). I dati sono mostrati come mezzi ± SD. I risultati dei test post-hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Infiammazione intestinale misurata come concentrazione fecale del biomarcatore lipocalina-2 (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Lo stato di Redox misurato in feci fresche di topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) differisce (p=0,005 test di probabilità del modello di effetti misti lineari). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.

Figura 1-figure supplement 2.Effetti del trattamento antibiotico sullo stato redox dell’intestino e sull’infiammazione dell’ospite.(A) Lo stato di Redox misurato in feci fresche di topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) è diverso (p=0,005, test di probabilità del modello di effetti lineari misti). I dati replicati sono presentati nella figura 1-figure supplement 2.(B) L’espressione genica per quattro geni associati all’infiammazione misurata con RT-qPCR di RNA isolato dalle feci nell’ultimo giorno di trattamento antibiotico (n = 6-7) differisce dai livelli di controllo (p<0,05, Bonferroni-corretto un-campione t-test).(C) L’infiammazione intestinale misurata come concentrazione fecale del biomarcatore lipocalina-2 (n = 9-10 per trattamento) differiva tra i topi trattati e quelli di controllo (p<0,001 linear mixed effects model likelihood tests). I dati sono mostrati come mezzi ± SD. I risultati dei test post-hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Infiammazione intestinale misurata come concentrazione fecale del biomarcatore lipocalina-2 (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Lo stato di Redox misurato in feci fresche di topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) differisce (p=0,005 test di probabilità del modello di effetti misti lineari). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
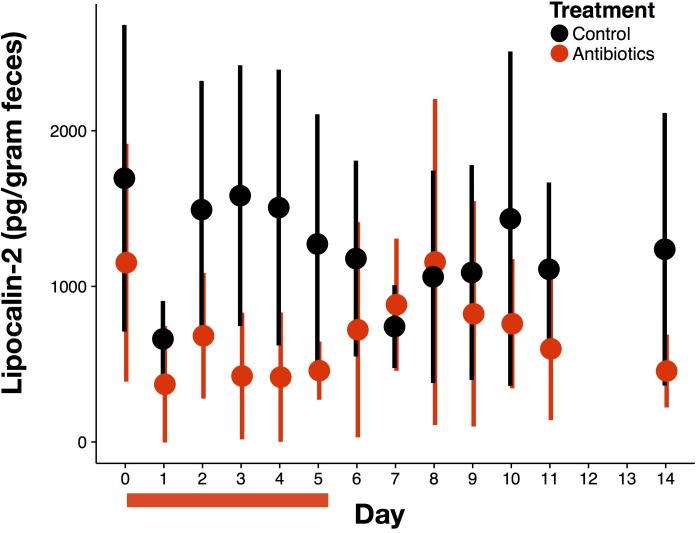
Figura 1-figure supplement 1.Effetti complessivi degli antibiotici sull’infiammazione.Infiammazione intestinale misurata come concentrazione fecale del biomarcatore lipocalina-2 (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.

Figura 1-figure supplement 2.Figura 1—supplemento di figura 2. Dati potenziali di Redox durante il trattamento da replicare corsa sperimentale.Redox stato misurato in feci appena annullate di feci trattate (rosso) e di controllo (nero) topi (n = 9-10 per trattamento) differivano (p = 0,005 lineare effetti misti modello di test di probabilità del modello di effetti lineari). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
Gli antibiotici di prova alterano la biologia dell’ospite per aumentare il potenziale redox
Abbiamo esaminato prima di tutto se gli antibiotici influenzano il potenziale redox attraverso gli effetti diretti dell’ospite mediati da una risposta immunitaria. L’espressione di tre geni dell’ospite nei campioni fecali era coerente con l’ipotesi che gli spostamenti redox fossero associati all’attivazione immunitaria. Abbiamo misurato gli aumenti relativi ai controlli in Nos2, che è legato ai livelli di specie di azoto reattivo(Dedon e Tannenbaum, 2004; Winterbourn, 2008); aumenti relativi ai controlli in Rela, che è stato trovato per contribuire a infiammazione di tipo IBD nei topi attraverso una risposta proinfiammatoria citochina(Waddell et al., 2013); e, diminuisce rispetto ai controlli in Apoa4, che ha una nota funzione antinfiammatoria(Broedl et al., 2007) (p<0,05, Bonferroni-corretto un-campione t-test; Figura 1B). Tuttavia, altri biomarcatori non hanno associato il trattamento antibiotico con una risposta immunitaria. L’espressione Nfkb1, che è associata alla soppressione dell’infiammazione(Cartwright et al., 2016), è aumentata dopo il trattamento antibiotico. Il trattamento antibiotico è stato anche seguito da una piccola, ma significativa, diminuzione dei livelli di lipocalina-2, che è un biomarcatore proteico dell’infiammazione(Chassaing et al., 2012) (p<0,001, linear mixed effects model likelihood tests; Figura 1C). Presi nel loro insieme, i nostri test sui biomarcatori hanno fornito prove equivoche per l’infiammazione intestinale nei topi trattati con antibiotici.
Risposte batteriche agli antibiotici associate a spostamento potenziale redox
Per testare se l’inibizione delle popolazioni batteriche da parte degli antibiotici potesse contribuire direttamente alla dinamica del potenziale redox, abbiamo utilizzato un sistema intestinale umano ex vivo basato su un bioreattore a flusso continuo(McDonald et al., 2013), che ha permesso la propagazione di una comunità microbica stabile rappresentativa del microbiota intestinale umano con tutti i principali phyla rappresentati(Figura 2-figure supplement 1). Il trattamento di questo sistema con lo stesso cocktail antibiotico utilizzato nello studio sui topi ha portato ad un aumento del potenziale redox rispetto ad un controllo non trattato (p=0,005, test di probabilità del modello di effetti lineari misti; figura 2A). Il potenziale redox è aumentato di 59 ± 47 mV entro quindici ore dalla prima dose di antibiotico, rispecchiando la rapidità con cui gli spostamenti redox si sono verificati in vivo (Figura 1A). Il potenziale redox nel nostro modello ex vivo è aumentato di nuovo di altri 141 ± 37 mV dopo una seconda dose di antibiotico (Figura 2-figuresupplement 1). Così, in assenza di interazioni dirette tra gli antibiotici e un ospite, il trattamento antibiotico ha prodotto cambiamenti nel potenziale redox ambientale sperimentato da una comunità microbica intestinale.
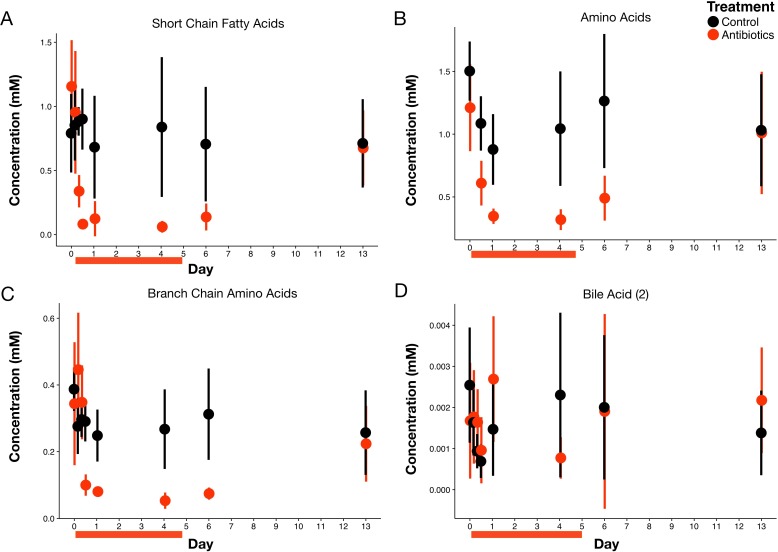
Figura 2-figure supplemento 3.Figura 2—figura 3. Risposta microbica agli antibiotici ex vivo e in vivo.gli esperimenti dei bioreattori ex vivo dimostrano che il solo cambiamento microbico può alterare i livelli di redox.gli effetti complessivi degli antibiotici sulla carica batterica.gli effetti complessivi degli antibiotici sui metaboliti.(A) Il potenziale di redox dal controllo (nero) e dai vasi del bioreattore trattati con antibiotici (rosso) che coltivano le comunità microbiche dell’intestino umano (n = 3 per trattamento) è diverso (p=0,005, test di probabilità del modello lineare di effetti misti).(B) Il carico microbico, misurato come numero di copie del gene 16S rRNA nei topi trattati con antibiotici e nei topi di controllo è diminuito durante le prime 24 ore di trattamento per i topi trattati con antibiotici (n = 9-10 per trattamento; p<0,001, Bonferroni-corretto Mann-Whitney U test).(C) I metaboliti misurati con la spettrometria NMR delle feci (n = 9-10 per trattamento; vedi Figura 2-figure supplement 3 e Supplementary file 1) sono diminuiti nei topi trattati con antibiotici durante le prime 24 ore (p<0.001, Bonferroni-corretto Mann-Whitney U test). I dati sono indicati come mezzi ± SD. I risultati dei test ad hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del Giorno 0; le barre rosse indicano la durata del trattamento.(A) L’impostazione sperimentale ha comportato l’acclimatazione dei vasi per 2 giorni (il primo giorno senza l’alimentazione di nuovi media) prima di sperimentare due dosi di antibiotici di un cocktail tra cui ampicillina, vancomicina, metronidazolo e neomicina (vedi Materiali e metodi).(B) Composizione comunitaria dei vasi di controllo per ogni replicare eseguito durante il periodo di sperimentazione.(C) Potenziale di Redox misurato in continuo con elettrodi per due giorni con dosi di antibiotici (se del caso) a 0 e 24 (N = 3 per trattamento). Le frecce rosse indicano la dose di antibiotico.Copia il gene 16S rRNA come quantificato da qPCR nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Le classi di metaboliti misurate con la spettrometria NMR delle feci (n = 9-10 per trattamento) comprendono gli acidi grassi a catena corta (A), gli amminoacidi (B), gliamminoacidi a catena ramificata (C) e una sottoclasse di acidi biliari (D). Vedere il file supplementare 1 per i risultati dei singoli metaboliti con analisi statistiche. I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.

Figura 2-figure supplement 3.Figura 2—figura 3. Risposta microbica agli antibiotici ex vivo e in vivo.gli esperimenti dei bioreattori ex vivo dimostrano che il solo cambiamento microbico può alterare i livelli di redox.gli effetti complessivi degli antibiotici sulla carica batterica.gli effetti complessivi degli antibiotici sui metaboliti.(A) Il potenziale di redox dal controllo (nero) e dai vasi del bioreattore trattati con antibiotici (rosso) che coltivano le comunità microbiche dell’intestino umano (n = 3 per trattamento) è diverso (p=0,005, test di probabilità del modello lineare di effetti misti).(B) Il carico microbico, misurato come numero di copie del gene 16S rRNA nei topi trattati con antibiotici e nei topi di controllo è diminuito durante le prime 24 ore di trattamento per i topi trattati con antibiotici (n = 9-10 per trattamento; p<0,001, Bonferroni-corretto Mann-Whitney U test).(C) I metaboliti misurati con la spettrometria NMR delle feci (n = 9-10 per trattamento; vedi Figura 2-figure supplement 3 e Supplementary file 1) sono diminuiti nei topi trattati con antibiotici durante le prime 24 ore (p<0.001, Bonferroni-corretto Mann-Whitney U test). I dati sono indicati come mezzi ± SD. I risultati dei test ad hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del Giorno 0; le barre rosse indicano la durata del trattamento.(A) L’impostazione sperimentale ha comportato l’acclimatazione dei vasi per 2 giorni (il primo giorno senza l’alimentazione di nuovi media) prima di sperimentare due dosi di antibiotici di un cocktail tra cui ampicillina, vancomicina, metronidazolo e neomicina (vedi Materiali e metodi).(B) Composizione comunitaria dei vasi di controllo per ogni replicare eseguito durante il periodo di sperimentazione.(C) Potenziale di Redox misurato in continuo con elettrodi per due giorni con dosi di antibiotici (se del caso) a 0 e 24 (N = 3 per trattamento). Le frecce rosse indicano la dose di antibiotico.Copia il gene 16S rRNA come quantificato da qPCR nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Le classi di metaboliti misurate con la spettrometria NMR delle feci (n = 9-10 per trattamento) comprendono gli acidi grassi a catena corta (A), gli amminoacidi (B), gliamminoacidi a catena ramificata (C) e una sottoclasse di acidi biliari (D). Vedere il file supplementare 1 per i risultati dei singoli metaboliti con analisi statistiche. I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
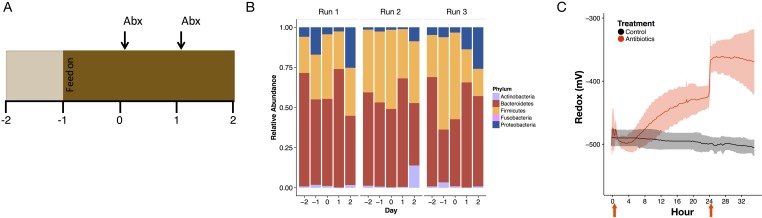
Figura 2-figure supplement 1.Esperimenti di bioreattore ex vivo dimostrano che il solo cambiamento microbico può alterare i livelli di redox.(A) La configurazione sperimentale ha comportato l’acclimatazione dei vasi per 2 giorni (il primo giorno senza nuovi media di alimentazione in) prima di sperimentare due dosi di antibiotici di un cocktail tra cui ampicillina, vancomicina, metronidazolo, e neomicina (vedi Materiali e metodi).(B) Composizione comunitaria dei vasi di controllo per ogni replicare eseguito durante il periodo di sperimentazione.(C) Potenziale di Redox misurato in continuo con elettrodi per due giorni con dosi di antibiotici (se del caso) a 0 e 24 (N = 3 per trattamento). Le frecce rosse indicano la dose di antibiotico.
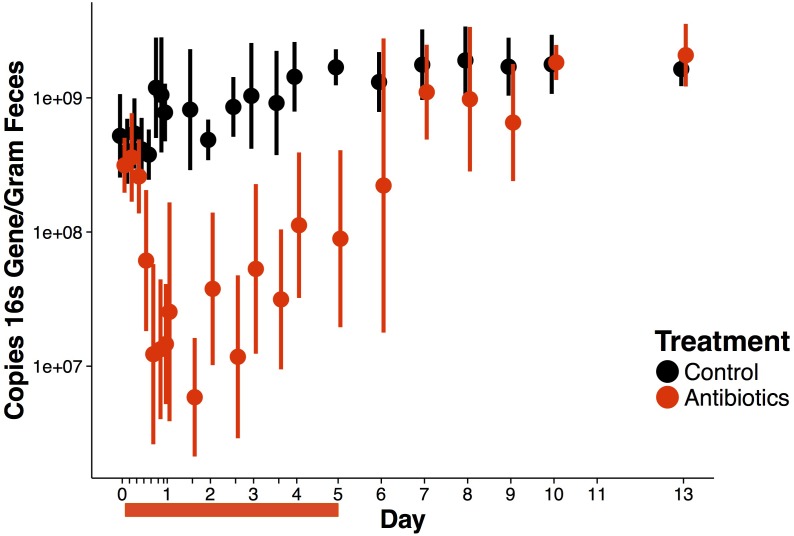
Figura 2-figure supplement 2.Effetti complessivi degli antibiotici sulla carica batterica.Copia il gene 16S rRNA come quantificato da qPCR nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.

Figura 2-figure supplement 3.Effetti antibiotici complessivi sui metaboliti.Le classi di metaboliti misurate con la spettrometria NMR delle feci (n = 9-10 per trattamento) comprendevano gli acidi grassi a catena corta (A), gli aminoacidi (B), gliaminoacidi a catena ramificata (C) e una sottoclasse di acidi biliari (D). Vedere il file supplementare 1 per i risultati dei singoli metaboliti con analisi statistiche. I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
Il potenziale redox fecale sotto antibiotici è associato al metabolismo batterico e alla respirazione
Per capire come gli effetti degli antibiotici sulle popolazioni batteriche possano portare a cambiamenti nel potenziale redox intestinale, abbiamo studiato la dinamica e il metabolismo dei microbioti attraverso il trattamento nei topi. Abbiamo osservato livelli depressi di carica batterica e di attività metabolica che si sono verificati entro poche ore dagli spostamenti iniziali del potenziale redox. Le concentrazioni batteriche fecali sono diminuite significativamente entro dodici ore dal trattamento antibiotico (p=0,01, test di Mann-Whitney U corretto da Bonferroni; Figura 2B), e sono rimaste significativamente al di sotto dei controlli durante i cinque giorni di trattamento (p<0,001, test di Mann-Whitney U corretto da Bonferroni; Figura 2-figure supplement 2). Utilizzando la metabolomica basata su NMR, abbiamo scoperto che gli acidi grassi a catena corta propionato e acetato, prodotti finali delle principali vie metaboliche microbiche, sono diminuiti otto ore dopo il trattamento antibiotico (p<0,05, Bonferroni-corretto Mann-Whitney U test; Figura 2-figure supplement 3, file supplementare 1). Ventuno dei ventotto metaboliti misurati, compresi altri acidi grassi a catena corta, amminoacidi, amminoacidi a catena ramificata e un gruppo di acidi biliari, in seguito sono diminuiti significativamente in seguito al trattamento antibiotico (p<0,05, Bonferroni-corretto Mann-Whitney U test; Figura 2C, Figura 2-figure supplement 3, file supplementare 1). La dinamica di dodici di questi metaboliti è stata associata in modo significativo al trattamento complessivo (p<0,05, test di probabilità del modello di effetti lineari misti; file supplementare 1). Così, la tempistica e la persistenza di una diminuzione del carico intestinale e dell’attività microbica ha coinciso con l’aumento del potenziale redox fecale.
Successivamente, abbiamo misurato la concentrazione di tre principali accettatori di elettroni utilizzati durante la respirazione microbica per valutare se i cambiamenti in questi percorsi hanno contribuito all’aumento del potenziale redox. In primo luogo, la misurazione dell’ossigeno luminale con un nuovo sistema di sensori in vivo, un sensore idrogel con covalentemente attaccato derivato Pd-porfirina sensibile all’ossigeno è stato inserito rettalmente e poi letto otticamente attraverso la pelle, abbiamo osservato un significativo aumento dei livelli di ossigeno luminale il giorno dopo il trattamento antibiotico (3.7 ± 3,6 Torr al basale a 26,5 ± 26,1 Torr; p=0,02, Bonferroni-corretto Mann-Whitney U test; Figura 3A). I lavori precedenti(Kelly et al., 2015; Rivera-Chávez et al., 2016) hanno mostrato un’elevata ossigenazione dei tessuti sotto antibiotici, che potrebbe portare a una maggiore diffusione e quindi a un maggiore ossigeno luminoso. Inoltre, la riduzione della respirazione aerobica dovuta alla mortalità per antibiotici o allo stress potrebbe anche aumentare i livelli di ossigeno intestinale. Tuttavia, i livelli di ossigeno intestinale nei topi trattati con antibiotici sono tornati ai livelli di controllo di quarantotto ore dopo la prima dose (p>0,05, test di Mann-Whitney U corretto da Bonferroni), e gli antibiotici non sono stati associati a un effetto complessivo sui livelli di ossigeno intestinale (p>0,05 test di probabilità del modello di effetti misti lineari). Inoltre, tra i topi trattati, i livelli di ossigeno intestinale non sono stati correlati con il potenziale redox (p> 0,05, correlazione misure ripetute; Figura 3-figure supplemento 1). Questi dati insieme suggeriscono che l’aumento dell’ossigenazione luminale durante il trattamento antibiotico può contribuire a turni iniziali del potenziale redox, ma non sono stati responsabili di turni redox persistente.
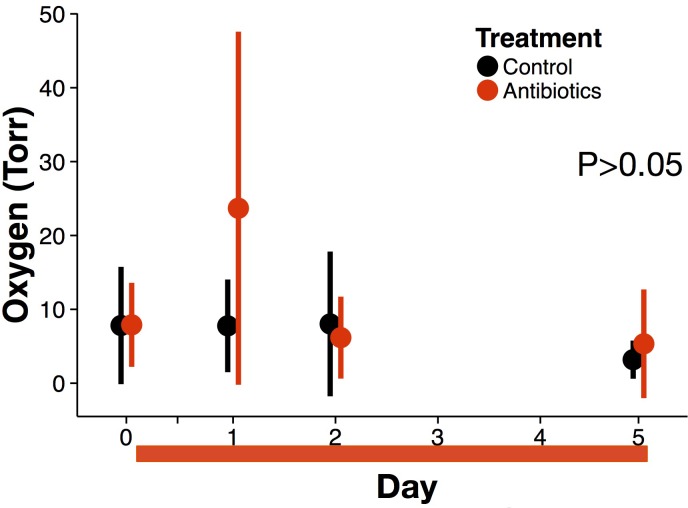
Figura 3-figure supplement 3.Il livello di accettabilità degli elettroni durante il trattamento antibiotico.il livello di redox non è determinato dalla concentrazione di ossigeno.gli effetti complessivi degli antibiotici sull’ossigeno.i dati relativi all’ossigeno durante il trattamento da replicare durante l’esecuzione sperimentale.(A) Intestino crasso concentrazione di ossigeno luminale misurata in vivo per i topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) non differiscono nel complesso durante il trattamento (p> 0,05, lineare effetti misti modello di test di probabilità del modello di effetti). I dati replicati sono presentati nella Figura 3-figure supplement 2.(B) Concentrazione di nitrato fecale (n = 2-9 per gruppo per punto di tempo) ha fatto differenza tra i topi trattati e di controllo durante il trattamento (p = 0,02, lineare misto effetti misti modello test di probabilità del modello di effetti).(C) Anche la concentrazione di fumarato fecale (n = 2-9 per gruppo per punto temporale) è risultata diversa (p<0,001, linear mixed effects model likelihood tests). I livelli degli accettatori di elettroni al di sotto del livello di rilevazione non sono qui tracciati. Per le misurazioni del fumarato in cui tutti gli animali trattati erano al di sotto del limite di rilevazione, i valori sono stati impostati a 200 nmol per i test statistici. I dati sono mostrati come mezzi ± SD. I risultati dei test post-hoc per i singoli punti temporali (* indica p<0,05 Bonferroni-corretto Mann-Whitney U test) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Ripetute misure di correlazione tra la concentrazione di ossigeno luminale e il potenziale redox intestinale dei topi trattati (rosso) e di controllo (nero) (n = 72).L’ossigeno luminale è stato misurato in vivo durante e dopo il trattamento antibiotico per tutti i topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.L’ossigeno misurato in vivo dei topi trattati (rosso) e dei topi di controllo (nero) (n = 9-10 per trattamento) non differisce (p>0,05 linear mixed effects model likelihood tests). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
Al contrario, abbiamo osservato un aumento sostenuto e significativo dei livelli di nitrati durante il trattamento antibiotico. Il nitrato è uno degli accettatori di elettroni più usati nell’intestino(Ravcheev e Thiele, 2014; Fischer e Lindenmayer, 2007) e la sua riduzione ad azoto gassoso (a pH 7) ha un potenziale di +0,75 V che lo rende uno dei più potenti accettatori di elettroni dopo l’ossigeno. L’aumento dell’espressione di Nos2(Figura 1B) ci ha portato a ipotizzare che il nitrato aumenterebbe in quanto è un gene che codifica l’ossido nitrico sintasi inducibile e la cui espressione è stata trovata per conferire un vantaggio di crescita alla respirazione dei nitrati competente ceppo E. coli(Winter et al., 2013). Livelli più bassi di respirazione microbica potrebbero anche portare ad un accumulo di nitrato separato dai cambiamenti nell’espressione dell’ospite. Abbiamo misurato i livelli di nitrato fecale con il metodo dell’acido cromotropico(West e Ramachandran, 1966) per un sottoinsieme di punti temporali quando redox variava, e abbiamo trovato un aumento significativo di nitrato (p = 0,03, Mann-Whitney U test; Figura 3B) a partire da meno di 24 ore dopo la prima dose di antibiotici. Questo aumento è persistito per tutta la durata del trattamento, e il trattamento antibiotico complessivo ha prodotto un aumento significativo del nitrato (p=0,02, test di probabilità del modello di effetti lineari misti).
Infine, abbiamo utilizzato un dosaggio enzimatico colorimetrico per misurare i livelli di fumarato fecale per un sottoinsieme di punti temporali durante il trattamento antibiotico. Le riduttasi di fumarato sono state trovate in oltre un terzo dei genomi dei microbi intestinali, e il fumarato è il più comune accettore terminale di elettroni per la respirazione anaerobica batterica(Kröger et al., 1992). A differenza del nitrato e dell’ossigeno, tuttavia, gran parte del fumarato nell’intestino è probabilmente prodotto dal microbiota stesso(Fischbach e Sonnenburg, 2011; El Aidy et al., 2013). Nel complesso, c’è stato un effetto significativo del trattamento sui livelli di fumarato (p<0,001, linear mixed effects model likelihood tests; Figura 3C). Abbiamo osservato un picco nei livelli di fumarato 4 ore dopo la prima dose di antibiotici (p=0,004, test di Mann-Whitney U corretto da Bonferroni); tuttavia, questo aumento si era dissipato di 8 ore dopo la prima dose e successivamente, durante il trattamento, abbiamo osservato una significativa diminuzione (al di sotto del limite di rilevazione) dei livelli di fumarato. Così, non tutti gli accettatori di elettroni sono aumentati in abbondanza durante il trattamento antibiotico.

Figura 3-figure supplemento 3.I livelli degli accettori di elettroni durante il trattamento antibiotico.il livello di Redox non è determinato dalla concentrazione di ossigeno.gli effetti complessivi degli antibiotici sull’ossigeno.i dati relativi all’ossigeno durante il trattamento da replicare durante l’esecuzione sperimentale.(A) Intestino crasso concentrazione di ossigeno luminale misurata in vivo per i topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) non differiscono nel complesso durante il trattamento (p> 0,05, lineare effetti misti modello di test di probabilità del modello di effetti). I dati replicati sono presentati nella Figura 3-figure supplement 2.(B) Concentrazione di nitrato fecale (n = 2-9 per gruppo per punto di tempo) ha fatto differenza tra i topi trattati e di controllo durante il trattamento (p = 0,02, lineare misto effetti misti modello test di probabilità del modello di effetti).(C) Anche la concentrazione di fumarato fecale (n = 2-9 per gruppo per punto temporale) è risultata diversa (p<0,001, linear mixed effects model likelihood tests). I livelli degli accettatori di elettroni al di sotto del livello di rilevazione non sono qui tracciati. Per le misurazioni del fumarato in cui tutti gli animali trattati erano al di sotto del limite di rilevazione, i valori sono stati impostati a 200 nmol per i test statistici. I dati sono mostrati come mezzi ± SD. I risultati dei test post-hoc per i singoli punti temporali (* indica p<0,05 Bonferroni-corretto Mann-Whitney U test) sono inclusi ai fini della visualizzazione. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.Ripetute misure di correlazione tra la concentrazione di ossigeno luminale e il potenziale redox intestinale dei topi trattati (rosso) e di controllo (nero) (n = 72).L’ossigeno luminale è stato misurato in vivo durante e dopo il trattamento antibiotico per tutti i topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.L’ossigeno misurato in vivo dei topi trattati (rosso) e dei topi di controllo (nero) (n = 9-10 per trattamento) non differisce (p>0,05 linear mixed effects model likelihood tests). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
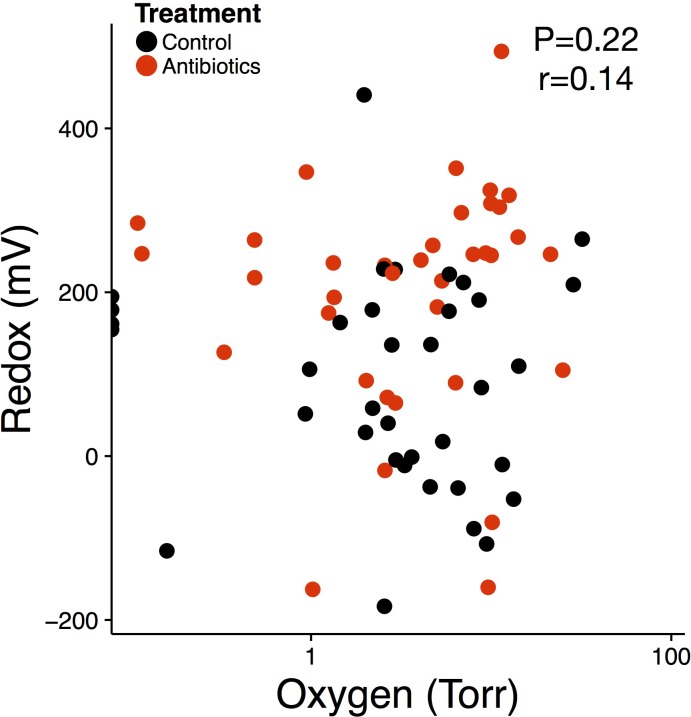
Figura 3-figure supplement 1.Il livello di Redox non è guidato dalla concentrazione di ossigeno.2. Ripetute misure di correlazione tra la concentrazione di ossigeno luminale e il potenziale redox intestinale dei topi trattati (rosso) e di controllo (nero) (n = 72).
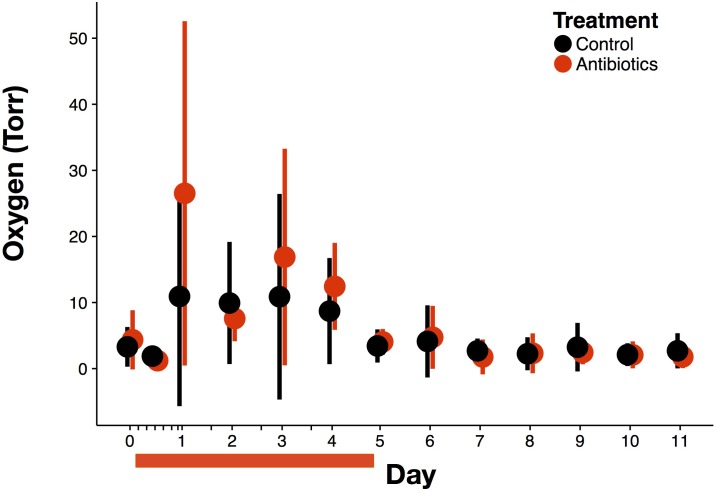
Figura 3-figure supplemento 2.Effetti antibiotici complessivi sull’ossigeno.L’ossigeno luminoso è stato misurato in vivo durante e dopo il trattamento antibiotico per tutti i topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.

Figura 3-figure supplement 3.Figura 3—supplemento di figura 3. Dati sull’ossigeno durante il trattamento da replicare la corsa sperimentale.Ossigeno misurato in vivo dei topi trattati (rosso) e di controllo (nero) (n = 9-10 per trattamento) non differiscono (p>0,05 lineare effetti misti modello di test di probabilità del modello di effetti lineari). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
La composizione della comunità e l’ambiente intestinale recuperano dopo il trattamento antibiotico
La successione ecologica, la dinamica “un po’ ordinata e prevedibile” (Fierereret al., 2010) di una comunità dopo una perturbazione, è nota per seguire il trattamento antibiotico (Antonopoulos et al., 2009; David et al., 2015). Ciò che è meno noto è come le condizioni abiotiche cambino durante la successione post-antibiotica e, per estensione, come le dinamiche biotiche e abiotiche si interfacciano in quel periodo. Dopo aver dimostrato che sia la comunità che l’ambiente intestinale non sono resistenti ai disturbi da antibiotici, abbiamo cercato di determinare quanto sono resilienti, cioè quanto velocemente si sono ripresi(Shade et al., 2012; Allison e Martiny, 2008). In questo studio, abbiamo infatti osservato cambiamenti biotici consistenti nel tempo nei topi che hanno ricevuto antibiotici(Figura 4-figure supplement 1). Carica batterica intestinale recuperato rapidamente dopo il trattamento su tutti i topi trattati, non mostrando alcuna differenza rispetto ai topi di controllo di due giorni (p> 0,05, Bonferroni-corretto Mann-Whitney U test; Figura 2-figure supplement 2). Un recupero riproducibile della diversità beta è stato osservato dopo una settimana (p>0,05, Bonferroni-corretto Mann-Whitney U test di Bonferroni; Figura 4-figure supplement 2).
Per caratterizzare il recupero di specifici taxa microbici, abbiamo raggruppato i generi batterici fecali in gruppi secondo i loro modelli dinamici attraverso il trattamento antibiotico(Figura 4A,B). Ci siamo concentrati sui cinque gruppi con un’abbondanza media di almeno l’1% nel nostro set di dati(file supplementare 2). Tra questi cinque gruppi, tre differivano significativamente durante il trattamento (p<0,05, test di probabilità del modello di effetti lineari misti; Figura 4A,B). In particolare, i due gruppi elevati durante il trattamento hanno incluso molti taxa anaerobici facoltativi(file supplementare 2). Uno di questi, il cluster composto principalmente da membri del Enterobacteriacaeae, è rimasto elevato anche all’inizio del periodo di recupero (p<0,05, Bonferroni-corretto Mann-Whitney U test corretti). Questo arricchimento è in linea con le precedenti scoperte che le Enterobacteriaceae fioriscono spesso dopo il trattamento antibiotico(Antonopoulos et al., 2009; David et al., 2015; Young e Schmidt, 2004; Theriot et al., 2014; Peterfreund et al., 2012; Dethlefsen et al., 2008; Jakobsson et al., 2010; Looft e Allen, 2012). Quel cluster, così come uno composto principalmente da Akkermansia e uno di un consorzio di tipici taxa commensali, ha mostrato significativi effetti residui del trattamento durante il periodo di recupero (p<0,05, linear mixed effects model likelihood tests; Figura 4A,B). Tuttavia, alla fine del periodo di recupero di una settimana, l’abbondanza di tutti i cluster era indistinguibile tra gli animali trattati e quelli di controllo (p>0,05, test di Mann-Whitney U corretti da Bonferroni).
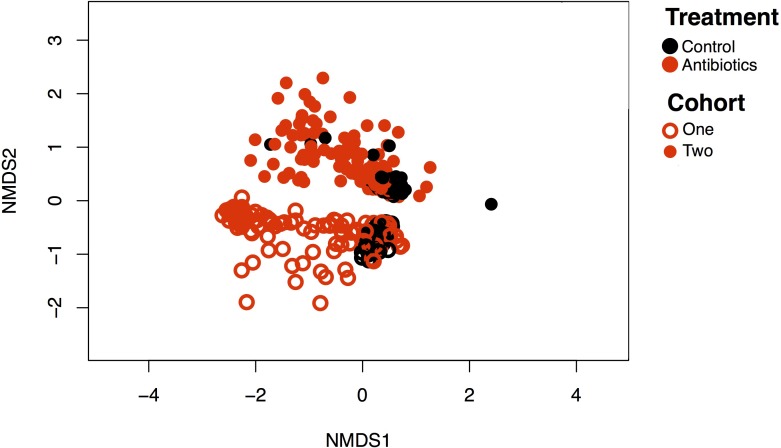
Figura 4-figure supplemento 5.La comunità intestinale è resistente ai disturbi da antibiotici.la composizione della comunità guidata dagli antibiotici cambia.gli effetti generali degli antibiotici sulla biodiversità beta.l’abbondanza delle enterobatteriacee è associata a condizioni abiotiche e a cambiamenti nello stato redox.la successione post-antibiotica può avvenire entro una settimana ma è mediata dallo stato di cohousing dei topi.le coorti di topi differiscono nello stato originale della comunità ma non nelle tendenze di risposta.(A-B) Dinamiche composizionali dei cluster più abbondanti durante e dopo il trattamento per il controllo (A) e i topi trattati (B) (n = 9-10 per trattamento). Tre gruppi (Tipico, Staph / Strep, Enterobacteriaceae) differivano significativamente tra i gruppi (p<0,05, lineare effetti misti test di probabilità del modello di effetti misti) durante il trattamento e due (Enterobacteriaceae, Akkermansia) durante il recupero.(C) Il potenziale di Redox misurato nelle feci appena svuotati di topi alloggiati singolarmente durante il periodo di recupero (n = 5 per trattamento) era inferiore a quello dei topi trattati inizialmente (p<0,05, Bonferroni-corretto Mann-Whitney U test).(D) La correlazione delle misure ripetute è stata significativa (p=0,02) tra la variazione dell’abbondanza di Enterobacteriaceae e la variazione del potenziale redox durante i primi tre giorni di recupero post-antibiotico (n = 64).(E) Giorni fino a quando non vi è alcuna differenza significativa (p>0,05 test di Mann Whitney U corretto da Bonferroni) tra i gruppi di controllo e gli animali trattati per l’abbondanza di redox e di Enterobacteriaceae, sia in alloggiamenti singoli che in coabitazione. Le barre rosse indicano la durata del trattamento(A-B). Le frecce rosse indicano l’ultima dose di antibiotico; le misurazioni del sesto giorno sono più di 24 ore dopo l’ultima dose(C). I dati sono indicati come mezzi ± SD. I risultati dei test ad hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione.NMDS delle dinamiche della comunità durante e dopo il trattamento antibiotico (i cerchi rossi pieni sono trattati i topi durante il trattamento; i cerchi rossi vuoti sono trattati i topi durante il recupero; i cerchi neri sono i controlli). Il trattamento ha avuto un effetto significativo sulla composizione (p<0,05, PERMANOVA).Bray-curtis dissimile alla composizione di base della comunità batterica nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.(A) Ripetute misure di correlazione tra l’abbondanza relativa di Enterobacteriaceae nelle feci e il potenziale redox dei topi trattati (rosso) e di controllo (nero) (n = 85).(B) Ripetute misure di correlazione tra l’abbondanza relativa delle Enterobacteriaceae nelle feci e la concentrazione di ossigeno (n = 116).Cambiamenti della comunità misurati come dissimiglianza Bray-Curtis rispetto alla composizione del giorno 0 per i topi trattati singolarmente rispetto a quelli trattati in co-cohoused (rosso) e di controllo (nero) (n = 9-10 per trattamento). I topi co-alloggiati trattati con antibiotici erano indistinguibili dai topi di controllo alla fine del periodo di recupero (p>0,05, Bonferroni-corretto Mann-Whitney U test), ma i topi trattati singolarmente non lo erano. I dati sono mostrati come mezzi ± SD. Le barre rosse indicano la durata del trattamento antibiotico.NMDS delle dinamiche di comunità per le due coorti di topi Charles River C57BL/6 acquistati in tempi diversi (i cerchi pieni e i cerchi vuoti sono coorti separate). I simboli neri sono animali di controllo e i simboli rossi sono animali trattati con antibiotici. La coorte ha avuto un effetto significativo sulla composizione (p<0,05, R2 = 0,17 PERMANOVA) così come il trattamento (p<0,05, R2 = 0,07), ma non vi è stata alcuna interazione significativa (p=0,3).
Anche le condizioni abiotiche nell’intestino del topo erano resilienti e il recupero era prevedibile. Il potenziale di Redox è tornato ai livelli di controllo entro una settimana dalla fine del trattamento (p> 0,05, Bonferroni-corretto Mann-Whitney U test; Figura 4C). In particolare, però, abbiamo osservato il giorno dopo la cessazione della terapia antibiotica, i potenziali redox fecali nei topi trattati erano significativamente diminuiti rispetto ai topi di controllo (-138,3 ± 149,8 mV vs 54,6 ± 211 mV; p = 0,006, Bonferroni-corretto Mann-Whitney U test), e non è stato fino a più tardi nel recupero che il potenziale redox è tornato a livelli di controllo in molti topi. I livelli di ossigeno e di biomarker dell’infiammazione sono rimasti bassi per tutto il periodo di recupero (p>0,05, Bonferroni-corretto Mann-Whitney U test; Figura 3-figure supplement 2, Figura 1-figure supplement 1, rispettivamente) coerente con i loro cambiamenti minimi durante il trattamento. Inoltre, molti prodotti metabolici microbici misurati da NMR avevano recuperato due giorni dopo le ultime dosi, e tutti avevano recuperato entro la fine della settimana di recupero(Figura 2-figure supplement 3; file supplementare 1). Di concerto, un recupero biotico e abiotico ampiamente riproducibile ha avuto luogo tra i topi trattati con antibiotici nei giorni successivi al trattamento antibiotico, indicando la resilienza complessiva di questo ecosistema ai disturbi antibiotici.

Figura 4-figure supplement 5.L’abbondanza delle enterobatteriacee è associata a condizioni abiotiche e a cambiamenti nello stato redox.la successione post-antibiotica può avvenire entro una settimana, ma è mediata dallo stato di cohousing dei topi.le coorti di topi differiscono nello stato originale della comunità, ma non nelle tendenze di risposta.(A-B) Dinamiche composizionali dei cluster più abbondanti durante e dopo il trattamento per il controllo (A) e i topi trattati (B) (n = 9-10 per trattamento). Tre gruppi (Tipico, Staph / Strep, Enterobacteriaceae) differivano significativamente tra i gruppi (p<0,05, lineare effetti misti test di probabilità del modello di effetti misti) durante il trattamento e due (Enterobacteriaceae, Akkermansia) durante il recupero.(C) Il potenziale di Redox misurato nelle feci appena svuotati di topi alloggiati singolarmente durante il periodo di recupero (n = 5 per trattamento) era inferiore a quello dei topi trattati inizialmente (p<0,05, Bonferroni-corretto Mann-Whitney U test).(D) La correlazione delle misure ripetute è stata significativa (p=0,02) tra la variazione dell’abbondanza di Enterobacteriaceae e la variazione del potenziale redox durante i primi tre giorni di recupero post-antibiotico (n = 64).(E) Giorni fino a quando non vi è alcuna differenza significativa (p>0,05 test di Mann Whitney U corretto da Bonferroni) tra i gruppi di controllo e gli animali trattati per l’abbondanza di redox e di Enterobacteriaceae, sia in alloggiamenti singoli che in coabitazione. Le barre rosse indicano la durata del trattamento(A-B). Le frecce rosse indicano l’ultima dose di antibiotico; le misurazioni del sesto giorno sono più di 24 ore dopo l’ultima dose(C). I dati sono indicati come mezzi ± SD. I risultati dei test ad hoc per i singoli punti temporali (* indica p<0,05 test di Mann-Whitney U corretto da Bonferroni) sono inclusi ai fini della visualizzazione.NMDS delle dinamiche della comunità durante e dopo il trattamento antibiotico (i cerchi rossi pieni sono trattati i topi durante il trattamento; i cerchi rossi vuoti sono trattati i topi durante il recupero; i cerchi neri sono i controlli). Il trattamento ha avuto un effetto significativo sulla composizione (p<0,05, PERMANOVA).Bray-curtis dissimile alla composizione di base della comunità batterica nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.(A) Ripetute misure di correlazione tra l’abbondanza relativa di Enterobacteriaceae nelle feci e il potenziale redox dei topi trattati (rosso) e di controllo (nero) (n = 85).(B) Ripetute misure di correlazione tra l’abbondanza relativa delle Enterobacteriaceae nelle feci e la concentrazione di ossigeno (n = 116).Cambiamenti della comunità misurati come dissimiglianza Bray-Curtis rispetto alla composizione del giorno 0 per i topi trattati singolarmente rispetto a quelli trattati in co-cohoused (rosso) e di controllo (nero) (n = 9-10 per trattamento). I topi co-alloggiati trattati con antibiotici erano indistinguibili dai topi di controllo alla fine del periodo di recupero (p>0,05, Bonferroni-corretto Mann-Whitney U test), ma i topi trattati singolarmente non lo erano. I dati sono mostrati come mezzi ± SD. Le barre rosse indicano la durata del trattamento antibiotico.NMDS delle dinamiche di comunità per le due coorti di topi Charles River C57BL/6 acquistati in tempi diversi (i cerchi pieni e i cerchi vuoti sono coorti separate). I simboli neri sono animali di controllo e i simboli rossi sono animali trattati con antibiotici. La coorte ha avuto un effetto significativo sulla composizione (p<0,05, R2 = 0,17 PERMANOVA) così come il trattamento (p<0,05, R2 = 0,07), ma non vi è stata alcuna interazione significativa (p=0,3).
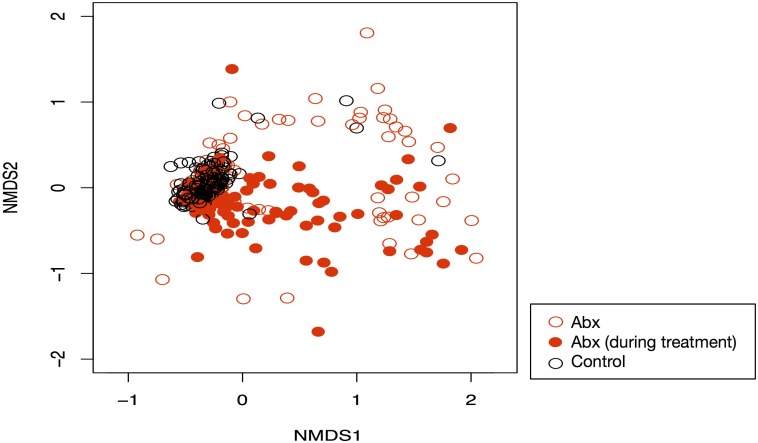
Figura 4-figure supplemento 1.Cambiamenti nella composizione della comunità guidata dagli antibiotici.NMDS delle dinamiche della comunità durante e dopo il trattamento antibiotico (i cerchi rossi pieni sono trattati i topi durante il trattamento; i cerchi rossi vuoti sono trattati i topi durante il recupero; i cerchi neri sono i controlli). Il trattamento ha avuto un effetto significativo sulla composizione (p<0,05, PERMANOVA).
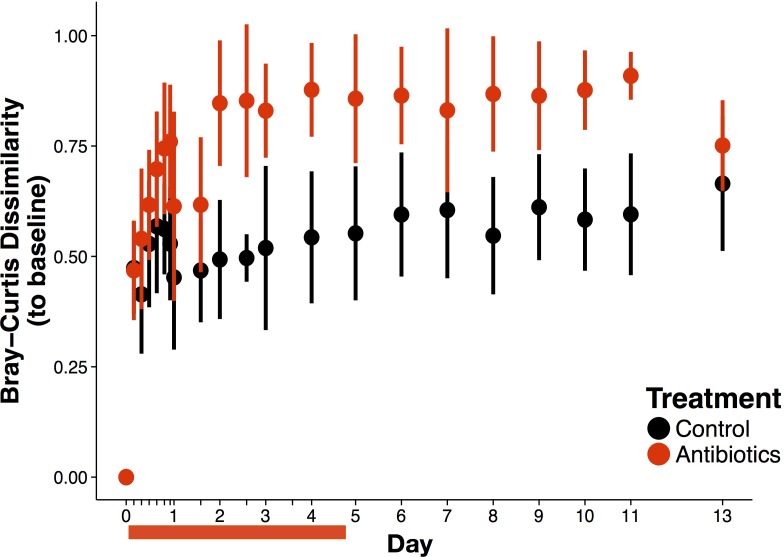
Figura 4-figure supplemento 2.Effetti complessivi antibiotici sulla biodiversità beta.Bray-curtis dissimile alla composizione di base della comunità batterica nel tempo (n = 9-10 per trattamento). I dati sono indicati come mezzi ± SD. Il trattamento antibiotico è iniziato dopo la misurazione del giorno 0; le barre rosse indicano la durata del trattamento.
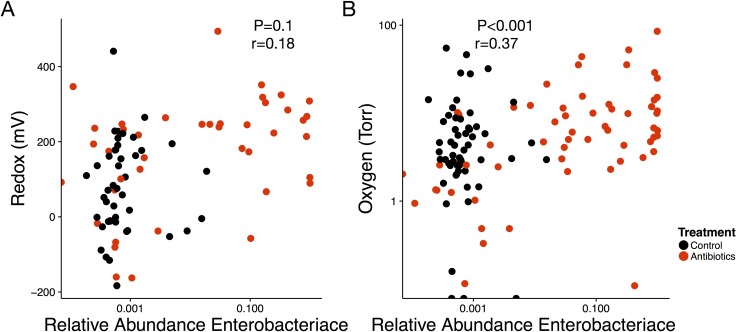
Figura 4-figure supplement 3.L’abbondanza delle Enterobacteriaceae è associata a condizioni abiotiche e cambiamenti nello stato redox.(A) Ripetute misure di correlazione tra l’abbondanza relativa delle Enterobacteriaceae nelle feci e il potenziale redox dei topi trattati (rosso) e di controllo (nero) (n = 85).(B) Ripetute misure di correlazione tra l’abbondanza relativa delle Enterobacteriaceae nelle feci e la concentrazione di ossigeno (n = 116).
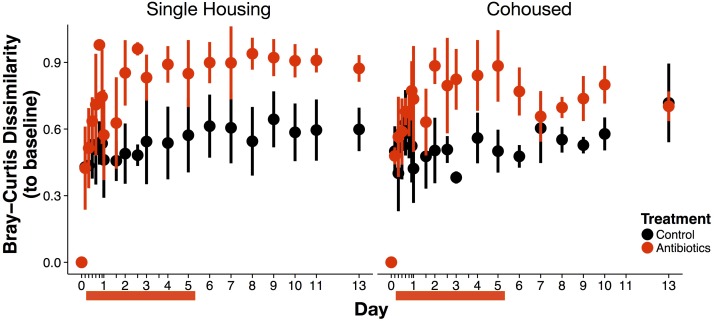
Figura 4-figure supplement 4.La successione post-antibiotica può avvenire entro una settimana, ma è mediata dallo stato di cohousing dei topi.Cambiamenti della comunità misurati come la dissimiglianza Bray-Curtis rispetto alla composizione del giorno 0 per i topi trattati singolarmente rispetto a quelli trattati in cohousing (rosso) e di controllo (nero) (n = 9-10 per trattamento). I topi co-alloggiati trattati con antibiotici erano indistinguibili dai topi di controllo alla fine del periodo di recupero (p>0,05, Bonferroni-corretto Mann-Whitney U test), ma i topi trattati singolarmente non lo erano. I dati sono mostrati come mezzi ± SD. Le barre rosse indicano la durata del trattamento antibiotico.

Figura 4-figure supplemento 5.Le coorti di topi differiscono per lo stato originale della comunità, ma non per le tendenze di risposta.NMDS delle dinamiche della comunità per le due coorti di topi Charles River C57BL/6 acquistati in tempi diversi (i cerchi pieni e i cerchi vuoti sono coorti separate). I simboli neri sono animali di controllo e i simboli rossi sono animali trattati con antibiotici. La coorte ha avuto un effetto significativo sulla composizione (p<0,05, R2 = 0,17 PERMANOVA) così come il trattamento (p<0,05, R2 = 0,07), ma non vi è stata alcuna interazione significativa (p=0,3).
La successione post-antibiotica è il risultato dell’interazione tra recupero biotico e abiotico
Nello studio delle potenziali interazioni tra i processi abiotici e biotici nel recupero degli antibiotici, abbiamo ipotizzato che le Enterobacteriaceae potrebbero prosperare in condizioni di alto potenziale redox e avere un impatto diretto sullo stato redox dell’intestino. I membri di questa famiglia batterica sono stati arricchiti al termine del trattamento nella nostra analisi di clustering(Figura 4B). Le Enterobacteriaceae possono anche impiegare molti accettatori di elettroni terminali per eseguire la respirazione aerobica e anaerobica(Ravcheev e Thiele, 2014) e quindi potrebbero probabilmente prosperare in condizioni di alto potenziale redox e di impatto diretto dello stato redox dell’intestino. Abbiamo trovato che l’abbondanza relativa delle Enterobacteriaceae era marginalmente correlata con il potenziale redox (p = 0,1, r =, 0,18, correlazione misure ripetute; Figura 4-figure supplemento 3) durante il trattamento e il recupero. Inoltre, la variazione del potenziale redox durante il periodo di recupero precedente è stata correlata negativamente con le variazioni dell’abbondanza assoluta delle Enterobacteriaceae (calcolata come numero totale di copie del gene 16S rRNA moltiplicato per l’abbondanza relativa) per entrambi gli animali trattati (p=0,03, r = -0,42, correlazione misure ripetute) e per tutti gli animali (p=0,02, r = -0,34, correlazione misure ripetute; Figura 4D). Insieme, queste osservazioni suggeriscono che l’aumento della respirazione da parte delle Enterobacteriaceae dopo il trattamento antibiotico può portare i potenziali redox a diminuire al di sotto dei livelli convenzionali. Più in generale, le nostre osservazioni sostengono l’ipotesi che le dinamiche delle Enterobacteriaceae contribuiscono alla naturale variazione del potenziale redox nell’intestino.
Abbiamo poi indagato se il recupero abiotico ha portato al recupero composizionale o se la dispersione(cioè la migrazione dei microbi) da popolazioni microbiche non colpite è stata necessaria affinché i microbioti mostrassero resilienza e tornassero ad uno stato di pretrattamento. Ricerche precedenti hanno dimostrato che i topi trattati con antibiotici in un solo alloggiamento presentano una composizione comunitaria alterata per settimane dopo il trattamento(Antonopoulos et al., 2009). Abbiamo quindi coabitato la metà dei topi trattati con animali di controllo durante il periodo di recupero e ne abbiamo tenuto la metà in alloggiamenti singoli. Poiché i topi praticano la coprofagia(cioè l’ingestione di feci), ci aspetteremmo che il cohousing reintroducesse i normali microbi commensali in competizione con le Enterobacteriaceae e altre specie precoci di successione, riducendo così la loro abbondanza. Alla fine del periodo di trattamento di una settimana, abbiamo infatti osservato un successo differenziale nel recupero della comunità tra i gruppi di alloggiamento. Le comunità di topi co-alloggiati erano tornate a controllare la composizione, mentre i topi alloggiati singolarmente non lo avevano fatto. Per i topi co-alloggiati, alla fine del periodo di recupero, il microbiota intestinale degli animali trattati era altrettanto dissimile dalla linea di base come lo era per i topi di controllo, indicando che la comunità aveva recuperato (p>0,05, Bonferroni-corretto Mann-Whitney U test; Figura 4-figure supplemento 4). Sia il potenziale redox fecale che i livelli di Enterobacteriaceae sono tornati ai livelli convenzionali più rapidamente nei topi co-alloggiati rispetto ai topi alloggiati singolarmente(Figura 4E). Al contrario, i topi trattati in alloggiamenti singoli hanno continuato ad essere più dissimili alla fine del recupero rispetto ai topi di controllo in alloggiamenti singoli (p<0,001, Bonferroni-corretto Mann-Whitney U test; Figura 4-figure supplement 4). Tale deriva è qualitativamente coerente con gli argomenti che la variazione stocastica può influenzare il modo in cui le comunità rispondono alle perturbazioni, come si è visto nelle comunità microbiche del suolo(Zhang et al., 2016). Più in generale, le differenze tra topi singoli e topi co-alloggiati evidenziano qui che il recupero ambientale non è sufficiente a produrre il recupero biotico – la dispersione è necessaria anche per una comunità resiliente.
Discussione
Qui abbiamo scoperto che una caratteristica chiave dell’ambiente microbico, il potenziale redox, può essere rapidamente e drammaticamente alterato nell’intestino sotto trattamento antibiotico. Questo cambiamento è stato accompagnato da cambiamenti nella disponibilità degli accettatori di elettroni. La direzione e i tempi dello spostamento del potenziale redox erano associati alla dinamica microbica e potevano essere riprodotti con un modello ex vivo dell’intestino umano in assenza di effetti dell’ospite. Inoltre, abbiamo trovato prove che la cinetica del potenziale redox era guidata da spostamenti del carico microbico e del metabolismo, in particolare tra gli anaerobi facoltativi della famiglia delle Enterobacteriaceae. Anche se c’erano prove di un aumento di alcune vie dell’ospite della produzione di accettatori di elettroni, non abbiamo trovato cambiamenti persistenti e significativi nell’infiammazione dell’ospite. Pertanto, proponiamo la respirazione microbica degli accettatori di elettroni provenienti dall’ospite agisce come un importante fattore determinante del potenziale redox nell’intestino, oltre alla produzione dell’ospite stesso. Il potenziale redox ha di conseguenza modellato le dinamiche di successione dopo la cessazione del trattamento, ma il ritorno al controllo delle condizioni redox è stato insufficiente a produrre un recupero comunitario. La dispersione è stata necessaria per la struttura della comunità per mostrare la piena resilienza ai disturbi antibiotici.
Le nostre scoperte implicano che gli accettatori di elettroni sono una risorsa critica normalmente in competizione con i batteri residenti e, come tali, sono fattori chiave che strutturano l’ecologia microbica intestinale. La capacità di respirare un’ampia gamma di accettatori di elettroni è già nota per essere cruciale per gli agenti patogeni per colonizzare l’intestino(Winter et al., 2010; 2013; Faber et al., 2016). Tuttavia, la ricerca ecologica finora condotta sui microbioti commensali si è concentrata principalmente sulla disponibilità di substrati(Donaldson et al., 2016 e relativi riferimenti, Pereira e Berry, 2017) o, in misura minore, sull’ossigeno(Espey, 2013). I nostri risultati qui supportano l’ipotesi che la disponibilità di altri accettatori di elettroni sia un importante fattore ecologico che guida anche la struttura delle popolazioni batteriche residenti(Jones et al., 2007; 2011). Se fosse vera, questa ipotesi implicherebbe che la resistenza alla colonizzazione dei patogeni da parte dei microbi commensali potrebbe essere mediata dalla competizione batterica che coinvolge le vie respiratorie.
Il lavoro futuro sarà necessario per tracciare un quadro completo delle dinamiche e del panorama chimico attraverso il quale i batteri nell’intestino competono per gli accettatori di elettroni. Mentre qualsiasi molecola attiva redox potrebbe contribuire a cambiamenti potenziali redox (quindi testare tutti i possibili driver chimici di redox è impossibile), ci siamo concentrati qui su tre specie di accettatori di elettroni rilevanti (ossigeno, nitrato e fumarato) che derivano da percorsi indipendenti e possono essere rappresentativi della dinamica complessiva. I nostri dati ci portano a congetturare che la disponibilità di ossigeno sia aumentata in modo transitorio a causa della ridotta capacità respiratoria di una comunità batterica vincolata da antibiotici. Ma, con l’aumentare della disponibilità di ossigeno, gli anaerobi facoltativi rimanenti sono passati alla respirazione aerobica, lasciando accumulare altri accettatori di elettroni di origine ospitante, come il nitrato. Gli accettatori di elettroni accumulati, in particolare il nitrato, possono anche aver contribuito a modificare la fisiologia dell’ospite, come evidenziato da un upregulation del gene Nos2, che è legato ai livelli di specie reattive dell’azoto(Dedon e Tannenbaum, 2004; Winterbourn, 2008). Tuttavia, non tutti gli accettatori di elettroni accumulati durante il trattamento antibiotico. In particolare, i nostri dati suggeriscono che quelli prodotti dal microbiota piuttosto che dall’ospite, come il fumarato, erano meno disponibili sotto pressione antibiotica.
I nostri dati supportano anche un modello in cui il consumo da parte di specifici taxa batterici forma omeostasi redox nell’intestino. Osserviamo che gli anaerobi facoltativi come le Enterobacteriaceae si comportano come taxa pionieri le cui dinamiche tracciano inversamente la dinamica redox dopo il trattamento antibiotico, fornendo una nuova comprensione del meccanismo deterministico che guida la fioritura consistente di questi batteri dopo il trattamento antibiotico(Antonopoulos et al., 2009; David et al., 2015; Young e Schmidt, 2004; Theriot et al., 2014; Peterfreund et al., 2012; Dethlefsen et al., 2008; Jakobsson et al., 2010; Looft e Allen, 2012). Le enterobatteriacee sono particolarmente adatte a sfruttare la disponibilità di accettatori di elettroni perché il loro diverso repertorio di percorsi di respirazione(Ravcheev e Thiele, 2014; Jones et al., 2007; 2011) consente tassi di crescita rapida in un regime ad alto redox. Le enterobatteriacee e più in generale i geni delle vie respiratorie sono più diffusi anche nelle viscere dei topi infiammati(Hughes et al., 2017), un altro ambiente in cui è previsto un elevato potenziale redox. In caso di infiammazione sperimentale, tuttavia, l’espansione delle Enterobacteriaceae può essere prevenuta con il trattamento con tungstato, un inibitore della respirazione(Zhu et al., 2018). Questo risultato conferma il nostro modello secondo cui i membri delle Enterobacteriaceae usano la respirazione per sbocciare in condizioni di disturbo e mette in evidenza il potenziale di manipolazione della disponibilità o dell’usabilità degli accettatori di elettroni per ingegnerizzare il microbiota.
Un aumento della disponibilità di risorse è comune in seguito a disturbi ecologici e può promuovere la crescita di taxa pionieri, che sono i più adatti a rispondere rapidamente a un picco di risorse, ma che attirano la risorsa portando così al loro stesso spostamento(Connell e Slatyer ,1977; Peet e Christensen, 1980; Tilman, 1985). Qui, come in molti casi di successione ecologica, le Enterobacteriaceae sono state alla fine sostituite da membri della comunità più convenzionali che tipicamente crescono meglio in condizioni più restrittive; infatti, la relazione tra il cambiamento dell’abbondanza delle Enterobacteriaceae e il cambiamento dello stato redox è coerente con questi taxa che facilitano il successo dei colonizzatori secondari. Tuttavia, il motivo per cui il recupero del redox è stato in ultima analisi isterico(cioè, l’intestino è diventato temporaneamente un ambiente riducente dopo la fine del trattamento) rimane poco chiaro. È stato precedentemente dimostrato che i membri delle Enterobacteriaceae producono agenti riducenti, come l’H2S, in risposta al trattamento antibiotico(Shatalin et al., 2011), ma questa teoria richiede ulteriori indagini in un contesto di comunità e in un contesto associato all’ospite.
Oltre a identificare un ruolo per i microbioti nella modellazione dei potenziali redox intestinali, i nostri risultati illustrano in modo più ampio come i fattori microbici possano essere importanti, oltre ai fattori ospiti, per determinare le condizioni abiotiche nell’intestino attraverso il bilanciamento della produzione e del consumo di risorse. Mentre l’ospite è in ultima analisi la fonte di tutti i substrati nell’intestino, l’azione microbica determina l’ambiente realizzato che modella la composizione e la funzione della comunità. Tale ingegneria dell’ecosistema microbico(Jones et al., 1994; Wright e Jones, 2006) è coerente con la nostra comprensione di come i microbi possono plasmare i loro ambienti in sistemi a vita libera(Gerbersdorf et al., 2009; Goddard, 2008). Le alterate disponibilità di risorse che risultano dall’azione microbica contribuiscono poi a strutturare la comunità microbica stessa. Tali feedback possono contribuire a una serie di fenomeni nei sistemi associati all’ospite. I costituenti comuni dell’intestino(ad esempio, i phyla Firmicutes e i Batteroidetes) potrebbero perpetuare per sé stessi ambienti ospitali simili, indipendentemente dalla biologia dell’ospite, portando a un’ampia coerenza di composizione a livello di phylum vista tra gli esseri umani, indipendentemente dalla dieta, dalla geografia o dalla genetica(David et al., 2014; Claesson et al., 2012; Goodrich et al., 2014; De Filippo et al., 2010; Yatsunenko et al., 2012; Arumugam et al., 2011). Le variazioni nel modo in cui l’ambiente intestinale è dettato dai microbi possono anche aiutare a spiegare le differenze interindividuali in risposta agli interventi in assenza di effetti ospite coerenti (ad esempio, Venkataraman et al., 2016). La segnalazione metabolica microbica può inoltre alterare la produzione dell’ospite di substrati, comprese le molecole attive redox. In particolare, è stato dimostrato che i livelli di butirrato ridotti, un cambiamento qui osservato, aumentano i livelli di nitrato intestinale(Byndloss et al., 2017) e l’ossigenazione epiteliale dell’ospite(Rivera-Chávez et al., 2016).
Conclusioni
Insieme, le nostre scoperte suggeriscono nuovi modelli ecologici per come gli antibiotici rimodellano il microbiota intestinale e per come i cambiamenti del redox potrebbero essere associati alle malattie enteriche. Gli antibiotici sono trionfi della medicina moderna che hanno ridotto drasticamente la mortalità per malattie infettive(Armstrong et al., 1999). Ma, stiamo sempre più imparando che gli antibiotici rimodellano in modo significativo anche il microbiota intestinale residente, lasciando un’impronta che può durare da mesi ad anni dopo il trattamento(Dethlefsen e Relman, 2011; Jakobsson et al., 2010) e predisponendo gli ospiti all’obesità(Cho et al., 2012), alle allergie alimentari(Stefka et al., 2014), alle malattie autoimmuni (Russell et al., 2012) e all’aumento del rischio di infezione(Stecher et al., 2007; Buffie et al., 2012; Wiström et al., 2001). Mentre questi farmaci riducono i livelli di organismi sensibili(Keeney et al., 2014), un ulteriore meccanismo ecologico di azione sta riducendo la competizione microbica e permettendo l’accumulo di metaboliti primari(ad esempio, acidi biliari primari, zuccheri)(Ng et al., 2013; Theriot et al., 2014), così come gli accettatori di elettroni di origine ospitante come l’ossigeno e il nitrato. Questo concetto integra le recenti scoperte che gli accettatori di elettroni facilitano la colonizzazione di patogeni enterici associati agli antibiotici(Rivera-Chávez et al., 2016; Winter et al., 2013; 2010). Tali aumenti nella disponibilità degli accettatori di elettroni non sono probabilmente unici per il trattamento antibiotico e potrebbero generalizzarsi a vari disturbi enterici. Infatti, gli animali privi di germi(Phillips et al., 1958; Celesk et al., 1976) e gli esseri umani che soffrono di malattie infiammatorie(Circu and Aw, 2011) e malnutrizione(Million et al., 2016) mostrano un aumento del potenziale redox intestinale. Pertanto, i nuovi trattamenti per i disturbi microbici o per prevenire le infezioni associate agli antibiotici possono includere alterazioni chimiche del potenziale redox o l’introduzione di concorrenti per gli accettatori di elettroni in eccesso. Più in generale, proponiamo di aggiungere il potenziale redox all’elenco delle condizioni abiotiche frequentemente saggiate e manipolate per migliorare il benessere dell’ospite.
Materiali e metodi
Esperimenti sugli animali
Topi
Tutti gli esperimenti sugli animali sono stati condotti in conformità con il National Institute of Health Guide for the Care and Use of Laboratory Animals utilizzando protocolli approvati dal Duke University Institutional Animal Care and Use Committee. Per tutti gli esperimenti sono stati utilizzati topi maschi C57BL/6 (Charles River Laboratories, Wilmington MA) di 8-10 settimane di età con un microbiota nativo. I topi sono stati tenuti in una struttura convenzionale per animali da laboratorio presso la Duke University. Misure di base e campioni fecali sono stati raccolti almeno ventiquattro ore prima della prima dose e poi i topi sono stati collocati in alloggiamenti individuali con arricchimento supplementare.
Trattamento antibiotico
I topi sono stati gavattati per via orale con 0,25 ml di acqua deionizzata in autoclave (controllo, N = 10) o 0,25 ml di un cocktail antibiotico (trattato, N = 10) al giorno per cinque giorni (Figura 5A). I topi sono stati assegnati in modo casuale ad un gruppo e i ricercatori che raccoglievano i dati sono stati accecati dai raggruppamenti fino a dopo la somministrazione della dose finale. La dimensione del campione è stata scelta a seguito di un’analisi di potenza per consentire β inferiore a 0,1. Il cocktail antibiotico era composto da ampicillina (Gold Biotechnology, St. Louis MO) 1 mg/ml, vancomicina (Alfa Aesar, Ward Hill MA) 5 mg/ml, neomicina (EMD Millipore, Burlington MA) 10 mg/ml, e metronidazolo (Alfa Aesar) 10 mg/ml (dopo Reikvam et al., 2011). Ogni giorno sono stati preparati cocktail di antibiotici freschi. Durante tutto l’esperimento sono stati raccolti campioni fecali appena svuotati e conservati a -80°C per le analisi successive.
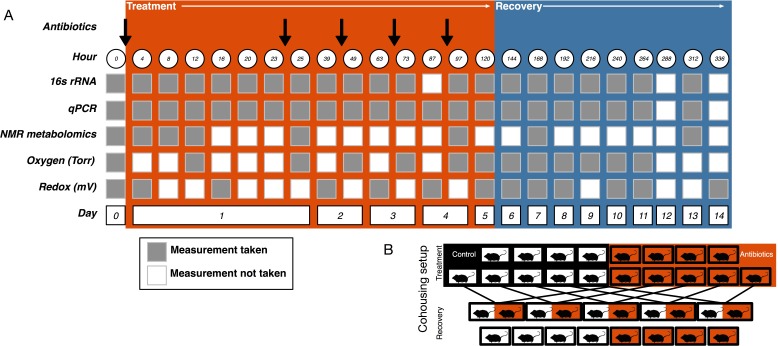
Figura 5.Impostazione sperimentale.(A) Regime di campionamento sperimentale.(B) Impostazione del trattamento di cohousing.

Figura 5.Configurazione sperimentale.(A) Regime di campionamento sperimentale. (B) Impostazione del trattamento di cohousing.
Cohousing di recupero
Un giorno dopo il gavage finale, i topi sono stati assegnati in modo casuale a gruppi di cohousing(Figura 5B). Le gabbie di controllo del cohousing (N = 8, quattro ciascuna da trattati e controllo) contenevano topi singoli che venivano tenuti in alloggiamenti individuali per tutto il periodo di recupero. Le gabbie di trattamento di cohousing (N = 5) contenevano un topo di controllo e un topo trattato con antibiotici della stessa cucciolata che sono stati messi insieme nella gabbia del topo di controllo e tenuti insieme per tutto il periodo di recupero. La dimensione del campione è stata determinata dal numero di topi disponibili alla fine del periodo di trattamento. I topi trattati con antibiotici in gabbie cohoused sono stati contrassegnati da una parziale rasatura per consentire la continuazione del campionamento. Durante il recupero, i campioni fecali appena svuotati sono stati raccolti e conservati a -80°C per le analisi successive. Mentre l’esperimento di trattamento antibiotico è stato eseguito più volte, il cohousing durante l’esperimento di recupero è stato eseguito solo con una singola coorte replicata.
Misurazioni del potenziale Redox
Abbiamo misurato il potenziale redox in pellet fecali freschi utilizzando un elettrodo redox con un diametro della punta di 500 µm in tandem con un elettrodo di riferimento Ag/AgCl (Unisense, Aarhus Denmark). I valori sono indicati in relazione all’elettrodo di idrogeno standard (SHE) e sono stati determinati misurando l’offset dell’elettrodo di riferimento in soluzioni tampone di quinidrone saturo (pH 4 e pH 7) con potenziali redox noti. Le calibrazioni giornaliere sono state effettuate a temperatura ambiente; tuttavia, a causa della potenziale variazione non misurata delle procedure di calibrazione(ad esempio, la fluttuazione della temperatura ambiente), abbiamo concentrato le nostre analisi sulle differenze tra gli animali di controllo e gli animali trattati in un dato momento. L’elettrodo ha un limite di rilevazione di 0,10 mV(Pang e Zhang, 1998).
I pellet fecali sono stati posti su una piastra di agar, appoggiati contro l’elettrodo di riferimento. L’elettrodo è stato inserito nel pellet con un micromanipolatore e i dati sono stati raccolti continuamente per tre minuti. I dati presentati sono la media su tre minuti. I dati Redox sono stati raccolti su due coorti di topi con risultati comparabili. I dati replicati sono mostrati nella Figura 1-figure supplement 2.
Misurazioni dell’ossigeno
Abbiamo misurato la concentrazione di ossigeno in vivo utilizzando un microsensore di ossigeno idrogel accoppiato ad un lettore ottico. I microsensori sono composti da un idrogel biocompatibile, poli (2-idrossietilmetacrilato di metile) (pHEMA) e una molecola di palladio-benzoporfirina (Pd-MABP) sensibile all’ossigeno nel vicino infrarosso (NIR)(Montero-Baker et al., 2015; Wisniewski et al., 2017; Chien et al.,2017). Il microsensore misura l’ossigeno sulla base del principio di estinzione della fosforescenza delle metalloporfirine, una tecnica consolidata con eccellente sensibilità e specificità all’ossigeno fisiologico(Wilson et al., 2006; Rumsey et al., 1988; Lo et al., 1996; Vinogradov et al., 2003). L’idrogel pHEMA è biocompatibile, ha una buona permeabilità all’ossigeno, eccellenti proprietà meccaniche e una lunga storia di utilizzo in dispositivi medici(Montheard et al., 1992). Le molecole di Pd-MABP sono attaccate in modo covalente all’idrogel pHEMA, assicurando che la chimica di rilevamento sia mantenuta nella struttura dell’idrogel. I sensori miniaturizzati (0,75 x 0,75 x 2,5 mm) sono morbidi e simili ai tessuti per ridurre al minimo lo stress all’interfaccia materiale-tessuto.
Il lettore ottico non invasivo è stato posizionato manualmente sopra il sensore e una luce di illuminazione a LED pulsata nella pelle sopra il sensore. Un fotorilevatore ha raccolto la luce di emissione emessa dal sensore. La durata di vita fosforescente, una proprietà del colorante sensibile all’ossigeno, è stata misurata, fornendo così un segnale non influenzato dalle permutazioni del percorso ottico come la profondità del sensore, le fluttuazioni del flusso sanguigno, il rapporto ossi/deossiemoglobina, il contenuto di melanina e l’idratazione(Montheard et al., 1992). L’attuale sistema di rilevamento dell’ossigeno misura la durata di vita entro 2 µs o superiore, che equivale a ~1 µM O2 in condizioni fisiologiche di ossigeno. La temperatura è stata ipotizzata essere a 37°C per convertire la misurazione della durata di vita in concentrazione di ossigeno.
Il sensore di ossigeno è stato posizionato tramite inserimento rettale nell’intestino crasso distale (~ 1 cm dall’ano) di topi sotto anestesia isofluoranica. Il segnale fosforescente è stato raccolto per due minuti con il lettore ottico e tutti i valori sono stati mediati prima della conversione alla concentrazione di ossigeno. Al risveglio dall’anestesia, il sensore è stato passato naturalmente dal mouse attraverso la peristalsi. I dati di ossigeno sono stati raccolti su due coorti di topi con risultati comparabili. I dati replicati sono mostrati nella figura 3-figure supplement 3.
Misurazioni dei nitrati
Abbiamo misurato il nitrato in campioni fecali di topo congelati utilizzando il NitraVer X Nitrogen-Nitrate Reaction Set (Hach Company, Loveland CO) seguendo le istruzioni del produttore. I dati al di sotto del limite inferiore di rilevazione di 0,00036 mg non sono stati inclusi nei lotti o nei test statistici. Queste misurazioni sono state raccolte solo per una singola coorte di topi a causa della disponibilità di campioni.
Misurazioni del fumarato
Abbiamo misurato il fumarato in campioni fecali di topo congelati utilizzando il Fumarate Colorimetric Assay Kit (Biovision, Milpitas CA) seguendo le istruzioni del produttore. I punti di dati al di sotto del limite di rilevazione inferiore di 200 nmol non sono stati mostrati sulle trame, ma sono stati inclusi nelle analisi statistiche post-hoc come pseudo-conteggio di 200 nmol quando tutte le misure per un gruppo di trattamento erano al di sotto del limite di rilevazione. Queste misurazioni sono state raccolte solo per una singola coorte di topi a causa della disponibilità di campioni.
Isolamento del DNA da campioni fecali di topo
Il DNA metagenomico è stato isolato da campioni fecali congelati utilizzando il kit di estrazione del DNA PowerSoil MoBio (ora Qiagen, Hilden Germany) seguendo le istruzioni del produttore (David et al., 2014; 2015 ).
Sequenziamento ed elaborazione del gene 16S rRNA
Abbiamo eseguito il sequenziamento dell’amplicon del gene 16S rRNA utilizzando primer personalizzati con codice a barre per la regione V4 del gene(Caporaso et al., 2011) e protocolli pubblicati(Caporaso et al., 2011; 2012; Maurice et al., 2013). Il sequenziamento è stato condotto su un MiSeq Illumina (San Diego, CA) con l’estremità accoppiata 250 bp letto utilizzando il kit V3. Tutti i campioni con meno di 5000 letture sono stati scartati. I dati di sequenziamento sono stati elaborati utilizzando QIIME (versione 1.8) (Caporasoet al., 2010) per produrre una tabella OTU con il 97% di somiglianza dei cluster. Il sequenziamento è stato eseguito su campioni provenienti da due coorti di topi con risultati comparabili, ma tutti i risultati presentati qui sono da una singola coorte che corrisponde ai dati abiotici presentati.
Poiché il microbioma di base dovrebbe differire tra le cucciolate di topo nel tempo così come tra i fornitori(Rosshart et al., 2017; Rausch et al., 2016; Ivanov et al., 2009; Campbell et al., 2012), abbiamo esaminato se gli effetti osservati erano riproducibili tra le due coorti sequenziate. Un’analisi complessiva della biodiversità beta ha mostrato che ci sono state differenze significative tra le coorti nel loro microbioma a livello OTU durante l’esperimento (p<0,01, R2 = 0,17, PERMANOVA; Figura 4-figure supplement 5). Questo effetto coorte non ha interagito in modo significativo con l’effetto del trattamento, tuttavia (p = 0,3). Poiché la risposta della fioritura delle Enterobacteriaceae agli antibiotici è stata trovata coerente tra gli esseri umani e i topi, così come tra i fornitori di topi(Antonopoulos et al., 2009; David et al., 2015; Young e Schmidt, 2004; Theriot et al., 2014; Peterfreund et al., 2012; Dethlefsen et al., 2008; Jakobsson et al., 2010; Looft e Allen, 2012), riteniamo che le tendenze generali qui osservate siano indicative di ciò che può accadere in altri contesti convenzionali di microbiota intestinale.
Clustering
I generi batterici sono stati raggruppati utilizzando una pipeline bioinformatica precedentemente descritta per identificare i taxa con dinamiche simili(David et al., 2014; 2015). Anche i generi rari (definiti come quelli osservati in cinque o meno campioni) sono stati esclusi dal clustering. I generi rari comprendono 194 dei 306 generi totali osservati, ma rappresentano solo lo 0,019% delle letture. Tutti i generi rimanenti(n= 112) sono stati infine assegnati a un cluster. Una soglia di clustering di 0,9 è stata scelta manualmente per bilanciare la semplicità del modello(cioè, costruire cluster con un numero interpretabile di generi) e la fedeltà(cioè, catturare le dinamiche uniche di generi distinti). Solo i cluster con un’abbondanza media di >1% (cinque cluster in totale) sono stati inclusi nelle analisi statistiche.
qPCR: Primer e condizioni
Per stimare l’abbondanza batterica totale, la PCR è stata eseguita su DNA estratto dalle feci utilizzando i seguenti primer: avanti, 5′-ACTCCTACGGGGGAGGCAGCAGCAGT-3′, indietro, 5′-GTATTACCGGCGGGCTGGGCTGGCCAC-3′ (Bergström et al..,2012). i saggi qPCR sono stati eseguiti utilizzando SYBR FAST qPCR Master Mix (KAPA, Wilmington MA) su un sistema PCR Fast Real-Time PCR 7900HT (Applied Biosystems, Foster City CA). I valori Ct sono stati standardizzati rispetto ad una curva di diluizione di concentrazione nota e poi regolati per il peso della materia fecale estratta. Le misurazioni del carico sono state eseguite su campioni provenienti da due coorti di topi con risultati comparabili, ma tutti i dati qui inclusi provengono da una singola coorte.
Saggi di infiammazione
La lipocalina fecale 2 (Lcn-2) è stata utilizzata come biomarcatore non invasivo per l’infiammazione intestinale dell’ospite. Lcn-2 è stato quantificato dall’ELISA dopo Chassaing et al.(Chassaing et al., 2012). I saggi sono stati condotti con il kit ELISA del duo lipocalina-2/ngal di topo del kit R e D Systems (Minneapolis, MN) utilizzando una rondella per piastre BioTek ELx405rs e un lettore di piastre BioTek Synergy HT (Bio-Rad, Hercules CA) presso il Centro UNC Center for Gastrointestinal Biology and Disease Advanced Analytics Core. Queste misurazioni sono state raccolte solo per una singola coorte di topi a causa della disponibilità di campioni.
Isolamento dell’RNA e RT-PCR
L’RNA totale è stato isolato dai pellet fecali conservati in RNALater (Thermo Fisher, Waltham, MA) utilizzando il kit di isolamento dell’RNA del MoBio PowerMicrobiome con una fase di estrazione del cloroformio fenolico aggiunta. ≤15 ng di RNA sono stati trascritti al contrario utilizzando cDNA Prep Reverse Transcription Master Mix (Fluidigm, South San Francisco, CA) seguendo le istruzioni del produttore. Trascrizioni di destinazione sono stati preamplificati per 18 cicli e poi diluito 10x. RT-PCR è stata eseguita utilizzando un BioMark (Fluidigm) su un chip 48 × 48 con Taqman Fast Advanced Master Mix (Thermo Fisher). Tre ERCC RNA Spike-in Mix (Thermo Fisher) controlli positivi e un controllo negativo non target di acqua priva di nucleasi sono stati anche eseguiti sul chip. I livelli di espressione sono stati quantificati per trenta geni(file supplementare 3). Di questi, sei geni potevano essere quantificati per almeno 20 punti di tempo, e quindi solo questi geni sono stati inclusi nelle analisi statistiche. I livelli di espressione sono stati normalizzati all’espressione Actb del mouse (ΔCt) per ogni punto temporale per ogni mouse e poi confrontati con la media dei mouse di controllo per quel punto temporale (ΔΔCt). Viene rappresentato il cambiamento di piega (2^ΔΔΔCt). Queste misurazioni sono state raccolte solo per una singola coorte di topi a causa della disponibilità di campioni.
Metabolomica NMR
I campioni sono stati preparati mescolando pellet fecali con 200 μl di tampone fosfato salino (PBS) preparato al 95% D2O, pH 7,5 e omogeneizzante per 5 minuti a 30 Hz. I detriti delle cellule sono stati rimossi centrifugando i campioni per 10 minuti a 14K x g a 4°C. Il supernatante è stato filtrato attraverso un filtro centrifugo di 0,22 μm. Per massimizzare l’estrazione del metabolita, ogni pellet dalla prima centrifugazione è stato miscelato con 200 μl PBS in 95% D2O,pH 7,5 e omogeneizzato per 5 minuti a 30 Hz. Le soluzioni sono state filtrate attraverso un filtro centrifugo di 0,22 μm come prima. I supernatanti sono stati combinati e 300 microlitri è stato preso e mescolato con 300 microlitri di 99,9% D2O contenente 500 μM 3-(trimetilsilililil)-1-propanesulfonico acido-d6, sale sodico (DSS-d6).
Gli spettri NMR sono stati raccolti su uno spettrometro NMR Avance III 700MHz (Bruker Biospin, Billerica, MA, USA) con una sonda a tripla risonanza TXI funzionante a 25C. È stata usata una sequenza di impulsi 1D NOESY con un’ampiezza spettrale di 12 ppm. Ogni spettro è stato digitalizzato con 32768 punti su un tempo di acquisizione totale di 3,9 s. La sequenza ha utilizzato un tempo di miscelazione di 100 ms e un ritardo di rilassamento interscansione di 2,0 s. Queste misure sono state raccolte solo per una singola coorte di topi a causa della disponibilità di campioni.
Gli spettri sono stati elaborati utilizzando l’Advanced Chemistry Development Spectrus Processor (versione 2016.1, Toronto, ON, Canada). I parametri di elaborazione comprendevano il riempimento a zero fino a 65526 punti, la moltiplicazione esponenziale a 0,3 Hz in decadimento. La correzione di fase è stata applicata utilizzando gli algoritmi automatizzati del software ACD e una correzione della linea di base polinomiale di terzo ordine è stata applicata a tutti i campioni. La profilazione quantitativa dei metaboliti è stata effettuata con la Chenomx NMR Suite (versione 8.2, Edmonton, AL, Canada). Gli spostamenti chimici e le concentrazioni sono stati referenziati utilizzando DSS-d6 aggiunto ad ogni campione ad una concentrazione di 250 μM e impostato su uno spostamento chimico di 0,00 ppm. La libreria Chenomx 700MHz comprendeva acetato, acetoacetato, acetoino, alanina, asparagina, aspartato, B-idrossibutirrato, butirrato, DSS-d6, etanolo, formiato, glucosio, glutammina, glutammato, glicina, isoleucina, isopropanolo, lattato, leucina, lisina, metionina, propionato, sarcosina, treonina e valina. I segnali nelle regioni degli acidi biliari dello spettro, da circa 0,6 a 0,9 ppm, erano adatti come funzioni sintetiche Lorentizan e ad ogni segnale è stata data un’assegnazione generica come gruppo metilico degli acidi biliari. La concentrazione per gli acidi biliari è presentata come unità di concentrazione arbitraria e si dovrebbero fare dei confronti relativi. L’adattamento di ogni spettro è stato effettuato in modalità batch. La regolazione manuale dell’adattamento è stata effettuata in alcuni campioni per correggere gli errori derivanti dalla sovrapposizione spettrale.
Esperimenti ex vivo
Condizioni culturali e trattamento
I microbioti intestinali sono stati coltivati da feci umane in un sistema di bioreattore a flusso continuo (Infors, Bottmingen Svizzera) in seguito a lavori precedenti(McDonald et al., 2013). Soggetti umani sani (n = 3) che si sono autodenunciati senza uso di antibiotici entro un mese dall’iscrizione hanno fornito una singola donazione di feci. La dimensione del campione è stata calcolata utilizzando un’analisi di potenza per consentire un β inferiore a 0,1. Questo esperimento è stato eseguito solo con una singola coorte di donatori. Il consenso informato è stato ottenuto da tutti i soggetti e il protocollo è stato approvato dalla Duke Health Institutional Review Board. I soggetti hanno raccolto i campioni collocando i contenitori monouso per i campioni da toilette (Fisher Scientific, Hampton NH) sotto i sedili della toilette prima dei movimenti intestinali. I campioni di feci intatte sono stati spostati entro circa 15 minuti in condizioni anaerobiche dopo il prelievo. Tutti i contenitori del bioreattore avevano un volume totale di 400 ml e un turnover continuo di 400 ml al giorno per i media per emulare il tempo di passaggio medio di 24 ore dell’intestino umano. Le comunità di bioreattori sono state sottoposte ad un periodo di adattamento di due giorni prima dell’inizio del trattamento(Figura 2-figure supplement 1). Le navi sono state poi assegnate in modo casuale una condizione di trattamento. Le colture trattate con antibiotici sono state sottoposte a dosi singole giornaliere di 3,896 ml di trattamenti antibiotici in due giorni consecutivi. Gli antibiotici sono stati preparati come sopra per i topi. I vasi di controllo e gli antibiotici avevano una bolla di gas N2 da 1 L al minuto. Redox è stato misurato in continuo con le sonde Hamilton (Reno NV) EasyFerm Plus ORP K8 225, che ha un campo di misura di ± 2000 mV. Le sonde Redox sono state calibrate nel buffer ORP di Hamilton prima di sterilizzare in autoclave la configurazione del bioreattore all’inizio di ogni corsa di replicazione. A causa delle differenze nella costruzione e nella calibrazione tra le sonde redox in vivo e ex vivo, non si dovrebbero effettuare confronti diretti tra i valori redox in vivo e ex vivo. I campioni sono stati raccolti giornalmente e congelati immediatamente a -80°C per la successiva analisi della composizione della comunità microbica. I dati abiotici sono stati mediati per incrementi di cinque minuti per l’analisi.
Analisi statistiche
Abbiamo eseguito l’analisi lineare del modello di effetti misti per determinare gli effetti degli antibiotici sul potenziale redox, la concentrazione di ossigeno, la concentrazione di lipocalina 2 fecale e la dissimilarità di Bray-Curtis. Come effetti fissi, abbiamo inserito nel modello il trattamento antibiotico e il tempo con un termine di interazione. Abbiamo incluso l’identità del topo come effetto casuale. I valori di P sono stati ottenuti con test di likelihood ratio confrontando il modello completo con un modello che includeva solo il tempo e l’identità del topo e sono stati eseguiti con la funzione ‘anova’ nel pacchetto ‘lme4’. Sono state utilizzate correlazioni di misure ripetute per valutare le correlazioni in cui più punti temporali dello stesso mouse sono stati inclusi nell’analisi statistica. Le correlazioni di misure ripetute sono state calcolate con la funzione ‘rmcorr’ nel pacchetto ‘rmcorr’. Queste e tutte le altre analisi statistiche sono state effettuate in R (R core team, versione 3.3). Tutti i test statistici eseguiti erano non parametrici, tranne quando un test di Shapiro-Wilks indicava che i dati erano normalmente distribuiti, nel qual caso sono stati utilizzati test parametrici. Tutti i punti di dati sono stati inclusi nelle analisi e gli outlier non sono stati trattati in alcun modo.
References
- Albenberg L, Esipova TV, Judge CP, Bittinger K, Chen J, Laughlin A, Grunberg S, Baldassano RN, Lewis JD, Li H, Thom SR, Bushman FD, Vinogradov SA, Wu GD. Correlation between intraluminal oxygen gradient and radial partitioning of intestinal Microbiota. Gastroenterology. 2014; 147:1055-1063. DOI | PubMed
- Allison SD, Martiny JB. Colloquium paper: resistance, resilience, and redundancy in microbial communities. PNAS. 2008; 105 Suppl 1:11512-11519. DOI | PubMed
- Antonopoulos DA, Huse SM, Morrison HG, Schmidt TM, Sogin ML, Young VB. Reproducible community dynamics of the gastrointestinal Microbiota following antibiotic perturbation. Infection and Immunity. 2009; 77:2367-2375. DOI | PubMed
- Armstrong GL, Conn LA, Pinner RW. Trends in infectious disease mortality in the United States during the 20th century. Jama. 1999; 281:61-66. DOI | PubMed
- Arumugam M, Raes J, Pelletier E, Le Paslier D, Yamada T, Mende DR, Fernandes GR, Tap J, Bruls T, Batto JM, Bertalan M, Borruel N, Casellas F, Fernandez L, Gautier L, Hansen T, Hattori M, Hayashi T, Kleerebezem M, Kurokawa K, Leclerc M, Levenez F, Manichanh C, Nielsen HB, Nielsen T, Pons N, Poulain J, Qin J, Sicheritz-Ponten T, Tims S, Torrents D, Ugarte E, Zoetendal EG, Wang J, Guarner F, Pedersen O, de Vos WM, Brunak S, Doré J, Antolín M, Artiguenave F, Blottiere HM, Almeida M, Brechot C, Cara C, Chervaux C, Cultrone A, Delorme C, Denariaz G, Dervyn R, Foerstner KU, Friss C, van de Guchte M, Guedon E, Haimet F, Huber W, van Hylckama-Vlieg J, Jamet A, Juste C, Kaci G, Knol J, Lakhdari O, Layec S, Le Roux K, Maguin E, Mérieux A, Melo Minardi R, M’rini C, Muller J, Oozeer R, Parkhill J, Renault P, Rescigno M, Sanchez N, Sunagawa S, Torrejon A, Turner K, Vandemeulebrouck G, Varela E, Winogradsky Y, Zeller G, Weissenbach J, Ehrlich SD, Bork P, MetaHIT Consortium. Enterotypes of the human gut microbiome. Nature. 2011; 473:174-180. DOI | PubMed
- Bergström A, Licht TR, Wilcks A, Andersen JB, Schmidt LR, Grønlund HA, Vigsnaes LK, Michaelsen KF, Bahl MI. Introducing GUt low-density array (GULDA): a validated approach for qPCR-based intestinal microbial community analysis. FEMS Microbiology Letters. 2012; 337:38-47. DOI | PubMed
- Broedl UC, Schachinger V, Lingenhel A, Lehrke M, Stark R, Seibold F, Göke B, Kronenberg F, Parhofer KG, Konrad-Zerna A. Apolipoprotein A-IV is an independent predictor of disease activity in patients with inflammatory bowel disease. Inflammatory Bowel Diseases. 2007; 13:391-397. DOI | PubMed
- Buffie CG, Jarchum I, Equinda M, Lipuma L, Gobourne A, Viale A, Ubeda C, Xavier J, Pamer EG. Profound alterations of intestinal microbiota following a single dose of clindamycin results in sustained susceptibility to Clostridium difficile-induced colitis. Infection and Immunity. 2012; 80:62-73. DOI | PubMed
- Byndloss MX, Olsan EE, Rivera-Chávez F, Tiffany CR, Cevallos SA, Lokken KL, Torres TP, Byndloss AJ, Faber F, Gao Y, Litvak Y, Lopez CA, Xu G, Napoli E, Giulivi C, Tsolis RM, Revzin A, Lebrilla CB, Bäumler AJ. Microbiota-activated PPAR-γ signaling inhibits dysbiotic Enterobacteriaceae expansion. Science. 2017; 357:570-575. DOI | PubMed
- Campbell JH, Foster CM, Vishnivetskaya T, Campbell AG, Yang ZK, Wymore A, Palumbo AV, Chesler EJ, Podar M. Host genetic and environmental effects on mouse intestinal microbiota. The ISME Journal. 2012; 6:2033-2044. DOI | PubMed
- Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods. 2010; 7:335-336. DOI | PubMed
- Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Huntley J, Fierer N, Owens SM, Betley J, Fraser L, Bauer M, Gormley N, Gilbert JA, Smith G, Knight R. Ultra-high-throughput microbial community analysis on the illumina HiSeq and MiSeq platforms. The ISME Journal. 2012; 6:1621-1624. DOI | PubMed
- Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Lozupone CA, Turnbaugh PJ, Fierer N, Knight R. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. PNAS. 2011; 108:4516-4522. DOI | PubMed
- Cartwright T, Perkins ND, L Wilson C. NFKB1: a suppressor of inflammation, ageing and cancer. The FEBS Journal. 2016; 283:1812-1822. DOI | PubMed
- Celesk RA, Asano T, Wagner M. The size, pH, and redox potential of the cecum in mice associated with various microbial floras. Experimental Biology and Medicine. 1976; 151:260-263. DOI
- Chassaing B, Srinivasan G, Delgado MA, Young AN, Gewirtz AT, Vijay-Kumar M. Fecal lipocalin 2, a sensitive and broadly dynamic non-invasive biomarker for intestinal inflammation. PLoS ONE. 2012; 7DOI | PubMed
- Chen J, Hanke A, Tegetmeyer HE, Kattelmann I, Sharma R, Hamann E, Hargesheimer T, Kraft B, Lenk S, Geelhoed JS, Hettich RL, Strous M. Impacts of chemical gradients on microbial community structure. The ISME Journal. 2017; 11:920-931. DOI | PubMed
- Chien JS, Mohammed M, Eldik H, Ibrahim MM, Martinez J, Nichols SP, Wisniewski N, Klitzman B. Injectable Phosphorescence-based oxygen biosensors identify post ischemic reactive hyperoxia. Scientific Reports. 2017; 7DOI | PubMed
- Cho I, Yamanishi S, Cox L, Methé BA, Zavadil J, Li K, Gao Z, Mahana D, Raju K, Teitler I, Li H, Alekseyenko AV, Blaser MJ. Antibiotics in early life alter the murine colonic microbiome and adiposity. Nature. 2012; 488:621-626. DOI | PubMed
- Circu ML, Aw TY. Redox biology of the intestine. Free Radical Research. 2011; 45:1245-1266. DOI | PubMed
- Claesson MJ, Jeffery IB, Conde S, Power SE, O’Connor EM, Cusack S, Harris HM, Coakley M, Lakshminarayanan B, O’Sullivan O, Fitzgerald GF, Deane J, O’Connor M, Harnedy N, O’Connor K, O’Mahony D, van Sinderen D, Wallace M, Brennan L, Stanton C, Marchesi JR, Fitzgerald AP, Shanahan F, Hill C, Ross RP, O’Toole PW. Gut microbiota composition correlates with diet and health in the elderly. Nature. 2012; 488:178-184. DOI | PubMed
- Connell JH, Slatyer RO. Mechanisms of succession in natural communities and their role in community stability and organization. The American Naturalist. 1977; 111:1119-1144. DOI
- Cowley ES, Kopf SH, LaRiviere A, Ziebis W, Newman DK. Pediatric cystic fibrosis sputum can be chemically dynamic, anoxic, and extremely reduced due to hydrogen sulfide formation. mBio. 2015; 6DOI | PubMed
- David LA, Maurice CF, Carmody RN, Gootenberg DB, Button JE, Wolfe BE, Ling AV, Devlin AS, Varma Y, Fischbach MA, Biddinger SB, Dutton RJ, Turnbaugh PJ. Diet rapidly and reproducibly alters the human gut microbiome. Nature. 2014; 505:559-563. DOI | PubMed
- David LA, Weil A, Ryan ET, Calderwood SB, Harris JB, Chowdhury F, Begum Y, Qadri F, LaRocque RC, Turnbaugh PJ. Gut microbial succession follows acute secretory diarrhea in humans. mBio. 2015; 6DOI | PubMed
- De Filippo C, Cavalieri D, Di Paola M, Ramazzotti M, Poullet JB, Massart S, Collini S, Pieraccini G, Lionetti P. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa. PNAS. 2010; 107:14691-14696. DOI | PubMed
- Dedon PC, Tannenbaum SR. Reactive nitrogen species in the chemical biology of inflammation. Archives of Biochemistry and Biophysics. 2004; 423:12-22. DOI | PubMed
- Dethlefsen L, Huse S, Sogin ML, Relman DA. The pervasive effects of an antibiotic on the human gut microbiota, as revealed by deep 16S rRNA sequencing. PLoS Biology. 2008; 6DOI | PubMed
- Dethlefsen L, Relman DA. Incomplete recovery and individualized responses of the human distal gut microbiota to repeated antibiotic perturbation. PNAS. 2011; 108:4554-4561. DOI | PubMed
- Dhall S, Do D, Garcia M, Wijesinghe DS, Brandon A, Kim J, Sanchez A, Lyubovitsky J, Gallagher S, Nothnagel EA, Chalfant CE, Patel RP, Schiller N, Martins-Green M. A novel model of chronic wounds: importance of redox imbalance and biofilm-forming Bacteria for establishment of chronicity. PLoS ONE. 2014; 9DOI | PubMed
- Donaldson GP, Lee SM, Mazmanian SK. Gut biogeography of the bacterial microbiota. Nature Reviews Microbiology. 2016; 14:20-32. DOI | PubMed
- El Aidy S, Derrien M, Merrifield CA, Levenez F, Doré J, Boekschoten MV, Dekker J, Holmes E, Zoetendal EG, van Baarlen P, Claus SP, Kleerebezem M. Gut bacteria-host metabolic interplay during conventionalisation of the mouse germfree colon. The ISME Journal. 2013; 7:743-755. DOI | PubMed
- Espey MG. Role of oxygen gradients in shaping redox relationships between the human intestine and its microbiota. Free Radical Biology and Medicine. 2013; 55:130-140. DOI | PubMed
- Faber F, Tran L, Byndloss MX, Lopez CA, Velazquez EM, Kerrinnes T, Nuccio SP, Wangdi T, Fiehn O, Tsolis RM, Bäumler AJ. Host-mediated sugar oxidation promotes post-antibiotic pathogen expansion. Nature. 2016; 534:697-699. DOI | PubMed
- Fierer N, Nemergut D, Knight R, Craine JM. Changes through time: integrating microorganisms into the study of succession. Research in Microbiology. 2010; 161:635-642. DOI | PubMed
- Fischbach MA, Sonnenburg JL. Eating for two: how metabolism establishes interspecies interactions in the gut. Cell Host & Microbe. 2011; 10:336-347. DOI | PubMed
- Fischer J, Lindenmayer DB. Landscape modification and habitat fragmentation: a synthesis. Global Ecology and Biogeography. 2007; 16:265-280. DOI
- Friedman N, Shriker E, Gold B, Durman T, Zarecki R, Ruppin E, Mizrahi I. Diet-induced changes of redox potential underlie compositional shifts in the rumen archaeal community. Environmental Microbiology. 2017; 19:174-184. DOI | PubMed
- Gerbersdorf SU, Bittner R, Lubarsky H, Manz W, Paterson DM. Microbial assemblages as ecosystem engineers of sediment stability. Journal of Soils and Sediments. 2009; 9:640-652. DOI
- Gobbetti M. The sourdough microflora: interactions of lactic acid bacteria and yeasts. Trends in Food Science & Technology. 1998; 9:267-274. DOI
- Goddard MR. Quantifying the complexities of Saccharomyces cerevisiae’s ecosystem engineering via fermentation. Ecology. 2008; 89:2077-2082. DOI | PubMed
- Goodrich JK, Waters JL, Poole AC, Sutter JL, Koren O, Blekhman R, Beaumont M, Van Treuren W, Knight R, Bell JT, Spector TD, Clark AG, Ley RE. Human genetics shape the gut microbiome. Cell. 2014; 159:789-799. DOI | PubMed
- Hooper LV, Littman DR, Macpherson AJ. Interactions between the Microbiota and the immune system. Science. 2012; 336:1268-1273. DOI | PubMed
- Hughes ER, Winter MG, Duerkop BA, Spiga L, Furtado de Carvalho T, Zhu W, Gillis CC, Büttner L, Smoot MP, Behrendt CL, Cherry S, Santos RL, Hooper LV, Winter SE. Microbial respiration and formate oxidation as metabolic signatures of inflammation-associated dysbiosis. Cell Host & Microbe. 2017; 21:208-219. DOI | PubMed
- Ivanov II, Atarashi K, Manel N, Brodie EL, Shima T, Karaoz U, Wei D, Goldfarb KC, Santee CA, Lynch SV, Tanoue T, Imaoka A, Itoh K, Takeda K, Umesaki Y, Honda K, Littman DR. Induction of intestinal Th17 cells by segmented filamentous bacteria. Cell. 2009; 139:485-498. DOI | PubMed
- Jakobsson HE, Jernberg C, Andersson AF, Sjölund-Karlsson M, Jansson JK, Engstrand L. Short-term antibiotic treatment has differing long-term impacts on the human throat and gut microbiome. PLoS ONE. 2010; 5DOI | PubMed
- Jones CG, Lawton JH, Shachak M. Organisms as ecosystem engineers. Oikos. 1994; 69:373-386. DOI
- Jones SA, Chowdhury FZ, Fabich AJ, Anderson A, Schreiner DM, House AL, Autieri SM, Leatham MP, Lins JJ, Jorgensen M, Cohen PS, Conway T. Respiration of Escherichia coli in the mouse intestine. Infection and Immunity. 2007; 75:4891-4899. DOI | PubMed
- Jones SA, Gibson T, Maltby RC, Chowdhury FZ, Stewart V, Cohen PS, Conway T. Anaerobic respiration of Escherichia coli in the mouse intestine. Infection and Immunity. 2011; 79:4218-4226. DOI | PubMed
- Keeney KM, Yurist-Doutsch S, Arrieta MC, Finlay BB. Effects of antibiotics on human microbiota and subsequent disease. Annual Review of Microbiology. 2014; 68:217-235. DOI | PubMed
- Kelly CJ, Zheng L, Campbell EL, Saeedi B, Scholz CC, Bayless AJ, Wilson KE, Glover LE, Kominsky DJ, Magnuson A, Weir TL, Ehrentraut SF, Pickel C, Kuhn KA, Lanis JM, Nguyen V, Taylor CT, Colgan SP. Crosstalk between microbiota-derived short-chain fatty acids and intestinal epithelial HIF augments tissue barrier function. Cell Host & Microbe. 2015; 17:662-671. DOI | PubMed
- Kröger A, Geisler V, Lemma E, Theis F, Lenger R. Bacterial fumarate respiration. Archives of Microbiology. 1992; 158:311-314. DOI
- Lo LW, Koch CJ, Wilson DF. Calibration of oxygen-dependent quenching of the phosphorescence of Pd-meso-tetra (4-Carboxyphenyl) porphine: a phosphor with general application for measuring oxygen concentration in biological systems. Analytical Biochemistry. 1996; 236:153-160. DOI | PubMed
- Looft T, Allen HK. Collateral effects of antibiotics on mammalian gut microbiomes. Gut Microbes. 2012; 3:463-467. DOI | PubMed
- Maurice CF, Haiser HJ, Turnbaugh PJ. Xenobiotics shape the physiology and gene expression of the active human gut microbiome. Cell. 2013; 152:39-50. DOI | PubMed
- McDonald JA, Schroeter K, Fuentes S, Heikamp-Dejong I, Khursigara CM, de Vos WM, Allen-Vercoe E. Evaluation of microbial community reproducibility, stability and composition in a human distal gut chemostat model. Journal of Microbiological Methods. 2013; 95:167-174. DOI | PubMed
- Million M, Tidjani Alou M, Khelaifia S, Bachar D, Lagier JC, Dione N, Brah S, Hugon P, Lombard V, Armougom F, Fromonot J, Robert C, Michelle C, Diallo A, Fabre A, Guieu R, Sokhna C, Henrissat B, Parola P, Raoult D. Increased gut redox and depletion of anaerobic and methanogenic prokaryotes in severe acute malnutrition. Scientific Reports. 2016; 6DOI | PubMed
- Montero-Baker MF, Au-Yeung KY, Wisniewski NA, Gamsey S, Morelli-Alvarez L, Mills JL, Campos M, Helton KL. The First-in-Man "Si Se Puede" Study for the use of micro-oxygen sensors (MOXYs) to determine dynamic relative oxygen indices in the feet of patients with limb-threatening ischemia during endovascular therapy. Journal of Vascular Surgery. 2015; 61:1501-1510. DOI | PubMed
- Montheard J-P, Chatzopoulos M, Chappard D. 2-Hydroxyethyl methacrylate (HEMA): chemical properties and applications in biomedical fields. Journal of Macromolecular Science, Part C: Polymer Reviews. 1992; 32:1-34. DOI
- Morris RL, Schmidt TM. Shallow breathing: bacterial life at low O(2). Nature Reviews Microbiology. 2013; 11:205-212. DOI | PubMed
- Ng KM, Ferreyra JA, Higginbottom SK, Lynch JB, Kashyap PC, Gopinath S, Naidu N, Choudhury B, Weimer BC, Monack DM, Sonnenburg JL. Microbiota-liberated host sugars facilitate post-antibiotic expansion of enteric pathogens. Nature. 2013; 502:96-99. DOI | PubMed
- Noll M, Matthies D, Frenzel P, Derakshani M, Liesack W. Succession of bacterial community structure and diversity in a paddy soil oxygen gradient. Environmental Microbiology. 2005; 7:382-395. DOI | PubMed
- Orcutt BN, Sylvan JB, Knab NJ, Edwards KJ. Microbial ecology of the dark ocean above, at, and below the seafloor. Microbiology and Molecular Biology Reviews. 2011; 75:361-422. DOI | PubMed
- Osono T. Colonization and succession of fungi during decomposition of Swida controversa leaf litter. Mycologia. 2005; 97:589-597. DOI | PubMed
- Pang H, Zhang TC. Fabrication of redox potential microelectrodes for studies in vegetated soils or biofilm systems. Environmental Science & Technology. 1998; 32:3646-3652. DOI
- Peet RK, Christensen NL. Succession: a population process. Vegetatio. 1980; 43:131-140. DOI
- Pereira FC, Berry D. Microbial nutrient niches in the gut. Environmental Microbiology. 2017; 19:1366-1378. DOI | PubMed
- Peterfreund GL, Vandivier LE, Sinha R, Marozsan AJ, Olson WC, Zhu J, Bushman FD. Succession in the gut microbiome following antibiotic and antibody therapies for Clostridium difficile. PLoS ONE. 2012; 7DOI | PubMed
- Phillips BP, Wolfe PA, Bartgis IL. Studies on the ameba-bacteria relationship in Amebiasis. II. some concepts on the etiology of the disease. The American Journal of Tropical Medicine and Hygiene. 1958; 7:392-399. DOI | PubMed
- Rausch P, Basic M, Batra A, Bischoff SC, Blaut M, Clavel T, Gläsner J, Gopalakrishnan S, Grassl GA, Günther C, Haller D, Hirose M, Ibrahim S, Loh G, Mattner J, Nagel S, Pabst O, Schmidt F, Siegmund B, Strowig T, Volynets V, Wirtz S, Zeissig S, Zeissig Y, Bleich A, Baines JF. Analysis of factors contributing to variation in the C57BL/6J fecal microbiota across german animal facilities. International Journal of Medical Microbiology. 2016; 306:343-355. DOI | PubMed
- Ravcheev DA, Thiele I. Systematic genomic analysis reveals the complementary aerobic and anaerobic respiration capacities of the human gut microbiota. Frontiers in Microbiology. 2014; 5DOI | PubMed
- Reikvam DH, Erofeev A, Sandvik A, Grcic V, Jahnsen FL, Gaustad P, McCoy KD, Macpherson AJ, Meza-Zepeda LA, Johansen FE. Depletion of murine intestinal microbiota: effects on gut mucosa and epithelial gene expression. PLoS ONE. 2011; 6DOI | PubMed
- Rivera-Chávez F, Zhang LF, Faber F, Lopez CA, Byndloss MX, Olsan EE, Xu G, Velazquez EM, Lebrilla CB, Winter SE, Bäumler AJ. Depletion of Butyrate-Producing clostridia from the gut Microbiota drives an aerobic luminal expansion of salmonella. Cell Host & Microbe. 2016; 19:443-454. DOI | PubMed
- Rosshart SP, Vassallo BG, Angeletti D, Hutchinson DS, Morgan AP, Takeda K, Hickman HD, McCulloch JA, Badger JH, Ajami NJ, Trinchieri G, Pardo-Manuel de Villena F, Yewdell JW, Rehermann B. Wild mouse gut microbiota promotes host fitness and improves disease resistance. Cell. 2017; 171:1015-1028. DOI | PubMed
- Rui J, Peng J, Lu Y. Succession of bacterial populations during plant residue decomposition in rice field soil. Applied and Environmental Microbiology. 2009; 75:4879-4886. DOI | PubMed
- Rumsey WL, Vanderkooi JM, Wilson DF. Imaging of phosphorescence: a novel method for measuring oxygen distribution in perfused tissue. Science. 1988; 241:1649-1651. DOI | PubMed
- Russell SL, Gold MJ, Hartmann M, Willing BP, Thorson L, Wlodarska M, Gill N, Blanchet MR, Mohn WW, McNagny KM, Finlay BB. Early life antibiotic-driven changes in microbiota enhance susceptibility to allergic asthma. EMBO Reports. 2012; 13:440-447. DOI | PubMed
- Shade A, Peter H, Allison SD, Baho DL, Berga M, Bürgmann H, Huber DH, Langenheder S, Lennon JT, Martiny JB, Matulich KL, Schmidt TM, Handelsman J. Fundamentals of microbial community resistance and resilience. Frontiers in Microbiology. 2012; 3DOI | PubMed
- Shatalin K, Shatalina E, Mironov A, Nudler E. H2S: a universal defense against antibiotics in bacteria. Science. 2011; 334:986-990. DOI | PubMed
- Shi W, Norton JM. Microbial control of nitrate concentrations in an agricultural soil treated with dairy waste compost or ammonium fertilizer. Soil Biology and Biochemistry. 2000; 32:1453-1457. DOI
- Spees AM, Wangdi T, Lopez CA, Kingsbury DD, Xavier MN, Winter SE, Tsolis RM, Bäumler AJ. Streptomycin-induced inflammation enhances Escherichia coli gut colonization through nitrate respiration. mBio. 2013; 4DOI | PubMed
- Stecher B, Robbiani R, Walker AW, Westendorf AM, Barthel M, Kremer M, Chaffron S, Macpherson AJ, Buer J, Parkhill J, Dougan G, von Mering C, Hardt W-D. Salmonella enterica serovar typhimurium exploits inflammation to compete with the intestinal Microbiota. PLoS Biology. 2007; 5DOI
- Stefka AT, Feehley T, Tripathi P, Qiu J, McCoy K, Mazmanian SK, Tjota MY, Seo GY, Cao S, Theriault BR, Antonopoulos DA, Zhou L, Chang EB, Fu YX, Nagler CR. Commensal bacteria protect against food allergen sensitization. PNAS. 2014; 111:13145-13150. DOI | PubMed
- Theriot CM, Koenigsknecht MJ, Carlson PE, Hatton GE, Nelson AM, Li B, Huffnagle GB, Z Li J, Young VB. Antibiotic-induced shifts in the mouse gut microbiome and metabolome increase susceptibility to Clostridium difficile infection. Nature Communications. 2014; 5DOI | PubMed
- Tilman D. The resource-ratio hypothesis of plant succession. The American Naturalist. 1985; 125:827-852. DOI
- Venkataraman A, Sieber JR, Schmidt AW, Waldron C, Theis KR, Schmidt TM. Variable responses of human microbiomes to dietary supplementation with resistant starch. Microbiome. 2016; 4DOI | PubMed
- Vinogradov SA, Grosul P, Rozhkov V, Dunphy I, Shuman L, Dugan BW, Evans S, Wilson DF. Oxygen distributions in tissue measured by phosphorescence quenching. Advances in Experimental Medicine and Biology. 2003; 510:181-185. DOI | PubMed
- Waddell A, Ahrens R, Tsai YT, Sherrill JD, Denson LA, Steinbrecher KA, Hogan SP. Intestinal CCL11 and eosinophilic inflammation is regulated by myeloid cell-specific RelA/p65 in mice. The Journal of Immunology. 2013; 190:4773-4785. DOI | PubMed
- West PW, Ramachandran TP. Spectrophotometric determination of nitrate using chromotropic acid. Analytica Chimica Acta. 1966; 35:317-324. DOI
- Wilson DF, Vinogradov SA, Grosul P, Sund N, Vacarezza MN, Bennett J. Imaging oxygen pressure in the rodent retina by phosphorescence lifetime. Advances in Experimental Medicine and Biology. 2006; 578:119-124. DOI | PubMed
- Winter SE, Thiennimitr P, Winter MG, Butler BP, Huseby DL, Crawford RW, Russell JM, Bevins CL, Adams LG, Tsolis RM, Roth JR, Bäumler AJ. Gut inflammation provides a respiratory electron acceptor for Salmonella. Nature. 2010; 467:426-429. DOI | PubMed
- Winter SE, Winter MG, Xavier MN, Thiennimitr P, Poon V, Keestra AM, Laughlin RC, Gomez G, Wu J, Lawhon SD, Popova IE, Parikh SJ, Adams LG, Tsolis RM, Stewart VJ, Bäumler AJ. Host-derived nitrate boosts growth of E. coli in the inflamed gut. Science. 2013; 339:708-711. DOI | PubMed
- Winterbourn CC. Reconciling the chemistry and biology of reactive oxygen species. Nature Chemical Biology. 2008; 4:278-286. DOI | PubMed
- Wisniewski NA, Nichols SP, Gamsey SJ, Pullins S, Au-Yeung KY, Klitzman B, Helton KL. Advances in Experimental Medicine and Biology. Springer International Publishing; 2017.
- Wiström J, Norrby SR, Myhre EB, Eriksson S, Granström G, Lagergren L, Englund G, Nord CE, Svenungsson B. Frequency of antibiotic-associated diarrhoea in 2462 antibiotic-treated hospitalized patients: a prospective study. Journal of Antimicrobial Chemotherapy. 2001; 47:43-50. DOI | PubMed
- Wright JP, Jones CG. The concept of organisms as ecosystem engineers ten years on: progress, limitations, and challenges. BioScience. 2006; 56:203-209. DOI
- Yatsunenko T, Rey FE, Manary MJ, Trehan I, Dominguez-Bello MG, Contreras M, Magris M, Hidalgo G, Baldassano RN, Anokhin AP, Heath AC, Warner B, Reeder J, Kuczynski J, Caporaso JG, Lozupone CA, Lauber C, Clemente JC, Knights D, Knight R, Gordon JI. Human gut microbiome viewed across age and geography. Nature. 2012; 486:222-227. DOI | PubMed
- Young VB, Schmidt TM. Antibiotic-associated diarrhea accompanied by large-scale alterations in the composition of the fecal microbiota. Journal of Clinical Microbiology. 2004; 42:1203-1206. DOI | PubMed
- Zhang X, Johnston ER, Liu W, Li L, Han X. Environmental changes affect the assembly of soil bacterial community primarily by mediating stochastic processes. Global Change Biology. 2016; 22:198-207. DOI | PubMed
- Zhu W, Winter MG, Byndloss MX, Spiga L, Duerkop BA, Hughes ER, Büttner L, de Lima Romão E, Behrendt CL, Lopez CA, Sifuentes-Dominguez L, Huff-Hardy K, Wilson RP, Gillis CC, Tükel Ç, Koh AY, Burstein E, Hooper LV, Bäumler AJ, Winter SE. Precision editing of the gut microbiota ameliorates colitis. Nature. 2018; 553:208-211. DOI | PubMed
Fonte
Reese AT, Cho EH, Klitzman B, Nichols SP, Wisniewski NA, et al. () Antibiotic-induced changes in the microbiota disrupt redox dynamics in the gut. eLife 7e35987. https://doi.org/10.7554/eLife.35987





