Introduzione
La risonanza magnetica (RMN) del ginocchio è la modalità di imaging standard per valutare i disturbi del ginocchio, e gli esami di RMN più muscoloscheletrici (MSK) vengono eseguiti sul ginocchio che su qualsiasi altra regione del corpo[1- 3]. La RM ha ripetutamente dimostrato un’elevata accuratezza per la diagnosi di patologie del menisco e del legamento crociato[4- 7] ed è utilizzata di routine per identificare coloro che trarrebbero beneficio da un intervento chirurgico [8- 10]. Inoltre, il valore predittivo negativo della RM del ginocchio è quasi del 100%, quindi la RM serve come metodo non invasivo per escludere disturbi chirurgici come le lacerazioni del legamento crociato anteriore (LCA)[11]. A causa della quantità e del dettaglio delle immagini in ogni esame di risonanza magnetica del ginocchio, l’interpretazione accurata della risonanza magnetica del ginocchio richiede molto tempo ed è soggetta a variabilità inter- e intra-reviewer, anche quando viene eseguita da radiologi MSK certificati dal consiglio[12]. Un sistema automatizzato per l’interpretazione delle immagini della risonanza magnetica del ginocchio ha una serie di potenziali applicazioni, come la rapida assegnazione di priorità ai pazienti ad alto rischio nel flusso di lavoro del radiologo e l’assistenza ai radiologi nell’effettuare le diagnosi[13]. Tuttavia, le proprietà multidimensionali e multiplanari della RM hanno finora limitato l’applicabilità dei metodi tradizionali di analisi delle immagini alla RM del ginocchio[13,14].
Gli approcci di apprendimento profondo, essendo in grado di apprendere automaticamente i livelli di caratteristiche, si prestano bene per modellare le complesse relazioni tra le immagini mediche e le loro interpretazioni[15,16]. Recentemente, tali approcci hanno superato i tradizionali metodi di analisi delle immagini e hanno permesso un significativo progresso nelle attività di imaging medico, tra cui la classificazione del cancro della pelle[17], la rilevazione della retinopatia diabetica[18] e la rilevazione dei noduli polmonari [19]. Le precedenti applicazioni dell’apprendimento profondo della risonanza magnetica al ginocchio sono state limitate alla segmentazione della cartilagine e al rilevamento delle lesioni cartilaginee[20- 22].
In questo studio, presentiamo MRNet, un modello di apprendimento profondo completamente automatizzato per l’interpretazione della RM del ginocchio, e confrontiamo le prestazioni del modello con quelle dei radiologi generici. Inoltre, valutiamo i cambiamenti nelle prestazioni diagnostiche degli esperti clinici quando le previsioni del modello di apprendimento profondo automatizzato sono fornite durante l’interpretazione. Infine, valutiamo le prestazioni del nostro modello su un set di dati esterni pubblicamente disponibili di esami di risonanza magnetica del ginocchio etichettati per lesioni del legamento crociato anteriore.
Metodi
Dataset
I rapporti degli esami di risonanza magnetica del ginocchio eseguiti presso lo Stanford University Medical Center tra il 1° gennaio 2001 e il 31 dicembre 2012 sono stati esaminati manualmente per curare un set di dati di 1.370 esami di risonanza magnetica del ginocchio. Il dataset conteneva 1.104 (80,6%) esami anomali, con 319 (23,3%) lacerazioni dell’ACL e 508 (37,1%) lacerazioni del menisco. Lacrime ACL e lacrime meniscali si sono verificate contemporaneamente in 194 (38,2%) esami. Le indicazioni più comuni per gli esami di risonanza magnetica del ginocchio in questo studio hanno incluso il dolore acuto e cronico, il follow-up o la valutazione preoperatoria, le lesioni/traumi e altri/non forniti. Gli esami sono stati eseguiti con scanner GE (GE Discovery, GE Healthcare, Waukesha, WI) con bobina di risonanza magnetica standard per il ginocchio e un protocollo di routine di risonanza magnetica del ginocchio senza contrasto che comprendeva le seguenti sequenze: coronale T1 ponderata, coronale T2 con saturazione del grasso, densità protonica sagittale (PD) ponderata, sagittale T2 con saturazione del grasso, e assiale PD ponderata con saturazione del grasso. Un totale di 775 (56,6%) esami ha utilizzato un campo magnetico 3.0-T; il rimanente ha utilizzato un campo magnetico 1.5-T. Vedere la tabella S1 per i parametri dettagliati della sequenza di risonanza magnetica. Per questo studio, da ogni esame sono state estratte serie pesate sul piano sagittale T2, serie pesate sul piano coronale T1 e serie pesate sul piano assiale PD per l’uso nel modello. Il numero di immagini di queste serie variava da 17 a 61 (media 31,48, SD 7,97).
Gli esami sono stati suddivisi in un set di training (1.130 esami, 1.088 pazienti), un set di tuning (120 esami, 111 pazienti) e un set di validazione (120 esami, 113 pazienti)(Fig 1). Per formare i set di validazione e di sintonizzazione, è stato utilizzato un campionamento casuale stratificato per assicurare che almeno 50 esempi positivi di ogni etichetta (anormale, lacerazione ACL e lacerazione meniscale) fossero presenti in ogni set. Tutti gli esami di ogni paziente sono stati inseriti nella stessa suddivisione. La tabella 1 contiene la patologia e le statistiche demografiche dei pazienti per ogni set di dati.
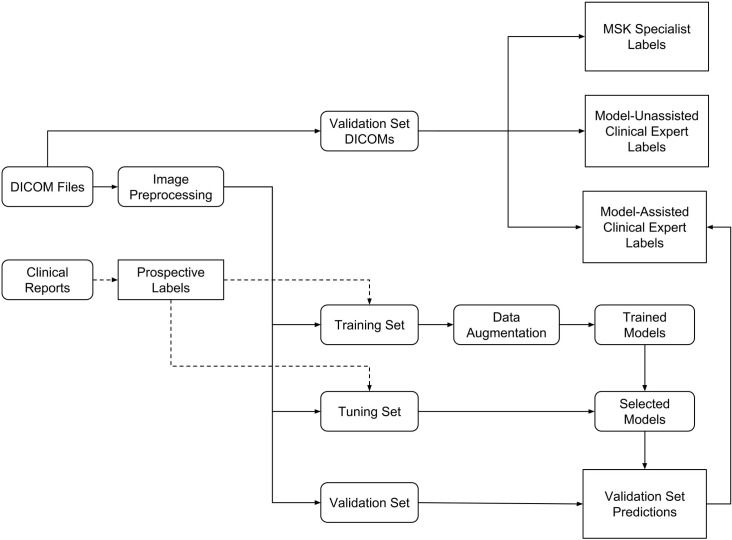
Fig. 1.Diagramma di flusso della configurazione sperimentale.Abbiamo raccolto retrospettivamente un set di dati di 1.370 esami di risonanza magnetica del ginocchio utilizzati per sviluppare il modello e per valutare il modello e gli esperti clinici. Le etichette sono state ottenute prospetticamente attraverso l’estrazione manuale dai rapporti clinici. Le immagini sono state estratte da file DICOM, preelaborate, poi collegate ai referti. Il set di dati è stato suddiviso in un set di training (per sviluppare il modello), un set di tuning (per scegliere tra i modelli) e un set di validazione (per valutare il modello e gli esperti clinici migliori). Il set di validazione DICOM corrisponde agli stessi esami del set di validazione, ma le immagini del set di validazione sono state preprocessate prima di essere inserite nel modello. Questi esami di validazione sono stati annotati in modo indipendente da radiologi muscoloscheletrici (MSK), esperti clinici non assistiti dal modello ed esperti clinici assistiti dal modello.
| Statistica | Formazione | Sintonizzazione | Convalida | ||
|---|---|---|---|---|---|
| Tutti | Etichette prospettiche1 | Etichette standard di riferimento2 | |||
| Numero di esami | 1,130 | 120 | 120 | ||
| Numero di pazienti | 1,0883 | 111 | 113 | ||
| Numero di pazienti di sesso femminile (%) | 480 (42.5) | 50 (41.7) | 39 (32.5) | ||
| Età, media (SD) | 38.3 (16.9) | 36.3 (16.9) | 37.1 (14.8) | ||
| Numero con anomalia (%) | 913 (80.8) | 95 (79.2) | 96 (80.0) | 99 (82.5) | |
| Numero con strappo ACL (%) | 208 (18.4) | 54 (45.0) | 57 (47.5) | 58 (48.3) | |
| Numero con strappo meniscale (%) | 397 (35.1) | 52 (43.3) | 59 (49.2) | 65 (54.2) | |
| Numero con ACL e strappo meniscale (%) | 125 (11.1) | 31 (25.8) | 38 (31.7) | 40 (33.3) | |

Fig. 1.Diagramma di flusso della configurazione sperimentale.Abbiamo raccolto retrospettivamente un set di dati di 1.370 esami di risonanza magnetica al ginocchio utilizzati per sviluppare il modello e per valutare il modello e gli esperti clinici. Le etichette sono state ottenute prospetticamente attraverso l’estrazione manuale dai rapporti clinici. Le immagini sono state estratte da file DICOM, preelaborate, poi collegate ai referti. Il set di dati è stato suddiviso in un set di training (per sviluppare il modello), un set di tuning (per scegliere tra i modelli) e un set di validazione (per valutare il modello e gli esperti clinici migliori). Il set di validazione DICOM corrisponde agli stessi esami del set di validazione, ma le immagini del set di validazione sono state preprocessate prima di essere inserite nel modello. Questi esami di validazione sono stati annotati in modo indipendente da radiologi muscoloscheletrici (MSK), esperti clinici non assistiti dal modello ed esperti clinici assistiti dal modello.
Convalida esterna
Abbiamo ottenuto un set di dati pubblicamente disponibile da Štajduhar et al. [23], composto da 917 esami sagittali pesati in PD di uno scanner Siemens Avanto 1.5-T presso il Clinical Hospital Centre Rijeka, Croazia. Dai referti dei radiologi, gli autori avevano estratto le etichette per 3 livelli di malattia ACL: non ferito (690 esami), parzialmente ferito (172 esami) e completamente rotto (55 esami). Abbiamo diviso gli esami in un rapporto di 60:20:20 in training, tuning e set di validazione utilizzando un campionamento casuale stratificato. Abbiamo prima applicato MRNet senza riqualificazione sui dati esterni, poi abbiamo ottimizzato MRNet utilizzando i set di training e tuning esterni. Il compito della classificazione è stato quello di discriminare tra ACL non feriti e ACL feriti (parzialmente feriti o completamente strappati).
Modello
Pretrattamento
Le immagini sono state estratte da file di Digital Imaging and Communications in Medicine (DICOM), scalate a 256 × 256 pixel e convertite in formato Portable Network Graphics (PNG) utilizzando il linguaggio di programmazione Python (versione 2.7) [24]e la libreria pydicom (versione 0.9.9) [25].
Per tener conto delle scale di intensità dei pixel variabili all’interno della serie MRI, è stato applicato alle immagini un algoritmo di standardizzazione dell’intensità basato su istogrammi[26]. Per ogni serie, una distribuzione rappresentativa dell’intensità è stata appresa dagli esami di formazione. Poi, i parametri di questa distribuzione sono stati utilizzati per regolare l’intensità dei pixel degli esami in tutti i set di dati (training, tuning e validazione). Sotto questa trasformazione, i pixel con valori simili corrispondono a tipi di tessuto simili. Dopo la standardizzazione dell’intensità, i valori dei pixel sono stati tagliati tra 0 e 255, la gamma standard per le immagini PNG.
MRNet
Il blocco principale del nostro sistema di previsione è MRNet, una rete neurale convoluzionale (CNN) che mappa una serie di MRI tridimensionale in base ad una probabilità[15](Fig 2). L’ingresso a MRNet ha dimensioni s × 3 × 256 × 256, dove s è il numero di immagini nella serie MRI (3 è il numero di canali di colore). In primo luogo, ogni fetta di immagine MRI bidimensionale è stata passata attraverso un estrattore di caratteristiche basato su AlexNet per ottenere un tensore s × 256 × 7 × 7 contenente caratteristiche per ogni fetta. È stato poi applicato uno strato di pooling globale medio per ridurre queste caratteristiche a s × 256. Abbiamo poi applicato il pooling massimo attraverso le fette per ottenere un vettore a 256 dimensioni, che è stato passato ad uno strato completamente collegato e la funzione di attivazione sigmoid per ottenere una previsione nell’intervallo 0 a 1. Abbiamo ottimizzato il modello utilizzando la perdita di crossentropia binaria. Per tener conto delle dimensioni di classe sbilanciate su tutti i compiti, la perdita per un esempio è stata scalata in modo inversamente proporzionale alla prevalenza della classe di quell’esempio nel dataset.
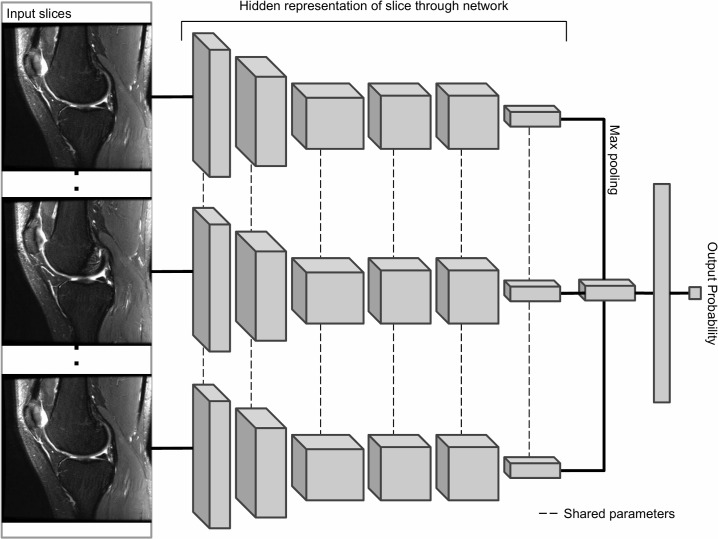
Fig. 2.Architettura MRNet.La MRNet è una rete neurale convoluzionale (CNN) che prende come input una serie di immagini MRI ed emette una previsione di classificazione. Le caratteristiche di AlexNet di ogni fetta della serie MRI sono combinate usando un’operazione di max pooling (massimo per elementi). Il vettore risultante viene alimentato attraverso uno strato completamente collegato per produrre una singola probabilità di uscita. Abbiamo addestrato un diverso MRNet per ogni compito (anomalia, lacerazione del legamento crociato anteriore [ACL], lacerazione del menisco) e tipo di serie (sagittale, coronale, assiale), con il risultato di 9 diversi MRNet (per la validazione esterna, usiamo solo il piano sagittale ACL lacerazione MRNet). Per ogni modello, la probabilità di uscita rappresenta la probabilità che il modello assegna alla serie per la presenza della diagnosi.
Durante l’addestramento, il gradiente della perdita è stato calcolato su ogni esempio di addestramento utilizzando l’algoritmo di backpropagazione, e i parametri MRNet sono stati regolati nella direzione opposta al gradiente [15]. Ogni esempio di allenamento è stato ruotato in modo casuale tra -25 e 25 gradi, spostato in modo casuale tra -25 e 25 pixel, e capovolto orizzontalmente con il 50% di probabilità ogni volta che appariva in allenamento. I parametri del modello sono stati salvati dopo ogni passaggio completo attraverso il set di allenamento, e il modello con la perdita media più bassa sul set di sintonizzazione è stato scelto per la valutazione sul set di validazione. La Fig. 2 descrive più dettagliatamente l’architettura MRNet. L’addestramento di ogni MRNet per 50 iterazioni attraverso il set di addestramento ha richiesto in media 6 ore con una GPU NVIDIA GeForce GTX 1070 8GB. MRNet è stato implementato con Python 3.6.3[27] e PyTorch 0.3.0[28].
La formazione di una CNN per la classificazione delle immagini da zero richiede in genere un set di dati più grande di 1.130 esempi. Per questo motivo, abbiamo inizializzato i pesi della porzione AlexNet della MRNet a valori ottimizzati sul database ImageNet[29] di 1,2 milioni di immagini su 1.000 classi, poi abbiamo messo a punto questi pesi per adattarli al nostro dataset MRI. Questo ha permesso ai primi strati della rete, che sono più difficili da ottimizzare rispetto ai livelli successivi, di riconoscere immediatamente caratteristiche generiche come linee e bordi. Questo approccio di “transfer learning” è stato applicato in modo simile al cancro della pelle [17] e alla retinopatia diabetica [18].

Fig 2.Architettura MRNet.La MRNet è una rete neurale convoluzionale (CNN) che prende come input una serie di immagini MRI ed emette una previsione di classificazione. Le caratteristiche di AlexNet di ogni fetta della serie MRI sono combinate usando un’operazione di max pooling (massimo per elementi). Il vettore risultante viene alimentato attraverso uno strato completamente collegato per produrre una singola probabilità di uscita. Abbiamo addestrato un diverso MRNet per ogni compito (anomalia, lacerazione del legamento crociato anteriore [ACL], lacerazione del menisco) e tipo di serie (sagittale, coronale, assiale), con il risultato di 9 diversi MRNet (per la validazione esterna, usiamo solo il piano sagittale ACL lacerazione MRNet). Per ogni modello, la probabilità di uscita rappresenta la probabilità che il modello assegna alla serie per la presenza della diagnosi.
Interpretazione MRNet
Per garantire che i modelli MRNet imparassero le caratteristiche pertinenti, abbiamo generato delle mappature di attivazione delle classi (CAM)[30](Fig 3). Per generare una CAM per un’immagine, abbiamo calcolato una media ponderata attraverso le 256 mappe delle caratteristiche CNN utilizzando i pesi del livello di classificazione per ottenere un’immagine 7 × 7. La CAM è stata poi mappata su uno schema di colori, campionata a 256 × 256 pixel e sovrapposta all’immagine originale in ingresso. Utilizzando i parametri del livello finale della rete per pesare le feature map, le feature map più predittive appaiono più luminose. Quindi, le aree più luminose delle CAM sono le regioni che più influenzano la previsione del modello.
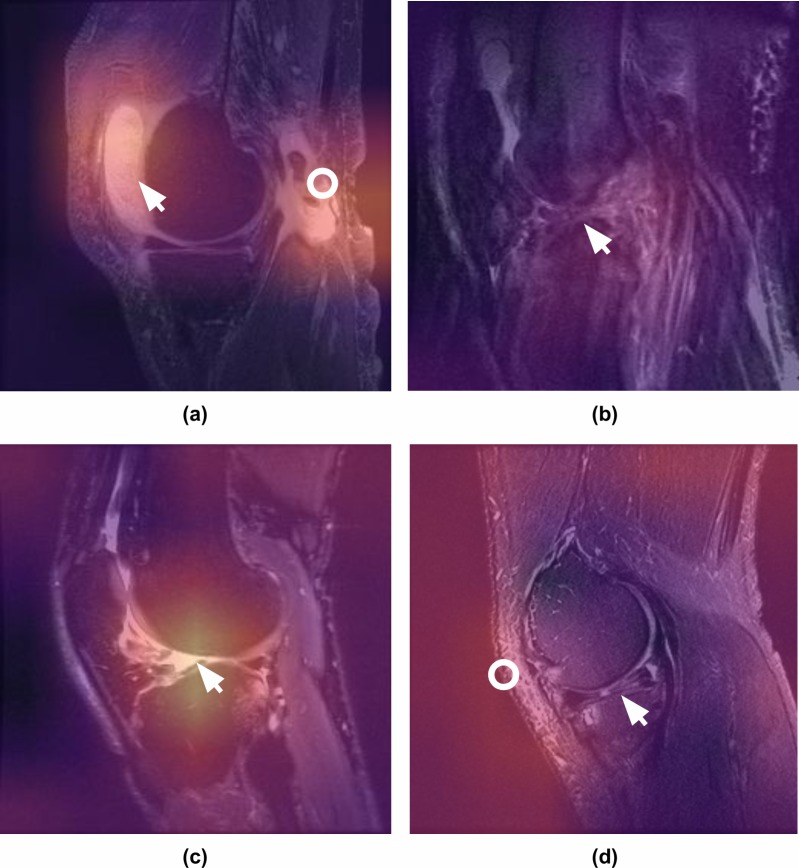
Fig 3.Mappature di attivazione di classe per l’interpretazione di MRNet.Le mappature di attivazione di classe (CAM) evidenziano quali pixel nelle immagini sono importanti per la decisione di classificazione del modello. Uno dei radiologi muscoloscheletrici certificati dalla commissione ha annotato le immagini (frecce e cerchi bianchi) e ha fornito le seguenti didascalie. a) Immagine sagittale T2-pesata del ginocchio che mostra un grande versamento (freccia) e la rottura del tendine gastrocnemio (anello), che sono stati correttamente localizzati dal modello e classificati come anormale. Si noti che il modello non è stato specificamente addestrato a rilevare queste patologie, ma è stato in grado di riconoscere le anomalie in base al contrasto con gli esami del ginocchio normali. (b) Immagine del ginocchio ponderata in T2 sagittale complicata da un significativo artefatto di movimento che dimostra una lacerazione completa del legamento crociato anteriore (ACL) (freccia), che è stata correttamente classificata e localizzata dal modello. Poiché speravamo di approssimare al meglio la realtà della pratica clinica – in cui la prevalenza di artefatti (cioè il movimento, metallico) e altri disturbi tecnici disturba l’interpretazione della risonanza magnetica del ginocchio – non abbiamo escluso i casi rumorosi dai dati di formazione o di convalida. c) Immagine del ginocchio pesata in T2 sagittale che dimostra la completa perturbazione dell’ACL, che è stata correttamente identificata dal modello come anormale e classificata come lacerazione dell’ACL. La CAM indica il punto focale del modello in corrispondenza dell’attaccamento anormale dell’ACL (freccia). (d) Immagine del ginocchio ponderata in T2 sagittale che mostra una lacerazione complessa che coinvolge il corno posteriore del menisco laterale (freccia). Mentre il modello ha classificato questo esame come anormale, la CAM indica che l’aumento del segnale sottocutaneo (anello) nei tessuti molli anteriore/laterale ha contribuito alla decisione, ma la lacerazione del menisco no.

Fig. 3.Mappature di attivazione di classe per l’interpretazione MRNet.Le mappature di attivazione di classe (CAM) evidenziano quali pixel nelle immagini sono importanti per la decisione di classificazione del modello. Uno dei radiologi muscoloscheletrici certificati dalla commissione ha annotato le immagini (frecce e cerchi bianchi) e ha fornito le seguenti didascalie. a) Immagine sagittale T2-pesata del ginocchio che mostra un grande versamento (freccia) e la rottura del tendine gastrocnemio (anello), che sono stati correttamente localizzati dal modello e classificati come anormale. Si noti che il modello non è stato specificamente addestrato a rilevare queste patologie, ma è stato in grado di riconoscere le anomalie in base al contrasto con gli esami del ginocchio normali. (b) Immagine del ginocchio ponderata in T2 sagittale complicata da un significativo artefatto di movimento che dimostra una lacerazione completa del legamento crociato anteriore (ACL) (freccia), che è stata correttamente classificata e localizzata dal modello. Poiché speravamo di approssimare al meglio la realtà della pratica clinica – in cui la prevalenza di artefatti (cioè il movimento, metallico) e altri disturbi tecnici disturba l’interpretazione della risonanza magnetica del ginocchio – non abbiamo escluso i casi rumorosi dai dati di formazione o di convalida. c) Immagine del ginocchio pesata in T2 sagittale che dimostra la completa perturbazione dell’ACL, che è stata correttamente identificata dal modello come anormale e classificata come lacerazione dell’ACL. La CAM indica il punto focale del modello in corrispondenza dell’attaccamento anormale dell’ACL (freccia). (d) Immagine del ginocchio ponderata in T2 sagittale che mostra una lacerazione complessa che coinvolge il corno posteriore del menisco laterale (freccia). Mentre il modello ha classificato questo esame come anormale, la CAM indica che l’aumento del segnale sottocutaneo (anello) nei tessuti molli anteriori/laterali ha contribuito alla decisione, ma la lacerazione del menisco no.
Combinando le previsioni MRNet
Date le previsioni del T2 sagittale, del T1 coronale e del PD assiale MRNets sul set di allenamento, insieme alle loro corrispondenti etichette originali, abbiamo allenato una regressione logistica per pesare le previsioni delle 3 serie e generare una singola uscita per ogni esame(Fig 4). Le serie più vantaggiose, determinate dai coefficienti della regressione logistica montata, sono state la PD assiale per le anomalie e le lacerazioni meniscali e la T1 coronale per le lacerazioni ACL. Dopo l’addestramento, la regressione logistica è stata applicata alle previsioni dei 3 MRNets per il set di validazione interna per ottenere le previsioni finali. Abbiamo addestrato 3 modelli di regressione logistica in totale 1 per ogni compito (rilevamento di anomalie, lacerazioni ACL e lacerazioni meniscali). Questi modelli sono stati implementati in Python[24] utilizzando il pacchetto scikit-learn[31]. Per la validazione esterna, dato che c’era solo 1 serie nel dataset, abbiamo usato la previsione da un singolo MRNet direttamente come output finale.

Fig. 4.Combinando le previsioni di serie usando la regressione logistica.Ogni esame contiene 3 tipi di serie: sagittale, coronale e assiale. Per ogni compito (anomalia, lacerazione ACL, lacerazione meniscale), abbiamo addestrato un classificatore di regressione logistica per combinare le 3 probabilità emesse dagli MRNets per produrre una singola probabilità prevista per l’esame. Le probabilità previste da un esame nel set di validazione interno sono mostrate come esempio.ACL, legamento crociato anteriore; CNN, rete neurale convoluzionale; LR, regressione logistica.

Fig. 4.Fig. 4. Combinazione di previsioni di serie utilizzando la regressione logistica.Ogni esame contiene 3 tipi di serie: sagittale, coronale e assiale. Per ogni compito (anomalia, lacerazione ACL, lacerazione meniscale), abbiamo addestrato un classificatore di regressione logistica per combinare le 3 probabilità prodotte dagli MRNets per produrre una singola probabilità prevista per l’esame. Le probabilità previste da un esame nel set di validazione interno sono mostrate come esempio.ACL, legamento crociato anteriore; CNN, rete neurale convoluzionale; LR, regressione logistica.
Valutazione
Le etichette standard di riferimento sono state ottenute sul set di validazione interna dal voto di maggioranza di 3 radiologi MSK certificati dal consiglio direttivo presso un grande studio accademico (anni in pratica 6-19 anni, media 12 anni). I radiologi MSK hanno avuto accesso a tutte le serie DICOM, al rapporto originale, alla storia clinica e agli esami di follow-up durante l’interpretazione. Tutti i lettori che hanno partecipato allo studio hanno utilizzato un ambiente di archiviazione delle immagini cliniche e del sistema di comunicazione (PACS) (GE Centricity) in una sala di lettura diagnostica, e la valutazione è stata effettuata sulle immagini cliniche DICOM presentate su un display di grado medico di almeno 3 megapixel con una luminanza minima di 1 cd/m2, luminanza massima di 400 cd/m2, dimensione dei pixel di 0,2, e risoluzione nativa di 1.500 × 2.000 pixel. Gli esami sono stati ordinati in ordine cronologico inverso. Ad ogni esame sono state assegnate 3 etichette binarie per la presenza o l’assenza di (1) qualsiasi anomalia, (2) uno strappo ACL e (3) uno strappo meniscale. Le definizioni per le etichette erano le seguenti:
- Anomalia: normale (tutte le immagini esaminate sono prive di anomalie) o anormale (i risultati anormali nel set di validazione interna che non erano lacerazioni del legamento crociato anteriore o del menisco includevano osteoartrite, versamento, sindrome della banda iliotibiale, lacerazione del legamento crociato posteriore, frattura, contusione, plica e distorsione del legamento collaterale mediale);
- ACL: intatto (normale, degenerazione mucoide, cisti gangliare, distorsione) o lacerazione (lacerazione parziale di basso grado con <50% di fibre lacerate, lacerazione parziale di alto grado con >50% di fibre lacerate, lacerazione completa) [32];
- Menisco: intatto (alterazioni normali, degenerative senza lacerazione, alterazioni post-chirurgiche senza lacerazione) o lacerazione (aumento del segnale che raggiunge la superficie articolare su almeno 2 fette o deformità morfologica)[33,34].
Indipendentemente dai radiologi MSK, 7 radiologi generici certificati dal consiglio direttivo e 2 chirurghi ortopedici praticanti presso lo Stanford University Medical Center (3-29 anni di pratica, in media 12 anni) hanno etichettato il set di validazione interna, non vedendo i rapporti e le etichette originali. Le etichette di questi esperti clinici sono state misurate rispetto alle etichette standard di riferimento stabilite dal consenso dei radiologi MSK. I radiologi generici sono stati divisi casualmente in 2 gruppi, con 4 radiologi nel Gruppo 1 e 3 radiologi nel Gruppo 2. I 2 chirurghi ortopedici erano anche nel Gruppo 1. Il Gruppo 1 ha esaminato per primo il set di validazione senza l’assistenza del modello, e il Gruppo 2 ha esaminato per primo il set di validazione con l’assistenza del modello. Per le revisioni con l’assistenza del modello, le previsioni del modello sono state fornite come probabilità previste di una diagnosi positiva (ad esempio, 0,98 lacerazione dell’ACL). Dopo un periodo di dilavamento di 10 giorni, il Gruppo 1 ha poi rivisto il set di validazione in un ordine diverso con l’assistenza di modello, e il Gruppo 2 ha rivisto il set di validazione senza l’assistenza di modello. Il comitato di revisione istituzionale di Stanford ha approvato questo studio.
Metodi statistici
Le misure di prestazione per il modello, i radiologi generici e i chirurghi ortopedici includevano sensibilità, specificità e precisione. Abbiamo anche calcolato la micro-media di queste statistiche solo per i radiologi generici e per tutti gli esperti clinici (radiologi generici e chirurghi). Abbiamo valutato le prestazioni del modello con l’area sotto la curva caratteristica operativa del ricevitore (AUC). Per valutare la variabilità delle stime, forniamo intervalli di confidenza dei punteggi Wilson del 95%[35] per sensibilità, specificità e accuratezza e intervalli di confidenza DeLong del 95% per AUC[36,37]. Una soglia di 0,5 è stata utilizzata per dicotomizzare le previsioni del modello. Le prestazioni del modello sul set di convalida esterno sono state valutate con gli intervalli di confidenza AUC e DeLong al 95%.
Poiché in questo studio abbiamo effettuato confronti multipli per valutare le prestazioni del modello rispetto a quelle dei radiologi generici praticanti e anche per valutare l’utilità clinica di fornire assistenza al modello, abbiamo controllato il tasso di falsificazione complessivo (FDR) a 0,05 [38] e abbiamo riportato sia i valori p non corretti che i valori q corretti. In linea di massima, FDR < 0,05 può essere interpretato come la proporzione prevista (0,05) di false affermazioni di rilevanza in tutti i risultati significativi. Pertanto, invece di utilizzare il valore pnon rettificato per valutare la significatività statistica, un valore q< 0,05 rappresenta correttamente questi confronti multipli. Per valutare le prestazioni del modello rispetto a quelle dei radiologi generici, abbiamo utilizzato un test chi-squared di Pearson su due lati per valutare se ci fossero differenze significative nella specificità, sensibilità e accuratezza tra il modello e la micro-media dei radiologi generici. I chirurghi ortopedici non sono stati inclusi in questo confronto.
Abbiamo valutato l’utilità clinica di fornire le previsioni del modello agli esperti clinici testando se le metriche delle prestazioni di tutti i 7 radiologi generici e 2 chirurghi ortopedici sono aumentate quando è stata fornita loro l’assistenza del modello. Esiste una variabilità naturale quando un esperto clinico valuta lo stesso studio di risonanza magnetica del ginocchio in tempi diversi, quindi non è inaspettato che le metriche delle prestazioni di un esperto clinico siano leggermente migliori o leggermente peggiori quando vengono testate in due occasioni, indipendentemente dall’assistenza del modello. Così, abbiamo eseguito robusti test di ipotesi per valutare se gli esperti clinici (come gruppo) hanno dimostrato un miglioramento statisticamente significativo con l’assistenza del modello. Abbiamo utilizzato un test a 1 coda t sul cambiamento (differenza) delle metriche di performance per i 9 esperti clinici per tutte e 3 le etichette. Per valutare se questi risultati dipendevano specificamente dal miglioramento dei chirurghi ortopedici, abbiamo eseguito un’analisi di sensibilità: abbiamo ripetuto il test a 1 coda t sulla variazione delle metriche di performance solo per i radiologi generici, esclusi i chirurghi ortopedici, per determinare se c’era ancora un miglioramento significativo.
L’esatta Fleiss kappa[39,40] è stata riportata per valutare il livello di consenso dei 3 radiologi MSK, il cui voto di maggioranza è stato utilizzato per le etichette standard di riferimento. Inoltre, per valutare se l’assistenza del modello può migliorare l’affidabilità tra le classificazioni, riportiamo l’esatto Fleiss kappa del set di 9 esperti clinici con e senza l’assistenza del modello per ognuno dei 3 compiti.
Tutte le analisi statistiche sono state completate in ambiente R per il calcolo statistico[41], utilizzando i pacchetti irr, pROC, binom e qvalue[38,42- 44], e il codice R è stato fornito con la presentazione.
Risultati
L’accordo inter-rater sulla validazione interna stabilita tra i 3 radiologi MSK, misurata dal punteggio esatto di Fleiss kappa, è stato di 0,508 per il rilevamento di anomalie, 0,800 per il rilevamento di lacerazioni ACL, e 0,745 per il rilevamento di lacerazioni meniscali.
Prestazioni del modello
Per la rilevazione di anomalie, la rilevazione di lacerazioni ACL e la rilevazione di lacerazioni meniscali, il modello ha raggiunto AUCs di 0,937 (95% CI 0,895, 0,980), 0,965 (95% CI 0,938, 0,993) e 0,847 (95% CI 0,780, 0,914), rispettivamente(Fig 5). Nel rilevare le anomalie, non ci sono state differenze significative nelle metriche delle prestazioni del modello e dei radiologi generici(Tabella 2). La specificità del modello per il rilevamento delle anomalie era inferiore alla micro-media dei radiologi generici, rispettivamente a 0,714 (95% CI 0,500, 0,862) e 0,844 (95% CI 0,776, 0,893). Il modello ha raggiunto una sensibilità di 0,879 (95% CI 0,800, 0,929) e una precisione di 0,850 (95% CI 0,775, 0,903), mentre i radiologi generici hanno raggiunto una sensibilità di 0,905 (95% CI 0,881, 0,924) e una precisione di 0,894 (95% CI 0,871, 0,913)(Tabella 2).
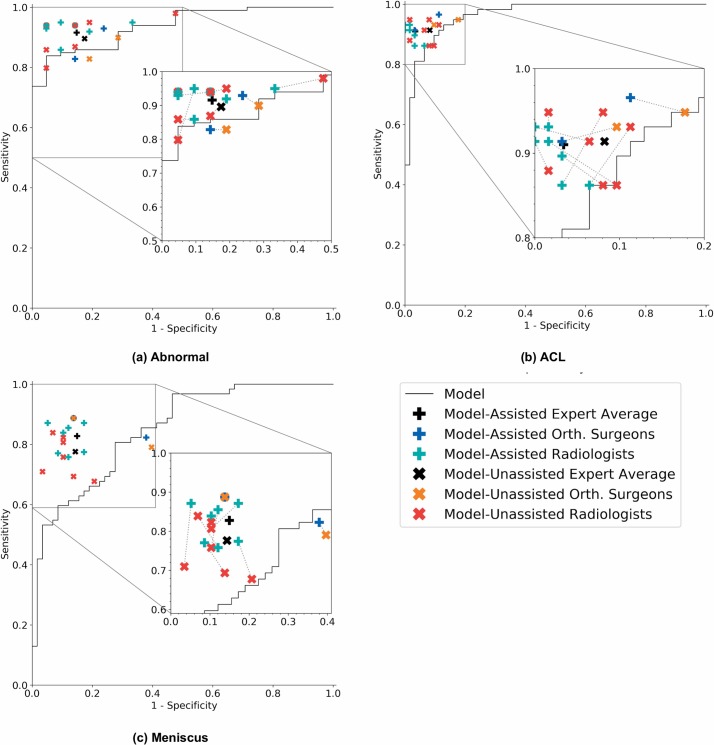
Fig. 5.Curve caratteristiche di funzionamento del ricevitore del modello e punti di funzionamento di esperti clinici non assistiti e assistiti.Ogni grafico illustra la curva caratteristica operativa del ricevitore (ROC) dell’algoritmo (curva nera) sull’insieme di validazione impostato per (a) l’anomalia, (b) lo strappo del legamento crociato anteriore (ACL) e (c) lo strappo del menisco. La curva ROC è generata variando la soglia di discriminazione (usata per convertire le probabilità di uscita in previsioni binarie). Vengono anche tracciati i singoli punti clinici esperti (specificità, sensibilità), dove le x rosse rappresentano i radiologi generici non assistiti, le x arancioni rappresentano i chirurghi ortopedici non assistiti, i segni verdi più rappresentano i radiologi generici assistiti e i segni blu più rappresentano i chirurghi ortopedici assistiti. Tracciamo anche la macro-media degli esperti clinici non assistiti (x nere) e la macro-media degli esperti clinici assistiti (segni neri più). Ogni punto operativo del perito clinico non assistito è collegato al corrispondente punto operativo assistito dal modello con una linea tratteggiata.
| Previsione | Specificità (95% CI) | p-Valueq-valueq | Sensibilità (95% CI) | p-Valueq-valueq | Precisione (95% CI) | p-Valueq-valueq |
|---|---|---|---|---|---|---|
| Anomalia | ||||||
| Modello, soglia = 0,5 | 0.714 (0.500, 0.862) | — | 0.879 (0.800, 0.929) | — | 0.850 (0.775, 0.903) | — |
| Micro-media generale non assistita di radiologo generale | 0.844 (0.776, 0.893) | 0.247 0.344 | 0.905 (0.881, 0.924) | 0.528 0.620 | 0.894 (0.871, 0.913) | 0.201 0.301 |
| Strappo ACL | ||||||
| Modello, soglia = 0,5 | 0.968 (0.890, 0.991) | — | 0.759 (0.635, 0.850) | — | 0.867 (0.794, 0.916) | — |
| Micro-media generale non assistita di radiologo generale | 0.933 (0.906, 0.953) | 0.441 0.566 | 0.906 (0.874, 0.931) | 0.002 0.019 | 0.920 (0.900, 0.937) | 0.075 0.173 |
| Strappo meniscale | ||||||
| Modello, soglia = 0,5 | 0.741 (0.616, 0.837) | — | 0.710 (0.587, 0.808) | — | 0.725 (0.639, 0.797) | — |
| Micro-media generale non assistita di radiologo generale | 0.882 (0.847, 0.910) | 0.003 0.019 | 0.820 (0.781, 0.853) | 0.504 0.619 | 0.849 (0.823, 0.871) | 0.015 0.082 |
Il modello era altamente specifico per la rilevazione delle lacerazioni ACL, raggiungendo una specificità di 0,968 (95% CI 0,890, 0,991), che è superiore alla micro-media dei radiologi generici, a 0,933 (95% CI 0,906, 0,953), ma questa differenza non era statisticamente significativa(Tabella 2). I radiologi generici hanno raggiunto una sensibilità significativamente più elevata rispetto al modello nel rilevamento delle lacerazioni ACL(valore p= 0,002, q= 0,019); la sensibilità micro-media dei radiologi generici è stata di 0,906 (95% CI 0,874, 0,931), mentre il modello ha raggiunto una sensibilità di 0,759 (95% CI 0,635, 0,850). I radiologi generici hanno anche raggiunto una specificità significativamente più alta nella rilevazione delle lacerazioni meniscali(p-valore= 0,003, q-valore= 0,019), con una specificità di 0,892 (95% CI 0,858, 0,918) rispetto ad una specificità di 0,741 (95% CI 0,616, 0,837) per il modello. Non ci sono state altre differenze significative nelle metriche di performance(Tabella 2). Sintesi delle stime metriche delle prestazioni e degli intervalli di confidenza si possono trovare nella Tabella 2, e le metriche delle prestazioni individuali per i 7 radiologi generici certificati dalla commissione e i 2 chirurghi ortopedici di questo studio si possono trovare nella Tabella S2.

Fig. 5.Curve caratteristiche operative del ricevitore del modello e punti operativi di esperti clinici non assistiti e assistiti.Ogni grafico illustra la curva caratteristica operativa del ricevitore (ROC) dell’algoritmo (curva nera) sull’insieme di validazione impostato per (a) l’anomalia, (b) la lacerazione del legamento crociato anteriore (ACL) e (c) la lacerazione del menisco. La curva ROC è generata variando la soglia di discriminazione (usata per convertire le probabilità di uscita in previsioni binarie). Vengono anche tracciati i singoli punti clinici esperti (specificità, sensibilità), dove le x rosse rappresentano i radiologi generici non assistiti, le x arancioni rappresentano i chirurghi ortopedici non assistiti, i segni verdi più rappresentano i radiologi generici assistiti e i segni blu più rappresentano i chirurghi ortopedici assistiti. Tracciamo anche la macro-media degli esperti clinici non assistiti (x nere) e la macro-media degli esperti clinici assistiti (segni neri più). Ogni punto operativo del perito clinico non assistito è collegato al corrispondente punto operativo assistito dal modello con una linea tratteggiata.
Utilità clinica del modello di assistenza
L’utilità clinica di fornire modelli di previsione agli esperti clinici durante il processo di etichettatura è illustrata nella Fig 6, e i valori numerici forniti nella Tabella 3. Quando agli esperti clinici è stata fornita l’assistenza del modello, c’è stato un aumento statisticamente significativo della specificità degli esperti clinici nell’identificazione delle lacerazioni del legamento crociato anteriore (p-valore < 0,001, q-valore =0,006). L’aumento medio della specificità dell’ACL è stato di 0,048 (4,8%), e poiché il set di validazione conteneva 62 esami negativi per la lacerazione dell’ACL, questo aumento della specificità nel setting clinico ottimale significherebbe potenzialmente 3 pazienti in meno inviati in chirurgia per sospetta lacerazione dell’ACL inutilmente. Sebbene sia sembrato che l’assistenza del modello abbia anche aumentato significativamente l’accuratezza degli esperti clinici nel rilevare le lacrime ACL (p-valore p = 0,020) e la sensibilità nel rilevare le lacrime del menisco (p-valore p = 0,028), questi risultati non erano più significativi dopo l’aggiustamento per i confronti multipli controllando il FDR (q-valori = 0,092 e 0,110, rispettivamente). Non ci sono stati altri miglioramenti statisticamente significativi delle prestazioni degli esperti clinici con l’assistenza del modello. I risultati individuali, i valori pnon corretti e i valori qcorretti sono forniti nella tabella S3.
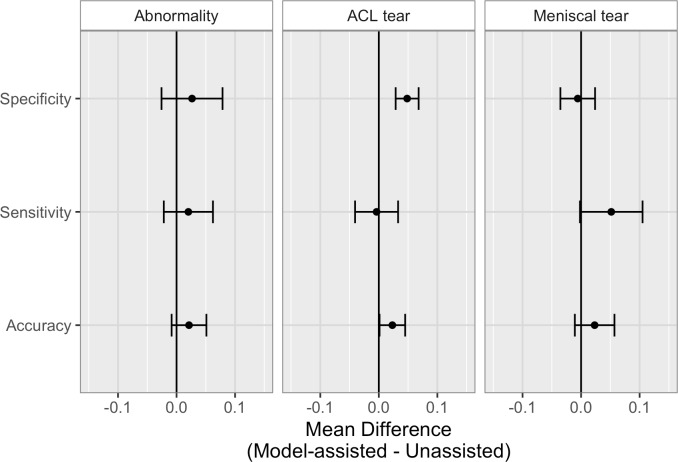
Fig. 6.Fig. 6. Confronto tra le metriche di performance non assistita e model-assisted degli esperti clinici sul set di validazione.Differenze medie (con barre di errore CI del 95%) nelle metriche di performance degli esperti clinici (model-assisted meno unassisted) per l’anomalia, la lacerazione del legamento crociato anteriore (ACL) e la rilevazione della lacerazione meniscale. I valori numerici sono forniti nella tabella 3 e i valori individuali nella tabella S2.
| Metrico | Anomalia | Strappo ACL | Strappo meniscale | |||
|---|---|---|---|---|---|---|
| Differenza media (95% CI) | p-Valueq-valueq | Differenza media (95% CI) | p-Valueq-valueq | Differenza media (95% CI) | p-Valueq-valueq | |
| Specificità | 0.026 (−0.026, 0.079) | 0.138 0.248 | 0.048 (0.029, 0.068) | <0.001 0.006 | −0.006 (−0.035, 0.024) | 0.667 0.692 |
| Sensibilità | 0.020 (−0.022, 0.062) | 0.150 0.253 | −0.004 (−0.041, 0.033) | 0.592 0.639 | 0.052 (−0.002, 0.105) | 0.028 0.110 |
| Precisione | 0.021 (−0.008, 0.051) | 0.069 0.173 | 0.023 (0.001, 0.045) | 0.020 0.092 | 0.023 (−0.011, 0.057) | 0.077 0.173 |
Per determinare se il miglioramento statisticamente significativo della specificità nell’identificazione delle lacerazioni della LCA con l’assistenza del modello dipendeva dalle metriche di performance dei chirurghi ortopedici, abbiamo valutato il miglioramento solo dei radiologi generici, escludendo i chirurghi ortopedici. Questa analisi di sensibilità ha confermato che anche tra i soli radiologi generici, c’è stato un significativo aumento della specificità nell’identificazione delle lacerazioni ACL(p-valore= 0,003, q-valore= 0,019; vedi tabella S4). Inoltre, abbiamo calcolato il Fleiss kappa per i 9 esperti clinici con e senza l’assistenza del modello, e anche se non abbiamo valutato la significatività statistica, abbiamo osservato che l’assistenza del modello ha aumentato la misura del Fleiss kappa di affidabilità inter-rater per tutti e 3 i compiti. Con l’assistenza del modello, la misura Fleiss kappa per la rilevazione delle anomalie è aumentata da 0,571 a 0,640, per la rilevazione delle lacrime ACL è aumentata da 0,754 a 0,840, e per la rilevazione delle lacrime meniscali è aumentata da 0,526 a 0,621.

Fig. 6.Confronto tra la metrica delle prestazioni non assistita e la metrica delle prestazioni assistita da modelli di esperti clinici sul set di validazione.Differenze medie (con barre di errore CI del 95%) nelle metriche di performance degli esperti clinici (model-assisted meno unassisted) per l’anomalia, la lacerazione del legamento crociato anteriore (ACL) e la rilevazione della lacerazione meniscale. I valori numerici sono forniti nella tabella 3 e i valori individuali nella tabella S2.
Convalida esterna
L’MRNet si è allenato sulla serie di etichette a strappo Stanford sagittal pesate T2 e Stanford ACL ha ottenuto un AUC di 0,824 (95% CI 0,757, 0,892) sul set di convalida Štajduhar et al. senza alcuna formazione aggiuntiva. Inoltre, abbiamo addestrato 3 MRNets a partire dai pesi ImageNet sul set di formazione Štajduhar et al. con diversi semi casuali. Abbiamo selezionato la MRNet con la perdita media più bassa sul set di sintonia e poi abbiamo valutato questo modello sul set di validazione. Questo modello ha raggiunto un AUC di 0,911 (95% CI 0,864, 0,958) sul set di validazione di Štajduhar et al. Štajduhar et al. hanno registrato un AUC di 0,894 per il loro modello migliore, un approccio semi-automatico che utilizza macchine vettoriali di supporto, anche se è stato valutato utilizzando uno schema di validazione incrociata di 10 volte [23]. MRNet ha richiesto meno di 30 minuti per l’addestramento e meno di 2 minuti per valutare il set di dati di Štajduhar et al. con una GPU NVIDIA GeForce GTX 12GB.
Discussione
Lo scopo di questo studio è stato quello di progettare e valutare un modello di apprendimento profondo per la classificazione delle patologie del ginocchio e di confrontare le prestazioni con quelle degli esperti clinici umani, sia con che senza l’assistenza del modello durante l’interpretazione in un design crossover. I nostri risultati dimostrano che un approccio di apprendimento profondo può raggiungere elevate prestazioni nelle attività di classificazione clinica sulla RM del ginocchio, con AUC per il rilevamento delle anomalie, il rilevamento delle lacerazioni ACL e il rilevamento delle lacerazioni del menisco di 0,937 (95% CI 0,895, 0,937), 0,965 (95% CI 0,938, 0,965) e 0,847 (95% CI 0,780, 0,847), rispettivamente. In particolare, il modello ha raggiunto un’elevata specificità nel rilevare le lacerazioni ACL sul set di validazione interno, il che suggerisce che tale modello, se utilizzato nel flusso di lavoro clinico, può avere il potenziale per escludere efficacemente le lacerazioni ACL. Su un set di dati esterno che utilizza serie pesate T1 invece di serie pesate T2 e una diversa convenzione di etichettatura per le lesioni ACL, lo stesso modello di lacerazione ACL ha ottenuto un AUC di 0,824 (95% CI 0,757, 0,892). La riqualificazione sul set di dati esterni ha migliorato l’AUC a 0,911 (95% CI 0,864, 0,958). Il nostro modello di apprendimento profondo ha raggiunto risultati all’avanguardia sul set di dati esterni, ma solo dopo la riqualificazione. Resta da vedere se il modello sarebbe meglio generalizzare ad un set di dati esterni con più serie di MRI e un protocollo di MRI più simile. Abbiamo anche scoperto che fornire le previsioni del modello di apprendimento profondo agli esperti clinici umani come aiuto diagnostico ha portato a specificità significativamente più elevate nell’identificazione delle lacerazioni del legamento crociato anteriore. Infine, a differenza degli esperti umani, che hanno richiesto in media più di 3 ore per rivedere completamente 120 esami, il modello di apprendimento profondo ha fornito tutte le classificazioni in meno di 2 minuti. I nostri risultati suggeriscono che l’apprendimento profondo può essere applicato con successo alla risonanza magnetica MSK avanzata per generare rapide classificazioni automatizzate della patologia e che l’output del modello può migliorare le interpretazioni cliniche.
Ci sono molte interessanti applicazioni potenziali di un modello di apprendimento profondo automatizzato per la diagnosi della RM del ginocchio nella pratica clinica. Ad esempio, il modello descritto potrebbe essere immediatamente applicato per la prioritizzazione delle liste di lavoro diagnostiche, in cui gli esami individuati come anomali potrebbero essere spostati in avanti nel flusso di lavoro di interpretazione delle immagini, e a quelli identificati come normali potrebbe essere automaticamente assegnata una lettura preliminare di “normale”. Con il suo alto valore predittivo negativo per le anomalie, il modello potrebbe portare a un rapido feedback preliminare per i pazienti i cui esami risultano “normali”. Inoltre, fornendo risultati rapidi al clinico ordinante potrebbe migliorare la disposizione in altre aree del sistema sanitario. In questo lavoro abbiamo notato che la specificità per il rilevamento delle lacerazioni del legamento crociato anteriore è migliorata sia per i radiologi generici che per i chirurghi ortopedici, il che implica che questo modello potrebbe aiutare a ridurre gli esami e gli interventi chirurgici aggiuntivi non necessari. La previsione e la localizzazione automatizzata delle anomalie potrebbe aiutare i radiologi generici o anche i clinici non radiologi (chirurghi ortopedici) a interpretare l’imaging medico per i pazienti al punto di cura piuttosto che aspettare l’interpretazione del radiologo specializzato, il che potrebbe aiutare a un’interpretazione efficiente, ridurre gli errori e aiutare a standardizzare la qualità delle diagnosi quando i radiologi specializzati in MSK non sono prontamente disponibili. In definitiva, sono necessari ulteriori studi per valutare l’integrazione ottimale di questo modello e di altri modelli di apprendimento profondo nel contesto clinico. Tuttavia, i nostri risultati forniscono un supporto precoce per un futuro in cui i modelli di apprendimento profondo possono giocare un ruolo significativo nell’assistenza ai medici e ai sistemi sanitari.
Per esaminare l’effetto che un modello di apprendimento profondo può avere sulle prestazioni interpretative dei medici, il nostro studio ha deliberatamente reclutato radiologi generici per interpretare gli esami di risonanza magnetica del ginocchio con e senza previsioni del modello. Abbiamo riscontrato un miglioramento statisticamente significativo della specificità per il compito di rilevamento delle lacrime ACL con l’assistenza del modello e, anche se non statisticamente significativo, una maggiore accuratezza per il rilevamento delle lacrime ACL e una maggiore sensibilità per il rilevamento delle lacrime meniscali. Sia per i radiologi generici che per i clinici non radiologi (chirurghi ortopedici), abbiamo riscontrato un miglioramento della sensibilità e/o della specificità per tutti e 3 i compiti con l’assistenza del modello(Fig 5; Tabella 3), anche se il gruppo di chirurghi era troppo piccolo per l’analisi formale. È importante notare che l’assistenza del modello ha anche portato ad una maggiore affidabilità tra gli esperti clinici per tutti e 3 i compiti, con misure di Fleiss kappa più elevate con l’assistenza del modello che senza. A nostra conoscenza, questo è il primo studio ad esplorare la possibilità di fornire output di modelli di apprendimento profondo per assistere radiologi e clinici non radiologi nel compito di interpretazione delle immagini. Sarà necessario un maggiore lavoro per capire se e come i modelli di apprendimento profondo possano ottimizzare le prestazioni di interpretazione dei radiologi praticanti e dei clinici non radiologi.
Una difficoltà nell’apprendimento profondo per l’imaging medico è la cura di grandi set di dati contenenti esempi della grande varietà di anomalie che possono verificarsi su un dato esame di imaging per formare un classificatore accurato, che è una strategia che abbiamo impiegato per il rilevamento di ACL e lacerazioni meniscali. Tuttavia, l’altro nostro compito di classificazione è stato quello di distinguere “normale” da “anormale” con l’intenzione che se il modello potesse imparare il range di normalità per una data popolazione di esami di risonanza magnetica del ginocchio, allora teoricamente qualsiasi anomalia, non importa quanto rara, potrebbe essere rilevata dal modello. Un esempio è mostrato nella Fig 3A di una relativamente rara ma grave rottura completa del tendine gastrocnemio, che è stata correttamente classificata e localizzata come “anormale” dal modello, nonostante non ci fossero altri esempi di questa specifica anomalia nei dati di formazione anormale. E’ possibile che con un approccio binario e dati di formazione “normali” a sufficienza, un modello sia in grado di rilevare qualsiasi anomalia, per quanto non comune. Tuttavia, è necessario più lavoro per esplorare se le anomalie più sottili richiederebbe dati specifici di formazione.
Questo studio ha dei limiti. La nostra convalida ha stabilito la verità di base non è stata governata strettamente dalla conferma chirurgica in tutti i casi. Il modello di apprendimento profondo descritto è stato sviluppato e formato su dati di risonanza magnetica da una grande istituzione accademica. Mentre MRNet ha funzionato bene sul set di validazione esterno senza formazione supplementare (AUC 0.824), abbiamo visto un sostanziale miglioramento (AUC 0.911) dopo la formazione sul set di dati esterno. Questo risultato suggerisce che il raggiungimento di prestazioni ottimali del modello può richiedere un ulteriore sviluppo del modello utilizzando dati più simili a quelli che il modello probabilmente vedrà nella pratica. Sono necessarie ulteriori ricerche per determinare se i modelli formati su set di dati più grandi e multi-istituzionali possono raggiungere prestazioni elevate senza riqualificazione. Il potere di rilevare guadagni statisticamente significativi nelle prestazioni degli esperti clinici con l’assistenza del modello è stato limitato dalle dimensioni del panel, e uno studio più ampio che includa un maggior numero di esperti clinici così come un maggior numero di esami di risonanza magnetica può rilevare guadagni più piccoli nell’utilità. Ciononostante, abbiamo dimostrato che anche in questo piccolo gruppo di esperti clinici, fornendo le previsioni del modello, abbiamo aumentato significativamente la specificità del rilevamento delle lacerazioni ACL, anche dopo aver corretto per i confronti multipli.
In conclusione, abbiamo sviluppato un modello di apprendimento approfondito che raggiunge elevate prestazioni nelle attività di classificazione clinica sulla risonanza magnetica del ginocchio e abbiamo dimostrato il beneficio, in un esperimento retrospettivo, di fornire previsioni modello ai medici durante l’attività di imaging diagnostico. Sono necessari studi futuri per migliorare le prestazioni e la generalizzabilità dei modelli di apprendimento profondo per la RM e per determinare l’effetto dell’assistenza del modello nel contesto clinico.
Informazioni di supporto
References
- Nacey NC, Geeslin MG, Miller GW, Pierce JL. Magnetic resonance imaging of the knee: an overview and update of conventional and state of the art imaging. J Magn Reson Imaging. 2017; 45:1257-75. Publisher Full Text | PubMed
- Naraghi AM, White LM. Imaging of athletic injuries of knee ligaments and menisci: sports imaging series. Radiology. 2016; 281:23-40. Publisher Full Text | PubMed
- Helms CA, Brant WE, Helms CA. Fundamentals of diagnostic radiology. Lippincott Williams & Wilkins: Philadelphia; 2007.
- Oei EH, Nikken JJ, Verstijnen AC, Ginai AZ, Myriam Hunink MG. MR Imaging of the menisci and cruciate ligaments: a systematic review. Radiology. 2003; 226:837-48. Publisher Full Text | PubMed
- Rangger C, Klestil T, Kathrein A, Inderster A, Hamid L. Influence of magnetic resonance imaging on indications for arthroscopy of the knee. Clin Orthop Relat Res. 1996; 330:133-42.
- Cheung LP, Li KC, Hollett MD, Bergman AG, Herfkens RJ. Meniscal tears of the knee: accuracy of detection with fast spin-echo MR imaging and arthroscopic correlation in 293 patients. Radiology. 1997; 203:508-12. Publisher Full Text | PubMed
- Mackenzie R, Palmer CR, Lomas DJ, Dixon AK. Magnetic resonance imaging of the knee: diagnostic performance studies. Clin Radiol. 1996; 51:251-7. PubMed
- McNally EG, Nasser KN, Dawson S, Goh LA. Role of magnetic resonance imaging in the clinical management of the acutely locked knee. Skeletal Radiol. 2002; 31:570-3. Publisher Full Text | PubMed
- Feller JA, Webster KE. Clinical value of magnetic resonance imaging of the knee. ANZ J Surg. 2001; 71:534-7. PubMed
- Elvenes J, Jerome CP, Reikerås O, Johansen O. Magnetic resonance imaging as a screening procedure to avoid arthroscopy for meniscal tears. Arch Orthop Trauma Surg. 2000; 120:14-6. PubMed
- Crawford R, Walley G, Bridgman S, Maffulli N. Magnetic resonance imaging versus arthroscopy in the diagnosis of knee pathology, concentrating on meniscal lesions and ACL tears: a systematic review. Br Med Bull. 2007; 84:5-23. Publisher Full Text | PubMed
- Kim A, Khoury L, Schweitzer M, Jazrawi L, Ishak C, Meislin R. Effect of specialty and experience on the interpretation of knee MRI scans. Bull NYU Hosp Jt Dis. 2008; 66:272-5. PubMed
- Doi K. Computer-aided diagnosis in medical imaging: historical review, current status and future potential. Comput Med Imaging Graph. 2007; 31:198-211. Publisher Full Text | PubMed
- Oakden-Rayner L, Carneiro G, Bessen T, Nascimento JC, Bradley AP, Palmer LJ. Precision radiology: predicting longevity using feature engineering and deep learning methods in a radiomics framework. Sci Rep. 2017; 7:1648. Publisher Full Text | PubMed
- LeCun Y, Bengio Y, Hinton G. Deep learning. Nature. 2015; 521:436-44. Publisher Full Text | PubMed
- Zeiler MD, Fergus R, Fleet D, Pajdla T, Schiele B, Tuytelaars T, Springer. Computer vision—ECCV 2014. Berlin; 2014.
- Esteva A, Kuprel B, Novoa RA, Ko J, Swetter SM, Blau HM. Dermatologist-level classification of skin cancer with deep neural networks. Nature. 2017; 542:115-8. Publisher Full Text | PubMed
- Gulshan V, Peng L, Coram M, Stumpe MC, Wu D, Narayanaswamy A. Development and validation of a deep learning algorithm for detection of diabetic retinopathy in retinal fundus photographs. JAMA. 2016; 316:2402-10. Publisher Full Text | PubMed
- Golan R, Jacob C, Denzinger J. Lung nodule detection in CT images using deep convolutional neural networks. 2016 International Joint Conference on Neural Networks; 2016 Jul 24–29; Vancouver, BC, Canada.
- Litjens G, Kooi T, Bejnordi BE, Setio AA, Ciompi F, Ghafoorian M. A survey on deep learning in medical image analysis. Med Image Anal. 2017; 42:60-88. Publisher Full Text | PubMed
- Prasoon A, Petersen K, Igel C, Lauze F, Dam E, Nielsen M, Mori K, Sakuma I, Sato Y, Barillot C, Navab N. Medical Image Computing and Computer-Assisted Intervention—MICCAI 2013. Springer: Berlin; 2013.
- Liu F, Zhou Z, Samsonov A, Blankenbaker D, Larison W, Kanarek A. Deep learning approach for evaluating knee MR images: achieving high diagnostic performance for cartilage lesion detection. Radiology. 2018; 289:160-9. Publisher Full Text | PubMed
- Štajduhar I, Mamula M, Miletić D, Ünal G. Semi-automated detection of anterior cruciate ligament injury from MRI. Comput Methods Programs Biomed. 2017; 140:151-64. Publisher Full Text | PubMed
- van Rossum G. Python 2.7.10 language reference. Wickford (UK): Samurai Media; 2015.
- Mason D. SU-E-T-33: Pydicom: an open source DICOM library. Med Phys. 2011; 38:3493.
- Nyúl LG, Udupa JK. On standardizing the MR image intensity scale. Magn Reson Med. 1999; 42:1072-81. PubMed
- van Rossum G, Drake FL. Python 3 reference manual. Paramount (CA): CreateSpace; 2009.
- Paszke A, Gross S, Chintala S, Chanan G, Yang E, DeVito Z, et al. Automatic differentiation in PyTorch. 31st Conference on Neural Information Processing Systems; 2017 Dec 4–9; Long Beach, CA, US.
- Deng J, Dong W, Socher R, Li L, Li K, Fei-Fei L. ImageNet: a large-scale hierarchical image database. 2009 IEEE Conference on Computer Vision and Pattern Recognition; 2009 Jun 20–25; Miami, FL, US.
- Zhou B, Khosla A, Lapedriza À, et al. Learning deep features for discriminative localization. 2016 IEEE Conference on Computer Vision and Pattern Recognition; 2016 Jun 26–Jul 1; Las Vegas, NV, US.
- Pedregosa F, Varoquaux G, Gramfort A, Michel V, Thirion B, Grisel O. Scikit-learn: machine learning in Python. J Mach Learn Res. 2011; 12:2825-30.
- Hong SH, Choi JY, Lee GK, Choi JA, Chung HW, Kang HS. Grading of anterior cruciate ligament injury. Diagnostic efficacy of oblique coronal magnetic resonance imaging of the knee. J Comput Assist Tomogr. 2003; 27:814-9. PubMed
- De Smet AA, Tuite MJ. Use of the ‘two-slice-touch’ rule for the MRI diagnosis of meniscal tears. AJR Am J Roentgenol. 2006; 187:911-4. Publisher Full Text | PubMed
- Nguyen JC, De Smet AA, Graf BK, Rosas HG. MR imaging-based diagnosis and classification of meniscal tears. Radiographics. 2014; 34:981-99. Publisher Full Text | PubMed
- Wilson EB. Probable inference, the law of succession, and statistical inference. J Am Stat Assoc. 1927; 22:209-12.
- DeLong ER, DeLong DM, Clarke-Pearson DL. Comparing the areas under two or more correlated receiver operating characteristic curves: a nonparametric approach. Biometrics. 1988; 44:837-45. PubMed
- Sun X, Xu W. Fast implementation of DeLong’s algorithm for comparing the areas under correlated receiver operating characteristic curves. IEEE Signal Process Lett. 2014; 21:1389-93.
- .Publisher Full Text
- Conger AJ. Integration and generalization of kappas for multiple raters. Psychol Bull. 1980; 88:322-8.
- Fleiss JL. Measuring nominal scale agreement among many raters. Psychol Bull. 1971; 76:378-82.
- .Publisher Full Text
- .Publisher Full Text
- Robin X, Turck N, Hainard A, Tiberti N, Lisacek F, Sanchez JC. pROC: an open-source package for R and S+ to analyze and compare ROC curves. BMC Bioinformatics. 2011; 12:77. Publisher Full Text | PubMed
- .Publisher Full Text
Fonte
Bien N, Rajpurkar P, Ball RL, Irvin J, Park A, et al. (2018) Deep-learning-assisted diagnosis for knee magnetic resonance imaging: Development and retrospective validation of MRNet. PLoS Medicine 15(11): e1002699. https://doi.org/10.1371/journal.pmed.1002699





