Abstract
Introduzione
L’ipotesi dell’iper-nutrizione evolutiva suggerisce meccanismi attraverso i quali le condizioni intrauterine legate alla maggiore adiposità materna potrebbero influenzare il rischio di ingrassamento della prole per tutta la vita[1]. L’indice di massa corporea materna (IMC) è positivamente associato ad un maggiore aumento del glucosio circolante, dei lipidi e degli acidi grassi in gravidanza[2,3], e a sua volta livelli gestazionali materni più elevati di questi nutrienti sono associati ad una maggiore dimensione della nascita[4- 6]. A sostegno di questa ipotesi, è stata recentemente dimostrata una forte evidenza di un effetto causale di una maggiore adiposità gestazionale materna e del glucosio circolante a digiuno, ma non dei trigliceridi, sul peso alla nascita e sull’indice ponderale alla nascita, utilizzando un approccio di randomizzazione mendeliana (MR)[7]. Poiché la dimensione della nascita è correlata alla dimensione successiva, è possibile che questi effetti si estendano alla successiva vita postnatale della prole. Oltre a questo effetto di tracciamento proposto, è stato suggerito che l’esposizione intrauterina a livelli più elevati di sostanze nutritive correlate alle adiposità, come il glucosio, provoca cambiamenti permanenti nel controllo dell’appetito della prole, nel funzionamento neuroendocrino o nel metabolismo energetico, che successivamente si traduce in una maggiore adiposità nella vita adulta, indipendentemente da qualsiasi effetto sulle dimensioni della nascita[8- 10]. Ascertare se una maggiore adiposità gestazionale materna si traduce in una maggiore adiposità della prole attraverso effetti intrauterini è importante perché se così fosse, questo meccanismo potrebbe portare ad un’accelerazione dell’epidemia di obesità attraverso le generazioni[11,12] e sottolineerebbe l’importanza di interventi preconcettuali o prenatali nelle donne in età riproduttiva per fermare e invertire l’epidemia di obesità[13,14].
Diversi studi di coorte di grandi dimensioni hanno dimostrato che l’IMC materno pre-gravidanza o precoce è positivamente associato al grasso della prole, misurato con l’IMC, la circonferenza della vita, o valutazioni più dirette della massa grassa, attraverso l’intera distribuzione dell’IMC materno[1,15- 18]. Tuttavia, a causa dell’elevata ereditarietà dell’adiposità e delle caratteristiche ambientali e comportamentali condivise tra le madri e la loro prole, è impossibile determinare specifici effetti intrauterini da tali studi[1,19,20]. Un disegno di RM intergenerazionale in cui le varianti genetiche materne sono utilizzate come variabili strumentali (IV) per le esposizioni intrauterine modificabili dal punto di vista ambientale, come l’esposizione a una maggiore adiposità materna, può essere utile per fornire indicazioni sull’effetto causale di queste esposizioni sui risultati successivi della prole(Fig 1)[21]. A nostra conoscenza, questo approccio è stato utilizzato solo una volta per esaminare l’effetto causale dell’IMC materno sull’adiposità della prole nell’infanzia[22]. In quello studio, una variante del gene FTO è stata utilizzata come IV per l’IMC pre-gravidanza. I risultati hanno suggerito nessun effetto causale di BMI materno gestazionale sulla prole a doppia energia a raggi X assorbitori di energia (DXA)-massa grassa determinata all’età di 10 y una volta genotipo della prole è stato preso in considerazione. Tuttavia, l’IC per la stima IV era molto ampia, e non poteva essere statisticamente distinta dall’associazione positiva multivariabile dell’IMC materno gestazionale con la massa grassa della prole. Quello studio MR e, in effetti, la maggior parte degli studi di associazione multivariabile convenzionale hanno esaminato le associazioni solo nell’infanzia o nella prima infanzia piuttosto che nell’età adulta[1]. Esaminare le associazioni in età più avanzata è importante perché il potenziale di questo meccanismo per accelerare l’epidemia di obesità si riferisce all’adiposità gestazionale materna, influenzando l’adiposità delle loro figlie durante gli anni riproduttivi, in modo tale che esse entrano in gravidanza un po’ più grasse e influenzano la generazione successiva e così via attraverso le generazioni [1].
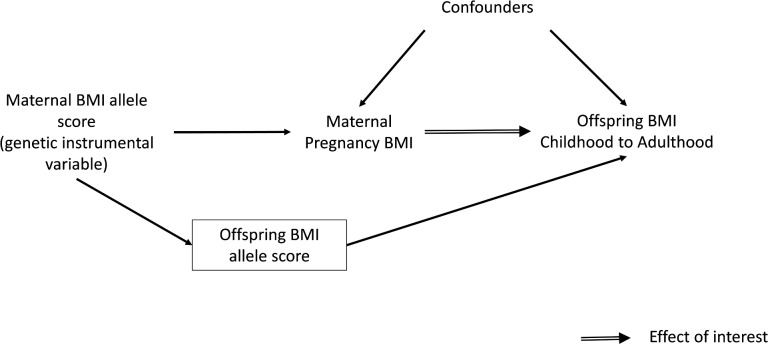
Fig. 1.Fig 1. Analisi MR intergenerazionale per indagare un effetto causale intrauterino dell’IMC materno sull’adiposità della prole.
Il nostro obiettivo era quello di utilizzare la risonanza magnetica per indagare l’effetto causale dell’esposizione intrauterina a un maggiore IMC materno pre/pre-gravidanza sulla prole e sulla massa grassa, utilizzando misurazioni ripetute dall’infanzia alla prima età adulta. L’uso di un punteggio allelico, generato da un gran numero di varianti genetiche dell’IMC identificate in studi di associazione a livello genomico (GWAS), può agire come uno strumento genetico più forte di una singola variante[23,24]. Inoltre, abbiamo cercato di replicare i risultati in uno studio indipendente per esplorare se i risultati potrebbero essere dovuti al caso e, se i risultati sono stati replicati, per aumentare ulteriormente la potenza dello studio mettendo insieme i risultati dei due studi.

Fig. 1.Fig 1. Analisi MR intergenerazionale per indagare un effetto causale intrauterino dell’IMC materno sull’adiposità della prole.
Metodi
Coorte e selezione dei partecipanti
Abbiamo usato i dati delle coppie madre-figlia che hanno partecipato allo Studio Avon Longitudinale dei Genitori e dei Figli (ALSPAC) [25,26] nell’analisi principale e dallo Studio di Generazione R [27]nell’analisi di replicazione. In entrambi gli studi, sono state incluse solo le nascite singolari a causa dei modelli di crescita intrauterina notevolmente diversi tra le nascite singolari e quelle multiple. In ALSPAC, erano disponibili dati sulla massa grassa della prole BMI- e DXA-determinata in età multipla tra 7 e 18 anni(n = 2.521-3.720 coppie madre-figlia per diverse età), mentre nella Generazione R (n =2.337), era disponibile la massa grassa della prole BMI a circa 6 anni di età.
ALSPAC
L’ALSPAC è uno studio di coorte prospettica basato sulla popolazione che ha iscritto 14.541 donne incinte residenti nella ex Contea di Avon, Regno Unito, con una data di parto prevista tra il 1° aprile 1991 e il 31 dicembre 1992[26,27]. Il sito web dello studio contiene i dettagli di tutti i dati disponibili attraverso un dizionario di dati completamente ricercabile(http://www.bristol.ac.uk/alspac/researchers/data-access/data-dictionary/). L’approvazione etica dello studio è stata ottenuta dal Comitato etico e giuridico dell’ALSPAC e dai Comitati locali di etica della ricerca. Dei 13.678 figli nati vivi in ALSPAC, i dati sul genotipo erano disponibili per 5.206 coppie madre-figlia. Per l’analisi principale, l’IMC pre-gravidanza dichiarato è stato disponibile per 4.629 delle coppie madre-figlia incluse, e di queste, 3.720 misure dell’IMC della prole sono state ottenute in una clinica quando la prole aveva un’età media di 7,5 anni (chiamata valutazione a 7 anni). In ALSPAC, le misure di adiposità sono state valutate in altre cinque cliniche di follow-up. Queste cliniche hanno avuto luogo quando la prole aveva un’età media di 9,8, 11,7, 13,8, 15,4 e 17,8 anni (d’ora in poi indicati come 10, 12, 14, 16 e 18 anni). Il numero di individui idonei in queste cliniche successive per l’inclusione in questo studio variava da 3.496 all’età di 10 anni a 2.521 all’età di 18 anni a causa della perdita del follow-up negli ultimi tempi.
Generazione R
Lo studio Generation R è uno studio prospettico di coorte multietnica basato sulla popolazione a partire dalla prima gravidanza, con sede a Rotterdam, Paesi Bassi[27]. Tutte le donne incinte che vivono nell’area dello studio con una data di parto compresa tra l’aprile 2002 e il gennaio 2006 erano idonee all’iscrizione. Il protocollo di studio è stato approvato dal Comitato etico medico del Centro medico Erasmus di Rotterdam. La coorte comprende 9.778 madri e i loro figli (9.749 bambini nati vivi). Di queste, 8.880 (91%) donne sono state iscritte durante la gravidanza e 8.633 hanno partorito da sole. I dati sul genotipo erano disponibili per 3.909 coppie madre-figlia. Per l’analisi principale, l’IMC pre-gravidanza auto-riferito era disponibile per 3.199 delle coppie madre/figlia incluse e, di queste, erano disponibili 2.337 misure dell’IMC della prole in una clinica a 6 anni (età media della prole 6,2 anni).
Antropometria
Il peso e l’altezza pre-gravidanza autodichiarati sono stati ottenuti per le madri di entrambe le coorti durante la gravidanza e utilizzati come esposizione principale. In entrambi gli studi, il peso registrato alla prima visita in clinica prenatale era molto correlato con il loro rapporto materno di peso pre-gravidanza al momento del reclutamento (coefficienti di correlazione = 0,96). Per ogni studio, l’età (in categorie di 1-y) z-score per l’IMC materno sono stati ricavati utilizzando la standardizzazione interna. Nell’ALSPAC e nella generazione R, l’altezza e il peso della prole sono stati misurati nelle cliniche di ricerca e utilizzati per calcolare l’IMC della prole. Per ogni studio, sono stati calcolati i punti z per il sesso e l’età standardizzati internamente (nelle categorie mensili) per l’IMC della prole e (solo in ALSPAC) l’indice di massa grassa (FMI) determinato dalla DXA.
ALSPAC
Dopo l’iscrizione, alla madre è stato chiesto di indicare la sua altezza e il suo peso pre-gravidanza in un questionario somministrato a 12 settimane di gestazione, dal quale è stato calcolato l’IMC pre-gravidanza (come peso [in chilogrammi] diviso per l’altezza [in metri)] al quadrato). La correlazione tra il peso di pre-gravidanza ottenuto tramite questionario e il peso misurato alla prima visita prenatale (10-12 settimane di gestazione) è stata di 0,96. I partner delle donne (padri dei bambini) hanno riportato la propria altezza e il proprio peso in questionari compilati contemporaneamente alle madri; questi sono stati utilizzati per determinare il loro IMC.
Dall’età di 7 anni in poi, i figli sono stati invitati in cliniche di follow-up dove è stata misurata l’antropometria. Il peso e l’altezza sono stati misurati con il bambino in abiti leggeri e senza scarpe. Il peso è stato misurato con un analizzatore di grasso corporeo Tanita (modello TBF 305; Tanita UK Limited, Viewsley, UK) e l’altezza con un Harpenden Stadiometer (Holtain Limited, Dyfed, UK) con un’approssimazione di 0,1 cm. L’IMC (kg/m2) è stato quindi calcolato come peso (in chilogrammi) diviso per l’altezza (in metri) al quadrato. L’età del bambino quando ha frequentato la clinica di ricerca è stata registrata in mesi.
A partire dalla clinica a 9 anni, la massa grassa totale (kg) e la massa magra totale (kg) sono state valutate con l’uso di uno scanner DXA Lunar Prodigy DXA (GE Medical Systems Lunar, Madison, WI). Le scansioni sono state ispezionate visivamente e, se necessario, riallineate. Una volta completata, il tester ha esaminato la scansione per garantirne la qualità e, se necessario, ha ripetuto la scansione. L’FMI è stato successivamente calcolato come massa grassa totale (in chilogrammi) divisa per l’altezza (in metri) al quadrato.
Generazione R
Le informazioni sul peso materno appena prima della gravidanza sono state ottenute tramite questionario, e l’altezza è stata misurata senza scarpe e abbigliamento pesante all’atto dell’iscrizione, da cui è stato calcolato l’IMC (kg/m2). La correlazione tra il peso pre-gravidanza ottenuto tramite questionario e il peso misurato alla prima visita prenatale è stata di 0,96. Anche l’altezza (cm) e il peso del partner (kg) sono stati misurati al momento dell’iscrizione.
A partire dai 5 anni i bambini sono stati invitati a partecipare a una valutazione pratica di antropometria. La loro altezza e il loro peso senza scarpe e vestiti pesanti sono stati misurati e utilizzati per calcolare l’IMC. Il peso (kg) è stato misurato con una bilancia meccanica personale (SECA, Almere, Paesi Bassi) e l’altezza (m) è stata determinata in posizione eretta con uno stadiometro Harpenden (Holtain Limited, Dyfed, Regno Unito). L’età del bambino quando ha frequentato la clinica di ricerca è stata registrata in mesi.
Genotipizzazione
Le procedure di genotipizzazione e i dettagli degli SNP BMI ottenuti dalla genotipizzazione per le madri e la prole nelle due coorti sono descritti di seguito.
ALSPAC
Le madri ALSPAC sono state genotipizzate utilizzando il chip Illumina 660K quad SNP (Illumina Inc., San Diego, CA, USA) presso il Centre National de Genotypage, Parigi. Il software PLINK[28] (v1.07) è stato utilizzato per effettuare misure di controllo qualità (QC).Sono stati esclusi gli individui con assegnazioni sessuali errate, eterozigosi insolite del genoma o del cromosoma X, livelli sproporzionati di missingness individuale (>5%), prove di parentela criptica (>12,5% identità per discendenza [IBD]), o quelli di ascendenza non europea determinata da analisi di scala multidimensionale seminata con individui del progetto internazionale HapMap [29]. Il set di dati risultante era costituito da 8340 individui[30].
Sono stati eliminati gli SNP con una frequenza di allele minore (MAF) di <1%, un tasso di chiamata di <95%, o quelli non in equilibrio Hardy-Weinberg (HWE, p < 1 x 10-6 )[30]. L’imputazione dei dati direttamente genotipizzati è stata condotta con il software MaCH (v1.0.16) Markov Chain Haplotyping[31,32] utilizzando gli individui CEU di HapMap fase 2 (release 22) come set di riferimento per l’imputazione autosomica[29].
I discendenti di ALSPAC sono stati genotipizzati utilizzando la piattaforma di genotipizzazione SNP SNP a livello genomico quadruplo di Illumina HumanHap550 (Illumina Inc., San Diego, CA, USA) dal Wellcome Trust Sanger Institute (Cambridge, Regno Unito) e dalla Laboratory Corporation of America (Burlington, NC, USA). È stata effettuata una procedura di QC simile a quella eseguita nelle madri ALSPAC. Sono stati esclusi gli individui con assegnazioni sessuali errate, estrema eterozigosi (<0,320 e >0,345 per i dati del Wellcome Trust Sanger Institute e <0,310 e >0,330 per i dati del LabCorp), livelli sproporzionati di missingness individuale (>3%), prove di parentela criptica (>10% IBD) o di ascendenza non europea. Il set di dati risultante era composto da 8.365 individui. [33,34]
Sono stati eliminati gli SNP con un MAF di <1%, un tasso di chiamata di <95%, o quelli non in HWE (p < 5x 10-7) [33,34]. L’imputazione dei dati direttamente genotipizzati è stata effettuata allo stesso modo delle madri.
Le varianti dell’IMC stabilito[35,36] nelle madri e nella prole ALSPAC sono state estratte dal set di dati imputati utilizzando gli individui HapMap come set di riferimento (in cui erano presenti tutti i genotipi per l’IMC).
Generazione R
I dati a livello genomico non sono attualmente disponibili per le madri nella Generazione R, ma il DNA è disponibile per la genotipizzazione in studi su geni candidati o di replicazione. Pertanto, la genotipizzazione personalizzata di 32 SNP identificati in una meta-analisi GWAS su larga scala per il BMI[35] è stata effettuata da LGC Genomics (ex Kbiosciences) utilizzando un test di discriminazione allelica Taqman (Applied Biosystems, Foster City, CA) e Abgene QPCR ROX mix (Abgene, Amburgo, Germania) con un tasso di chiamata di 99.3%, una doppia concordanza del 99,8% e tutti gli SNP considerati trovati in HWE, ad eccezione di rs4836133(ZNF608; p = 3 x 10-15). Per confermare l’accuratezza dei risultati della genotipizzazione, 276 campioni selezionati a caso sono stati genotipizzati per una seconda volta con lo stesso metodo, con un tasso di errore <1%.
La genotipizzazione a livello genomico nella prole è stata eseguita utilizzando i chip Illumina HumanHap 610 o 660 Quad (Illumina Inc., San Diego, USA) a seconda del tempo di raccolta del DNA[37,38]. Il QC del genotipo e il processo di imputazione è stato eseguito nello studio come descritto in precedenza[38,39]. In breve, sono stati esclusi gli individui con rilevamento di duplicati, bassi tassi di chiamata (<97,5%), disadattamento del sesso e alta eterozigosi (>4 deviazioni standard [SDs]), esono stati esclusi gli SNP con bassi tassi di chiamata (<98,0%), non in HWE (p < 1 x 10-6), con un basso MAF(< 0,1%) o quelli con missingness differenziale tra i due chip (p < 1 x 10-7).
La composizione etnica del campione è stata stimata mediante l’analisi Identity-By-State utilizzando l’analisi dei componenti principali (PCA) seminata con l’International HapMap Phase 2 release 22 individui[29]. I partecipanti sono stati definiti di origine non europea quando hanno deviato più di 4 SD dal valore medio del panel CEU in uno dei primi quattro componenti principali. Le relazioni familiari criptiche sono state identificate attraverso l’analisi dell’IBD.
L’imputazione MaCH[32] dei dati genetici della discendenza è stata effettuata utilizzando sia i pannelli di riferimento HapMap Fase 2 release 22[29] che 1000 Genomi Fase 3[40], utilizzando tutti gli aplotipi disponibili delle diverse popolazioni in un approccio “cosmopolita” [38]. Le varianti BMI stabilite[35] sono state estratte dal set di dati imputati 1000 Genomi a causa della mancata disponibilità di tre degli SNP nella serie di dati HapMap fase 2.
Dai dati del genotipo materno e della prole sono stati generati punteggi allelici ponderati dell’IMC utilizzando 32 varianti indipendenti che hanno dimostrato di essere associate in modo affidabile all’IMC in entrambe le coorti[41]. In ALSPAC, è stato anche possibile generare un punteggio allelico aggiuntivo composto da 97 varianti indipendenti associate al BMI in un GWAS più recente[36], in quanto ALSPAC ha dati completi a livello genomico sia sulle madri che sulla prole. I punteggi allelici sono stati ricavati utilizzando la dose dell’effetto (BMI-increscente) dell’allele in ogni SNP, che è stato prima ponderato dalla dimensione dell’effetto della variante in GWAS[36,41] e poi sommato:WeightedBMIscore=w1×SNP1+w2×SNP2+⋯wn×SNPndove w è il peso (cioè il coefficiente beta di associazione dell’SNP con l’IMC dal GWAS pubblicato) e SNP è il dosaggio degli alleli che aumentano l’IMC in quel locus (cioè 0, 1, o 2 alleli che aumentano l’IMC). I pesi utilizzati sono riportati nella tabella S1. Il punteggio è stato poi ridimensionato per riflettere il numero medio di alleli che aumentano l’IMC trasportati da un individuo utilizzando la formula descritta in Lin et al.[42]:RescaledweightedBMIscore=Weightedscore×NumberofSNPsavailableSumofweightsovailableSNPs
Altre variabili
La classe sociale e l’educazione dei genitori, il fumo materno durante la gravidanza, la parità e l’IMC paterno sono stati considerati come potenziali fattori di confusione nelle analisi di regressione multivariabile. L’etnia è stata considerata anche come potenziale cofondatore nella Generazione R, una coorte multietnica (nell’ALSPAC, il 95% dei partecipanti era di origine bianca europea). I dettagli di come ciascuno di questi confonditori è stato valutato nelle due coorti sono descritti di seguito.
ALSPAC
La parità, definita come il numero di gravidanze precedenti che hanno portato a un parto vivo o morto, è stata registrata in un questionario compilato a 18 settimane di gestazione. In un questionario compilato a 32 settimane di gestazione, le madri hanno registrato l’occupazione e l’educazione di se stesse e dei loro partner. L’occupazione più alta della madre o del partner è stata utilizzata per assegnare i gruppi di classe sociale della famiglia (classi I [professioni professionali], II [professioni manageriali e tecniche], III non manuali [professioni non manuali qualificate], III manuali [professioni manuali qualificate], IV [professioni parzialmente qualificate], V [professioni non qualificate]) utilizzando la classificazione dell’Office of Population Censuses and Surveys 1991 del British Office of Population. I titoli di studio più alti della madre e del padre sono stati trattati come variabili separate, e ciascuno di essi è stato suddiviso in una delle quattro categorie: istruzione fino a 16 anni con formazione professionale o certificato di istruzione secondaria, istruzione fino a 16 anni con certificazione generale dell’istruzione (livello ordinario), istruzione fino a 18 anni con certificato generale dell’istruzione (livello avanzato), e diploma universitario. Le informazioni sullo stato di salute delle madri durante la gravidanza sono state ottenute tramite questionari somministrati a 18 e 32 settimane di gestazione. I dati sono stati utilizzati per generare una variabile categorica: mai fumato durante la gravidanza, fumato solo all’inizio della gravidanza e fumato durante tutta la gravidanza. L’età materna al momento del parto è stata derivata dalla data di nascita della madre, che è stata registrata al momento del reclutamento, e dalla data di nascita della sua prole. Il sesso della prole è stato registrato in sala parto ed estratto dai registri ostetrici e/o dalle notifiche di nascita.
Generazione R
Le informazioni sulla parità della madre, definita come il numero di volte che la donna ha partorito un feto con un’età gestazionale di 24 o più settimane, sono state ottenute tramite questionario all’atto dell’iscrizione. Le informazioni sul reddito familiare (euro/mese) sono state ottenute da un questionario somministrato durante la gravidanza. Il livello di istruzione più alto completato (scuola primaria, scuola secondaria, istruzione superiore) sia per le madri che per i padri è stato ottenuto da un questionario somministrato al momento dell’iscrizione. Le informazioni sul fumo materno in gravidanza sono state valutate tramite questionario in ogni trimestre e sono state classificate in mai fumato durante la gravidanza, fumato solo all’inizio della gravidanza e fumato durante tutta la gravidanza. L’età materna è stata ottenuta nel questionario somministrato al momento dell’iscrizione. Utilizzando questo e le informazioni sull’età gestazionale del feto all’iscrizione e sull’età gestazionale alla nascita, è stata ricavata l’età della madre al momento del parto. Il sesso della prole è stato ottenuto dai registri dell’ostetrica e dell’ospedale al momento della nascita.
Analisi statistica
Abbiamo esaminato le associazioni di regressione multivariabile e gli effetti MR di BMI materno pre-gravidanza BMI sulla prole BMI a 6-7 anni nelle due coorti e sia BMI e DXA massa grassa a tutte le età con i dati disponibili fino all’età di 18 anni in ALSPAC. Non siamo stati in grado di trovare qualsiasi altro studio con dati rilevanti sulle varianti genetiche materne e della prole, l’IMC materno pre- o precoce gravidanza, e l’IMC della prole o la massa grassa nell’adolescenza / prima età adulta e quindi non abbiamo potuto esplorare la replicazione dei risultati ALSPAC in età più avanzata. I risultati sono presentati nei maschi e nelle femmine combinati come stime puntiformi e sembravano molto simili in entrambi i sessi, e non c’era una forte evidenza statistica per una interazione per sesso sia nelle analisi multivariabili o MR (tutti i valori p> 0,07).
Abbiamo seguito un piano di analisi, che è stato scritto prima di analizzare i dati per le analisi principali, mentre alcune analisi di sensibilità sono state effettuate post hoc(Appendice S1). Tutte le analisi sono state effettuate utilizzando Stata (Stata Corp, TX, US), versione 13.
Analisi dell’effetto dell’IMC materno pre-gravidanza sull’adiposità della prole attraverso l’infanzia alla giovane età adulta in ALSPAC
La regressione lineare multivariabile è stata eseguita per esaminare l’associazione dell’IMC materno pre-gravidanza con l’IMC della prole dai 7 ai 18 anni e l’IMC della prole dai 10 ai 18 anni. Nel primo modello, l’età materna, l’età della prole, e il sesso sono stati controllati nella esposizione standardizzata e il risultato. Nel secondo modello, abbiamo inoltre regolato per potenziale confusione da parte della classe sociale dei genitori, l’educazione dei genitori, il fumo materno durante la gravidanza, la parità, e l’IMC paterno.
L’analisi MR è stata prima effettuata utilizzando il punteggio allelico materno ponderato BMI composto da 32 SNPs come IV per il suo BMI pre-pre-gravidanza per valutare il suo effetto causale sulla prole BMI (dai 7 ai 18 anni) e FMI della prole (dai 10 ai 18 anni). Entrambi gli approcci multivariabile e IV hanno esaminato la stessa relazione, vale a dire, la variazione di SD nel risultato per 1 aumento di SD nell’IMC materno pre/pre-gravidanza precoce.
Per l’approccio MR abbiamo usato l’analisi dei minimi quadrati a due stadi (TSLS) IV. Abbiamo assunto un modello genetico additivo come supportato dal GWAS originale[41]. La forza della IV è stata valutata esaminando le statistiche R2 e F dalla regressione del primo stadio per ogni analisi[43]. L’età materna, l’età della prole e il sesso sono stati controllati in tutte le analisi di risonanza magnetica con l’uso di misure di esposizione e di risultato standardizzate per queste caratteristiche. Nonostante l’evidenza che lo stile di vita e le caratteristiche socioeconomiche che comunemente confondono gli studi osservazionali sono distribuite in modo casuale rispetto al genotipo[44], abbiamo testato questa ipotesi nel nostro studio esaminando le associazioni tra il punteggio allelico e i potenziali confonditori dell’associazione osservazionale[45].
Per ottenere una stima causale dell’influenza intrauterina dell’IMC materno sull’IMC della progenie, è importante adeguarsi al genotipo della progenie per escludere la possibilità di un altro percorso indipendente tra lo strumento genetico e l’esito, cioè attraverso la trasmissione genetica dalla madre alla progenie(Fig 1). Questo risultato è stato ottenuto aggiungendo il punteggio dell’allele BMI ponderato della prole ai modelli IV. Un test z-test è stato utilizzato per verificare la differenza tra questa analisi MR IV e l’analisi di regressione multivariabile corretta per il cofondatore, con l’evidenza che la differenza tra i due è indicativa della possibilità di confusione non osservata nell’analisi multivariabile. La statistica z è stata calcolata stimando la covarianza tra la regressione multivariabile e le stime IV utilizzando una procedura di bootstrapping.
Abbiamo generato un punteggio allelico addizionale ponderato da 97 varianti genetiche, usando pesi specifici SNP presi dalla recente meta-analisi GWAS in cui sono stati tutti identificati[36], al fine di esplorare se i nostri risultati principali sono stati coerenti quando abbiamo usato questo punteggio, con una potenza potenzialmente maggiore.
Replicazione in una coorte indipendente e meta-analisi
Abbiamo intrapreso le analisi di regressione multivariabile e di MR IV nello studio di replicazione Generation R utilizzando gli stessi metodi descritti sopra per ALSPAC, con l’ulteriore aggiustamento per l’etnia, includendo le prime 20 componenti principali ottenute dai dati del genotipo della prole come covariate in entrambe le analisi. Siamo stati in grado di completare le analisi IV utilizzando solo il punteggio allelico ponderato 32-SNP in questa coorte.
Abbiamo usato una meta-analisi a effetti fissi per combinare i risultati di regressione multivariabile di ALSPAC e Generazione R e abbiamo usato il test Q di Cochran e la statistica I2 per esplorare l’eterogeneità tra i risultati di queste due coorti [46]. Abbiamo adottato un approccio simile per mettere in comune i risultati della MR IV. Abbiamo fatto questo mettendo in comune i risultati delle due fasi delle analisi IV separatamente: (i) il punteggio allelico materno (con e senza aggiustamento per il punteggio allelico della prole) associazione con l’IMC materno (prima regressione) e (ii) il punteggio allelico materno (con e senza aggiustamento per il punteggio allelico della prole) associazione con l’IMC della prole a 6 anni (seconda regressione). Abbiamo poi combinato queste due stime messe in comune per ottenere l’effetto causale IV MR utilizzando la stima del rapporto, cioè i risultati messi in comune di (ii) ÷ i risultati messi in comune di (i) [47]. Gli errori standard di queste stime sono stati calcolati utilizzando un’approssimazione della serie di Taylor[48]. Abbiamo messo in comune i risultati utilizzando i punteggi di 32-SNP generati in entrambi gli studi e anche i risultati utilizzando il punteggio di 97-SNP in ALSPAC con il punteggio di 32-SNP nella Generazione R. Abbiamo confrontato i risultati MR IV messi in comune con il punteggio allele-regolato dei figli con le stime multivariabili messe in comune con i confounder-regolati con un test z e il bootstrapping, come descritto sopra per ALSPAC.
Per fornire risultati più interpretabili per uso clinico e di salute pubblica, abbiamo convertito i risultati sulla scala SD in unità BMI moltiplicandole per un valore rappresentativo della SD di BMI pre-gravidanza (3,7 kg/m2) e BMI della prole (2,0 kg/m2) preso dal Discovery and largest study (ALSPAC).
Analisi della sensibilità
Nell’analisi MR, è necessario regolare il punteggio dell’allele BMI della prole per separare l’influenza dell’ereditarietà genetica dall’effetto intrauterino dell’adiposità materna durante la gravidanza. Tuttavia, come è stato evidenziato in una risposta al precedente documento in cui è stato utilizzato questo metodo[49], l’aggiustamento può introdurre una forma di polarizzazione nota come polarizzazione del collisore nella stima dell’associazione esposizione al risultato. Questo perché aggiustando il punteggio dell’allele della prole nelle analisi MR IV, possiamo indurre un’associazione attraverso varianti genetiche paterne (su cui non abbiamo dati; S1 Fig). Spieghiamo questa possibilità in modo più dettagliato nell’appendice(Appendice S1) e descriviamo anche come abbiamo intrapreso studi di simulazione per esplorare la probabilità di questa distorsione dei nostri risultati principali con l’aggiustamento del punteggio allelico della prole. Inoltre, un metodo alternativo per evitare possibili distorsioni da collisione è quello di utilizzare solo gli alleli materni non trasmessi[50]. Abbiamo esplorato l’uso di questo approccio all’interno di ALSPAC e presentiamo i risultati nell’appendice(Appendice S1) solo data la potenza statistica relativamente bassa associata a questo metodo.
Un alto grado di eterogeneità tra le stime causali dei singoli SNP che compongono i punteggi allelici potrebbe indicare una violazione dell’ipotesi di MR che non vi sia pleiotropia. Abbiamo quindi eseguito una meta-analisi a variazione inversa ponderata (IVW) delle stime degli SNP individuali sia nell’ALSPAC che nella Generazione R e abbiamo calcolato la statistica Q e I2 di Cochran per stimare il grado di eterogeneità nella meta-analisi degli effetti fissi [51]. Abbiamo anche studiato il potenziale bias dovuto alla pleiotropia eseguendo la regressione MR Egger[52] per il modello regolato per il punteggio allelico della prole. L’intercetta in questa analisi fornisce un test per la pleiotropia direzionale complessiva, e il coefficiente fornisce una stima causale valida in presenza di pleiotropia. I risultati sono stati ottenuti sia per ALSPAC (utilizzando entrambi i set di 32 SNPs e 97 SNPs) che per la Generazione R (utilizzando il set di 32 SNPs). Le intercettazioni e le pendenze sono state meta-analizzate e confrontate con quelle ottenute utilizzando IVW dei singoli SNP. L’analisi è stata effettuata utilizzando il pacchetto mrrobust Stata[53].
Infine, abbiamo indagato la possibile non linearità dell’associazione sia nell’analisi di regressione multivariabile che in quella di MR, sovrapponendo un loess non parametrico più liscio e una linea di miglior adattamento su un grafico residuo parziale aumentato.
Risultati
Latabella 1 mostra le caratteristiche chiave della coorte di Discovery e Replication.
| ALSPAC (Scoperta) | Generazione R (Replica) | Generazione R (europei)* | |
|---|---|---|---|
| n | 3,720 | 2,337 | 1,280 |
| Maschi (%) | 48.6% | 49.6% | 49.4% |
| Età dei figli in mesi (SD) | 89.6 (1.9) | 74.3 (6.1) | 73.2 (4.6) |
| Peso alla nascita della prole in gs (SD) | 3,465 (511) | 3,479 (507) | 3,561 (506) |
| Peso della prole in kg (SD) | 25.6 (4.4) | 23.2 (4.1) | 22.8 (3.4) |
| Altezza della prole in cm (SD) | 125.7 (5.4) | 119.5 (5.9) | 119.5 (5.4) |
| IMC della prole in kg/m2 (SD) | 16.2 (2.0) | 16.2 (1.8) | 15.9 (1.4) |
| IMC materno in kg/m2 (SD) | 22.9 (3.7) | 23.5 (4.1) | 23.2 (3.8) |
Analisi dall’infanzia alla prima età adulta in ALSPAC
La dimensione del campione ad ogni età da 7 a 18 anni è diminuita a seguito della perdita di follow-up, ma la proporzione di maschi e femmine e la distribuzione del peso alla nascita (di quelli rimasti nella coorte) erano simili in ogni gruppo di età(Tabella S2). Altezza, peso, IMC, BMI, massa grassa, FMI e massa magra sono aumentati con l’aumentare dell’età da 7 a 18 anni come previsto(Tabella S2).
L’IMC materno è stato associato a caratteristiche che a priori abbiamo considerato come probabili confonditori(Tabella S3). Con una sola eccezione, il punteggio dell’allele dell’IMC materno non è stato associato a questi fattori di confusione. C’è stata una debole associazione inversa con l’educazione paterna (suggerendo che in media ogni aumento di categoria dell’educazione paterna è stato associato a un cambiamento di SD nel punteggio allelico ponderato [S3Table]) di -0,03 [95% CI -0,07-0,00]. Il punteggio allelico materno dell’IMC è stato normalmente distribuito ed è stato fortemente associato con l’IMC materno pre-gravidanza, spiegando il 2,2% della variazione dell’IMC(Tabella S4), e con le statistiche F di primo stadio per ciascuna delle analisi MR, tutte >45 (Tabella S5).
LaTabella 2 mostra le associazioni multivariabili confounder-adjusted delle associazioni multivariabili di IMC materno pre-gravidanza con la prole IMC e IMC FMI a ogni età e gli equivalenti risultati MR (con aggiustamento per i punteggi allele della prole). Le tabelle S6 e S7 mostrano risultati più dettagliati, compresi i risultati multivariabili non corretti per i punteggi allelici della prole e i risultati MR. Nella regressione multivariabile confounder-adjusted, un IMC materno di 1 SD (equivalente a 3,7 kg/m2) più alto aggiustato per l’età è stato associato a un IMC materno di 0,25 (0,21, 0,29) SD più alto per la prole a 7 anni e a 0,33 SD più alto per la prole a 18 anni (equivalente a 0,56 e 0,76 kg/m2, rispettivamente; Tabella 2). I risultati equivalenti per le analisi genetiche IV con aggiustamento per il punteggio dell’allele della prole sono stati 0,04 (-0,21, 0,30) SD a 7 anni e -0,03 (-0,32, 0,26) SD a 18 anni(Tabella 2). I risultati per l’IMC dai 10 ai 18 anni sono stati simili sia per la regressione multivariabile che per i risultati di RM a quelli visti con l’IMC(Tabella 2). Abbiamo inoltre valutato, in modo post hoc, l’impatto di un ulteriore aggiustamento per il punteggio dell’allele materno e della prole nei modelli di regressione multivariabile e abbiamo trovato che questi aggiustamenti aggiuntivi non hanno sostanzialmente modificato le stime dei punti per la differenza media(Tabella S14).
| Risultato della prole | Confoundera-risultati di regressione multivariabile regolati | Risultati MR (punteggio allelico materno genetico IV adattato per il punteggio allelico della prole) | p-differenzab | ||
|---|---|---|---|---|---|
| n | Differenza nel risultato medio della prole (SD) per 1SD aumento dell’IMC materno (95%CI) | n | Differenza nel risultato medio della prole (SD) per 1SD aumento dell’IMC materno (95%CI) | ||
| IMC età 7 anni | 2,565 | 0.25 (0.21–0.29) | 3,720 | 0.04 (-0.21–0.30) | 0.13 |
| IMC età 10 | 2,507 | 0.31 (0.27–0.35) | 3,657 | 0.03 (-0.23–0.29) | 0.03 |
| IMC età 12 anni | 2,411 | 0.32 (0.29–0.36) | 3,496 | 0.00 (-0.26–0.26) | 0.02 |
| IMC età 14 anni | 2,254 | 0.32 (0.28–0.36) | 3,227 | -0.07 (-0.34–0.20) | 0.01 |
| IMC età 16 anni | 1,979 | 0.34 (0.30–0.39) | 2,806 | -0.10 (-0.41–0.20) | 0.003 |
| IMC età 18 anni | 1,798 | 0.33 (0.28–0.37) | 2,521 | -0.03 (-0.32–0.26) | 0.01 |
| Età FMI 10 | 2,413 | 0.30 (0.26–0.33) | 3,495 | 0.13 (-0.13–0.39) | 0.221 |
| FMI età 12 anni | 2,375 | 0.31 (0.27–0.35) | 3,444 | 0.04 (-0.22–0.30) | 0.053 |
| Età FMI 14 anni | 2,233 | 0.30 (0.26–0.34) | 3,192 | 0.03 (-0.23–0.29) | 0.043 |
| FMI età 16 anni | 1,927 | 0.33 (0.29–0.38) | 2,715 | -0.10 (-0.40–0.21) | 0.001 |
| Età FMI 18 anni | 1,739 | 0.32 (0.27–0.37) | 2,430 | 0.03 (-0.27–0.32) | 0.033 |
C’era una forte evidenza statistica che i risultati dell’analisi MR IV differivano dai risultati dell’analisi di regressione multivariabile, con l’eccezione della progenie BMI a 7 anni e FMI a 10 anni, dove le associazioni IV avevano stime puntuali che erano più piccole dei risultati dell’analisi di regressione multivariabile, ma erano statisticamente coerenti con quei risultati(Tabella 2). I risultati sono stati simili quando il punteggio dell’allele 97-SNP BMI è stato utilizzato come IV( tabelleS8 e S9 ).
Replicazione e meta-analisi con una Coorte Indipendente Aggiuntiva
L’IMC materno è stato associato a potenziali confonditori nella Generazione R(Tabella S10). Il punteggio allelico materno dell’IMC è stato normalmente distribuito con media e SD simili a quelli dell’ALSPAC ed è stato fortemente associato all’IMC materno pre-gravidanza(Tabella S4). Simile ad ALSPAC, il punteggio allelico dell’IMC materno era debolmente inversamente correlato all’educazione paterna nella Generazione R, ma non ad altri confonditori osservati(Tabella S10).
I risultati aggiustato per età e sesso erano simili a quelli dell’ALSPAC, anche se c’era evidenza di eterogeneità nei risultati di regressione multivariabile aggiustato per confondere (I2 = 82%; S2 Fig). I risultati di entrambi gli studi hanno mostrato che un aumento di 1 SD (equivalente a 3,7 kg/m2) dell’IMC materno corretto per l’età è stato associato a un aumento di 0,22 (0,19, 0,25) SD (equivalente a 0,44 [0,38, 0,50] kg/m2) dell’IMC della prole nel modello corretto per i fondatori. Il punteggio allelico materno dell’IMC è stato analogamente associato positivamente all’IMC della prole in entrambi gli studi, ma con l’aggiustamento del punteggio allelico della prole le associazioni si sono attenuate al nullo, senza alcuna evidenza di eterogeneità tra le stime(S3 Fig).
Ci sono state associazioni simili del punteggio allelico materno con l’IMC materno e con l’IMC della prole tra le due coorti (ALSPAC e Generazione R; S3 Fig). Anche se l’effetto MR quando si mettono in comune i risultati rettificati per la prole in base al punteggio allelico 32-SNP in entrambe le coorti è apparso più debole rispetto alla nostra stima multivariabile rettificata per il cofondatore, 0,10 (-0,11, 0,31) SD (equivalente a 0,20 [-0,22, 0.62] kg/m2) contro 0,22 (0,19, 0,25) SD (equivalente di 0,44 [0,38, 0,50) kg/m2] per 1 SD maggiore IMC materno pre-gravidanza, non c’era una forte evidenza statistica che queste due stime differissero tra loro(p(diff) = 0,34; Fig 2). Quando abbiamo messo insieme i risultati di MR usando il punteggio dell’allele 97-SNP di ALSPAC con quelli della Generazione R usando il punteggio dell’allele 32-SNP, le stime dei punti erano più vicine a zero e sono state stimate con maggiore precisione (0.05 [-0.11, 0.21] SD per 1 SD maggiore BMI materno pre-gravidanza materna), e c’era una crescente evidenza statistica per una differenza tra questi risultati di MR e la stima multivariabile aggiustata per il cofondatore(p(diff) = 0.05; Fig 2).
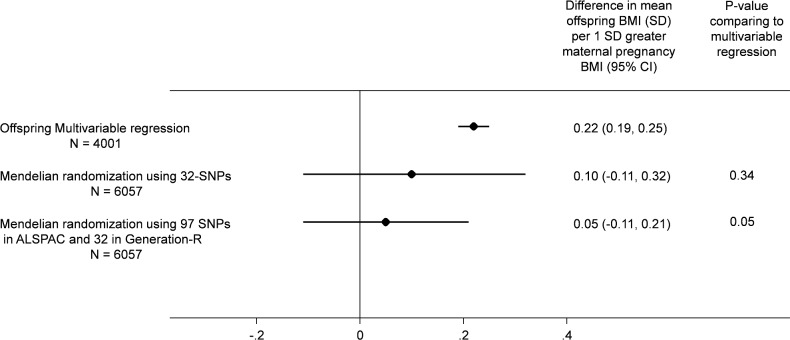
Fig 2.Relazione tra l’IMC della gravidanza materna e l’IMC della prole all’età di 7 anni: analisi multivariabile ALSPAC e analisi multivariabile Generazione-R e MR messe in comune.
Tutti i risultati sono la differenza di IMC medio della prole a 7 anni (unità SD) per ogni aumento di 1 IMC materno-gravidanza SD mettendo in comune i risultati dell’ALSPAC (coorte Discovery) con quelli dello studio Generation R (coorte Replication). In tutte le analisi, l’età della prole e il sesso sono controllati per attraverso la standardizzazione dell’IMC della prole, e l’età materna è controllata per la standardizzazione dell’IMC sull’età. I risultati delle analisi di regressione multivariabile sono controllati per la classe sociale dei genitori, l’educazione dei genitori, il fumo materno durante la gravidanza, la parità e l’IMC paterno e nella Generazione R solo per l’etnia (regolando per i primi 20 componenti principali dei dati di matrice genomica della prole). I risultati della risonanza magnetica sono corretti per il punteggio dell’allele della prole e nella generazione R solo per l’etnia (con una correzione per le prime 20 componenti principali dei dati a livello genomico della prole).
Data la debole associazione del punteggio allelico materno con l’educazione paterna in entrambe le coorti, ri-analizziamo tutte le analisi IV con un ulteriore aggiustamento per l’educazione paterna. L’aggiustamento aggiuntivo per l’educazione paterna non ha alterato la stima di MR nulla in seguito all’aggiustamento del punteggio dell’allele della prole(S4 Fig). Inoltre, le analisi limitate a quelle di ascendenza europea nella Generazione R erano simili a quelle che includono tutti i partecipanti alla Generazione R(S5 Fig).

Fig. 2.Relazione tra IMC materno-infantile e IMC della prole all’età di 7 anni: analisi multivariabile ALSPAC e analisi MR e multivariabile della Generazione R messe in comune.
Analisi di sensibilità
La nostra indagine sulla possibilità di introdurre il bias attraverso il genotipo paterno ha suggerito che la stima di MR allele-regolata del punteggio della prole era il risultato del minor bias dell’effetto causale dell’IMC materno pre-gravidanza sulla prole IMC. Anche se l’incapacità di regolare anche per le varianti genetiche paterne significava che questo era un po ‘di parte verso il nulla, era improbabile che abbia cambiato in modo significativo le nostre scoperte(S1 Appendice S1 e S11 Tabella). Inoltre, abbiamo esplorato l’uso dell’approccio aplotipo non trasmesso (alla prole)[50] in ALSPAC e sia il punteggio aplotipo 32-SNP che 97-SNP ha rivelato che il punteggio aplotipo materno non trasmesso non mostrava forti prove di associazione con la prole, mentre il punteggio aplotipo materno trasmesso era fortemente associato, che indica l’influenza genetica prevista per la prole sul proprio IMC, ma che fornisce scarso supporto per un effetto intrauterino materno dell’IMC sulla prole(Appendice S1 e Tabella S12).
I risultati dell’approccio IVW MR hanno mostrato stime approssimativamente uguali all’approccio del punteggio allelico TSLS. Inoltre, questo approccio non ha mostrato alcuna chiara evidenza di eterogeneità delle stime di effetto dei singoli SNPs che compongono il punteggio in ALSPAC o Generazione R (I2 = 0%; S6 Fig, S13 Table). Inoltre, il metodo MR Egger non ha dato alcuna indicazione di pleiotropia direzionale che influenza i risultati dell’analisi MR (intercetta = 0,005 [-0,003, 0,013], p = 0,13) per la meta-analisi di 97 SNPs in ALSPAC e 32 SNPs nella Generazione R) e ha fornito la prova di una mancanza di un effetto causale coerente di IMC materno sulla prole IMC nei modelli corretti per il genotipo della prole (coefficiente = 0.08 SD [-0.12, 0.28], p = 0.07) nella meta-analisi delle stime di ALSPAC e della Generazione R, anche se c’era un certo grado di eterogeneità tra i due studi (I2 per la pendenza = 70%; S6 Fig, S13 Table). Non c’era una forte evidenza di scostamento dalla linearità nella relazione tra IMC materno e IMC della prole nelle analisi multivariabili o MR(S7 Fig e S8 Fig).
Discussione
In ALSPAC, abbiamo trovato associazioni positive di BMI materno pre-gravidanza materna con la prole BMI e FMI a tutte le età, dall’infanzia alla prima età adulta, in analisi di regressione multivariabili confounder-adjusted multivariabile. Tuttavia, non abbiamo trovato alcuna prova che queste associazioni siano causali nelle analisi MR IV utilizzando un punteggio allelico ponderato di 32 (o 97) varianti genetiche note per essere robustamente associate all’IMC. C’era la prova statistica che le analisi multivariabili aggiustate per il cofondatore differivano dalle analisi MR della progenie, e i risultati erano virtualmente identici quando abbiamo usato l’FMI misurato con la scansione DXA. I risultati ALSPAC sono stati replicati nello studio indipendente Generation R con BMI valutato all’età di 6 anni. Presi insieme, questi risultati non supportano un importante effetto causale intrauterino di un maggiore BMI materno su una maggiore adiposità della prole. Ciò è in contrasto con l’evidenza di un effetto causale di una maggiore adiposità materna sul peso alla nascita e sull’indice ponderale alla nascita identificato utilizzando la RM in uno studio precedente che includeva entrambe le coorti qui utilizzate[7], indicando potenzialmente un effetto decrescente di questa esposizione intrauterina nel corso della vita(Fig 3).
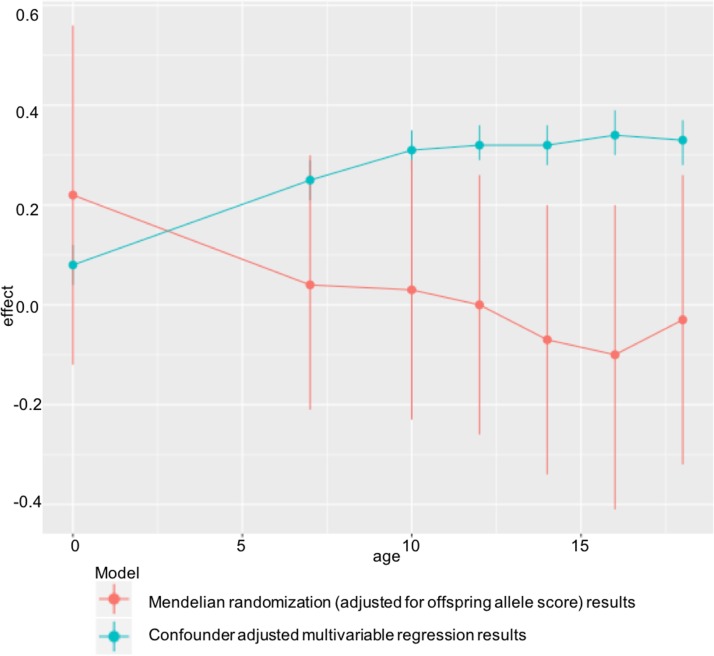
Fig 3.Fig. 3. Effetto causale decrescente dell’iper-nutrizione dello sviluppo nel corso della vita.Stime dell’effetto multivariabile e dell’effetto IV da ALSPAC all’età di 7-18 anni (Tabella 2) rispetto a quelle ottenute quando si indaga l’effetto dell’IMC materno sull’indice ponderale della prole (kg/m3)alla nascita nella stessa coorte.
I risultati di un effetto causale nullo dall’analisi MR sia in ALSPAC (a tutte le età dall’infanzia alla prima età adulta) e la generazione R (nell’infanzia) sono coerenti con la maggior parte degli studi di controllo negativo, che mostrano una magnitudo simile di associazione tra IMC materno pre-gravidanza e IMC della prole come quella tra IMC paterno e IMC della prole[54- 59], anche se nessuno di questi studi ha valutato i risultati in età adulta. Essi sono anche coerenti con un’ampia analisi all’interno di una grande all’interno di una famiglia, che ha suggerito che le caratteristiche familiari condivise confondevano l’associazione positiva tra IMC materno precoce gravidanza e IMC della prole all’età di 18 anni, in quanto questo è scomparso quando le analisi sono state condotte all’interno di fratelli e sorelle con una stretta corrispondenza delle caratteristiche familiari condivise [60]. Tuttavia, questo studio è stato eseguito solo nella prole maschile. Infine, i risultati sono coerenti con il precedente studio di risonanza magnetica in ALSPAC condotto utilizzando la variante FTO solo come IV per l’IMC materno ed esaminando la massa grassa determinata dall’IMC e dalla DXA della prole all’età di 10 anni[22]. Combinando un gran numero di varianti genetiche in un punteggio allelico, in questo studio, abbiamo aumentato la potenza statistica e mostrato una differenza tra l’analisi multivariabile e IV in ALSPAC a più età (dall’infanzia alla prima età adulta), che non era il caso con la precedente pubblicazione ALSPAC. Abbiamo anche mostrato una replica indipendente dei nostri risultati nella Generazione R, abbiamo studiato l’uso di più strumenti e testato i potenziali effetti pleiotropici.
Per l’analisi MR, abbiamo indagato a fondo su qualsiasi violazione delle principali ipotesi di questo approccio, che sono che la IV è fortemente associata all’esposizione di interesse, non legata a fattori di confusione dell’associazione esposizione-risultato, e non legata al risultato indipendente dal suo effetto sull’esposizione [45].
I punteggi allelici derivati da varianti stabilite per l’IMC sono stati fortemente associati con l’esposizione di interesse (IMC materno) nelle coorti complete, e grandi statistiche F hanno indicato una forza sufficiente come IVs, consentendo una valutazione più precisa dell’effetto dell’IMC materno rispetto a quanto fosse possibile in precedenza con l’uso di una singola variante genetica[22].
I punteggi allelici non sono stati associati in modo coerente con una serie di fattori socioeconomici, e questo illustra un punto di forza chiave dell’approccio MR. Inoltre, regolando per il genotipo della prole, la RM intergenerazionale è in grado di distinguere gli effetti materni causali sull’adiposità della prole dalla trasmissione genetica delle varianti di adiposità.
L’esistenza della pleiotropia, in cui uno strumento genetico ha un effetto su un risultato (IMC della prole) indipendente dal suo effetto sull’esposizione (IMC materno), avrebbe implicazioni per le ipotesi fatte nell’analisi della RM. Allo stesso modo, se una variante genetica nel punteggio fosse in disequilibrio di collegamento con un’altra variante genetica che influenza l’esito attraverso un percorso che non è correlato all’esposizione, questo potrebbe influenzare la stima causale. Il fatto che alcune varianti dell’IMC si riferiscano al glucosio e ad altri metaboliti[61,62] è un esempio di pleiotropia di tipo 2 o spuria ed è improbabile che influenzi gli effetti IV[63]. Abbiamo cercato di controllare l’effetto del genotipo della prole, che è correlato al genotipo materno, sul BMI della prole regolando il genotipo della prole. È difficile determinare un vero e proprio effetto pleiotropico che produca gli effetti nulli visti in seguito a questo aggiustamento. Inoltre, la coerenza delle stime IV ottenute utilizzando due punteggi allelici in questo studio suggerisce che la pleiotropia è improbabile. Inoltre, non vi erano forti prove di eterogeneità delle stime SNP individuali che comprendono i punteggi allelici, e il metodo MR Egger non ha dato alcuna indicazione di pleiotropia direzionale in ALSPAC o nella Generazione R.
Anche se le varianti genetiche materne vengono utilizzate come proxy infondato per un’esposizione intrauterina, queste varianti avranno un impatto anche sull’IMC materno dopo la gravidanza e quindi qualsiasi effetto causale, se fosse stato identificato, potrebbe anche incorporare effetti postnatali. Ad esempio, le madri geneticamente predisposte ad avere un IMC più elevato potrebbero influenzare l’adiposità della loro prole attraverso caratteristiche postnatali come il comportamento alimentare materno [64]. L’eterogeneità osservata nei modelli multivariabili, confounder-adjusted, potrebbe rappresentare una variazione nei contesti ambientali postnatali prevalenti, per esempio le influenze culturali sul comportamento alimentare dei genitori [65]. Tuttavia, gli effetti MR erano nulli sia in ALSPAC che nella Generazione R, suggerendo che l’effetto causale dell’IMC materno postnatale sull’adiposità della prole è al massimo molto piccolo. La debole associazione inversa dei punteggi allelici dell’IMC materno con i loro partner (ma non con la propria) è intrigante e vale la pena di essere esplorata ulteriormente, ma l’aggiustamento per questo non ha alterato i nostri risultati di MR.
E’ importante adattarsi al genotipo della prole nelle analisi MR, ma così facendo potremmo introdurre un percorso tra il genotipo materno e l’IMC della prole attraverso il genotipo paterno. Le nostre simulazioni suggerivano che questo avrebbe portato ad un debole bias verso il nullo, ma questo bias non spiegherebbe completamente gli effetti nulli che osserviamo, poiché i risultati dello studio di simulazione con aggiustamento per il punteggio dell’allele della prole erano vicini al vero risultato simulato(Tabella S11). Inoltre, i nostri risultati sono stati corroborati con i risultati dell’analisi aplotipica non trasmessa[50], applicata in ALSPAC, che ha rivelato che il punteggio aplotipico materno non trasmesso non mostrava forti prove di associazione con l’IMC della prole.
Nella meta-analisi MR utilizzando il punteggio allelico 97-SNP in ALSPAC e il punteggio allelico 32-SNP nella Generazione R, con aggiustamento per il genotipo della prole(n = 6.057), abbiamo avuto l’82% di potere di rilevare un effetto causale della dimensione di quello visto nell’analisi multivariabile controllata per età e sesso (0,28 SD) con un α su due lati = 0,05 nella nostra analisi MR. Pertanto, siamo stati adeguatamente potenziati per rilevare un effetto della dimensione di quello visto in modo osservativo, e l’eterogeneità che vediamo tra i risultati multivariabili e la risonanza magnetica è improbabile che sia dovuta al caso. Tuttavia, avremmo bisogno di un numero molto maggiore di partecipanti per escludere completamente l’evidenza di un effetto causale più debole.
Inoltre, il potere è stato limitato per alcuni dei test di sensibilità come la valutazione dell’eterogeneità SNP, l’analisi MR Egger, e la valutazione dell’eterogeneità nelle stime causali tra ALSPAC e Generazione R. Ulteriori studi con dati rilevanti su genotipo materno, prole e paterno sono necessari per ottenere stime causali più precise (e imparziali).
Ulteriori limitazioni all’analisi della risonanza magnetica includono la possibilità di stratificazione e canalizzazione della popolazione[66]. Anche se in ALSPAC la stratificazione della popolazione è improbabile perché i partecipanti sono individui non imparentati di ascendenza europea, lo Studio Generazione R è una coorte multietnica. Tuttavia, i tentativi di adattarsi alla stratificazione della popolazione includendo le componenti principali e le analisi limitate a quelle delle ascendenze europee sono stati nel complesso simili(S3 Fig). La canalizzazione dello sviluppo, in cui i sistemi si sviluppano in modo diverso per controbilanciare gli effetti di un particolare genotipo, può rappresentare un problema per la RM convenzionale. Tuttavia, questo è meno problematico nell’analisi della risonanza magnetica intergenerazionale. Questo perché quando il genotipo materno viene utilizzato come indicatore dell’ambiente intrauterino, allora questo influenzerà l’ambiente di sviluppo della prole solo attraverso l’esposizione di interesse e non attraverso meccanismi concorrenti[67].
L’IMC materno pre-gravidanza è stato dichiarato in entrambe le coorti. Anche se queste misure di auto-rapporto hanno dimostrato di essere fortemente correlate con l’IMC misurato nella prima gravidanza[68-70], la possibilità di sotto-rapporto sistematico, per esempio, se le persone che sono più pesanti sistematicamente sotto-rapportano il loro peso, potrebbe influenzare i risultati. Tuttavia, l’analisi precedente in ALSPAC ha dimostrato che l’errata dichiarazione è simile per la maggior parte dei partecipanti e non è influenzata in modo marcato dal peso medio[71]. Inoltre, l’ampiezza dell’associazione del punteggio allelico ponderato con l’IMC materno in questo studio era simile a quella vista per il suo rapporto con l’IMC basato sul peso misurato e l’altezza da altri studi[7].
Un limite generale delle misure longitudinali in ALSPAC è la perdita di follow-up del campione nel tempo, da 3.720 partecipanti all’età di 7 anni a 2.521 all’età di 17 anni. Ciononostante, la distribuzione del peso alla nascita era simile in gruppi ad ogni età. Inoltre, è improbabile che la perdita di bias di follow-up influisca sulle stime della RM, poiché è improbabile che le associazioni genetiche siano influenzate da dati mancanti[72]. Ciò è supportato dal fatto che i punteggi allelici utilizzati in questo studio non erano fortemente associati a una serie di fattori di rischio associati alla perdita al follow-up.
In questo studio abbiamo esaminato i risultati in ogni punto temporale separatamente, al fine di esplorare se le magnitudini sono simili a ogni età. Potrebbe anche essere utile esaminare se le esposizioni materne si riferiscono al tasso di variazione dell’adiposità della prole nell’infanzia e nell’età adulta utilizzando modelli multilivello. Di recente sono stati sviluppati metodi per applicare a tali modelli dati a livello genomico[73]. Tuttavia, non siamo a conoscenza del fatto che siano utilizzati in un quadro di MR IV, e riteniamo che l’approccio che abbiamo adottato sia pertinente al nostro obiettivo e produca risultati di facile interpretazione.
Il nostro studio ha esaminato l’effetto di aumenti lineari (incrementali) dell’IMC materno con esiti sulla prole. Sebbene siano stati recentemente sviluppati metodi per valutare gli effetti non lineari utilizzando la RM[74], questi richiedono campioni di dimensioni molto grandi e non siamo in grado di applicarli ai nostri dati. Pertanto, non possiamo escludere la possibilità, ad esempio, che un effetto soglia non lineare dell’obesità materna estrema abbia un effetto causale intrauterino sull’adiposità della prole. Tuttavia, non abbiamo trovato alcun chiaro scostamento dalla linearità né per la regressione multivariabile né per le analisi di risonanza magnetica(S7 Fig e S8 Fig). Infine, anche se il modello di regressione multivariabile positiva e di risultati di MR nulli è stato coerente in tutte le età in ALSPAC, riconosciamo che sarebbe utile mostrare un’ulteriore replicazione dei nostri risultati in grandi coorti indipendenti, in particolare nelle età più avanzate.
Dati i nostri risultati, insieme a quelli del confronto tra fratelli e sorelle[60] e degli studi di controllo negativo[54- 59,65], sembra improbabile che sottili differenze incrementali nel BMI materno pre- o precoce gravidanza giocino un ruolo chiave nell’avviare o perpetuare l’epidemia di obesità [11,12]. Anche se alcuni controlli negativi[22,75– 77] e confronti tra fratelli e sorelle [78,79] suggeriscono deboli effetti positivi dell’IMC materno pre-gravidanza o precoce sulla progenie, in generale, questi studi sono più piccoli di quelli che trovano effetti nulli e non hanno esplorato le associazioni in età adulta [1]. I nostri risultati non hanno mostrato alcun effetto fino all’età di 18 anni nelle femmine e nei maschi della coorte ALSPAC. Questa constatazione è importante, dato che la mancanza di effetti nella prole che entra nel loro corso della vita riproduttiva suggerisce che un’adiposità materna incrementalmente maggiore nella popolazione in gravidanza difficilmente alimenterà l’epidemia di obesità attraverso le generazioni, anche se non possiamo escludere un effetto di fenotipi più estremi, come l’obesità estrema o il diabete gestazionale[1]. Inoltre, non siamo stati in grado di indagare l’effetto causale dell’aumento di peso gestazionale durante la gravidanza sull’adiposità della prole utilizzando la risonanza magnetica, data l’attuale assenza di solidi strumenti genetici per questa esposizione[80].
Questi risultati suggeriscono che l’eccessiva dipendenza dagli interventi in gravidanza per ridurre l’obesità della popolazione potrebbe non essere giustificata e che le dichiarazioni di consenso[14], che indirizzano gli interventi di salute pubblica a tutti i membri della famiglia[81,82] e in diverse fasi del corso della vita, e non solo intrauterina o nella prima infanzia[13], sono probabilmente importanti. Ad esempio, gli interventi rivolti all’intera popolazione (cioè a tutti i membri della famiglia in tutte le fasi del ciclo vitale), come le proposte di tasse in eccesso sugli alimenti obesogeni[83], hanno potenzialmente maggiori probabilità di arrestare l’epidemia di obesità rispetto a un focus sull’IMC materno pre-gravidanza.

Fig. 3.Diminuzione dell’effetto causale dell’eccesso di nutrizione per lo sviluppo in tutto il corso della vita.Stime dell’effetto multivariabile e dell’effetto IV da ALSPAC all’età di 7-18 anni (Tabella 2) rispetto a quelle ottenute quando si indaga l’effetto dell’IMC materno sulla prole indice ponderale (kg/m3)alla nascita nella stessa coorte.
Informazioni di supporto
References
- Lawlor DA. The Society for Social Medicine John Pemberton Lecture 2011. Developmental overnutrition—an old hypothesis with new importance?. International journal of epidemiology. 2013; 42(1):7-29. DOI | PubMed
- Shin D, Song WO. Prepregnancy body mass index is an independent risk factor for gestational hypertension, gestational diabetes, preterm labor, and small- and large-for-gestational-age infants. Journal of Maternal-Fetal & Neonatal Medicine. 2014;1-8.
- Harmon KA, Gerard L, Jensen DR, Kealey EH, Hernandez TL, Reece MS. Continuous glucose profiles in obese and normal-weight pregnant women on a controlled diet: metabolic determinants of fetal growth. Diabetes care. 2011; 34(10):2198-204. DOI | PubMed
- Pederson J. Weight and length at birth of infants of diabetic mothers. Acta Endocrinologica. 1954; 16:330-42. PubMed
- Jackson WP. The prediabetic syndrome; large babies and the (pre) diabetic father. Journal of Clinical Endocrinology and Metabolism. 1954; 14(2):177-83. DOI | PubMed
- Freinkel N. Banting Lecture 1980. Of pregnancy and progeny. Diabetes. 1980; 29(12):1023-35. PubMed
- Tyrrell J, Richmond RC, Palmer TM, Feenstra B, Rangarajan J, Metrustry S. Genetic evidence for causal relationships between maternal obesity-related traits and birth weight. JAMA. 2015.
- Gillman MW, Kuh D, Ben-Shlomo Y. A Lifecourse Approach to Chronic Disease Epidemiology. Oxford University Press: Oxford; 2004.
- Taylor PD, Poston L. Developmental programming of obesity in mammals. Experimental Physiology. 2007; 92(2):287-98. DOI | PubMed
- Remmers F, Delemarre-van de Waal HA. Developmental programming of energy balance and its hypothalamic regulation. Endocrine Reviews. 2011; 32(2):272-311. DOI | PubMed
- Pettitt DJ, Knowler WC. Diabetes and obesity in the Pima Indians: a cross-generational vicious cycle. Journal of Obesity and Weight Regulation. 1988; 7:61-75.
- Ebbeling CB, Pawlak DB, Ludwig DS. Childhood obesity: public-health crisis, common sense cure. Lancet. 2002; 360(9331):473-82. DOI | PubMed
- Lawlor DA, Chaturvedi N. Treatment and prevention of obesity—are there critical periods for intervention?. International journal of epidemiology. 2006; 35(1):3-9. DOI | PubMed
- Poston L, Harthoorn LF, Van Der Beek EM, Workshop CttIE. Obesity in pregnancy: implications for the mother and lifelong health of the child. A consensus statement. Pediatric Research. 2011; 69(2):175-80. DOI | PubMed
- Nelson SM, Matthews P, Poston L. Maternal metabolism and obesity: modifiable determinants of pregnancy outcome. Human Reproduction Update. 2010; 16(3):255-75. DOI | PubMed
- Drake AJ, Reynolds RM. Impact of maternal obesity on offspring obesity and cardiometabolic disease risk. Reproduction. 2010; 140(3):387-98. DOI | PubMed
- Lawlor DA, Relton C, Sattar N, Nelson SM. Maternal adiposity-a determinant of perinatal and offspring outcomes?. Nat Rev Endocrinol. 2012; 8(11):679-88. DOI | PubMed
- Yu Z, Han S, Zhu J, Sun X, Ji C, Guo X. Pre-pregnancy body mass index in relation to infant birth weight and offspring overweight/obesity: a systematic review and meta-analysis. PLoS ONE. 2013; 8(4):e61627. DOI | PubMed
- Davey Smith G. Assessing intrauterine influences on offspring health outcomes: can epidemiological studies yield robust findings?. Basic and Clinical Pharmacology & Toxicology. 2008; 102(2):245-56. PubMed
- Gaillard R, Felix JF, Duijts L, Jaddoe VW. Childhood consequences of maternal obesity and excessive weight gain during pregnancy. Acta Obstetricia et Gynecologica Scandinavica. 2014; 93(11):1085-9. DOI | PubMed
- Davey Smith G, Ebrahim S. "Mendelian randomisation": can genetic epidemiology contribute to understanding environmental determinants of disease?. International journal of epidemiology. 2003; 32:1-22. PubMed
- Lawlor DA, Timpson NJ, Harbord RM, Leary S, Ness A, McCarthy MI. Exploring the developmental overnutrition hypothesis using parental-offspring associations and FTO as an IV. PLoS Med. 2008; 5(3):484-93.
- Burgess S, Thompson SG. Use of allele scores as instrumental variables for Mendelian randomization. International journal of epidemiology. 2013; 42(4):1134-44. DOI | PubMed
- Evans DM, Brion MJA, Paternoster L, Kemp JP, McMahon G, Munafo M. Mining the Human Phenome Using Allelic Scores That Index Biological Intermediates. PLoS Genet. 2013; 9(10)
- Boyd A, Golding J, Macleod J, Lawlor DA, Fraser A, Henderson J. Cohort Profile: The ‘Children of the 90s’—the index offspring of the Avon Longitudinal Study of Parents and Children. Int J Epidemiol. 2013; 42(1):111-27. DOI | PubMed
- Fraser A, Macdonald-Wallis C, Tilling K, Boyd A, Golding J, Davey Smith G. Cohort Profile: The Avon Longitudinal Study of Parents and Children: ALSPAC mothers cohort. Int J Epidemiol. 2013; 42(1):97-110. DOI | PubMed
- Jaddoe VW, van Duijn CM, Franco OH, van der Heijden AJ, van Iizendoorn MH, de Jongste JC. The Generation R Study: design and cohort update 2012. European journal of epidemiology. 2012; 27(9):739-56. DOI | PubMed
- Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D. PLINK: a tool set for whole-genome association and population-based linkage analyses. American journal of human genetics. 2007; 81(3):559-75. DOI | PubMed
- International HapMap Consortium, Frazer KA, Ballinger DG, Cox DR, Hinds DA, Stuve LL. A second generation human haplotype map of over 3.1 million SNPs. Nature. 2007; 449(7164):851-61. DOI | PubMed
- Evans DM, Zhu G, Dy V, Heath AC, Madden PAF, Kemp JP. Genome-wide association study identifies loci affecting blood copper, selenium and zinc. Hum Mol Genet. 2013; 22(19):3998-4006. DOI | PubMed
- Li Y, Willer C, Sanna S, Abecasis G. Genotype Imputation. Annu Rev Genom Hum G. 2009; 10:387-406.
- Li Y, Willer CJ, Ding J, Scheet P, Abecasis GR. MaCH: Using Sequence and Genotype Data to Estimate Haplotypes and Unobserved Genotypes. Genet Epidemiol. 2010; 34(8):816-34. DOI | PubMed
- Paternoster L, Zhurov AI, Toma AM, Kemp JP, St Pourcain B, Timpson NJ. Genome-wide association study of three-dimensional facial morphology identifies a variant in PAX3 associated with nasion position. American journal of human genetics. 2012; 90(3):478-85. DOI | PubMed
- Kemp JP, Medina-Gomez C, Estrada K, St Pourcain B, Heppe DH, Warrington NM. Phenotypic dissection of bone mineral density reveals skeletal site specificity and facilitates the identification of novel loci in the genetic regulation of bone mass attainment. PLoS Genet. 2014; 10(6):e1004423. DOI | PubMed
- Speliotes EK, Willer CJ, Berndt SI, Monda KL, Thorleifsson G, Jackson AU. Association analyses of 249,796 individuals reveal 18 new loci associated with body mass index. Nature genetics. 2010; 42(11):937-48. DOI | PubMed
- Locke AE, Kahali B, Berndt SI, Justice AE, Pers TH, Day FR. Genetic studies of body mass index yield new insights for obesity biology. Nature. 2015; 518(7538):197-206. DOI | PubMed
- Kruithof CJ, Kooijman MN, van Duijn CM, Franco OH, de Jongste JC, Klaver CC. The Generation R Study: Biobank update 2015. European journal of epidemiology. 2014; 29(12):911-27. DOI | PubMed
- Medina-Gomez C, Felix JF, Estrada K, Peters MJ, Herrera L, Kruithof CJ. Challenges in conducting genome-wide association studies in highly admixed multi-ethnic populations: the Generation R Study. European journal of epidemiology. 2015; 30(4):317-30. DOI | PubMed
- Jaddoe VW, van Duijn CM, van der Heijden AJ, Mackenbach JP, Moll HA, Steegers EA. The Generation R Study: design and cohort update 2010. European journal of epidemiology. 2010; 25(11):823-41. DOI | PubMed
- 1000 Genomes Project Consortium, Abecasis GR, Auton A, Brooks LD, DePristo MA, Durbin RM. An integrated map of genetic variation from 1,092 human genomes. Nature. 2012; 491(7422):56-65. DOI | PubMed
- Speliotes EK, Willer CJ, Berndt SI, Monda KL, Thorleifsson G, Jackson AU. Association analyses of 249,796 individuals reveal 18 new loci associated with body mass index. Nature Genetics. 2010; 42(11):937-48. DOI | PubMed
- Lin X, Song K, Lim N, Yuan X, Johnson T, Abderrahmani A. Risk prediction of prevalent diabetes in a Swiss population using a weighted genetic score-the CoLaus Study. Diabetologia. 2009; 52(4):600-8. DOI | PubMed
- Staiger D, Stock JH. Instrumental variables regression with weak instruments. Econometrica. 1997; 65(3):557-86.
- Davey Smith G, Lawlor DA, Harbord R, Timpson N, Day I, Ebrahim S. Clustered environments and randomized genes: a fundamental distinction between conventional and genetic epidemiology. PLoS Med. 2007; 4(12):e352. DOI | PubMed
- Didelez V, Meng S, Sheehan NA. Assumptions of IV Methods for Observational Epidemiology. Stat Sci. 2010; 25(1):22-40.
- Higgins JPT, Thompson SG, Deeks JJ, Altman DG. Measuring inconsistency in meta-analyses. Brit Med J. 2003; 327(7414):557-60. DOI | PubMed
- Palmer TM, Lawlor DA, Harbord RM, Sheehan NA, Tobias JH, Timpson NJ. Using multiple genetic variants as instrumental variables for modifiable risk factors. Statistics in medicine. 2012; 21(3):223-242. DOI | PubMed
- Thomas DC, Lawlor DA, Thompson JR. Re: Estimation of bias in nongenetic observational studies using "Mendelian triangulation" by Bautista et al.. Annals of epidemiology. 2007; 17(7):511-3. DOI | PubMed
- Ding EL, Hu FB. Determining origins and causes of childhood obesity via Mendelian randomization analysis. PLoS Med. 2008; 5(3):e65. DOI | PubMed
- Zhang G, Bacelis J, Lengyel C, Teramo K, Hallman M, Helgeland O. Assessing the Causal Relationship of Maternal Height on Birth Size and Gestational Age at Birth: A Mendelian Randomization Analysis. PLoS Med. 2015; 12(8)
- Del Greco MF, Minelli C, Sheehan NA, Thompson JR. Detecting pleiotropy in Mendelian randomisation studies with summary data and a continuous outcome. Stat Med. 2015; 34(21):2926-40. DOI | PubMed
- Bowden J, Davey Smith G, Burgess S. Mendelian randomization with invalid instruments: effect estimation and bias detection through Egger regression. International journal of epidemiology. 2015; 44(2):512-25. DOI | PubMed
- .Publisher Full Text
- Davey Smith G, Steer C, Leary S, Ness A. Is there an intrauterine influence on obesity? Evidence from parent-child associations in the Avon Longitudinal Study of Parents and Children (ALSPAC). Arch Dis Child. 2007; 92(10):876-80. DOI | PubMed
- Patel R, Martin RM, Kramer MS, Oken E, Bogdanovich N, Matush L. Familial associations of adiposity: findings from a cross-sectional study of 12,181 parental-offspring trios from Belarus. PLoS ONE. 2011; 6(1):e14607. DOI | PubMed
- Subramanian SV, Ackerson LK, Davey Smith G. Parental BMI and childhood undernutrition in India: an assessment of intrauterine influence. Pediatrics. 2010; 126(3):e663-71. DOI | PubMed
- Fleten C, Nystad W, Stigum H, Skjaerven R, Lawlor DA, Davey Smith G. Parent-offspring body mass index associations in the Norwegian Mother and Child Cohort Study: a family-based approach to studying the role of the intrauterine environment in childhood adiposity. Am J Epidemiol. 2012; 176(2):83-92. DOI | PubMed
- Vik KL, Romundstad P, Carslake D, Davey Smith G, Nilsen TI. Comparison of father-offspring and mother-offspring associations of cardiovascular risk factors: family linkage within the population-based HUNT Study, Norway. International journal of epidemiology. 2014; 43(3):760-71. DOI | PubMed
- Corsi DJ, Subramanian SV, Ackerson LK, Davey Smith G. Is there a greater maternal than paternal influence on offspring adiposity in India?. Arch Dis Child. 2015.
- Lawlor DA, Lichtenstein P, Langstrom N. Association of maternal diabetes mellitus in pregnancy with offspring adiposity into early adulthood: sibling study in a prospective cohort of 280,866 men from 248,293 families. Circulation. 2011; 123(3):258-65. DOI | PubMed
- Freathy RM, Timpson NJ, Lawlor DA, Pouta A, Ben-Shlomo Y, Ruokonen A. Common variation in the FTO gene alters diabetes-related metabolic traits to the extent expected given its effect on BMI. Diabetes. 2008; 57(5):1419-26. DOI | PubMed
- Wurtz P, Wang Q, Kangas AJ, Richmond RC, Skarp J, Tiainen M. Metabolic Signatures of Adiposity in Young Adults: Mendelian Randomization Analysis and Effects of Weight Change. PLoS Med. 2014; 11(12)
- Davey Smith G, Hemani G. Mendelian randomization: genetic anchors for causal inference in epidemiological studies. Hum Mol Genet. 2014; 23(R1):R89-R98. DOI | PubMed
- Baker JL, Michaelsen KF, Rasmussen KM, Sorensen TIA. Maternal prepregnant body mass index, duration of breastfeeding, and timing of complementary food introduction are associated with infant weight gain. Am J Clin Nutr. 2004; 80(6):1579-88. PubMed
- Patro B, Liber A, Zalewski B, Poston L, Szajewska H, Koletzko B. Maternal and paternal body mass index and offspring obesity: a systematic review. Annals of Nutrition & Metabolism. 2013; 63(1–2):32-41. PubMed
- Lawlor DA, Harbord RM, Sterne JAC, Timpson NJ, Davey Smith G. Mendelian randomization: using genes as instruments for making causal inferences in epidemiology. Statistic in Medicine. 2008; 27:1133-63.
- Davey Smith G. Mendelian Randomization for Strengthening Causal Inference in Observational Studies: Application to Gene x Environment Interactions. Perspect Psychol Sci. 2010; 5(5):527-45. DOI | PubMed
- Brion MJ, Zeegers M, Jaddoe V, Verhulst F, Tiemeier H, Lawlor DA. Intrauterine effects of maternal prepregnancy overweight on child cognition and behavior in 2 cohorts. Pediatrics. 2011; 127(1):e202-11. DOI | PubMed
- Gaillard R, Durmus B, Hofman A, Mackenbach JP, Steegers EA, Jaddoe VW. Risk factors and outcomes of maternal obesity and excessive weight gain during pregnancy. Obesity. 2013; 21(5):1046-55. DOI | PubMed
- Nohr EA, Timpson NJ, Andersen CS, Davey Smith G, Olsen J, Sorensen TI. Severe obesity in young women and reproductive health: the Danish National Birth Cohort. PLoS ONE. 2009; 4(12):e8444. DOI | PubMed
- Sharp GC, Lawlor DA, Richmond RC, Fraser A, Simpkin A, Suderman M. Maternal pre-pregnancy BMI and gestational weight gain, offspring DNA methylation and later offspring adiposity: findings from the Avon Longitudinal Study of Parents and Children. International journal of epidemiology. 2015; 10
- Bhatti P, Sigurdson AJ, Wang SS, Chen J, Rothman N, Hartge P. Genetic variation and willingness to participate in epidemiologic research: data from three studies. Cancer Epidemiology, Biomarkers & Prevention. 2005; 14(10):2449-53.
- Warrington NM, Howe LD, Paternoster L, Kaakinen M, Herrala S, Huikari V. A genome-wide association study of body mass index across early life and childhood. Int J Epidemiol. 2015; 44(2):700-12. DOI | PubMed
- Silverwood RJ, Holmes MV, Dale CE, Lawlor DA, Whittaker JC, Davey Smith G. Testing for non-linear causal effects using a binary genotype in a Mendelian randomization study: application to alcohol and cardiovascular traits. International journal of epidemiology. 2014; 43(6):1781-90. DOI | PubMed
- Catalano PM, Farrell K, Thomas A, Huston-Presley L, Mencin P, de Mouzon SH. Perinatal risk factors for childhood obesity and metabolic dysregulation. Am J Clin Nutr. 2009; 90(5):1303-13. DOI | PubMed
- Lawlor DA, Davey Smith G, O’Callaghan M, Alati R, Mamun AA, Williams GM. Epidemiologic evidence for the fetal overnutrition hypothesis: findings from the mater-university study of pregnancy and its outcomes. Am J Epidemiol. 2007; 165(4):418-24. DOI | PubMed
- Gaillard R, Steegers EA, Duijts L, Felix JF, Hofman A, Franco OH. Childhood cardiometabolic outcomes of maternal obesity during pregnancy: the Generation R Study. Hypertension. 2014; 63(4):683-91. DOI | PubMed
- Kral JG, Biron S, Simard S, Hould FS, Lebel S, Marceau S. Large maternal weight loss from obesity surgery prevents transmission of obesity to children who were followed for 2 to 18 years. Pediatrics. 2006; 118(6):e1644-9. DOI | PubMed
- Smith J, Cianflone K, Biron S, Hould FS, Lebel S, Marceau S. Effects of maternal surgical weight loss in mothers on intergenerational transmission of obesity. Journal of Clinical Endocrinology and Metabolism. 2009; 94(11):4275-83. DOI | PubMed
- Warrington NN, Evans DM, Lawlor DA, Consortium EGG. Does genetic variation contribute to the architecture of gestational weight gain?. Journal of Developmental Origins of Health and Disease. 2015; 6(S2):S6.
- Richardson SS, Daniels CR, Gillman MW, Golden J, Kukla R, Kuzawa C. Don’t blame the mothers. Nature. 2014; 512(7513):131-2. DOI | PubMed
- Warin M, Zivkovic T, Moore V, Davies M. Mothers as smoking guns: Fetal overnutrition and the reproduction of obesity. Fem Psychol. 2012; 22(3):360-75.
- Tedstone A, Targett V, Allen R, England PH. Sugar Reduction: The evidence for action. Public Health England: London; 2015.
Fonte
Richmond RC, Timpson NJ, Felix JF, Palmer T, Gaillard R, et al. (2017) Using Genetic Variation to Explore the Causal Effect of Maternal Pregnancy Adiposity on Future Offspring Adiposity: A Mendelian Randomisation Study. PLoS Medicine 14(1): e1002221. https://doi.org/10.1371/journal.pmed.1002221





