Ricerca nel contestoEvidenza prima di questo studioSi ipotizza che le infezioni da dengue naturali primarie siano in grado di offrire una protezione a vita contro il sierotipo infettante, in parte, grazie alla produzione di anticorpi specifici per quel sierotipo, ma l’ontogenesi di questa risposta a livello delle cellule B non è definita. La determinazione accurata dello stato di infezione e l’acquisizione di campioni longitudinali da un’infezione primaria naturale può essere problematica. Per il nostro studio abbiamo cercato di utilizzare un modello di infezione umana controllata per la dengue per definire la specificità della risposta delle cellule B all’infezione primaria da dengue 2 dalla fase acuta fino alla convalescenza. All’inizio del nostro studio, nel 2015, abbiamo cercato su Pubmed (filtro linguistico disattivato) le “cellule B dell’infezione da dengue di tipo 2” e abbiamo recuperato 15 voci, nessuna delle quali utilizzava un modello di infezione umana controllata per la dengue.Valore aggiuntoIl nostro lavoro è il primo a mostrare chiaramente le origini e il mantenimento della specificità dei sierotipi della risposta delle cellule B all’infezione primaria da dengue in soggetti sani e ingenui.Implicazioni di tutte le prove disponibili Questi risultati sono rilevanti per la salute umana perché i quattro sierotipi di dengue sono una delle principali minacce per la salute globale. Un vaccino contro la dengue che protegge da tutti e quattro i sierotipi nei soggetti affetti da flavivirus-naïve è urgentemente necessario. Questi risultati servono come linea di base contro cui valutare le risposte del vaccino contro la dengue in soggetti flavivirus-naïve sia in termini di cinetica della risposta cellulare, ampiezza e specificità: Box non etichettato
Prove prima di questo studio
Si ipotizza che le infezioni primarie naturali da dengue siano in grado di offrire una protezione a vita contro il sierotipo infettante, in parte attraverso la produzione di anticorpi specifici per quel sierotipo, ma l’ontogenesi di questa risposta a livello delle cellule B non è definita. La determinazione accurata dello stato di infezione e l’acquisizione di campioni longitudinali da un’infezione primaria naturale può essere problematica. Per il nostro studio abbiamo cercato di utilizzare un modello di infezione umana controllata per la dengue per definire la specificità della risposta delle cellule B all’infezione primaria da dengue 2 dalla fase acuta fino alla convalescenza. All’inizio del nostro studio nel 2015 abbiamo cercato Pubmed (filtro linguistico spento) per “cellule B dell’infezione da dengue di tipo 2” e abbiamo recuperato 15 voci, nessuna delle quali utilizzava un modello di infezione umana controllata per la dengue.
Valore aggiunto
Il nostro lavoro è il primo a mostrare chiaramente le origini e il mantenimento della specificità del sierotipo della risposta delle cellule B all’infezione primaria da dengue in soggetti sani e ingenui.
Implicazioni di tutte le prove disponibili
Questi risultati sono rilevanti per la salute umana perché i quattro sierotipi di dengue sono una delle principali minacce per la salute globale. È urgentemente necessario un vaccino contro la dengue che protegga da tutti e quattro i sierotipi nei soggetti affetti da flavivirus-naïve. Questi risultati servono come linea di base contro cui valutare le risposte del vaccino contro la dengue in soggetti flavivirus-naïve sia in termini di cinetica di risposta cellulare, ampiezza e specificità.
Introduzione
I virus della dengue (DENV) trasmessi dai vettori delle zanzare sono gli agenti eziologici della febbre dengue e della febbre emorragica dengue[1,2]. Si stima che i DENV causino ogni anno 390 milioni di infezioni, soprattutto nelle regioni tropicali e subtropicali del mondo[3]. Con >100 milioni di casi apparenti, le infezioni da DENV sono la principale causa di ospedalizzazione pediatrica in molti paesi colpiti.
Il complesso della DENV è costituito da quattro sierotipi strettamente correlati (DENV1-4). A seguito di un’infezione primaria naturale da DENV, vengono generati elevati livelli di anticorpi omotipici neutralizzanti circolanti contro il sierotipo infettante[4] e mantenuti per decenni[5], suggerendo un contributo alla protezione a vita contro il sierotipo infettante. Gli anticorpi cross-reattivi del sierotipo, anch’essi provocati da un’infezione primaria, sono utilizzati per fornire una protezione incrociata transitoria contro l’infezione eterotipica secondaria con un nuovo sierotipo[6]. Le infezioni eterotipiche secondarie più remote (anni) sono associate a un maggiore rischio di sviluppare una grave febbre emorragica dengue [[7], [8], [ 9], [ 10], [ 11]]. L’aumento del rischio di malattia grave nei casi secondari sembra essere guidato, almeno in parte, da anticorpi cross-reattivi e non neutralizzanti che possono aumentare l’infezione virale[12]. La relazione temporale tra risposte precoci e tardive delle cellule B e il mantenimento della specificità del sierotipo a seguito di un’infezione primaria da DENV è incompleta.
I plasmablasti sono una popolazione di cellule B che secernono anticorpi, presenti a bassi livelli nel sangue periferico sano, ma possono aumentare drasticamente in numero a seguito di infezione o vaccinazione [13], [ 14], [ 15], [ 16], [ 17], [ 18]]. Sono stati documentati alti livelli di plasmablasti durante l’infezione acuta in corso da DENV [[19], [ 20], [ 21]]]. Più tardi, dopo il recupero, le risposte anticorpali protettive a lungo termine sono mantenute per molti anni dalle cellule B della memoria specifica dell’antigene (MBC) e dalle plasmacellule a lunga vita (LLPC) [[22], [23], [ 24]]]. Circa lo 0,1-0,5% del MBC nel sangue periferico produce anticorpi DENV-specifici [25,26]. Studi precedenti hanno dimostrato che la maggior parte degli MBC specifici per la DENV producono anticorpi cross-reattivi del sierotipo e scarsamente neutralizzanti. Una piccola frazione di MBC specifico della DENV codifica per gli anticorpi specifici del sierotipo e fortemente neutralizzanti [[27], [28], [29], [ 30]]]. La maggior parte degli anticorpi fortemente neutralizzanti si lega a complessi epitopi conformazionali della proteina dell’involucro virale (E) che vengono visualizzati in modo efficiente sui virioni intatti ma non sui monomeri della proteina E [25,[31], [ 32], [ 33], [ 34]]]. La risposta anticorpale policlonale LLPC-derivata dal siero contiene sia anticorpi cross-reattivi scarsamente neutralizzanti che una piccola frazione di anticorpi fortemente neutralizzanti specifici del sierotipo[25, 28,31].
A differenza delle risposte MBC e LLPC, la risposta precoce del plasmablast all’infezione primaria da DENV è meno chiara. Diversi gruppi hanno caratterizzato la risposta precoce del plasmablasto a seguito di infezioni secondarie da DENV[19,20,35,36]. I plasmablastomi DENV-specifici rappresentano il 30-50% delle cellule B circolanti durante un’infezione secondaria in corso [19,35,37,38]. Nelle infezioni secondarie, l’entità della risposta del plasmablasto è stata correlata positivamente con la gravità della malattia[35]. Nella dengue secondaria la stragrande maggioranza del pool di plasmablast è cross-reattiva[38,39] e non viene mantenuta in convalescenza[40]. Qui si mira a caratterizzare la cinetica del plasmblablablasto, l’entità e la specificità dell’infezione primaria da DENV.
Per definire longitudinalmente la funzionalità dei compartimenti del plasmablasto, MBC e siero a seguito di un’infezione primaria da DENV, abbiamo analizzato le risposte DENV-specifiche in ciascuno di questi compartimenti negli stessi individui a seguito di un’infezione da virus DENV2 challenge[41]. In questo modello di infezione controllata, un’infezione primaria da DENV2 ha stimolato una risposta rapida del plasmablasto in cui una grande frazione di queste cellule ha prodotto anticorpi specifici del sierotipo contro il DENV2. La dominanza degli anticorpi specifici del sierotipo DENV2 è stata osservata anche durante la convalescenza tardiva (6 months dopo l’infezione) nei compartimenti MBC e LLPC. Nel complesso, le proprietà degli anticorpi antigene-specifici nella fase iniziale si riflettono anche nella fase di convalescenza tardiva.
Materiali e metodi
Partecipanti allo studio
Per questo studio, i soggetti hanno ricevuto103 unità di messa a fuoco del virus dell’infezione umana parzialmente attenuato DENV2 rDEN2Δ30, ceppo Tonga/74 per iniezione sottocutanea e seguito per sei mesi. Dieci soggetti hanno partecipato ad uno studio autonomo per la sicurezza e l’immunogenicità del ceppo rDEN2Δ30/Tonga/74 (CIR286, Clinicaltrials .govNCT01931176, rif.42] e nove hanno partecipato al braccio placebo/challenge di uno studio clinico di Fase II per testare la sicurezza e l’immunogenicità del vaccino tetravalente TV003 (CIR287, Clinicaltrials .govNCT02021968) seguito dalla sfida rDEN2Δ30/Tonga/74[41]. Tutti i soggetti sono stati confermati sierologicamente come flavivirus-naïve al momento della sfida rDEN2Δ30, come descritto in precedenza [41]. Gli studi sono stati approvati dalle commissioni di revisione istituzionale dell’Università del Vermont e/o della Johns Hopkins University. Il consenso informato è stato ottenuto in conformità alle normative federali e internazionali (21CFR50 e ICHE6). Il monitoraggio esterno indipendente è stato effettuato dal National Institute of Allergy and Infectious Diseases Data Safety Monitoring Board ogni 6 months.
Procedure cliniche e raccolta di campioni
Durante le visite di studio, il sangue è stato raccolto mediante venipuntura in tubi separatori di siero per l’analisi della viremia e della sierologia, e in tubi EDTA per l’isolamento delle cellule mononucleari del sangue periferico (PBMC). Il siero è stato isolato e congelato a -20 °C fino all’uso. PBMC sono stati isolati da Ficoll-paque densità di separazione gradiente di densità Ficoll-paque, contati e congelati in 10% DMSO/40% di siero fetale bovino (FBS) in mezzo di coltura e crioconservati in fase di vapore di azoto liquido.
Quantificazione del virus e risposta sierologica nei soggetti
I campioni di siero raccolti durante le visite di follow-up ogni due giorni fino al 16° giorno sono stati testati per il virus infettivo mediante titolazione diretta su cellule Vero-81 (American Type Culture Collection; CCL-81, RRID: CVCL_0059), e i focolai sono stati rilevati con anticorpi monoclonali specifici del sierotipo come descritto[43]. Le risposte anticorpali neutralizzanti contro DENV1 (WestPac/74), DENV2 (Nuova Guinea C e Tonga/74), DENV3 (Slemen/78), e DENV4 (Dominica/81) sono state misurate con il test di neutralizzazione della riduzione del fuoco (FRNT), utilizzando la più bassa diluizione sierica che ha dato una riduzione del 50% dei focolai virali (FRNT50) come descritto[43]. Se non diversamente specificato, questi virus sono stati utilizzati per la determinazione della specificità del sierotipo DENV2 e della reattività crociata degli anticorpi derivati dai plasmablasti, MBC e siero nei test ELISA o di neutralizzazione.
Analisi dei plasmablasti e delle cellule B della memoria
Fenotipizzazione del plasmablast
PBMC ai giorni 0, 4, 8,14, 21, 28, 56, 56, 90, 150 e 180 dopo la sfida rDEN2Δ30 sono stati macchiati: anti-CD19 (clone HIB19, PE-Dazzle 594, BioLegend), anti-CD20 (2H7, PE-Cy7, BioLegend), anti-CD27 (O323, Brilliant Violet 510, BioLegend), anti-CD38 (HIT2, AlexaFluor 647, BioLegend), anti-CD3 (UCHT1, FITC, BioLegend), e anti-CD14 (HCD14, FITC, BioLegend). I plasmablasti sono stati fenotipizzati mediante gating per CD3- CD14- CD19 + CD20- CD27 + CD38hi celle di sabbiaturadal vivo.I dati sono stati raccolti su un BD LSR II (QC’d daily e set di compensazione con perline di compensazione (BD Biosciences) e i dati sono stati analizzati con FlowJo v10 (TreeStar).
Smistamento al plasma
PBMC ai giorni 8, 14 e 21 dopo la sfida rDEN2Δ30 sono stati colorati con: anti-CD19 (HIB19, Brilliant Violet 421, BioLegend), anti-CD20 (2H7, PE-Cy7, BioLegend o L27, PerCP-Cy5.5, BD Biosciences), anti-CD27 (O323, Brilliant Violet 510, BioLegend), anti-CD38 (HIT2, Alexa647, BioLegend), anti-IgA (IS11-8E10, FITC, Miltenyi Biotec), anti-IgM (MHM-88, FITC, BioLegend), anti-CD3 (UCHT1, FITC, BioLegend), e anti-CD14 (HCD14, FITC, BioLegend). L’ordinamento per plasmablasti IgG+ (una cellula per pozzetto in piastre a 96 pozzetti) è stato eseguito mediante selezione di cellule attivate a fluorescenza (FACS) per CD3- CD14- IgA- IgM- CD19 + CD20low/- CD27 + CD38hicellule utilizzando unsoftware BD FACSJazz e BDFACS Diva. Le cellule sono state classificate a cella singola in piastre PCR a 96 pozzetti contenenti tampone di lisi (10 mM Tris-HCl, pH 7.6), con 2 mM dNTPs (New England Biolabs), 5 μM oligo(dT)20VN, e 1 unità/μl di RiboLock RNase Inhibitor (ThermoFisher Scientific). Le piastre a cella singola ordinata sono state conservate a -80 °C fino all’uso per la trascrizione inversa (RT).
La trascrizione inversa, la PCR, l’assegnazione del codice a barre, l’assemblaggio della sequenza, l’assegnazione V(D)J e l’identificazione delle mutazioni sono state eseguite come descritto in precedenza[44,45] con le seguenti modifiche: per la trascrizione inversa è stato utilizzato oligo(dT) biotinilato, il cDNA è stato estratto utilizzando perle di Streptavidina C1 (Life Technologies), le concentrazioni di DNA sono state determinate utilizzando qPCR (KAPA SYBR® FAST qPCR Kit per Titanio, Kapabiosystems), ed è stata richiesta una copertura minima di 10 letture da ogni assemblaggio a catena da includere nei repertori di sequenza. L’assegnazione V(D)J e l’identificazione delle mutazioni sono state eseguite utilizzando un’implementazione di SoDA[46]. Le sequenze accoppiate della catena H e della catena L all’interno di ogni repertorio di plasmablast del destinatario rDEN2Δ30 sono state assegnate allo stesso lignaggio se l’uso del gene H-catena V, la lunghezza CDRH3, l’uso del gene L-catena V e la lunghezza CDRL3 erano identici. I CDR a catena H e L, come definiti [47], sono stati identificati allineando le sequenze proteiche ad un modello Markov nascosto [48]. Le sequenze sono state ulteriormente separate in lignaggi putativi in base al grado di identità delle sequenze CDRH3 e CDRL3.
Selezione, clonazione di geni anticorpali e l’espressione di anticorpi monoclonali da plasmablasti
I diversi lignaggi anticorpali sono stati classificati in base alle prove di espansione e convergenza tra i soggetti, come descritto[49]. In breve, i criteri utilizzati per classificare i lignaggi sono stati (1) il numero di cloni di plasmablastoma distinti all’interno di ogni lignaggio indicativi di espansione o di risposta distorta all’infezione, (2) il numero di mutazioni che suggeriscono la maturazione dell’affinità, (3) la sovrapposizione di lignaggi tra i tre soggetti che suggeriscono un’evoluzione convergente, e (4) lignaggi clonali con apparente somiglianza di sequenza tra i membri del lignaggio, indicativi della condivisione di progenitori comuni. Da ciascuno dei 96 lignaggi a più alta priorità, abbiamo selezionato un membro del lignaggio per l’espressione ricombinante e la purificazione. Le sequenze selezionate erano o dal clone plasmablast del lignaggio con la più alta identità alla sequenza di consenso del lignaggio, o dal clone espresso dal maggior numero di plasmablast del lignaggio.
Le 96 coppie di geni a catena pesante e leggera di anticorpi sono state clonate in vettori di espressione mammiferi (Lake Pharma, Belmont, California). Ogni costruzione completa è stata confermata dal sequenziamento. Una piccola scala (0.01 L) produzione transitoria è stato fatto in cellule HEK293 cellule che sono stati seminati in un matraccio di agitazione e ampliato utilizzando chimicamente definito siero libero mezzo. Per ogni anticorpo, sia il pesante e la catena di luce che la catena di codifica del DNA costruisce sono stati transitoriamente co-trasferiti in cellule. Le cellule sono state mantenute come una coltura alimentata in batch fino alla fine della produzione. Le proteine sono state purificate con la purificazione della Proteina A. I mezzi condizionati dalla corsa di produzione transitoria è stato raccolto e chiarito da centrifugazione e filtrazione. Il supernatante è stato caricato in una colonna di Proteina A pre-equilibrata con tampone legante. Il tampone di lavaggio è stato fatto passare attraverso la colonna fino a quando il valore di OD280 (NanoDrop, ThermoScientific) è stato misurato a zero. La proteina bersaglio è stato eluito con un tampone a basso pH; frazioni sono state raccolte e filtrate attraverso un filtro a membrana 0.2 μm. Gli anticorpi erano in 200 mM HEPES, 100 mM NaCl, 50 mM NaOAc, pH 7.0 buffer. La concentrazione di proteine è stata calcolata a partire dal valore di OD280 e dal coefficiente di estinzione calcolato. La resa media è stata di 0,117 mg e la resa mediana è stata di 0,08 mg. Novantadue delle 96 coppie selezionate IGH/IGL hanno prodotto proteine sufficienti per i test funzionali.
Memoria B isolamento delle cellule e l’immortalizzazione
Le celle B di memoria commutata sono state isolate dalle PBMC crioconservate raccolte il giorno 180 a seguito della sfida rDEN2Δ30. Dopo lo scongelamento, la vitalità di PBMC è stata >80% come valutato dalla mancanza di colorazione DAPI (4, 6-diamidino-2-fenilindolo, 5 μg per campione in PBS – analizzato con citometria a flusso su un autocampionatore Miltenyi VYB). Le cellule B sono state arricchite con l’etichettatura PBMC con anticorpo anti-CD22 coniugato con microsfere (Miltenyi, catalogo n. 130-046-401) seguito da separazione del campo magnetico (colonne Miltenyi MS) ad una purezza media dell’85%. Le cellule B a memoria commutata sono state purificate dalle cellule B arricchite con CD22 mediante etichettatura con anti-CD3 (UCHT1, FITC, Biolegend), anti-CD19 (HIB19, PE-Dazzle594, Biolegend), anti-CD27 (O323, PE-Cy7, Biolegend) e anti-IgM (MHM-88, PerCP-Cy5.5, BioLegend). Le cellule sono state ordinate in un mezzo di coltura completo (vedi sotto) da FACS dal vivo linfocita (FSC x SSC) gate per CD3- CD19 + CD27+IgM- cellulesu un BD FACSAriaIII utilizzando il software BD FACSDiva. La purezza post-sorta era ≥ 95%.
Le cellule B della memoria purificata sono state immortalate con l’introduzione di BCL-6 e Bcl-xL utilizzando la trasduzione retrovirale come descritto in precedenza[50]. In breve, le cellule B della memoria purificata sono state attivate in una piastra trattata con 24 pozzetti di cultura tissutale con 1 mL di Iscove’s Modified Dulbecco’s Medium (IMDM) (Gibco, ThermoFisher) contenente l’8% di FBS (Atlanta Biologicals), penicillina (100 units/mL, Gibco), streptomicina (100 μg/mL, Gibco), 1 × 105 irradiato(50 Gy ) umano CD40L (CD154)-espressione di fibroblasti cellulari L del topo e 50 ng/mL interleuchina umana ricombinante 50 ng/mL (rhIL)-21 (Peprotech) per 36-48 h a 37 °C, 5% di CO2. Le cellule sono state poi sospese in 0.25 mL di IMDM senza siero in 24 pozzetti trattati con coltura non-tessuto piastre rivestite con 30 ng/mL retronectina (Takara) e bloccate con il 2% di albumina sierica umana. La sospensione di cellule B attivate in mezzo privo di siero è stato miscelato con lo stesso volume di retrovirus (in DMEM/F-12 con 8% FBS) e aggiunto ai pozzetti. Le piastre sono state centrifugate a temperatura ambiente per un’ora a 700 × g e poi incubate a 37 °C per 6 h a notte. Dopo l’incubazione, le cellule sono state lavate con IMDM completo e mantenuto con rhIL-21 e CD40L-L cellule. Al 7-10 days di cultura, l’espressione GFP è stata utilizzata come marcatore di trasduzione e abbiamo osservato un’efficienza specifica del soggetto del 25-70%. CD19 + GFP+ Le cellule B sono state poi ordinate a 50 cellule/pozzetti in piastre a 96 pozzetti contenenti rhIL-21 e 1 × 104 CD40L- L cellule in IMDM completo utilizzando un BD FACSAriaIII (utilizzando la modalità “single cell sort mask”, che fornisce la massima precisione possibile (<4% di varianza) nelle cellule di erogazione. Dopo la cultura per 2-3 weeks, le colture policlonali sono stati schermati per DENV-reattivo IgG da ELISA (vedi sotto). Il recupero di cellule policlonali ordinate era del 100% e la secrezione di IgG in coltura supernatante (media di 110 ng/mL) è stata confermata da ELISA di cattura (Capture Ab: AffiniPure goat anti-human IgG (Fcγ), Jackson Immunoresearch cat n. 109-005-008; rilevazione: Perossidasi AffiniPure Goat Anti-Human IgG (Fcγ), Fcγ fragment-specific, Jackson Immunoresearch cat no.109-035-008).
Clonazione mAb da MBC immortalato
Le colture policlonali selezionate specifiche per DENV2 sono state selezionate in monocolture monoclonali (stessa modalità di cui sopra) e sono state coltivate su CD40L e rhIL-21 e poi sottoposte a screening come sopra dopo tre settimane. Colture monoclonali DENV-specifiche DENV sono state ulteriormente testate qualitativamente per la neutralizzazione del DENV mediante incubazione del virus con 30 μL di supernatanti di coltura prima dell’infezione delle cellule Vero e la valutazione dell’attività di neutralizzazione mediante il saggio di micronutrizione (vedi sotto). Da pellet di cellule congelate di colture monoclonali abbiamo isolato l’RNA, eseguito la PCR nidificata per i geni IGH e IGL e sequenziato utilizzando primer specifici come descritto[51,52]. Le sequenze sono state inserite in IgBLAST(https://www.ncbi.nlm.nih.gov/igblast/) e confrontate con il germline per determinare l’uso della catena pesante e leggera variabile, l’uso del gene V(D)J, le ipermutazioni somatiche, la sequenza di regioni determinanti complementari (CDR)3 e il sottotipo IgG. Abbiamo clonato il VH in IgG1 umano (Genbank FJ475055) e VL in vettori di espressione Ig-λ o Ig-κ (FJ517647, FJ475056, rispettivamente). Pesanti e leggeri plasmidi a catena di codifica a catena sono stati verificati mediante sequenziamento e co-trasferiti in cellule HEK-293F (RRID: CVCL_6642) per produrre mAbs come descritto[51,52].
rDENV epitope-mappatura dei virus rDENV
I virus ricombinanti sono costruiti utilizzando una strategia di clonazione a quattro CDNA come descritto in precedenza[53]. In breve, il rDENV4/2 è stato creato introducendo la proteina dell’involucro (E) dominio III (EDIII) residui di DENV2 nel subclone DENV4 A (Sri Lanka 1992; GenBank KJ160504.1) e sostituendo i nucleotidi E da 900 a 1179 con i corrispondenti nucleotidi che codificano la variante degli amminoacidi DENV2[53]. rDENV2/1 è stato anche ottenuto in modo simile su una spina dorsale di clone infettivo DENV2 S16803. L’anticorpo monoclonale umano (hmAb) 1F4 è un anticorpo specifico del sierotipo DENV1 fortemente neutralizzante. Utilizzando la microscopia crio-elettronica, l’impronta di hmAb 1F4 è stata mappata su un epitopo quaternario che abbraccia i monomeri E di un dimero vicino alla regione della cerniera del dominio I/II dell’involucro. L’epitopo DENV1 1F4 epitopo è stato trapiantato nel clone infettivo DENV2 (S16803) cambiando 30 amminoacidi in DENV2 alle loro controparti in DENV1. Il trapianto risulta in un virus, rDENV2/1, che mantiene la neutralizzazione hmAb 2D22 specifica per il sierotipo DENV2 e guadagna completamente la neutralizzazione hmAb 1F4. I cDNA a lunghezza piena sono stati trascritti in RNA a lunghezza genomica utilizzando la polimerasi T7, come descritto in precedenza [[53], [54], [55]]]. Queste trascrizioni sono stati elettroporati in cellule C6/36 (RRID: CVCL_Z230), e la coltura cellulare supernatante contenente virus vitale è stato raccolto. Il virus è stato trasmesso due volte su colture monostrato di cellule C6/36 e conservato a 80 °C. Il virus Passage 3 è stato utilizzato come materiale di lavoro.
ELISA per rilevare virus, anticorpi rE e anticorpi rEDIII-binding
I virus non chimerici utilizzati negli schermi ELISA e nei test di neutralizzazione sono stati coltivati in cellule Vero-81 a 37 °C, come precedentemente descritto [26]. La proteina ricombinante dell’involucro (recE) (80% della proteina E) del ceppo di riferimento DENV2 S16803 dell’OMS è stata prodotta nel nostro laboratorio o acquistata da Hawaii Biotech, Inc.[56]. La proteina EDIII ricombinante da DENV2 è stata prodotta nel nostro laboratorio come descritto in precedenza[57]. Per la rilevazione di IgG reattivo a DENV intero da ELISA, ogni lotto di ogni sierotipo è stato titolato separatamente da ELISA utilizzando una quantità fissa di campione di siero altamente reattivo incrociato (DT000) per raggiungereOD405 = 1.0 per normalizzare tra i sierotipi e tra i lotti nei saggi ELISA. Quantità equivalenti di virus DENV sono state catturate da un topo legato a piastre anti-E mAb 4G2 durante la notte a 4 °C. Le proteine E ricombinanti sono state rivestite direttamente (rE – 100 ng/well; rEDIII – 200 ng/well) su piastre ELISA durante la notte a 4 °C [53]. Le piastre sono state bloccate con il 3% (vol/vol) di siero normale di capra (Gibco – ThermoFisher, USA), in Tris-buffered saline (TBS) contenente 0,05% (vol/vol) Tween 20 (buffer di blocco). IgG1 mAbs derivati da plasmablast sono stati testati ad una concentrazione fissa di 20 μg/ml. 50 μL di supernatante policlonale da cellule B di memoria immortalata è stato testato. Dopo quattro lavaggi, alcalino-fosfatasi coniugati con anticorpi secondari anti-IgG umane coniugati con alcalina sono stati utilizzati per rilevare il legame degli anticorpi primari con il substrato di p-nitrofenil fosfato, e le variazioni di colore di reazione a OD405 sono state quantificate dalla spettrofotometria.
Determinazione della qualità degli anticorpi neutralizzanti mediante esaurimento del siero immunitario con virus DENV
Per determinare se gli anticorpi specifici del sierotipo DENV2 o gli anticorpi cross-reattivi contribuiscono alla neutralizzazione globale del siero immunitario, abbiamo eseguito studi di deplezione degli anticorpi come descritto in precedenza[26]. I ceppi di riferimento dell’OMS per gli antigeni virali purificati sono i seguenti: DENV1 (genotipo americano; ceppo West Pac74), DENV2 (genotipo asiatico; ceppo S16803), (fornito da Robert Putnak, Walter Reed Army Institute of Research, Silver Spring, MD). Questi antigeni virali sono stati utilizzati per l’esecuzione delle deplezioni e anche ulteriormente utilizzati per i saggi di conferma ELISA e di neutralizzazione.
DENV purificato è stato adsorbito su microsfere di polistirene (Polysciences, Inc.) di 4,5 μm di diametro in microsfere di polistirolo (Polysciences, Inc.) con un rapporto di 5:2 perlina (μL) a ligando (μg). Le microsfere di polistirolo sono state lavate tre volte con tampone al borato 0.1 M (pH 8.5) e incubate con il relativo DENV purificato (DENV2 per impoverimenti omotipici e DENV1 per impoverimenti eterotipici) durante la notte a temperatura ambiente (RT). Le microsfere di controllo sono state incubate durante la notte con una quantità equivalente di sieroalbumina bovina (BSA). Le perle di controllo e le perle di virus-adsorbite sono state bloccate con tampone BSA (10 mg/ml)-borato per 30 min a RT tre volte e lavate quattro volte con soluzione fisiologica a base di fosfato (PBS). I sieri immuni DENV2 dei tre soggetti selezionati sono stati impoveriti di anticorpi specifici del virus incubando i campioni con perline virus-adsorbite per 1 h a 37 °C con miscelazione finale. I campioni sono stati sottoposti ad almeno tre cicli sequenziali di esaurimento prima che la rimozione dei rispettivi anticorpi fosse confermata dal test ELISA. La capacità dei campioni esauriti di neutralizzare i virus di tutti e quattro i sierotipi è stata testata dopo l’ELISA di conferma. La capacità di neutralizzazione dei sieri immuni DENV2 impoveriti è stata misurata utilizzando un test di neutralizzazione basato sulla citometria a flusso con cellule U937-DC-SIGN come descritto in precedenza[31].
Vero test di micronutrizione
Abbiamo adattato il FRNT come descritto in precedenza[53] a un formato a 96 pozzetti[58]. Le piastre a 96 pozzetti sono state rivestite con 2 × 104 Vero-81 celle/pozzetti e incubate a 37 °C per 24 h. Titoli di neutralizzazione sono stati determinati da tre diluizioni seriali di sieri miscelati con 50-100 unità di messa a fuoco per pozzetto in DMEM/F-12 integrato con il 2% di FBS. Attività di neutralizzazione di mAbs è stato testato in modo simile su un intervallo di 1-1000 ng/mL. Le miscele anticorpo-virus sono state incubate a 37 °C per 1 h prima di essere trasferite alle piastre a 96 pozzetti contenenti monostrato Vero-81 confluente. A seguito di un ulteriore incubazione di 1 -h a 37 °C, i monostrato sono stati sovrapposti con Opti-MEM (Gibco, ThermoFisher) contenente 2% FBS e 1% (wt / vol) carbossimetilcellulosa (Sigma). Le piastre infette sono state incubate per 2 days a 37 °C con il 5% di CO2, seguito dalla fissazione con 50 μl 4% di paraformaldeide per ogni pozzo. Le cellule fisse sono state poi permeabilizzate e bloccate con latte secco non grasso in tampone di permeabilizzazione. Poi le piastre sono state incubate con una miscela di anticorpi primari 4G2 e 2H2 per un’ora a 37 °C, lavate, seguite da perossidasi secondaria di rafano (HRP)- coniugata anti-topo IgG (KPL) per un’ora a 37 °C, lavate di nuovo, e sviluppate con substrato di perossidasi True Blue (KPL). La reazione è stata fermata con acqua e le piastre sono state asciugate all’aria prima di contare i focolai su un analizzatore Immunospot® S6 (Cellular Technology Limited) utilizzando il software di conteggio doppio Immunospot®. Abbiamo calcolato i valori IC50 utilizzando l’equazione della risposta della dose sigmoidale (pendenza variabile) di Prisma 6 (Software Graph Pad). I valori riportati sono stati richiesti per avere un R2 > 0.75, una pendenza di collina > 0.5, e un IC50 all’interno del range delle diluizioni.
Risultati
Abbiamo utilizzato un modello di infezione umana da DENV per analizzare le risposte degli anticorpi delle cellule B e del siero all’infezione da DENV. Il modello di infezione umana si basa su soggetti difficili con un ceppo parzialmente attenuato di DENV2 (rDEN2Δ30/Tonga/74). È stata pubblicata una descrizione completa delle caratteristiche virologiche e cliniche di questo modello[41,42]. Sono stati seguiti per un periodo di sei mesi soggetti sani e privi di flavivirus infettati da rDEN2Δ30 (Fig. S1).
Risposta al plasmablast a rDEN2Δ30 nei soggetti affetti da flavivirus-naïve
Ci siamo concentrati per la prima volta sui plasmablasti, una popolazione di cellule B che secernono anticorpi e che aumentano transitoriamente nel sangue precocemente dopo le infezioni virali[13], incluso il DENV[19,20,35,36]. Per determinare se un aumento di plasmablasti è stato indotto nel modello di infezione primaria da DENV2, abbiamo valutato le frequenze delle cellule CD19 + CD20- CD27 + CD38hi inPBMC crioconservatedi dodici soggetti infettati con rDEN2Δ30. I livelli di plasmablast sono aumentati a partire dall’ottavo giorno dopo l’infezione, hanno raggiunto il picco al quattordicesimo giorno e sono diminuiti al ventunesimo giorno(Fig. 1A, Fig. S2A), oscillando tra l’1,5 e il 20% della popolazione totale di cellule B CD19+ tra i soggetti, e aumentando in media di 8,7 volte (range 2,5-30 volte) dal giorno 0 alla risposta di picco (Fig. S2A). Abbiamo valutato la percentuale di cellule B periferiche nel totale PBMC per determinare se la variazione della percentuale di plasmablast era una funzione delle cellule B totali. In generale, la percentuale di cellule B periferiche è rimasta stabile prima e durante i 6 mesi successivi all’infezione(Fig. 1B, Fig. S2B e S3).Fig. 1Kinetics della risposta del plasmablasto alla DENV2 primaria. Analisi citometrica a flusso di(A) CD3- CD19 + CD20low/-CD27 + CD38hi plasmablasti e(B) totale CD19+ cellule Bin PBMC da soggetti infettati con rDEN2Δ30. Box e baffi trame (Tukey) sono mostrati (n = 12).(C,D) Il fold-rise in plasmablasts il giorno 14 contro la linea di base è stata tracciata contro(C) picco DENV2 carico virale e (D) DENV2 anticorpi neutralizzanti DENV2 misurata al picco dopo l’infezione (giorni 28-56) o al giorno 180 dopo l’infezione. Sono mostrati i coefficienti di correlazione R-squared di Pearson e i valori P.Fig. 1
Nelle persone esposte a infezioni naturali da DENV, i livelli di plasmablast sono stati correlati con il titolo virale di picco[19,59]. Analogamente, in questo modello di sfida, la carica virale di picco è stata correlata positivamente con l’aumento del plasmablasto(Fig. 1C). Abbiamo osservato una debole correlazione tra le variazioni dei livelli di plasmablasto e il picco del titolo anticorpale neutralizzante del siero(Fig. 1D), anche se il grado di correlazione non ha raggiunto un significato(P =0 .07).
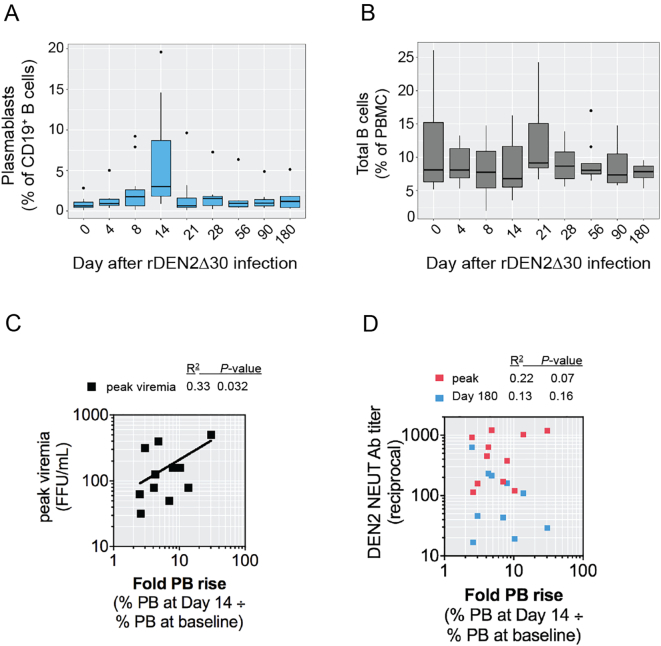
Fig. 1.Cinetica della risposta del plasmablastoma all’infezione primaria da DENV2. Analisi citometrica a flusso di(A) CD3- CD19 + CD20low/-CD27 + CD38hi plasmablasti e(B) totale CD19+ cellule Bin PBMC da soggetti infettati con rDEN2Δ30. Box e baffi trame (Tukey) sono mostrati (n = 12).(C,D) Il fold-rise in plasmablasts il giorno 14 contro la linea di base è stata tracciata contro(C) picco DENV2 carico virale e (D) DENV2 anticorpi neutralizzanti DENV2 misurata al picco dopo l’infezione (giorni 28-56) o al giorno 180 dopo l’infezione. Sono mostrati i coefficienti di correlazione R-squared di Pearson e i valori P.
La risposta del plasmablast all’infezione primaria da DENV2 è ampliata clonalmente e mostra un’evoluzione convergente tra i soggetti
Per tre individui in una gamma di viremia rDEN2Δ30 e dinamica dei plasmablasti (Tabella1) abbiamo eseguito una valutazione longitudinale approfondita di plasmablasti, MBC e anticorpi sierici negli stessi soggetti nel tempo. Per caratterizzare il repertorio degli anticorpi plasmablasti, i plasmablasti di ogni individuo sono stati ordinati a singola cellula in piastre a 96 pozzetti sulla base della colorazione della superficie cellulare per CD19 + CD20low/- CD27 + CD38hi IgA- IgM-. Un metodo di codifica a barre del DNA è stato utilizzato per ottenere le sequenze di anticorpi a catena pesante e leggera accoppiate da un totale di 1690 singole cellule dei tre soggetti (Tabella S1)[44,60]. Alberi filogenetici dei repertori di plasmablast dei soggetti sono stati generati sulla base dell’omologia della sequenza delle sequenze accoppiate a catena pesante e leggera (i rami colorati indicano l’uso di gene variabile a catena pesante, Fig. 2A). Solo lo 0,6% di queste cellule ha espresso anticorpi ingenui e non mutilati (dati non mostrati). I plasmablasti rimanenti hanno mostrato un tasso medio di 34 mutazioni nucleotidiche per coppia VH e VL(Fig. 2B), con una media di 21 sostituzioni di aminoacidi attraverso VH e VL per cellula per soggetto (non mostrato).Tabella 1Viremia e plasmablasti in tre riceventi rDEN2Δ30.Tabella 1SubjectDENV2 ViremiaDENV2 indotta da plasmablasti indotti da ViremiaDENV2Onset (giorno)Titolo del picco (log10 FFU/mL)Giorno del picco% Baseline% al giorno 14Fold-rise (D14/Baseline)Giorno del picco% al piccoFold-rise (Peak/Baseline)01081.5100.51.32.32.6212.75.402562.161.59.66.66.5149.66.503842.940.33.110.110.3143.110.3Fig. 2Evidenza di ipermutazione somatica e diversità nella risposta del plasmablasto alla rDEN2Δ30. Plasmablast dai punti di tempo di risposta di picco da tre destinatari rDEN2Δ30 sono stati ordinati a cella singola e il sequenziamento IGH / IGL è stato eseguito. Le sequenze IGH/IGL nativamente accoppiate sono state confrontate con le sequenze di germline e i filogrammi(A) sono stati costruiti per visualizzare la diversità IGH/IGL. La distanza dal centro è proporzionale all’ipermutazione somatica VH/VL accoppiata rispetto al germline. L’uso del gene VH è mostrato come rami colorati. Il numero di plasmablasti unici sequenziati da ogni donatore è indicato. Le stelle indicano le coppie VH/VL di sequenza anticorpale selezionate per l’espressione IgG1 ricombinante.(B) I valori mediani, medi e massimi per la somma delle mutazioni nucleotidiche tra le regioni VH e VL accoppiate in tutte le plasmablasti sono mostrati per ogni soggetto.(C) diagramma di Venn del numero totale di plasmablasti in sequenza (con il corrispondente numero di lignaggi), il numero di cellule contenute all’interno di lignaggi che mostrano segni di espansione (cioè lignaggi rappresentati da almeno 2 cellule), e il numero di cellule all’interno dei lignaggi espansi che sono stati selezionati (n = 96), e da cui i mAb sono stati ricombinanti espressi. Si noti che i mAbs espressi rappresentavano il 76% delle cellule dei lignaggi con evidenza di espansione e il 43% del repertorio complessivo di plasmablast in sequenza.(D) Alta identità di sequenza di aminoacidi attraverso IGH e IGL regioni variabili da singoli plasmablasti di due diversi soggetti sfidati con rDEN2Δ30. L’ombreggiatura del grigio nella sequenza allineata indica un disallineamento. Colorazione del logo della sequenza: verde, polare/neutro; nero, non polare/neutro; rosso, polare/acido; blu, polare/basico.(E) Sulla base delle caratteristiche della sequenza e del repertorio (vedi metodi), 96 lignaggi sono stati classificati come prioritari per la generazione di una libreria di screening degli anticorpi. Un singolo clone di anticorpi dei primi 96 di questi lignaggi è stato selezionato per l’espressione IgG ricombinante, incluse 12 coppie convergenti (#) tra due o più soggetti (notare i diversi colori che rappresentano i diversi soggetti in barre sovrapposte). Quattro mAbs non si sono espresse come proteine in quantità sufficiente (si noti con Ø), lasciando 92 mAbs (di cui 11 convergenti) per i test funzionali. (Per l’interpretazione dei riferimenti al colore in questa legenda di figura, il lettore è riferito alla versione web di questo articolo.)Fig. 2
In tutti e tre i soggetti sono stati osservati esempi di lignaggi di plasmablast clonalmente espansi. Le 1690 sequenze della coppia IGH/IGL rappresentavano 923 lignaggi basati sulla somiglianza della sequenza nucleotidica (clonale)(Fig. 2C). Un quarto dei lignaggi (211 di 923) sono stati rappresentati da almeno due plasmablasti e si è ritenuto che abbiano subito un’espansione proliferativa (cioè una selezione in vivo ). Mentre la maggior parte dei plasmablasti sequenziati (978/1690, 58% del totale) appartenevano a lignaggi espansi, il resto delle cellule (712/1690, 42% del totale) apparteneva a lignaggi composti da una sola cellula (singoletti) (Tabella S2). Dei lignaggi espansi, abbiamo identificato 48 lignaggi che mostrano un’evoluzione convergente. I membri dei lignaggi convergenti hanno condiviso i geni della linea germinale IGHV/IGLV, le lunghezze CDRH3 e CDRL3, ed hanno mostrato un’identità ≥75% attraverso il peptide VH o VL maturo. Un esempio di queste coppie convergenti IGH/IGL è mostrato nella Fig.2D. Presi insieme, questi dati mostrano che l’infezione primaria DENV2 ha suscitato un pool diversificato di plasmablasti che mostrano l’espansione clonale, l’ipermutazione somatica e la popolazione di lignaggi convergenti condivisi tra i soggetti.
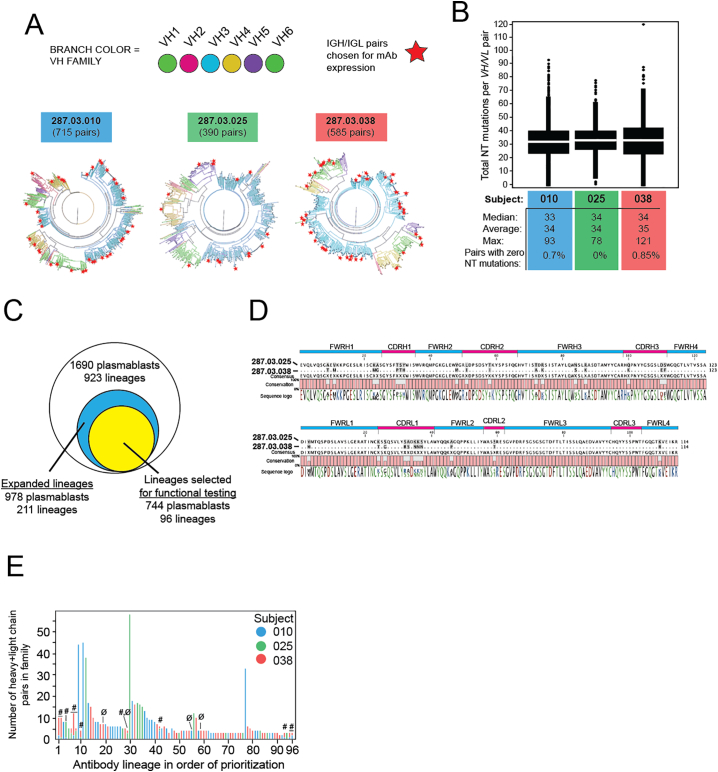
Fig. 2.Fig. 2. Evidenza di ipermutazione somatica e diversità nella risposta del plasmablasto alla rDEN2Δ30. I plasmablasti dai punti di picco dei tempi di risposta da tre riceventi rDEN2Δ30 sono stati ordinati a singola cellula e il sequenziamento IGH/IGL è stato eseguito. Le sequenze IGH/IGL nativamente accoppiate sono state confrontate con le sequenze di germline e i filogrammi(A) sono stati costruiti per visualizzare la diversità IGH/IGL. La distanza dal centro è proporzionale all’ipermutazione somatica VH/VL accoppiata rispetto al germline. L’uso del gene VH è mostrato come rami colorati. Il numero di plasmablasti unici sequenziati da ogni donatore è indicato. Le stelle indicano le coppie VH/VL di sequenza anticorpale selezionate per l’espressione IgG1 ricombinante.(B) I valori mediani, medi e massimi per la somma delle mutazioni nucleotidiche tra le regioni VH e VL accoppiate in tutte le plasmablasti sono mostrati per ogni soggetto.(C) diagramma di Venn del numero totale di plasmablasti in sequenza (con il corrispondente numero di lignaggi), il numero di cellule contenute all’interno di lignaggi che mostrano segni di espansione (cioè lignaggi rappresentati da almeno 2 cellule), e il numero di cellule all’interno dei lignaggi espansi che sono stati selezionati (n = 96), e da cui i mAb sono stati ricombinanti espressi. Si noti che i mAbs espressi rappresentavano il 76% delle cellule dei lignaggi con evidenza di espansione e il 43% del repertorio complessivo di plasmablast in sequenza.(D) Alta identità di sequenza di aminoacidi attraverso IGH e IGL regioni variabili da singoli plasmablasti di due diversi soggetti sfidati con rDEN2Δ30. L’ombreggiatura del grigio nella sequenza allineata indica un disallineamento. Colorazione del logo della sequenza: verde, polare/neutro; nero, non polare/neutro; rosso, polare/acido; blu, polare/basico.(E) Sulla base delle caratteristiche della sequenza e del repertorio (vedi metodi), 96 lignaggi sono stati classificati come prioritari per la generazione di una libreria di screening degli anticorpi. Un singolo clone di anticorpi dei primi 96 di questi lignaggi è stato selezionato per l’espressione IgG ricombinante, incluse 12 coppie convergenti (#) tra due o più soggetti (notare i diversi colori che rappresentano i diversi soggetti in barre sovrapposte). Quattro mAbs non si sono espresse come proteine in quantità sufficiente (si noti con Ø), lasciando 92 mAbs (di cui 11 convergenti) per i test funzionali. (Per l’interpretazione dei riferimenti al colore in questa legenda di figura, il lettore è riferito alla versione web di questo articolo).
Proprietà leganti degli anticorpi derivati dal plasmablast
Per stimare la misura in cui i plasmablasti che si sono espansi a seguito dell’infezione primaria da DENV2 sono stati in grado di riconoscere l’antigene virale, abbiamo selezionato 96 coppie IGH/IGL per l’espressione ricombinante e il test per il legame e la neutralizzazione della DENV(Fig. 2C). Questo numero è stato scelto come equilibrio tra un’adeguata copertura dei lignaggi espansi (96 su 211, ovvero il 45%), le risorse e la conformità a una procedura di clonazione basata su micropiastra. All’interno di questo gruppo di lignaggi espansi selezionati per i test funzionali, abbiamo identificato 12 lignaggi convergenti(Fig. 2E, Tabella S3). 92 sequenze hanno prodotto proteine sufficienti per ulteriori test. Questi 92 lignaggi contenevano un totale di 741 cellule, che è il 76% del numero totale di cellule dei 211 lignaggi espansi (978 cellule) nel set di dati (Tabella S2). Anche se tutti i 92 anticorpi sono stati espressi in modo ricombinante come IgG1, essi sono stati derivati da anticorpi nativi che erano o IgG1 (83/92, 90%), IgG2 (3/92, 3%), o isotipi IgG3 (6/92, 7%). La selezione finale del test includeva mAbs di tutti e tre i soggetti, con 11 sequenze convergenti condivise tra almeno due soggetti(Fig. 2E). Il quarantacinque per cento (41/92) degli anticorpi ricombinanti espressi legati all’antigene DENV2 Tonga/74(Fig. 3A). Il 32% degli anticorpi legati alla DENV, il 32% erano cross-reattivi al sierotipo DENV, mentre il 68% erano legati solo alla DENV2 (sierotipo-specifico)(Fig. 3B). Delle undici sequenze convergenti, sei anticorpi (55%) legati all’antigene DENV2 e uno era neutralizzante (Tabella S3). Inoltre, per ogni soggetto, abbiamo determinato la percentuale di plasmablasti nei lignaggi che producono anticorpi legati al DENV tra il totale dei plasmablasti nei lignaggi selezionati per l’espressione ricombinante (Tabella S2). Nei soggetti 010, 025 e 038, le proporzioni dei plasmablasti nei lignaggi positivi al DENV erano rispettivamente del 32%, 70% e 60%. Nel complesso, i lignaggi che abbiamo testato per il legame che conteneva cloni reattivi DENV2 rappresentavano il 54% dei plasmablasti tra i plasmablasti in tutti i lignaggi testati.Fig. 3Proprietà di legame degli anticorpi derivati dai plasmablasti.(A) Sono mostrate la frazione di colpi reattivi del plasmablast derivato dal DENV reattivo e anticorpi non reattivi e la proporzione di leganti specifici del sierotipo DENV2 e di leganti cross-reattivi del DENV2.(B) Specificità del sierotipo di mAbs DENV2-positivo (a sinistra) e indicazione delle specificità del ceppo DENV2 (al centro). Di quei mAbs che si legano al ceppo di riferimento OMS S16803, viene mostrata la proporzione che si lega al ceppo S16803-derivato recE (a destra).(C). Sono raffigurate le proporzioni dei mAb cross-reattivi che si legano a recE.(D) Modello strutturale del dimero della proteina DENV4 E, con residui scambiati di EDIII di DENV2 (Nicaragua-694) colorati in verde per rappresentare la glicoproteina rDENV4/2 (virus DENV2 EDIII) E e il sierotipo DENV2 specifico del mAb 2D22 sfuggono ai residui mutanti evidenziati in magenta.(E) Modello strutturale di DENV2 (S16803) dimero proteico E con DENV1-derivato EDI e EI/II residui di cerniera scambiato colorato in verde per rappresentare il virus rDENV2/1 virus E glicoproteina E.(F) Otto mAb specifici DENV2 specifici per DENV2 che si legano ai virus S16803 e Nicaragua-694 DENV2 sono stati testati per il legame con EDIII ricombinante (derivato da S16803) così come rDENV4/2 e rDENV2/1 virus chimerico. Sono indicati il numero e la proporzione di mAbs che si legano agli antigeni indicati. D: mAbs epitope-targeting quaternario. (Per l’interpretazione dei riferimenti al colore in questa legenda di figura, il lettore è rinviato alla versione web di questo articolo.)Fig. 3
Successivamente, abbiamo confrontato il legame dei mAbs specifici del sierotipo DENV2 con diversi ceppi di DENV2 rappresentati da Tonga/74 (ceppo di sfida, genotipo americano), Nuova Guinea C (genotipo asiatico II), S16803 (genotipo asiatico I) e Nicaragua 694 (genotipo asiatico/americano). Il cinquantasei percento (14/25) degli anticorpi specifici del sierotipo DENV2 testato legati esclusivamente al ceppo Tonga/74, mentre i restanti anticorpi legati ad almeno due ceppi DENV2 (Fig. 3B). Questi risultati dimostrano che la risposta precoce del plasmablastoma all’infezione primaria da DENV2 è una miscela di anticorpi specifici del sierotipo DENV e di anticorpi cross-reattivi. Inoltre, quasi la metà degli anticorpi specifici del sierotipo sono specifici del ceppo e riconoscono gli epitopi visualizzati sul virus DENV2 Tonga/74 ma non altri ceppi DENV2 strettamente correlati.
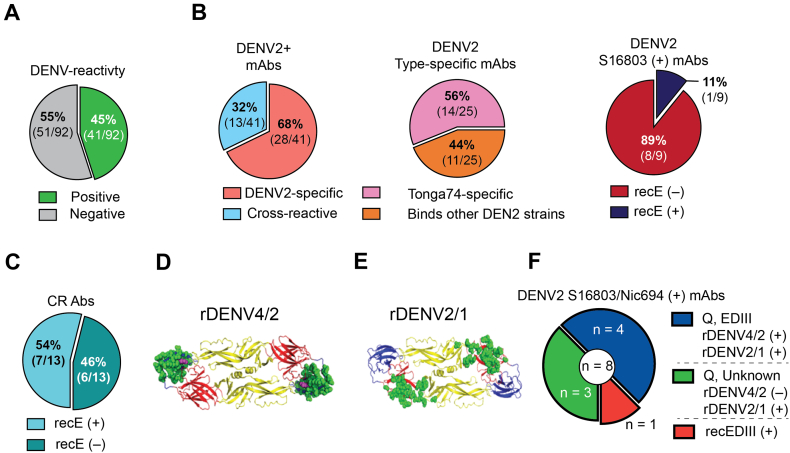
Fig. 3.Proprietà di legame degli anticorpi derivati dal plasmablasto.(A) Sono mostrate la frazione di colpi reattivi del DENV plasmablast-derivato da plasmablast e anticorpi non reattivi e la proporzione di leganti specifici del sierotipo DENV2 e di leganti cross-reattivi del DENV2.(B) Specificità del sierotipo di mAbs DENV2-positivo (a sinistra) e indicazione delle specificità del ceppo DENV2 (al centro). Di quei mAbs che si legano al ceppo di riferimento OMS S16803, viene mostrata la proporzione che si lega al ceppo S16803-derivato recE (a destra).(C). Sono raffigurate le proporzioni dei mAb cross-reattivi che si legano a recE.(D) Modello strutturale del dimero della proteina DENV4 E, con residui scambiati di EDIII di DENV2 (Nicaragua-694) colorati in verde per rappresentare la glicoproteina rDENV4/2 (virus DENV2 EDIII) E e il sierotipo DENV2 specifico del mAb 2D22 sfuggono ai residui mutanti evidenziati in magenta.(E) Modello strutturale di DENV2 (S16803) dimero proteico E con DENV1-derivato EDI e EI/II residui di cerniera scambiato colorato in verde per rappresentare il virus rDENV2/1 virus E glicoproteina E.(F) Otto mAb specifici DENV2 specifici per DENV2 che si legano ai virus S16803 e Nicaragua-694 DENV2 sono stati testati per il legame con EDIII ricombinante (derivato da S16803) così come rDENV4/2 e rDENV2/1 virus chimerico. Sono indicati il numero e la proporzione di mAbs che si legano agli antigeni indicati. D: mAbs epitope-targeting quaternario. (Per l’interpretazione dei riferimenti al colore in questa legenda di figura, il lettore è rinviato alla versione web di questo articolo).
Mappatura degli epitopi riconosciuti dagli anticorpi DENV specifici per il plasmablasto
Abbiamo eseguito studi di mappatura più dettagliati con gli anticorpi specifici del sierotipo DENV2. Utilizzando proteine ricombinanti e reagenti del virus chimerico basati sul ceppo DENV2 S16803, abbiamo mappato gli epitopi di 9 anticorpi specifici del sierotipo DENV2 che si legano a S16803 (dei 25 cloni specifici del sierotipo che si legano a DENV2 Tonga/74). La maggior parte (8/9) di questi mAb specifici del sierotipo non si è legata ai monomeri della proteina E ricombinante espressa (recE) derivati dallo stesso ceppo, indicando che gli epitopi E della struttura quaternaria visualizzati sui virioni intatti sono probabili bersagli di questi anticorpi(Fig. 3B). L’unico anticorpo specifico del sierotipo che ha legato la recE ha riconosciuto un semplice epitopo su EDIII (dati non mostrati). Abbiamo anche testato i 13 anticorpi cross-reattivi DENV per il legame con la recE. Il 54 per cento (7/13) di questi anticorpi legati alla recE dimostra che alcuni anticorpi cross-reattivi legati a epitopi semplici sulla proteina E monomerica, mentre altri hanno riconosciuto la proteina prM o epitopi della proteina E solo su virioni intatti(Fig. 3C).
Per mappare ulteriormente gli epitopi bersaglio degli anticorpi specifici del sierotipo DENV2, abbiamo eseguito test di legame utilizzando virus DENV chimerici ricombinanti DENV con glicoproteine E mutate. rDENV4/2 è un ceppo DENV4 in cui l’intero DENV4 EDIII è stato sostituito con EDIII da DENV2-Nicaragua 694(Fig.3D). Questo virus ricombinante è stato utilizzato per mappare il legame degli anticorpi specifici del sierotipo che colpiscono gli epitopi semplici o complessi concentrati su DENV2 EDIII[53]. rDENV2/1 è un virus DENV2 (S16803) in cui i residui nell’EDI e nella regione di cerniera tra EDI ed EDII sono stati mutati per visualizzare gli epitopi per l’epitopo per il sierotipo specifico del DENV1 e neutralizzare l’anticorpo umano 1F4(Fig. 3E). Abbiamo quindi selezionato 8 mAbs che riconoscono sia il ceppo S16803 che quello Nicaragua-694 per l’analisi. Quattro degli 8 mAb diretti a epitopi quaternari specifici per DENV2(Fig. 3B) hanno mostrato un legame con rDENV4/2 e non hanno mostrato alcuna perdita apprezzabile di legame con il ceppo rDENV2/1, stabilendo epitopi quaternari centrati su DENV2 EDIII come obiettivo principale del legame(Fig. 3F, mostrata in blu). Un mAb era DENV2 recE-reattivo e mirava a semplici epitopi monomerici semplici su EDIII ricombinante(Fig. 3F, indicata in rosso). I rimanenti tre mAb legati a rDENV2/1 ma non a rDENV4/2 indicano che i residui su EDIII o sulla regione dei cardini EDI/II non erano probabilmente residui critici per il legame(Fig. 3F, in verde). Nel complesso, questi risultati dimostrano che la maggior parte dei mAb cross-reattivi derivati dal plasmablast hanno preso di mira gli epitopi semplici sulla proteina recE mentre i mAb specifici del sierotipo riconoscono gli epitopi della struttura quaternaria centrati su EDIII, comprese le nuove regioni che non mappano gli epitopi mAb conosciuti specifici di DENV2 come quelli di 2D22 [61].
Neutralizzazione funzionale della DENV2 mediante mAb derivati dal plasmablasto
Abbiamo testato tutti i mAb legati al DENV in un test di neutralizzazione DENV2 e abbiamo identificato cinque mAb neutralizzanti, con potenze (valutate da concentrazioni IC50) che vanno da 501700 ng/mL (Fig. 4A).Quattro di cinque erano specifici per il sierotipo DENV2(Tabella 2), compreso il mAb 1662, con una potenza che rivaleggia con il 2D22, un forte mAb neutralizzante DENV2[31]. Questi cinque mAb neutralizzanti non erano specifici per il ceppo DENV2 Tonga74. Quattro dei cinque mAb legati a tutti i ceppi/genotipi DENV2 testati nello studio. Solo un mAb (1615) non si è legato al ceppo S16803, ma a tutti gli altri ceppi. Inoltre, i tre mAb neutralizzanti specifici per sierotipo che si sono legati a epitopi su tutti i virioni DENV2 e non a recE hanno mostrato un guadagno di legame al virus rDENV4/2 e non hanno perso il legame a rDENV2/1. Questi risultati mostrano che i tre mAbs neutralizzanti specifici del sierotipo hanno preso di mira gli epitopi quaternari nella regione EDIII. Nella Fig. 4B-D, abbiamo mappato tutti i colpi di neutralizzazione, cross-reattivi e specifici del sierotipo DENV2 da ciascuno dei tre soggetti agli alberi filogenici dei plasmablasti, ma non siamo stati in grado di discernere l’uso particolare di VH o le caratteristiche del lignaggio clonale predittivo dell’attività funzionale.Fig. 4DENV2 neutralizza gli anticorpi neutralizzanti dei plasmablasti.(A) I saggi di microneutralizzazione di Vero sono stati condotti con tutti i colpi reattivi di DENV2 per determinare la neutralizzazione di DENV2 Tonga 74. Sono state mostrate curve di neutralizzazione per cinque anticorpi neutralizzanti. Ogni punto rappresenta il valore medio di neutralizzazione ± errore standard della media di almeno due esperimenti.,.(B-D) Sono mostrati gli alberi filogenetici che rappresentano le proprietà degli anticorpi testati in tutti e tre i soggetti.Fig. 4Table 2Detailed epitope mapping e la caratterizzazione funzionale dei mAb neutralizzanti DENV2 derivati da plasmablasti.Tabella 2mAbSubjectBinds DEN2 TongaarecE/QbrEDIII bindingcTS/CRdNEUT50 (μg/mL)e1662010+Q-TS0.0541652038+recE+CR0.2451671038+QNBTS0.281615010+NDNDNDTS0.591628038+QNBTS1.764aIntero virus ELISA, “+” = O.D. ≥ 2 volte sopra vuoto.bbbbinding a ricombinante E da DENV2 (recE). Se positivo, annotato come “recE”. Se negativo, annotato come epitopo quaternario (Q). ND, non determinato in quanto questo anticorpo non si lega al ceppo parentale WHO S16803 da cui deriva recE.cbinding to recombinant E domain III from DENV2 (rEDIII). NB, nessun legame; ND, non determinato.d whole virus ELISA. Se legato solo a DENV2, annotato come sierotipo specifico (TS); annotato come cross-reattivo (CR) se legato a DENV2 più almeno un altro sierotipo found.emicroneutralization test utilizzando cellule Vero, NEUT50 è la concentrazione di mAb necessaria per una riduzione ≥50% dell’infezione virale rispetto al controllo negativo.
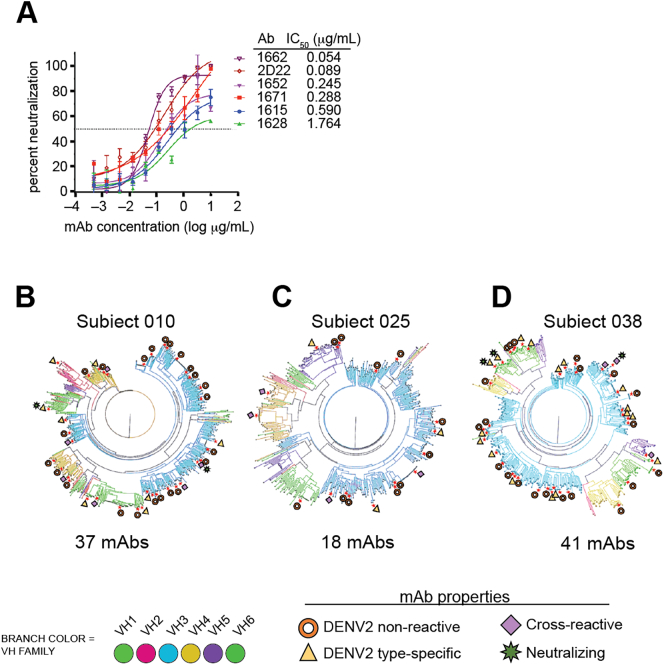
Fig. 4.DENV2 neutralizzare gli anticorpi da plasmablasti.(A) I saggi di microneutralizzazione Vero sono stati condotti con tutti i colpi reattivi DENV2 per determinare la neutralizzazione di DENV2 Tonga 74. Sono state mostrate curve di neutralizzazione per cinque anticorpi neutralizzanti. Ogni punto rappresenta il valore medio di neutralizzazione ± errore standard della media di almeno due esperimenti.,.(B-D) Sono mostrati gli alberi filogenetici che rappresentano le proprietà degli anticorpi testati in tutti e tre i soggetti.
Memoria cellule B a seguito di infezione primaria DENV2
Il nostro obiettivo successivo era quello di valutare le proprietà delle cellule B della memoria (MBC) sei mesi dopo l’infezione primaria da DENV2 in questi tre soggetti. Abbiamo utilizzato in precedenza descritto l’approccio di riprogrammazione genetica basata sulla trasduzione retrovirale del linfoma a cellule B (BCL)-6 e la proteina anti-apoptotica BCL-xL (insieme noto come 6XL) per immortalare MBC dai tre soggetti[50]. Per stimare la frazione del MBC che produce gli anticorpi che legano la DENV, abbiamo schermato i supernatanti di coltura contenenti immunoglobuline da cellule trasdotte 6XL da DENV capture ELISA (Fig. S4, Tabella S4)[26,31]. Le frequenze di MBC specifiche per la DENV sono state calcolate utilizzando il numero di colture positive e il numero totale di cellule B trasdotte ordinate. Abbiamo stimato una media dello 0,4% (219/52800) di MBC totali specifici per la DENV, con il più alto responder, il soggetto 287.03.025, che mostra una frequenza dello 0,74% (89 colture positive su 12.000 cellule totali 6XL+ placcate) MBC DENV2 reattivo seguito da 287.03.010 con una frequenza dello 0,34% (98 positivi su 28.800 cellule 6XL+), e 287.03.038 con una frequenza dello 0,28% (32 positivi su 12.000 cellule 6XL+)(Fig. 5A).Fig. 5Elicitazione di cellule B della memoria specifica della DENV da infezione da rDEN2Δ30.(A) Totale CD19 + IgM-CD27+ switched MBC sono stati isolati sei mesi dopo l’infezione rDEN2Δ30 negli stessi tre soggetti (287.03.010, 287.03.025, e 287.03.038) da cui sono state condotte le analisi del plasmablasto. MBC da ogni soggetto sono stati immortalati con un retrovirus codifica BCL6, Bcl-xL (6XL) e la proteina fluorescente verde marker di trasduzione (GFP). 6XL-immortalized MBC sono stati ordinati da FACS per GFP e coltivate a 50 cellule / pozzo; supernatanti sono stati sottoposti a screening per il legame IgG a DENV2 Tonga/74. Per ogni soggetto, la frequenza di DENV-immortalizzato MBC è stato calcolato dal numero di pozzi positivi diviso per il numero totale di 6XL + cellule coltivate e basato sulla presenza di una media di un clone positivo che produce il segnale positivo per 50 cellule in ingresso.(B) Da ogni soggetto, DENV2-binding MBC sono stati controllati per la reattività ad altri sierotipi da ELISA. Le proporzioni (e numeri) di 219 colture MBC che erano DENV2 sierotipo specifico o cross-reattivo con almeno un altro sierotipo (cross-reattivo) sono presentati. Vengono inoltre mostrate le proporzioni medie delle colture specifiche per sierotipo rispetto a quelle cross-reattive nei tre soggetti. (Per l’interpretazione dei riferimenti al colore in questa legenda di figura, il lettore è rinviato alla versione web di questo articolo)Fig. 5
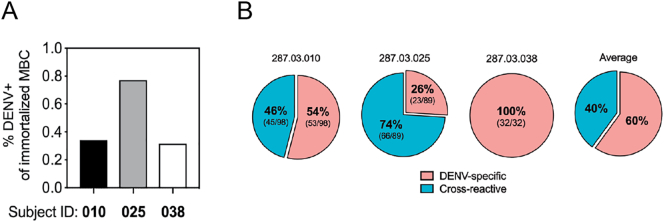
Fig. 5.Elicitazione delle cellule B della memoria specifica della DENV mediante infezione da rDEN2Δ30.(A) Totale CD19 + IgM-CD27+ switched MBC sono stati isolati sei mesi dopo l’infezione rDEN2Δ30 negli stessi tre soggetti (287.03.010, 287.03.025, e 287.03.038) da cui sono state condotte le analisi del plasmablast. MBC da ogni soggetto sono stati immortalati con un retrovirus codifica BCL6, Bcl-xL (6XL) e la proteina fluorescente verde marker di trasduzione (GFP). 6XL-immortalized MBC sono stati ordinati da FACS per GFP e coltivate a 50 cellule / pozzo; supernatanti sono stati sottoposti a screening per il legame IgG a DENV2 Tonga/74. Per ogni soggetto, la frequenza di DENV-immortalizzato MBC è stato calcolato dal numero di pozzi positivi diviso per il numero totale di 6XL + cellule coltivate e basato sulla presenza di una media di un clone positivo che produce il segnale positivo per 50 cellule in ingresso.(B) Da ogni soggetto, DENV2-binding MBC sono stati controllati per la reattività ad altri sierotipi da ELISA. Le proporzioni (e numeri) di 219 colture MBC che erano DENV2 sierotipo specifico o cross-reattivo con almeno un altro sierotipo (cross-reattivo) sono presentati. Vengono inoltre mostrate le proporzioni medie delle colture specifiche per sierotipo rispetto a quelle cross-reattive nei tre soggetti. (Per l’interpretazione dei riferimenti al colore in questa legenda delle figure, il lettore è rinviato alla versione web di questo articolo).
Specificità vincolante degli anticorpi derivati da MBC
Abbiamo esaminato i 219 supernatanti della cultura DENV2-leganti MBC di questi tre soggetti per determinare la misura in cui questi anticorpi hanno reagito in modo incrociato con altri sierotipi DENV. Abbiamo trovato sia MBC specifici per sierotipo che MBC cross-reattivi con reattività bi-, tri- e tetravalente. La frazione specifica del sierotipo della risposta MBC DENV2-reattiva era dipendente dal donatore; e su tre soggetti, una media del 60% di MBC DENV2-reattivo era specifica del sierotipo DENV2 e il 40% era cross-reattiva con DENV2 e almeno un altro sierotipo(Fig. 5B).
Da uno dei soggetti (287.03.010) abbiamo isolato quattro mAb DENV2-specifici, uno dei quali neutralizzante, che rappresentava quattro lignaggi distinti (Tabella S5). Nessuno di questi accoppiato IGH / IGL sequenze è stato identificato nei plasmablasti campionati da questo soggetto. Questa mancanza di sovrapposizione clonale tra i due compartimenti cellulari B è stata osservata in uno studio precedente di dengue secondaria[40]. Presi insieme, questi dati dimostrano il mantenimento funzionale della risposta umorale specifica del sierotipo in convalescenza dopo l’infezione primaria da DENV.
Qualità degli anticorpi neutralizzanti del siero policlonale convalescente dopo l’infezione da DENV2
Successivamente, abbiamo caratterizzato le proprietà della fase di convalescenza (6 months dopo l’infezione) anticorpi neutralizzanti del siero. Tutti e tre i soggetti hanno sviluppato anticorpi neutralizzanti che hanno neutralizzato il DENV2 ma non il DENV1, 3 e 4(Fig. 6A). I sieri immunitari hanno neutralizzato efficacemente il ceppo infettivo DENV2 (Tonga/74) e altri due ceppi appartenenti a diversi genotipi di DENV2 (NGC e S16803)(Fig. 6B, Tabella S6). Abbiamo eseguito studi di deplezione degli anticorpi per definire ulteriormente i livelli di anticorpi neutralizzanti specifici del sierotipo DENV2 e di anticorpi neutralizzanti cross-reattivi. Sono state incubate perline di polistirolo rivestite con i sierotipi omotipici (DENV2) o eterotipici (DENV1) per esaurire specifiche popolazioni di anticorpi. L’esaurimento degli anticorpi è stato confermato dall’ELISA prima di utilizzare i campioni nei test di neutralizzazione (dati non mostrati). L’esaurimento dei campioni di siero Day 180 con l’antigene omotipico DENV2 ha portato alla rimozione di quasi tutti gli anticorpi specifici del DENV (DENV2 sierotipo-specifici e reattivi incrociati) nei campioni, oltre a una significativa perdita di attività neutralizzante. Quando solo gli anticorpi cross-reattivi del sierotipo DENV sono stati esauriti in ogni campione (esaurimento con l’antigene DENV1), c’è stata una riduzione minima della neutralizzazione del DENV2(Fig. 6C-E). In media ~89% dell’attività di neutralizzazione del siero tracciata con anticorpi specifici del sierotipo DENV2(Fig. 6F).Fig. 6 Le proprietà degli anticorpi neutralizzanti del siero convalescente dopo l’infezione da DENV2.(A) 6 months dopo l’infezione, i sieri dei tre soggetti sono stati testati contro il DENV1-4 in un test di neutralizzazione.(B) I sieri provenienti da punti temporali al giorno 0, 56 e 180 dei tre soggetti sono stati testati per la capacità di neutralizzare il DENV2 Tonga/74 (ceppo di sfida, genotipo americano) e la Nuova Guinea C (genotipo asiatico II) in un test di micronutrizione. Per A e B, le linee tratteggiate indicano il fattore di diluizione del siero più basso utilizzato (1:10) nei saggi di neutralizzazione e anche la soglia oltre la quale un titolo sierico è considerato positivo.(C-E) 6 months I sieri post infezione erano privi di anticorpi che si legano all’antigene DENV2 o all’antigene DENV1. Le deplezioni di controllo sono state eseguite utilizzando l’albumina di siero bovino come antigene. I risultati presentati qui sono da 2 repliche tecniche in un esperimento. Le curve di neutralizzazione sono contro l’antigene DENV2 per ogni soggetto. Ogni punto delle curve di neutralizzazione rappresenta il valore medio di neutralizzazione dei due replicati e le barre di errore rappresentano la deviazione standard. Le linee tratteggiate rappresentano la neutralizzazione al 50%.(F) Tutti e tre i soggetti sviluppano anticorpi specifici del sierotipo DENV2 che contribuiscono principalmente alla neutralizzazione DENV2.Fig. 6
Per caratterizzare ulteriormente gli epitopi bersaglio degli anticorpi neutralizzanti specifici per il sierotipo DENV2, ottenuti dall’infezione da rDEN2Δ30, abbiamo eseguito dei test di neutralizzazione utilizzando i virus rDENV2/1 e rDENV4/2. Abbiamo anche eseguito i test con un pannello aggiuntivo di dieci campioni di siero raccolti da individui che hanno ricevuto la sfida rDEN2Δ30. I campioni raccolti all’indirizzo 180 days dopo l’infezione sono stati testati per la capacità di neutralizzazione contro i virus rDENV2/1 e rDENV4/2 e i virus parentali (Fig.7). Tutti i campioni analizzati hanno neutralizzato bene il DENV2 e il DENV1 e quattro malamente. Il virus rDENV2/1 che mostrava l’epitopo DENV1 1F4 è stato neutralizzato allo stesso livello del DENV2, dimostrando che la regione EDI sul DENV2 perturbata dal trapianto dell’epitopo DENV1 1F4 non era un bersaglio importante per la neutralizzazione degli anticorpi indotti nelle persone infettate dal rDEN2Δ30. La risposta anticorpale neutralizzante indotta da rDEN2Δ30 è stata diretta principalmente agli epitopi centrati su EDIII perché rDENV4/2 che mostrava EDIII da DENV2 è stato neutralizzato allo stesso livello della DENV2.Fig. 7 Gli anticorpi neutralizzanti specifici del sierotipo DENV2 sono stati indirizzati agli epitopi centrati nella regione EDIII. I saggi di neutralizzazione sono stati eseguiti utilizzando un pannello di sieri policlonali di soggetti che hanno ricevuto il ceppo Tonga 74 DENV2 utilizzando i virus chimerici e di tipo selvaggio rDENV4/2 e rDENV2/1. Ogni colore rappresenta un singolo campione e la neutralizzazione contro ogni virus (asse x). Le barre indicano i titoli geometrici medi e gli intervalli di confidenza del 95% dei campioni raggruppati. I valori P sono stati calcolati da Kruskal-Wallis seguito dal test post-hoc di Dunn.Fig. 7
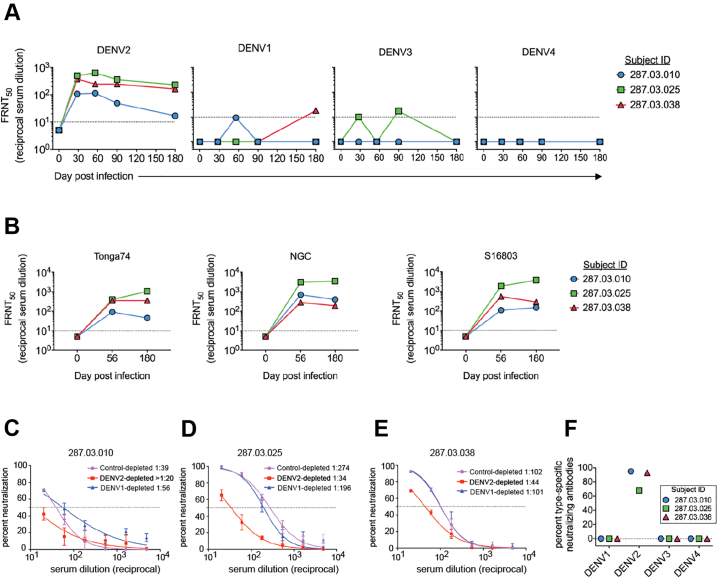
Fig. 6.Le proprietà degli anticorpi neutralizzanti del siero convalescente dopo l’infezione da DENV2.(A) 6 months dopo l’infezione, i sieri dei tre soggetti sono stati testati contro il DENV1-4 in un test di neutralizzazione.(B) I sieri provenienti da punti temporali al giorno 0, 56 e 180 dei tre soggetti sono stati testati per la capacità di neutralizzare il DENV2 Tonga/74 (ceppo di sfida, genotipo americano) e la Nuova Guinea C (genotipo asiatico II) in un test di micronutrizione. Per A e B, le linee tratteggiate indicano il fattore di diluizione del siero più basso utilizzato (1:10) nei saggi di neutralizzazione e anche la soglia oltre la quale un titolo sierico è considerato positivo.(C-E) 6 months I sieri post infezione erano privi di anticorpi che si legano all’antigene DENV2 o all’antigene DENV1. Le deplezioni di controllo sono state eseguite utilizzando l’albumina di siero bovino come antigene. I risultati presentati qui sono da 2 repliche tecniche in un esperimento. Le curve di neutralizzazione sono contro l’antigene DENV2 per ogni soggetto. Ogni punto delle curve di neutralizzazione rappresenta il valore medio di neutralizzazione dei due replicati e le barre di errore rappresentano la deviazione standard. Le linee tratteggiate rappresentano la neutralizzazione al 50%.(F) Tutti e tre i soggetti sviluppano anticorpi specifici del sierotipo DENV2 che contribuiscono principalmente alla neutralizzazione DENV2.
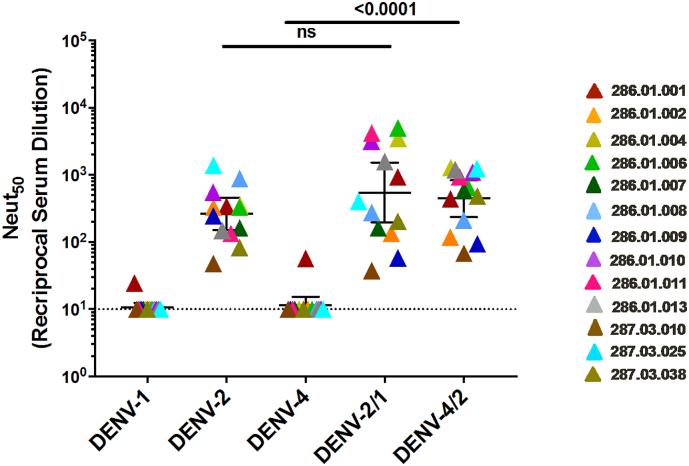
Fig. 7.Gli anticorpi neutralizzanti specifici del sierotipo DENV2 mirano ad epitopi concentrati nella regione EDIII. I saggi di neutralizzazione sono stati eseguiti utilizzando un pannello di sieri policlonali di soggetti che hanno ricevuto il ceppo Tonga 74 DENV2 utilizzando i virus chimerici e di tipo selvaggio rDENV4/2 e rDENV2/1. Ogni colore rappresenta un singolo campione e la neutralizzazione contro ogni virus (asse x). Le barre indicano i titoli geometrici medi e gli intervalli di confidenza del 95% dei campioni raggruppati. I valori P sono stati calcolati da Kruskal-Wallis seguito dal test post-hoc di Dunn.
Discussione
Gli anticorpi fortemente neutralizzanti svolgono un ruolo essenziale nella clearance virale e quindi contribuiscono alla protezione a lungo termine dell’infezione da DENV[31]. Tuttavia, l’evoluzione di questi rari anticorpi neutralizzanti è poco conosciuta. Utilizzando un modello unico di infezione umana da DENV2, abbiamo fornito un’analisi longitudinale del continuum della risposta delle cellule B all’infezione primaria da DENV. Nel complesso, abbiamo scoperto che la funzionalità in termini di specificità del sierotipo DENV2 è conservata dalla risposta acuta del plasmablasto attraverso il MBC convalescente e i compartimenti sierologici (che probabilmente riflettono gli anticorpi derivati da LLPC). Proponiamo che questo quadro di riferimento per tracciare lo sviluppo delle cellule B specifiche del DENV, comprese quelle che producono anticorpi specifici del sierotipo, sia utile per la valutazione dei candidati vivi attenuati al vaccino DENV, e per aiutare a deconvolgere la complessa sierologia che spesso si trova nell’infezione naturale.
Durante un’infezione acuta secondaria da DENV, la risposta del plasmablasto può comprendere una frazione sostanziale (36-95%) della risposta totale delle cellule B CD19+ [19,35]. Nel nostro modello di infezione primaria DENV2 abbiamo osservato un picco medio di plasmablasto del 6,0% della popolazione totale di cellule B. I nostri risultati sono simili a quelli di un recente studio che ha riportato un picco di plasmablasto del 2,5% delle cellule B periferiche tra le persone esposte a Zika come infezione primaria da flavivirus[62]. In un altro studio su 84 pazienti con infezioni DENV primarie o secondarie confermate in laboratorio, l’entità della risposta dei plasmablastomi è stata sostanzialmente maggiore nelle infezioni secondarie rispetto a quelle primarie[35]. Questi studi suggeriscono che le infezioni primarie da flavivirus provocano una risposta al plasmablasto inferiore rispetto all’infezione secondaria. La risposta più elevata nell’infezione secondaria da DENV può essere dovuta a una risposta di richiamo in seguito all’incontro con antigeni DENV simili. Un’altra possibilità derivante dall’associazione tra la risposta del plasmablasto e la viremia, qui e altrove, suggerisce la replicazione virale come un regolatore deterministico putativo della risposta delle cellule B[19]. Sarà essenziale determinare se il basso livello di replicazione di un virus tetravalente vivo attenuato del vaccino dengue rispetto al virus della sfida rDEN2Δ30 [41,63], predice una modesta risposta al plasmablasto. È anche possibile, tuttavia, che la somministrazione simultanea di quattro virus provochi una risposta plasmablast distinta dall’infezione naturale o dalla sfida primaria con rDEN2Δ30.
L’espansione del plasmablasto all’infezione DENV2 può essere la manifestazione di meccanismi di attivazione immunitaria multipli che agiscono sul compartimento delle cellule B. Durante l’infezione acuta da DENV le cellule mieloidi come i monociti si colorano positive per l’antigene NS3 che segna la replicazione di DENV[64]. I monociti CD14+CD16+ attivati sono stati collegati alla differenziazione e alla proliferazione dei plasmablast[59]. Inoltre, l’interazione diretta tra DENV e cellule B senza l’impegno del recettore cellulare B di superficie / immunoglobulina potrebbe anche portare all’attivazione globale delle cellule B. Anticorpi sierici neutralizzanti contro il DENV2 sviluppati in tutti i soggetti a seguito della risposta transitoria del plasmablasto, suggerendo un’attivazione antigene-specifica della risposta delle cellule B. In linea con questo, quasi la metà delle linee di plasmablasto testate ha prodotto anticorpi che si legano al DENV2. Inoltre, l’analisi del repertorio IGH/IGL accoppiato della risposta plasmablast ha rivelato che oltre la metà dei plasmablasti di sangue appartengono a lignaggi che mostrano segni di espansione clonale. Abbiamo anche osservato prove di espansione di lignaggi convergenti tra tre soggetti. Il livello complessivo dei leganti DENV2 identificati, la clonalità e i lignaggi condivisi tra i soggetti hanno suggerito che la proliferazione dei plasmablasti è stata guidata da antigeni DENV cognato oltre che da effetti puramente astanti.
Per stimare il grado di specificità dell’antigene della risposta del plasmablasto, abbiamo caratterizzato un pannello di mAb derivati da una vasta gamma di lignaggi che comprende lignaggi grandi, piccoli e convergenti. Utilizzando DENV binding ELISA abbiamo determinato che il 45% dei mAbs prodotti erano DENV-specifici. Ci sono alcune limitazioni alla nostra analisi. In primo luogo, il nostro approccio non ha coperto completamente il panorama dei plasmablast. I 92 lignaggi selezionati per l’espressione anticorpale, pur rappresentando circa il 76% dei plasmablasti in tutti i lignaggi espansi (741 su 978 cellule), rappresentavano il 44% di tutti i plasmablasti sequenziati. Abbiamo ragionato che i lignaggi espansi (che rappresentano 978 di 1690 cellule totali) avrebbero un’alta probabilità di esibire un’attività specifica DENV quando vengono espressi. Tuttavia, è possibile che alcuni dei lignaggi singleton (n = 712) potrebbero essere DENV-specifici. Infine, è stato selezionato per l’analisi solo un clone di anticorpi da ciascuno di questi 92 lignaggi. È in corso uno sforzo continuo per caratterizzare ulteriori cloni di questi lignaggi espansi e singoli plasmablast per affinare ulteriormente la nostra stima della specificità dell’antigene nella risposta del plasmablast. Dal momento che abbiamo testato mAbs da lignaggi che rappresentano il 76% di quelli con evidenza di espansione, e abbiamo trovato che il 45% di questi DENV legati, la nostra analisi suggerisce che almeno il 34% dei plasmablast in lignaggi che si sono espansi in risposta all’infezione primaria rDEN2Δ30 sono antigene-specifici.
Abbiamo ulteriormente affinato la specificità, le regioni antigeniche e le proprietà funzionali del nostro pannello mAb derivato dall’infezione primaria rDEN2Δ30. Lo screening per la reattività crociata del sierotipo ha rivelato che il 68% dei mAb derivati dal plasmablast ha riconosciuto esclusivamente il sierotipo infettante DENV2, e la frazione rimanente ha mostrato reattività crociata a DENV2 e ad almeno un altro sierotipo. La maggior parte degli anticorpi specifici del sierotipo hanno preso di mira epitopi quaternari su virioni intatti, piuttosto che semplici epitopi sulla proteina E virale. Ciò è in contrasto con un rapporto sugli anticorpi derivati da plasmablast a seguito di infezioni secondarie da dengue, dove questi anticorpi erano principalmente epitopi monomerici E semplici reattivi incrociati e mirati e non epitopi complessi[40]. Abbiamo trovato mAb specifici per DENV2 con attività funzionale simile al mAb 2D22 isolato dall’infezione naturale[25,61,65,66] ma anche mAb le cui specificità non potevano essere mappate a questi epitopi o anche a EDIII, suggerendo la possibilità che il nostro pannello di mAb sia indirizzato a specificità DENV2 sconosciute. Cinque dei 41 mAbs DENV-reattivi DENV2 neutralizzati; ciò indica che l’attività di neutralizzazione osservata nel siero può basarsi solo su una piccola frazione delle cellule B specifiche della DENV, nonostante l’ampia attività di legame del repertorio totale. Questa scoperta e nuovi mAbs specifici per la DENV2 aumentano il nostro repertorio di reagenti specifici per la DENV2 e possono fornire nuovi spunti per i meccanismi di neutralizzazione della DENV2 oltre ai pochi mAbs neutralizzanti specifici per la DENV2 attualmente conosciuti[25,65,66]. Abbiamo recentemente dimostrato che il DENV1 passato da linee cellulari di laboratorio era strutturalmente immaturo e ipersensibile alla neutralizzazione da parte di anticorpi umani, in particolare quelli eterotipici, rispetto al DENV1 circolante nei soggetti umani[67]. Con l’avvertenza che la stessa nozione deve ancora essere testata per il DENV2, noi ipotizziamo che i mAbs DENV2 di tipo specifico che si trovano qui neutralizzeranno il DENV2 in circolazione, mentre gli anticorpi cross-reattivi DENV2-binding anticorpi possono non farlo.
È stato sorprendente che un alto livello di mAbs DENV specifici per il plasmablast abbia mostrato la specificità del ceppo DENV2 (verso il DENV 2 Tonga/74). Al momento del lavoro di mappatura dell’epitopo, erano disponibili solo gli antigeni ricombinanti basati sul ceppo WHO DENV2 (S16803) che indirizzavano le nostre analisi ai mAb che legavano il ceppo S16803. Sulla base di recenti pubblicazioni riguardanti le capacità di neutralizzazione differenziale degli anticorpi contro diversi genotipi o ceppi all’interno di un sierotipo e il fallimento dei vaccini contro le varianti genotipiche[26,68], è importante comprendere gli epitopi bersaglio di questi anticorpi specifici del ceppo e come questo possa cambiare nel tempo. Tuttavia, questa specificità del ceppo DENV2 non è stata vista in MBC (dati non mostrati)(Fig. 6B), indicando che la risposta della memoria è più ampia della risposta del plasmablasto. Queste osservazioni suggeriscono che potrebbe essere una proprietà intrinseca della risposta del plasmablasto di concentrarsi sul ceppo DENV infettante, mentre l’MBC può evolvere per mantenere una più ampia specificità. Due limiti del nostro studio sono il piccolo numero di soggetti in cui abbiamo caratterizzato la risposta del plasmablasto e della memoria all’infezione primaria rDEN2Δ30 e che rDEN2Δ30 è un modello di infezione primaria da dengue. Ciononostante, Purtha e colleghi hanno riferito di differenze simili nell’ampiezza dei compartimenti di plasmablasto e MBC in un modello murino di infezione da virus del Nilo occidentale[69].
La presenza di MBC specifici per l’antigene in circolazione è uno dei marcatori dell’immunità adattiva antivirale a lungo termine[70]. Per interrogare ulteriormente la risposta delle cellule B generata dall’infezione primaria da DENV2, abbiamo analizzato il repertorio DENV-specifico di MBC immortalato 6 mesi dopo l’infezione. Tra i tre soggetti abbiamo stimato una media dello 0,4% del pool di MBC immortalato come DENV-specifico rispetto allo 0,5-8,1% riportato per le infezioni secondarie da DENV da Appanna e colleghi [40]. La nostra stima è stata anche simile a una frequenza riportata (0,39%) di cellule DENV-specifiche nel pool di MBC a seguito di un’infezione primaria naturale da DENV e superiore alla stima della vaccinazione (0,08%) di MBC immortalato[71]. Questo livello di specificità dell’antigene nel pool di MBC rispetto all’infezione naturale supporta l’applicabilità di rDEN2∆30 per l’uso come virus di sfida fisiologicamente rilevante, ma sicuro [41,42].
Negli studi sull’infezione naturale, la risposta MBC specifica per il DENV è stata caratterizzata come cross-reattiva in modo schiacciante[25,28,66,71]. Troviamo qui che, in modo simile alla risposta del plasmablasto, la risposta MBC all’infezione primaria rDEN2Δ30 è stata influenzata dalla specificità del sierotipo DENV2. Una possibile spiegazione di questa discrepanza può essere data dai punti temporali dopo l’infezione in cui sono stati studiati i MBC; abbiamo immortalato i MBC sei mesi dopo l’infezione, mentre gli studi precedenti utilizzavano campioni di infezione naturale raccolti da diversi anni a decenni dopo l’infezione. Inoltre, la determinazione definitiva dello stato di esposizione alla DENV nell’infezione naturale può essere inesatta. È anche possibile che l’MBC e la risposta sierologica convergano ulteriormente dopo molti anni a causa della popolazione del compartimento LLPC che nel tempo si è riempita di cloni derivati dall’MBC[72].
Per quanto riguarda la possibilità di sovrapposizione clonale tra i compartimenti plasmablast e MBC, abbiamo eseguito il sequenziamento IGH/IGL su MBC di uno dei soggetti e abbiamo identificato quattro distinti lignaggi basati su sequenze CDR3. Non abbiamo trovato questi lignaggi nel repertorio del plasmablasto dello stesso soggetto. Una possibilità può essere attribuita al numero limitato di sequenze MBC uniche isolate da quel soggetto. Un’altra possibilità è che ci possa essere una scarsa condivisione di cloni tra il plasmablasto e i bracci MBC, implicando diverse fonti di questi cloni durante lo sviluppo e la differenziazione delle cellule B. Infatti, Appanna e colleghi hanno riferito che i plasmablasti durante l’infezione acuta secondaria da DENV rappresentano un piccolo sottoinsieme del pool MBC[40]. Inoltre, un recente lavoro di Ellebedy e colleghi ha indicato che il sottoinsieme attivato delle cellule B era fenotipicamente, trascrizionalmente e funzionalmente distinto dai plasmablasti e contribuisce al pool di MBC in misura maggiore rispetto al pool di plasmablasti[14].
La predominanza della risposta specifica del sierotipo osservata nel repertorio dei plasmablastomi è stata corroborata anche nei dati sierologici. All’interno del sito 4 weeks a seguito dell’infezione, i nostri soggetti hanno sviluppato anticorpi neutralizzanti del siero contro la DENV2 ma non contro la DENV 1, 3 e 4, indicando che l’infezione primaria da DENV in soggetti flavivirus-naïve ha portato a una risposta omotipica neutralizzante focalizzata sul sierotipo infettante. Ciò è coerente con il nostro lavoro e con quello di altri che dimostrano l’importanza degli anticorpi specifici del sierotipo e il loro contributo nell’attività di neutralizzazione del siero nell’infezione naturale[6,26,73]. Questa risposta sierologica è stata sostenuta in convalescenza a 6 mesi dopo l’infezione, nonostante la natura di breve durata della popolazione di plasmablast. L’intuizione unica offerta dall’uso di campioni di modello di sfida è stata che la risposta precoce delle cellule B all’infezione da DENV contribuisce al pool di plasmacellule a lunga vita che producono anticorpi sierici.
In sintesi, abbiamo definito le caratteristiche temporali e la specificità della risposta delle cellule B precoci all’infezione primaria da DENV2 e abbiamo dimostrato che questi anticorpi precoci prodotti dai plasmablasti in fase acuta si sovrappongono funzionalmente a quelli prodotti nei compartimenti MBC e LLPC a lungo termine. Con l’avvertenza che questo studio è stato fatto in un piccolo numero di soggetti, i nostri risultati aiutano a convalidare ulteriormente il modello di sfida umana rDEN2Δ30 dell’infezione primaria da DENV, come è stato fatto anche dall’analisi della risposta delle cellule T [74]. Successivamente, i nostri dati che collegano la risposta di picco del plasmablasto con la viremia possono anche fornire un mezzo secondario per monitorare indirettamente la replicazione virale in vivo, che sarà importante per il continuo sviluppo di vaccini vivi attenuati contro la dengue. Nel contesto sia delle infezioni naturali da DENV che dei vaccini vivi attenuati, profilando la risposta al plasmablasto in fase acuta, può essere possibile collegare la replicazione virale con i primi plasmablasto e potenzialmente identificare le risposte protettive a livello cellulare. Proponiamo che il quadro qui presentato possa essere utile per la sua capacità di identificare nuovi modelli di risposta immunitaria, molti dei quali eventi di fenocopia visti nelle infezioni naturali, e di prevedere la qualità e la durata delle risposte anticorpali a lungo termine alla vaccinazione.
Fonti di finanziamento
Questo studio è stato sostenuto dalla Bill and Melinda Gates Foundation (OPP110470), dagli Istituti Nazionali di Allergia e Malattie Infettive (R01 AI107731-05 a A.DS, e T32AI055402 a H.T.), e dagli Istituti Nazionali di Scienze Mediche Generali (P20GM125498 a B.D.K. e S.A.D.). Le sperimentazioni cliniche da cui sono stati ottenuti i campioni sono state finanziate dal contratto intramurale NIAID HHSN272200900010C. I finanziatori non hanno avuto alcun ruolo nel disegno dello studio; nella raccolta, analisi e interpretazione dei dati, nella stesura del rapporto o nella decisione di presentare il documento per la pubblicazione.
Conflitto di interessi
NN e DEE sono dipendenti di Atreca Inc. Altri autori non dichiarano alcun conflitto di interessi.
Contributi degli autori
SAD, DEE e AMD hanno concepito e progettato esperimenti. UKN, HAT, MJD, JS, BP e NN hanno condotto esperimenti. UNK, HAT, NN, NN, DEE, RSB, AMD e SAD hanno analizzato i dati. APD, SSW, KKP e BDK hanno fornito i campioni. UKN, HAT, AMD e SAD hanno scritto il manoscritto. Tutti gli autori hanno partecipato al processo editoriale e hanno approvato il manoscritto.
References
- Guzman M.G., Gubler D.J., Izquierdo A., Martinez E., Halstead S.B.. Dengue infection. Nat Rev Dis Primers. 2016; 2
- Guzman M.G., Dengue Harris E.. Lancet.. 2015; 385(9966):453-465. PubMed
- Bhatt S., Gething P.W., Brady O.J., Messina J.P., Farlow A.W., Moyes C.L.. The global distribution and burden of dengue. Nature.. 2013; 496(7446):504-507. PubMed
- Sabin A.B.. Research on dengue during world war II. Am J Trop Med Hyg. 1952; 1(1):30-50. PubMed
- Imrie A., Meeks J., Gurary A., Sukhbaatar M., Truong T.T., Cropp C.B.. Antibody to dengue 1 detected more than 60 years after infection. Viral Immunol. 2007; 20(4):672-675. PubMed
- Patel B., Longo P., Miley M.J., Montoya M., Harris E., de Silva A.M.. Dissecting the human serum antibody response to secondary dengue virus infections. PLoS Negl Trop Dis. 2017; 11(5)
- Halstead S.B., Nimmannitya S., Cohen S.N.. Observations related to pathogenesis of dengue hemorrhagic fever. IV. Relation of disease severity to antibody response and virus recovered. Yale J Biol Med. 1970; 42(5):311-328. PubMed
- Burke D.S., Nisalak A., Johnson D.E., Scott R.M.. A prospective study of dengue infections in Bangkok. Am J Trop Med Hyg. 1988; 38(1):172-180. PubMed
- Sangkawibha N., Rojanasuphot S., Ahandrik S., Viriyapongse S., Jatanasen S., Salitul V.. Risk factors in dengue shock syndrome: a prospective epidemiologic study in Rayong, Thailand. I. The 1980 outbreak. Am J Epidemiol. 1984; 120(5):653-669. PubMed
- Thein S., Aung M.M., Shwe T.N., Aye M., Zaw A., Aye K.. Risk factors in dengue shock syndrome. Am J Trop Med Hyg. 1997; 56(5):566-572. PubMed
- Guzman M.G., Alvarez M., Halstead S.B.. Secondary infection as a risk factor for dengue hemorrhagic fever/dengue shock syndrome: an historical perspective and role of antibody-dependent enhancement of infection. Arch Virol. 2013; 158(7):1445-1459. PubMed
- Katzelnick L.C., Gresh L., Halloran M.E., Mercado J.C., Kuan G., Gordon A.. Antibody-dependent enhancement of severe dengue disease in humans. Science.. 2017; 358(6365):929-932. PubMed
- Wrammert J., Smith K., Miller J., Langley W.A., Kokko K., Larsen C.. Rapid cloning of high-affinity human monoclonal antibodies against influenza virus. Nature.. 2008; 453(7195):667-671. PubMed
- Ellebedy A.H., Jackson K.J., Kissick H.T., Nakaya H.I., Davis C.W., Roskin K.M.. Defining antigen-specific plasmablast and memory B cell subsets in human blood after viral infection or vaccination. Nat Immunol. 2016; 17(10):1226-1234. PubMed
- Obermoser G., Presnell S., Domico K., Xu H., Wang Y., Anguiano E.. Systems scale interactive exploration reveals quantitative and qualitative differences in response to influenza and pneumococcal vaccines. Immunity.. 2013; 38(4):831-844. PubMed
- Wrammert J., Koutsonanos D., Li G.M., Edupuganti S., Sui J., Morrissey M.. Broadly cross-reactive antibodies dominate the human B cell response against 2009 pandemic H1N1 influenza virus infection. J Exp Med. 2011; 208(1):181-193. PubMed
- Rahman A., Rashu R., Bhuiyan T.R., Chowdhury F., Khan A.I., Islam K.. Antibody-secreting cell responses after Vibrio cholerae O1 infection and oral cholera vaccination in adults in Bangladesh. Clin Vaccine Immunol. 2013; 20(10):1592-1598. PubMed
- McElroy A.K., Akondy R.S., Davis C.W., Ellebedy A.H., Mehta A.K., Kraft C.S.. Human Ebola virus infection results in substantial immune activation. Proc Natl Acad Sci U S A. 2015; 112(15):4719-4724. PubMed
- Wrammert J., Onlamoon N., Akondy R.S., Perng G.C., Polsrila K., Chandele A.. Rapid and massive virus-specific plasmablast responses during acute dengue virus infection in humans. J Virol. 2012; 86(6):2911-2918. PubMed
- Xu M., Hadinoto V., Appanna R., Joensson K., Toh Y.X., Balakrishnan T.. Plasmablasts generated during repeated dengue infection are virus glycoprotein-specific and bind to multiple virus serotypes. J Immunol. 2012; 189(12):5877-5885. PubMed
- Priyamvada L., Cho A., Onlamoon N., Zheng N.Y., Huang M., Kovalenkov Y.. B cell responses during secondary Dengue virus infection are dominated by highly cross-reactive, memory-derived Plasmablasts. J Virol. 2016; 90(12):5574-5585. PubMed
- Tangye S.G., Tarlinton D.M.. Memory B cells: effectors of long-lived immune responses. Eur J Immunol. 2009; 39(8):2065-2075. PubMed
- Schmidlin H., Diehl S.A., Blom B.. New insights into the regulation of human B-cell differentiation. Trends Immunol. 2009; 30(6):277-285. PubMed
- Smith T., Cunningham-Rundles C.. Primary B-cell immunodeficiencies. Hum Immunol. 2018.
- Smith S.A., de Alwis A.R., Kose N., Jadi R.S., de Silva A.M., Crowe J.E.. Isolation of dengue virus-specific memory B cells with live virus antigen from human subjects following natural infection reveals the presence of diverse novel functional groups of antibody clones. J Virol. 2014; 88(21):12233-12241. PubMed
- Nivarthi U.K., Kose N., Sapparapu G., Widman D., Gallichotte E., Pfaff J.M.. Mapping the human memory B cell and serum neutralizing antibody responses to Dengue virus serotype 4 infection and vaccination. J Virol. 2017; 91(5)
- Schieffelin J.S., Costin J.M., Nicholson C.O., Orgeron N.M., Fontaine K.A., Isern S.. Neutralizing and non-neutralizing monoclonal antibodies against dengue virus E protein derived from a naturally infected patient. Virol J. 2010; 7:28. PubMed
- Beltramello M., Williams K.L., Simmons C.P., Macagno A., Simonelli L., Quyen N.T.. The human immune response to Dengue virus is dominated by highly cross-reactive antibodies endowed with neutralizing and enhancing activity. Cell Host Microbe. 2010; 8(3):271-283. PubMed
- Dejnirattisai W., Jumnainsong A., Onsirisakul N., Fitton P., Vasanawathana S., Limpitikul W.. Cross-reacting antibodies enhance dengue virus infection in humans. Science. 2010; 328(5979):745-748. PubMed
- de Alwis R., Beltramello M., Messer W.B., Sukupolvi-Petty S., Wahala W.M., Kraus A.. In-depth analysis of the antibody response of individuals exposed to primary dengue virus infection. PLoS Negl Trop Dis. 2011; 5(6):e1188. PubMed
- de Alwis R., Smith S.A., Olivarez N.P., Messer W.B., Huynh J.P., Wahala W.M.. Identification of human neutralizing antibodies that bind to complex epitopes on dengue virions. Proc Natl Acad Sci U S A. 2012; 109(19):7439-7444. PubMed
- Teoh E.P., Kukkaro P., Teo E.W., Lim A.P., Tan T.T., Yip A.. The structural basis for serotype-specific neutralization of dengue virus by a human antibody. Sci Transl Med. 2012; 4(139):139ra83.
- Friberg H., Jaiswal S., West K., O’Ketch M., Rothman A.L., Mathew A.. Analysis of human monoclonal antibodies generated by dengue virus-specific memory B cells. Viral Immunol. 2012; 25(5):348-359. PubMed
- Rouvinski A., Guardado-Calvo P., Barba-Spaeth G., Duquerroy S., Vaney M.C., Kikuti C.M.. Recognition determinants of broadly neutralizing human antibodies against dengue viruses. Nature. 2015; 520(7545):109-113. PubMed
- Garcia-Bates T.M., Cordeiro M.T., Nascimento E.J., Smith A.P., Soares de Melo K.M., McBurney S.P.. Association between magnitude of the virus-specific plasmablast response and disease severity in dengue patients. J Immunol. 2013; 190(1):80-87. PubMed
- Dejnirattisai W., Wongwiwat W., Supasa S., Zhang X., Dai X., Rouvinski A.. A new class of highly potent, broadly neutralizing antibodies isolated from viremic patients infected with dengue virus. Nat Immunol. 2015; 16(2):170-177. PubMed
- Balakrishnan T., Bela-Ong D.B., Toh Y.X., Flamand M., Devi S., Koh M.B.. Dengue virus activates polyreactive, natural IgG B cells after primary and secondary infection. PLoS One. 2011; 6(12)
- Zompi S., Montoya M., Pohl M.O., Balmaseda A., Harris E.. Dominant cross-reactive B cell response during secondary acute dengue virus infection in humans. PLoS Negl Trop Dis. 2012; 6(3):e1568. PubMed
- Mathew A., West K., Kalayanarooj S., Gibbons R.V., Srikiatkhachorn A., Green S.. B-cell responses during primary and secondary dengue virus infections in humans. J Infect Dis. 2011; 204(10):1514-1522. PubMed
- Appanna R., Kg S., Xu M.H., Toh Y.X., Velumani S., Carbajo D.. Plasmablasts during acute Dengue infection represent a small subset of a broader virus-specific memory B cell Pool. EBioMedicine. 2016; 12:178-188. PubMed
- Kirkpatrick B.D., Whitehead S.S., Pierce K.K., Tibery C.M., Grier P.L., Hynes N.A.. The live attenuated dengue vaccine TV003 elicits complete protection against dengue in a human challenge model. Sci Transl Med. 2016; 8(330):330ra36.
- Larsen C.P., Whitehead S.S., Durbin A.P.. Dengue human infection models to advance dengue vaccine development. Vaccine. 2015; 33(50):7075-7082. PubMed
- Durbin A.P., Karron R.A., Sun W., Vaughn D.W., Reynolds M.J., Perreault J.R.. Attenuation and immunogenicity in humans of a live dengue virus type-4 vaccine candidate with a 30 nucleotide deletion in its 3′-untranslated region. Am J Trop Med Hyg. 2001; 65(5):405-413. PubMed
- Tan Y.C., Kongpachith S., Blum L.K., Ju C.H., Lahey L.J., Lu D.R.. Barcode-enabled sequencing of plasmablast antibody repertoires in rheumatoid arthritis. Arthritis Rheumatol. 2014; 66(10):2706-2715. PubMed
- Tan Y.C., Blum L.K., Kongpachith S., Ju C.H., Cai X., Lindstrom T.M.. High-throughput sequencing of natively paired antibody chains provides evidence for original antigenic sin shaping the antibody response to influenza vaccination. Clin Immunol. 2014; 151(1):55-65. PubMed
- Volpe J.M., Cowell L.G., Kepler T.B.. SoDA: implementation of a 3D alignment algorithm for inference of antigen receptor recombinations. Bioinformatics.. 2006; 22(4):438-444. PubMed
- Kabat E.A., Wu T.T., Reid-Miller M., Perry H.M., Gottesman K.S.. 1987.
- Glanville J., Zhai W., Berka J., Telman D., Huerta G., Mehta G.R.. Precise determination of the diversity of a combinatorial antibody library gives insight into the human immunoglobulin repertoire. Proc Natl Acad Sci U S A. 2009; 106(48):20216-20221. PubMed
- Scally S.W., McLeod B., Bosch A., Miura K., Liang Q., Carroll S.. Molecular definition of multiple sites of antibody inhibition of malaria transmission-blocking vaccine antigen Pfs25. Nat Commun. 2017; 8(1):1568. PubMed
- Kwakkenbos M.J., Diehl S.A., Yasuda E., Bakker A.Q., van Geelen C.M., Lukens M.V.. Generation of stable monoclonal antibody-producing B cell receptor-positive human memory B cells by genetic programming. Nat Med. 2010; 16(1):123-128. PubMed
- Ho I.Y., Bunker J.J., Erickson S.A., Neu K.E., Huang M., Cortese M.. Refined protocol for generating monoclonal antibodies from single human and murine B cells. J Immunol Methods. 2016; 438:67-70. PubMed
- Smith K., Garman L., Wrammert J., Zheng N.Y., Capra J.D., Ahmed R.. Rapid generation of fully human monoclonal antibodies specific to a vaccinating antigen. Nat Protoc. 2009; 4(3):372-384. PubMed
- Gallichotte E.N., Widman D.G., Yount B.L., Wahala W.M., Durbin A., Whitehead S.. A new quaternary structure epitope on dengue virus serotype 2 is the target of durable type-specific neutralizing antibodies. MBio.. 2015; 6(5)
- Messer W.B., Yount B., Hacker K.E., Donaldson E.F., Huynh J.P., de Silva A.M.. Development and characterization of a reverse genetic system for studying dengue virus serotype 3 strain variation and neutralization. PLoS Negl Trop Dis. 2012; 6(2):e1486. PubMed
- Messer W.B., Yount B.L., Royal S.R., de Alwis R., Widman D.G., Smith S.A.. Functional transplant of a Dengue virus serotype 3 (DENV3)-specific human monoclonal antibody epitope into DENV1. J Virol. 2016; 90(10):5090-5097. PubMed
- Modis Y., Ogata S., Clements D., Harrison S.C.. Structure of the dengue virus envelope protein after membrane fusion. Nature. 2004; 427(6972):313-319. PubMed
- Wahala W.M., Kraus A.A., Haymore L.B., Accavitti-Loper M.A., de Silva A.M.. Dengue virus neutralization by human immune sera: role of envelope protein domain III-reactive antibody. Virology. 2009; 392(1):103-113. PubMed
- Swanstrom J.A., Plante J.A., Plante K.S., Young E.F., McGowan E., Gallichotte E.N.. Dengue virus envelope dimer epitope monoclonal antibodies isolated from Dengue patients are protective against Zika virus. MBio. 2016; 7(4)
- Kwissa M., Nakaya H.I., Onlamoon N., Wrammert J., Villinger F., Perng G.C.. Dengue virus infection induces expansion of a CD14(+)CD16(+) monocyte population that stimulates plasmablast differentiation. Cell Host Microbe. 2014; 16(1):115-127. PubMed
- Lu D.R., Tan Y.C., Kongpachith S., Cai X., Stein E.A., Lindstrom T.M.. Identifying functional anti-Staphylococcus aureus antibodies by sequencing antibody repertoires of patient plasmablasts. Clin Immunol. 2014; 152(1–2):77-89. PubMed
- Gallichotte E.N., Baric T.J., Yount B.L., Widman D.G., Durbin A., Whitehead S.. Human dengue virus serotype 2 neutralizing antibodies target two distinct quaternary epitopes. PLoS Pathog. 2018; 14(2)
- Rogers T.F., Goodwin E.C., Briney B., Sok D., Beutler N., Strubel A.. Zika virus activates de novo and cross-reactive memory B cell responses in dengue-experienced donors. Sci Immunol. 2017; 2(14)
- Kirkpatrick B.D., Durbin A.P., Pierce K.K., Carmolli M.P., Tibery C.M., Grier P.L.. Robust and balanced immune responses to all 4 Dengue virus serotypes following Administration of a Single Dose of a live attenuated tetravalent Dengue vaccine to healthy, Flavivirus-naive adults. J Infect Dis. 2015; 212(5):702-710. PubMed
- Durbin A.P., Vargas M.J., Wanionek K., Hammond S.N., Gordon A., Rocha C.. Phenotyping of peripheral blood mononuclear cells during acute dengue illness demonstrates infection and increased activation of monocytes in severe cases compared to classic dengue fever. Virology. 2008; 376(2):429-435. PubMed
- Fibriansah G., Ibarra K.D., Ng T.S., Smith S.A., Tan J.L., Lim X.N.. Dengue Virus. Cryo-EM structure of an antibody that neutralizes dengue virus type 2 by locking E protein dimers. Science. 2015; 349(6243):88-91. PubMed
- Smith S.A., Zhou Y., Olivarez N.P., Broadwater A.H., de Silva A.M., Crowe J.E.. Persistence of circulating memory B cell clones with potential for dengue virus disease enhancement for decades following infection. J Virol. 2012; 86(5):2665-2675. PubMed
- Raut R., Corbett K.S., Tennekoon R.N., Premawansa S., Wijewickrama A., Premawansa G.. Dengue type 1 viruses circulating in humans are highly infectious and poorly neutralized by human antibodies. Proc Natl Acad Sci U S A. 2019; 116(1):227-232. PubMed
- Gallichotte E.N., Baric T.J., Nivarthi U., Delacruz M.J., Graham R., Widman D.G.. Genetic variation between Dengue virus type 4 strains impacts human antibody binding and neutralization. Cell Rep. 2018; 25(5):1214-1224. PubMed
- Purtha W.E., Tedder T.F., Johnson S., Bhattacharya D., Diamond M.S.. Memory B cells, but not long-lived plasma cells, possess antigen specificities for viral escape mutants. J Exp Med. 2011; 208(13):2599-2606. PubMed
- Crotty S., Felgner P., Davies H., Glidewell J., Villarreal L., Ahmed R.. Cutting edge: long-term B cell memory in humans after smallpox vaccination. J Immunol. 2003; 171(10):4969-4973. PubMed
- Smith S.A., de Alwis R., Kose N., Durbin A.P., Whitehead S.S., de Silva A.M.. Human monoclonal antibodies derived from memory B cells following live attenuated dengue virus vaccination or natural infection exhibit similar characteristics. J Infect Dis. 2013; 207(12):1898-1908. PubMed
- Amanna I.J., Slifka M.K.. Mechanisms that determine plasma cell lifespan and the duration of humoral immunity. Immunol Rev. 2010; 236:125-138. PubMed
- Katzelnick L.C., Montoya M., Gresh L., Balmaseda A., Harris E.. Neutralizing antibody titers against dengue virus correlate with protection from symptomatic infection in a longitudinal cohort. Proc Natl Acad Sci U S A. 2016; 113(3):728-733. PubMed
- Grifoni A., Angelo M., Sidney J., Paul S., Peters B., de Silva A.D.. Patterns of cellular immunity associated with experimental infection with rDEN2Delta30 (Tonga/74) support its suitability as a human Dengue virus challenge strain. J Virol. 2017; 91(8)
Fonte
Nivarthi UK, Tu HA, Delacruz MJ, Swanstrom J, Patel B, et al. (2019) Longitudinal analysis of acute and convalescent B cell responses in a human primary dengue serotype 2 infection model. EBioMedicine 41. https://doi.org/10.1016/j.ebiom.2019.02.060





