Introduzione
L’attivazione di CD95/Fas attraverso l’interazione con il suo cognato ligando CD95L o anticorpi che attivano i recettori induce l’apoptosi nelle cellule sensibili(Suda et al., 1993). Praticamente tutte le ricerche su CD95 e CD95L si sono concentrate sull’interazione fisica tra le due proteine e le successive cascate di segnalazione basate sulle proteine(Algeciras-Schimnich et al., 2002; Fu et al., 2016; Nisihara et al., 2001; Schneider et al., 1997). Tuttavia, abbiamo recentemente dimostrato che l’mRNA di CD95 e CD95L sequenze di porto che, se convertiti in piccoli interferenti (si) o corti RNA, causano una tossicità massiccia e robusta in tutte le cellule tumorali testate. Questi si/shRNA derivati da CD95/CD95L hanno come bersaglio una rete di geni di sopravvivenza, con conseguente attivazione simultanea di più vie di morte cellulare attraverso l’interferenza dell’RNA (RNAi) in un processo che abbiamo chiamato DISE (Death Induced by Survival gene Elimination)(Putzbach et al., 2017). Abbiamo determinato che per un si/shRNA per provocare questa forma di tossicità, sono necessarie solo le posizioni 2-7 del filamento guida, la sequenza di semi 6mer (Putzbachet al., 2017). Più recentemente, uno screening di tutti i 4096 semi 6mer ha rivelato che la tossicità ottimale dei semi 6mer richiede semi ricchi di G che mirano alle regioni ricche di C nei 3’UTR dei geni di sopravvivenza(Gao et al., 2018).
In questo rapporto, mostriamo che l’espressione dell’mRNA CD95L è tossica per le cellule anche senza una previa conversione in piccoli (s)RNA. Questa tossicità è indipendente dalla proteina CD95L a lunghezza intera o dall’espressione del recettore CD95L e assomiglia a DISE. La tossicità coinvolge anche l’RNAi. Abbiamo scoperto che sRNA multipli sono generati dall’mRNA di CD95L all’interno delle cellule e sono caricati nel complesso di silenziamento indotto dall’RNA (RISC), il mediatore chiave dell’RNAi(Liu et al., 2004). Inoltre, forniamo la prova che gli mRNA endogeni possono essere elaborati in sRNA e caricati nel RISC, specialmente nelle cellule con bassa espressione di miRNA. Il sottoinsieme dei geni che si trovano nel RISC sono regolatori della traduzione delle proteine e della proliferazione cellulare.
Risultati
CD95L mRNA è tossico per le cellule
Testando ogni possibile shRNA derivato dal CD95L open reading frame (ORF) o dal suo 3’UTR, abbiamo recentemente riscontrato un elevato arricchimento di si/shRNA tossici derivati dal CD95L ORF(Putzbach et al., 2017). Più recentemente abbiamo determinato che la tossicità dei semi 6mer osservata in molti si/shRNA è in gran parte dovuta alla loro composizione nucleotidica, con i semi ricchi di G che sono i più tossici(Gao et al., 2018)(https://6merdb.org). Quando abbiamo rianalizzato gli shRNA derivati da CD95L ORF, abbiamo trovato una correlazione significativa tra la tossicità degli shRNA più tossici derivati da CD95L(Putzbach et al., 2017) e la tossicità del corrispondente seme 6mer che abbiamo recentemente determinato in uno screening di tutti i 4096 semi 6mer(Gao et al., 2018). Questo suggerisce che i si/shRNA derivati dal CD95L uccidono le cellule tumorali, in gran parte attraverso la tossicità dei semi 6mer.
Ci siamo quindi chiesti se l’espressione dell’mRNA ORF CD95L – senza pretrattamento in siRNA artificiali – sarebbe tossica per le cellule. L’espressione della proteina CD95L nella maggior parte delle cellule uccide attraverso l’induzione dell’apoptosi. Di conseguenza, l’espressione di CD95L nelle cellule HeyA8, che sono altamente sensibili all’apoptosi mediata da CD95, uccide le cellule entro poche ore dall’infezione con un lentivirus che codifica CD95L(Figura 1A, pannello sinistro). È interessante notare che una grave riduzione della crescita è stata vista senza alcun segno di apoptosi (non mostrato) quando un mutante CD95L, incapace di legare il CD95, è stato espresso (CD95LMUT in Figura 1A, pannello sinistro). Questo mutante è portatore di una mutazione puntiforme Y218R, che impedisce alla proteina CD95L di legarsi al CD95(Schneider et al., 1997), ed è espressa ad un livello simile a quello del tipo selvaggio (wt) CD95L(Figura 1B). Per evitare che il mRNA CD95L mRNA produca la proteina CD95L a lunghezza intera, abbiamo anche introdotto un codone di arresto prematuro subito dopo il codone di inizio nel vettore CD95LMUT (CD95LMUTNP). Questo costrutto (contenente quattro mutazioni puntiformi e confermato per produrre mRNA senza la proteina CD95L full-length rilevabile, Figura 1B) è stato analogamente attivo nel ridurre la crescita delle cellule HeyA8 rispetto al vettore CD95LMUT(Figura 1A, pannello sinistro). CD95LMUTNP produceva ancora CD95L troncato e probabilmente solubile(Figura 1B), probabilmente a causa dell’uso di un codone di partenza alternativo. Tuttavia, il supernatante delle cellule 293T infettate da questo virus non ha indotto l’apoptosi quando è stato aggiunto alle cellule HeyA8 (dati non mostrati), confermando che non aveva alcuna attività che induceva l’apoptosi.
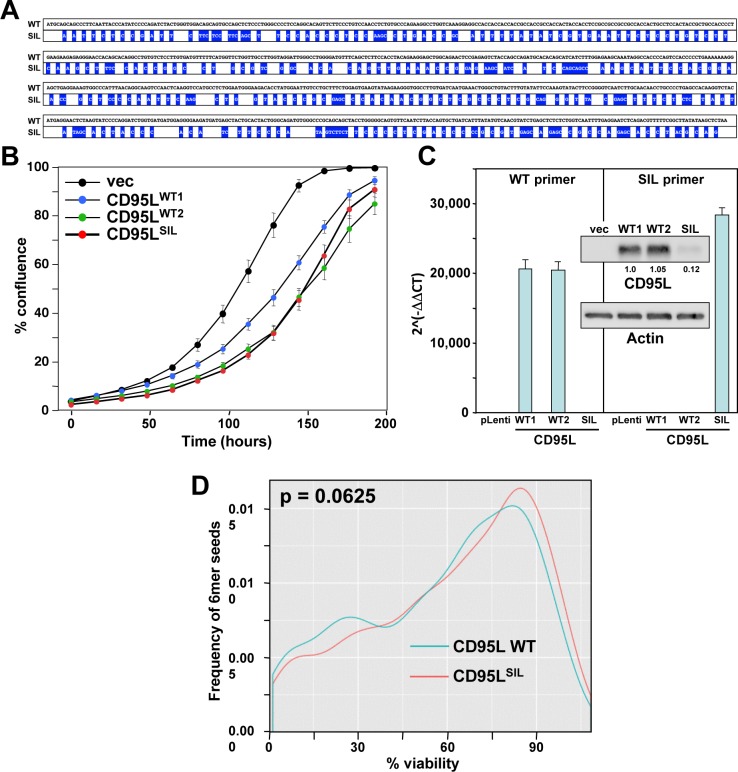
Figura 1-figure supplemento 3.Figura 1—figura supplemento 3. Il mRNA CD95L è tossico per le cellule. La generazione di cellule complete CD95 k.o. MCF-7. Mutazione del codone di inizio alternativo in CD95LMUTNP construct.Tossicità del mRNA CD95L è indipendente dall’espressione della proteina CD95L.(A) A sinistra: schema dei diversi mutanti CD95L utilizzati. A destra: Percentuale di confluenza cellulare nel tempo di cellule parentali HeyA8 in assenza(pannello di sinistra) o in presenza di 20 μM zVAD-fmk (pannello centrale), o di cellule CD95 k.o. (pannello didestra) dopo l’espressione dei costrutti CD95L. I dati sono rappresentativi di uno o tre esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice o quadruplice mostrato come media ±SE.(B) A sinistra: analisi Western blot di cellule HeyA8 che sovraesprimono diversi RNA mutanti CD95L. Le cellule che esprimono CD95LMUT o CD95L sono state pretrattate con 20 μM zVAD-fmk. Si noti la piccola quantità di CD95L troncato nelle cellule infettate con CD95L MUTNPnon ha attività legante CD95. Dati molto simili sono stati ottenuti quando i costrutti sono stati espressi in cellule CD95 k.o. HeyA8 (clone #11) o NB-7, che mancano di espressione di caspase-8, entrambi senza trattamento con zVAD (dati non mostrati). Esatto: Analisi RT-qPCR per CD95L degli stessi campioni. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(C, D) Quantificazione della morte cellulare(C) e della produzione di ROS (D) in cellule CD95 k.o. HeyA8 (clone #11) che esprimono pLenti (v) o pLenti-CD95L (L) in diversi punti temporali (giorni dopo l’infezione). I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±SE di tre repliche. *p<0.05, **p<0.001, ***p<0.0001, t-test non accoppiato.(E) Confluenza nel tempo del clone MCF-7 completo CD95 k.o. FA4 (a destra) o un clone MCF-7 #21 in cui abbiamo eliminato il sito target shR6 con conseguente spostamento fuori fotogramma dopo l’infezione con il controllo vettoriale pLenti (vec) o wt CD95L. I dati sono rappresentativi di due esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche.(A) Schema delle posizioni genomiche e le sequenze dei gRNA utilizzati per accise l’intero gene CD95 in MCF-7 cellule. I siti PAM sono sottolineati.(B) PCR con i primer laterali (pannelli superiori) e interni(pannelli inferiori) utilizzati per confermare l’assenza del gene CD95 nei cloni MCF-7. Le cellule parentali (Par.) e tre cloni infettati solo da Cas9 (Cas9) e due cloni omozigoti completi CD95 k.o. (F2 e FA4) sono mostrati.(C) RT-qPCR analisi RT-qPCR dei cloni indicati utilizzando primer che coprono sia esone 1/2 o esone 2/3 del gene CD95.(D) Colorazione superficiale per CD95 di un clone di un wt e di un k.o..(E) Analisi Western blot di tutti i cloni.(A) A sinistra: schema dei diversi mutanti CD95L utilizzati. Per la proteina mutante M86I la mutazione puntiforme a livello di DNA è tra parentesi.(B) Analisi del Western blot delle cellule HeyA8 CD95 k.o. che sovraesprimono diversi mutanti CD95L. Nota, per ragioni sconosciute in questo esperimento l’espressione della proteina CD95LMUTNP è stata più efficiente rispetto all’esperimento mostrato in Figura 1B.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) dopo l’espressione dei diversi costrutti CD95L. I valori sono stati calcolati a partire da campioni placcati in triplice copia mostrati come media ±SE.(A) Schema che mostra le posizioni delle mutazioni silenziose ottimizzate del codone del mutante CD95LSIL rispetto al tipo selvaggio CD95L (mutazioni evidenziate in blu).(B) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) che sovraesprimono il vettore pLenti vuoto (vec), il wild-type CD95L (da due virus clonati separatamente), o il CD95LSIL.(C) RT-qPCR analisi RT-qPCR e Western blot(inset) di mRNA mutanti di tipo selvaggio CD95L e CD95LSIL nelle cellule sovraespressive mostrate in B.(D) Trama di densità di probabilità che confronta la tossicità di tutti i possibili semi 6mer situati nel mRNA WT o SIL CD95L. p-valore è stato calcolato utilizzando un due campioni su due lati K-S test.
Questo risultato ha suggerito che l’mRNA CD95L potrebbe essere tossico per le cellule HeyA8 senza la proteina CD95L che induce apoptosi. Ciò è stato confermato dall’espressione dei tre costrutti CD95L in presenza dell’inibitore dell’oligo-caspasi zVAD-fmk(Figura 1A, pannello centrale). Con l’apoptosi soppressa, tutti e tre i costrutti erano ora ugualmente tossici per le cellule HeyA8. Infine, abbiamo testato un clone HeyA8 CD95 k.o. confermato di non esprimere alcuna proteina CD95(Putzbach et al., 2017). In queste cellule, senza l’aggiunta di zVAD-fmk, wt CD95L e CD95LMUTNP erano di nuovo ugualmente attivi nel ridurre la crescita delle cellule(Figura 1A, pannello di destra). Insieme, questi dati suggeriscono che è il mRNA CD95L che ha ucciso le cellule. La morte delle cellule è stata confermata dalla quantificazione della frammentazione nucleare(Figura 1C). Abbiamo anche rilevato un aumento significativo del ROS nelle cellule che esprimono CD95LMUTNP(Figura 1D), che è una caratteristica caratteristica di DISE(Hadji et al., 2014; Patel e Peter, 2018). Per escludere la possibilità che la proteina CD95 troncata o qualsiasi parte dell’mRNA CD95 abbia un ruolo in questa tossicità, abbiamo eliminato il gene CD95 nelle cellule MCF-7(Figura 1-figure supplement 1). La sovraespressione del tipo selvaggio CD95L ha ucciso le cellule del clone FA4, che ospitano una completa delezione omozigote dell’intero gene CD95 (esone 1-9), così come la proteina CD95 k.o. clone #21 cellule che conservano una certa espressione di mRNA troncata (Figura 1E).
Infine, per escludere che la proteina troncata prodotta dal costrutto CD95LMUTNP abbia avuto un’attività non voluta, abbiamo mutato il codone di partenza ATG che molto probabilmente ha dato origine al CD95L troncato in ATA (Figura 1-figure supplement 2A). Questo costrutto mutante CD95LMUTNP (G258A) non produceva più alcuna proteina rilevabile(Figura 1-figure supplement 2B) ma era tossico per le cellule CD95 k.o. HeyA8 come CD95LMUTNP(Figura 1-figure supplement 2C).
I nostri esperimenti hanno dimostrato che un piccolo numero di mutazioni puntiformi introdotte in vari mutanti CD95L non hanno influito in modo significativo sulla tossicità del mRNA CD95L. Per determinare se un gran numero di mutazioni avrebbe ridotto la sua tossicità, abbiamo generato un mutante CD95LMUTNP con 308 mutazioni puntiformi silenziose ottimizzate per il codone (CD95LSIL)(Figura 1-figure supplement 3A). L’attività di questo costrutto mutante per influenzare negativamente la crescita cellulare di cellule CD95 k.o. HeyA8 è stata confrontata con due costrutti CD95L wt clonati indipendentemente (WT1 e WT2 in figura 1-figure supplement 3B). Il costrutto SIL mutante è stato ugualmente efficace nel sopprimere la crescita cellulare e ha prodotto circa la stessa quantità di mRNA CD95L(Figura 1-figure supplement 3C). Tuttavia, la costruzione SIL ha prodotto solo circa il 12% della proteina WT CD95L(Figura 1-figure supplement 3C), sempre a sostegno dell’osservazione che è l’mRNA CD95L e non la proteina che provoca tossicità.
Abbiamo recentemente determinato che una tossicità generale di si/shRNA è in gran parte basata sulla composizione della sequenza di semi 6mer del filamento guida(Gao et al., 2018). Abbiamo ora tracciato la tossicità di tutti gli sRNA che possono essere derivati dal WT o dal SIL mutante CD95L e abbiamo trovato che la tossicità generale prevista sulla base del nostro schermo di tossicità eseguito nelle cellule HeyA8 non era statisticamente diversa tra i due costrutti del CD95L, (Figura 1-figure supplement 3D) suggerendo che le sequenze tossiche sono incorporate nel CD95LSIL ORF. Presi insieme, i nostri dati indicano che anche la mutazione di più di 300 posizioni in mRNA CD95L permette ancora di formare sRNA tossici, suggerendo che la tossicità dell’mRNA CD95L è un fenomeno molto robusto.

Figura 1-figure supplemento 3.Figura 1—figura supplemento 3. Il mRNA CD95L mRNA è tossico per le cellule.generazione di completo CD95 k.o. MCF-7 cells.Mutation del codone di inizio alternativo in CD95LMUTNP construct.Toxicity di mRNA CD95L mRNA è indipendente dall’espressione della proteina CD95L.(A) A sinistra: schema dei diversi mutanti CD95L utilizzati. A destra: Percentuale di confluenza cellulare nel tempo di cellule parentali HeyA8 in assenza(pannello di sinistra) o in presenza di 20 μM zVAD-fmk (pannello centrale), o di cellule CD95 k.o. (pannello didestra) dopo l’espressione dei costrutti CD95L. I dati sono rappresentativi di uno o tre esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice o quadruplice mostrato come media ±SE.(B) A sinistra: analisi Western blot di cellule HeyA8 che sovraesprimono diversi RNA mutanti CD95L. Le cellule che esprimono CD95LMUT o CD95L sono state pretrattate con 20 μM zVAD-fmk. Si noti la piccola quantità di CD95L troncato nelle cellule infettate con CD95L MUTNPnon ha attività legante CD95. Dati molto simili sono stati ottenuti quando i costrutti sono stati espressi in cellule CD95 k.o. HeyA8 (clone #11) o NB-7, che mancano di espressione di caspase-8, entrambi senza trattamento con zVAD (dati non mostrati). Esatto: Analisi RT-qPCR per CD95L degli stessi campioni. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(C, D) Quantificazione della morte cellulare(C) e della produzione di ROS (D) in cellule CD95 k.o. HeyA8 (clone #11) che esprimono pLenti (v) o pLenti-CD95L (L) in diversi punti temporali (giorni dopo l’infezione). I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±SE di tre repliche. *p<0.05, **p<0.001, ***p<0.0001, t-test non accoppiato.(E) Confluenza nel tempo del clone MCF-7 completo CD95 k.o. FA4 (a destra) o un clone MCF-7 #21 in cui abbiamo eliminato il sito target shR6 con conseguente spostamento fuori fotogramma dopo l’infezione con il controllo vettoriale pLenti (vec) o wt CD95L. I dati sono rappresentativi di due esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche.(A) Schema delle posizioni genomiche e le sequenze dei gRNA utilizzati per accise l’intero gene CD95 in MCF-7 cellule. I siti PAM sono sottolineati.(B) PCR con i primer laterali (pannelli superiori) e interni(pannelli inferiori) utilizzati per confermare l’assenza del gene CD95 nei cloni MCF-7. Le cellule parentali (Par.) e tre cloni infettati solo da Cas9 (Cas9) e due cloni omozigoti completi CD95 k.o. (F2 e FA4) sono mostrati.(C) RT-qPCR analisi RT-qPCR dei cloni indicati utilizzando primer che coprono sia esone 1/2 o esone 2/3 del gene CD95.(D) Colorazione superficiale per CD95 di un clone di un wt e di un k.o..(E) Analisi Western blot di tutti i cloni.(A) A sinistra: schema dei diversi mutanti CD95L utilizzati. Per la proteina mutante M86I la mutazione puntiforme a livello di DNA è tra parentesi.(B) Analisi del Western blot delle cellule HeyA8 CD95 k.o. che sovraesprimono diversi mutanti CD95L. Nota, per ragioni sconosciute in questo esperimento l’espressione della proteina CD95LMUTNP è stata più efficiente rispetto all’esperimento mostrato in Figura 1B.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) dopo l’espressione dei diversi costrutti CD95L. I valori sono stati calcolati a partire da campioni placcati in triplice copia mostrati come media ±SE.(A) Schema che mostra le posizioni delle mutazioni silenziose ottimizzate del codone del mutante CD95LSIL rispetto al tipo selvaggio CD95L (mutazioni evidenziate in blu).(B) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) che sovraesprimono il vettore pLenti vuoto (vec), il wild-type CD95L (da due virus clonati separatamente), o il CD95LSIL.(C) RT-qPCR analisi RT-qPCR e Western blot(inset) di mRNA mutanti di tipo selvaggio CD95L e CD95LSIL nelle cellule sovraespressive mostrate in B.(D) Trama di densità di probabilità che confronta la tossicità di tutti i possibili semi 6mer situati nel mRNA WT o SIL CD95L. p-valore è stato calcolato utilizzando un due campioni su due lati K-S test.
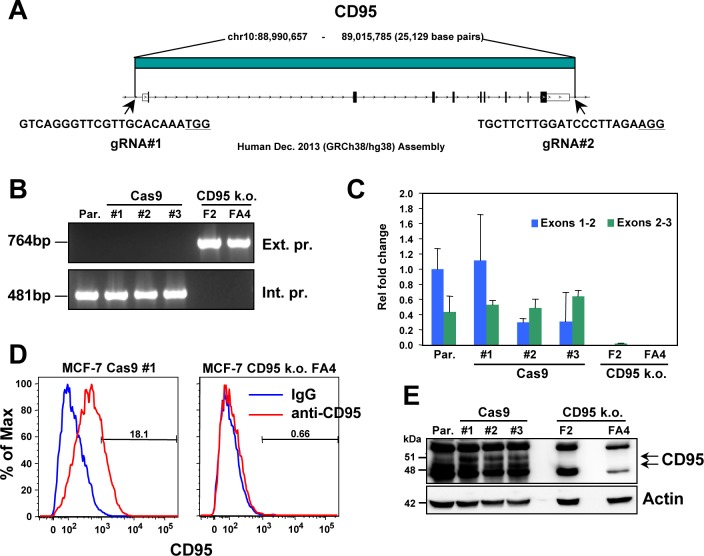
Figura 1-figure supplement 1.Generazione di cellule complete CD95 k.o. MCF-7.(A) Schema delle posizioni genomiche e le sequenze dei gRNA utilizzati per accise l’intero gene CD95 in MCF-7 cellule. I siti PAM sono sottolineati.(B) PCR con i primer laterali (pannelli superiori) e interni(pannelli inferiori) utilizzati per confermare l’assenza del gene CD95 nei cloni MCF-7. Le cellule parentali (Par.) e tre cloni infettati solo da Cas9 (Cas9) e due cloni omozigoti completi CD95 k.o. (F2 e FA4) sono mostrati.(C) RT-qPCR analisi RT-qPCR dei cloni indicati utilizzando primer che coprono sia esone 1/2 o esone 2/3 del gene CD95.(D) Colorazione superficiale per CD95 di un clone di un wt e di un k.o..(E) Analisi Western blot di tutti i cloni.
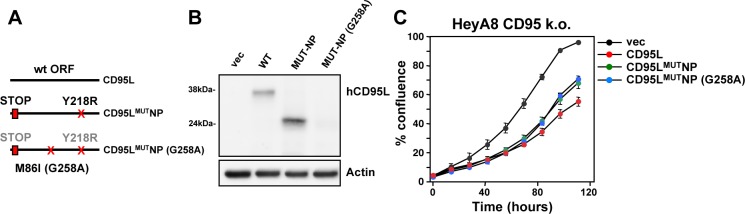
Figura 1-figure supplement 2.Mutazione del codone di partenza alternativo nel costrutto CD95LMUTNP.(A) A sinistra: Schema dei diversi mutanti CD95L utilizzati. Per la proteina M86I mutante la mutazione del punto a livello di DNA è tra parentesi.(B) Western blot analisi di HeyA8 CD95 k.o. cellule CD95 k.o. che sovraesprimono diversi mutanti CD95L. Nota, per ragioni sconosciute in questo esperimento l’espressione della proteina CD95LMUTNP è stata più efficiente rispetto all’esperimento mostrato in Figura 1B.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) dopo l’espressione dei diversi costrutti CD95L. I valori sono stati calcolati a partire da campioni placcati in triplice copia mostrati come media ±SE.

Figura 1-figure supplement 3.Tossicità di mRNA CD95L mRNA è indipendente dall’espressione della proteina CD95L.(A) Schema che mostra le posizioni delle mutazioni silenziose ottimizzate codone del mutante CD95LSIL rispetto al tipo selvaggio CD95L (mutazioni evidenziate in blu).(B) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. (cl. #11) che sovraesprimono il vettore pLenti vuoto (vec), il wild-type CD95L (da due virus clonati separatamente), o il CD95LSIL.(C) RT-qPCR analisi RT-qPCR e Western blot(inset) di mRNA mutanti di tipo selvaggio CD95L e CD95LSIL nelle cellule sovraespressive mostrate in B.(D) Trama di densità di probabilità che confronta la tossicità di tutti i possibili semi 6mer situati nel mRNA WT o SIL CD95L. p-valore è stato calcolato utilizzando un due-campione a due lati K-S test.
CD95L mRNA CD95L uccide le cellule attraverso DISE
Dopo l’infezione con CD95L, le cellule CD95 k.o. HeyA8 hanno mostrato cambiamenti morfologici sorprendentemente simili ai cambiamenti osservati nelle cellule HeyA8 dopo l’introduzione di un shRNA derivato da CD95L (shL3)(Figura 2A, Video 1- 4) che suggeriscono che le cellule sono morte attraverso un meccanismo simile. Per determinare la causa della morte cellulare indotta da mRNA CD95L in cellule HeyA8 CD95 k.o. molecolarmente, abbiamo eseguito un’analisi RNA-Seq. Abbiamo trovato che l’espressione di CD95L ha causato un downregulation preferenziale dei geni critici di sopravvivenza e non dei geni non sopravvissuti in un set di controllo(Figura 2B). Inoltre, la morte cellulare indotta da mRNA CD95L ha causato una perdita sostanziale di 11 dei 12 istoni rilevati per essere downregolati nelle cellule trattate con CD95 e CD95L-derivato sh/siRNA(Figura 2C). La perdita di istoni è un evento precoce durante la DISE(Putzbach et al., 2017). Un’analisi di Metascape ha dimostrato che l’assemblaggio dei nucleosomi, la regolazione della mitosi e i geni coerenti con il coinvolgimento degli istoni erano tra gli RNA più significativamente downregolati in tutte le cellule in cui la DISE è stata indotta da uno qualsiasi dei quattro sh/siRNA o dall’espressione di mRNA CD95L(Figura 2D). Questo suggerisce che l’mRNA CD95L uccide le cellule nello stesso modo di CD95/L-derivato si/shRNA.
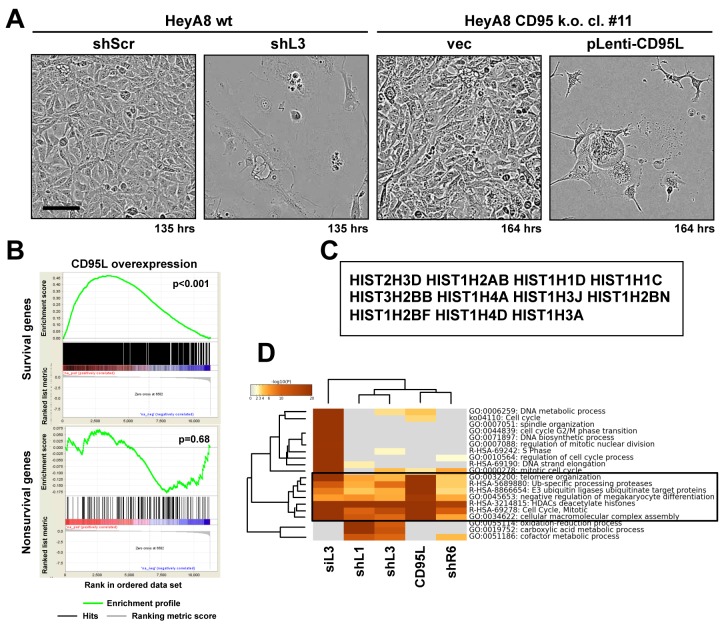
Figura 2-video 4.La tossicità indotta dalla sovraespressione di CD95L ricorda le cellule DISE.CD95 k.o. HeyA8 (cl. #11) infettate dal virus di controllo pLenti.CD95 k.o. HeyA8 (cl. #11) infettate dal virus pLenti-CD95L.HeyA8 infettate dalle cellule pLKO-shScr.HeyA8 infettate dalle cellule pLKO-shL3.(A) Immagini in contrasto di fase delle cellule HeyA8 e HeyA8 CD95 k.o. (cl. #11) dopo l’infezione con pLKO-shScr/shL3 o pLenti (vec)/pLenti-CD95L, rispettivamente, nel punto temporale indicato.(B) Gene set analisi di arricchimento per i 1846 geni di sopravvivenza (pannello superiore) e 416 geni non di sopravvivenza(pannello inferiore) identificati nello studio Sabatini(Putzbach et al., 2017; Wang et al., 2015) di mRNA downregulated in CD95L che esprime le cellule HeyA8 CD95 k.o. rispetto alle cellule HeyA8 CD95 k.o. infettate con il virus pLenti. p-valori p indicano il significato di arricchimento.(C) Geni comuni downregulated in tutti gli esperimenti RNA-Seq da (HeyA8) cellule trattate con uno dei quattro si/shRNA(Putzbach et al., 2017) derivati da CD95 o CD95L (vedi Figura 2D) e cellule che esprimono in eccesso CD95L ORF come descritto in(B).(D) Analisi Metascape di 5 set di dati RNA Seq analizzati. I cluster di termine GO in scatola sono stati altamente arricchiti in tutti e cinque i set di dati.

Figura 2-video 4.La tossicità indotta dalla sovraespressione di CD95L ricorda le cellule DISE.CD95 k.o. HeyA8 (cl. #11) infettate dal virus di controllo pLenti.CD95 k.o. HeyA8 (cl. #11) infettate dal virus pLenti-CD95L.HeyA8 infettate dalle cellule pLKO-shScr.HeyA8 infettate dalle cellule pLKO-shL3.(A) Immagini in contrasto di fase delle cellule HeyA8 e HeyA8 CD95 k.o. (cl. #11) dopo l’infezione con pLKO-shScr/shL3 o pLenti (vec)/pLenti-CD95L, rispettivamente, nel punto temporale indicato.(B) Gene set analisi di arricchimento per i 1846 geni di sopravvivenza (pannello superiore) e 416 geni non di sopravvivenza(pannello inferiore) identificati nello studio Sabatini(Putzbach et al., 2017; Wang et al., 2015) di mRNA downregulated in CD95L che esprime le cellule HeyA8 CD95 k.o. rispetto alle cellule HeyA8 CD95 k.o. infettate con il virus pLenti. p-valori p indicano il significato di arricchimento.(C) Geni comuni downregulated in tutti gli esperimenti RNA-Seq da (HeyA8) cellule trattate con uno dei quattro si/shRNA(Putzbach et al., 2017) derivati da CD95 o CD95L (vedi Figura 2D) e cellule che esprimono in eccesso CD95L ORF come descritto in(B).(D) Analisi Metascape di 5 set di dati RNA Seq analizzati. I cluster di termine GO in scatola sono stati altamente arricchiti in tutti e cinque i set di dati.

Figura 2-video 1.CD95 k.o. HeyA8 cellule (cl. #11) infettate dal virus del controllo pLenti.

Figura 2-video 2.CD95 k.o. HeyA8 cellule (cl. #11) infettate dal virus pLenti-CD95L.

Figura 2-video 3.HeyA8 cellule HeyA8 infettate con pLKO-shScr.

Figura 2-video 4.4. HeyA8 cellule HeyA8 infettate con pLKO-shL3.
CD95L mRNA uccide le cellule attraverso l’RNAi
Dato il nostro precedente lavoro sulla tossicità del si/shRNA derivato dal CD95L, abbiamo ipotizzato che l’mRNA del CD95L uccida le cellule attraverso un meccanismo basato sull’RNAi, forse elaborando gli sRNA che sono incorporati nel RISC. Le cellule Drosha k.o. prive della maggior parte dei miRNA endogeni, ma che conservano l’espressione delle proteine Ago, hanno dimostrato di essere ipersensibili al DISE indotto da si- e shRNA(Putzbach et al., 2017). Abbiamo interpretato questo effetto come causato da un aumento del pool di macchine RNAi non occupate a causa dell’assenza della maggior parte dei miRNA. Drosha k.o. cellule Drosha erano anche ipersensibili all’espressione di CD95LMUTNP(Figura 3A, p = 0,014, secondo un modello di raccordo polinomiale); Praticamente tutte le cellule sono morte (inserire in Figura 3A). Per determinare direttamente se il RISC è coinvolto nella tossicità, abbiamo introdotto il CD95L nelle cellule CD95 k.o. HeyA8 dopo aver abbattuto l’AGO2(Figura 3B). La tossicità provocata dal CD95L è stata bloccata in seguito all’abbattimento dell’AGO2, suggerendo che l’AGO2 era necessario per l’mRNA del CD95L per essere tossico. Questo è stato anche il caso quando le cellule CD95 k.o. HeyA8 sono state infettate con il CD95MUTNP o il CD95SIL(Figura 3-figure supplement 1A), confermando che tutte e tre le versioni del CD95L hanno ucciso le cellule con l’aiuto del RISC. Quando le cellule parentali HeyA8 sono state infettate con CD95L WT o CD95LSIL sono morte così rapidamente che le cellule hanno dovuto essere placcate prima del trattamento con la puromicina utilizzata per selezionare le cellule infettate dal virus(Figura 3—figura supplemento 1B). Come previsto in queste cellule che sono morte per apoptosi canonica, l’abbattimento di AGO2 non ha avuto alcun effetto sulla vitalità cellulare. Al contrario, la morte cellulare indotta da CD95LMUTNP, che in questo esperimento ha ucciso in modo molto efficiente le cellule parentali HeyA8, è stata gravemente smussata dall’abbattimento di AGO2(Figura 3-figure supplement 1C).
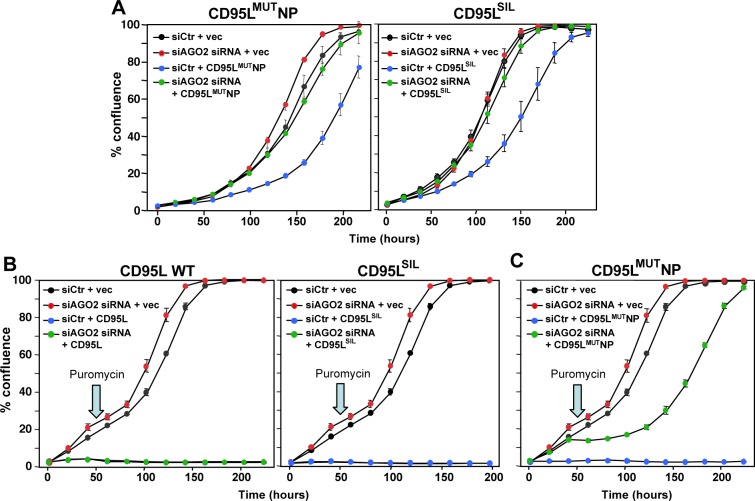
Figura 3-figure supplemento 1.Tutti i mutanti mRNA CD95L sono tossici attraverso l’RNAi.(A) Percentuale di confluenza cellulare nel tempo di cellule parentali HCT116 (sinistra) o Drosha k.o.(destra) dopo l’infezione con CD95MUTNP. I dati sono rappresentativi di tre esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche. Inset: Immagini di contrasto di fase delle cellule k.o. Drosha 9 giorni dopo l’infezione con vettore vuoto o CD95LMUTNP.(B) Percentuale di confluenza cellulare di HeyA8 CD95 k.o. cellule HeyA8 trasfettate con siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira AGO2 a seguito di infezione successiva con pLenti (vec) o pLenti CD95L vuoti. Inset: Western blot che mostra l’abbattimento dell’AGO2 umano.(C) In alto: autoradiografo sugli RNA abbattuti con il peptide legante Ago. In basso: Western blot analisi delle proteine Ago abbattute. v, pLenti; L, pLenti-CD95L che esprime le cellule.(D) Grafici a torta che mostrano il rapporto relativo di sRNA tirato giù con le proteine di Ago in wt e Drosha k.o. cellule. Rappresentati sono tutti gli importi di tutti gli sRNA che hanno contribuito almeno lo 0,01% al contenuto totale di RNA. Solo nelle cellule k.o. di Drosha è stata trovata una quantità significativa di CD95L derivati da Ago bound. Esse rappresentavano la 75a specie di sRNA più abbondante (freccia). Il numero medio di letture totali in sequenza (di due duplicati) è mostrato per ogni condizione.(E) In alto: numero di letture (normalizzate per milione) dei sei sRNA più abbondanti nel RISC delle cellule HCT116 wt-pLenti o -pLenti-CD95L. In basso: Numero di letture (per milione) dei primi cinque geni con sRNA più abbondanti nel RISC o di CD95L nel RISC delle celle HCT116 Drosha k.o. pLenti (v), o -pLenti-CD95L (L). Nota: il miR-21 non è incluso poiché è già mostrato nella riga superiore. Pannello in basso a destra: Abbondanza di sRNA derivati da CD95L di Ago bound. In tutti i pannelli è mostrata l’abbondanza di RNA nei quattro campioni. Rep 1 e Rep 2, replicare 1 e 2.(A) Percentuale di confluenza cellulare di cellule HeyA8 CD95 k.o. (cl. #11) trasfettate con siRNA non bersaglio (siCtr) o un pool di 4 siRNA bersaglio AGO2 a seguito di infezione successiva con pLenti vuoti (vec), pLenti CD95LMUTNP (sinistra), o pLenti CD95LSIL (destra).(B) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, pLenti CD95L WT (sinistra) o pLenti CD95LSIL (destra).(C) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, o pLenti CD95LMUTNP. In A le cellule sono state placcate dopo la selezione di puromicina. In(B) e(C) le cellule sono state placcate direttamente dopo l’infezione virale e la puromicina è stata aggiunta 50 ore dopo l’infezione (freccia).
Per testare l’ipotesi che le cellule Drosha k.o. erano più sensibili perché il loro RISC non era occupato da grandi quantità di miRNA non tossici e per determinare se CD95L mRNA potrebbe dare origine a sRNA che sono incorporati nel RISC, abbiamo tirato giù AGO1-4-associati RNA e analizzato la loro composizione in wt e Drosha k.o. cellule dopo aver espresso il mRNA CD95LMUTNP. Per il pull-down, abbiamo usato un peptide derivato dalla proteina GW182/TNRC6B GW182/TNRC6B, recentemente descritto per legarsi a tutte e quattro le proteine Ago(Hauptmann et al., 2015). Come previsto nelle cellule HCT116 wt, grandi quantità di sRNA (19-23nt in lunghezza) sono stati rilevati legati alle proteine Ago(Figura 3C). Sia AGO1 che AGO2 sono stati efficacemente abbattuti. Al contrario, nelle cellule k.o. Drosha, che non possono generare miRNA canonici, è stata rilevata solo una bassa quantità di sRNA, confermando l’assenza di miRNA nel RISC. Sorprendentemente, la quantità di proteine Ago abbattute è stata gravemente ridotta nonostante il fatto che queste cellule Drosha k.o. esprimano livelli comparabili di AGO2(Putzbach et al., 2017). Questo suggerisce che il peptide non ha avuto accesso alle proteine di Ago nelle cellule di Drosha k.o., presumibilmente perché si lega in modo efficiente solo alle proteine di Ago complessate con RNA come recentemente dimostrato(Elkayam et al., 2017).
L’analisi di tutti gli RNA di Ago-bound ha mostrato che nelle cellule wt, >98,4% degli RNA legati erano miRNA. Al contrario, solo il 34% degli RNA legati erano miRNA nelle cellule k.o. Drosha(Figura 3D e dati non mostrati). Questi includono miRNA che vengono elaborati indipendentemente da Drosha come miR-320a(Kim et al., 2016). Di conseguenza, questo miRNA è diventato una delle principali specie di RNA legate alle proteine Ago nelle cellule di Drosha k.o.(Figura 3D). Sia nelle cellule wt che in quelle k.o. di Drosha, è stato rilevato un aumento significativo di sRNA derivati da CD95L legati alle proteine Ago nelle cellule infettate dal virus CD95L rispetto alle cellule infettate dal vettore pLenti vuoto, come previsto. Essi corrispondono allo 0,0006% e allo 0,043% di tutti gli RNA legati a Ago nelle cellule wt e nelle cellule k.o. Drosha, rispettivamente. La tossicità dell’mRNA CD95L non era, quindi, dovuta al sovraccarico del RISC. In assenza della maggior parte dei miRNA, la quantità totale di RNA legati alle proteine di Ago nelle cellule k.o. di Drosha era circa il 10% della quantità legata ad Ago nelle cellule wt(Figura 3D). La riduzione dei miRNA legati ad Ago nelle cellule k.o. di Drosha(Figura 3E, riga superiore) è stata accompagnata da un sostanziale aumento del legame di altri sRNA alle proteine di Ago(Figura 3E, riga inferiore). È interessante notare che la quantità di sRNA derivati da Ago-bound CD95L era >100 volte superiore nelle cellule k.o. Drosha rispetto alle cellule wt (colonne rosse in Figura 3E ). Questi dati supportano la nostra ipotesi che le cellule del k.o. di Drosha siano più sensibili alla tossicità mediata da mRNA di CD95L a causa della loro capacità di assorbire nel RISC piccoli RNA derivati da CD95L più tossici in assenza della maggior parte dei miRNA.

Figura 3-figure supplement 1.Tutti i mRNA mutanti mRNA CD95L sono tossici attraverso l’RNAi.(A) Percentuale di confluenza cellulare nel tempo di cellule parentali HCT116 (a sinistra) o Drosha k.o.(a destra) dopo l’infezione con CD95MUTNP. I dati sono rappresentativi di tre esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche. Inset: Immagini a contrasto di fase delle cellule k.o. Drosha 9 giorni dopo l’infezione con vettore vuoto o CD95LMUTNP.(B) Percentuale di confluenza cellulare di HeyA8 CD95 k.o. cellule HeyA8 trasfettate con siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira AGO2 a seguito di infezione successiva con pLenti (vec) o pLenti CD95L vuoti. Inset: Western blot che mostra l’abbattimento dell’AGO2 umano.(C) In alto: autoradiografo sugli RNA abbattuti con il peptide legante Ago. In basso: Western blot analisi delle proteine Ago abbattute. v, pLenti; L, pLenti-CD95L che esprime le cellule.(D) Grafici a torta che mostrano il rapporto relativo di sRNA tirato giù con le proteine di Ago in wt e Drosha k.o. cellule. Rappresentati sono tutti gli importi di tutti gli sRNA che hanno contribuito almeno lo 0,01% al contenuto totale di RNA. Solo nelle cellule k.o. di Drosha è stata trovata una quantità significativa di CD95L derivati da Ago bound. Esse rappresentavano la 75a specie di sRNA più abbondante (freccia). Il numero medio di letture totali in sequenza (di due duplicati) è mostrato per ogni condizione.(E) In alto: numero di letture (normalizzate per milione) dei sei sRNA più abbondanti nel RISC delle cellule HCT116 wt-pLenti o -pLenti-CD95L. In basso: Numero di letture (per milione) dei primi cinque geni con sRNA più abbondanti nel RISC o di CD95L nel RISC delle celle HCT116 Drosha k.o. pLenti (v), o -pLenti-CD95L (L). Nota: il miR-21 non è incluso poiché è già mostrato nella riga superiore. Pannello in basso a destra: Abbondanza di sRNA derivati da CD95L di Ago bound. In tutti i pannelli è mostrata l’abbondanza di RNA nei quattro campioni. Rep 1 e Rep 2, replicare 1 e 2.(A) Percentuale di confluenza cellulare di cellule HeyA8 CD95 k.o. (cl. #11) trasfettate con siRNA non bersaglio (siCtr) o un pool di 4 siRNA bersaglio AGO2 a seguito di infezione successiva con pLenti vuoti (vec), pLenti CD95LMUTNP (sinistra), o pLenti CD95LSIL (destra).(B) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, pLenti CD95L WT (sinistra) o pLenti CD95LSIL (destra).(C) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, o pLenti CD95LMUTNP. In A le cellule sono state placcate dopo la selezione di puromicina. In(B) e(C) le cellule sono state placcate direttamente dopo l’infezione virale e la puromicina è stata aggiunta 50 ore dopo l’infezione (freccia).

Figura 3-figure supplemento 1.Tutti i mutanti mRNA CD95L mRNA sono tossici attraverso l’RNAi.(A) Percentuale di confluenza cellulare di cellule HeyA8 CD95 k.o. (cl. #11) trasfettate con siRNA non bersaglio (siCtr) o un pool di 4 siRNA bersaglio AGO2 a seguito di infezione successiva con pLenti vuoti (vec), pLenti CD95LMUTNP (sinistra), o pLenti CD95LSIL (destra).(B) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, pLenti CD95L WT (sinistra) o pLenti CD95LSIL (destra).(C) Percentuale di confluenza cellulare di cellule parentali HeyA8 trasfettate con un pool di 4 siRNA non bersaglio (siCtr) o un pool di 4 siRNA che prendono di mira l’AGO2 in seguito a successiva infezione con pLenti (vec) vuoti, o pLenti CD95LMUTNP. In A le cellule sono state placcate dopo la selezione di puromicina. In(B) e(C) le cellule sono state placcate direttamente dopo l’infezione virale e la puromicina è stata aggiunta 50 ore dopo l’infezione (freccia).
CD95L ORF è degradato in frammenti di sRNA che vengono poi caricati nel RISC
È interessante notare che non solo le proteine di Ago nelle cellule di Drosha k.o. legano molti più sRNA derivati da CD95L che nelle cellule di wt, ma anche la lunghezza di picco delle più abbondanti specie di RNA legate ad Ago è aumentata da 20 a 23 nt(Figura 4A, pannello superiore). Per determinare i siti all’interno dell’mRNA CD95L che hanno dato origine a piccoli RNA ingo-bound, abbiamo allineato tutti i piccoli RNA ingo-bound rilevati in tutte le condizioni alla sequenza ORF CD95L(Figura 4B e C). Abbiamo identificato 22 regioni nell’ORF CD95L che hanno dato origine a sRNA che erano legati dalle proteine di Ago (Figura 4B). Per determinare se questi sRNA si sono formati nel citosol e poi caricati nel RISC, abbiamo anche allineato tutti gli sRNA nella frazione di RNA totale isolato da CD95LMUTNP che esprime le cellule HCT116 Drosha k.o. con CD95L(Figura 4C). È interessante notare che sono state trovate regioni molto simili di piccoli RNA. Inoltre, la media così come il picco della distribuzione delle lunghezze di lettura degli sRNA legati alle proteine Ago era più piccola rispetto alla frazione totale di piccoli RNA(Figura 4A, pannello centrale), suggerendo che questi frammenti sono stati tagliati alla lunghezza appropriata sia prima di essere caricati nel RISC o dal RISC stesso. Questo era più ovvio per gli sRNA nel cluster 3(Figura 4B e C). Abbiamo anche notato che alcuni sRNA erano più abbondanti nella frazione di Ago-bound rispetto all’RNA totale rispetto a tutti gli altri RNA. Per determinare se questo tipo di elaborazione era specifico per le cellule HCT116 Drosha k.o., abbiamo analizzato gli sRNA derivati da Ago-bound CD95L nelle cellule HeyA8 CD95 k.o. dopo l’espressione di wt CD95L(Figura 4D) e li ha confrontati con la frazione di RNA totale (Figura 4E). Mentre abbiamo trovato meno letture di CD95L-derivate in queste cellule, la posizione generale di alcuni dei cluster di lettura si è sovrapposta a quella delle cellule k.o. Drosha e ancora una volta sia la media che il picco della distribuzione delle lunghezze di RNA era più piccola nella frazione di Ago-bound rispetto alla frazione di RNA totale(Figura 4A, pannello in basso). Insieme, questi dati suggeriscono che l’mRNA CD95L può essere elaborato in frammenti di RNA più piccoli, che vengono poi tagliati ad una lunghezza adeguata per l’incorporazione nel RISC.
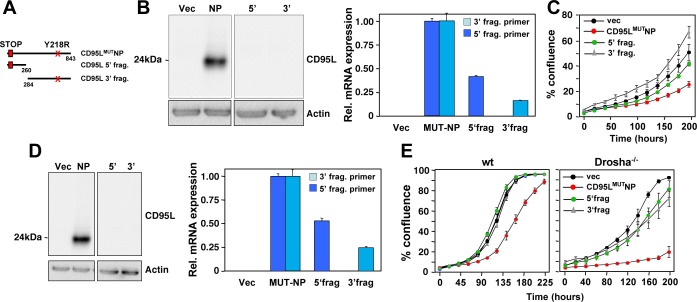
Figura 4-figure supplement 2.L’intero mRNA del CD95L dà origine a sRNA che si legano al RISC.struttura secondaria prevista dell’ORF del CD95L e tossicità degli sRNA derivati dal CD95L dopo la conversione in siRNA.i frammenti di CD95L sono meno tossici dell’mRNA del CD95L a lunghezza piena.(A) La distribuzione della lunghezza del CD95L derivato da CD95L viene letta in varie analisi.(B, C) Allineamento di lettura con CD95LMUTNP ORF delle analisi di sRNA tirato giù con le proteine Ago da HCT116 wt (B, in alto) e Drosha k.o. (B, in basso) cellule e di sRNA totali da HCT116 Drosha k.o. cellule (C) dopo l’infezione con CD95LMUTNP.(D, E) Leggere l’allineamento con il wt CD95L ORF delle analisi degli sRNA abbattuti con le proteine Ago (D) o degli sRNA totali(E) dalle cellule HeyA8 CD95 k.o. dopo l’infezione con il wt CD95L.(F) Percentuale di confluenza cellulare nel tempo di cellule parentali HCT116 (in alto) o Dicer k.o. (clone #43)(in basso) dopo l’infezione con CD95MUTNP. (Dicer k.o. clone #45, ha dato un risultato simile, dati non mostrati). I dati sono rappresentativi di due esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche.(G) RT-qPCR analisi RT-qPCR dei cluster 8 e 21 in HCT116 parentale, Dicer k.o. (clone # 43), e Drosha k.o. cellule dopo l’infezione con CD95MUTNP. Ogni barra rappresenta la media ± S.D. di tre repliche. v, vettore, L, CD95L che esprime le cellule.(A) L’RNA CD95LMUTNP è stato sottoposto ad un’analisi della struttura secondaria dell’RNA(http://rna.tbi.univie.ac.at) utilizzando le impostazioni predefinite. Sono mostrate le posizioni di 22 letture rappresentative dei 22 cluster di lettura. Le regioni con potenziale formazione duplex sono inscatolate. Gli oligonucleotidi che sono risultati tossici quando espressi come siRNA sono cerchiati.(B) Tossicità degli otto siRNA progettati utilizzando i CD95L-derivato piccoli frammenti di RNA legati ad Ago come le sequenze di filo antisenso 96 hr post-trasformazione nelle linee cellulari indicate. Ogni punto di dati rappresenta la media ±SE di tre repliche.(A) Schema dei diversi frammenti CD95L utilizzati.(B) A sinistra: analisi Western blot di HeyA8 CD95 k.o. cellule CD95L che sovraesprimono diversi RNA mutanti CD95L. A destra: Analisi RT-qPCR per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.(D) A sinistra: analisi Western blot di HCT116 Drosha k.o. cellule k.o. che esprimono in eccesso diversi RNA mutanti CD95L. A destra: RT-qPCR analisi per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(E) Percentuale di confluenza cellulare nel tempo di cellule HCT116 Drosha k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.
I nostri dati suggeriscono che l’mRNA CD95L, quando è sovraespresso, è tossico per le cellule a causa della formazione di sRNA Ago-bound che sono incorporati nel RISC e uccidono le cellule attraverso l’RNAi. Questo processo è indipendente da Drosha. Per determinare se Dicer è necessario per l’elaborazione di mRNA CD95L o per il caricamento degli sRNA nel RISC, abbiamo espresso CD95LMUTNP in wt e Dicer k.o. cellule HCT116(Figura 4F). Le cellule k.o. Dicer erano ancora sensibili alla tossicità indotta dall’espressione dell’mRNA CD95L, suggerendo che la tossicità dell’mRNA CD95L non richiede l’elaborazione da parte di Drosha o Dicer. Utilizzando primer qPCR personalizzati in tempo reale progettati per rilevare specificamente gli sRNA dai cluster 8 e 21, abbiamo rilevato, sia in wt e Dicer k.o. cellule che sovraesprimono CD95LMUTNP, frammenti da questi cluster(Figura 4G), dimostrando che Dicer non è coinvolto nell’elaborazione di mRNA CD95L.
Tutti i piccoli RNA riportati, derivati da CD95L, corrispondevano al filamento di senso dell’mRNA espresso, sollevando la questione di come possano essere elaborati in sRNA a doppio filamento in assenza di un filamento antisenso. Per ottenere una risposta preliminare a questa domanda, abbiamo sottoposto la sequenza di mRNA ORF CD95L ORF ad una previsione della struttura secondaria(Figura 4-figure supplement 1A). Secondo questa analisi, l’mRNA ORF CD95L forma una struttura strettamente piegata con molti degli sRNA dei 22 cluster che si giustappongono l’un l’altro in strutture simili a steli creando regioni di significativa complementarità. Queste possono fornire i duplex necessari per essere elaborati e caricati nel RISC. È interessante notare che alcune delle letture di giustapposizione sono state trovate in strutture duplex con sporgenze di 3′. Tre di questi oligonucleotidi (derivati dai cluster 7, 15 e 22) quando espressi come siRNA erano tossici per le cellule HeyA8, H460, M565 e 3LL(Figura 4-figure supplement 1B).
Per rispondere alla domanda se la sequenza completa di CD95L ORF fosse necessaria per produrre sRNA tossici, abbiamo generato due frammenti di CD95L, un frammento da 5′ (280 nt) e un frammento da 3′ (559 nt)(Figura 4-figure supplement 2A). Quando espresso in cellule CD95 k.o. HeyA8, nessuna proteina è stata rilevata con l’anticorpo anti-CD95L utilizzato, ma entrambi sono stati entrambi espressi in modo significativo a livello di RNA, anche se a livelli inferiori rispetto al wt CD95L(Figura 4-figure supplement 2B). Solo il frammento di 5′ ha causato una piccola riduzione della crescita delle cellule rispetto al CD95L a piena lunghezza(Figura 4-figure supplement 2C). Mentre nessuno dei due frammenti erano tossici per le cellule HCT116 wt, nel ipersensibile Drosha k.o. cellule HCT116 Drosha entrambi i frammenti hanno mostrato una debole tossicità(Figura 4-figure supplement 2D e E). Questi dati suggeriscono che i frammenti di CD95L mostrano una tossicità ridotta che potrebbe essere dovuta ai loro livelli di espressione più bassi e / o la loro incapacità di piegare e formare correttamente sRNA.
Per affrontare la questione se oltre al CD95L espresso in modo esogeno, gli mRNA di geni endogeni sarebbero anche elaborati e caricati nel RISC, abbiamo interrogato i set di dati RNA-Seq che abbiamo avuto da Ago pull-down e le analisi totali di piccoli RNA-Seq in HCT116 wt e HCT116 Drosha k.o. celle. I geni elaborati in modo simile all’mRNA CD95L dovrebbero essere espressi in modo significativo a livello di mRNA, e darebbero origine a sRNA multipli che si legano al RISC in modo più efficace nelle cellule k.o. Drosha rispetto alle cellule wt. Abbiamo interrogato tutti i set di dati utilizzando i seguenti parametri: Abbiamo contato ogni gene che aveva almeno una lettura unica che si allineava solo all’interno del suo mRNA con un conteggio di lettura di 10 o più. Abbiamo iniziato analizzando le letture di Ago bound e poi abbiamo confrontato la posizione e la dimensione delle letture in questi geni identificati con l’analisi del piccolo RNA totale delle cellule k.o. di Drosha che esprimono CD95L. Abbiamo trovato letture di Ago bound di 10 o più letture/posizione che si allineavano con 5629 geni nel genoma umano. Di questi geni ~ 10% erano non codificanti, 558 (~ 10%) erano geni codificanti proteine che soddisfacevano i nostri criteri per l’elaborazione, e 4498 geni (~ 80%) erano geni codificanti proteine che non lo erano. I dieci geni di codifica più abbondantemente elaborati con più letture univoche nel RISC sono mostrati nella Figura 5A e B e nella Figura 5-figure supplement 1A e B. Quelli più elaborati in modo più uniforme sono mostrati nella Figura 5B. In quasi tutti i casi, in modo simile al CD95L elaborato, la lunghezza media delle letture legate alle proteine Ago era più piccola di quella trovata nell’RNA totale(Figura 5A e Figura 5-figure supplement 1A). Mentre questi geni sono stati espressi in abbondanza, non erano gli mRNA più abbondanti in queste cellule(Figura 5C). Dei geni che codificano le proteine con letture legate alle proteine Ago, molti di essi erano altamente espressi, ma non sono stati elaborati in questa forma(Figura 5D). Quando tutte le piccole letture derivate da questi cinque geni sono stati allineati con il genoma umano, abbiamo osservato che: 1) le letture erano tra 5-10 volte più abbondanti nel RISC delle cellule prive di espressione Drosha e 2) erano caricate in modo altrettanto efficiente nelle cellule che esprimono il controllo vettoriale vuoto o il virus che esprime il CD95L (Figura 5-figure supplement 2).
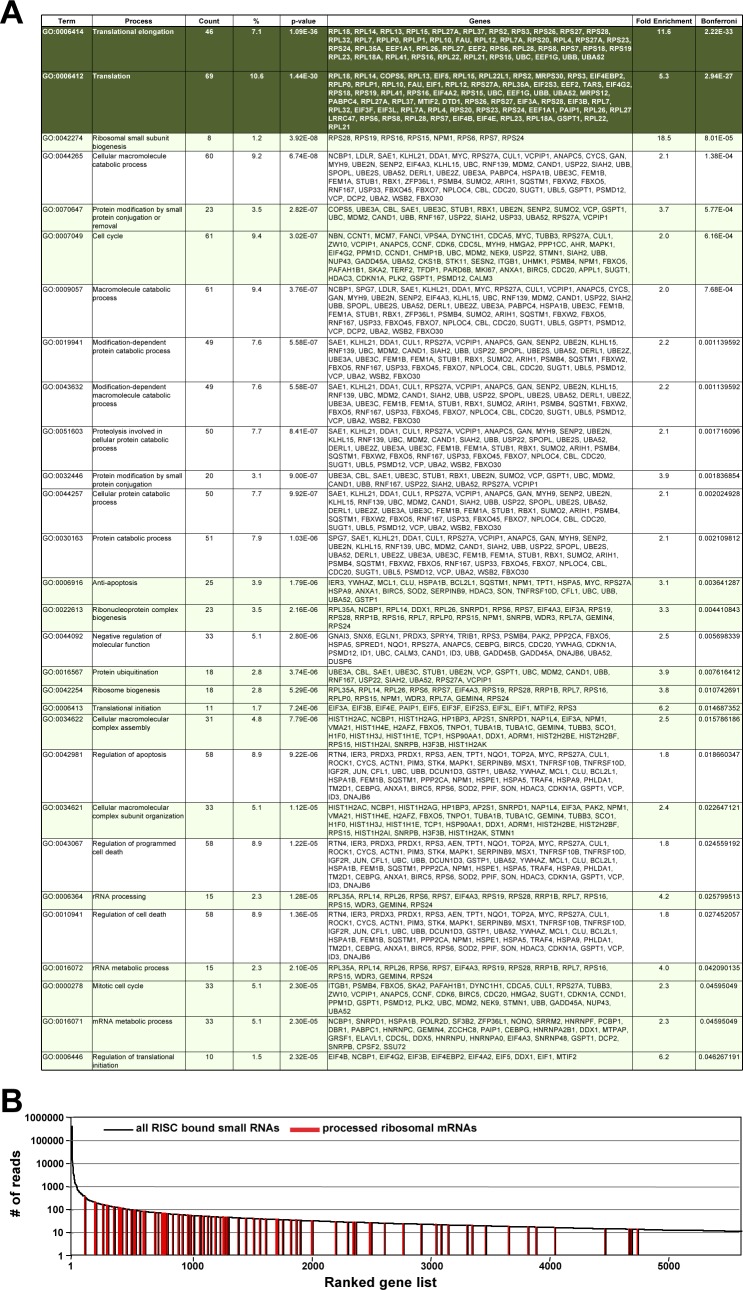
Figura 5-figure supplement 3.Gli mRNA endogeni sono elaborati e caricati nel RISC.gli mRNA endogeni sono elaborati e caricati nel RISC – geni aggiuntivi.la mappatura delle letture di Ago bound da cinque geni elaborati al genoma umano.i geni con letture multiple legate alle proteine di Ago sono coinvolti nella crescita delle cellule e nella traduzione delle proteine.(A) Distribuzione in lunghezza delle letture derivate da cinque dei dieci geni più abbondanti caricati nel RISC del CD95L che esprime le cellule HCT116 Drosha k.o.. I numeri tra parentesi indicano la classifica dei primi dieci geni più abbondanti con letture rilegate in Ago.(B) Allineamento delle letture dei cinque geni mostrati in A con linee orizzontali blu che rappresentano le posizioni mappate delle letture. Ogni linea blu rappresenta una singola lettura, con la sua lunghezza nel grafico proporzionale alla lunghezza della lettura. Piccoli RNA tirati giù con le proteine di Ago (in alto) o sRNA totali (in basso) da HCT116 e cellule k.o. Drosha dopo l’infezione con wt CD95L. *Questa pila contiene 14899 letture di cui 3000 sono state scelte a caso e tracciate.(C) Tutti i 4262 geni nelle cellule HCT116 Drosha k.o. che esprimono CD95L sono classificati secondo la massima espressione con più di 10 letture espresse come letture per kb per milione (RPKM). L’abbondanza dei sei geni mostrati in A e B è etichettata.(D) I geni classificati secondo la più alta abbondanza nel RISC delle cellule k.o. di Drosha. Le letture derivate dai cinque geni in A sono etichettate così come la posizione delle letture derivate dal CD95L.(E) Percentuale di confluenza cellulare nel tempo delle cellule parentali HCT116 e Drosha k.o..(F) Tossicità media dei semi di tutti i miRNA di Ago-bound e non miRNA (Altro) nelle cellule parentali HCT116 e Drosha k.o.. Le letture sono indicate come letture per milione (RPM). Il test Chi al quadrato è stato utilizzato per calcolare il valore p.(G) Percentuale di confluenza cellulare nel tempo di Drosha k.o. HCT116 cellule 24 ore dopo la trasfezione con 25 nM di SMARTpool non bersaglio (siCtr) o AGO2 SMARTpool siRNAs. Ogni punto di dati rappresenta la media ±SE di tre repliche. L’esperimento è rappresentativo di tre repliche biologiche.(A) Distribuzione della lunghezza di lettura derivata da 5 dei dieci geni più abbondanti tra i primi dieci caricati nel RISC di CD95L che esprime le cellule HCT116 Drosha k.o.. I numeri tra parentesi indicano la classifica dei dieci geni più abbondanti con letture rilegate in Ago.(B) Allineamento delle letture dei cinque geni mostrati in A con i loro rispettivi mRNA derivati da piccoli RNA estratti con le proteine di Ago (in alto) o piccoli RNA totali (in basso) da cellule HCT116 e Drosha k.o. dopo l’infezione con WT CD95L.Allineamento di tutte le letture derivate dai cinque geni mostrati in Figura 5A e B con il genoma umano. Sono mostrate tutte le 8 tracce di HCT116 wt e HCT116 Drosha k.o. cellule HCT116 infettate con il vettore di controllo pLenti (vec) o pLenti-CD95L in duplicato. Per ognuno dei geni l’asse Y è stato fissato sulla stessa scala.(A) Tutti i 558 geni di codifica delle proteine che sono stati elaborati in modo simile a CD95L e che hanno avuto letture legate alle proteine Ago sono stati sottoposti ad un’analisi DAVID GO. Mostrato sono tutti i cluster di geni significativamente arricchiti. I due cluster principali (verde scuro) si sono distinti con valori p molto bassi di arricchimento. I cluster evidenziati in verde sono collegati alla proliferazione cellulare (ciclo cellulare, antiapoptosi o traduzione delle proteine).(B) Tutti i 5629 geni con Ago rilegato legge (10 o più conteggi) classificati secondo la più alta abbondanza. Le posizioni degli mRNA delle proteine ribosomiali sono indicate in rosso negli mRNA classificati.
Il fatto che molti geni codificanti delle proteine altamente espressi non sono stati elaborati ha sollevato la questione se i geni elaborati e i geni caricati RISC sono caduti in alcuni cluster funzionali. Per iniziare ad affrontare questa domanda, abbiamo sottoposto tutti i geni codificanti elaborati con sRNA legati al RISC a un’analisi ontologica del gene DAVID. Sorprendentemente, i geni trovati in molti di questi cluster arricchiti sono stati coinvolti nella crescita delle cellule (righe verdi nella Figura 5-figure supplement 3A). Infatti, i geni più significativamente arricchiti sono stati coinvolti nella traduzione delle proteine (righe verdi scure in figura 5-figure supplement 3A). È interessante notare che, oltre alla ciclina D1, MYC(Figura 5B) e CDK6(Supplementary File 1) e molti fattori di iniziazione e di allungamento, 38 degli mRNA delle 50 proteine ribosomiali (76%) nell’analisi sono stati elaborati in questo modo(Figura 5-figure supplement 3B). Questo suggerisce che le proteine coinvolte nella traduzione delle proteine possono essere contrassegnate per essere elaborate e caricate nel RISC.
I nostri dati suggeriscono che gli mRNA dei geni codificanti delle proteine endogene possono essere elaborati e caricati nel RISC soprattutto in condizioni di basso miRNA. Ciò ha sollevato la questione se gli sRNA caricati nel RISC possano regolare negativamente la crescita delle cellule. Abbiamo notato che le cellule k.o. di Drosha sono cresciute costantemente più lentamente delle loro cellule wt parentali(Figura 5E). Sulla base dei nostri dati recenti sulla tossicità dei semi 6mer ci siamo chiesti se, in assenza della maggior parte delle cellule miRNA non tossiche e quindi protettive, gli sRNA sarebbero stati incorporati nel RISC che influenzano negativamente la crescita cellulare attraverso un basso livello di targeting del gene di sopravvivenza. Per affrontare questa domanda, abbiamo confrontato la tossicità dei semi 6mer di tutti gli sRNA legati alle proteine Ago nelle cellule wt o Drosha k.o.(Figura 5F). La differenza nella tossicità dei semi prevista è stata sorprendente. La maggior parte degli sRNA legati al RISC nelle cellule wt erano miRNA con bassa tossicità dei semi (colonne blu nella Figura 5F). Al contrario, nelle cellule k.o. di Drosha la maggior parte degli sRNA legati al RISC avevano un seme 6mer piuttosto tossico (colonne rosse nella Figura 5F). Questo suggerisce che tali sRNA possono costantemente mettere sotto pressione l’espressione dei geni della sopravvivenza. Per testare questo, abbiamo abbattuto l’AGO2 nelle cellule Drosha k.o. e questo trattamento ha portato ad un aumento del loro tasso di crescita che era ora simile a quello osservato nelle cellule wt(Figura 5G). Questi dati suggeriscono che gli sRNA endogeni legati al RISC possono regolare la crescita delle cellule attraverso il meccanismo DISE.
In sintesi, i nostri dati suggeriscono che gli si- e/o shRNA con determinate sequenze di semi presenti in CD95 e CD95L e l’intero CD95L ORF sono tossici per le cellule tumorali. L’mRNA del CD95L viene elaborato in sRNA che vengono caricati nel RISC che poi prendono di mira i geni critici di sopravvivenza. Ciò provoca la morte delle cellule in gran parte attraverso la tossicità dei semi 6mer. Questo processo è indipendente sia da Drosha che da Dicer. Inoltre, i nostri dati suggeriscono che un elevato contenuto di miRNA, “riempiendo” il RISC, potrebbe rendere le cellule meno sensibili a questa forma di morte cellulare e che gli mRNA endogeni multipli vengono elaborati e caricati nel RISC e possono regolare il destino delle cellule.

Figura 4-figure supplement 2.L’intero mRNA CD95L dà origine a sRNA che si legano al RISC.struttura secondaria prevista dell’ORF CD95L e tossicità degli sRNA derivati da CD95L dopo la conversione in siRNA.i frammenti CD95L sono meno tossici degli mRNA CD95L a lunghezza intera.(A) La distribuzione della lunghezza del CD95L derivato da CD95L viene letta in varie analisi.(B, C) Allineamento di lettura con CD95LMUTNP ORF delle analisi di sRNA tirato giù con le proteine Ago da HCT116 wt (B, in alto) e Drosha k.o. (B, in basso) cellule e di sRNA totali da HCT116 Drosha k.o. cellule (C) dopo l’infezione con CD95LMUTNP.(D, E) Leggere l’allineamento con il wt CD95L ORF delle analisi degli sRNA abbattuti con le proteine Ago (D) o degli sRNA totali(E) dalle cellule HeyA8 CD95 k.o. dopo l’infezione con il wt CD95L.(F) Percentuale di confluenza cellulare nel tempo di cellule parentali HCT116 (in alto) o Dicer k.o. (clone #43)(in basso) dopo l’infezione con CD95MUTNP. (Dicer k.o. clone #45, ha dato un risultato simile, dati non mostrati). I dati sono rappresentativi di due esperimenti indipendenti. Ogni punto di dati rappresenta la media ±SE di tre repliche.(G) RT-qPCR analisi RT-qPCR dei cluster 8 e 21 in HCT116 parentale, Dicer k.o. (clone # 43), e Drosha k.o. cellule dopo l’infezione con CD95MUTNP. Ogni barra rappresenta la media ± S.D. di tre repliche. v, vettore, L, CD95L che esprime le cellule.(A) L’RNA CD95LMUTNP è stato sottoposto ad un’analisi della struttura secondaria dell’RNA(http://rna.tbi.univie.ac.at) utilizzando le impostazioni predefinite. Sono mostrate le posizioni di 22 letture rappresentative dei 22 cluster di lettura. Le regioni con potenziale formazione duplex sono inscatolate. Gli oligonucleotidi che sono risultati tossici quando espressi come siRNA sono cerchiati.(B) Tossicità degli otto siRNA progettati utilizzando i CD95L-derivato piccoli frammenti di RNA legati ad Ago come le sequenze di filo antisenso 96 hr post-trasformazione nelle linee cellulari indicate. Ogni punto di dati rappresenta la media ±SE di tre repliche.(A) Schema dei diversi frammenti CD95L utilizzati.(B) A sinistra: analisi Western blot di HeyA8 CD95 k.o. cellule CD95L che sovraesprimono diversi RNA mutanti CD95L. A destra: Analisi RT-qPCR per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.(D) A sinistra: analisi Western blot di HCT116 Drosha k.o. cellule k.o. che esprimono in eccesso diversi RNA mutanti CD95L. A destra: RT-qPCR analisi per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(E) Percentuale di confluenza cellulare nel tempo di cellule HCT116 Drosha k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.
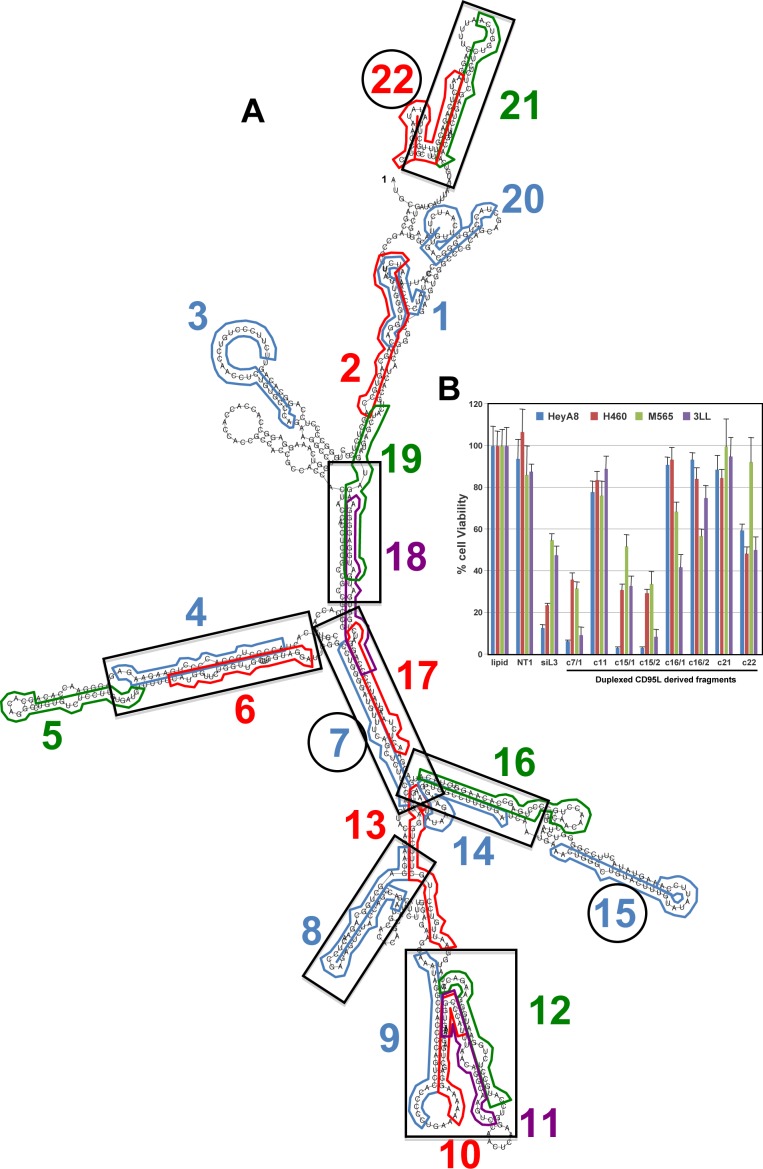
Figura 4-figure supplemento 1.Struttura secondaria prevista di CD95L ORF e la tossicità di CD95L-derivato sRNA dopo la conversione in siRNA.(A) L’RNA CD95LMUTNP è stato sottoposto ad un’analisi della struttura secondaria dell’RNA(http://rna.tbi.univie.ac.at) utilizzando le impostazioni predefinite. Sono indicate le posizioni di 22 letture rappresentative dei 22 cluster di lettura. Le regioni con potenziale formazione duplex sono inscatolate. Gli oligonucleotidi che sono risultati tossici quando espressi come siRNA sono cerchiati.(B) Tossicità degli otto siRNA progettati utilizzando i CD95L-derivato piccoli frammenti di RNA legati ad Ago come le sequenze di filo antisenso 96 hr post-trasformazione nelle linee cellulari indicate. Ogni punto di dati rappresenta la media ±SE di tre repliche.

Figura 4-figure supplemento 2.CD95L frammenti sono meno tossici di mRNA CD95L mRNA a piena lunghezza.(A) Schema dei diversi frammenti di CD95L utilizzati.(B) A sinistra: analisi Western blot di HeyA8 CD95 k.o. cellule CD95 k.o. che sovraesprimono diversi RNA mutanti CD95L. A destra: Analisi RT-qPCR per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(C) Percentuale di confluenza cellulare nel tempo delle cellule HeyA8 CD95 k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.(D) A sinistra: analisi Western blot di HCT116 Drosha k.o. cellule k.o. che esprimono in eccesso diversi RNA mutanti CD95L. A destra: RT-qPCR analisi per CD95L degli stessi campioni utilizzando primer che rilevano la metà 5′ o 3′ dell’mRNA. I dati sono rappresentativi di due esperimenti indipendenti. Ogni barra rappresenta la media ±S.D. di tre repliche.(E) Percentuale di confluenza cellulare nel tempo di cellule HCT116 Drosha k.o. dopo aver espresso il mutante CD95L o frammenti. I dati sono rappresentativi di due esperimenti indipendenti. I valori sono stati calcolati a partire da campioni fatti in triplice copia indicati come media ±SE.

Figura 5-figure supplemento 3.Gli mRNA endogeni sono elaborati e caricati nel RISC.gli mRNA endogeni sono elaborati e caricati nel RISC – geni aggiuntivi.la mappatura delle letture di Ago bound da cinque geni elaborati al genoma umano.i geni con letture multiple legate alle proteine di Ago sono coinvolti nella crescita delle cellule e nella traduzione delle proteine.(A) Distribuzione in lunghezza delle letture derivate da cinque dei dieci geni più abbondanti caricati nel RISC del CD95L che esprime le cellule HCT116 Drosha k.o.. I numeri tra parentesi indicano la classifica dei primi dieci geni più abbondanti con letture rilegate in Ago.(B) Allineamento delle letture dei cinque geni mostrati in A con linee orizzontali blu che rappresentano le posizioni mappate delle letture. Ogni linea blu rappresenta una singola lettura, con la sua lunghezza nel grafico proporzionale alla lunghezza della lettura. Piccoli RNA tirati giù con le proteine di Ago (in alto) o sRNA totali (in basso) da HCT116 e cellule k.o. Drosha dopo l’infezione con wt CD95L. *Questa pila contiene 14899 letture di cui 3000 sono state scelte a caso e tracciate.(C) Tutti i 4262 geni nelle cellule HCT116 Drosha k.o. che esprimono CD95L sono classificati secondo la massima espressione con più di 10 letture espresse come letture per kb per milione (RPKM). L’abbondanza dei sei geni mostrati in A e B è etichettata.(D) I geni classificati secondo la più alta abbondanza nel RISC delle cellule k.o. di Drosha. Le letture derivate dai cinque geni in A sono etichettate così come la posizione delle letture derivate dal CD95L.(E) Percentuale di confluenza cellulare nel tempo delle cellule parentali HCT116 e Drosha k.o..(F) Tossicità media dei semi di tutti i miRNA di Ago-bound e non miRNA (Altro) nelle cellule parentali HCT116 e Drosha k.o.. Le letture sono indicate come letture per milione (RPM). Il test Chi al quadrato è stato utilizzato per calcolare il valore p.(G) Percentuale di confluenza cellulare nel tempo di Drosha k.o. HCT116 cellule 24 ore dopo la trasfezione con 25 nM di SMARTpool non bersaglio (siCtr) o AGO2 SMARTpool siRNAs. Ogni punto di dati rappresenta la media ±SE di tre repliche. L’esperimento è rappresentativo di tre repliche biologiche.(A) Distribuzione della lunghezza di lettura derivata da 5 dei dieci geni più abbondanti tra i primi dieci caricati nel RISC di CD95L che esprime le cellule HCT116 Drosha k.o.. I numeri tra parentesi indicano la classifica dei dieci geni più abbondanti con letture rilegate in Ago.(B) Allineamento delle letture dei cinque geni mostrati in A con i loro rispettivi mRNA derivati da piccoli RNA estratti con le proteine di Ago (in alto) o piccoli RNA totali (in basso) da cellule HCT116 e Drosha k.o. dopo l’infezione con WT CD95L.Allineamento di tutte le letture derivate dai cinque geni mostrati in Figura 5A e B con il genoma umano. Sono mostrate tutte le 8 tracce di HCT116 wt e HCT116 Drosha k.o. cellule HCT116 infettate con il vettore di controllo pLenti (vec) o pLenti-CD95L in duplicato. Per ognuno dei geni l’asse Y è stato fissato sulla stessa scala.(A) Tutti i 558 geni di codifica delle proteine che sono stati elaborati in modo simile a CD95L e che hanno avuto letture legate alle proteine Ago sono stati sottoposti ad un’analisi DAVID GO. Mostrato sono tutti i cluster di geni significativamente arricchiti. I due cluster principali (verde scuro) si sono distinti con valori p molto bassi di arricchimento. I cluster evidenziati in verde sono collegati alla proliferazione cellulare (ciclo cellulare, antiapoptosi o traduzione delle proteine).(B) Tutti i 5629 geni con Ago rilegato legge (10 o più conteggi) classificati secondo la più alta abbondanza. Le posizioni degli mRNA delle proteine ribosomiali sono indicate in rosso negli mRNA classificati.
Figura 5-figure supplemento 1.Gli mRNA endogeni sono elaborati e caricati nel RISC – geni aggiuntivi.(A) Distribuzione in lunghezza delle letture derivate da 5 dei dieci geni più abbondanti caricati nel RISC di CD95L che esprimono le cellule HCT116 Drosha k.o.. I numeri tra parentesi indicano la classifica dei dieci geni più abbondanti con letture rilegate in Ago.(B) Allineamento delle letture dei cinque geni mostrati in A con i loro rispettivi mRNA derivati da piccoli RNA estratti con le proteine di Ago (in alto) o piccoli RNA totali (in basso) da cellule HCT116 e Drosha k.o. dopo l’infezione con WT CD95L.

Figura 5-figure supplemento 2.Mappatura di Ago legato legge da cinque geni elaborati al genoma umano.Allineamento di tutte le letture derivate dai cinque geni mostrati in Figura 5A e B con il genoma umano. Sono mostrate tutte le 8 tracce di cellule HCT116 wt e HCT116 Drosha k.o. infettate con il vettore di controllo pLenti (vec) o pLenti-CD95L in duplicato. Per ognuno dei geni l’asse Y è stato fissato sulla stessa scala.

Figura 5-figure supplemento 3.I geni con letture multiple legate alle proteine Ago sono coinvolti nella crescita delle cellule e nella traduzione delle proteine.(A) Tutti i 558 geni codificanti delle proteine che sono stati elaborati in modo simile al CD95L e che avevano letture legate alle proteine Ago sono stati sottoposti ad un’analisi DAVID GO. Mostrato sono tutti i cluster di geni significativamente arricchiti. I due cluster principali (verde scuro) si sono distinti con valori p molto bassi di arricchimento. I cluster evidenziati in verde sono collegati alla proliferazione cellulare (ciclo cellulare, antiapoptosi o traduzione delle proteine).(B) Tutti i 5629 geni con Ago rilegato legge (10 o più conteggi) classificati secondo la più alta abbondanza. Le posizioni degli mRNA delle proteine ribosomiali sono indicate in rosso negli mRNA classificati.
Discussione
Abbiamo recentemente riportato una nuova forma di morte cellulare che è stata osservata dopo l’espressione dei si/shRNA progettati dalle sequenze di CD95/CD95L mRNA(Putzbach et al., 2017). Più recentemente abbiamo descritto che le cellule muoiono a causa di una perdita di geni multipli di sopravvivenza attraverso un meccanismo che chiamiamo 6mer seed toxicity(Gao et al., 2018). I si/shRNA più tossici derivati da CD95 o CD95L sono stati trovati nell’ORF di CD95L(Putzbach et al., 2017). Questo indicava che l’mRNA del CD95L era tossico.
Ora mostriamo che l’espressione del mRNA CD95L mRNA a lunghezza intera innesca una tossicità indipendente dal prodotto proteico e dall’apoptosi canonica. Questo è intrigante se si considera che studi precedenti hanno dimostrato che l’espressione transgenica del CD95L tramite virus ha ucciso più cellule tumorali che erano completamente resistenti all’apoptosi mediata dal CD95(ElOjeimy et al., 2006; Hyer et al., 2000; Sudarshan et al., 2005; Sun et al., 2012). Questi risultati sono stati interpretati come CD95L intracellulare che scatena l’apoptosi in modo più efficiente rispetto a quando viene aggiunto alle cellule. Tuttavia, ora forniamo una spiegazione alternativa: sia la proteina CD95L che l’mRNA sono tossici per le cellule attraverso meccanismi distinti. La proteina induce l’apoptosi, e l’mRNA induce la tossicità attraverso un meccanismo basato sull’RNAi.
Dimostriamo che Dicer e Drosha non sono coinvolti nella generazione dei frammenti derivati dal CD95L di Ago-bound, ma ci sono diversi RNasi candidati che sono in grado di elaborare gli mRNA. Date le differenze nella distribuzione della lunghezza tra i frammenti di RNA citosolico e quelli di Agobound, è probabile che i frammenti intermedi derivati da CD95L siano incorporati nel RISC e poi tagliati alla lunghezza appropriata da Ago. In effetti, un meccanismo simile è noto che si verifica durante la maturazione del miR-451 eritropoietico, dove il pre-miRNA viene prima scisso da AGO2 e poi tagliato alla fine dei 3′ alla forma finale matura dall’esoribonucleasi PARN (Yodaet al., 2013). Inoltre, un processo simile si verifica con la classe di RNA di Ago-bound, recentemente identificata, chiamata agotrons(Hansen et al., 2016), che consiste in un introne excised caricato nel RISC in modo indipendente dal pre-processing Drosha o Dicer. Una volta tagliati alla dimensione appropriata, gli RNA guida in complesso con il RISC possono regolare l’espressione genica attraverso l’RNAi.
I nostri dati forniscono la prima prova della tossicità di un cDNA sovraespresso che esercita una tossicità attraverso un meccanismo dipendente dall’RNAi. È stato dimostrato per la prima volta nelle piante che i transgeni sovraespressivi possono essere convertiti in brevi sequenze di RNAi attivo RNAi(Hamilton e Baulcombe, 1999). I nostri dati sugli effetti dell’RNA CD95L sovraespresso, sebbene meccanicamente distinti da quanto riportato nelle piante, possono essere il primo esempio di un transgene che determina il destino delle cellule attraverso il meccanismo RNAi nelle cellule dei mammiferi. Gli sRNA derivati dal CD95L agiranno probabilmente in modo simile al miRNA prendendo di mira 3’UTR di geni di sopravvivenza attraverso la tossicità dei semi 6mer(Gao et al., 2018). È stato dimostrato che gli mRNA contenenti CAG-repeat-contenenti mRNA inducono la formazione di sRNA e la tossicità cellulare attraverso l’RNAi(Bañez-Coronel et al., 2012). Tuttavia, abbiamo recentemente riportato che questi sCAG probabilmente colpiscono regioni CUG completamente complementari contenenti ripetizioni di CUG negli ORF di geni critici per la sopravvivenza cellulare in un meccanismo simile a quello degli siRNA(Murmann et al., 2018a; Murmann et al., 2018b).
Oltre all’attività di mRNA CD95L aggiunto esogeneamente, forniamo anche la prova che alcuni mRNA codificanti endogeni possono essere elaborati in sRNA multipli che vengono poi caricati nel RISC. Piccoli mRNA derivati da mRNA sono stati segnalati per essere legati a tutte e quattro le proteine Ago prima(Burroughs et al., 2011). Tuttavia, erano molto piccoli in numero e quantità e nessuna specifica funzione cellulare poteva essere attribuita ad essi. Al contrario, ora dimostriamo che nelle cellule con l’elaborazione di miRNA disabilitati, circa il 3% dei geni codificanti delle proteine può essere elaborato in questo modo e che questi geni sono fortemente arricchiti in termini GO associati alla traduzione delle proteine (iniziazione e allungamento) così come alla regolazione del ciclo cellulare e alla proliferazione cellulare. Essi contengono un gran numero di geni 5′ TOP (cioè il 76% di tutte le proteine ribosomiali) necessari per la traduzione delle proteine(Yamashita et al., 2008). I nostri risultati possono essere rilevanti per situazioni in cui i livelli di miRNA cellulari sono bassi, come nelle cellule staminali somatiche(Morin et al., 2008) o nei tumori avanzati che sono caratterizzati da una riduzione globale dei miRNA(Lu et al., 2005). È intrigante notare che i geni che fanno parte di questo gruppo rientrano in cluster GO che si sovrappongono a quelli che abbiamo trovato downregolati nelle cellule sottoposte a DISE(Gao et al., 2018; Putzbach et al., 2017).
Una domanda importante che sorge dai nostri dati è se l’mRNA CD95L è tossico in vivo. Noi e altri abbiamo notato l’upregulation del CD95L in condizioni di stress multiplo, come dopo il trattamento con chemioterapia ((Friesen et al., 1999) e dati non mostrati). Mentre la quantità di mRNA CD95L e il livello di upregulation da solo potrebbe non essere sufficiente per essere tossico, potrebbe essere la combinazione di frammenti di RNA multipli, derivati da mRNA multipli diversi che vengono generati per uccidere le cellule(Putzbach et al., 2018). Noi consideriamo il CD95L come uno dei tanti RNA che hanno questa attività. È improbabile che il CD95L sia l’unico gene il cui mRNA è tossico per le cellule, poiché questo livello di tossicità basato sull’mRNA sarebbe ridondante con la potente capacità di uccidere la proteina CD95L. Inoltre, l’upregistrazione di un mRNA che, da solo, potrebbe decimare le cellule che altrimenti avrebbero bisogno di upregolare l’mRNA per svolgere la loro funzione biologica in primo luogo, come ad esempio nelle cellule T attivate che upregolano il CD95L per montare una risposta immunitaria, sarebbe autolesionista. Pertanto, la natura ha probabilmente distribuito questa capacità tossicologica basata sull’mRNA su molti geni del genoma per evitare di attivarla quando uno qualsiasi di questi geni viene upregolato durante specifici processi cellulari. È più probabile che esista un’intera rete di questi geni in grado di rilasciare sRNA tossici quando si incontra lo stimolo appropriato. Coerentemente con questa ipotesi abbiamo recentemente identificato altri geni che contengono sequenze che, una volta convertiti in shRNA, uccidono le cellule tumorali attraverso la tossicità dei semi 6mer(Patel e Peter, 2018). Il lavoro futuro sarà finalizzato allo studio dei geni codificanti che si trovano nel RISC, all’identificazione di altri geni e del meccanismo attraverso il quale vengono elaborati e in quali condizioni uccidono le cellule.
Materiali e metodi
| Tipo di reagente (specie) o risorsa | Designazione | Fonte o riferimento | Identificatori | Ulteriori informazioni |
|---|---|---|---|---|
| Gene (Homo sapiens) | CD95L | NA | NM_000639 | |
| Gene (H. sapiens) | CD95 | NA | NM_00000043 | |
| Linea cellulare (H. sapiens) | MCF-7 | ATCC | ATCC: HTB-22 | Adenocarcinoma umano della ghiandola mammaria, seno; derivato da sede metastatica: versamento pleurico |
| Linea cellulare (H. sapiens) | MCF-7 CD95 ΔshR6 clone #21 | questo documento | NA | MCF-7 CD95 ΔshR6 clone #21 con cancellazione omozigote 227 nucleotide del sito target shR6 in CD95 (chr10:89,008,920-89,009,146; Human Dec. 2013 GRCh38/hg38 assembly) prodotto con tecnologia CRISPR/Cas9; verificato omozigote CD95 knockout della proteina CD95 |
| Linea cellulare (H. sapiens) | MCF-7 CD95 cancellazione clone FA4 | questo documento | NA | MCF-7 CD95 clone di cancellazione FA4 con una delezione omozigote dell’intero gene CD95 (chr10:88,990,657-89,015,785; Human Dec. 2013 GRCh38/hg38 assembly) prodotto utilizzando la tecnologia CRISPR/Cas9; verificato knockout della proteina omozigote CD95 |
| Linea cellulare (H. sapiens) | HeyA8 | PMID: 4016745 | RRID: CVCL_8878 | Adenocarcinoma sieroso ovarico umano adenocarcinoma ovarico di alto grado; derivato dalle cellule di Hey dei genitori (RRID: CVCL_0297) |
| Linea cellulare (H. sapiens) | HeyA8 shR6 k.o. clone #11, HeyA8 CD95 k.o. | PMID: 29063830 | NA | HeyA8 CD95 k.o. clone con una delezione nucleotidica omozigote 227 del sito target shR6 in CD95 (chr10:89,008,920-89,009,146; Human Dec. 2013 GRCh38/hg38 assembly) prodotto con tecnologia CRISPR/Cas9; verificato knockout della proteina omozigote CD95 |
| Linea cellulare (H. sapiens) | HCT116 | Collezione coreana per le culture di tipo (KCTC) | KCTC: cat#HC19023; ATCC: CCL_247 | Carcinoma colorettale umano |
| Linea cellulare (H. sapiens) | Drosha-/-; Drosha-/-clone #40 | Collezione coreana per le culture di tipo (KCTC); PMID: 26976605 | KCTC: cat#HC19020 | HCT116 clone #40 con knockout omozigote della proteina Drosha; knockout ottenuto utilizzando CRISPR/Cas9 che ha portato ad un singolo inserimento nucleotidico in un allele e ad una delezione di 26 nucleotidi nell’altro |
| Linea cellulare (H. sapiens) | Dicer-/-; Dicer-/-clone #43 | Collezione coreana per le culture di tipo (KCTC); PMID: 26976606 | KCTC: cat#HC19023 | HCT116 clone #43 con knockout omozigote della proteina Dicer; knockout ottenuto utilizzando CRISPR/Cas9 che ha portato ad un inserimento di tre nucleotidi e ad una delezione di 14 nucleotidi in un allele e ad una delezione di 35 nucleotidi nell’altro |
| Linea cellulare (H. sapiens) | Dicer-/-; Dicer-/- clone #45 | Collezione coreana per le culture di tipo (KCTC); PMID: 26976607 | KCTC: cat#HC19024 | HCT116 clone #45 con knockout omozigote della proteina Dicer; knockout ottenuto utilizzando CRISPR/Cas9 che ha portato ad una delezione di 53 nucleotidi in un allele e ad una delezione di 28 nucleotidi nell’altro |
| Linea cellulare (H. sapiens) | 293T | ATCC | ATCC: CRL-3216 | Derivato da cellule HEK293 (ATCC: CRL-1573); antigene T espresso di grandi dimensioni; utilizzato per il confezionamento di virus |
| Linea cellulare (H. sapiens) | H460 | ATCC | ATCC: #HTB-177 | Carcinoma pleurico da versamento polmonare umano |
| Linea cellulare (Mus muscolo muscoloso) | 3LL | ATCC | ATCC #CRL-1642 | Carcinoma polmonare di Lewis del topo |
| Linea cellulare (Mus muscolo muscoloso) | M565 | PMID: 25366259 | NA | Carcinoma epatocellulare del topo isolato da un tumore naturale in un fondo floxato CD95 |
| Anticorpo | anti-umano AGO1 (coniglio monoclonale) | Segnalazione cellulare | Segnalazione della cella #5053 | 1:2000; per western blot; primario Ab |
| Anticorpo | anti-umano AGO1 (coniglio policlonale) | Abcam | Abcam #98056 | 1:2000; per western blot; primario Ab |
| Anticorpo | anti-umano AGO2 (coniglio policlonale) | Abcam | Abcam #32381 | 1:500; per western blot; primario Ab |
| Anticorpo | Capra anti-coniglio, IgG-HRP | Biotecnologie del Sud | Southern Biotech: cat#SB-4030-05 | 1:5000; per western blot; secondario Ab |
| Anticorpo | Anticorpo antiargonauta-2 (monoclonale di coniglio) [EPR10411] | Abcam | Abcam #186733 | 1:1200; per western blot; primario Ab |
| Anticorpo | Anticorpo CD178 anti-umano (Mouse IgG1) Clone G247-4 | BD Pharmingen | BD Pharmingen #556387 | 1 μg/ml; per western blot; Ab primario |
| Anticorpo | Anti-CD95 (coniglio policlonale, C-20) | Santa Cruz | Santa Cruz #sc-715 (da quando non è più in produzione) | 1:1000; per western blot; primario Ab |
| Anticorpo | Capra anti-coniglio, IgG-HRP | Segnalazione cellulare | Segnalazione cellulare: cat#7074 | 1:2000; per western blot; secondario Ab |
| Anticorpo | Capra anti-topo; IgG1-HRP | Biotecnologie del Sud | Southern BioTech: cat#1070-05 | 1:5000; per western blot; secondario Ab |
| Reagente proteico ricombinante | LzCD95L | PMID: 14504390 | NA | Leucina cerniera con etichetta CD95L; proteina ricombinante |
| Composto chimico | CellTiter-Glo | Promega | Promega #G7570 | Rileva il rilascio di ATP come surrogato della morte cellulare; la lettura è in fluorescenza |
| Composto chimico | ioduro di propidio | Sigma-Aldrich | Sigma-Aldrich: cat#P4864 | Utilizzato per l’analisi citometrica a flusso subG1 |
| Composto chimico | puromicina | Sigma-Aldrich | Sigma-Aldrich: cat#P9620 | Utilizzato per la selezione delle cellule che esprimono le cassette di resistenza alla puromicina |
| Composto chimico | 2′,7′-diclorodiidrofluoresceina diacetato di 2′,7′ -diclorodiidrofluoresceina | Termofisher Scientific | Termofisher Scientific #D399 | Colorante utilizzato per rilevare la produzione di ROS |
| Composto chimico | zVAD-fmk | Sigma-Aldrich | Sigma-Aldrich: cat#V116 | Usato a 20 uM; inibitore della pan caspasi |
| Reagente DNA ricombinante | pLenti-GIII-CMV- RFP-2A-Puro vettore; pLenti | ABM Inc. | NA | pLenti controllano il vettore lentivirale vuoto; trasporta una cassetta di resistenza RFP-2a-puromicina |
| Reagente DNA ricombinante | pLenti-CD95L | questo documento | NA | pLenti-GIII-CMV-RFP-2A-Puro vettore che esprime il cDNA cDNA di tipo selvatico umano CD95L (NM_000639.2); utilizzato per esprimere il CD95L umano in caso di infezione da particelle lentivirali |
| Reagente DNA ricombinante | pLenti-CD95LMUT | questo documento | NA | pLenti-GIII-CMV-RFP-2A-Puro vettore che esprime il cDNA CD95L umano (NM_000639.2) con due sostituzioni nucleotidiche nel codone 218 (TAT – > CGT) con conseguente sostituzione della tirosina con l’arginina (mutazione Y218R); incapace di legare CD95 |
| Reagente DNA ricombinante | pLenti-CD95LMUTNP | questo documento | NA | pLenti-GIII-CMV-RFP-2A-Puro vettore che esprime il cDNA CD95L umano (NM_000639.2) sia con la mutazione Y218R che con una singola sostituzione nucleotidica al secondo codone (CAG – > TAG), con conseguente arresto prematuro del codone subito dopo il codone di partenza |
| Reagente DNA ricombinante | pLenti-CD95LSIL | questo documento | NA | pLenti-GIII-CMV-RFP-2A-Puro vettore che esprime il cDNA CD95L umano (NM_000639.2) con tutti i codoni contenenti mutazioni sinonimetiche ad eccezione dei codoni selezionati nel dominio ricco di prolina per soddisfare i criteri di sintesi IDT |
| Costruttore trasformato | ponteggio gRNA | PMID: 23287722 | IDT: sintetizzato come blocco genetico | 455 nucleotide CRISPR/Cas9 gRNA scaffold sintetizzato come blocco genetico; contiene promotore, scaffold gRNA, sequenza target e sequenza di terminazione; scaffold trascrive i gRNA che bersagliano l’endonucleasi Cas9 da tagliare nei siti target; le sequenze target consistono di 19 nucleotidi che sono complementari al sito target di scelta; co-trasformati con Cas9 per catalizzare la scissione. |
| Reagente DNA ricombinante | pLenti-CD95LMUTNP (G258A) | Questo documento | NA | pLenti-GIII-CMV-RFP-2A-Puro vettore che esprime il cDNA CD95L umano (NM_000639.2) con la mutazione Y218R, e due ulteriori sostituzioni di nucleotidi singoli; uno al secondo codone (CAG – > TAG), con conseguente arresto prematuro del codone subito dopo il codone di partenza, e un altro, G258A, con conseguente sostituzione di una metionina con un’isoleucina, eliminando così il sito alternativo di partenza traslazionale. |
| Costruttore trasformato | pMJ920 Plasmide Cas9 | Addgene; PMID: 23386978 | Addgene: cat#42234 | Plasmide che esprime un Cas9 ottimizzato per il codone umano, contrassegnato con GFP e HA; utilizzato per esprimere il Cas9 per le cancellazioni mediate da CRISPR. |
| Composto chimico | Lipofectamina 2000 | ThermoFisher Scientific | ThermoFisher Scientific: cat#11668019 | Reagente di trasformazione |
| Composto chimico | Lipofectamina RNAiMAX | ThermoFisher Scientific | ThermoFisher Scientific: cat#13778150 | Reagente di trasfezione; utilizzato per la trasfezione di piccoli RNA come i siRNA |
| Saggio commerciale o kit | Kit di clonazione PCR StrataClone Blunt PCR | Tecnologie Agilent | Agilent Technologies: cat#240207 | Utilizzato per clonare le impalcature in gRNA nel plasmide pSC-B |
| Reagente genetico | Taqman Gene expression master mix | ThermoFisher Scientific | #4369016 | |
| Reagente a base di sequenza | shR6 affiancare il primer di Fr. | IDT | IDT: DNA oligo personalizzato | Fr primer che affianca il sito shR6; utilizzato per rilevare la cancellazione di 227 nt shR6; 5′-GGTGTCATGATGCTGTGACTGGTTG-3′. |
| Reagente a base di sequenza | shR6 primer per il montaggio di Rev Primer | IDT | IDT: DNA oligo personalizzato | Rev primer che affianca il sito shR6; utilizzato per rilevare la cancellazione di 227 nt shR6; 5′-TTTAGCTTAAGTGGGCCAGCAA-3′. |
| Reagente a base di sequenza | shR6 primer interno Rev primer | IDT | IDT: DNA oligo personalizzato | Rev primer che si sovrappone al sito shR6; utilizzato per rilevare la cancellazione di 227 nt shR6; 5′-AAGTTGGGTTTACATCTGCAC-3′. |
| Reagente a base di sequenza | CD95 che affianca il primer di Fr. | IDT | IDT: DNA oligo personalizzato | Fr primer che affianca il gene CD95; utilizzato per rilevare la delezione del gene CD95; 5′-TGTTTAATATAGCTGGGGGGGCTATGC-3′. |
| Reagente a base di sequenza | CD95 primer per il Rev Primer di fiancata | IDT | IDT: DNA oligo personalizzato | Rev primer che affianca il gene CD95; utilizzato per rilevare la delezione del gene CD95; 5′-TGGGGGACTCATGGGTTAAATAGAAT-3′. |
| Reagente a base di sequenza | CD95 primer interno Rev primer | IDT | IDT: DNA oligo personalizzato | Rev primer interno che mira all’interno del gene CD95; utilizzato per rilevare la delezione del gene CD95; 5′-GACCAGTCTTCTCATTTCATTTCAGAGGT-3′. |
| Reagente basato sulla sequenza | siScr/siNT1 | IDT; | IDT: DNA oligo personalizzato | controllare il siRNA non bersaglio; senso: UGGUUUACAUGUCGACUAA-3′. |
| Reagente a base di sequenza | c7/1 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 7 CD95L | antisenso: 5′-AUUGGGGGCCUG GGGAUGUUUU-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c7/2 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 7 CD95L | antisenso: 5′-CCUGGGGGAU GUUUCAGCUC-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c11 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 11 CD95L | antisenso: 5′-CCAACUCAAGG UCCAUGCC-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c15/1 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 15 CD95L | antisenso: 5′-AAAACUGGGGCUGU ACUUUUGU-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c15/2 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 15 CD95L | antisenso: 5′- AACUGGGGCUGUGU ACUUUUGUA-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c16/1 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 16 CD95L | antisenso: 5′- CAACAACCUGCC CCUGAGC-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c16/2 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 16 CD95L | antisenso: 5′- AACUCUAAGCG UCCCCCCAGG-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c21 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 21 CD95L | antisenso: 5′- UCAACGUAUC UGAGCUCUC-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | c22 | IDT | siRNA personalizzato; il filo antisenso corrisponde alla sequenza del cluster 22 CD95L | antisenso: 5′- AAUCUCAGACG UUUUUUCGG-3′; filo antisenso progettato con 3′ deoxy AA; il filo di senso complementare ha 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni |
| Reagente a base di sequenza | siCtr piscina | Dharmacon | D-001810–10 | controllo del pool di siRNA non bersaglio |
| Reagente a base di sequenza | SMARTpool siRNA punta su AGO2 | Dharmacon | L-004639-00-0005 | pool di siRNA progettato per colpire l’AGO2 |
| Reagente a base di sequenza (umano) | Primer GAPDH | Termofisher Scientific | Hs00266705_g1 | RT-qPCR; sonda di controllo |
| Reagente a base di sequenza (umano) | Primer CD95L | Termofisher Scientific | Hs00181226_g1; Hs00181225_m1 | RT-qPCR |
| Reagente a base di sequenza (umano) | Primer CD95 | Termofisher Scientific | Hs00531110_m1; Hs00236330_m1 | RT-qPCR |
| Reagente a base di sequenza (umano) | CD95LSIL primer | Termofisher Scientific | saggio d’identificazione: APNKTUD | Primer RT-qPCR personalizzato progettato utilizzando lo strumento di progettazione Thermofisher Scientific per il rilevamento di mRNA CD95LSIL |
| Reagente a base di sequenza (umano) | Cluster 8 CD95L piccolo primer RNA | Termofisher Scientific | Sonda personalizzata Saggio ID: CT7DPEM | Primer RT-qPCR personalizzato progettato utilizzando lo strumento di progettazione Thermofisher Scientific all’indirizzo https://www.thermofisher.com/order/custom-genomic-products/tools/small-rnaper rilevare specificamente piccoli RNA dal cluster 8 del CD95L (5′- AAGGAGCTGGGCAGAGAACTCCGAGA-3′) |
| Reagente a base di sequenza (umano) | Cluster 21 CD95L piccolo primer RNA | Termofisher Scientific | Sonda personalizzata Saggio ID: CTAAADA | Primer RT-qPCR personalizzato progettato utilizzando lo strumento di progettazione Thermofisher Scientific all’indirizzo https://www.thermofisher.com/order/custom-genomic-products/tools/small-rnaper rilevare specificamente piccoli RNA dal cluster 21 del CD95L (5′- TCAACGTATCTGAGCTCTCTCTC-3′) |
| Reagente a base di sequenza (umano) | z30 primer | Termofisher Scientific | ThermoFisher Scientific #4427975 | RT-qPCR per piccoli RNA; sonda di controllo |
| Peptide, proteina ricombinante | Peptide Flag-GST-T6B | PMID: 26351695 | NA | Peptide derivato da GW182/TNRC6B utilizzato per abbattere AGO1 a 4 |
| Saggio commerciale o kit | perline magnetiche anti-bandiera M2 | Sigma-Aldrich | Sigma-Aldrich #M8823 |
Reagenti e anticorpi
Tutti i reagenti e gli anticorpi sono stati descritti in precedenza(Putzbach et al., 2017) ad eccezione di quelli citati nei paragrafi seguenti.
Linee cellulari
HeyA8 (RRID:CVCL_8878) e HeyA8 CD95 celle knock-out, HCT116 (ATCC #CCL-247; RRID:CVCL_0291) e HCT116 celle knock-out Drosha e Dicer knock-out, celle MCF-7 (ATCC #HTB-22; RRID:CVCL_0031), e 293T (ATCC #CRL-3216; RRID:CVCL_0063) le celle sono state coltivate come descritto in precedenza(Putzbach et al., 2017). Le cellule MCF-7 CD95 knock-out e di eliminazione sono state coltivate in RPMI 1640 medium (Cellgro #10-040 CM), 10% FBS inattivati dal calore (Sigma-Aldrich), 1% L-glutammina (Mediatech Inc), e 1% penicillina /streptomicina (Mediatech Inc). Le cellule H460 (ATCC #HTB-177; RRID:CVCL_0459) sono state coltivate in RPMI1640 medium (Cellgro Cat#10-040) integrato con 10% FBS (Sigma Cat#14009C) e 1% L-glutammina (Corning Cat#25-005). Le cellule 3LL (ATCC #CRL-1642; RRID:CVCL_4358) sono state coltivate in medium DMEM (Gibco Cat#12430054) integrato con 10% FBS e 1% L-Glutammina. Le cellule del carcinoma epatocellulare del topo M565 sono state descritte in precedenza(Ceppi et al., 2014) e sono state coltivate in DMEM/F12 (Gibco Cat#11330) integrate con 10% FBS, 1% L-Glutammina e ITS (Corning #25-800-CR). Tutte le linee cellulari sono state autenticate usando il profilo STR e testate mensilmente per il micoplasma usando il PlasmoTest (Invitrogen).
Plasmidi e costrutti
Il pLenti-CD95L è stato sintetizzato subclonando un inserto contenente il CD95L ORF (NM_000639.2; sintetizzato dall’IDT come minigene con il sito 5′ NheI RE affiancato da 5′ NheI RE e 3′ XhoI RE in pIDTblue vector) nel vettore pLenti-GIII-CMV-RFP-2A-Puro (ABM Inc). L’inserto e la spina dorsale sono stati digeriti con gli enzimi di restrizione NheI (NEB #R0131) e XhoI (NEB #R0146). Legatura successiva con T4 DNA ligasi T4 ha creato il vettore pLenti-CD95L. Il vettore pLenti-CD95LMUT è stato creato subclonando un inserto cDNA CD95L con due sostituzioni nucleotidiche nel codone 218(TAT – > CGT) con conseguente sostituzione della tirosina per l’arginina, che è stato descritto per inibire il legame al CD95 (Schneider etal., 1997) nel vettorepLenti-GIII-CMV-RFP-2A-Puro. Il vettore pLenti-CD95LMUTNP è stato creato inserendo nel vettore pLenti-GIII-CMV-RFP-2A-Puro una sequenza di cDNA ORF CD95L contenente sia la mutazione Y218R che una singola sostituzione nucleotidica al secondo codone(CAG – > TAG), con conseguente arresto prematuro del codone subito dopo il codone di partenza. Il pLenti-CD95LSIL è stato creato subclonando una sequenza di cDNA CD95L ORF mutante con codoni mutati sinonimamente(Figura 1-figure supplement 2A) al codone successivo più altamente utilizzato nelle cellule umane (eccezioni sono state fatte all’interno del dominio ricco di prolina (Novoradovskyet al., 2005) per soddisfare i criteri di progettazione di sintesi genica) nel pLenti-GIII-CMV-RFP-2A-Puro vettore. Il GenScript Codon Usage Frequency Table Tool for Expression Host Organism: Human(https://www.genscript.com/tools/codon-frequency-table) è stato utilizzato per determinare le frequenze di utilizzo del codone.
Il mutante pLenti-CD95LMUTNP (G258A) è stato generato dalla mutagenesi diretta dal sito. L’identificazione dell’ATG che ha dato origine al peptide 24 kDa CD95LMUTNP è stata prevista utilizzando la Sequence Manipulation Suite(Stothard, 2000). La metionina successiva a valle del codone di arresto precoce è stato previsto per produrre una proteina 22,27 kDa, ed è stato quindi mutato in isoleucina (ATG >ATA). Lo strumento Agilent QuikChange Primer Design è stato utilizzato per progettare i primer della mutagenesi(Novoradovsky et al., 2005). I seguenti primer sono stati sintetizzati da IDT con fosforilazione 5′ e sono stati purificati in PAGE 5′- GCCTGTGTGTCTCTCTCTGTGATATATGTTTCATGGGTTTTTTGGGTTGT-3′ e 5′- CAACCAGAACCATGAAAAAAAAAAAAAAAGGAGAGACACACACGGG-3′. La reazione PCR è stata eseguita utilizzando NEB Phusion High-Fidelity DNA Polymerase (#M0530) con HF Buffer (#B0518) con il 3% DMSO secondo il protocollo del produttore. Il termociclatore BioRad T100 è stato utilizzato per la reazione di mutagenesi utilizzando le seguenti condizioni di ciclo: (fase 1) 98°C, 30 s; (fase 2) 25 cicli: 98°C, 10 s; 68°C, 30 s; 72°C, 6 min; (fase tre) 72°C, 10 min. Il DNA modello è stato digerito con DpnI (Thermo Scientific F541), e il plasmide mutagenizzato è stato legato con T4 ligasi (Thermo Scientific F541) e trasformato in Stbl3 (Invitrogen C737303). I mutanti di successo sono stati sottoposti a screening di crescita su piastre di Kanamicina, e confermato dal sequenziamento Sanger (ACGT, Inc).
Il vettore del frammento 5′ pLenti-CD95L 5′ è stato creato subclonando il frammento 5′ della sequenza CD95LMUTNP ORFcDNA(5′-GCTAGCCTGACTCACCCCAGCTGGATGATGATTTCACCCCCCATTTCAATTACCCATCATATCCATCCATCCCCATATCCCCATTTTA 3’) nel vettore pLenti-GIII-CMV-RFP-2A-Puro. Il vettore del frammento pLenti-CD95L 3′ è stato creato subclonando la metà di 3′ della sequenza CD95LMUTNP ORFcDNA(5′-3’) nel vettore pLenti-GIII-CMV-RFP-2A-Puro.
Sovraespressione di cDNA CD95L
Tutti i costrutti lentivirali sono stati generati in cellule 293T come descritto in precedenza(Putzbach et al., 2017). HeyA8 e MCF-7 (e tutte le linee cellulari derivate) cellule di tipo selvaggio sovraespressivo CD95L e cDNA CD95L mutanti sono stati generati da cellule di semina a 100.000 cellule per pozzo in una piastra a 6 pozzetti e cellule infettanti con lentivirus generato in cellule 293T (500 microlitri di surnatante virale per pozzo) con 8 microgrammi / ml di polibrene e successiva centrifugazione a 2700 rpm per 0,5-1 ore. Media è stato cambiato il giorno successivo, e pozzi confluenti sono stati ampliati a piatti più grandi. La selezione è stata avviata il giorno successivo con 3 μg/ml puromicina. HCT116, HCT116 Drosha knockout, e HCT116 cellule knockout Dicer(Kim et al., 2016) overexpressing CD95L cDNA sono stati generati da cellule di semina a 100.000 cellule per pozzo in una piastra a 24 pozzetti o 500.000 cellule per pozzo in una piastra a 6 pozzetti e cellule infettanti con lentivirus generato in cellule 293T (100 microlitri di virus per 24 pozzetti o 500 microlitri per 6 pozzetti) in presenza di 8 microgrammi / ml di polibrene. Media è stato cambiato il giorno successivo, e le cellule sono state selezionate con 3 μg/ml puromicina il giorno successivo. L’infezione con pLenti vuote è stata sempre inclusa come controllo.
Per valutare la tossicità dei cDNA CD95L sovraespressivi, le cellule infettate con questi costrutti sono state placcate in una piastra a 96 pozzetti 1 giorno dopo la selezione in presenza di puromicina (le cellule non infette erano tutte morte dopo 1 giorno in presenza di puromicina); La confluenza cellulare è stata valutata nel tempo utilizzando l’IncuCyte come descritto in precedenza(Putzbach et al., 2017).
Per valutare la sovraespressione di CD95L cDNA in cellule HeyA8 sensibili all’apoptosi in Figura 1A, le infezioni da lentivirus CD95L sono state fatte in 96 pozzetti con 50 μl di virus in presenza di 20 μM zVAD-fmk (Sigma-Aldrich # V116) e 8 μg/ml Polibrene; media è stato cambiato il giorno successivo in presenza di 20 μM zVAD-fmk; 3 μg/ml puromicina è stato aggiunto il giorno successivo. L’infezione con i costrutti CD95L per la RT-qPCR e Western blot in Figura 1B sono stati fatti in una piastra a 6 pozzetti in presenza di 20 μM zVAD-fmk.
Per l’esperimento in Figura 3B, Figura 3-figure supplemento 1, e Figura 5G, le cellule sono stati invertiti trasfettati in una piastra a 6 pozzetti; 100.000 cellule sono state placcate in pozzi con il On-TargetPlus non-targeting siRNA (Dharmacon # D-001810-10) o siAGO2 piscina (Dharmacon # # # L-004639-00-00-00-0005) a 25 nM complessato con 1 microlitri RNAiMax. Dopo ~ 24 ore, le cellule sono state infettate con pLenti o diversi pLenti-CD95L WT o costrutti mutanti (500 μl di surnatante virale [25% del volume totale]) come descritto sopra. Il giorno dopo, i media è stato sostituito, e le cellule sono state ampliate a 10 cm piastre. Il giorno seguente, 3 μg/ml di puromicina è stato aggiunto. Quando la selezione puromicina era completa (un giorno dopo), 750-3000 cellule sono state placcate per pozzo in una piastra a 96 pozzetti e messo nella macchina IncuCyte per valutare la confluenza delle cellule nel tempo. Quando le cellule HeyA8 erano cellule infette sono state placcate un’ora dopo l’infezione e la centrifugazione. Lauromicina è stata aggiunta ai pozzi 50 ore dopo l’infezione.
Cancellazioni CRISPR
Abbiamo co-trasformato un plasmide Cas9-espressivo(Jinek et al., 2013) e due gRNA che mirano a monte e a valle per eliminare un’intera sezione di DNA come descritto in precedenza(Putzbach et al., 2017). L’impalcatura di gRNA è stata utilizzata come descritto(Mali et al., 2013). I gRNA sono stati progettati utilizzando l’algoritmo trovato su http://crispr.mit.edu; solo i gRNA con un punteggio superiore a 50 sono stati considerati.
Una delezione di 227 nucleotidi nell’esone 4 del CD95 nelle celle MCF-7 (ΔshR6, clone #21) è stata generata utilizzando gRNA descritti in precedenza (Putzbachet al., 2017). La cancellazione di questo sito provoca una mutazione frame-shift che causa un knock-out a livello proteico(Putzbach et al., 2017). PCR con primer esterni affiancati (Fr: 5′-GGTGTGTCATGCTGTGTGACTGGTTTTG-3‘ e Rev: 5′-TTTAGCTTAAGTGGGCCAGCAA-3‘) e primer interni (Fr primer Fr e il primer interno Rev primer 5′-AAGTTGGTTTTTACATCTGCAC-3‘) è stato utilizzato per lo screening di cloni a cellula singola che ospitano una delezione omozigote.
Le due sequenze mirate dai gRNA di fianco per la cancellazione dell’intero gene CD95 erano 5′-GTCAGGGGTTCGTTCGGTTGCACAAA-3′ e 5′-TGCTTCTTCTTGGGATCCCTTAGA-3′. Per il rilevamento della delezione del gene CD95, i primer esterni affiancati erano 5′-TGTTTAATATAGCTGGGGGGGGCTATGC-3‘(primer di Fr) e 5′-TGGGGACTCATGGGGTTAAATAGAAT-3‘ (primer di Rev), e il primer interno inverso era 5′-GACCAGTCTTCTCATTTCAGGAGGT-3‘. Dopo lo screening dei cloni, è stato eseguito il sequenziamento Sanger per confermare la corretta cancellazione si era verificato.
PCR quantitativa in tempo reale
L’espressione relativa degli mRNA specifici è stata quantificata come descritto in precedenza(Gao et al., 2018). I primer/sonde acquistati da ThermoFisher Scientific sono stati GAPDH (Hs00266705_g1), CD95L umano (Hs00181226_g1 e Hs00181225_m1), CD95 umano (Hs00531110_m1 e Hs00236330_m1) e un primer/sonda personalizzato per rilevare l’mRNA CD95LSIL (progettato utilizzando lo strumento di progettazione personalizzato di Thermofisher Scientific; ID del test: APNKTUD) e CD95L 5′ mRNA a frammento (progettato utilizzando lo strumento di progettazione personalizzata Thermofisher Scientific; test ID: APEPTMU).
Le sonde RT-qPCR personalizzate progettate per rilevare specificamente le specie di sRNA sono state utilizzate per rilevare i frammenti di CD95L nella Figura 4G. Abbondanti sequenze di sRNA CD95L legate a Ago sono state identificate dai dati di sequenziamento di Drosha k.o. Ago pull-down. Le sequenze di sRNA mappate a FASLG sono state ordinate in base alla loro posizione finale di 3′. Il numero di letture di sRNA che terminano nella stessa posizione nucleotidica sono stati contati, e le sequenze più abbondanti sono state selezionate per la progettazione della sonda qPCR dai cluster 8 e 21. Queste sonde sono state progettate utilizzando il Custom TaqMan TaqMan Small RNA Assay Design Tool(https://www.thermofisher.com/order/custom-genomic-products/tools/small-rna/) di ThermoFisherper mirare alla sequenza del cluster 8 (5′- AAGGAGCTGGCAGAGAACTCCGAGA-3′) e alla sequenza del cluster 21 (5′- TCAACGTATCTGAGCTCTCTCTC-3′). Il rilevamento di questi frammenti comporta un protocollo di amplificazione in due fasi utilizzato per rilevare i miRNA. Nella prima fase, il kit di trascrizione inversa di cDNA ad alta capacità viene utilizzato per trascrivere selettivamente in modo inverso i due cluster da quantificare utilizzando primer specifici e 100 ng di RNA in ingresso. Il cDNA è stato diluito 1:5. La miscela di reazione qPCR è composta dal cDNA diluito, dalle sonde personalizzate e dalla Taqman Universal PCR Master Mix (Applied Biosystems #43240018). Le reazioni sono state eseguite in triplice copia. I valori Ct sono stati determinati utilizzando il sistema di PCR in tempo reale Applied Biosystems 7500 con un profilo di termociclo di 50°C per due minuti (fase uno), 95°C per 10 minuti (fase due), e poi 40 cicli di 95°C per 15 s (fase tre) e 60°C per 1 minuto (fase quattro). I valori ΔΔCt tra l’sRNA di interesse e il controllo sono stati calcolati per determinare l’abbondanza relativa dell’sRNA. I campioni sono stati normalizzati a Z30 (ThermoFisher Scientific #4427975).
Analisi Western blot
Il rilevamento di CD95, CD95L, AGO1 e AGO2 umani è stato effettuato tramite Western blot come descritto in precedenza(Putzbach et al., 2017).
Colorazione della superficie del CD95
La citometria a flusso è stata utilizzata per quantificare il livello di CD95 localizzato a membrana come descritto in precedenza(Putzbach et al., 2017).
Quantificazione della morte cellulare (frammentazione del DNA) e produzione di ROS
La percentuale di nuclei subG1 (DNA frammentato) è stata determinata con la citometria PI colorante / di flusso come descritto in precedenza(Putzbach et al., 2017). La produzione di ROS è stata quantificata utilizzando l’indicatore cellulare-permeabile 2′,7′-diclorodiidrofluoresceina diacetato (ThermoFisher Scientific #D399) come descritto in precedenza(Hadji et al., 2014).
Valutazione della crescita cellulare e della fluorescenza nel tempo
Dopo il trattamento / infezione, 750-3000 cellule sono state seminate in una piastra a 96 pozzetti almeno in triplice copia. Le immagini sono state catturate in punti di tempo indicati utilizzando un sistema di imaging a cellule vive IncuCyte ZOOM (Essen BioScience) con un obiettivo 10x lente. La percentuale di confluenza e l’intensità totale di fluorescenza integrata è stata calcolata utilizzando il software IncuCyte ZOOM (versione 2015A).
Infezione delle cellule per Ago-pull down e piccole analisi RNA-Seq
HeyA8 ΔshR6 clone # 11 cellule sono stati seminati a 75.000 cellule per pozzo su piastre a 6 pozzetti, e le cellule HCT116 e HCT116 Drosha knock-out sono stati entrambi seminati a 500.000 per pozzo su piastre a 6 pozzetti. Il HeyA8 ΔshR6 ΔshR6 clone # 11 cellule sono state infettate con 0,5 mL di pLenti vuoto o pLenti-CD95L-WT surnatante virale per pozzo. Le cellule HCT116 e HCT116 Drosha knockout sono state infettate con 0,5 mL di pLenti vuoto o pLenti-CD95LMUTNP supernatante virale per pozzetto. I media sono stati cambiati il giorno dopo e le cellule sono state messe in comune ed espanse a più piatti da 15 cm. La selezione con 3 μg/mL di puromicina è iniziata il giorno successivo. Il giorno dopo, il HeyA8 ΔshR6 ΔshR6 clone # 11 cellule infette sono state seminate a 600.000 cellule per piatto in più piatti da 15 cm, il HCT116 e HCT116 Drosha knock-out cellule sono state seminate a 5 milioni di cellule per piatto in più piatti da 15 cm. Due giorni dopo, ognuno dei campioni è stato pellettato e diviso in due: un pellet è stato lisato ed elaborato per il sequenziamento del piccolo RNA, e l’altro pellet è stato congelato in flash in azoto liquido. I pellet sono stati conservati a -80°C fino a quando non hanno potuto essere utilizzati per l’esperimento di Ago pull-down. Lo scopo di dividere il campione era quello di poter confrontare il pool cellulare totale di sRNA con la frazione che era legata al RISC. In questo modo, è stato possibile mappare i frammenti di elaborazione derivati dal CD95L dal mRNA a tutta lunghezza nel citosol fino alla forma finale matura legata al RISC. Tutto questo è stato fatto in duplicato.
Analisi dell’RNA-Seq
L’RNA totale è stato isolato utilizzando il miRNeasy Mini Kit (Qiagen, #74004) seguendo le istruzioni del produttore. È stata inclusa una fase di digestione su colonna utilizzando il set di DNasi senza RNase-free (Qiagen #79254). Sono state generate e sequenziate sia le librerie mRNA piccole che quelle grandi come descritto in precedenza(Putzbach et al., 2017). Le letture sono state tagliate con TrimGalore e poi allineate all’assemblaggio hg38 del genoma umano con Tophat. I conteggi delle letture grezze sono stati assegnati ai geni utilizzando HTSeq e l’espressione genica differenziale è stata analizzata con il pacchetto R Bioconductor EdgeR(Robinson et al., 2010).
Ago pull down e RNA-Seq analisi di sRNA legati
I pellet cellulari sono stati raccolti a 50 ore dopo la placcatura (122 ore dopo l’infezione) e sono stati congelati in azoto liquido. I pellet sono stati conservati a -80°C fino a quando non sono stati pronti per un’ulteriore lavorazione. Tra 10 e 25 × 106 cellule sono state lisate in tampone di lisi NP40 (20 mM Tris, pH 7,5, 150 mM NaCl, 2 mM EDTA, 1% (v/v) NP40, integrato con inibitori della fosfatasi) su ghiaccio per 15 min. Il lisato è stato sonicato 3 volte per 30 s al 60% di ampiezza (Sonics, VCX130) e purificato per centrifugazione a 12.000 g per 20 min. AGO1-4 sono stati abbattuti utilizzando 500 μg di peptide Flag-GST-T6B (Hauptmannet al., 2015) e con 60 μl di perle magnetiche anti-bandiera M2 (Sigma-Aldrich) per 2 ore a 4°C. Il pull-down è stato lavato tre volte in buffer di lisi NP40. Durante l’ultimo lavaggio, il 10% delle perle sono stati rimossi e incubati a 95 ° C a 95 ° C per 5 minuti in 2x SDS-PAGE tampone campione. I campioni sono stati eseguiti su un 4-12% SDS-PAGE e trasferiti su membrana di nitrocellulosa. L’efficienza pull-down è stata determinata da immunoblotting contro AGO1 (Cell Signaling # 5053; RRID: AB_10695871 e Abcam #98056; RRID: AB_10680548) e AGO2 (Abcam # 32381; RRID: AB_867543). Alle restanti perle sono stati aggiunti 500 µl di reagente TRIzol e l’RNA estratto secondo le istruzioni del produttore. Il pellet RNA è stato diluito in 20 µl di acqua. Il campione è stato diviso, e la metà del campione è stato dephosphorylated con 0,5 U / ml di fosfatasi alcalina CIP a 37 ° C per 15 minuti e successivamente radiomarcato con 0,5 μCi γ-32P-ATP e1U / ml di T4 PNK chinasi PNK per 20 minuti a 37 ° C. Gli RNA interagenti AGO1-4 sono stati visualizzati su un 15% di urea-PAGE. L’RNA rimanente è stato preso attraverso una piccola preparazione biblioteca RNA come descritto in precedenza(Hafner et al., 2012). In breve, l’RNA è stato legato con 3 ‘adattatori adenilati e separati su un 15% di denaturazione urea-PAGE. L’RNA corrispondente alla dimensione dell’inserto di 19-35 nt è stato eluito dal gel, etanolo precipitato seguito da 5 ‘legatura adattatore. I campioni sono stati separati su un 12% di Urea-PAGE ed estratti dal gel. Trascrizione inversa è stata eseguita utilizzando trascrizione inversa Superscript III trascriptasi e il cDNA amplificato da PCR. Il cDNA è stato sequenziato su Illumina HiSeq 3000. Sequenze dell’adattatore: Adattatore 1 – NNTGACTGTGGAATTGGAATTCTCGGGGGTGCCAAGG; Adattatore 2 – NNACACTCTGGAATTCTCGGGGGTGCCAAGG, Adattatore 3 – NNACAGAGTGGAATTGGAATTCTCGGGGGTGCCAAGG, Adattatore 4 – NNGCGATATGGAATTGGAATTCGGGGTGCCAAGG, Adattatore 47 – NNTCTGTGTGGGAATTGGAATTCTCGGGGGGTGCCAAGG, adattatore 48 – NNCAGCATTGGAATTGGAATTCTCGGGGGGTGCCAAGG, adattatore 49 – NNATAGTATGGAATTGGAATTCTCGGGGGTGCCAAGG, adattatore 50 – NNTCATAGTGGAATTGGAATTCGGGGTGCCAAGG. Sequenza di primer RT: GCCTTGGGCACCCGGAGAATTCCAATTCCA; sequenze di primer PCR: CAAGCAGAAGACACACACCATACGATACGAGATTTGGATGTGATGTGATGTGACTGGGGGGGGGGCACCCCCGAGAATTCCA. Per identificare gli sRNA derivati da CD95L tra le letture sequenziate, è stato generato un database BLAST da ogni set di letture, e blastn è stato usato per interrogare l’ORF CD95L (derivato da NM_000639.2) contro le letture da cellule infettate con pLenti-CD95L e per interrogare la sequenza ORF CD95LMUTNP contro le letture da cellule infettate con CD95LMUTNP. Le uniche letture prese in considerazione sono state quelle corrispondenti a una sequenza CD95L con un valore di e-valore inferiore allo 0,05% e un’identità del 100% per tutta la lunghezza della lettura. Ciò ha comportato la perdita di alcune letture di lunghezza inferiore a 19/20 nt. I risultati del BLAST filtrato sono stati convertiti in un file letto formattato, descrivendo le posizioni delle letture relative alla relativa sequenza CD95L, e il pacchetto R Sushi è stato utilizzato per tracciare i file letto e generare la Figura 4B-E.
Valutazione della tossicità degli sRNA derivati da CD95L
Per determinare se gli RNA guida derivati dall’mRNA CD95L sovraespresso potrebbero evocare tossicità, il piccolo RNA derivato dal CD95L legge (corrispondente a diversi cluster mostrati in Figura 4C) legato ad AGO dalle cellule knock-out HCT116 Drosha sono stati convertiti in siRNA. In primo luogo, tutte le letture meno di 18 nucleotidi sono stati filtrati, in quanto questi non incorporano in modo efficiente nel RISC. gli siRNA sono stati progettati con fili antisenso completamente complementari alle sequenze derivate dal CD95L che mappano le aree della struttura secondaria mRNA CD95L(Figura 4-figure supplement 1A) che si prevede formino duplex. Queste sequenze sono state progettate come 19 oligo nucleotidici con un 3′ deoxy AA. Il filamento di senso complementare è stato progettato con un 3′ deoxy TT e 2′-O-metilazione nelle prime due posizioni per impedire la sua incorporazione nel RISC. Questi oligo sono stati ordinati all’IDT e ricotti per formare i siRNA finali. Le sequenze dei filamenti antisenso (corrispondenti ai frammenti di mRNA derivati da mRNA CD95L) erano le seguenti: 5′-AUUGGGGGCCUGGGGAUGGAUGUUUU-3′ (c7/1), 5′-CCUGGGGAUGUUUUCAGCUC-3′ (c7/2), 5′.-CCAACUCAAGGGUUCCAUGCCAUGCC-3″ (c11), 5′-AACUGGGGCUGUACUUUUUGU-3″ (c15/1), 5′-AACUGGGGUUACUUUUUUGUUUGU-3″ (c15/1), 5′-AACUGGGGGUUUUGUUUUGUUGUU-3″. (c15/2), 5′- CAACAACCUGCCCCCCUUGGAGC-3′ (c16/1), 5′- AACUCUAAGCGUCCUCCCCAGG-3′ (c16/2), 5′- UCAACGUAUUGGAGCUCUC-3′ (c21), e 5′- AAUCUCAGACGUUUUUUUUUCGG-3′ (c22).
Questi otto siRNA sono stati trasfettati al contrario in HeyA8, H460, M565, e 3LL cellule utilizzando il reagente di trasfezione RNAiMAX (ThermoFisher Scientific) a 10 nM in triplice copia come descritto in precedenza(Murmann et al., 2018a). Il non-targeting (NT) e siRNA siL3 siRNA, come descritto in precedenza(Putzbach et al., 2017), sono stati utilizzati come controllo negativo e positivo, rispettivamente. La morte cellulare è stata quantificata tramite il rilascio di ATP 96 ore dopo la trasfezione con CellTiter-Glo (Promega). La vitalità % è stata calcolata in relazione alla struttura di trattamento solo RNAiMAX(Figura 4-figure supplement 1B).
Per analizzare la distribuzione della tossicità dei semi di tutti gli sRNA legati al RISC nelle cellule k.o. wt e Drosha, sono stati utilizzati i dati di pull down Ago basati sulla lettura. Le sequenze di semi 6mer (posizione 2-7) sono state estratte da tutte le sequenze uniche presenti nelle cellule wt o Drosha k.o.. La corrispondente tossicità media dei semi 6mer (tra le cellule HeyA8 e H460) è stata aggiunta per ogni sequenza unica. Queste sequenze sono state aggregate in sei gruppi in base ai loro diversi livelli di tossicità del seme (i numeri indicati sono percentuali di vitalità cellulare): (1)<20%; (2) 20 ~ 40%; (3) 40 ~ 60%; (4) 60 ~ 80%; (5) 80 ~ 100%; (6)>100%. Le letture totali (RPM) in ogni gruppo di tossicità dei semi sono state sommate e tracciate(Figura 5F). Inoltre, la distribuzione della tossicità dei semi di tutti i miRNA legati al RISC in wt e cellule k.o. Drosha sono stati analizzati in modo simile, tranne per il fatto che sono state incluse solo le letture che potevano essere allineate ai miRNA.
Analisi della tossicità teorica del mutante CD95LSIL
Tutti i 6mers possibili che potevano essere generati attraverso l’elaborazione della sequenza 846 nt di CD95L WT o CD95LSIL sono stati estratti in R, per un totale di 841 6mers possibili. I corrispondenti dati di fattibilità HeyA8 della schermata dei 4096 6mers sono stati uniti ai corrispondenti 6mers teorici CD95L 6mers(Gao et al., 2018). Dai dati di vitalità è stato generato un diagramma di densità. R pacchetti utilizzati: dplyr, data.table, ggplot2. Un test a due campioni di Kolmogorov-Smirnov è stato eseguito in R per determinare la significatività statistica.
Analisi degli sRNA legati al RISC derivati da geni codificanti
Tutte le letture di Ago pull down dei campioni di Drosha k.o. con pLenti o CD95L sovraespressi sono state allineate al genoma hg38 come descritto in precedenza(Putzbach et al., 2017) (vedi Figura 5-figure supplement 2). Sono state estratte le letture allineate in modo univoco al genoma per ogni coppia di replicati, e raggruppate in un unico set per tipo di campione (CD95L o pLenti). Le letture identiche sono state collassate e contate e sono state eliminate le letture <15 bp o >50 bp di lunghezza, così come le letture contenenti N. Il numero di letture trovate almeno 10 volte per ogni coppia di campioni replicati è stato di 10.839 per le cellule che esprimono CD95L e di 10.617 per le cellule infette da pLenti. I risultati sono stati filtrati in modo da contare solo il 100% delle corrispondenze su tutta la lunghezza della lettura, in direzione della trascrizione. La posizione di partenza è stata riportata per ogni mappatura univoca, molto abbondante (>=10 copie) letta. Il numero di tali posizioni di partenza uniche è stato contato per ottenere un numero putativo di pile per ogni trascrizione. Qualsiasi gene con almeno una pila è stato contato, e i dieci geni con il più alto numero di pile sono tracciati nella Figura 5B (e Figura 5-figure supplemento 1B). Una volta identificati i dieci geni che contenevano la maggior parte delle cellule che esprimono il RISC, è stata estratta una singola trascrizione per ogni gene. In ogni caso è stata utilizzata la trascrizione con il più lungo 3’UTR: 1 – ZC3HAV1 (ENST00000242351), 2 – FAT1 (ENST00000441802), 3 – SRRM2 (ENST00000301740), 4 – TNFRSF10D (ENST00000312584), 5 – PABPC1 (ENST00000318607), 6 – CCND1 (ENST0000000227507), 7 – MYC (ENST00000621592), 8 – YWHAG (ENST00000307630), 9 – DKKK1 (ENST00000373970), 10 – AKAP12 (ENST00000402676). Queste letture sono state confrontate con tutti i dati RNA-seq grezzi. Sono state considerate solo le corrispondenze al 100% per l’intera lunghezza della lettura, nella direzione della trascrizione, ma tutte le letture che corrispondevano alla trascrizione a quella stringa sono state tracciate, indipendentemente dalla loro abbondanza o unicità individuale, con ogni singola linea blu che rappresentava una singola lettura, con la sua lunghezza nel grafico proporzionale alla lunghezza letta.
Determinare l’abbondanza dei geni codificanti che danno origine alle letture di Ago-bound
Per determinare l’abbondanza di geni codificanti con letture legate ad Ago(Figura 5C), abbiamo isolato tutti i geni che avevano un RPKM di almeno 10 usando letture mappate in modo univoco da un set di dati RNA-Seq lungo convenzionale. Questi hanno portato a 4262 geni nel CD95L che esprimono le cellule HCT116 Drosha k.o. e 4256 geni nelle cellule infette pLenti. Entrambi i set di dati erano molto simili con una sovrapposizione >90% dei due set.
Analisi statistiche
I dati continui sono stati riassunti come mezzi e deviazioni standard (ad eccezione di tutti gli esperimenti di IncuCyte dove vengono mostrati gli errori standard) e i dati dicotomici come proporzioni. I dati continui sono stati confrontati utilizzando test t per due gruppi indipendenti e ANOVA unidirezionale per tre o più gruppi. Per la valutazione dei risultati continui nel tempo, è stata utilizzata l’ANOVA bidirezionale con un fattore per le condizioni di trattamento di interesse primario e un secondo fattore per il tempo trattato come variabile categoriale per consentire la non linearità.
Gli effetti del trattamento sulle cellule wild-type rispetto alle cellule knock-out Drosha sono stati valutati statisticamente con modelli di regressione che includevano termini lineari e quadratici per il valore nel tempo, gli effetti principali per il trattamento e il tipo di cellula, e le interazioni a due e tre vie per il trattamento, il tipo di cellula e il tempo. L’interazione a tre vie sui termini polinomiali con il trattamento e il tipo di cellula è stata valutata per la significatività statistica in quanto rappresenta la differenza degli effetti del trattamento nel corso dell’esperimento per i diversi tipi di cellule.
GSEA utilizzato in Figura 2B è stato eseguito utilizzando il software GSEA v2.2.4 del Broad Institute(http://software.broadinstitute.org/gsea); sono state utilizzate 1000 permutazioni. Le liste di geni Sabatini sono stati impostati come set di geni personalizzati per determinare l’arricchimento dei geni di sopravvivenza rispetto ai geni di controllo non di sopravvivenza in geni non di controllo in geni downregulated dai dati RNA-Seq come fatto in precedenza(Putzbach et al., 2017); p-valori inferiori a 0,05 sono stati considerati significativamente arricchiti. I geni con un’espressione di lettura normalizzata media (su entrambe le coppie di duplicati) inferiore a tre sono stati esclusi in modo da includere solo i geni realmente espressi. L’analisi di arricchimento GO mostrata nella figura 2D è stata eseguita con tutti i geni che dopo l’allineamento e la normalizzazione sono risultati essere almeno 1,5 volte regolati con un valore p regolato di <0,05 utilizzando il software disponibile su www.Metascape.org e i parametri di funzionamento predefiniti. Gli altri set di dati utilizzati in questa analisi (cellule HeyA8 trasfettate con un siRNA tossico che colpisce CD95L siL3 e 293T infettate con shRNA tossici che colpiscono CD95L siL1 e shL3 e cellule HeyA8 infettate con shRNA tossici che colpiscono CD95L shL6) sono stati precedentemente descritti(Putzbach et al., 2017).
Tutte le analisi statistiche sono state condotte in Stata 14 o R 3.3.1.
Disponibilità dei dati
I dati di sequenziamento dell’RNA generati per questo studio sono disponibili nel repository GEO: GSE103631(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE103631) e GSE114425(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE114425).
References
- Algeciras-Schimnich A, Shen L, Barnhart BC, Murmann AE, Burkhardt JK, Peter ME. Molecular ordering of the initial signaling events of CD95. Molecular and Cellular Biology. 2002; 22:207-220. DOI | PubMed
- Bañez-Coronel M, Porta S, Kagerbauer B, Mateu-Huertas E, Pantano L, Ferrer I, Guzmán M, Estivill X, Martí E. A pathogenic mechanism in Huntington’s disease involves small CAG-repeated RNAs with neurotoxic activity. PLoS Genetics. 2012; 8DOI | PubMed
- Burroughs AM, Ando Y, de Hoon MJ, Tomaru Y, Suzuki H, Hayashizaki Y, Daub CO. Deep-sequencing of human Argonaute-associated small RNAs provides insight into miRNA sorting and reveals Argonaute association with RNA fragments of diverse origin. RNA Biology. 2011; 8:158-177. DOI | PubMed
- Ceppi P, Hadji A, Kohlhapp FJ, Pattanayak A, Hau A, Liu X, Liu H, Murmann AE, Peter ME. CD95 and CD95L promote and protect cancer stem cells. Nature Communications. 2014; 5DOI | PubMed
- Elkayam E, Faehnle CR, Morales M, Sun J, Li H, Joshua-Tor L. Multivalent Recruitment of Human Argonaute by GW182. Molecular Cell. 2017; 67:646-658. DOI | PubMed
- ElOjeimy S, McKillop JC, El-Zawahry AM, Holman DH, Liu X, Schwartz DA, Day TA, Dong JY, Norris JS. FasL gene therapy: a new therapeutic modality for head and neck cancer. Cancer Gene Therapy. 2006; 13:739-745. DOI | PubMed
- Friesen C, Fulda S, Debatin KM. Cytotoxic drugs and the CD95 pathway. Leukemia. 1999; 13:1854-1858. DOI | PubMed
- Fu Q, Fu TM, Cruz AC, Sengupta P, Thomas SK, Wang S, Siegel RM, Wu H, Chou JJ. Structural Basis and Functional Role of Intramembrane Trimerization of the Fas/CD95 Death Receptor. Molecular Cell. 2016; 61:602-613. DOI | PubMed
- Gao QQ, Putzbach W, Murmann AE, Chen S, Ambrosini G, Peter JM, Peter ME. 6mer seed toxicity in tumor suppressive miRNAs. Nature Communications. 2018. DOI
- Hadji A, Ceppi P, Murmann AE, Brockway S, Pattanayak A, Bhinder B, Hau A, De Chant S, Parimi V, Kolesza P, Richards J, Chandel N, Djaballah H, Peter ME. Death Induced by CD95 or CD95 Ligand Elimination. Cell Reports. 2014; 7:208-222. DOI | PubMed
- Hafner M, Renwick N, Farazi TA, Mihailović A, Pena JT, Tuschl T. Barcoded cDNA library preparation for small RNA profiling by next-generation sequencing. Methods. 2012; 58:164-170. DOI | PubMed
- Hamilton AJ, Baulcombe DC. A species of small antisense RNA in posttranscriptional gene silencing in plants. Science. 1999; 286:950-952. DOI | PubMed
- Hansen TB, Venø MT, Jensen TI, Schaefer A, Damgaard CK, Kjems J. Argonaute-associated short introns are a novel class of gene regulators. Nature Communications. 2016; 7DOI | PubMed
- Hauptmann J, Schraivogel D, Bruckmann A, Manickavel S, Jakob L, Eichner N, Pfaff J, Urban M, Sprunck S, Hafner M, Tuschl T, Deutzmann R, Meister G. Biochemical isolation of Argonaute protein complexes by Ago-APP. PNAS. 2015; 112:11841-11845. DOI | PubMed
- Hyer ML, Voelkel-Johnson C, Rubinchik S, Dong J, Norris JS. Intracellular Fas ligand expression causes Fas-mediated apoptosis in human prostate cancer cells resistant to monoclonal antibody-induced apoptosis. Molecular Therapy. 2000; 2:348-358. DOI | PubMed
- Jinek M, East A, Cheng A, Lin S, Ma E, Doudna J. RNA-programmed genome editing in human cells. eLife. 2013; 2DOI | PubMed
- Kim YK, Kim B, Kim VN. Re-evaluation of the roles of DROSHA, Export in 5, and DICER in microRNA biogenesis. PNAS. 2016; 113:E1881-E1889. DOI | PubMed
- Liu J, Carmell MA, Rivas FV, Marsden CG, Thomson JM, Song JJ, Hammond SM, Joshua-Tor L, Hannon GJ. Argonaute2 is the catalytic engine of mammalian RNAi. Science. 2004; 305:1437-1441. DOI | PubMed
- Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, Sweet-Cordero A, Ebert BL, Mak RH, Ferrando AA, Downing JR, Jacks T, Horvitz HR, Golub TR. MicroRNA expression profiles classify human cancers. Nature. 2005; 435:834-838. DOI | PubMed
- Mali P, Yang L, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9. Science. 2013; 339:823-826. DOI | PubMed
- Morin RD, O’Connor MD, Griffith M, Kuchenbauer F, Delaney A, Prabhu AL, Zhao Y, McDonald H, Zeng T, Hirst M, Eaves CJ, Marra MA. Application of massively parallel sequencing to microRNA profiling and discovery in human embryonic stem cells. Genome Research. 2008; 18:610-621. DOI | PubMed
- Murmann AE, Gao QQ, Putzbach WE, Patel M, Bartom ET, Law CY, Bridgeman B, Chen S, McMahon KM, Thaxton CS, Peter ME. Small interfering RNAs based on huntingtin trinucleotide repeats are highly toxic to cancer cells. EMBO reports. 2018a; 19DOI | PubMed
- Murmann AE, Yu J, Opal P, Peter ME. Trinucleotide repeat expansion diseases, RNAi, and Cancer. Trends in Cancer. 2018b; 4:684-700. DOI | PubMed
- Nisihara T, Ushio Y, Higuchi H, Kayagaki N, Yamaguchi N, Soejima K, Matsuo S, Maeda H, Eda Y, Okumura K, Yagita H. Humanization and epitope mapping of neutralizing anti-human Fas ligand monoclonal antibodies: structural insights into Fas/Fas ligand interaction. The Journal of Immunology. 2001; 167:3266-3275. DOI | PubMed
- Novoradovsky A, Zhang V, Ghosh M, Hogrefe H, Sorge JA, Gaasterland T. Computational principles of primer design for site directed mutagenesis. Chapter 9: Computational Biology. 2005;532-535.
- Patel M, Peter ME. Identification of DISE-inducing shRNAs by monitoring cellular responses. Cell Cycle. 2018; 17:506-514. DOI | PubMed
- Putzbach W, Gao QQ, Patel M, van Dongen S, Haluck-Kangas A, Sarshad AA, Bartom ET, Kim KA, Scholtens DM, Hafner M, Zhao JC, Murmann AE, Peter ME. Many si/shRNAs can kill cancer cells by targeting multiple survival genes through an off-target mechanism. eLife. 2017; 6DOI | PubMed
- Putzbach W, Gao QQ, Patel M, Haluck-Kangas A, Murmann AE, Peter ME. DISE: A Seed-Dependent RNAi Off-Target Effect That Kills Cancer Cells. Trends in Cancer. 2018; 4:10-19. DOI | PubMed
- Robinson MD, McCarthy DJ, Smyth GK. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 2010; 26:139-140. DOI | PubMed
- Schneider P, Bodmer JL, Holler N, Mattmann C, Scuderi P, Terskikh A, Peitsch MC, Tschopp J. Characterization of Fas (Apo-1, CD95)-Fas ligand interaction. Journal of Biological Chemistry. 1997; 272:18827-18833. DOI | PubMed
- Stothard P. The sequence manipulation suite: JavaScript programs for analyzing and formatting protein and DNA sequences. BioTechniques. 2000; 28:1102-1104. DOI | PubMed
- Suda T, Takahashi T, Golstein P, Nagata S. Molecular cloning and expression of the Fas ligand, a novel member of the tumor necrosis factor family. Cell. 1993; 75:1169-1178. DOI | PubMed
- Sudarshan S, Holman DH, Hyer ML, Voelkel-Johnson C, Dong JY, Norris JS. In vitro efficacy of Fas ligand gene therapy for the treatment of bladder cancer. Cancer Gene Therapy. 2005; 12:12-18. DOI | PubMed
- Sun H, Liu Y, Bu D, Liu X, Norris JS, Xiao S. Efficient growth suppression and apoptosis in human laryngeal carcinoma cell line HEP-2 induced by an adeno-associated virus expressing human FAS ligand. Head & Neck. 2012; 34:1628-1633. DOI | PubMed
- Wang T, Birsoy K, Hughes NW, Krupczak KM, Post Y, Wei JJ, Lander ES, Sabatini DM. Identification and characterization of essential genes in the human genome. Science. 2015; 350:1096-1101. DOI | PubMed
- Yamashita R, Suzuki Y, Takeuchi N, Wakaguri H, Ueda T, Sugano S, Nakai K. Comprehensive detection of human terminal oligo-pyrimidine (TOP) genes and analysis of their characteristics. Nucleic Acids Research. 2008; 36:3707-3715. DOI | PubMed
- Yoda M, Cifuentes D, Izumi N, Sakaguchi Y, Suzuki T, Giraldez AJ, Tomari Y. Poly(A)-specific ribonuclease mediates 3′-end trimming of Argonaute2-cleaved precursor microRNAs. Cell Reports. 2013; 5:715-726. DOI | PubMed
Fonte
Putzbach W, Haluck-Kangas A, Gao QQ, Sarshad AA, Bartom ET, et al. () CD95/Fas ligand mRNA is toxic to cells. eLife 7e38621. https://doi.org/10.7554/eLife.38621





