Abstract
Introduzione
I veleni e le tossine di cui sono composti sono usati soprattutto dagli animali per interazioni antagoniste, come la cattura delle prede e la difesa dai predatori(Fry et al., 2009; Casewell et al., 2013). La ricerca farmacologica si è concentrata quasi esclusivamente sui veleni degli stadi adulti, nonostante il fatto che molti animali mostrano notevoli trasformazioni nelle architetture del corpo e nell’ecologia durante il loro sviluppo(Ruppert et al., 2004). Tali grandi differenze possono dettare diverse interazioni interspecifiche per i diversi stadi della vita(Wilbur, 1980). Poiché si ipotizza che il veleno sia metabolicamente costoso e in molti casi altamente specifico(Nisani et al., 2012; Casewell et al., 2013), è plausibile che la sua composizione possa cambiare tra i diversi stadi di vita. In effetti, sono state segnalate alcune variazioni ontogenetiche nei veleni dei serpenti(Gibbs et al., 2011), dei ragni(Santana et al., 2017) e delle lumache a cono(Safavi-Hemami et al., 2011), ma finora questo fenomeno non è stato studiato a fondo in un animale con un ciclo vitale complesso durante tutto il suo sviluppo.
Il più antico gruppo di animali velenosi esistente è il phylum Cnidaria marino, che comprende anemoni di mare, coralli, meduse e idroidi. Gli Cnidari sono caratterizzati dalla loro cellula pungente, il nematocita, che ospita un organulo unico e altamente complesso, il nematocita(Kass-Simon e Scappaticci, 2002; David et al., 2008). Questo organulo proteico è utilizzato come sistema di diffusione del veleno in miniatura(Thomason, 1991; Lotan et al., 1995). La maggior parte degli cnidari ha un ciclo di vita complesso che comprende sia gli stadi di vita bentonica sessile che quelli pelagici mobili, di ampia distribuzione dimensionale e di interazioni ecologiche. Per esempio, il ciclo vitale canonico degli antozoi (coralli e anemoni di mare) comprende uno stadio larvale natatorio (planula) che si trasforma in uno stadio di polipo sessile che matura nell’adulto riproduttivo.
L’anemone di mare stellato, Nematostella vectensis, sta diventando un modello di laboratorio cnidario leader, poiché a differenza di molte altre specie cnidarie può essere coltivato in laboratorio durante tutto il suo ciclo di vita. Questo rende Nematostella un sistema unico per studiare il veleno di un animale con un ciclo vitale complesso. Un altro vantaggio è che l’elevata omogeneità genetica del comune ceppo di Nematostella lab minimizza la variazione genetica individuale, che è tutt’altro che banale nella maggior parte degli altri animali velenosi raccolti dall’ambiente naturale in numero limitato. Inoltre, Nematostella ha un genoma sequenziato e trascrizioni specifiche per stadio e vari strumenti molecolari sono disponibili per la sua manipolazione genetica(Wikramanayake et al., 2003; Putnam et al., 2007; Renfer et al., 2010; Helm et al., 2013; Layden et al., 2016). La cassetta degli attrezzi per la sperimentazione di Nematostella è unica, non solo per gli cnidari, ma anche per gli animali velenosi in generale.
Durante il ciclo di vita di Nematostella, le femmine rilasciano un pacchetto di uova gelatinose e i maschi rilasciano sperma nell’acqua(Hand and Uhlinger, 1992). Dopo la fecondazione si verifica, la scissione zigote inizia, formando una blastula e meno di 24 ore dopo la fecondazione (hpf) gastrulazione è completata. Una larva di planula emerge dal pacchetto di uova 48-72 hpf e inizia a nuotare in acqua. Da sei a sette giorni dopo la fecondazione, la planula si deposita in substrato morbido e inizia a metamorfosare in un polipo primario e la maturazione sessuale richiede circa 4 mesi in condizioni di laboratorio(Hand and Uhlinger, 1992)(Figura 1A). Mentre l’uovo e la planula sono approssimativamente sferici e misurano solo circa 250 µm, il polipo adulto adulto morfologicamente differenziato e allungato può raggiungere la lunghezza di 4 cm in natura e fino a 20 cm in laboratorio (Williams,1975; Hand e Uhlinger, 1992). Il veleno è prodotto in Nematostella dalle cellule della ghiandola ectodermica e dai nematociti(Moran et al., 2012b; Moran et al., 2013). Sebbene la cattura delle prede e la produzione di Nv1, una delle principali tossine modulatrici dei canali del sodio(Moran et al., 2008a; Moran et al., 2012b), inizia allo stadio di polipo primario sessile, le nematociti appaiono già a 48 hpf nella planula natatoria(Marlow et al., 2009). Queste sono forti indicazioni che il veleno è probabilmente presente già nelle fasi iniziali della vita e la sua composizione potrebbe cambiare drasticamente in tutto il ciclo di vita dei Nematostella. Un’ulteriore complessità nella regolazione della produzione di veleno può essere dovuta alla Nematostella che produce diversi tipi di cellule che sono distribuite in modo non uniforme tra i vari tessuti(Moran et al., 2013)(Figura 1B).

Figura 1.Espressione di tossine attraverso lo sviluppo di Nematostella e dei tessuti.(A) Il ciclo di vita di N. vectensis.(B) Dissezionato polipo femminile Nematostella. (C) Nematocisti planula scaricate trovato nel mezzo dopo un incontro con A. nauplii salina.(D) Nematocisti di Nematostella planula appuntato nella cuticola di un A. sal ina nauplii.(E-F) Mappe di calore dei livelli di espressione differenziale nCounter di geni che codificano le tossine e altre proteine nematocisti in vari stadi di sviluppo e tessuti femminili adulti.(G) Un grafico su scala logaritmica di nCounter ha normalizzato i livelli di espressione dei geni che codificano NEP3, NvePTx1 e Nv1 in ogni stadio dello sviluppo. Ogni punto è la media di tre repliche biologiche e le barre di errore rappresentano la deviazione standard.(H) Un grafico su scala logaritmica dei valori iBAQ (Intensity-Based Absolute Quantification) misurati in spettrometria di massa dei peptidi NEP3, NvePTx1 e Nv1 in cinque stadi di sviluppo. Ogni punto è la media di quattro repliche tecniche e le barre di errore rappresentano la deviazione standard. Chiave per i pannelli E e G: UE = Uovo non fecondato; B = Blastula; G = Gastrula; EP = Planula precoce; LP = Planula tardiva; M = metamorfosi; PP = Polipo primario; JP = Polipo giovanile; MA = Adulti misti; AM = Maschio adulto; AF = Femmina adulta. Tasto per il pannello F: M = Mesenterie; Px = Faringe; Pa = Physa; T = Tentacoli.
Utilizzando un approccio integrato e comparativo, quantifichiamo e caratterizziamo attentamente l’espressione spazio-temporale delle note e nuove tossine di Nematostella attraverso diversi stadi di sviluppo e utilizziamo la transgenesi per comprendere le dinamiche di produzione del veleno in una specie con un ciclo di vita complesso. Correliamo le dinamiche di espressione delle tossine negli stadi di vita con gli esperimenti di comportamento a livello organismico che evidenziano le risposte differenziali delle prede e dei predatori quando sono esposti a questi stadi.

Figura 1.Espressione delle tossine attraverso lo sviluppo di Nematostella e dei tessuti.(A) Il ciclo di vita di N. vectensis.(B) Dissezionato polipo femminile Nematostella. (C) Nematocisti planula scaricate trovato nel mezzo dopo un incontro con A. nauplii salina.(D) Nematocisti di Nematostella planula appuntato nella cuticola di un A. sal ina nauplii.(E-F) Mappe di calore dei livelli di espressione differenziale nCounter di geni che codificano le tossine e altre proteine nematocisti in vari stadi di sviluppo e tessuti femminili adulti.(G) Un grafico su scala logaritmica di nCounter ha normalizzato i livelli di espressione dei geni che codificano NEP3, NvePTx1 e Nv1 in ogni stadio dello sviluppo. Ogni punto è la media di tre repliche biologiche e le barre di errore rappresentano la deviazione standard.(H) Un grafico su scala logaritmica dei valori iBAQ (Intensity-Based Absolute Quantification) misurati in spettrometria di massa dei peptidi NEP3, NvePTx1 e Nv1 in cinque stadi di sviluppo. Ogni punto è la media di quattro repliche tecniche e le barre di errore rappresentano la deviazione standard. Chiave per i pannelli E e G: UE = Uovo non fecondato; B = Blastula; G = Gastrula; EP = Planula precoce; LP = Planula tardiva; M = metamorfosi; PP = Polipo primario; JP = Polipo giovanile; MA = Adulti misti; AM = Maschio adulto; AF = Femmina adulta. Tasto per il pannello F: M = Mesenterie; Px = Faringe; Pa = Physa; T = Tentacoli.
Risultati
Le larve di nematostella sono velenose
Per valutare il potenziale velenoso delle larve di Nematostella abbiamo incubato delle planule di nuoto vecchie di 4 giorni con i nauplii del gamberetto in salamoia Artemia salina. Sorprendentemente, entro 10 minuti dall’inizio dell’incubazione 3 su 8 Artemie erano paralizzate o morte, ed entro 90 minuti 7 su 8 erano morte(Video 1), mentre in un gruppo di controllo senza planule tutte le Artemie erano vive. Questo esperimento ha rivelato che le Nematostelle planule sono in grado di uccidere rapidamente un crostaceo più grande di loro. L’effetto relativamente rapido e la differenza di dimensioni suggeriscono che il veleno è coinvolto nel processo. Numerose nematocisti scaricate sono state trovate nell’acqua intorno ai nauplii morti così come nella loro cuticola(Figura 1C-D), suggerendo inoltre che le capsule pungenti sono coinvolti nel processo di avvelenamento.

Video 1.L’interazione tra Nematostellaplanulae e Artemia nauplii.Le panule stanno rapidamente paralizzando i nauplii dopo l’interazione fisica. Questo filmato è collegato alla Figura 1.

Video 1.L’interazione tra Nematostellaplanule e Artemia nauplii.Le panule stanno rapidamente paralizzando i nauplii dopo l’interazione fisica. Questo filmato è collegato alla Figura 1.
Modelli di espressione distinti e dinamici dei geni delle tossine in Nematostella
Per misurare con precisione i livelli di espressione di geni di tossine note, tossine putativi e geni che codificano le proteine strutturali delle nematocisti, abbiamo utilizzato la piattaforma nCounter a media produttività (vedi materiali e metodi), che in precedenza ha dimostrato di mostrare un’elevata sensibilità e precisione simile a quella della PCR quantitativa in tempo reale(Prokopec et al., 2013). Abbiamo analizzato i livelli di espressione dell’RNA dei geni che codificano la neurotossina modulatrice dei canali del sodio Nv1(Moran et al., 2008a), le tossine putativi NvePTx1, NEP3, NEP3-like, NEP4, NEP8 e NEP16(Moran et al., 2013; Orts et al., 2013), le metallopeptidasi putativi NEP6 e NEP14(Moran et al.,2013), NvLysin1b, una tossina citolitica che può servire anche per la digestione degli alimenti(Moran et al., 2012a), e i componenti strutturali della capsula nematocitaria Ncol1, Ncol3 e Ncol4(David et al., 2008; Zenkert et al., 2011), così come la presunta componente strutturale nematocitaria NR2 (Moran et al., 2014). Le misurazioni dell’RNA sono state eseguite su nove stadi di sviluppo(Figura 1E), adulti di ogni sesso e quattro tessuti sezionati di una femmina adulta(Figura 1F; File supplementare 1). L’analisi nCounter ha rivelato che molti dei geni formano dei cluster informativi(Figura 1E-F). E ‘evidente che i modelli di espressione di NEP3, NEP3-like e NEP4 fortemente raggruppati con quelli dei geni che codificano i componenti strutturali nematocistici componenti strutturali in entrambe le analisi dello sviluppo e dei tessuti(Figura 1E-F), che è coerente con la constatazione che queste tossine putativo sono prodotti in nematociti e vengono rilasciati dalla capsula al momento della scarica (Moranet al., 2013). Altre tossine come Nv1, che è stato dimostrato essere espresso in cellule della ghiandola polipocitaria ectodermica(Moran et al., 2012b), o Nvlysin1b, prodotto da cellule della ghiandola grande nella faringe e mesenterie a partire dalle prime fasi dello sviluppo(Moran et al., 2012a), ha avuto diversi modelli di espressione nell’analisi nCounter e non ha formato grandi cluster (Figura 1E-F). I livelli di espressione di NEP3, Nv1 e NvePTx1 erano sorprendentemente distinti nello sviluppo(Figura 1G). L’espressione di Nv1 è relativamente bassa nelle fasi iniziali dello sviluppo e poi picchi netti nei polipi giovani e adulti a livelli straordinari che sono più alti di quasi due ordini di grandezza rispetto alle altre tossine(Figura 1G). Questi livelli di espressione possono essere spiegati dal fatto che il genoma di Nematostella contiene più di una dozzina di copie del gene codificante Nv1(Moran et al., 2008b) e il picco di espressione trascrizionale in ritardo nello sviluppo è coerente con le osservazioni precedenti a livello proteico(Moran et al., 2012b). A differenza di Nv1, NEP3 è espresso a livelli elevati già a livello di gastrulazione, picchi nella planula iniziale e rimane più o meno stabile per tutto il resto dello sviluppo in età adulta. A differenza delle altre tossine, l’espressione dei picchi di NvePTx1 in corrispondenza dell’uovo non fecondato, scende bruscamente attraverso lo sviluppo e sale nuovamente nella femmina adulta(Figura 1G).
Come variazioni significative tra l’espressione delle tossine a livello trascrizionale e il livello proteico sono stati recentemente riportati(Madio et al., 2017), abbiamo completato l’espressione dei geni delle tossine in Nematostella utilizzando la proteomica shotgun (spettrometria di massa tandem MS/MS). Abbiamo eseguito queste quantificazioni in quattro repliche su lisati da quattro stadi di sviluppo distinti, così come i sessi separati dei polipi adulti(Figura 1H; File supplementare 2). I modelli di espressione dinamica che abbiamo osservato a livello proteico ben correlato con la dinamica che osserviamo a livello trascrittomico.
NvePTx1 è una tossina depositata maternamente
NvePTx1 è stato originariamente identificato come un omologo del bloccante dei canali di potassio di tipo cinque BCsTx3 dell’anemone di mare Bunodosoma caissarum(Orts et al., 2013). Abbiamo anche identificato bioinformamaticamente diverse sequenze omologhe negli anemoni di mare Anthopleura elegantissima e Metridium senile, e l’idrozoo Hydractinia symbiolongicarpus(Figura 2A). Ciò suggerisce che questa famiglia peptidica era già presente nell’ultimo antenato comune di tutti gli Cnidaria, ma si è persa più volte in vari lignaggi cnidari.
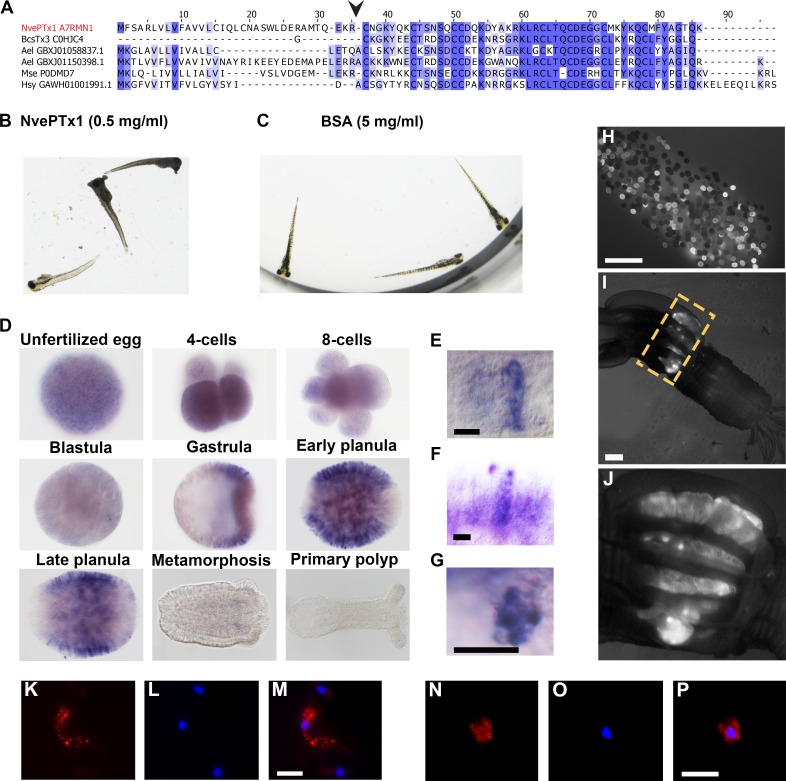
Figura 2.NvePTx1 è una tossina che si deposita maternamente nelle uova di Nematostella.(A) Allineamento di sequenza di NvePTx1 e dei suoi omologhi di altre specie cnidarie. La punta della freccia rappresenta il sito di proteolisi che separa la tossina matura dal peptide di segnale. I numeri di adesione appaiono vicino ad ogni sequenza. Nve = Nematostellavectensis; Bcs = Bunodosoma caissarum; Ael = Anthopleura elegantissima; Mse =Metridium senile; Hsy= Hydractinia symbiolongicurpus.(B-C) Larve dizebrafish 16 ore dopo l’incubazione in NvePTx1 e controllo BSA.(D) Rilevazione dell’espressione NvePTx1 da parte di ISH. Dalla fase di blastula in poi, il polo orale è a destra. Da vicino su planula grande allungata(E-F) e piccolo tondo (G) cellule ghiandolari tinte da ISH per NvePTx1 . Cellule della ghiandola, che sono riconoscibili dalle loro vescicole rotonde sono privi di macchia, barra di scala è di 10 µm.(H-I) Un adulto F0 polipo femmina F0 che come uno zigote è stato iniettato da un NvePTx1 ::mOrange2 costruire. mOrange2 espressione è evidente nelle gonadi. Il pannello G è un primo piano della regione indicata nel pannello H dal riquadro giallo punteggiato. La barra di scala è 1000 µm (J) Un pacchetto di uova deposte dalla femmina dal pannello H. Molte delle uova esprimono mOrange2. La scala è di 1000 µm.(K-M) Grande allungata e (N-P) piccole cellule ghiandolari rotonde colorate da anti mCherry. La scala è di 10 µm.
In primo luogo, per verificare se NvePTx1 è effettivamente una tossina abbiamo espresso in una forma ricombinante e incubato 20 zebrafish(Danio rerio) larve di pesce zebra(Danio rerio) con 0,5 mg / ml di peptide ricombinante altamente puro (saggiato mediante cromatografia a fase inversa) per 20 ore. All’aggiunta della tossina, il pesce ha reagito rapidamente, con un aumento della velocità di nuoto. Dopo 2 ore di incubazione 10 larve di pesce erano morte, con il resto dei pesci morti nel corso delle successive 18 ore (a quel punto l’esperimento era terminato) (Figura 2B). Il gruppo di controllo (incubato in 5 mg/ml di sieroalbumina bovina) si è comportato normalmente per tutta la durata dell’esperimento e tutte le larve erano vive dopo 20 ore di incubazione(Figura 2C). Per completare l’esperimento nCounter, abbiamo saggiato il modello di espressione spazio-temporale di NvePTx1 da ibridazione in situ (ISH). Abbiamo osservato che mentre NvePTx1 è espresso in modo uniforme in tutto l’uovo non fecondato e le prime fasi post-fertilizzazione, nel gastrula l’espressione diventa spazialmente localizzata e sembra essere assente dai poli orale e aborale(Figura 2D). Nel planula, l’espressione è chiaramente osservata nel ectoderma in cellule ghiandolari imballate assenti dai due poli del corpo, e su metamorfosi, l’espressione diminuisce(Figura 2D-E). L’ISH rivela due tipi di cellule ghiandolari, una grande e allungata(Figura 2E-F) e un’altra piccola e rotonda (Figura 2G). I risultati degli esperimenti ISH e nCounter hanno indicato che NvePTx1 è maternamente depositato a livello di RNA. Inoltre, abbiamo potuto rilevare i risultati del peptide NvePTx1 nei nostri dati di analisi MS/MS (Figura 1H) così come in set di dati supplementari disponibili da precedenti studi proteomici di uova di Nematostella(Lotan et al., 2014; Levitan et al., 2015), suggerendo la deposizione materna anche a livello proteico. Per testare direttamente questo, abbiamo iniettato negli zigoti di Nematostella un costrutto di transgenesi che porta il gene che codifica il reporter fluorescente mOrange2(Shaner et al., 2004) fuso in un peptide di segnale NvePTx1 a valle di un promotore NvePTx1 putativo. Si noti che diverse femmine della prima generazione iniettata (F0) hanno mostrato una forte espressione di mOrange2 in strutture rotonde nei loro mesenteri, che sono molto probabilmente le ovaie dove si formano le uova(Figura 2H-I). Questa osservazione è congruente con il fatto che il mesentere è l’unico tessuto femminile adulto in cui gli alti livelli di trascrizione NvePTx1 sono rilevati ad alti livelli dall’analisi nCounter(Figura 1F). Inoltre, dopo l’induzione della deposizione delle uova, i polipi femminili con il tessuto mesenteriale fluorescente hanno rilasciato confezioni di ovuli che contengono molti ovuli fluorescenti(Figura 2J), sostenendo fortemente la deposizione materna di NvePTx1. Successivamente, eseguendo un test di immunocolorazione su animali transgenici F1 siamo stati in grado di verificare la presenza di due distinti tipi di cellule della ghiandola ectodermica come identificato dai risultati ISH(Figura 2K-M).

Figura 2.NvePTx1 è una tossina che si deposita maternamente nelle uova di Nematostella.(A) Allineamento di sequenza di NvePTx1 e dei suoi omologhi di altre specie cnidarie. La punta della freccia rappresenta il sito di proteolisi che separa la tossina matura dal peptide di segnale. I numeri di adesione appaiono vicino ad ogni sequenza. Nve = Nematostellavectensis; Bcs = Bunodosoma caissarum; Ael = Anthopleura elegantissima; Mse =Metridium senile; Hsy= Hydractinia symbiolongicurpus.(B-C) Larve dizebrafish 16 ore dopo l’incubazione in NvePTx1 e controllo BSA.(D) Rilevazione dell’espressione NvePTx1 da parte di ISH. Dalla fase di blastula in poi, il polo orale è a destra. Da vicino su planula grande allungata(E-F) e piccolo tondo (G) cellule ghiandolari tinte da ISH per NvePTx1 . Cellule della ghiandola, che sono riconoscibili dalle loro vescicole rotonde sono privi di macchia, barra di scala è di 10 µm.(H-I) Un adulto F0 polipo femmina F0 che come uno zigote è stato iniettato da un NvePTx1 ::mOrange2 costruire. mOrange2 espressione è evidente nelle gonadi. Il pannello G è un primo piano della regione indicata nel pannello H dal riquadro giallo punteggiato. La barra di scala è 1000 µm (J) Un pacchetto di uova deposte dalla femmina dal pannello H. Molte delle uova esprimono mOrange2. La scala è di 1000 µm.(K-M) Grande allungata e (N-P) piccole cellule ghiandolari rotonde colorate da anti mCherry. La scala è di 10 µm.
Filogenesi, struttura primaria e l’attività di NEP3
I prodotti del gene NEP3, NEP4, e NEP8 sono stati precedentemente rilevati da MS / MS per essere rilasciato da nematocisti Nematostella al momento della dimissione e quindi sono stati ipotizzati per essere tossine putativo(Moran et al., 2013). Abbiamo rilevato un gene aggiuntivo che codifica un omologo NEP3 nel genoma di Nematostella e lo ha chiamato NEP3-like. Le quattro sequenze nucleotidiche sono state tradotte in silico e sono state suggerite per codificare i precursori delle proteine secretorie dotate di peptidi di segnale tipici (secondo lo strumento online SignalP)(Petersen et al., 2011). Un’ulteriore ricerca nel database Pfam(Finn et al., 2010) ha mostrato che ogni precursore contiene tre motivi di sequenza ShKT (PF01549, Figura 3A) tipici per diverse potenti tossine cnidarie(Aneiros et al., 1993; Castañeda e Harvey, 2009). Sulla base di queste caratteristiche comuni e la loro somiglianza di sequenza, abbiamo designato NEP3, NEP3-like, NEP4, e NEP8 come la ‘famiglia NEP3’. Abbiamo cercato nei database di montaggio del fucile da caccia trascriptomico pubblicamente disponibili e abbiamo identificato diverse sequenze di omologhi dagli anemoni di mare Edwardsiella lineata, Aiptasia pallida e Anthopleura elegantissima così come i coralli sassosi Acropora digitifera e Stylophora pistillata, mostrando una significativa somiglianza di sequenza e struttura di dominio identica ai membri della famiglia NEP3 in Nematostella(Figura 3-figure supplemento 1). Un’analisi filogenetica ha rivelato che ciascuno dei NEP3, NEP4 e NEP8 da Nematostella formato un forte sostegno (valori di bootstrap > 0,5) clade con una proteina molto simile da Edwardsiella (Figura 3B), cheindica l‘ortologia. Così, le nuove sequenze da Edwardsiella sono stati chiamati secondo i loro ortologi Nematostella. Poiché Nematostella ed Edwardsiella sono membri della famiglia di anemoni di mare Edwardsiidae (Rodríguezet al., 2014; Stefanik et al., 2014) e possiedono NEP3, NEP4 e NEP8, possiamo dedurre che queste tre proteine hanno probabilmente avuto origine nell’ultimo antenato comune degli Edwardsiidae. Altre specie cnidarie hanno membri della famiglia dei NEP3 più lontani, e la loro esatta natura ortologica o paralogica non è stata determinata a causa dei bassi valori di bootstrap(Figura 3B). Tuttavia, la loro presenza nei coralli sassosi suggerisce che queste proteine sono apparse già 500 milioni di anni fa nell’ultimo antenato comune dei coralli sassosi edegli anemoni di mare(Shinzato et al., 2011), ma si sono perse in molteplici lignaggi esacoralliani.
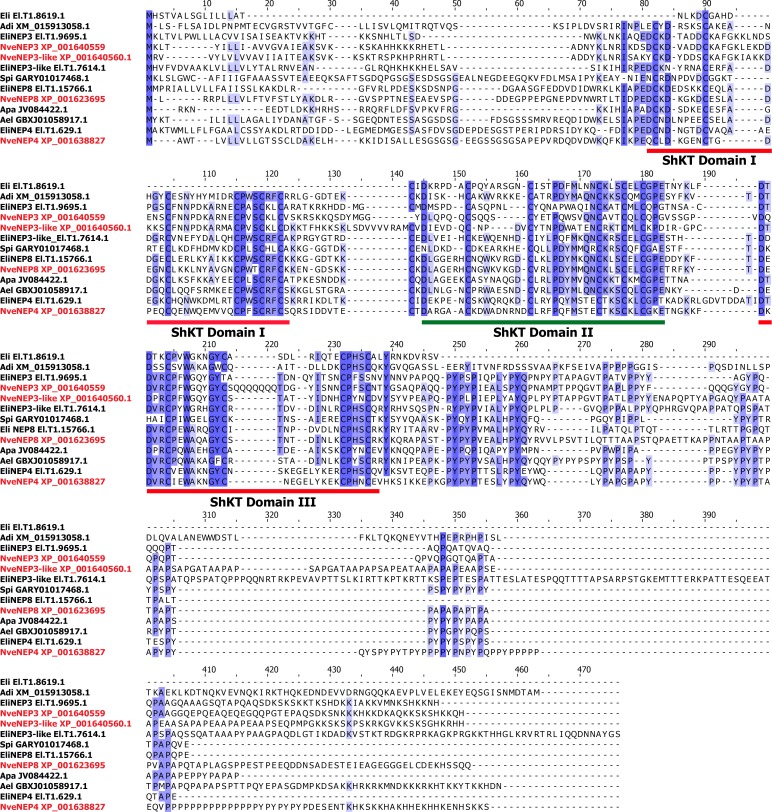
Figura 3-figure supplement 1.NEP3 è una tossina che viene elaborata da una proteina precursore lungo ed è parte di una grande famiglia.allineamento sequenza multipla dei membri della famiglia NEP3 dalla Figura 3B.(A) La struttura primaria del precursore NEP3.(B) Un albero di massima probabilità della famiglia NEP3. I numeri di adesione appaiono vicino ad ogni sequenza e i valori di bootstrap (frazione di 1000 bootstrap) appaiono vicino ad ogni nodo. Nve = Nematostellavectensis; Eli: Edwardsiella lineata; Ael = Anthopleuraelegans; Apa = Aiptasia pallida;Spi = Stylophora pistillata; Adi =Acropora digitifera.(C) Western blot con anticorpo anti-NEP3 di ratto. I campioni vengono scaricati estratto di nematocisti (NE), tossina nativa purificata (PT) e tossina ricombinante (RT).(D-E) Larve dizebrafish dopo 16 ore di incubazione nel controllo NEP3 e BSA. Le punte delle frecce puntano verso le code attorcigliate.I numeri di adesione appaiono vicino ad ogni sequenza. Sequenze di Nematostella sono in rosso.
Per verificare se NEP3 serve come tossina, abbiamo pianificato di esprimerla in forma ricombinante. Tuttavia, inizialmente non era chiaro in quale forma nativa questa proteina si trova nell’animale come abbiamo rilevato un potenziale tandem Lys-Arg, che è un segnale di scissione prominente nelle proteine nematocisti(Anderluh et al., 2000), tra il primo e il secondo dominio di NEP3. Quindi, abbiamo deciso di esplorare prima la struttura primaria del nativo maturo NEP3. A tal fine, abbiamo scaricato nematocisti liofilizzate e analizzato il peso molecolare di NEP3 facendo parte della miscela di proteine espulse da western blot con anticorpi policlonali personalizzati contro il primo dominio NEP3 ShKT. Tuttavia, il western blot ha portato a due bande principali (~ 10 e 12 kDa)(Figura 3C). Una graduale procedura FPLC a tre fasi di filtrazione su gel, scambio di anioni e cromatografia a fase inversa è stata applicata per purificare il frammento dominante NEP3, e in ogni fase abbiamo continuato con la frazione con il segnale più forte western blot. Questa procedura ha prodotto una frazione quasi pura di frammento NEP3 maturo con peso molecolare ~ 10 kDa. Il confronto tra la mobilità elettroforetica del NEP3 maturo nativo e un peptide ricombinante corrispondente al primo dominio ha mostrato che i due peptidi possiedono pesi molecolari molto simili(Figura 3C). Così, si conclude che un peptide nativo composto solo dal primo dominio di NEP3 si trova in Nematostella e viene rilasciato dal nematocisti alla scarica. A seguito di questa scoperta abbiamo effettuato un test di tossicità in cui abbiamo incubato le larve di zebrafish con 0,5 mg/ml di NEP3 maturo ricombinante. Mentre nel gruppo di controllo (incubato per 20 ore in 5 mg/ml di siero albumina bovina) tutti i 20 pesci sono sopravvissuti(Figura 3D), tutte le 17 larve di pesce Danio nel gruppo trattato con NEP3 sono morte entro 5 ore e le larve hanno mostrato una forte contrazione e spasmi della coda(Figura 3E) suggerendo che il peptide NEP3 maturo potrebbe essere neurotossico.

Figura 3-figure supplemento 1.NEP3 è una tossina che viene elaborato da una proteina precursore lungo ed è parte di una grande famiglia.allineamento sequenza multipla dei membri della famiglia NEP3 dalla Figura 3B.(A) La struttura primaria del precursore NEP3.(B) Un albero di massima probabilità della famiglia NEP3. I numeri di adesione appaiono vicino ad ogni sequenza e i valori di bootstrap (frazione di 1000 bootstrap) appaiono vicino ad ogni nodo. Nve = Nematostellavectensis; Eli: Edwardsiella lineata; Ael = Anthopleuraelegans; Apa = Aiptasia pallida;Spi = Stylophora pistillata; Adi =Acropora digitifera.(C) Western blot con anticorpo anti-NEP3 di ratto. I campioni vengono scaricati estratto di nematocisti (NE), tossina nativa purificata (PT) e tossina ricombinante (RT).(D-E) Larve dizebrafish dopo 16 ore di incubazione nel controllo NEP3 e BSA. Le punte delle frecce puntano verso le code attorcigliate.I numeri di adesione appaiono vicino ad ogni sequenza. Sequenze di Nematostella sono in rosso.

Figura 3-figure supplemento 1.Allineamento multiplo delle sequenze dei membri della famiglia NEP3 dalla Figura 3B.I numeri di adesione appaiono vicino ad ogni sequenza. Le sequenze di nematostella sono in rosso.
Nematociti diversi esprimono diversi membri della famiglia NEP3
Per ottenere una migliore risoluzione dell’espressione dei quattro membri della famiglia NEP3, abbiamo impiegato ISH per localizzare la loro espressione in cinque fasi di sviluppo. Tutti e quattro i geni sono espressi in nematociti sulla superficie della planula iniziale(Figura 4A; Figura 4-figure supplement 1). Tuttavia, a partire dalla fase tardiva planula l’espressione di NEP8 si sposta a solo una manciata di nematociti nella faringe inferiore(Figura 4A). A differenza di NEP8, gli altri tre membri della famiglia, NEP3, NEP3-like e NEP4 sono espressi in tutto l’ectoderma in nematociti, con alta concentrazione di cellule che esprimono nel polo orale. Nel polipo primario, l’espressione di NEP3, NEP3-like e NEP4 è evidente nei nematociti nella parete del corpo e nella fisa ectoderma e nella faringe superiore e inferiore(Figura 4A). Inoltre, nelle punte dei tentacoli, che sono molto ricche di nematociti, ci sono un gran numero di nematociti che esprimono le tre tossine, adattandosi bene ai nostri risultati nCounter(Figura 1F). Uno sguardo superficiale su questi risultati può dare l’impressione che in Nematostella ci sia una piccola popolazione di nematociti faringei che esprimono NEP8 e un’altra molto grande popolazione di nematociti che esprimono NEP3, NEP3-like e NEP4 in gran parte dell’ectoderma. Tuttavia, abbiamo deciso di utilizzare la doppia fluorescenza ISH (dFISH) per verificare se questo è veramente il caso.

Figura 4-figure supplemento 1.Figura 4—supplemento figura 1. Modelli di espressione parzialmente sovrapposti e distinti dei membri della famiglia NEP3.da vicino sulla colorazione ISH di NEP3, NEP3-like, NEP4 e NEP8 rivela che essi sono espressi in nematociti.(A) Espressione dei membri della famiglia NEP3 in cinque fasi di sviluppo della Nematostella come determinato da ISH.(B) Espressione dei membri della famiglia NEP3 in planule e polipi di Nematostella come determinato da dFISH. In tutti i pannelli, il polo orale è a destra.Le barre di scala sono di 20 µm.
Negli esperimenti dFISH, abbiamo localizzato le combinazioni mRNA di NEP3 con Ncol3, NEP3 con NEP3-like, NEP3 con NEP4, e NEP3-like con Ncol3. Inaspettatamente, tutte le combinazioni hanno mostrato solo sovrapposizione limitata nella loro espressione, con NEP3-come e Ncol3 che mostra la sovrapposizione più alta e NEP3 e NEP4 che mostra il più basso(Figura 4B). Questo risultato può essere spiegato da due spiegazioni non esclusive: la prima è che la nematogenesi (nematocisti di produzione) è un processo altamente dinamico che richiede l’espressione di geni diversi in tempi diversi lungo il processo di maturazione dei nematociti; la seconda è che i tre membri della famiglia sono per lo più espressi in diverse popolazioni di nematociti e solo pochi nematociti esprimono tutti i membri della famiglia.
Per testare quest’ultima ipotesi, abbiamo iniettato agli zigoti di Nematostella un costrutto che porta una chimera del peptide di segnale di NEP3 con mOrange2 a valle del promotore putativo di NEP3. Gli zigoti hanno iniziato ad esprimere mOrange2 in nematociti circa 4 giorni dopo l’iniezione. Abbiamo sollevato gli animali F0 positivi all’età adulta e poi li abbiamo incrociati con polipi di tipo selvatico per identificare i fondatori. Abbiamo trovato sei femmine e tre maschi fondatori che l’espressione mOrange2 nella loro progenie F1 imitato l’espressione del gene nativo(Figura 5A-C). Per verificare questo, abbiamo eseguito un doppio ISH su animali F1 e F2 (terza generazione) e abbiamo trovato una sovrapposizione quasi perfetta tra l’espressione trascrizionale del transgene mOrange2 e quella del gene della tossina NEP3(Figura 5D-F). mOrange2 espressione è stata osservata in molti nematociti con popolazioni particolarmente dense nelle punte dei tentacoli e nei mesenteri (Figura 5C). Forte espressione di mOrange2 è stato anche rilevato in numerosi nematociti all’interno dei nematosomi(Figura 5G-I), strutture difensive che Nematostella rilascia nei suoi dintorni e all’interno di confezioni di uova (Baboniset al., 2016).

Figura 5.Il NEP3 è espresso solo in un sottoinsieme di nematociti.(A) Tre giorni di planula di una linea transgenica F1 che esprime mOrange2 sotto un promotore NEP3.(B) 6 giorni di larva metamorfosante della stessa linea transgenica.(C) Polipi giovani di due mesi della stessa linea.(D-F) Doppio ISH di un polipo primario F2 della linea transgenica con sonde contro NEP3 e mOrange2. mOrange2 trascrizioni mOrange2 sono stati colorati con un NBT (nitro-blu tetrazolio cloruro di tetrazolio) e BCIP (5-bromo-4-cloro-3′-indolifosfato p-toluidina) soluzione che forma cristalli viola, mentre trascrizioni NEP3 sono stati colorati con FastRed che fornisce il segnale rosso fluorescente.(G-I) Un nematosoma della linea transgenica che mostra il segnale fluorescente mOrange2 all’interno dei suoi nematociti.(J-L) Una diffusione di cellule dissociate dei tentacoli di un polipo NEP3 transgenico F2. Un nematocita mOrange2-positivo è indicato da una freccia nera e spirociti negativi sono indicati da frecce bianche.(M-O) Un’immagine di un altro campo della cellula diffusa da pannelli precedenti. Un nematocita mOrange2-positivo è indicato da una freccia nera e un nematocita negativo è indicato da una freccia bianca. La barra di scala è di 10 µm nei pannelli G-O.(P) Nematociti di un polipo transgenico F2 polipo appuntato nella cuticola di A. salina. Solo un nematocita nella foto è mOrange2-positivo.(D) I nematociti di un polipo F2 della stessa linea transgenica sono appuntati nella pelle di una larva di pesce zebra. Solo alcuni di essi sono mOrange2 positivi. Le frecce bianche indicano esempi di nematociti negativi.
Successivamente, abbiamo dissociato i tentacoli di animali transgenici F1 da una miscela di proteasi commerciali in singole cellule e osservato ciò che le cellule esprimono mOrange2 e quindi NEP3. Come previsto, abbiamo osservato che NEP3 è espresso in nematociti, ma non in spirociti(Figura 5J-L), che si ritiene vengano utilizzati per impigliare le prede e non per la consegna del veleno (Mariscalet al., 1977). Inoltre, abbiamo anche osservato che il NEP3 è espresso solo in una sottopopolazione di nematociti(Figura 5M-O), suggerendo come gli esperimenti ISH e dFISH che diversi nematociti esprimono diverse tossine.
Tuttavia, a quel punto c’era la possibilità che il mOrange2 sia rilevabile solo nello sviluppo dei nematociti e che le capsule mature non brillino a causa di varie limitazioni tecniche come la parete matura della capsula che ostruisce la luce. Per verificare se ci sono capsule mature mOrange2-positive nella nostra linea transgenica, abbiamo sfidato i polipi F1 con i nauplii di Artemia e le larve di pesce zebra. Abbiamo poi preso gli oggetti delle prede attaccate e li abbiamo visualizzati con la microscopia a fluorescenza. Sorprendentemente, mOrange2-positivo capsule con tubuli luminosi sono stati appuntati nel crostaceo e pesce cuticola o pelle, rispettivamente(Figura 5P-Q), indicando che si tratta di capsule mature. Tuttavia, queste capsule sono state accompagnate da altre capsule che erano mOrange2-negativi, suggerendo ancora una volta fortemente che solo una certa sottopopolazione di nematociti in Nematostella sta esprimendo NEP3.

Figura 4-figure supplemento 1.Figura 4—supplemento figura 1. Parzialmente sovrapposti e modelli di espressione distinti dei membri della famiglia NEP3.close up sulla colorazione ISH di NEP3, NEP3-come, NEP4 e NEP8 rivela che sono espressi in nematociti.(A) Espressione dei membri della famiglia NEP3 in cinque fasi di sviluppo della Nematostella come determinato da ISH.(B) Espressione dei membri della famiglia NEP3 in planule e polipi di Nematostella come determinato da dFISH. In tutti i pannelli, il polo orale è a destra.Le barre di scala sono di 20 µm.

Figura 4-figure supplemento 1.Da vicino sulla colorazione ISH di NEP3, NEP3-like, NEP4 e NEP8 rivela che sono espressi in nematociti.Le barre di scala sono 20 µm.

Figura 5.NEP3 è espresso solo in un sottoinsieme di nematociti.(A) Tre giorni di planula di una linea transgenica F1 che esprime mOrange2 sotto un promotore NEP3.(B) 6 giorni di larva metamorfosante della stessa linea transgenica.(C) Polipi giovani di due mesi della stessa linea.(D-F) Doppio ISH di un polipo primario F2 della linea transgenica con sonde contro NEP3 e mOrange2. mOrange2 trascrizioni mOrange2 sono stati colorati con un NBT (nitro-blu tetrazolio cloruro di tetrazolio) e BCIP (5-bromo-4-cloro-3′-indolifosfato p-toluidina) soluzione che forma cristalli viola, mentre trascrizioni NEP3 sono stati colorati con FastRed che fornisce il segnale rosso fluorescente.(G-I) Un nematosoma della linea transgenica che mostra il segnale fluorescente mOrange2 all’interno dei suoi nematociti.(J-L) Una diffusione di cellule dissociate dei tentacoli di un polipo NEP3 transgenico F2. Un nematocita mOrange2-positivo è indicato da una freccia nera e spirociti negativi sono indicati da frecce bianche.(M-O) Un’immagine di un altro campo della cellula diffusa da pannelli precedenti. Un nematocita mOrange2-positivo è indicato da una freccia nera e un nematocita negativo è indicato da una freccia bianca. La barra di scala è di 10 µm nei pannelli G-O.(P) Nematociti di un polipo transgenico F2 polipo appuntato nella cuticola di A. salina. Solo un nematocita nella foto è mOrange2-positivo.(D) I nematociti di un polipo F2 della stessa linea transgenica sono appuntati nella pelle di una larva di pesce zebra. Solo alcuni di essi sono mOrange2 positivi. Le frecce bianche indicano esempi di nematociti negativi.
Interazione tra Nematostella e potenziali predatori e prede
In diversi stadi di sviluppo Nematostella abita varie nicchie ecologiche, e di conseguenza la sua interazione con i predatori e le prede può cambiare durante tutto il ciclo di vita. Qui, abbiamo testato le interazioni di Nematostella con il gamberetto d’erba Palaemonetes sp. e il killifish Fundulus heteroclitus a uovo, planula, polipo primario, e gli stadi di vita adulta(Tabella 1). I gamberi d’erba sono riferito predatori di Nematostella(Kneib, 1985; Kneib, 1988), tuttavia, nelle nostre osservazioni, quando si incontrano i tentacoli dei polipi adulti di Nematostella scavati nel substrato, i gamberi immediatamente ‘saltato’ lontano dai tentacoli (Video 2). Al contrario, in assenza del tipico substrato di fango, i gamberi in un ambiente privo di cibo consumerebbero i polipi adulti iniziando a nutrirsi a lato della loro colonna corporea, evitando il contatto con i tentacoli. Questi risultati sono in accordo con uno studio precedente che ha scoperto che i gamberi possono generalmente predare i polipi di Nematostella, ma non quando sono scavati nel substrato(Posey e Hines, 1991). La risposta dei gamberi d’erba al contatto con i tentacoli è ben correlata con l’elevata sensibilità (LD50 = 1,25 ng/100 mg di gambero) di questa specie alla tossina Nv1, che è altamente espressa nei tentacoli a partire dallo stadio di polipo giovanile.
| Confezione di uova | Uova individuali | Planulae | Polipi primari | Adulti (corpo esposto) | Adulti scavati nel substrato | |
|---|---|---|---|---|---|---|
| Gamberetto d’erba | ++ | ++ | – | + | + | — |
| Larve di pesce | — | ++* | — | — | — | – |
| Pesci adulti | – | – | – | + | – | NA |

Video 2.L’interazione tra una Nematostella adulta scavatacon gamberi erbivori.Solo i tentacoli di Nematostella sono esposti e possono essere incontrati dal gambero. Il gambero salta via facilmente una volta a contatto con i tentacoli.
I nostri risultati indicano che la Nematostella può essere una potenziale fonte di cibo per i gamberi d’erba nelle fasi iniziali dello sviluppo (uova dissociate, confezioni di uova, larve di planule e polipi primari). Gamberetti d’erba tenuti in un ambiente senza cibo per più di 2 giorni consumato uova dissociate e pacchetti di uova, che può essere dovuto a giovani stadi embrionali che non esprimono Nv1. Inoltre, iniezioni di NvePTx1 embrionalmente espresso e NEP3 (50 ng/100 mg gamberetti) non ha prodotto sintomi evidenti nei gamberetti d’erba. Non abbiamo osservato alcuna larva di planulae essere consumato da gamberetti. Tuttavia, è probabile che i gamberi erbivori non incontrino regolarmente larve di planule, in quanto hanno il potenziale di disperdersi lontano dal substrato in cui il gambero si nutre solitamente. Quando i gamberi d’erba sono stati forniti di polipi primari sono stati consumati dopo 7 giorni in cui non sono state fornite altre fonti di cibo, suggerendo che questa fonte di cibo probabilmente non è preferibile, ma viene ingerita.
Fundulus è onnivoro, ed è stato riportato che si nutre di una grande varietà di organismi bentonici(James-Pirri et al., 2001; McMahon et al., 2005) e forse preda di Nematostella(Wiltse et al., 1984). In laboratorio, i pesci adulti non hanno tentato di mangiare polipi adulti, né hanno tentato di mangiare uova dissociate o larve di planule, il che può essere dovuto alle loro piccole dimensioni rispetto ai pesci adulti e può anche non essere riconosciuto come potenziale fonte di cibo. Sorprendentemente, le larve di pesce si sono nutrite di uova quando sono state separate dalla porzione gelatinosa della confezione di uova mediante trattamento con cisteina, ma hanno espulso dalla bocca le uova che sono state separate meccanicamente dalla confezione(Video 3). Questo suggerisce che le uova potrebbero portare composti difensivi che vengono rimossi o inattivati dalla cisteina. I pesci non mangiano le uova se racchiuse all’interno dell’imballaggio dell’uovo e hanno tentato di rimuovere l’imballaggio gelatinoso delle uova dalla loro coda a seguito di incontri casuali(Video 3). Quando le planule fornite come prede, le larve di Fundulus hanno tentato di inghiottire le larve, ma le hanno immediatamente liberate e hanno nuotato via(Video 4).

Video 3.L’interazione dellelarve Fundulus con ilpacchetto di uova di Nematostella.Al contatto con il pacchetto di uova, Fundulus reagisce rapidamente e fugge.

Video 4.L’interazione dellelarve di Fundulus con leplanule di Nematostella.Fundulus espelle attivamente le planule dalle loro bocche, se sono state erroneamente percepite come cibo.
In laboratorio, le larve di Fundulus (1-3 giorni dopo la schiusa) sono state consumate da Nematostella adulta senza substrato, ma sono riuscite ad evitare la predazione da parte di Nematostella quando il substrato era presente, tuttavia, questo è probabilmente dovuto allo spazio verticale aggiunto fornito alle larve di pesce all’interno dei piatti contenenti substrato e anemoni. Con il passare del tempo, può verificarsi un incontro casuale, con conseguente predazione delle larve di Fundulus. Le larve di Fundulus evitano attivamente i polipi primari, tuttavia, gli adulti li consumano quando il cibo è limitato(Tabella 1). Per testare il significato di questa osservazione abbiamo esposto le larve di pesce a tre trattamenti: Artemia morta (fonte di cibo), perle di silice (trattamento inerte) e polipi primari. Le larve di Fundulus hanno trascorso più tempo sul fondo dell’acquario rispetto alla parte superiore quando erano presenti le perle di silice o l ‘Artemia e hanno trascorso più tempo sul fondo rispetto alla parte superiore quando erano presenti i polipi primari(Figura 6). Il tempo trascorso in ogni posizione era significativamente diverso nella nostra analisi ANOVA a senso unico per la parte superiore (valore p = 0,035) e inferiore (valore p = 0,01) per ogni posizione, con l’analisi Tukey che indica che la differenza significativa è stata trovata tra i polipi primari e gli altri due trattamenti, ma non quando si confrontano le perle di silice e Artemia in entrambe le posizioni ( Figura 6). Pertanto, possiamo concludere che le larve di Fundulus tendono ad evitare interazioni con i polipi primari di Nematostella.

Figura 6.Tempo (secondi) trascorso da ogni pesce esposto ad Artemia (rosso), perle di silice (nero), o polipi primari di Nematostella (blu) nella parte superiore (simboli aperti) o inferiore (simboli chiusi) terzo dell’acquario.Le differenze statisticamente significative tra i trattamenti identificati utilizzando ANOVA separati a senso unico e il test Tukey post hoc sono annotate con un asterisco.

Video 2.L’interazione tra una Nematostella adulta scavatacon gamberi erbivori.Solo i tentacoli di Nematostella sono esposti e possono essere incontrati dal gambero. Il gambero salta via facilmente una volta a contatto con i tentacoli.

Video 3.L’interazione dellelarve di Fundulus con ilpacchetto di uova di Nematostella.Al contatto con il pacchetto di uova, Fundulus reagisce rapidamente e fugge.

Video 4.L’interazione dellelarve di Fundulus con leplanule di Nematostella.Fundulus espelle attivamente le planule dalle loro bocche, se sono state erroneamente percepite come cibo.

Figura 6.Tempo (secondi) trascorso da ogni pesce esposto ad Artemia (rosso), perle di silice (nero), o polipi primari di Nematostella (blu) nella parte superiore (simboli aperti) o inferiore (simboli chiusi) terzo dell’acquario.Le differenze statisticamente significative tra i trattamenti identificati utilizzando ANOVA separati a senso unico e il test Tukey post hoc sono annotate con un asterisco.
Discussione
La nostra scoperta di diversi livelli di espressione delle tossine in diversi stadi di sviluppo e nei tessuti adulti suggerisce fortemente che la composizione del veleno cambia durante lo sviluppo e che ogni arsenale di tossine potrebbe essere stato modellato dalla selezione per le diverse interazioni biotiche. Come Nematostella si sviluppa da una larva nuotante non predatoria ad un polipo predatore sessile adulto che è 150 volte più grande della larva(Figura 1A), le sue interazioni interspecifiche cambiano notevolmente attraverso lo sviluppo. Ad esempio, le confezioni di uova di Nematostella possono essere consumate dai gamberi d’erba, ma è altamente improbabile che i polipi adulti facciano parte della dieta dei gamberi d’erba(Tabella 1; Video 2). Queste osservazioni sono correlate con la dinamica di espressione della Nv1 che è altamente tossica per i gamberi. A differenza dei gamberi erbivori, le larve di Fundulus non consumano le confezioni di uova di Nematostella ed espellono attivamente dalla bocca le planule, che sono state erroneamente percepite come cibo(Video 3 e 4). Consumano solo singole uova in seguito a lavaggi con cisteina, che non rappresentano condizioni native. La ridotta presenza di predazione può essere spiegata dalla presenza di varie tossine, come i membri della famiglia NEP3, che possono proteggere la Nematostella dai predatori di pesci anche in fasi di sviluppo molto precoci. Questa nozione è supportata dagli effetti tossici del NEP3 sulle larve dei pesci zebra.
Ipotizziamo che queste interazioni dinamiche, insieme al costo metabolico potenzialmente elevato delle tossine(Nisani et al., 2012), abbiano guidato l’evoluzione di una composizione velenosa distinta in ogni stadio dello sviluppo. Inoltre, nel caso degli cnidari, un costo metabolico aggiuntivo deriva dal fatto che le nematocisti sono apparati di somministrazione del veleno monouso che devono essere riprodotti in numero molto elevato dopo ogni interazione antagonista. Quindi, c’è un chiaro vantaggio nell’uso di un veleno altamente adattato in ogni fase dello sviluppo.
Poiché il veleno nelle planule è usato esclusivamente per la difesa, mentre il veleno nei polipi è usato sia per la difesa che per la cattura delle prede, i nostri risultati si riferiscono anche ai risultati precedenti negli scorpioni(Inceoglu et al., 2003) e nelle lumache a cono(Dutertre et al., 2014), dove è stato dimostrato che le composizioni del veleno per la difesa e la cattura delle prede sono diverse. Inoltre, alcune delle tossine che abbiamo localizzato possono essere attribuite per funzioni specifiche in base ai loro modelli di espressione. Ad esempio, è molto probabile che NvePTx1 sia una tossina difensiva per la sua presenza nell’uovo e nelle cellule della ghiandola ectodermica della planula(Figure 2D-H e 7), mentre NEP8 è probabilmente usato nel polipo per uccidere le prede dopo che sono state inghiottite, in quanto si esprime esclusivamente nella faringe inferiore e nei mesenteri (Figure4A e 7).
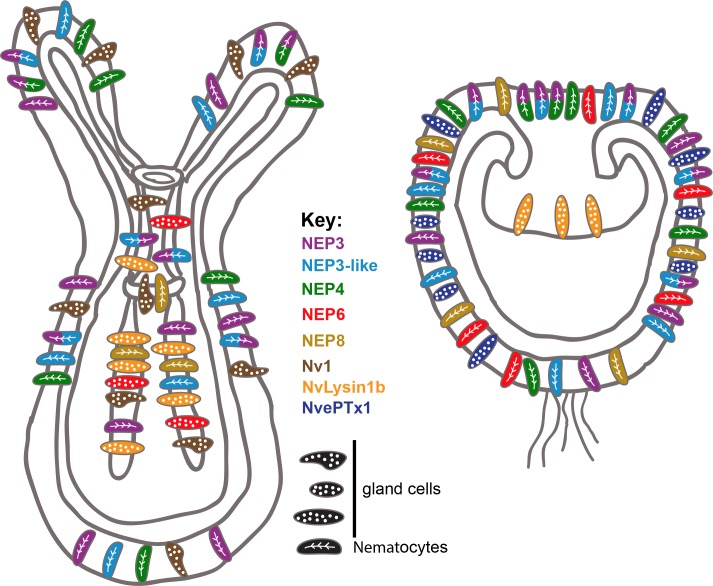
Figura 7.Sintesi delle attuali conoscenze sull’espressione della tossina spazio-temporale in planula precoce e polipo primario di Nematostella.Questa illustrazione riassume il lavoro attuale e i lavori precedenti(Moran et al., 2012a; Moran et al., 2012b; Moran et al., 2013). Il grado di sovrapposizione di espressione del NEP8 con altre tossine è attualmente sconosciuto.
La protezione chimica delle uova è stata riportata in diversi animali come i ragni vedova nera(Buffkin et al., 1971; Lei et al., 2015), lumache(Dreon et al., 2013), polpi e alcuni pesci e anfibi(Bane et al., 2014). I nostri dati su Nematostella NvePTx1 espressi in uova e stadi embrionali supportano ulteriormente l’idea dell’importanza ecologica della difesa chimica nelle fasi iniziali della vita in tutto il regno animale.
Le nostre scoperte che i membri della famiglia NEP3 sono espressi in diverse popolazioni di nematociti rivelano che la diversità dei nematociti in Nematostella supera di gran lunga le valutazioni basate sulla morfologia, che hanno rivelato solo due tipi di nematocisti in Nematostella: gli aplonemi basitrichi (chiamati anche basitrichi) e i mastigofori microbasici(Frank e Bleakney, 1976; Zenkert et al., 2011). Uno studio sull’Idra ha scoperto che due membri di una singola famiglia di tossine che formano i pori sono espressi in due tipi di nematociti morfologicamente distinti(Hwang et al., 2007), ma, per quanto ne sappiamo, non è mai stata segnalata prima d’ora una simile complessità dei modelli di espressione delle tossine in nematociti morfologicamente simili. Inoltre, meccanismi di biosintesi del veleno a livello cellulare non sono stati riportati anche in organismi velenosi più complessi. Tuttavia, a bassa risoluzione, alcuni rapporti suggeriscono che all’interno di una stessa ghiandola velenifera diverse unità secretorie sono specializzate nella produzione di un numero limitato di tossine(Dutertre et al., 2014; Undheim et al., 2015). Pertanto, le cellule secretorie velenifere specializzate sono probabilmente un tratto comune tra gli animali velenosi.
I distinti modelli di espressione della famiglia NEP3 forniscono anche importanti indicazioni sull’evoluzione delle tossine. Ad esempio, l’espressione di NEP8 nei nematociti faringei, e la sua assenza dai tentacoli e dalla parete esterna del corpo, dove sono espressi i suoi paraloghi NEP3, NEP3-like e NEP4(Figure 4A e 7), è un’indicazione per la sub o neo-funzionalizzazione. La specializzazione dei diversi membri della famiglia è anche supportata dalla loro conservazione in Edwardsiella(Figura 3B).
La variazione nei modelli di espressione dei membri della famiglia NEP3 e il fatto che almeno quattro diversi tipi di cellule ghiandolari in stadi di sviluppo e tessuti distinti esprimono diverse tossine (Nv1, Nvlysin1b, NEP6 e NvePTx1) in Nematostella suggerisce un paesaggio velenoso altamente complesso in questa specie(Figura 7). A prima vista, un tale sistema potrebbe sembrare relativamente inefficiente. Tuttavia, si ipotizza che la presenza di molti tipi diversi di cellule produttrici di tossine, fornisce modularità e consente una plasticità evolutiva dell’espressione delle tossine. Infatti, i nostri risultati, così come quelli di altri, suggeriscono che diverse specie di anemoni di mare esprimono tossine simili in diversi tipi di cellule(Moran et al., 2012b) ein diversi tessuti(Macrander et al., 2016). Questa plasticità evolutiva potrebbe essere uno dei fattori che hanno reso gli anemoni di mare un gruppo di successo che vive in tutti gli oceani del mondo negli ultimi 600 milioni di anni.

Figura 7.Sintesi delle attuali conoscenze sull’espressione della tossina spazio-temporale nei primi planula e nel polipo primario di Nematostella.Questa illustrazione riassume il lavoro attuale e i lavori precedenti(Moran et al., 2012a; Moran et al., 2012b; Moran et al., 2013). Il grado di sovrapposizione di espressione del NEP8 con altre tossine è attualmente sconosciuto.
Materiali e metodi
Cultura degli anemoni di mare
Gli embrioni dinematostella, le larve e i giovani sono stati coltivati in 16‰ di acqua salata a 22°C. Gli adulti sono stati cresciuti nella stessa salinità ma a 17°C. I polipi venivano alimentati con Artemia nauplii tre volte alla settimana. L’induzione della deposizione delle uova di gameti è stata effettuata secondo un protocollo pubblicato(Genikhovich e Technau, 2009b).
nAnalisi del contatore
RNA totale da diversi stadi di sviluppo e parti del corpo della femmina adulta Nematostella è stato estratto con Tri-Reagent (Sigma-Aldrich, St. Louis, MO) secondo il protocollo del produttore, trattato con Turbo DNAse (Thermo Fisher Scientific, Waltham, MA) e poi ri-estratto con Tri-Reagent. La qualità dell’RNA è stata valutata su Bioanalyzer Nanochip (Agilent, Santa Clara, CA) e sono stati utilizzati solo campioni con RNA Integrity Number (RIN) ≥8.0. Ogni campione è stato preparato da decine di campioni (polipi adulti e i loro tessuti) o da centinaia di campioni (tutti i più giovani stadi di sviluppo) al fine di normalizzare per ogni singola variazione. Questi campioni sono stati analizzati sulla piattaforma nCounter (NanoString Technologies, Seattle, WA, USA; eseguita da Agentek Ltd., Israele) in triplicati tecnici, ciascuno composto da un diverso lotto di campioni secondo un protocollo precedentemente descritto(Geiss et al., 2008). In breve, per ogni trascrizione da testare, sono state generate due sonde e ibridate al rispettivo mRNA. Gli mRNA sono stati immobilizzati su una cartuccia e i codici a barre su una delle sonde sono stati contati da un microscopio a fluorescenza automatico. Per la normalizzazione abbiamo usato una media geometrica dei livelli di espressione di 5 geni di riferimento con espressione stabile attraverso lo sviluppo. I geni sono stati selezionati come segue: abbiamo calcolato l’entropia di Shannon (come descritto in[Schug et al., 2005]) per ciascuno dei 23.041 geni di Nematostella sulla base di stime normalizzate dell’abbondanza delle trascrizioni per sei punti temporali dello sviluppo di Nematostella(Helm et al., 2013). Abbiamo poi classificato i geni in base all’entropia, che indica una variazione temporale minima dell’abbondanza, e tra i primi 20 abbiamo scelto cinque geni (Sequenze di riferimento NCBI XM_001629766.1, XM_001628650.1, XM_001625670.1, XM_001640487.1 e XM_001624235.1) con modelli genici completi e livelli di abbondanza media che coprono l’intervallo sperimentale previsto. Sequenze di sonde, punteggi di entropia e tutti i dati di lettura nCounter grezzi e normalizzati sono disponibili nel file supplementare 1.
Analisi semiquantitativa MS/MS
Centinaia di uova non fecondate, planule vecchie di 4 giorni, polipi primari vecchi di 9 giorni, maschi adulti (cinque individui) e femmine adulte (cinque individui) sono stati lisati in urea 8M, 400 mM soluzione di bicarbonato di ammonio e centrifugati (22000 × g, 20 min, 4°C). Le concentrazioni di proteine sono state misurate con il BCA Protein Assay Kit (Thermo Fisher Scientific). Dieci µg di proteine sono stati ridotti con DTT e alchilati con iodoacetamide. Digestione triptica (0,3 µg tripsina / campione) è stata eseguita durante la notte a 37 ° C, seguita da aggiunta di 0,05% ProteaseMAX tensioattivo (Promega Corp., USA) e l’ulteriore incubazione per 1 ora a 37 ° C. I peptidi triptici sono stati dissalati su C18 StageTips autoprodotto(Rappsilber et al., 2007). Un totale di 1,25 µg di peptidi di ogni campione sono stati iniettati nello spettrometro di massa.
L’analisi MS è stata eseguita in quattro repliche tecniche utilizzando uno spettrometro di massa Q Exactive Plus (Thermo Fisher Scientific) accoppiato on-line ad uno strumento UHPLC a nanoflusso (Ultimate 3000 Dionex, Thermo Fisher Scientific). I peptidi elusi sono stati separati su una corsa a gradiente di 180 minuti ad una portata di 0,2 µl/min su una colonna PepMap RSLC C18 a fase inversa (50 cm ×75 µm, 2 µm, 100 Å, Thermo Fisher Scientific). Le scansioni di indagine (380-2.000 m/z, valore target 3E6 cariche, tempi massimi di iniezione ionica 50 ms) sono state acquisite e seguite da una maggiore energia di dissociazione collisionale (HCD) basata sulla frammentazione (energia di collisione normalizzata 25). Una risoluzione di 70.000 è stata utilizzata per le scansioni di indagine e fino a 15 ioni precursori più abbondanti scelti dinamicamente sono stati frammentati (finestra di isolamento 1,6 m/z). Le scansioni MS/MS sono state acquisite con una risoluzione di 17.500 (valore target 5E4 cariche, massima iniezione di ioni 57 ms). L’esclusione dinamica è stata di 60 s.
I dati degli spettri di massa sono stati elaborati utilizzando la piattaforma di calcolo MaxQuant, versione 1.5.3.12(Cox e Mann, 2008). Le liste dei picchi sono state confrontate con le sequenze di codifica tradotte dei modelli genici di N. vectensis. La ricerca ha incluso la cisteina carbamidometilazione come modifica fissa e l’ossidazione della metionina e l’acetilazione dei terminali N come modifiche variabili. Sono stati considerati peptidi con un minimo di sette lunghezza amminoacidi e il FDR richiesto è stato impostato all’1% a livello di peptidi e proteine. L’identificazione delle proteine richiedeva almeno due peptidi unici o rasoio per gruppo proteico. Sono state utilizzate le opzioni peptide dipendente e match-between-runs. La quantificazione relativa delle proteine è stata eseguita utilizzando i valori iBAQ. I file grezzi MS/MS e i risultati dell’analisi MaxQuant sono stati depositati al Consorzio ProteomeXchange tramite il repository partner PRIDE(Vizcaíno et al., 2016) con l’identificatore di dati PXD008218.
Ibridazione in situ (ISH)
L’ISH singolo e doppio sono stati eseguiti come descritto in precedenza(Genikhovich e Technau, 2009a; Moran et al., 2013). dFISH è stato eseguito anche secondo i protocolli pubblicati(Nakanishi et al., 2012; Wolenski et al., 2013) con tiramide coniugata a coloranti fluorescenti Dylight 488 e Dylight 594 (Thermo Fisher Scientific). In ISH e FISH, gli embrioni più vecchi di 4 giorni sono stati trattati con 2 u/µl T1 RNAse (Thermo Fisher Scientific) dopo il lavaggio della sonda per ridurre lo sfondo. Gli embrioni e le larve macchiate sono stati visualizzati con un microscopio Eclipse Ni-U dotato di una telecamera DS-Ri2 e di un software Elements BR (Nikon, Tokyo, Giappone). Per ogni gene sono stati testati almeno 20 campioni di ogni stadio di sviluppo.
Transgenesi
Per generare costrutti transgenici, abbiamo sostituito il gene mCherry con mOrange2(Shaner et al., 2004) e abbiamo sostituito la sequenza promotrice del pNvT-MHC::mCH plasmide(Renfer et al., 2010). Per il gene NEP3, abbiamo inserito al plasmide 920 bp a monte del sito di partenza della trascrizione così come il primo esone non codificante, il primo introne e la parte del secondo esone che codifica il peptide di segnale di NEP3 (scaffold_7:1,219,288-1,221,320 del genoma di Nematostella). Per il gene NvePTX1, abbiamo inserito al plasmide 1033 bp a monte del sito di partenza della trascrizione così come il primo esone non codificante, il primo introne e la regione del secondo esone che codifica il peptide del segnale (scaffold_14:1,246,079-1,247,853 del genoma di Nematostella). I costrutti sono stati iniettati con il lievito meganucleasi I-SceI (New England Biolabs, Ipswich, MA) per facilitare l’integrazione genomica(Renfer et al., 2010). Gli animali transgenici sono stati visualizzati sotto uno stereomicroscopio SMZ18 dotato di una telecamera DS-Qi2 (Nikon).
Immunostaining
L’immunocolorazione è stata eseguita secondo un protocollo precedentemente descritto(Moran et al., 2012b), utilizzando un anticorpo policlonale di coniglio disponibile in commercio contro mCherry (Abcam) diluito a 1:400 e DAPI (Thermo Fisher Scientific) diluito a 1:500.
Filogenetica
Sequenze di famiglie di proteine NEP e NvePTx1 sono state recuperate utilizzando le ricerche BLAST(Altschul et al., 1990) contro il database di sequenze nucleotidiche non ridondanti del NCBI e la base EdwardsiellaBase (Stefaniket al., 2014). L’analisi di massima probabilità è stata impiegata per la ricostruzione delle storie evolutive molecolari. Gli alberi sono stati generati utilizzando PhyML 3.0(Guindon et al., 2010), e il supporto dei nodi è stato valutato con 1000 repliche di bootstrapping.
Dissociazione tentacolare
I tentacoli di Nematostella sono stati dissociati utilizzando una combinazione di papaina (2 mg/ml; Sigma-Aldrich: P4762), collagenasi (2 mg/ml; Sigma-Aldrich: C9407) e pronasi (4 mg/ml; Sigma-Aldrich: P5147) in DTT (1,3 mM) e soluzione di PBS (1,8 mM NaH2PO4, 8,41 mM Na2HPO4, 175 mM NaCl, pH 7,4). I tentacoli sono stati incubati con la miscela di proteasi a 22°C durante la notte. I tessuti sono stati poi dissociati in singole cellule da sfogliare le provette delicatamente e poi per centrifugazione a 400 × g per 15 minuti a 4 ° C, seguita da risospensione in PBS.
Purificazione di NEP3 da nematocisti
Nematocisti liofilizzate sono state ottenute dai Laboratori della Baia di Monterey (Cesarea, Israele). 2,5 g di nematocisti sono stati scaricati per incubazione con 80 ml di trifosfato di sodio all’1% (Sigma-Aldrich). Dopo la centrifugazione (21.000 × g, 20 min), l’estratto grezzo è stato concentrato con filtri centrifughi Amicon con 3 kDa tagliati (Merck Millipore, Billerica, MA) a 2 ml di volume, filtrato attraverso filtri centrifughi Amicon con 50 kDa tagliati (Merck Millipore) e utilizzato per un’ulteriore purificazione. Nella prima fase, l’estratto è stato frazionato per esclusione dimensionale FPLC su una colonna calibrata Superdex 75 (60 × 1,6 cm, GE Healthcare, Little Chalfont, UK) in buffer PBS. Frazioni di proteine con peso molecolare inferiore a 17,6 kDa sono state messe in comune e il buffer PBS è stato scambiato a 20 mM di etanolamina a pH nove utilizzando filtri centrifughi Amicone, tagliati fuori 3 kDa. Nella seconda fase, le frazioni SEC sono state separate mediante scambio anionico FPLC utilizzando una colonna HiTrapQ HP (1 ml, GE Healthcare) e un gradiente di concentrazione NaCl (0-750 mM NaCl in 30 volumi di colonna, 20 mM etanolamina pH 9,0). Le frazioni sono state analizzate da western blot con anticorpi anti-Nep3 e quelle positive sono state messe in comune. All’ultimo passo, Nep3 frammento è stato purificato da fase inversa FPLC su una colonna Resource RPC (3 ml, GE Healthcare) utilizzando gradiente di concentrazione di acetonitrile (8-60% CH3CN in 25 volumi di colonna, 0,1% di acido trifluoracetico). Le frazioni corrispondenti ai singoli picchi sono state raccolte e analizzate da western blot con anticorpi anti-NEP3.
Espressione ricombinante e purificazione delle tossine
Un frammento di DNA sintetico che codifica l’intero polipeptide NEP3 è stato acquistato da GeneArt (Regensburg, Germania). Il frammento corrispondente al primo dominio di NEP3 tra i siti di scissione di Lys-Arg è stato amplificato da PCR, clonato ed espresso come una proteina di fusione di His6-thioredoxin in Shuffle T7 Escherichia coli ceppo T7 (New England Biolabs).
I frammenti di DNA sintetico Nv1 e NvePTx1 sono stati acquistati da Integrated DNA Technologies (Coralville, IA) e clonati in un vettore pET40 modificato (il peptide del segnale DSBC è stato cancellato dal frammento che codifica il peptide del segnale DSBC da esso è stato cancellato da Protein Expression and Purification facility dell’Università Ebraica per consentire l’espressione citoplasmatica del DSBC). Nv1 e NvePTx1 sono stati espressi nel ceppo BL21(DE3) E. coli (Merck Millipore) come fusioni con His6-DSBC.
Il tag poliistidina delle proteine di fusione è stato utilizzato per la purificazione dal lisato di E. coli lisato per affinità con il nichel FPLC. Le proteine di fusione purificate sono state scisse in due frammenti da Tobacco Etch Virus (TEV) proteasi (temperatura ambiente, durante la notte) in un sito di scissione della proteasi TEV a monte dei frammenti di tossina. Le tossine ricombinanti sono state poi purificate mediante la fase inversa FPLC su una colonna RPC Resource RPC (GE Healthcare) utilizzando un gradiente di concentrazione di acetonitrile in acido trifluoroacetico allo 0,1%.
Western blot
Anticorpi policlonali personalizzati specifici per il primo dominio di NEP3 sono stati acquistati da GenScript (Piscataway, NJ). Il peptide sintetico contenente le posizioni amminoacidiche 47-91 è stato utilizzato come antigene per l’immunizzazione di due ratti. Gli anticorpi sono stati purificati affinità su una colonna rivestita con l’antigene. Le proteine sono state separate per elettroforesi su gel di Tris-tricina a gradiente 10-20% (Bio-Rad, Hercules, CA) e di conseguenza trasferite su membrane di 0,2 um PVDF (Bio-Rad). Le membrane sono state bloccate dal 5% di latte scremato in tampone TBST (50 mM Tris base, 150 mM NaCl, 0,1% Tween 20, pH 7,6) e incubate con anticorpi anti-NEP3 (1 ug/ml) in 5% di siero albumina bovina (BSA) in tampone TBST (4°C, durante la notte). Questo è stato seguito da incubazione con anticorpi di capra anti-ratto IgG anticorpi coniugati con perossidasi di rafano (0,1 ug/ml; Jackson ImmunoResearch, West Grove, PA) in 5% di latte scremato in TBST (temperatura ambiente, durante la notte). Il reagente ECL (GE Healthcare) è stato utilizzato per la visualizzazione delle bande proteiche che interagiscono con gli anticorpi anti-NEP3. La chemiluminescenza è stata registrata con un sistema di imaging Odyssey Fc (LI-COR Biosciences, Lincoln, NE) e il marcatore di dimensione fluorescente (Bio-Rad) è stato ripreso sullo stesso sistema.
Animali per prede-predatori e test di tossicità
Le larve diDanio rerio di età inferiore alle 120 ore sono state generosamente fornite dal Dr. Adi Inbal (la Scuola di Medicina dell’Università Ebraica). L’uso di queste giovani larve non richiede permessi etici secondo le leggi europee e israeliane.
Le uova fertilizzate Fundulus di Kings Creek, VA (37°18 16.2″N 76°24 58.9″W) e Scorton Creek, MA (41°43’52.1″N 70°24’51.3″W) sono state gentilmente fornite dal Dr. Rafael Trevisan (Duke University) e Diane Nacci (Environmental Protection Agency), rispettivamente. Sono stati tenuti in acqua di mare artificiale (ASW) del 15 ‰ a temperatura ambiente fino alla schiusa (circa 2-3 settimane) e utilizzati immediatamente per le analisi comportamentali. Esperimenti su F. heteroclitus sono stati effettuati in base al permesso n. 17-018 concesso dall’Institutional Animal Care and Use Committee (IACUC) presso l’Università del North Carolina a Charlotte secondo le norme etiche dell’Office of Laboratory Animal Welfare (National Institutes of Health, USA).
Il primo lotto di gamberi erbivori e di Fundulus adulti è stato raccolto in un estuario vicino a Georgetown, SC (33°21’01.0″N 79°11’26.1″W). Una seconda raccolta di gamberi erbivori è stata raccolta a Sippewissett Marsh, MA (41°35’22.9″N 70°38’17.0″W). Gli animali sono stati trasportati al laboratorio e tenuti in 15 ‰ ASW fino all’uso. Esperimenti di interazione sono stati condotti in 15 ‰ ASW in piatti 24 pozzetti o ciotole in pyrex (~ 150 ml di acqua, ~ 10 cm di diametro), a seconda delle dimensioni degli animali e le interazioni in corso di registrazione. Gamberetti d’erba sono stati alimentati ogni giorno con cozze o TetraMin cibo per pesci tropicali (Tetra Holding, USA). Fundulus sono stati alimentati due volte al giorno con Artemia allevati in laboratorio.
Saggi di tossicità
Per valutare la tossicità di NEP3 e NvePTx1 sui pesci, le larve di D. rerio di 4 giorni sono state incubate con 0,5 mg/ml di peptidi in 500 µl di pozzo. Ogni esperimento è stato condotto in duplicati. 5 mg/ml BSA è stato utilizzato come controllo negativo. Tre replicati sono stati eseguiti per trattamento e ogni replicato incluso 5-7 larve. Effetto è stato filmato e monitorato sotto uno stereomicroscopio SMZ18 (Nikon) dopo 5 min, 15 min, 1 ora e 15-17 ore di incubazione.
Per valutare la tossicità sui gamberi d’erba, Nv1, NEP3, e NvePTx1 sono stati disciolti in tampone PBS a 2,5-50 ng/µl concentrazione e 1 µl è stato iniettato nell’addome dal lato ventrale per ogni 200 mg di massa di gamberi. Dieci gamberi sono stati iniettati ad ogni concentrazione.
Saggi di interazione tra i diversi stadi di vita di Nematostella e le potenziali prede e specie di predatori
Per testare l’interazione di Fundulus con Nematostella, le larve di pesce sono state messe in 24 piastre di pozzo e sono stati condotti screening preliminari che coinvolgono duplicati di osservazioni sperimentali per 5-10 uova isolate, larve di planula, e polipi primari di Nematostella . Sulla base di queste osservazioni iniziali, abbiamo condotto ulteriori esperimenti osservazionali in triplicati, con ogni pozzetto contenente cinque larve Fundulus che interagiscono con diversi pacchetti di uova o decine di larve di planula. Per ogni saggio, i pesci sono stati osservati con porzioni della loro interazione registrate per documentare come i pesci hanno risposto alle confezioni di uova o alle larve di planula. Inoltre, per le uova isolate, gli esperimenti di osservazione sono stati condotti con una singola larva Fundulus in ogni pozzo insieme a 10-15 uova. Il numero di uova sono state annotate all’inizio dell’esperimento e osservate ogni 2 ore per un periodo di 8 ore. Esperimenti osservazionali che coinvolgono due larve Fundulus e due Nematostella adulti sono stati condotti in duplicati utilizzando piccoli piatti Pyrex (~ 50 ml di ASW) per un periodo di 2 giorni, con e senza substrato. Per il Fundulus adulto, esperimenti osservazionali che coinvolgono un singolo adulto accanto a uova (>100), confezioni di uova (4-5), larve (>100), polipi primari (>100), e polipi adulti Nematostella (3) sono stati condotti per oltre 48 ore in piccoli piatti in pyrex (~ 200 ml di acqua, ~ 10 cm di diametro). Le interazioni sono state saggiate in triplicati per gamberi, con un singolo gambero che interagisce con diversi stadi di vita: tre confezioni di uova, >100 uova, >100 larve di planula, >100 polipi primari, e tre adulti Nematostella (con e senza substrato). Attraverso tutti gli esperimenti osservazionali sono stati osservati modelli comportamentali simili attraverso repliche sperimentali, tuttavia, non siamo stati in grado di identificare esattamente quanti casi di una reazione avversa si è verificato durante le nostre osservazioni. A volte era difficile distinguere tra i pesci che mostravano modelli di nuoto sporadici, che non potevamo collegare con loro interagendo con Nematostella.
Per testare il potenziale effetto dei polipi primari di Nematostella sulle larve di Fundulus, le larve di Fundulus appena schiuse (N = 10) sono state collocate in un piccolo acquario di vetro (~3 ml) con 15 ‰ ASW. Comportamento dei pesci è stato registrato oltre 30 min per esperimento utilizzando il Moticam 580 (Motic, Hong Kong, Cina), 15 min che serve come controllo e 15 min con un trattamento. Artemia appena schiusa sono stati sacrificati dal congelamento e utilizzati come controllo positivo (N = 10). Perle di silice di 0,5 mm di diametro (BioSpec Products, Bartlesville, OK) sono state utilizzate come controllo negativo (N = 10). Per ogni video, la registrazione è stata suddivisa nelle seguenti sezioni in queste scale temporali: 0-5 min – acclimatazione, 5-15 min – controllo, 15-20 min – acclimatazione al trattamento, 20-30 min – comportamento registrato. Il contenitore dell’acqua è stato diviso in tre parti uguali (‘fondo’, ‘centro’ e ‘cima’) in base alla lunghezza del pesce. Se il pesce era nel ‘centro’ senza alcuna porzione del suo corpo che attraversava l’uno o l’altro dei due lati, non è stato registrato in quanto non è stata considerata una preferenza. ANOVA a senso unico sono stati effettuati separatamente per i punti di tempo superiore e inferiore per ogni trattamento. Trattamenti specifici che erano statisticamente significativi sono stati identificati utilizzando l’analisi Tukey post hoc. I risultati sono stati tracciati in GraphPad Prism versione 7.00 per Windows (GraphPad Software, La Jolla, CA).
References
- Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. Journal of Molecular Biology. 1990; 215:403-410. DOI | PubMed
- Anderluh G, Podlesek Z, Macek P. A common motif in proparts of Cnidarian toxins and nematocyst collagens and its putative role. Biochimica et Biophysica Acta (BBA) – Protein Structure and Molecular Enzymology. 2000; 1476:372-376. DOI | PubMed
- Aneiros A, García I, Martínez JR, Harvey AL, Anderson AJ, Marshall DL, Engström A, Hellman U, Karlsson E. A potassium channel toxin from the secretion of the sea anemone Bunodosoma granulifera. Isolation, amino acid sequence and biological activity. Biochimica et Biophysica Acta (BBA) – General Subjects. 1993; 1157:86-92. DOI | PubMed
- Babonis LS, Martindale MQ, Ryan JF. Do novel genes drive morphological novelty? An investigation of the nematosomes in the sea anemone Nematostella vectensis. BMC Evolutionary Biology. 2016; 16DOI | PubMed
- Bane V, Lehane M, Dikshit M, O’Riordan A, Furey A. Tetrodotoxin: chemistry, toxicity, source, distribution and detection. Toxins. 2014; 6:693-755. DOI | PubMed
- Buffkin DC, Russell FE, Deshmukh A. Preliminary studies on the toxicity of black widow spider eggs. Toxicon. 1971; 9:393-402. DOI | PubMed
- Casewell NR, Wüster W, Vonk FJ, Harrison RA, Fry BG. Complex cocktails: the evolutionary novelty of venoms. Trends in Ecology & Evolution. 2013; 28:219-229. DOI | PubMed
- Castañeda O, Harvey AL. Discovery and characterization of cnidarian peptide toxins that affect neuronal potassium ion channels. Toxicon. 2009; 54:1119-1124. DOI | PubMed
- Cox J, Mann M. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nature Biotechnology. 2008; 26:1367-1372. DOI | PubMed
- David CN, Ozbek S, Adamczyk P, Meier S, Pauly B, Chapman J, Hwang JS, Gojobori T, Holstein TW. Evolution of complex structures: minicollagens shape the cnidarian nematocyst. Trends in Genetics. 2008; 24:431-438. DOI | PubMed
- Dreon MS, Frassa MV, Ceolín M, Ituarte S, Qiu JW, Sun J, Fernández PE, Heras H. Novel animal defenses against predation: a snail egg neurotoxin combining lectin and pore-forming chains that resembles plant defense and bacteria attack toxins. PLoS One. 2013; 8DOI | PubMed
- Dutertre S, Jin AH, Vetter I, Hamilton B, Sunagar K, Lavergne V, Dutertre V, Fry BG, Antunes A, Venter DJ, Alewood PF, Lewis RJ. Evolution of separate predation- and defence-evoked venoms in carnivorous cone snails. Nature Communications. 2014; 5DOI | PubMed
- Finn RD, Mistry J, Tate J, Coggill P, Heger A, Pollington JE, Gavin OL, Gunasekaran P, Ceric G, Forslund K, Holm L, Sonnhammer EL, Eddy SR, Bateman A. The Pfam protein families database. Nucleic Acids Research. 2010; 38:D211-D222. DOI | PubMed
- Frank P, Bleakney JS. Histology and sexual reproduction of the anemone Nematostella vectensis Stephenson 1935. Journal of Natural History. 1976; 10:441-449. DOI
- Fry BG, Roelants K, Champagne DE, Scheib H, Tyndall JD, King GF, Nevalainen TJ, Norman JA, Lewis RJ, Norton RS, Renjifo C, de la Vega RC. The toxicogenomic multiverse: convergent recruitment of proteins into animal venoms. Annual Review of Genomics and Human Genetics. 2009; 10:483-511. DOI | PubMed
- Geiss GK, Bumgarner RE, Birditt B, Dahl T, Dowidar N, Dunaway DL, Fell HP, Ferree S, George RD, Grogan T, James JJ, Maysuria M, Mitton JD, Oliveri P, Osborn JL, Peng T, Ratcliffe AL, Webster PJ, Davidson EH, Hood L, Dimitrov K. Direct multiplexed measurement of gene expression with color-coded probe pairs. Nature Biotechnology. 2008; 26:317-325. DOI | PubMed
- Genikhovich G, Technau U. In situ hybridization of starlet sea anemone (Nematostella vectensis) embryos, larvae, and polyps. Cold Spring Harbor Protocols. 2009a; 2009DOI | PubMed
- Genikhovich G, Technau U. Induction of spawning in the starlet sea anemone Nematostella vectensis, in vitro fertilization of gametes, and dejellying of zygotes. Cold Spring Harbor Protocols. 2009b; 2009DOI | PubMed
- Gibbs HL, Sanz L, Chiucchi JE, Farrell TM, Calvete JJ. Proteomic analysis of ontogenetic and diet-related changes in venom composition of juvenile and adult Dusky Pigmy rattlesnakes (Sistrurus miliarius barbouri). Journal of Proteomics. 2011; 74:2169-2179. DOI | PubMed
- Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0. Systematic Biology. 2010; 59:307-321. DOI | PubMed
- Hand C, Uhlinger KR. The culture, sexual and asexual reproduction, and growth of the sea anemone Nematostella vectensis. The Biological Bulletin. 1992; 182:169-176. DOI | PubMed
- Helm RR, Siebert S, Tulin S, Smith J, Dunn CW. Characterization of differential transcript abundance through time during Nematostella vectensis development. BMC Genomics. 2013; 14DOI | PubMed
- Hwang JS, Ohyanagi H, Hayakawa S, Osato N, Nishimiya-Fujisawa C, Ikeo K, David CN, Fujisawa T, Gojobori T. The evolutionary emergence of cell type-specific genes inferred from the gene expression analysis of Hydra. PNAS. 2007; 104:14735-14740. DOI | PubMed
- Inceoglu B, Lango J, Jing J, Chen L, Doymaz F, Pessah IN, Hammock BD. One scorpion, two venoms: prevenom of Parabuthus transvaalicus acts as an alternative type of venom with distinct mechanism of action. PNAS. 2003; 100:922-927. DOI | PubMed
- James-Pirri MJ, Raposa KB, Catena JG. Diet composition of mummichogs, fundulus heteroclitus, from restoring and unrestricted regions of a New England (U.S.A.) Salt Marsh. Estuarine, Coastal and Shelf Science. 2001; 53:205-213. DOI
- Kass-Simon G, Scappaticci AA. The behavioral and developmental physiology of nematocysts. Canadian Journal of Zoology. 2002; 80:1772-1794. DOI
- Kneib RT. Predation and disturbance by grass shrimp, Palaemonetespugio Holthuis, in soft-substratum benthic invertebrate assemblages. Journal of Experimental Marine Biology and Ecology. 1985; 93:91-102. DOI
- Kneib RT. Testing for indirect effects of predation in an intertidal soft-bottom community. Ecology. 1988; 69:1795-1805. DOI
- Layden MJ, Rentzsch F, Röttinger E. The rise of the starlet sea anemone Nematostella vectensis as a model system to investigate development and regeneration. Wiley Interdisciplinary Reviews: Developmental Biology. 2016; 5:408-428. DOI | PubMed
- Lei Q, Yu H, Peng X, Yan S, Wang J, Yan Y, Wang X. Isolation and preliminary characterization of proteinaceous toxins with insecticidal and antibacterial activities from black widow spider (L. tredecimguttatus) eggs. Toxins. 2015; 7:886-899. DOI | PubMed
- Levitan S, Sher N, Brekhman V, Ziv T, Lubzens E, Lotan T. The making of an embryo in a basal metazoan: Proteomic analysis in the sea anemone Nematostella vectensis. Proteomics. 2015; 15:4096-4104. DOI | PubMed
- Lotan A, Fishman L, Loya Y, Zlotkin E. Delivery of a nematocyst toxin. Nature. 1995; 375DOI | PubMed
- Lotan T, Chalifa-Caspi V, Ziv T, Brekhman V, Gordon MM, Admon A, Lubzens E. Evolutionary conservation of the mature oocyte proteome. EuPA Open Proteomics. 2014; 3:27-36. DOI
- Macrander J, Broe M, Daly M. Tissue-specific venom composition and differential gene expression in sea anemones. Genome Biology and Evolution. 2016; 8:2358-2375. DOI | PubMed
- Madio B, Undheim EAB, King GF. Revisiting venom of the sea anemone Stichodactyla haddoni: Omics techniques reveal the complete toxin arsenal of a well-studied sea anemone genus. Journal of Proteomics. 2017; 166:83-92. DOI | PubMed
- Mariscal RN, McLean RB, Hand C. The form and function of cnidarian spirocysts. 3. Ultrastructure of the thread and the function of spirocysts. Cell and Tissue Research. 1977; 178:427-433. DOI | PubMed
- Marlow HQ, Srivastava M, Matus DQ, Rokhsar D, Martindale MQ. Anatomy and development of the nervous system of Nematostella vectensis, an anthozoan cnidarian. Developmental Neurobiology. 2009; 69:235-254. DOI | PubMed
- McMahon KW, Johnson BJ, Ambrose WG. Diet and movement of the killifish,Fundulus heteroclitus, in a Maine salt marsh assessed using gut contents and stable isotope analyses. Estuaries. 2005; 28:966-973. DOI
- Moran Y, Weinberger H, Reitzel AM, Sullivan JC, Kahn R, Gordon D, Finnerty JR, Gurevitz M. Intron retention as a posttranscriptional regulatory mechanism of neurotoxin expression at early life stages of the starlet anemone Nematostella vectensis. Journal of Molecular Biology. 2008a; 380:437-443. DOI | PubMed
- Moran Y, Weinberger H, Sullivan JC, Reitzel AM, Finnerty JR, Gurevitz M. Concerted evolution of sea anemone neurotoxin genes is revealed through analysis of the Nematostella vectensis genome. Molecular Biology and Evolution. 2008b; 25:737-747. DOI | PubMed
- Moran Y, Fredman D, Szczesny P, Grynberg M, Technau U. Recurrent horizontal transfer of bacterial toxin genes to eukaryotes. Molecular Biology and Evolution. 2012a; 29:2223-2230. DOI | PubMed
- Moran Y, Genikhovich G, Gordon D, Wienkoop S, Zenkert C, Ozbek S, Technau U, Gurevitz M. Neurotoxin localization to ectodermal gland cells uncovers an alternative mechanism of venom delivery in sea anemones. Proceedings of the Royal Society B: Biological Sciences. 2012b; 279:1351-1358. DOI | PubMed
- Moran Y, Praher D, Schlesinger A, Ayalon A, Tal Y, Technau U. Analysis of soluble protein contents from the nematocysts of a model sea anemone sheds light on venom evolution. Marine Biotechnology. 2013; 15:329-339. DOI | PubMed
- Moran Y, Fredman D, Praher D, Li XZ, Wee LM, Rentzsch F, Zamore PD, Technau U, Seitz H. Cnidarian microRNAs frequently regulate targets by cleavage. Genome Research. 2014; 24:651-663. DOI | PubMed
- Nakanishi N, Renfer E, Technau U, Rentzsch F. Nervous systems of the sea anemone Nematostella vectensis are generated by ectoderm and endoderm and shaped by distinct mechanisms. Development. 2012; 139:347-357. DOI | PubMed
- Nisani Z, Boskovic DS, Dunbar SG, Kelln W, Hayes WK. Investigating the chemical profile of regenerated scorpion (Parabuthus transvaalicus) venom in relation to metabolic cost and toxicity. Toxicon. 2012; 60:315-323. DOI | PubMed
- Orts DJ, Moran Y, Cologna CT, Peigneur S, Madio B, Praher D, Quinton L, De Pauw E, Bicudo JE, Tytgat J, de Freitas JC. BcsTx3 is a founder of a novel sea anemone toxin family of potassium channel blocker. FEBS Journal. 2013; 280:4839-4852. DOI | PubMed
- Petersen TN, Brunak S, von Heijne G, Nielsen H. SignalP 4.0: discriminating signal peptides from transmembrane regions. Nature Methods. 2011; 8:785-786. DOI | PubMed
- Posey MH, Hines AH. Complex predator-prey interactions within an estuarine benthic community. Ecology. 1991; 72:2155-2169. DOI
- Prokopec SD, Watson JD, Waggott DM, Smith AB, Wu AH, Okey AB, Pohjanvirta R, Boutros PC. Systematic evaluation of medium-throughput mRNA abundance platforms. RNA. 2013; 19:51-62. DOI | PubMed
- Putnam NH, Srivastava M, Hellsten U, Dirks B, Chapman J, Salamov A, Terry A, Shapiro H, Lindquist E, Kapitonov VV, Jurka J, Genikhovich G, Grigoriev IV, Lucas SM, Steele RE, Finnerty JR, Technau U, Martindale MQ, Rokhsar DS. Sea anemone genome reveals ancestral eumetazoan gene repertoire and genomic organization. Science. 2007; 317:86-94. DOI | PubMed
- Rappsilber J, Mann M, Ishihama Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2007; 2:1896-1906. DOI | PubMed
- Renfer E, Amon-Hassenzahl A, Steinmetz PR, Technau U. A muscle-specific transgenic reporter line of the sea anemone, Nematostella vectensis. PNAS. 2010; 107:104-108. DOI | PubMed
- Rodríguez E, Barbeitos MS, Brugler MR, Crowley LM, Grajales A, Gusmão L, Häussermann V, Reft A, Daly M. Hidden among sea anemones: the first comprehensive phylogenetic reconstruction of the order Actiniaria (Cnidaria, Anthozoa, Hexacorallia) reveals a novel group of hexacorals. PLoS One. 2014; 9DOI | PubMed
- Ruppert EE, Fox RS, Barnes RD. Invertebrate Zoology: A Functional Evolutionary Approach. Thomson-Brooks/Cole: Belmont; 2004.
- Safavi-Hemami H, Siero WA, Kuang Z, Williamson NA, Karas JA, Page LR, MacMillan D, Callaghan B, Kompella SN, Adams DJ, Norton RS, Purcell AW. Embryonic toxin expression in the cone snail Conus victoriae: primed to kill or divergent function?. The Journal of Biological Chemistry. 2011; 286:22546-22557. DOI | PubMed
- Santana RC, Perez D, Dobson J, Panagides N, Raven RJ, Nouwens A, Jones A, King GF, Fry BG. Venom profiling of a population of the theraphosid spider phlogius crassipes reveals continuous ontogenetic changes from juveniles through adulthood. Toxins. 2017; 9:116. DOI | PubMed
- Schug J, Schuller WP, Kappen C, Salbaum JM, Bucan M, Stoeckert CJ. Promoter features related to tissue specificity as measured by Shannon entropy. Genome Biology. 2005; 6DOI | PubMed
- Shaner NC, Campbell RE, Steinbach PA, Giepmans BN, Palmer AE, Tsien RY. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nature Biotechnology. 2004; 22:1567-1572. DOI | PubMed
- Shinzato C, Shoguchi E, Kawashima T, Hamada M, Hisata K, Tanaka M, Fujie M, Fujiwara M, Koyanagi R, Ikuta T, Fujiyama A, Miller DJ, Satoh N. Using the Acropora digitifera genome to understand coral responses to environmental change. Nature. 2011; 476:320-323. DOI | PubMed
- Stefanik DJ, Lubinski TJ, Granger BR, Byrd AL, Reitzel AM, DeFilippo L, Lorenc A, Finnerty JR. Production of a reference transcriptome and transcriptomic database (EdwardsiellaBase) for the lined sea anemone, Edwardsiella lineata, a parasitic cnidarian. BMC Genomics. 2014; 15DOI | PubMed
- Thomason JC. Cnida discharge and the mechanism of venom delivery in Anemonia viridis (Cnidaria, Actiniaria). Hydrobiologia. 1991; 216-217:649-654. DOI
- Undheim EA, Hamilton BR, Kurniawan ND, Bowlay G, Cribb BW, Merritt DJ, Fry BG, King GF, Venter DJ. Production and packaging of a biological arsenal: evolution of centipede venoms under morphological constraint. PNAS. 2015; 112:4026-4031. DOI | PubMed
- Vizcaíno JA, Csordas A, del-Toro N, Dianes JA, Griss J, Lavidas I, Mayer G, Perez-Riverol Y, Reisinger F, Ternent T, Xu QW, Wang R, Hermjakob H. 2016 update of the PRIDE database and its related tools. Nucleic Acids Research. 2016; 44:D447-D456. DOI | PubMed
- Wikramanayake AH, Hong M, Lee PN, Pang K, Byrum CA, Bince JM, Xu R, Martindale MQ. An ancient role for nuclear beta-catenin in the evolution of axial polarity and germ layer segregation. Nature. 2003; 426:446-450. DOI | PubMed
- Wilbur HM. Complex life cycles. Annual Review of Ecology and Systematics. 1980; 11:67-93. DOI
- Williams RB. A redescription of the brackish-water sea anemone Nematostella vectensis Stephenson, with an appraisal of congeneric species. Journal of Natural History. 1975; 9:51-64. DOI
- Wiltse WI, Foreman KH, Teal JM, Valiela I. Effects of predators and food resources on the macrobenthos of salt marsh creeks. Journal of Marine Research. 1984; 42:923-942. DOI
- Wolenski FS, Layden MJ, Martindale MQ, Gilmore TD, Finnerty JR. Characterizing the spatiotemporal expression of RNAs and proteins in the starlet sea anemone, Nematostella vectensis. Nature Protocols. 2013; 8:900-915. DOI | PubMed
- Zenkert C, Takahashi T, Diesner MO, Özbek S. Morphological and molecular analysis of the Nematostella vectensis cnidom. PLoS One. 2011; 6DOI | PubMed
Fonte
Columbus-Shenkar YY, Sachkova MY, Macrander J, Fridrich A, Modepalli V, et al. () Dynamics of venom composition across a complex life cycle. eLife 7e35014. https://doi.org/10.7554/eLife.35014





