Abstract
Introduzione
L’implementazione del sistema batterico CRISPR/Cas9 in eucarioti ha innescato un salto quantico nell’editing del genoma mirato in qualsiasi organismo con un genoma sequenziato o regione di destinazione(Cong et al., 2013; Jinek et al., 2012; Mali et al., 2013). Le rotture a doppio filamento (DSB) specifiche del sito sono catalizzate dall’enzima Cas9 guidato da un singolo RNA con una breve regione complementare al sito target. In risposta, le macchine per la riparazione del DNA endogeno non omologo (NHEJ) sigillano il DSB. Poiché la riparazione perfetta ripristinerà il sito target CRISPR/Cas9, vengono selezionate le mutazioni introdotte da NHEJ imprecise.
Per acquisire una precisa modifica del genoma, il DSB iniziale dovrebbe essere fissato tramite il meccanismo di riparazione a conduzione omologa (HDR) che è preferibilmente attivo durante la fase finale S/G2 del ciclo cellulare(Hustedt e Durocher, 2017). I modelli di DNA del donatore con regioni di affiancamento omologhe al locus bersaglio vengono utilizzati per introdurre mutazioni specifiche o particolari sequenze di DNA. Il dsDNA iniettato (lineare) si multimerizza rapidamente(Winkler et al., 1991), cosa che probabilmente accade anche negli approcci basati su CRISPR/Cas9. Inoltre, l’alta attività di NHEJ ri-ligating CRISPR/Cas9 mediato DSBs mediato può multimerizzare iniettato (lineare) dsDNA modelli di donatore dsDNA iniettato (lineare). Ciò pone un problema poiché, di conseguenza, la ricombinazione precisa HDR-mediata in HDR dei modelli di donatore a copia singola è piuttosto rara.
Sono state seguite diverse strategie per evitare l’NHEJ e/o favorire l’HDR. NHEJ è stato interferito dall’inibizione farmacologica della DNA ligasi IV(Maruyama et al., 2015). Al contrario, HDR doveva essere favorito dalla fusione della proteina HDR-mediante la proteina del lievito Rad52 a Cas9(Wang et al., 2017). Analogamente, la rimozione di Cas9 nella fase G1/S(Gutschner et al., 2016) collegandolo alla regione N-terminale dell’inibitore della replicazione del DNA Gemininin, dovrebbe limitare l’introduzione di tagli a doppio filamento nella fase G2, quando l’HDR è più evidente(Hustedt e Durocher, 2017). Questi approcci hanno migliorato l’integrazione mediata dall’HDR dei fianchi dell’omologia, mentre non hanno affrontato l’integrazione segnalata di multimer(Auer et al., 2014) che si verifica dopo l’iniezione di modelli di dsDNA come plasmidi o prodotti di PCR(Winkler et al., 1991).
Risultati
Per migliorare l’HDR senza interferire con i macchinari di riparazione del DNA endogeno, abbiamo mirato a stabilire modelli di donatori di DNA che sfuggano alla multimetrizzazione o agli eventi NHEJ. Abbiamo così bloccato entrambi i 5´end di PCR amplificati con cassette di donatore di dsDNA lunghe e amplificate usando moieties “ingombranti” come Biotina, Amino-dT (A-dT) e distanziatori di carbonio (ad es. Spacer C3, SpC3). Questo dovrebbe proteggere il donatore di DNA dalla multimerization e dall’integrazione tramite NHEJ, favorendo così un’integrazione precisa ed efficiente di una singola copia tramite HDR(Figura 1A).
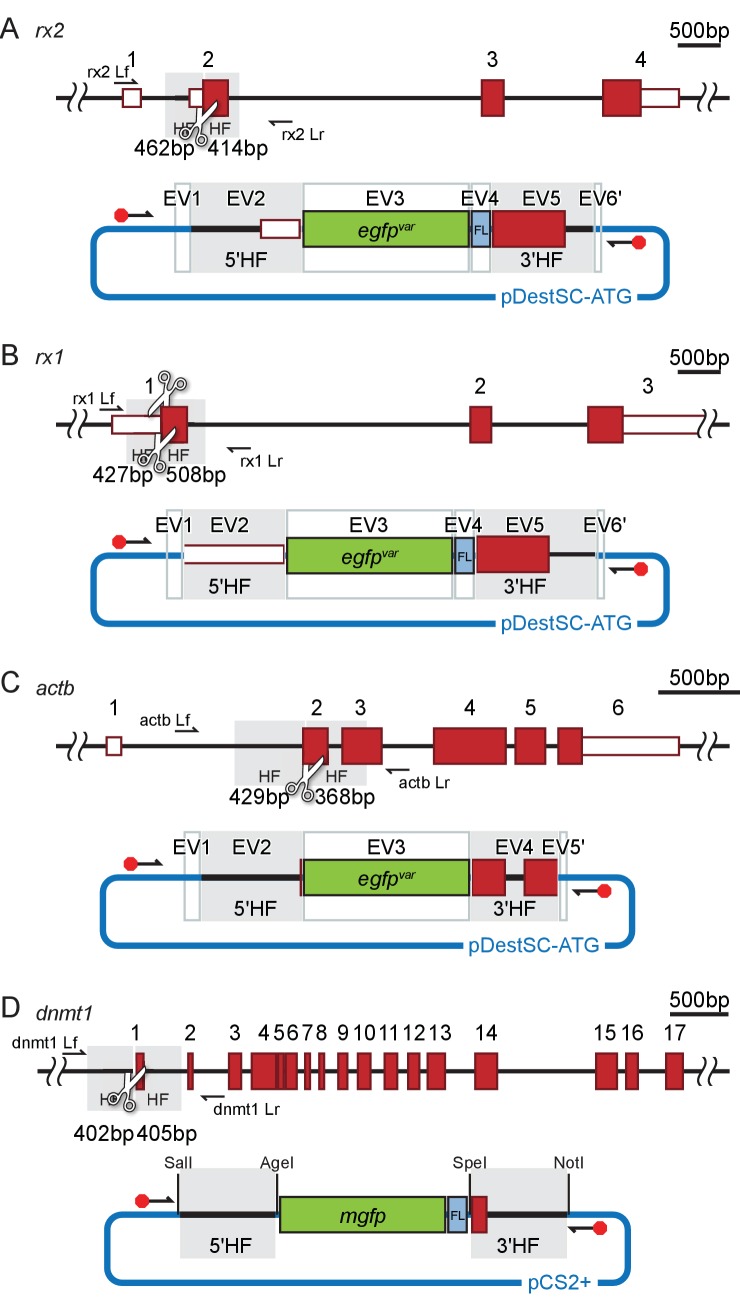
Figura 1-figure supplement 1.La modifica di 5′ estremità di frammenti di dsDNA lunghi impedisce la multimerization in vivo.rappresentazione schematica dei plasmidi del donatore.(A) Rappresentazione schematica di amplificazione lungo dsDNA donatore cassetta PCR con primer universale (frecce nere) complementare alla spina dorsale vettore clonazione al di fuori della cassetta donatore assemblato (ad esempio gfp con fianchi omologia). Moieties ingombranti come la Biotina alle estremità 5′ di entrambi i primer modificati (ottagono rosso) impediscono la multimerization/NHEJ del dsDNA, fornendo le condizioni ottimali per l’integrazione HDR-mediata a copia singola dopo CRISPR/Cas9-introdotto DSB al locus target (forbici grigie). Rappresentazione dei primer del locus (Lf/Lr) e dei primer gfp interni (Gf/Gr) per la genotipizzazione PCR di eventi di integrazione gfp presunti mediati in HDR.(B) L’analisi del Southern blot rivela lo stato monomerico dei frammenti di dsDNA iniettati in vivo per la modifica 5′ con Biotina o Spacer C3. I dsDNA lunghi generati con i primer di controllo non modificati o i primer Amino-dT attaccati si multimerizzano come indicato da una scala ad alto peso molecolare apparente già entro due ore dopo l’iniezione (hpi). Nota: 5′ moieties non ha migliorato la stabilità del DNA iniettato.(A-D) Rappresentazione schematica in scala dei rispettivi locus target (A, rx2; B , rx1; C , actb ; D, dnmt1 ) con UTR (scatole bianche con contorni rossi) ed esoni (scatole rosse) evidenziati. Sono indicati i fianchi dell’omologia (HF, grigio), i siti target sgRNA (forbici bianche) e i rispettivi primer del locus (frecce nere, Lf, Lr, vedi file supplementare 2). Il plasmide del donatore assemblato rispettabile (A, B, C Golden GATEway(Kirchmaier et al., 2013) o D, clonazione convenzionale) che serviva da modello per l’amplificazione PCR della cassetta del donatore dsDNA gfp lungo non modificato/modificato è rappresentato qui sotto (FL, linker flessibile; sequenza di backbone in blu). Sono indicati i vettori di ingresso (EV) o i siti di enzimi di restrizione per la clonazione. La posizione dei primer che affiancano la cassetta del donatore è indicata, la modifica è evidenziata da un ottagono rosso.
Per prima cosa abbiamo affrontato l’impatto della modifica 5′ del donatore sulla formazione di multimer in vivo. Abbiamo iniettato i donatori di dsDNA modificati e non modificati in embrioni di medaka a stadio monocellulare(Oryzias latipes) e abbiamo analizzato lo stato conformazionale del materiale iniettato durante lo sviluppo zigotico. donatori di dsDNA sono stati generati da PCR che impiegano 5′ primer modificati e non modificati rispettivamente, e fornendo inoltre tracce di DIG-dUTP per l’etichettatura del prodotto risultante PCR. La conformazione dei donatori di dsDNA iniettati è stata analizzata nel DNA totale estratto dopo 2, 4 e 6 ore dopo l’iniezione, rispettivamente. Il DNA è stato frazionato in base alle dimensioni per elettroforesi su gel e la conformazione del DNA del donatore è stata rilevata dopo il tamponamento del DNA su una membrana di nylon da anticorpi anti-DIG(Figura 1B). In donatori di controllo non modificati con etichetta DIG, abbiamo scoperto la multimerization già a 2 ore dopo l’iniezione come evidente da una scala di DNA donatore etichettato che rappresenta il numero di copie diverse multimer(Figura 1B). Al contrario, la modifica della biotina e della SpC3 dei donatori marcati con DIG ha impedito la multimerization entro sei ore dopo l’iniezione(Figura 1B). I donatori di dsDNA stabiliti da primati modificati A-dT, tuttavia, multimerizzati e hanno prodotto risultati simili a quelli dei donatori di dsDNA marcati con DIG non modificati (Figura 1B). I nostri risultati rivelano che le modifiche di Biotina e SpC3 5′ impediscono efficacemente la multimerization dei donatori in vivo. Pur bloccando fortemente la multimerization, la modifica 5′ del dsDNA non ha apparentemente migliorato la stabilità del dsDNA risultante (confrontare i donatori modificati e non modificati nel tempo, Figura 1B).
Per verificare se la modifica 5′ non solo riduce il grado di multimerization, ma ha anche un impatto sull’integrazione HDR-mediata da una singola copia di donatori di dsDNA lunghi, abbiamo progettato cassette di donatori contenenti gfp per una lettura visiva immediata. Abbiamo generato donatori di fusione gfp in-frame per quattro diversi geni: i fattori di trascrizione della omeobox retinica rx2 e rx1(Reinhardt et al., 2015), la beta-actina citoscheletrica non muscolare (actb)(Stemmer et al., 2015) e il DNA metiltransferasi 1(dnmt1). Le cassette dei donatori contenevano il rispettivo fianco di omologia 5′ (HF) (462 bp per rx2, 430 bp per rx1, 429 bp per actb, 402 bp per dnmt1 ), seguito dalla sequenza di codifica gfp in-frame, un linker flessibile nel caso di rx2, rx1 e dnmt1 , e i corrispondenti 3′ HF (414 bp per rx2, 508 bp per rx1, 368 bp per actb, 405 bp per dnmt1; rappresentazione schematica in Figura 1A, Figura 1-figure supplement 1 per la progettazione dettagliata del donatore). Per amplificare i donatori di dsDNA lunghi abbiamo impiegato una coppia di primer universali (5′ modificati o non modificati come controllo) complementari alla spina dorsale dei vettori di clonazione (pDestSC-ATG [Kirchmaieret al., 2013] o pCS2+ [Ruppet al., 1994]) che comprendono l’intera cassetta del donatore assemblata (Figura 1-figure supplement 1).
I donatori di dsDNA lunghi modificati o non modificati sono stati successivamente co-iniettati insieme a Cas9 mRNA e il rispettivo sgRNA specifico del locus in medaka a uno stadio di zigoti di medaka(Loosli et al., 1999; Rembold et al., 2006). Per tutti e quattro i quattro loci (e tutte le modifiche 5′) testati abbiamo osservato il targeting efficiente come evidente dall’espressione GFP all’interno del dominio di espressione previsto (Figura 2A, file supplementare 1, Figura 2-figure supplement 1).
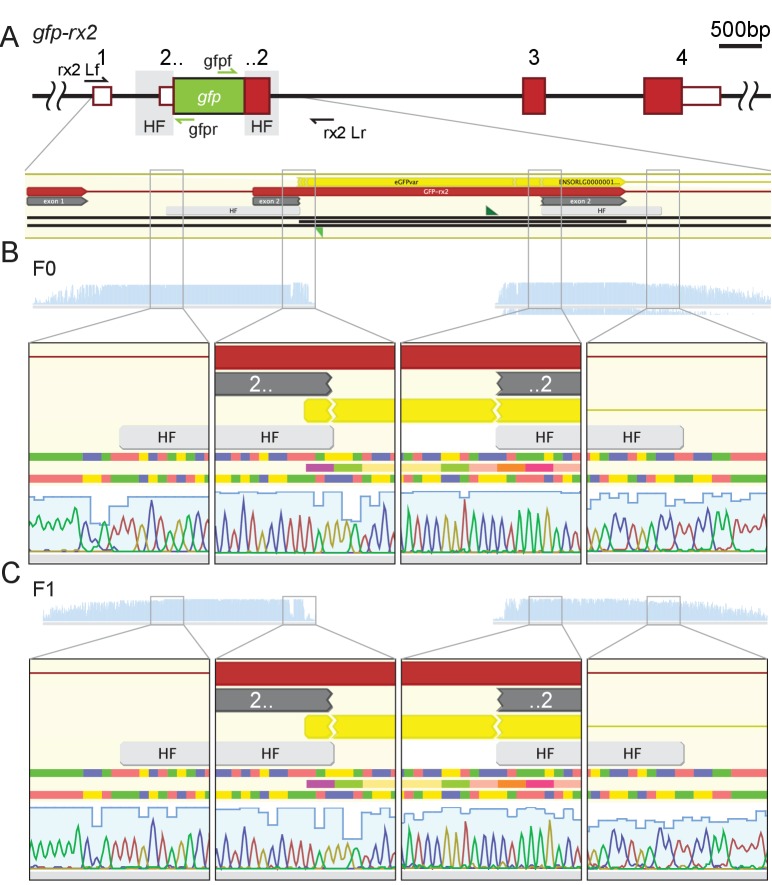
Figura 2-figure supplement 3.La modifica di 5′ estremità di frammenti di dsDNA lunghi promuove l’integrazione HDR-mediata da una singola copia.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.5′ La modifica della biotina dei donatori di dsDNA lunghi migliora fortemente l’integrazione HDR-mediata.la trasmissione stabile del germline della singola copia HDR-mediata da una precisa integrazione gfp.(A) GFP espressione nel rispettivo dominio di espressione dopo HDR-mediata integrazione HDR-mediata di dsDNA modificato gfp donatore cassette in rx2, rx1, actb e dnmt1 ORF nella generazione iniettata.(B) Singolo embrione PCR genotipizzazione PCR evidenzia efficiente HDR-mediata HDR-mediata integrazione singola copia di 5’Biotina modificati donatori dsDNA lungo, ma non cassette donatore non modificato. Locus PCR rivela la dimensione della banda indicativa dell’integrazione gfp a copia singola (asterisco) oltre agli alleli senza integrazione gfp (punta di freccia aperta). Amplificazione del donatore gfp (freccia bianca) per il controllo.Embrioni iniettati con Biotina non modificata o 5’Biotina, Amino-dT o Spacer C3 modificato lungo dsDNA gfp-rx2 cassette donatore sono stati segnati per la sopravvivenza ad un dpi, e per l’espressione GFP a due dpi. n, numero totale di embrioni iniettati.10.7554/eLife.39468.007Figure 2-figure supplement 1-source data 1.Quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.(A) rx2 locus PCR genotipizzazione PCR di singoli embrioni positivi GFP-Rx2 iniettati con cassette donatrici di gfp non modificate o 5’Biotina o Spacer C3 modificato long dsDNA gfp (asterisco verde, integrazione HDR-mediata a copia singola di gfp, 2547 bp, punta di freccia aperta, allele rx2 senza integrazione gfp, 1719 bp;). Barra orizzontale, singolo embrione; L, rx2 locus PCR con rx2 Lf/rx2 Lr; G, gfp PCR interna per il controllo con Gf/Gr.(B) Sintesi qualitativa dello spettro di banda (integrazione HDR-mediata in HDR a copia singola di gfp, allele rx2 senza integrazione gfp, altro) risultante dalla genotipizzazione PCR in(A). n, numero di GFP-Rx2 genotipizzati che esprimono gli embrioni.(A) Rappresentazione schematica in scala del locus gfp-rx2 con UTR (scatole bianche con contorni rossi), esoni (scatole rosse) e fianchi di omologia (HF, grigio) evidenziati.(B, C) I singoli GFP-Rx2 che esprimono gli embrioni (F0, B; F1, C) sono stati genotipizzati usando i primer rx2 Lf/rx2 Lr (frecce nere) e sequenziati con i primer gfpf e gfpr (frecce verdi).
I tassi di sopravvivenza degli embrioni iniettati con Biotina e SpC3 5′ donatori di dsDNA modificato non differivano significativamente dagli embrioni iniettati con i donatori di controllo dsDNA non modificati (file supplementare1, figura 2-figure supplement 1). Al contrario, l’iniezione di A-dT 5′ donatori dsDNA modificato dsDNA ha portato ad un’elevata letalità embrionale (file supplementare1, figura 2 e mdash;supplemento di figura 1).
Abbiamo poi analizzato la frequenza di eventi HDR a copia singola a seguito di un attento approccio PCR a ciclo limitato su DNA genomico di GFP che esprimono embrioni iniettati con donatori di dsDNA non modificati, Biotina o SpC3 5′ modificato dsDNA. Il nostro approccio ha permesso di distinguere gli alleli senza integrazione gfp (cioè la dimensione del locus wild-type) da quelli generati rispettivamente da HDR e NHEJ e ha affrontato la dimensione dell’integrazione con un locus che abbraccia la PCR con un numero ridotto di cicli di PCR (<=30) per omettere artefatti di fusione-PCR in vitro (dati propri e [Wone Dawid, 2017]). Per determinare il potere predittivo dell’espressione delle GFP per una perfetta integrazione, abbiamo genotipizzato in modo casuale gli embrioni che esprimono le GFP utilizzando i primer del locus (Lf/Lr) localizzati distalmente alle HF utilizzate (Figura 1-figure supplement 1) e abbiamo indirizzato la fusione della sequenza del donatore gfp verso i geni target (Figura 2B, Figura 2-figure supplement 2 e 3).
Impiegando donatori non modificati, il tasso di HDR era molto basso, come dimostra l’amplificazione predominante degli alleli senza integrazione del gfp(Figura 2B, Figura 2-figure supplement 2). In forte contrasto, i donatori di dsDNA lunghi modificati di 5′ hanno portato ad un HDR efficiente già rilevabile nella generazione iniettata per tutti i loci mirati. Per l’etichettatura gfp di rx2, 6 su 10 selezionati in modo casuale, GFP-espressione di embrioni ha mostrato una precisa HDR-mediata HDR-mediata integrazione singola copia in F0(Figura 2-figure supplemento 2) come sequenza confermata dall’analisi del locus (Figura 2-figure supplemento 3). Così, il 9,5% degli zigoti iniettati e dei sopravvissuti ha mostrato una precisa integrazione HDR-mediata in copia singola (il 15,8% degli zigoti iniettati ha espresso le PFP; il 60% di questi ha mostrato una precisa integrazione in copia singola; File supplementare 1). Per l’etichettatura gfp di actb il 46,5% degli zigoti iniettati ha espresso le GFP, il 35% dei quali (7 su 20 embrioni selezionati a caso che esprimono le GFP) ha mostrato una precisa integrazione HDR-mediata in una singola copia in F0, che rappresenta il 16,3% degli zigoti iniettati inizialmente(file supplementare 1).
Nel caso del dnmt1, il tasso di precisa integrazione HDR-mediata in una singola copia era ancora più alto, poiché la piena funzionalità del gene è necessaria per la progressione dello sviluppo e la sopravvivenza embrionale. Qui, sorprendentemente, tutti gli embrioni che esprimono GFP hanno mostrato la perfetta integrazione desiderata.
Abbiamo osservato la più alta efficienza del targeting HDR da 5′ dsDNA modificato per tutti i loci testati per i donatori 5’Biotin modificati (Figura 2-figuresupplement 2). Già nella generazione iniettata abbiamo rilevato e convalidato la fusione mediata da HDR dei donatori di dsDNA lunghi con il rispettivo locus(Figura 2-figure supplement 2). Pur continuando a dare origine ad un’alta percentuale di eventi HDR, i donatori di dsDNA modificati SpC3 5′ hanno anche portato a livelli elevati di bande aggiuntive indicative per l’integrazione di multimer di ordine superiore e di eventi NHEJ (Figura 2-figure supplement 2). Anche se sempre più popolare, nelle nostre mani l’uso di RNPs (proteina Cas9 e rispettivi sgRNA) non si è nemmeno avvicinato all’efficienza raggiunta dalla co-iniezione di Cas9 mRNA e il corrispondente sgRNA valutato dal gene targeting come descritto sopra.
L’integrazione precisa rilevata nella generazione iniettata è stata trasmessa con successo alla generazione successiva(Figura 3, Figura 2-figure supplement 3, Figura 3-figure supplement 1). Per il gfp-rx2, il 9% dei pesci provenienti dagli embrioni inizialmente iniettati ha trasmesso con successo il locus esattamente modificato alla generazione successiva (il 15,8% degli embrioni iniettati ha espresso le BPA; 4 su 7 BPA che trasmettevano i pesci fondatori trasmettevano anche la precisa integrazione singola della cassetta del donatore di BPA ). Per il gfp-rx1, il 3,9% dei pesci provenienti dagli embrioni inizialmente iniettati trasmetteva la precisa singola copia integrata alla generazione successiva (il 13,5% degli embrioni iniettati esprimeva le BPA; 2 dei 7 pesci fondatori che trasmettevano le BPA trasmettevano anche la precisa singola integrazione della cassetta del donatore gfp-rx1 ). Per il dnmt1, l’alto tasso di precisa integrazione della singola copia HDR-mediata in HDR osservato nella generazione iniettata è stato completamente mantenuto nella trasmissione alla generazione successiva a causa del requisito assoluto di una versione funzionale / funzionale tagged del locus. Come abbiamo trovato nel corso del nostro studio, l’integrazione precisa della cassetta del donatore gfp nel locus actb si traduce in letalità embrionale tardiva. Di conseguenza, non è stato possibile stabilire linee transgeniche stabili.
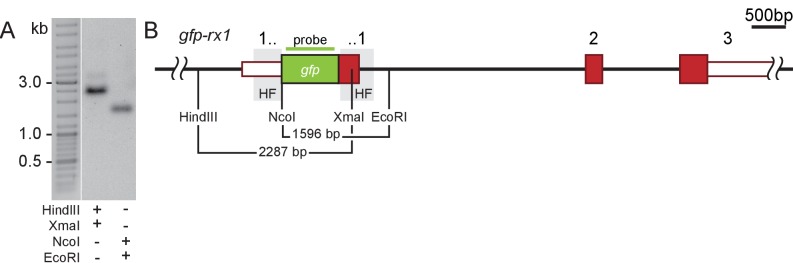
Figura 3-figure supplement 1.Figura 3—supplemento figura 1. L’integrazione a copia singola di un donatore di dsDNA lungo stabilisce il gene di fusione gfp-rx2 trasmesso in modo stabile; l’integrazione a copia singola trasmessa in modo stabile della cassetta del donatore gfp-rx1.(A) Gli embrioni omozigoti F2 mostrano l’espressione della proteina di fusione GFP-Rx2 nel modello del gene endogeno nella retina.(B) L’analisi Southern Blot di F2 embrioni gfp-rx2 rivela una singola banda per uno schema di digestione che taglia al di fuori della cassetta del donatore (BglII/HindIII) o all’interno della cassetta del donatore 5′ e nell’introne 2 (ScaI/HindIII) che indica una precisa integrazione del donatore a copia singola.(B” ) Rappresentazione schematica del locus modificato che indica i siti di restrizione e il dominio complementare alla sonda utilizzata in (B).(C) RT-PCR analisi RT-PCR su mRNA isolato da singoli embrioni omozigoti F3 indica la trascrizione di un singolo mRNA di fusione gfp-rx2 mRNA in confronto al più corto mRNA wild-type rx2 mRNA come schematicamente rappresentato in(C’ ).(A) L’analisi Southern Blot degli embrioni F2 gfp-rx1 rivela una singola banda per uno schema di digestione che taglia al di fuori della cassetta del donatore e all’interno della cassetta del donatore 3′ (HindIII/XmaI) o all’interno della cassetta del donatore 5′ e nell’introne 1 (NcoI/EcoRI) che indica l’integrazione precisa del donatore in una singola copia.(A’ ) Rappresentazione schematica del luogo modificato che indica i siti di restrizione e il dominio complementare alla sonda utilizzata in (A).
Per stimare il punto temporale degli eventi HDR negli embrioni iniettati, abbiamo studiato il tasso effettivo di mosaicismo nella linea germinale come riflesso dalla velocità di trasmissione della linea germinale. Per il gfp-rx2, questo tasso variava dallo 0,8% fino al 12,9%, indicando un evento HDR non prima dello stadio a 4 cellule (supponendo che si sia verificato solo un singolo evento per blastomero). Nel caso del gfp-rx1, l’HDR non si è verificato allo stadio a una cellula, ma piuttosto più tardi (stadio a 2-8 cellule come riflesso da tassi di trasmissione del germline del 23,9% e del 5,8% rispettivamente). Quindi, in entrambi i casi, le velocità di trasmissione del locus perfettamente marcato riflettono un livello di mosaicismo indicativo per un evento HDR tra lo stadio a 4 e quello a 32 cellule.
Abbiamo affrontato la natura dell’inserzione che si prevedeva fosse a copia singola mediante una combinazione di studi di PCR e di espressione. Abbiamo convalidato l’organizzazione genomica del gfp-rx2(Figura 3A) e gfp-rx1 knock-in in animali omozigoti F2 mediante sequenziamento genomico(Figura 2-figure supplement 3C) e Southern Blot (Southern, 2006) analisi (Figura 3B e B’ , Figura 3-figure supplement 1). In entrambi i casi, abbiamo rilevato una singola banda indicativa per un’integrazione HDR-mediata a copia singola, durante il sondaggio del DNA genomico digerito di F2 gfp-rx2+/+ e gfp-rx1+/+ rispettivamente pesciolini da banco. Abbiamo usato combinazioni di enzimi che rilasciano la regione gfp-rx2 includendo gli HF (BglII/HindIII, Figura 3B e B’ ) o tagliando nei 5′ HF (ScaI/HindIII, Figura 3B ,B’ ). Per il gfp-rx1 abbiamo rilasciato la regione di affiancamento 5′ inclusa la cassetta del donatore (HindIII/XmaI) o la rispettiva regione di affiancamento 3′ e la cassetta del donatore (NcoI/EcoRI) (Figura 3-figuresupplement 1).
Questa analisi ha validato in modo cruciale le previsioni basate sulla PCR e i risultati del sequenziamento.
Inoltre, l’analisi delle trascrizioni in embrioni gfp-rx2 omozigoti F3 ha scoperto esclusivamente una singola trascrizione di fusione gfp-rx2(Figura 3C e C’ ). Questa analisi molecolare ha confermato che la modificazione 5’Biotina del donatore dsDNA lungo ha promosso una precisa integrazione HDR-mediata a copia singola con alta efficienza.
Presi insieme la semplice modifica 5’Biotina ad entrambe le estremità dei donatori di dsDNA lungo da amplificazione PCR convenzionale qui presentato fornisce i mezzi per favorire HDR senza interferire con il macchinario di riparazione del DNA cellulare.

Figura 1-figure supplemento 1.Figura 1—figura 1. Modifica di 5′ estremità di frammenti di dsDNA lunghi impedisce la multimerization.rappresentazione schematica dei plasmidi donatori.(A) Rappresentazione schematica di amplificazione lungo dsDNA donatore cassetta PCR con primer universale (frecce nere) complementare alla spina dorsale vettore di clonazione al di fuori della cassetta donatore assemblato (ad esempio gfp con fianchi omologia). Moieties ingombranti come la Biotina alle estremità 5′ di entrambi i primer modificati (ottagono rosso) impediscono la multimerization/NHEJ del dsDNA, fornendo le condizioni ottimali per l’integrazione HDR-mediata di una singola copia dopo CRISPR/Cas9-introdotto DSB al locus target (forbici grigie). Rappresentazione dei primer del locus (Lf/Lr) e dei primer gfp interni (Gf/Gr) per la genotipizzazione PCR di eventi di integrazione gfp presunti mediati in HDR.(B) L’analisi del Southern blot rivela lo stato monomerico dei frammenti di dsDNA iniettati in vivo per la modifica 5′ con Biotina o Spacer C3. I dsDNA lunghi generati con i primer di controllo non modificati o i primer Amino-dT attaccati si multimerizzano come indicato da una scala ad alto peso molecolare apparente già entro due ore dopo l’iniezione (hpi). Nota: 5′ moieties non ha migliorato la stabilità del DNA iniettato.(A-D) Rappresentazione schematica in scala dei rispettivi locus target (A, rx2; B , rx1; C , actb ; D, dnmt1 ) con UTR (scatole bianche con contorni rossi) ed esoni (scatole rosse) evidenziati. Sono indicati i fianchi dell’omologia (HF, grigio), i siti target sgRNA (forbici bianche) e i rispettivi primer del locus (frecce nere, Lf, Lr, vedi file supplementare 2). Il plasmide del donatore assemblato rispettabile (A, B, C Golden GATEway(Kirchmaier et al., 2013) o D, clonazione convenzionale) che serviva da modello per l’amplificazione PCR della cassetta del donatore dsDNA gfp lungo non modificato/modificato è rappresentato qui sotto (FL, linker flessibile; sequenza dorsale in blu). Sono indicati i vettori di ingresso (EV) o i siti di enzimi di restrizione per la clonazione. La posizione dei primer che affiancano la cassetta del donatore è indicata, la modifica è evidenziata da un ottagono rosso.

Figura 1-figure supplement 1.Rappresentazione schematica dei plasmidi del donatore.(A-D) Rappresentazione schematica in scala dei rispettivi locus di destinazione (A, rx2; B , rx1; C , actb ; D, dnmt1 ) con UTR (scatole bianche con contorni rossi) ed esoni (scatole rosse) evidenziati. Sono indicati i fianchi dell’omologia (HF, grigio), i siti target sgRNA (forbici bianche) e i rispettivi primer del locus (frecce nere, Lf, Lr, vedi file supplementare 2). Il plasmide del donatore assemblato rispettabile (A, B, C Golden GATEway(Kirchmaier et al., 2013) o D, clonazione convenzionale) che serviva da modello per l’amplificazione PCR della cassetta del donatore dsDNA gfp lungo non modificato/modificato è rappresentato qui sotto (FL, linker flessibile; sequenza dorsale in blu). Sono indicati i vettori di ingresso (EV) o i siti di enzimi di restrizione per la clonazione. La posizione dei primer che affiancano la cassetta del donatore è indicata, la modifica è evidenziata da un ottagono rosso.

Figura 2-figure supplement 3.Figura 2—supplemento alla figura 3. La modifica di 5′ estremità di frammenti di dsDNA lunghi promuove l’integrazione HDR-mediata di una singola copia.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle GFP degli embrioni iniettati.5’La modifica della biotina dei donatori di dsDNA lunghi migliora fortemente l’integrazione HDR-mediata.la trasmissione germline stabile della singola copia HDR-mediata di una precisa integrazione gfp.(A) GFP espressione nel rispettivo dominio di espressione dopo HDR-mediata integrazione HDR-mediata di dsDNA modificato gfp donatore cassette in rx2, rx1, actb e dnmt1 ORF nella generazione iniettata.(B) Singolo embrione PCR genotipizzazione PCR evidenzia efficiente HDR-mediata HDR-mediata integrazione singola copia di 5’Biotina modificati donatori dsDNA lungo, ma non cassette donatore non modificato. Locus PCR rivela la dimensione della banda indicativa dell’integrazione gfp a copia singola (asterisco) oltre agli alleli senza integrazione gfp (punta di freccia aperta). Amplificazione del donatore gfp (freccia bianca) per il controllo.Embrioni iniettati con Biotina non modificata o 5’Biotina, Amino-dT o Spacer C3 modificato lungo dsDNA gfp-rx2 cassette donatore sono stati segnati per la sopravvivenza ad un dpi, e per l’espressione GFP a due dpi. n, numero totale di embrioni iniettati.10.7554/eLife.39468.007Figure 2-figure supplement 1-source data 1.Quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.(A) rx2 locus PCR genotipizzazione PCR di singoli embrioni positivi GFP-Rx2 iniettati con cassette donatrici di gfp non modificate o 5’Biotina o Spacer C3 modificato long dsDNA gfp (asterisco verde, integrazione HDR-mediata a copia singola di gfp, 2547 bp, punta di freccia aperta, allele rx2 senza integrazione gfp, 1719 bp;). Barra orizzontale, singolo embrione; L, rx2 locus PCR con rx2 Lf/rx2 Lr; G, gfp PCR interna per il controllo con Gf/Gr.(B) Sintesi qualitativa dello spettro di banda (integrazione HDR-mediata in HDR a copia singola di gfp, allele rx2 senza integrazione gfp, altro) risultante dalla genotipizzazione PCR in(A). n, numero di GFP-Rx2 genotipizzati che esprimono gli embrioni.(A) Rappresentazione schematica in scala del locus gfp-rx2 con UTR (scatole bianche con contorni rossi), esoni (scatole rosse) e fianchi di omologia (HF, grigio) evidenziati.(B, C) I singoli GFP-Rx2 che esprimono gli embrioni (F0, B; F1, C) sono stati genotipizzati usando i primer rx2 Lf/rx2 Lr (frecce nere) e sequenziati con i primer gfpf e gfpr (frecce verdi).
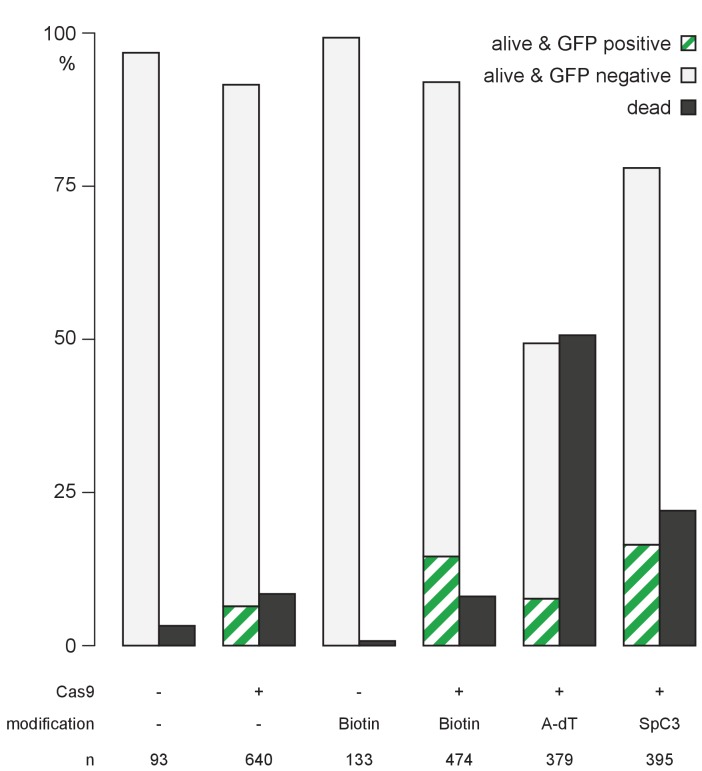
Figura 2-figure supplement 1-source data 1.Quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.Gli embrioni iniettati con biotina non modificata o 5’Biotina, Amino-dT o Spacer C3 modificato long dsDNA gfp-rx2 sono stati valutati per la sopravvivenza ad un dpi, e per l’espressione delle GFP a due dpi. n, numero totale di embrioni iniettati.10.7554/eLife.39468.007Figure 2-figure supplement 1-source data 1.Quantificazione della sopravvivenza e dell’espressione delle PFL degli embrioni iniettati.
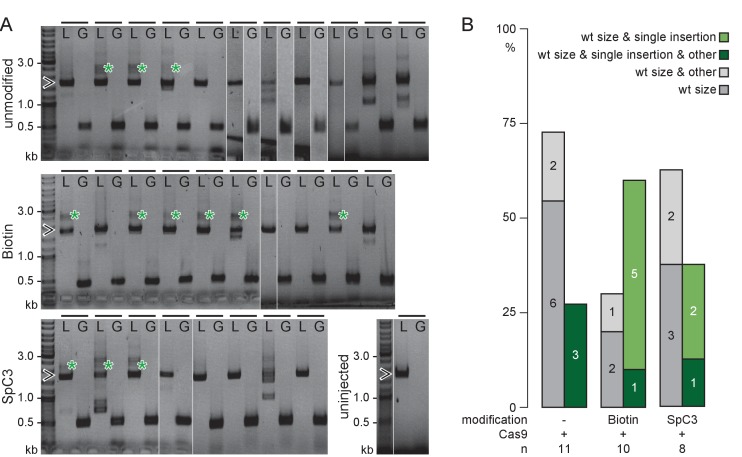
Figura 2-figure supplement 2.5’La modifica della biotina di donatori di dsDNA lunghi migliora fortemente l’integrazione mediata dall’HDR.(A) rx2 locus PCR genotipizzazione PCR di singoli embrioni positivi GFP-Rx2 iniettati con non modificato o 5’Biotina o Spacer C3 modificato lungo dsDNA gfp cassette donatore modificato (asterisco verde, singola copia HDR-mediata integrazione HDR-mediata di gfp, 2547 bp, punta di freccia aperta, allele rx2 senza integrazione gfp, 1719 bp;). Barra orizzontale, singolo embrione; L, rx2 locus PCR con rx2 Lf/rx2 Lr; G, gfp PCR interna per il controllo con Gf/Gr.(B) Sintesi qualitativa dello spettro di banda (integrazione HDR-mediata in HDR a copia singola di gfp, allele rx2 senza integrazione gfp, altro) risultante dalla genotipizzazione PCR in(A). n, numero di GFP-Rx2 genotipizzati che esprimono gli embrioni.

Figura 2-figure supplement 3.Figura 2—supplemento di figura 3. Trasmissione stabile del germline della singola copia HDR-mediata da una precisa integrazione gfp.(A) Rappresentazione schematica in scala del locus gfp-rx2 con UTR (scatole bianche con contorni rossi), esoni (scatole rosse) e fianchi di omologia (HF, grigio) evidenziati.(B, C) I singoli GFP-Rx2 che esprimono gli embrioni (F0, B; F1, C) sono stati genotipizzati usando i primer rx2 Lf/rx2 Lr (frecce nere) e sequenziati con i primer gfpf e gfpr (frecce verdi).

Figura 3-figure supplement 1.Figura 3—supplemento figura 1. L’integrazione a copia singola del donatore di dsDNA lungo stabilisce il gene di fusione gfp-rx2 a trasmissione stabile.l’integrazione a copia singola della cassetta del donatore gfp-rx1 a trasmissione stabile.(A) Gli embrioni omozigoti F2 mostrano l’espressione della proteina di fusione GFP-Rx2 nel modello del gene endogeno nella retina.(B) L’analisi Southern Blot di F2 embrioni gfp-rx2 rivela una singola banda per uno schema di digestione che taglia al di fuori della cassetta del donatore (BglII/HindIII) o all’interno della cassetta del donatore 5′ e nell’introne 2 (ScaI/HindIII) che indica una precisa integrazione del donatore a copia singola.(B” ) Rappresentazione schematica del locus modificato che indica i siti di restrizione e il dominio complementare alla sonda utilizzata in (B).(C) RT-PCR analisi RT-PCR su mRNA isolato da singoli embrioni omozigoti F3 indica la trascrizione di un singolo mRNA di fusione gfp-rx2 mRNA in confronto al più corto mRNA wild-type rx2 mRNA come schematicamente rappresentato in(C’ ).(A) L’analisi Southern Blot degli embrioni F2 gfp-rx1 rivela una singola banda per uno schema di digestione che taglia al di fuori della cassetta del donatore e all’interno della cassetta del donatore 3′ (HindIII/XmaI) o all’interno della cassetta del donatore 5′ e nell’introne 1 (NcoI/EcoRI) che indica l’integrazione precisa del donatore in una singola copia.(A’ ) Rappresentazione schematica del luogo modificato che indica i siti di restrizione e il dominio complementare alla sonda utilizzata in (A).

Figura 3-figure supplement 1.Integrazione della singola copia della cassetta del donatore gfp-rx1 trasmessa in modo stabile.(A) L’analisi Southern Blot di F2 embrioni gfp-rx1 rivela una singola banda per uno schema di digestione di taglio al di fuori della cassetta del donatore e all’interno della cassetta del donatore 3′ (HindIII/XmaI) o all’interno della cassetta del donatore 5′ e nell’introne 1 (NcoI/EcoRI) che indica una precisa integrazione del donatore a copia singola.(A’ ) Rappresentazione schematica del luogo modificato che indica i siti di restrizione e il dominio complementare alla sonda utilizzata in (A).
Discussione
Per una ricombinazione efficiente è essenziale la qualità dei primer modificati, cioè la frazione di primer effettivamente etichettati con la biotina e quindi la qualità del prodotto 5′ long ds DNA PCR protetto. L’integrazione di frammenti di 5′ PCR modificati di dimensioni maggiori di 2 kb non costituisce in linea di principio un problema. Abbiamo già integrato con successo cassette fino a 8,6 kb (dati non mostrati) tramite HDR mediante linearizzazione in vivo del plasmide del donatore(Stemmer et al., 2015). Nell’approccio qui presentato, è probabile che la qualità del prodotto di PCR protetto alla fine si riduca con una lunghezza maggiore, in parte a causa dell’intaccatura (UV-) nel processo di estrazione e di purificazione. Inoltre, la rapida convalida mediante PCR con locus spanning PCR ha dimensioni limitate, ma alla fine l’analisi Southern blot risolverà la questione di una singola copia, una perfetta integrazione. Indipendentemente dalle dimensioni della cassetta del donatore, i cicli di PCR utilizzati per sondare per la riuscita della singola integrazione tramite HDR non devono superare un totale di 30 per evitare artefatti di fusione in vitro-PCR fuorvianti (dati propri e[Won e Dawid, 2017]).
Come già accennato in precedenza, l’HDR si verifica preferibilmente durante la fase tardiva S/G2 del ciclo cellulare(Heyer et al., 2010; Hustedt e Durocher, 2017). Le diverse efficienze dell’editing del genoma tramite HDR potrebbero essere dovute a differenze specie-specifiche, in particolare della fase S/G2 del ciclo cellulare che probabilmente ha un impatto cruciale sul tasso di HDR. Come ampiamente riportato, non c’è una fase G2 apparente prima della metà della transizione della blastula in anfibia(Xenopus) e nei pesci(Danio rerio) con la marcata eccezione della prima scissione, dove è stata riportata una fase G2(Kimelman, 2014). Le velocità di trasmissione del germe ottenute per la marcatura di Rx1 e Rx2 hanno indicato eventi HDR dopo la fase a due cellule, prevalentemente nella fase a 4 cellule e nelle fasi successive. Il ciclo più lento del medaka rispetto al pesce zebra risulta in un’estensione della fase G2 nel primo ciclo cellulare di oltre il 150% (ciclo totale del pesce zebra: 45 min[Kimmel et al., 1995], medaka: 65 min[Iwamatsu, 2004]). Supponendo un tasso comparabile di sintesi del DNA tra le due specie, c’è più del doppio del tempo per la fase S e la fase M per replicare un genoma che comprende circa un terzo della dimensione del pesce zebra. Questa lunghezza estesa del ciclo cellulare è particolarmente evidente anche nei cicli successivi in medaka (15 min di pesce zebra[Kimmel et al., 1995], 40 min di medaka[Iwamatsu, 2004]). È interessante notare che la fosforilazione dell’RNA polimerasi, un prerequisito per l’espressione delle prime trascrizioni zigotiche, è stata segnalata per iniziare allo stadio a 64 cellule in medaka(Kraeussling et al., 2011) e le divisioni asincrone, un segno distintivo di MBT, sono osservate già al passaggio dallo stadio a 16 a quello a 32 cellule(Kraeussling et al., 2011), suggerendo differenze specie-specifiche nell’insorgenza della trascrizione zigotica e di conseguenza l’allungamento del ciclo cellulare.
Il nostro approccio facilita il rilevamento altamente efficiente degli eventi HDR mediante la marcatura GFP anche in geni non essenziali. Oltre che nei donatori di dsDNA non modificati, l’espressione locus-specifica delle GFP è un eccellente predittore per una precisa integrazione HDR-mediata da una singola copia già nella generazione iniettata. Ciò consente una facile selezione e consente di ottenere tassi di HDR negli embrioni positivi alle GFP fino al 60% nella generazione iniettata. Questo tasso può anche essere più alto in loci essenziali, come il dnmt1, dove la piena funzionalità del gene (modificato) in esame è necessaria per la sopravvivenza delle cellule, dei tessuti, degli organi o degli organismi colpiti, fornendo così i mezzi per un’efficace selezione positiva per singole integrazioni completamente funzionali. È interessante notare che circa 400 bp che affiancano il sito di inserimento in direzione 5′ e 3′ sono sufficienti per HDR. L’uso di primer universali modificati per i due vettori di template dei donatori utilizzati ha permesso un’applicazione flessibile della procedura e ha garantito un’alta qualità dei donatori di dsDNA lunghi modificati da 5′ generati dalla PCR. Sorprendentemente, le regioni non omologiche introdotte con il primer nella periferia dei donatori di dsDNA lunghi non hanno avuto un impatto negativo sull’efficienza dell’HDR.
Presi nel loro insieme, la semplicità e l’alta riproducibilità della nostra analisi di proof-of-concept evidenziano che la protezione presentata di entrambe le estremità 5′ dei donatori di dsDNA lunghi previene la multimetrizzazione e promuove l’inserimento/sostituzione precisa degli elementi del DNA, facilitando così gli studi funzionali nella ricerca di base così come gli interventi terapeutici.
Materiali e metodi
| Tipo di reagente (specie) o risorsa | Designazione | Fonte o riferimento | Identificatori | Ulteriori informazioni |
|---|---|---|---|---|
| Ceppo, fondo di ceppo(Oryzias latipes) | Cabina | altro | medaka Popolazione selvaggia del sud | |
| Ceppo, fondo di ceppo(Oryzias latipes) | rx2-gfp | questo documento | ||
| Ceppo, fondo di ceppo(Oryzias latipes) | rx1-gfp | questo documento | ||
| Ceppo, fondo di ceppo(Oryzias latipes) | actb-gfp | questo documento | ||
| Ceppo, fondo di ceppo(Oryzias latipes) | dnmt1-gfp | questo documento | ||
| Reagente DNA ricombinante | cassetta donatorerx2-gfp | questo documento | ||
| Reagente DNA ricombinante | cassetta donatorerx1-gfp | questo documento | ||
| Reagente DNA ricombinante | cassetta donatoreactb-gfp | questo documento | ||
| Reagente DNA ricombinante | cassetta donatorednmt1-gfp | questo documento | ||
| Reagente a base di sequenza | rx2 5’HF f | questo documento | con il sito di restrizione BamHI: GCCGGATCCAAGCATGTCAAAACGTAGAGAAGCG | |
| Reagente a base di sequenza | rx2 5’HF r | questo documento | con il sito di restrizione KpnI: GCCGGGGTACCCATTTTTTTTTTTTTTTTTGCC | |
| Reagente a base di sequenza | rx2 3’HF f | questo documento | con il sito di restrizione BamHI: GCCGGATCCCATTTTTTTTTCAATGGAC ACGCTTTTGGATGGATGGTGGACGAT | |
| Reagente a base di sequenza | rx2 3’HF r | questo documento | con il sito di restrizione KnpI: GCCGGTACCTGGTGGACTGGAAGTTATTTATTT | |
| Reagente a base di sequenza | rx2 sgRNA f | questo documento | i nucleotidi sostituiti per facilitare la trascrizione in vitro del T7 degli oligonucleotidi sgRNA sono indicati con lettere minuscole TAgGGCATTTGTTCAATGGATACCC | |
| Reagente a base di sequenza | rx2 sgRNA r | questo documento | AAACGGGTATTTATTGATTGACAAATG | |
| Reagente a base di sequenza | rx2 Lf/5’UTRf | questo documento | TGCATGTTTTTTTTGGCAACG | |
| Reagente a base di sequenza | rx2 Lr | questo documento | AGGGACCATACCTGACCTGACCCTC | |
| Reagente a base di sequenza | actb 5’HF f | questo documento | con il sito di restrizione BamHI: GGGGGATCCCAGAGCAACGACTTCGCCACAAA | |
| Reagente a base di sequenza | actb 5’HF r | questo documento | con il sito di restrizione KnpI: GGGGGTACCGGCAATGTCATCATCATCATCATCCATGGC | |
| Reagente a base di sequenza | actb 3’HF f | questo documento | con il sito di restrizione BamHI: GGGGGATCCGACGACGACGATATATAGATAGGGATAGGGATTTG CACTGGTTGTTGACAACGGATTTG | |
| Reagente a base di sequenza | actb 3’HF r | questo documento | con il sito di restrizione KnpI: GGGGGTACCCAGGGGGCAATTCAGCTCAGCTCA | |
| Reagente a base di sequenza | actb sgRNA f | questo documento | TAGGATGATGATGACATTGCCGCCAC | |
| Reagente a base di sequenza | actb sgRNA r | questo documento | AAACGTGGCGGGGCAATGTCATCAT | |
| Reagente a base di sequenza | actb Lf | questo documento | GTCCGGAGTTGAGGGTGTGTGTGTG | |
| Reagente a base di sequenza | actb Lr | questo documento | CATGTGCTCCACTGTGAGGT | |
| Reagente a base di sequenza | dnmt1 5’HF f | questo documento | con il sito di restrizione di SalI: AATTTGTCGGACGCTTTTTGA CAGTTAACCTACTACACG | |
| Reagente a base di sequenza | dnmt1 5’HF r | questo documento | con il sito di restrizione AgeI: AATTTACCGGTCGTAACTGCA AACTAAAAAAAATAAAAC | |
| Reagente a base di sequenza | dnmt1 3’HF f | questo documento | con il sito di restrizione SpeI: AATTTACTACTAGTATGATGCCATCCATCCATCCATCCAGAA CGTCCTTATTTATCTCTACCAGACGATGATG TCAGAAAAAGGTAC | |
| Reagente a base di sequenza | dnmt1 3’HF r | questo documento | con il sito di restrizione NotI: AATTTGGCGGCCGGCCCCTACACATA TTGTGTGTGTGATAC | |
| Reagente a base di sequenza | mgfpf | questo documento | con il sito di restrizione AgeI: AATTTACCGGGGTACTAGTACCATG AGTAAAGGAGAAGAACTTTTTTCAC | |
| Reagente a base di sequenza | mgfpr | questo documento | con il sito di restrizione SpeI: AATTTACTAGTCGCCGGCCGGGGCCCCACTTTTTOO CCACCCCCCCCCCATCCGATCCGATCCGATCCGCCACCAAC CCCCCTCCACCTCCACCCCCCATTTTTTTTTTTTTTTTOO | |
| Reagente a base di sequenza | dnmt1 sgRNA f | questo documento | i nucleotidi sostituiti per facilitare la trascrizione in vitro del T7 degli oligonucleotidi sgRNA sono indicati con lettere minuscole TAgGGACATCGTCTGGCAAAGAC | |
| Reagente a base di sequenza | dnmt1 sgRNA r | questo documento | AAACGTCTTTTTGCCCCAGACGATGT | |
| Reagente a base di sequenza | dnmt1 Lf | questo documento | CTCAATGTAAACTTTTTTTTTIIII DDDIII CTCAATTTTIII DDDIII CTCAATTTTIII DDDII CTCAATTTTIIII | |
| Reagente a base di sequenza | dnmt1 Lr | questo documento | TTGCATGATATATTTTTTTTIIII DDDIII TTGCATTTTIII DDDIII TTGATTTTII DDDII TTGATTTTIII | |
| Reagente a base di sequenza | rx1 5’HF f | questo documento | con il sito di restrizione BamHI: GCCGGATCCGATCCCATCCGAAAGG TAAGGACTGACTGCAAACC | |
| Reagente a base di sequenza | rx1 5’HF r | questo documento | con il sito di restrizione KpnI: GCCGGGGTACCCATGAGAGAGCCG TCTGGGCTCTCTGACC | |
| Reagente a base di sequenza | rx1 3’HF f | questo documento | con il sito di restrizione BamHI: GGCGGATCCCATTTATTTATCAC TCGATACCATGAGCA | |
| Reagente a base di sequenza | rx1 3’HF r | questo documento | con il sito di restrizione KpnI: GGCGGGGTACCTTTTTTTTTTTA AGAACATTTTTTTTTTOO | |
| Reagente a base di sequenza | rx1 sgRNA1 f | questo documento | i nucleotidi sostituiti per facilitare la trascrizione in vitro del T7 degli oligonucleotidi di sgRNA sono indicati con lettere minuscole TAggAAATGCATGAGGAGCGTTO | |
| Reagente a base di sequenza | rx1 sgRNA1 r | questo documento | AAACAGACGATTTTTATTTTOO | |
| Reagente a base di sequenza | rx1 sgRNA2 f | questo documento | nucleotidi sostituiti per facilitare la trascrizione in vitro del T7 degli oligonucleotidi sgRNA sono indicati in lettere minuscole TAggCTCTCATGCATTTATCAC | |
| Reagente a base di sequenza | rx1 sgRNA2 r | questo documento | AAACGTGTGATAAATGCATGCATGAGAGAG | |
| Reagente a base di sequenza | rx1 Lf | questo documento | CTTTGCTGTTTTGGAGAATTGCACC | |
| Reagente a base di sequenza | rx1 Lr | questo documento | GAGACCGAACGATGACAATAACAC | |
| Reagente a base di sequenza | pDest f (controllo) | questo documento | CGAGCCGCCAGCCAGTCAGTCAGTGAG | |
| Reagente a base di sequenza | pDest r (controllo) | questo documento | CATGTAATACGACTCACTATAG | |
| Reagente a base di sequenza | pDest f mod | questo documento | Gli asterischi indicano legami di fosforotioato, “5’moiety” era 5’Biotina, Amino-dT o Spacer C3. 5’moiety-C*G*G*A*G*G*C*GCAGCGGGGTCAGTGAGTGAG | |
| Reagente a base di sequenza | pDest r mod | questo documento | Gli asterischi indicano legami di fosforotioato, “5’moiety” era 5’Biotina, Amino-dT o Spacer C3. 5’moiety-C*A*T*T*G*T*T*AATACGACTCACTATAG | |
| Reagente a base di sequenza | pCS2 f | questo documento | CCATTCAGGCTGCGGCAACTG | |
| Reagente a base di sequenza | pCS2 r | questo documento | CACACACAGGAAACAGCTATGAC | |
| Reagente a base di sequenza | pCS2 f mod | questo documento | Gli asterischi indicano legami di fosforotioato, “5’moiety” era 5’Biotina, Amino-dT o Spacer C3. 5’moiety-C*C*C*A*T*T*T*CAGGGCTG CGCAACTG | |
| Reagente a base di sequenza | pCS2 r mod | questo documento | Gli asterischi indicano legami di fosforotioato, “5’moiety” era 5’Biotina, Amino-dT o Spacer C3. 5’moiety-C*A*C*C*A*C*AGGAAACAGAGCTATGAC | |
| Reagente a base di sequenza | Gf | questo documento | CTGCACCACCCCCCHHHIIOO DDII CTGCACCACCCCHHHIIOO DDII CTGCACCACCCHHHIIOO | |
| Reagente a base di sequenza | Gr | questo documento | CTCAGGTAGTGTGGTTTTGGGG | |
| Reagente a base di sequenza | gfpf | questo documento | GCTCGACCAGGATGGGCA | |
| Reagente a base di sequenza | gfpr | questo documento | CTGAGCAAAGACCCCAACGAGA AGCGCGGATCACATG | |
| Reagente a base di sequenza | sonda gfp f | questo documento | GTGAGCAAGGGCGAGGAGGAGCT | |
| Reagente a base di sequenza | sonda gfp r | questo documento | CTTGGTACAGCTCGTCCATG |
Manutenzione dei pesci
Tutti i pesci sono mantenuti in stock chiusi all’Università di Heidelberg. L’allevamento di Medaka(Oryzias latipes) (autorizzazione numero 35-9185.64/BH Wittbrodt) e gli esperimenti (autorizzazione numero 35-9185.81/G-145/15 Wittbrodt) sono stati eseguiti secondo le norme locali sul benessere degli animali (Tierschutzgesetz §11, Abs. 1, Nr. 1) e in conformità alle direttive dell’Unione Europea sul benessere degli animali (Bert et al.,2016). L’impianto ittico è sotto la supervisione del rappresentante locale dell’agenzia per il benessere degli animali. Gli embrioni di medaka del ceppo wild-type Cab sono stati utilizzati nelle fasi precedenti la schiusa. Medaka è stato allevato e mantenuto come descritto in precedenza(Koster et al., 1997).
Plasmidi donatori
I plasmidiRx2 e actb template plasmidi per l’amplificazione a cassette per donatori gfp sono descritti in Stemmer et al. (2015) e sono stati generati dall’assemblaggio GoldenGATE nel vettore di destinazione pGGGDestSC-ATG (addgene #49322) secondo Kirchmaier et al. (2013). Vedere il file supplementare 2 per i primer utilizzati per amplificare i rispettivi fianchi di omologia. Il plasmide dnmt1 gfp plasmide è stato clonato con i fianchi di omologia (5′ HF 402 bp, primer dnmt1 5’HF f/dnmt1 5’HF r; 3′ HF 405 bp, dnmt1 3’HF f/dnmt1 3’HF r) che sono stati amplificati con PCR con polimerasi Q5 (New England Biolabs, 30 cicli) da DNA genomico medaka wild-type. mgfp-flexible linker è stato amplificato con primer mgfpf/mgfpr. Il rispettivo enzima di restrizione è stato utilizzato per digerire gli ampliconi (5’HF: SalI HF (New England Biolabs), AgeI HF (New England Biolabs); mgfp-flexible linker: AgeI HF (New England Biolabs), SpeI HF (New England Biolabs); 3’HF: SpeI HF (New England Biolabs), NotI HF (New England Biolabs)) seguito da purificazione in gel (Analytik Jena) e legatura in pCS2+ (Ruppet al., 1994) (digerito con SalI HF (New England Biolabs), NotI HF (New England Biolabs)). Il plasmide rx1 gfp è stato clonato con fianchi di omologia (5’HF 430 bp, primer rx1 5’HF f/rx1 5’HF r; 3′ HF 508 bp, rx1 3’HF f/rx1 3’HF r) che sono stati amplificati con PCR con polimerasi Q5 (New England Biolabs, 30 cicli) da DNA genomico medaka wild-type. Tutti i primer sono stati ottenuti da Eurofins Genomics.
Amplificazione del donatore
Abbiamo progettato primer universali che corrispondono alla spina dorsale pGGGDestSC-ATG(Kirchmaier et al., 2013) (addgene #49322) o pCS2+(Rupp et al., 1994) che comprende gli inserti assemblati (cioè la cassetta del donatore gfp ). Primer di controllo non modificati (pDest f, pDest r, pCS2 f, pCS2 r) sono stati ordinati da Eurofins Genomics. I primer modificati ottenuti da Sigma-Aldrich (pDest f mod, pDest r mod, pCS2 f mod, pCS2 r mod) sono costituiti dalle stesse sequenze con legami fosforotiodici nei primi cinque nucleotidi e 5’moiety extension: 5’Biotina, 5’Amino-dT o 5’Spacer C3.
Le cassette donatore dsDNA sono state amplificate mediante PCR utilizzando 1x Q5 tampone di reazione, 200 µM dNTPs, 200 µM primer forward e reverse e 0,6 U/µl Q5 polimerasi (New England Biolabs). Condizioni utilizzate: denaturazione iniziale a 98°C 30 s, seguita da 35 cicli di: denaturazione a 98°C 10 s, ricottura a 62°C 20 s ed estensione a 72°C 30 s per kb e una fase finale di estensione di 2 min a 72°C. La reazione PCR è stata trattata con 20 unità di DpnI (New England Biolabs) per rimuovere qualsiasi template di plasmide dopo la purificazione del gel con il QIAquick Gel Extraction Kit (Qiagen, 28706) e l’eluizione con 20 µl di acqua priva di nucleasi.
La cassetta LacZ del pGGGDestSC-ATG(Kirchmaier et al., 2013) (addgene #49322) che serviva come frammento dsDNA marcato DIG per testare la multimerizzazione in vivo è stata amplificata tramite Q5-PCR come sopra utilizzando una miscela di 200 µM dATP, dCTP, dGTP, 170 µM dTTP e 30 µM DIG-dUTP e purificata come descritto in dettaglio.
selezione del sito target sgRNA
Gli sgRNADnmt1 sono stati progettati con CCTop come descritto in Stemmer et al. (2015). gli sgRNA per rx2 e actb erano gli stessi di Stemmer et al. (2015). Sono stati utilizzati i seguenti siti target vicini ai codici di partenza traslazionali (PAM tra parentesi): rx2 (GCATTTGTGTCAATGGGATACCC[TGGG]), actb (GGATGATGACATTGCCGCCAC[TGG]), dnmt1 (TGACATCGTCTGGCAAAGAC[AGG]) e rx1 (AAATGCATGAGAGCGGTCT[GGG] e CTCTCATGCATTTATCAC[TGGG]). La clonazione di modelli di sgRNA e la trascrizione in vitro è stata eseguita come descritto in Stemmer et al. (2015).
Trascrizione in vitro di mRNA
Ilplasmide pCS2 +Cas9 è stato linearizzato utilizzando NotI e l’mRNA è stato trascritto in vitro utilizzando il kit mMessage_mMachine SP6 (ThermoFisher Scientific, AM1340).
Microiniezione e screening
Gli zigoti Medaka sono stati iniettati con 10 ng di donatori con marchio DIG e sono stati autorizzati a svilupparsi fino a 2, 4 e 6 ore dopo l’iniezione. Per gli esperimenti CRISPR/Cas9, agli zigoti medaka sono stati iniettati 5 ng/µl di donatori di dsDNA lunghi non modificati e modificati insieme a 150 ng/µl di mRNA Cas9 e 15-30 ng/µl di sgRNA specifici del gene. Gli embrioni iniettati sono stati mantenuti a 28 ° C in mezzo di allevamento embrionale (ERM, 17 mM NaCl, 40 mM KCl, 0,27 mM CaCl2,0,66 mM MgSO4, 17 mM Hepes). Un giorno dopo l’iniezione (dpi) gli embrioni sono stati sottoposti a screening per la sopravvivenza, l’espressione GFP è stata segnata a due dpi.
Macchia meridionale
Per verificare la multimeritizzazione dei donatori non modificati e modificati, abbiamo utilizzato un approccio Southern Blot modificato. In breve, gli embrioni sono stati iniettati con donatori etichettati DIG che sono stati amplificati con PCR da pGGDestSC-ATG (addgene # 49322) utilizzando primer pDest f/pDest r( cassettaLacZ ) che non ospitano 5′ moiety o uno dei seguenti: 5’Biotina, Amino-dT o Spacer C3. 2, 4 e 6 ore dopo l’iniezione, 30 embrioni sono stati lisati in TEN tampone più proteinasi K (10 mM Tris pH 8, 1 mM EDTA, 100 mM NaCl, 1 mg / ml proteinasi K) a 60 ° C durante la notte. DNA è stato precipitato etanolo dopo la rimozione di lipidi e proteine per estrazione fenolo-cloroformio. DNA totale è stato risospeso in tampone TE (10 mM Tris HCl Tris pH 8,0, 1 mM EDTA pH 8,0). 200 ng di ogni campione è stato eseguito su un gel di agarosio 0,8%. Come controllo, sono stati caricati 100 pg di prodotto PCR del donatore non iniettato. Il gel di agarosio è stato trasferito su una membrana di nylon durante la notte utilizzando 10x SSC (1,5 M NaCl, 0,15 M C6H5Na3O7) come soluzione di trasferimento. La membrana reticolata è stato direttamente bloccato in 1% w / v bloccando reagente (Roche) in 1x DIG1 soluzione (0,1 M di acido maleico, 0,15 M NaCl, pH 7,5) e il DNA etichettato è stato rilevato utilizzando CDP star (Roche) seguendo le istruzioni del produttore.
Al fine di verificare la presenza di inserimenti di numero di copia nelle linee transgeniche gfp-rx2 e gfp-rx1, il DNA genomico è stato isolato come descritto in precedenza da embrioni F2 che esprimono GFP. 10 µg di DNA genomico digerito sono stati caricati per corsia su un gel di agarosio allo 0,8% e dimensioni frazionate per elettroforesi. Il gel è stato depurato in 0,25 N HCl per 30 minuti a temperatura ambiente, risciacquato conH2O, denaturato in 0,5 N NaOH, 1,5 M soluzione di NaCl per 30 minuti a temperatura ambiente e neutralizzato in 0,5 M Tris HCl, 1,5 M NaCl, pH 7,2 prima di essere trasferito durante la notte a temperatura ambiente su una membrana Hybond (Amersham). La membrana è stata lavata con 50 mM NaPi per 5 minuti a temperatura ambiente, poi reticolata e pre-ibridizzata in tampone di ibridazione Church (0,5 M NaPi, 7% SDS, 1 mM EDTA pH 8,0) a 65°C per almeno 30 minuti. La sonda è stata sintetizzata dal plasmide donatore con primer gfp sonda gfp f e gfp sonda r utilizzando il PCR DIG Probe Synthesis Kit (Roche, 11636090910) e il seguente protocollo PCR: denaturazione iniziale a 95 ° C per 2 min, 35 cicli di 95 ° C 30 s, 60 ° C 30 s, 72 ° C 40 s e l’estensione finale a 72 ° C 7 min. La sonda è stata bollita in tampone di ibridazione per 10 min a 95°C e la membrana è stata ibridata durante la notte a 65°C. La membrana è stata lavata con tampone di lavaggio della chiesa preriscaldato (65°C) (40 mM NaPi, 1% SDS) a 65°C per 10 min, poi a temperatura ambiente per 10 min e con 1x DIG1% e 0,3% Tween per 5 min a temperatura ambiente. La membrana è stata bloccata in 1% w / v bloccante reagente (Roche) in 1x DIG1 soluzione a temperatura ambiente per almeno 30 min. La membrana è stata incubata con 1:10.000 anti-igoxigenina-AP Fab frammenti (Roche) per 30 minuti a temperatura ambiente in 1% w / v bloccante reagente (Roche) in 1x DIG1 soluzione. Due fasi di lavaggio con 1x DIG1% e 0,3% Tween sono stati eseguiti per 20 minuti a temperatura ambiente, seguita da una fase di lavaggio 5 minuti in 1x DIG3 (0,1 M Tris pH 9,5, 0,1 M NaCl) a temperatura ambiente. La rilevazione è stata effettuata utilizzando 6 µl/ml CDP star (Roche).
Genotipizzazione
Singola iniezione di embrioni GFP positivi sono stati lisati in tampone di estrazione del DNA (0,4 M Tris / HCl pH 8,0, 0,15 M NaCl, 0,1% SDS, 5 mM EDTA pH 8,0, 1 mg / ml proteinasi K) a 60 ° C durante la notte. Proteinasi K è stato inattivato a 95 ° C per 10 minuti e la soluzione è stata diluita 1:2 con H2O. Genotipizzazione è stata eseguita in 1x tampone di reazione Q5, 200 µM dNTPs, 200 µM primer avanti e indietro e 0,012 U / µl Q5 polimerasi Q5 e 2 µl di campione di DNA diluito e dei rispettivi primer locus. Le condizioni erano: 98°C 30 s, 30 cicli di 98°C 10 s, ricottura per 20 s e 72°C 30 s per kb (il tempo di estensione utilizzato permetterebbe di rilevare potenziali eventi NHEJ su entrambe le estremità del donatore) (rx2 Lf/rx2 Lr: Ricottura a 68°C, tempo di estensione di 90 s; rx1 Lf/rx1 Lr: Ricottura a 66°C, tempo di estensione di 90 s; actb Lf/actb Lr: 66°C ricottura, tempo di estensione 84 s; dnmt1 Lf/dnmt1 Lr: Ricottura a 65°C, tempo di estensione di 90 s) e un’estensione finale di 2 min a 72°C. I prodotti PCR sono stati analizzati su un gel di agarosio all’1%.
Diagnostica GFP PCR qui: Ricottura a 63°C, 500 bp, estensione 15 s
RT-PCR
L’RNA totale è stato isolato da 60 embrioni omozigoti (stadio 32) mediante lisi in TRIzol (Ambion) ed estrazione di cloroformio secondo il protocollo del produttore. RNA è stato precipitato utilizzando isopropanolo e risospeso inH2O. cDNA è stato trascritto al contrario con Revert Aid Kit (Thermo Fisher Scientific) dopo la digestione del DNAse e l’inattivazione seguendo le istruzioni del produttore. La PCR è stata eseguita utilizzando 5’UTRf, 3’UTRr e Q5 polimerasi (New England Biolabs): 98°C 30 s, 35 cicli di 98°C 10 s, ricottura 65°C per 20 s e 72°C 210 s e un’estensione finale di 2 min a 72°C. I prodotti PCR sono stati analizzati su un gel di agarosio all’1,5%.
Sequenziamento
I plasmidi e i frammenti di PCR sono stati sequenziati con i primer indicati da un servizio commerciale (Eurofins Genomics).
References
- Auer TO, Duroure K, De Cian A, Concordet JP, Del Bene F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Research. 2014; 24:142-153. DOI | PubMed
- Bert B, Chmielewska J, Bergmann S, Busch M, Driever W, Finger-Baier K, Hößler J, Köhler A, Leich N, Misgeld T, Nöldner T, Reiher A, Schartl M, Seebach-Sproedt A, Thumberger T, Schönfelder G, Grune B. Considerations for a European animal welfare standard to evaluate adverse phenotypes in teleost fish. The EMBO Journal. 2016; 35:1151-1154. DOI | PubMed
- Cong L, Ran FA, Cox D, Lin S, Barretto R, Habib N, Hsu PD, Wu X, Jiang W, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013; 339:819-823. DOI | PubMed
- Gutschner T, Haemmerle M, Genovese G, Draetta GF, Chin L. Post-translational regulation of Cas9 during G1 enhances homology-directed repair. Cell Reports. 2016; 14:1555-1566. DOI | PubMed
- Heyer WD, Ehmsen KT, Liu J. Regulation of homologous recombination in eukaryotes. Annual Review of Genetics. 2010; 44:113-139. DOI | PubMed
- Hustedt N, Durocher D. The control of DNA repair by the cell cycle. Nature Cell Biology. 2017; 19:1-9. DOI
- Iwamatsu T. Stages of normal development in the medaka Oryzias latipes. Mechanisms of Development. 2004; 121:605-618. DOI | PubMed
- Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012; 337:816-821. DOI | PubMed
- Kimelman D. Cdc25 and the importance of G2 control. Cell Cycle. 2014; 13:2165-2171. DOI | PubMed
- Kimmel CB, Ballard WW, Kimmel SR, Ullmann B, Schilling TF. Stages of embryonic development of the zebrafish. Developmental Dynamics. 1995; 203:253-310. DOI | PubMed
- Kirchmaier S, Lust K, Wittbrodt J. Golden GATEway cloning–a combinatorial approach to generate fusion and recombination constructs. PLoS One. 2013; 8DOI | PubMed
- Koster R, Stick R, Loosli F, Wittbrodt J. Medaka Spalt acts as a target gene of hedgehog signaling. Development. 1997; 124:3147-3156. PubMed
- Kraeussling M, Wagner TU, Schartl M. Highly asynchronous and asymmetric cleavage divisions accompany early transcriptional activity in pre-blastula medaka embryos. PLoS One. 2011; 6DOI | PubMed
- Loosli F, Winkler S, Wittbrodt J. Six3 overexpression initiates the formation of ectopic retina. Genes & Development. 1999; 13:649-654. DOI | PubMed
- Mali P, Esvelt KM, Church GM. Cas9 as a versatile tool for engineering biology. Nature Methods. 2013; 10:957-963. DOI | PubMed
- Maruyama T, Dougan SK, Truttmann MC, Bilate AM, Ingram JR, Ploegh HL. Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining. Nature Biotechnology. 2015; 33:538-542. DOI | PubMed
- Reinhardt R, Centanin L, Tavhelidse T, Inoue D, Wittbrodt B, Concordet JP, Martinez-Morales JR, Wittbrodt J. Sox2, Tlx, Gli3, and Her9 converge on Rx2 to define retinal stem cells in vivo. The EMBO Journal. 2015; 34:1572-1588. DOI | PubMed
- Rembold M, Lahiri K, Foulkes NS, Wittbrodt J. Transgenesis in fish: efficient selection of transgenic fish by co-injection with a fluorescent reporter construct. Nature Protocols. 2006; 1:1133-1139. DOI | PubMed
- Rupp RA, Snider L, Weintraub H. Xenopus embryos regulate the nuclear localization of XMyoD. Genes & Development. 1994; 8:1311-1323. DOI | PubMed
- Southern E. Southern blotting. Nature Protocols. 2006; 1:518-525. DOI | PubMed
- Stemmer M, Thumberger T, Del Sol Keyer M, Wittbrodt J, Mateo JL. CCTop: an intuitive, flexible and reliable CRISPR/Cas9 target prediction tool. PLoS One. 2015; 10DOI | PubMed
- Wang L, Yang L, Guo Y, Du W, Yin Y, Zhang T, Lu H. Enhancing targeted genomic DNA editing in chicken cells using the CRISPR/Cas9 system. PLoS One. 2017; 12DOI | PubMed
- Winkler C, Vielkind JR, Schartl M. Transient expression of foreign DNA during embryonic and larval development of the medaka fish (Oryzias latipes). MGG Molecular & General Genetics. 1991; 226:129-140. DOI | PubMed
- Won M, Dawid IB. PCR artifact in testing for homologous recombination in genomic editing in zebrafish. PLoS One. 2017; 12DOI | PubMed
Fonte
Gutierrez-Triana JA, Tavhelidse T, Thumberger T, Thomas I, Wittbrodt B, et al. () Efficient single-copy HDR by 5’ modified long dsDNA donors. eLife 7e39468. https://doi.org/10.7554/eLife.39468





