Abstract
Introduzione
Lo stafilococco aureo è uno dei principali batteri patogeni umani che può causare un ampio spettro di infezioni da moderate a gravi che vanno da infezioni cutanee e ortopediche a polmonite necrotizzante mortale e sepsi. È considerato una delle cause più frequenti di morbilità e mortalità in tutto il mondo (Lowy, 1998). Spesso provoca reazioni iperinfiammatorie del sistema immunitario dell’ospite contribuendo al suo elevato tasso di mortalità nelle infezioni sistemiche. Gli stafilococchi possiedono uno spesso strato di peptidoglicano, a cui sono legati gli acidi teicoici e i polisaccaridi. Gli acidi teicoici alla parete cellulare includono gli acidi teicoici della parete (WTA) e gli acidi lipoteico (LTA). Questi agiscono come fattori di patogenicità e sono stabiliti ligandi TLR-2 (Travassos et al., 2004). Oltre ad altre, le tossine stafilococciche comprendono le enterotossine e le moduline fenolo-solubili (PSM), recentemente identificate. Di tutte le 20 o più enterotossine stafilococciche, le enterotossine stafilococciche A e B (SEA e SEB) sono state meglio caratterizzate. Essi sono considerati come super-antigeni a causa della loro capacità di reticolare le molecole MHC di classe II con i recettori delle cellule T e quindi stimolare grandi popolazioni di cellule T indipendenti dal legame specifico dell’antigene. Ciò si traduce in una massiccia proliferazione policlonale delle cellule T e in una secrezione infiammatoria di citochine (Pinchuk et al., 2010). I PSM sono solubili in fenolo e considerati importanti fattori di virulenza. Alcuni di questi peptidi sono in grado di lisare i neutrofili umani (Wang et al., 2007). In particolare, i ceppi di S. aureus meticillino-resistenti associati alla comunità altamente virulenti (CA-MRSA) rilasciano grandi quantità di peptidi PSM citolitici distinti (Peschel e Otto, 2013). È interessante notare che i PSM sono stati riportati anche come peptidi immunomodulatori per le cellule dendritiche che portano a una ridotta infiammazione delle cellule T (Schreiner et al., 2013).
Le cellule soppressori di derivazione mieloide (MDSC) rappresentano un nuovo meccanismo antinfiammatorio descritto per la prima volta in pazienti affetti da cancro (Schmielau e Finn, 2001). Negli ultimi anni è diventato chiaro che le MDSC svolgono un ruolo critico anche nella regolazione di diversi tipi di infiammazione che non sono direttamente associati al cancro, ad esempio nelle malattie infettive (Marigo et al., 2008; Gabrilovich e Nagaraj, 2009). Queste cellule mieloidi sono caratterizzate dalla loro capacità di sopprimere potentemente le risposte delle cellule T (Gabrilovich e Nagaraj, 2009). Le MDSC includono due sottoinsiemi principali basati sulle loro caratteristiche fenotipiche e morfologiche: polimorfonucleare (PMN-) e monocitico (M-)MDSC. Questi sottoinsiemi mostrano caratteristiche funzionali e biochimiche uniche, ma parzialmente sovrapposte (Gabrilovich e Nagaraj, 2009; Dumitru et al., 2012; Bronte et al., 2016). Fenotipicamente, il PMN-MDSC umano è stato determinato in modo più coerente come CD33+CD11b+CD14- CD15+ e M-MDSC come CD33+CD14+HLA-DRlow( Bronte et al., 2016 ).
MDSC nel contesto dell’interazione ospite-patogeno sono stati recentemente riportati per diversi patogeni batterici (Ost et al., 2016), ad esempio, per Klebsiella pneumoniae (Poe et al., 2013), Mycobacterium tuberculosis (du Plessis et al., 2013), e Pseudomonas aeruginosa (Rieber et al., 2013). Studi precedenti hanno anche fornito prove di un contributo di S. aureus sulla generazione e la funzione di MDSC: (i) Due gruppi di ricerca hanno riferito che l’MDSC è coinvolto nelle infezioni da biofilm ortopediche (Heim et al., 2014; Peng et al., 2017). Grazie alla loro azione antinfiammatoria, gli MDSC hanno contribuito alla cronicità delle infezioni da biofilm di S. a ureus (Heim et al., 2014). (ii) Tebartz et al. hanno descritto un effetto immunosoppressivo predominante di MDSC rispetto alle cellule T regolatorie per la cronicità delle infezioni di S. a ureus (Tebartz et al., 2015). (iii) D’altra parte sono stati descritti anche i decorsi della malattia migliorati sotto l’influenza di MDSC, ad esempio, in modelli murini di sindrome da shock tossico stafilococcico acuto causato da enterotossina B stafilococcica (Szabo et al., 2016) e di dermatite atopica con cute colonizzata da S. a ureus (Skabytska et al., 2014).
Sulla base di questi risultati precedenti, in questo studio in vitro abbiamo cercato di determinare ulteriormente l’impatto di diversi ceppi di S. a ureus e dei fattori di virulenza associati sulla generazione di MDSC nell’uomo. Qui dimostriamo per la prima volta che le enterotossine stafilococciche modulano in modo dose-dipendente la generazione di MDSC. L’interazione delle enterotossine stafilococciche con le cellule soppressori di derivazione mieloide potrebbe giocare un ruolo importante nella reazione infiammatoria di overshooting spesso vista nelle infezioni sistemiche di S. aureus.
Materiali e metodi
Ceppi batterici, condizioni di coltura e preparazione dei supernatanti stafilococcici per i test di stimolazione
Per analizzare l’induzione o l’inibizione della formazione di MDSC mediata da S. aureus, abbiamo usato una varietà di ceppi stafilococcici (Tabella 1). I batteri sono stati conservati come scorte di glicerolo a -80°C e coltivati durante la notte su piastre di agar TSB a 37°C (caseina peptone 17 g/l, soia peptone 3 g/l, glucosio 2,5 g/l, idrogeno fosfato dipotassico 2,5 g/l, cloruro di sodio 5 g/l, Sigma-Aldrich). Le singole colonie di ogni ceppo sono state inoculate e agitate per 16 h a 130 rpm a 37°C in mezzo RPMI 1640 (Biochrom), integrate con 4 mM di L-glutammina (Gibco/Life Technologies). Le cellule batteriche sono state rimosse per centrifugazione per 30 minuti a 5.000x g a 4°C e i surnatanti sono stati sterilizzati due volte con filtri non pirogenici da 0,2 μm. La crescita equivalente dei batteri è stata verificata da misurazioni di densità ottica a 600 nm e da CFU contando su piastre di agar TSB. P. aeruginosa è stato coltivato durante la notte in mezzo TSB invece di RPMI 1640, e sopranatanti sono stati preparati come descritto (Rieber et al., 2013). I supernatanti filtrati sono stati conservati in aliquote a -20°C e sono stati utilizzati per esperimenti di stimolazione.
| Ceppo (sostantivo) | Identificazione del ceppo [a] | Genotipo e descrizione [b] | Riferimenti |
|---|---|---|---|
| S. aureus | |||
| USA300 (FPR3757) | ATCC BAA-1516, NCBI 451515, NRS482 | In questo studio si parla di “USA300”; CA-MRSA; agr+ | McDougal et al., 2003; Diep et al., 2006 |
| USA300 (LAC) Δ agr | n.d. | CA-MRSA; cancellato agr locus agr | Cheung et al., 2011 |
| USA300 (JE2) | JE2 | CA-MRSA; derivato da USA300 (LAC) mediante la polimerizzazione di tre plasmidi; ceppo parentale di mutanti a inserzione transposon raccolti nella Nebraska Transposon Mutant Library | Fey et al., 2013 |
| USA300 (JE2) secA- | NE66 | Inserimento di Tn nel gene della translocazione delle preproteine SAUSA300_2584 | Fey et al., 2013 |
| USA300 (JE2) enterotossina presunta di tipo A- | NE309 | Inserimento di Tn in enterotossina presunta di tipo A gene SAUSA300_1559 | Fey et al., 2013 |
| USA300 (JE2) sek- | NE1255 | Inserimento di Tn nel gene dell’enterotossina K SAUSA300_0800 | Fey et al., 2013 |
| USA300 (JE2) seq- | NE1605 | Inserimento di Tn nel gene Q dell’enterotossina SAUSA300_0801 | Fey et al., 2013 |
| USA300 (JE2) selX- | NE1809 | Inserimento di Tn nel gene presunto dell’enterotossina selX SAUSA300_0370 | Fey et al., 2013 |
| USA300 (SF8300) | n.d. | CA-MRSA | Diep et al., 2008b |
| USA300 (SF8300) Δ ACME | n.d. | Locus ACME cancellato | Diep et al., 2008b |
| USA400 (MW2) | ATCC BAA-1707, NCBI 196620, NRS123 | CA-MRSA | Baba et al., 2002 |
| N315 | NCBI 158879, NRS70 | HA-MRSA | Kuroda et al., 2001 |
| Mu50 | ATCC 700699, NCBI 158878, NRS1 | Vancomicina-intermedio HA-MRSA | Kuroda et al., 2001 |
| COL | NCBI 93062, NRS100 | HA-MRSA, agr basso, sigB+ | Gill et al., 2005 |
| Newman | ATCC 13420, NCTC 8178, NCBI 426430 | MSSA; agr+; saeRS espresso in modo costitutivo; fenotipo PVL-; fnbA- e fnbB- | Baba et al., 2008 |
| Newman φSa2MW | n.d. | Newman lisogenizzato con φSa2MW che trasporta lukF/S-PV | Wirtz et al., 2009 |
| PS187 | ATCC 15564, NCTC 9754, NCBI 1323662 | MSSA; prototipo del lignaggio ST395; struttura WTA unica nel suo genere con una spina dorsale GroP-GalNAc | Winstel et al., 2013 |
| NCTC 8325; RN1 | NCTC 8325, NCBI 93061, NRS77 | MSSA; ceppo parentale di 8325-4, RN4220, HG003, SA113, RN6390; φ11+ φ12+ φ13+, agr+, rsbU-, tcaR- | Novick, 1967 |
| HG003 | n.d. | RN1 derivato; φ11+ φ12+ φ12+ φ13+, agr+, rsbU e tcaR riparato | Herbert et al., 2010 |
| SA113 | ATCC 35556, DSM 4910 | RN1 derivato; MNNG mutagenesi; φ11+ φ12+ φ13+, agr- rsbU- tcaR-, r- m– | Iordanescu e Surdeanu, 1976 |
| 8325-4; RN0450 | NRS135 | Derivato RN1; mutagenesi UV; φ11- φ12- φ13-, agr+, rsbU-, tcaR-, tcaR- | Novick, 1967 |
| RN6390 | n.d. | derivato RN1; mutagenesi UV, φ6390 lisogenesi e inserimento Tn554 erm; φ11- φ12- φ13-, agr+ rsbU- tcaR- | Peng et al., 1988 |
| RN4220 | NCBI 561307, NRS144, DSM 26309 | Derivato RN1; mutagenesi UV e MNNG; φ11- φ12- φ13-, agr-, rsbU-, tcaR-, r- m- | Kreiswirth et al., 1983 |
| S. carnosus | |||
| TM300 | BCN 396513 | Batterio di coltura di starter di carne, apatogeno | Rosenstein et al., 2009 |
| P. aeruginosa | |||
| PAO-1 | ATCC 15692, NCBI 208964, DSM 22644 | L’agente patogeno opportunistico che induce MDSC, persiste nella fibrosi cistica | Stover et al., 2000 |
2. Pretrattamento dei supernatanti stafilococcici e delle enterotossine
Dove indicato, i supernatanti stafilococcici da colture notturne RPMI sono stati diluiti 1:3 in RPMI 1640 e pretrattati con 20 μg/ml di proteinasi K (Promega) a 130 rpm a 37°C per 16 h. Il trattamento termico dei supernatanti stafilococcici o enterotossine stafilococciche è stato eseguito a 80°C per 20 min.
Per gli esperimenti di esclusione delle dimensioni, sono stati utilizzati i concentratori Vivaspin 15 con taglio del peso molecolare (MWCO) di 10.000 o 50.000 (Sartorius Stedim Biotech). I supernatanti stafilococcici sono stati centrifugati attraverso filtri MWCO 50.000 per 30 minuti a 4.000x g e 4°C, ottenendo una concentrazione di 300 volte. Il flusso attraverso il flusso è stato poi centrifugato in una seconda fase attraverso MWCO 10.000 filtri utilizzando le stesse condizioni. Il flusso passante e i concentrati residui sono stati regolati dopo ogni fase al volume iniziale con il mezzo utilizzato per la crescita batterica.
Isolamento del PBMC umano
Le cellule mononucleate del sangue periferico (PBMC) sono state preparate a partire da campioni di sangue umano eparinizzato da volontari sani mediante sedimentazione a gradiente di densità Ficoll (mezzo di separazione dei linfociti, Biochrom). PBMC sono stati lavati due volte in mezzo RPMI 1640 (Biochrom), risospesi in RPMI 1640 integrato con 10% FCS (Biochrom), 2 mM L-glutammina (Sigma-Aldrich) e 1% di penicillina /streptomicina (Biochrom), indicato come “mezzo completo”, e utilizzato nei saggi di stimolazione. Una vitalità di >90% è stato determinato per tutti i preparati PBMC utilizzando trypan colorazione blu.
MDSC generazione test di generazione e analisi citometriche a flusso
Subito dopo l’isolamento PBMC sono stati seminati in 24 piastre a fondo piatto (Corning) a 7,5 × 105 cellule per pozzetto in 1,5 ml di terreno completo, trattati con stimolanti come indicato, e coltivate in un’atmosfera umidificata a 37 ° C e 5% di CO2. Dopo 4 giorni, mezzo e stimolanti sono stati rinfrescati. Al sesto giorno di coltivazione, tutte le cellule sono state raccolte da colture PBMC e lavate e risospese in D-PBS (Sigma-Aldrich). Le cellule aderenti sono state raccolte utilizzando non-protease Detachin Cell Detachment Solution (Genlantis). SEA e SEB sono stati acquistati da Sigma-Aldrich. Stimolanti non batterici erano granulociti-macrofagi fattore stimolante colonia (GM-CSF, 10 ng/ml; Genzyme), Aspergillus fumigatus lisato (10 μg/ml; Miltenyi Biotec), e IL-2 (100 U/ml; MBL).
I numeri MDSC nelle colture PBMC sono stati quantificati mediante analisi citometriche a flusso. PMN-MDSC sono stati gated come SSChighCD33 + CD14- cellule. Queste cellule erano in parte positivi per CD16 (vedi Figura 1A per la strategia di gating). Ulteriori marcatore di superficie rappresentativo colorazione superficiale ha dimostrato una forte positività per CD11b, ma la colorazione negativa per CD15 (Figura 1A per la strategia di gating). Pertanto, queste cellule granulocitiche sono state considerate come PMN-come MDSC. Il numero totale di cellule apoptotiche e cellule morte è stato determinato dalla colorazione dell’annesso V e dello ioduro di propidio. Per la quantificazione del totale delle cellule vive, è stata eseguita la colorazione blu di tripan e le cellule vive sono state contate in una camera di Neubauer. Sono stati acquistati da Miltenyi Biotec anticorpi anti-CD33 marcati PE, anti-CD11b marcati APC e rispettivi anticorpi di controllo dell’isotipo; PI, FITC Annexin V, FITC anti-CD14, APC anti-CD14, FITC anti-CD15, PerCP anti-CD16, APC anti-CD3 e rispettivi anticorpi di controllo dell’isotipo sono stati ottenuti da BD Pharmingen. La citometria a flusso è stata eseguita utilizzando un FACSCalibur (BD), e le analisi delle percentuali di cellule sono state eseguite utilizzando il software BD CellQuest Pro. La percentuale di PMN-MDSC in colture di medie dimensioni è stata impostata a 1. Le percentuali di PMN-MDSC dopo la stimolazione sono presentate come x-fold rispetto ai controlli medi. I controlli non macchiati sono stati utilizzati per impostare i quadranti del diagramma a punti.
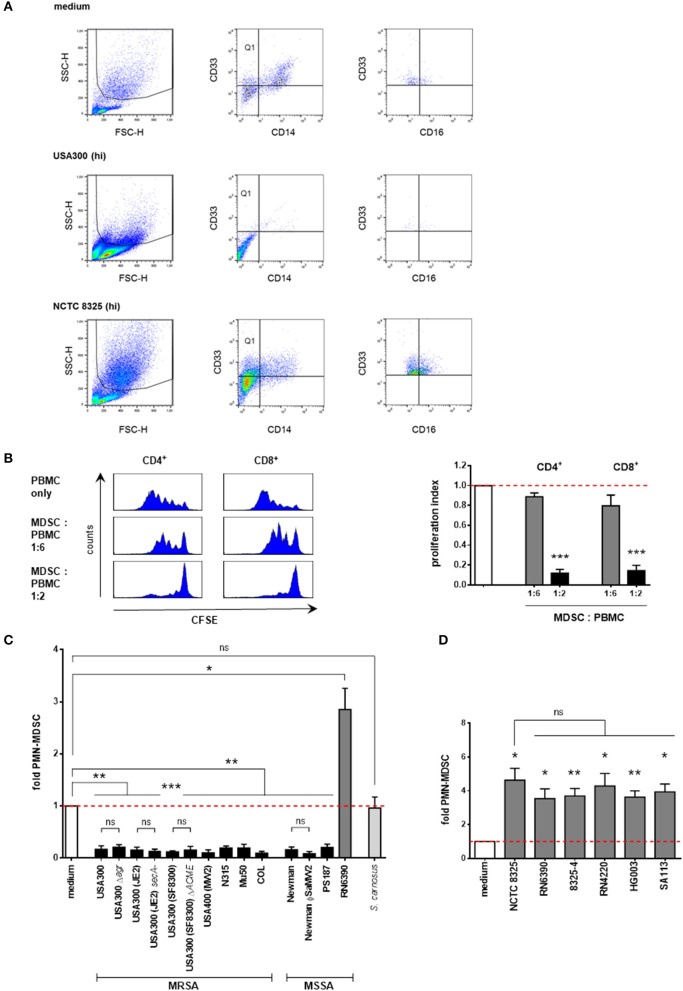
Figura 1.Supernatanti da ceppi di S. aureus modulano in modo differenziato i livelli di PMN-like MDSC. PBMC sono stati stimolati con supernatanti preparati da colture notturne dei ceppi stafilococcici indicati e PMN-come MDSC sono stati valutati con citometria a flusso. (A) Determinazione fenotipica di PMN-like MDSC in PBMC. La regione granulocitaria è stata gated nel lato anteriore-diffusione. In seguito CD33 + CD14- cellule (Quadrante Q1) sono stati gated e PMN-come MDSC sono stati determinati come SSChighCD33 + CD14- cellule. Un’ulteriore analisi citrometrica di flusso ha rivelato che queste cellule granulocitarie sono in parte CD16+. I diagrammi a punti illustrano la modulazione di PMN-like MDSC mediata da supernatanti da NCTC 8325 e USA300 [al 3% (hi) concentrazione] rispetto al solo mezzo. (B)S. aureo indotto PMN-MDSCaureo sopprime la proliferazione delle cellule T in modo dose-dipendente. PMN-like MDSC sono stati indotti utilizzando lo 0,02% di USA300 supernatanti, isolati con separazione MACS CD33 e co-coltura per 4 giorni con PBMC appena isolato, CFSE macchiato di CFSE a determinati rapporti. CFSE-fluorescenza intensità di CD4 + e CD8 + cellule T è stata analizzata con citometria a flusso. Pannello di sinistra: Istogrammi che mostrano la soppressione della proliferazione delle cellule T. Pannello di destra: Le barre rappresentano l’indice di proliferazione. I valori sono normalizzati alla proliferazione delle cellule CD4+ o CD8+ T senza aggiunta di MDSC. Le barre rappresentano significa ± SEM. Le differenze tra le co-culture MDSC e i controlli sono state analizzate con un t-test di un campione. (C) Screening di S. aureus supernatanti per la modulazione dell’induzione di PMN-like MDSC. PBMC sono stati stimolati utilizzando 3 vol.% di supernatanti preparati da colture notturne dei ceppi stafilococcici indicati. Ad eccezione di RN6390, tutti i ceppi di S. a ureus testati hanno inibito la formazione di PMN-like MDSC. I ceppi di S. aureus sono illustrati in barre nere ad eccezione dei membri NCTC 8325 (mostrati in barre grigio scuro). S. carnosus è mostrato in barre grigio chiaro. D) Screening dei ceppi derivati NCTC 8325. Tutti i membri testati della famiglia NCTC 8325 hanno costantemente indotto PMN-come MDSC. Le barre rappresentano i mezzi ± SEM. Le differenze tra le stimolazioni e i controlli (C,D) sono state analizzate con un t-test di un campione. Le differenze tra i diversi ceppi wild-type e mutanti di S. aureus (C,D) sono stati analizzati da un test di Mann-Whitney o da un t-test non accoppiato. *p < 0,05; **p < 0,01; ***p < 0, 001; ns-non significativo.

Figura 1.I surnatanti da ceppi di S. aureus modulano in modo differenziato i livelli di PMN-like MDSC. PBMC sono stati stimolati con supernatanti preparati da colture notturne dei ceppi stafilococcici indicati e PMN-come MDSC sono stati valutati con citometria a flusso. (A) Determinazione fenotipica di PMN-like MDSC in PBMC. La regione granulocitaria è stata gated nel lato anteriore-diffusione. In seguito CD33 + CD14- cellule (quadrante Q1) sono stati gated e PMN-come MDSC sono stati determinati come SSChighCD33 + CD14- cellule. Un’ulteriore analisi citrometrica di flusso ha rivelato che queste cellule granulocitarie sono in parte CD16+. I diagrammi a punti illustrano la modulazione di PMN-like MDSC mediata da supernatanti da NCTC 8325 e USA300 [al 3% (hi) concentrazione] rispetto al solo mezzo. (B)S. aureo indotto PMN-MDSCaureo sopprime la proliferazione delle cellule T in modo dose-dipendente. PMN-like MDSC sono stati indotti utilizzando lo 0,02% di USA300 supernatanti, isolati con separazione MACS CD33 e co-coltura per 4 giorni con PBMC appena isolato, CFSE macchiato di CFSE a determinati rapporti. CFSE-fluorescenza intensità di CD4 + e CD8 + cellule T è stata analizzata con citometria a flusso. Pannello di sinistra: Istogrammi che mostrano la soppressione della proliferazione delle cellule T. Pannello di destra: Le barre rappresentano l’indice di proliferazione. I valori sono normalizzati alla proliferazione delle cellule CD4+ o CD8+ T senza aggiunta di MDSC. Le barre rappresentano significa ± SEM. Le differenze tra le co-culture MDSC e i controlli sono state analizzate con un t-test di un campione. (C) Screening di S. aureus supernatanti per la modulazione dell’induzione di PMN-like MDSC. PBMC sono stati stimolati utilizzando 3 vol.% di supernatanti preparati da colture notturne dei ceppi stafilococcici indicati. Ad eccezione di RN6390, tutti i ceppi di S. a ureus testati hanno inibito la formazione di PMN-like MDSC. I ceppi di S. aureus sono illustrati in barre nere ad eccezione dei membri NCTC 8325 (mostrati in barre grigio scuro). S. carnosus è mostrato in barre grigio chiaro. D) Screening dei ceppi derivati NCTC 8325. Tutti i membri testati della famiglia NCTC 8325 hanno costantemente indotto PMN-come MDSC. Le barre rappresentano i mezzi ± SEM. Le differenze tra le stimolazioni e i controlli (C,D) sono state analizzate con un t-test di un campione. Le differenze tra i diversi ceppi wild-type e mutanti di S. aureus (C,D) sono stati analizzati da un test di Mann-Whitney o da un t-test non accoppiato. *p < 0,05; **p < 0,01; ***p < 0, 001; ns-non significativo.
Test di soppressione delle cellule T
MDSC sono stati generati da cellule mieloidi della frazione PBMC come descritto in precedenza e isolati da colture cellulari mediante selezione di cellule a microsfere magnetiche per CD33 (Miltenyi Biotec). Responder-PBMC sono stati ottenuti da sangue eparinizzato di volontari sani e colorati con CFSE (Life Technologies) secondo il protocollo del produttore. CFSE etichettati PBMC sono stati stimolati con 100 U / ml IL-2 (Sistemi di R & D) e 1 μg / ml OKT3 (Janssen-Cilag). Sia MDSC e CFSE etichettati PBMC sono stati aggiunti a RPMI 1640 medio integrato con il 10% di siero umano, 2 mM L-glutammina, 100 UI / ml di penicillina e 100 mg / ml di streptomicina. In una piastra di fondo rotonda a 96 pozzetti (Greiner Bio-One), sono stati aggiunti a 60.000 PBMC per pozzetto 10.000/30.000 MDSC o, solo come mezzo di controllo integrato, sono stati aggiunti a 60.000 PBMC per pozzetto. Le cellule sono state incubate in un’atmosfera umidificata a 37°C e al 5% di CO2. Il giorno 4 le cellule sono state raccolte e colorate con anticorpi anti-CD8a-APC, anti-CD4-PE (BioLegend), e ioduro di propidio (BD). Le cellule PI positive sono state escluse nella citometria a flusso. I segnali CFSE di CD4+ e CD8+ PBMC sono stati analizzati.
MS analisi di proteine stafilococciche surnatanti
Preparazione di proteine surnatanti stafilococciche per le analisi della SM
Per determinare il loro contenuto di enterotossine, i supernatanti stafilococcici sono stati preparati nello stesso modo come per i saggi di stimolazione di cui sopra, tranne che le colture notturne sono state coltivate in medium TSB (Sigma-Aldrich) per 14 h per ottenere maggiori rese di proteine sopranatanti. Quantità equivalenti di batteri sono stati confermati da misure di densità ottica a 600 nm e dal conteggio delle CFU. Le proteine sono state ottenute dai surnatanti mediante precipitazione di cloroformio/metanolo, utilizzando 4 vol. di metanolo, 1 vol. di cloroformio e 3 vol. H2Odd per vol. di coltura notturna. Dopo la centrifugazione per 45 minuti a 4.800x g e 12°C, è stata rimossa la fase acquosa e sono stati aggiunti 6 vol. di metanolo, seguita da una seconda fase di centrifugazione utilizzando le stesse condizioni per il pellet delle proteine. I pellet proteici sono stati essiccati all’aria e conservati a -20°C.
Digestione triptica delle proteine
Per l’analisi del proteoma i pellet proteici sono stati eseguiti su un gel, e dopo una breve colorazione di Coomassie, la digestione triptica delle proteine e l’analisi nano-MS/MS sono state fatte come descritto in precedenza (Burian et al., 2015), tranne per il fatto che abbiamo usato un gradiente segmentato di 230 minuti.
Elaborazione e analisi dei dati MS
Gli spettri MS acquisiti sono stati elaborati con il pacchetto software MaxQuant versione 1.2.2.9 (Olsen et al., 2005; Cox e Mann, 2008) con motore di ricerca integrato Andromeda. La ricerca nel database è stata eseguita contro un target-decoy di tutti i ceppi di S. aureus. Il database è stato ottenuto da UniProt (ID tassonomia 1280), contenente 126.225 voci proteiche e 247 contaminanti di laboratorio comunemente presenti. In base al contenuto medio di TSB, abbiamo inoltre ricercato il Bos taurus (bovini; tassonomia ID 9913) contenente 24.240 voci proteiche, e il Glycine max (soia; tassonomia ID 3847) contenente 64.601 voci proteiche, entrambi ottenuti da UniProt. La tripsina endoproteasi è stata fissata definita come la proteasi con un massimo di due scollature mancanti. L’ossidazione delle metionine e l’acetilazione N-terminale sono stati specificati come modifiche variabili, mentre la carbamidometilazione sulle cisteine è stata definita come una modifica fissa. La tolleranza di massa massima iniziale consentita è stata impostata a 6 ppm (per la scansione di indagine) e 0,5 Da per gli ioni frammento CID. Un tasso di falsa scoperta dell’1% è stato applicato a livello peptidico e proteico. Un minimo di due conteggi peptidici non modificati sono stati richiesti per la rispettiva quantificazione delle proteine. L’algoritmo senza etichetta è stato attivato, così come l’opzione “match between runs” (Luber et al., 2010).
Analisi statistica
L’analisi statistica è stata eseguita in GraphPad Prism versione 6.0 utilizzando un t-test a un campione, un test Mann-Whitney o un t-test non accoppiato come indicato. In tutti i test, le differenze sono state considerate significative a P < 0,05 (*P<0,05; **P < 0, 01; ***P < 0,001; ****P < 0 , 0001).
Risultati
I ceppi di S. aureus modulano in modo differenziato il PMN-come MDSC
Per determinare l’impatto di S. aureus sulla generazione di MDSC, ci siamo basati su un sistema di generazione di MDSC in vitro (Lechner et al., 2010; Rieber et al., 2013) per indurre il PMN-MDSC da sangue periferico di donatori sani e per quantificarli con la citometria a flusso. PMN-MDSC sono state inizialmente gated come cellule ad alta granularità (SSChigh), recanti il marcatore mieloide CD33, prive del marcatore monocitico CD14 (rappresentativo dei punti Figura 1A) e con la caratteristica di sopprimere le risposte delle cellule T (dati rappresentativi in Figura 1B). Un’ulteriore analisi citometrica a flusso ha rivelato che queste cellule mieloidi soppressive sono fortemente CD11b+, in parte CD16+, e CD15-( Figura 1A e Figura supplementare S1), perché preferiremmo definirle PMN-come MDSC.
Inizialmente abbiamo esaminato i supernatanti a concentrazioni del 3% da una vasta gamma di ceppi di S. aureus, compresi vari isolati clinici di MRSA e MSSA e diversi ceppi mutanti affetti nella loro espressione di geni di virulenza prominente per la loro capacità di modulare la generazione di MDSC. Inoltre, abbiamo testato i supernatanti di diversi ceppi di laboratorio stafilococco stabiliti e di un’altra specie stafilococcica, S. carnosus. Tutti i ceppi di S. aureus testati hanno abrogato la formazione di MDSC, ad eccezione del ceppo RN6390, che ha indotto MDSC (Figura 1C). Al contrario, il batterio non patogeno per uso alimentare S. carn osus non ha modulato i livelli cellulari di MDSC in modo significativo.
Rispetto al rispettivo ceppo wild-type USA300, il knock out del regolatore globale della virulenza Agr, che regola un’ampia varietà di determinanti della virulenza (Novick, 2003), non ha influenzato i livelli di MDSC. Inoltre, la mutagenesi transposon di secA, un componente del percorso Sec per la secrezione di proteine (Green e Mecsas, 2016), non ha avuto alcun effetto nel substrato USA300 JE2.
L’elemento mobile catabolico arginina ACME, un elemento genetico mobile che conferisce sopravvivenza e crescita negli ospiti fornendo resistenza alla poliammina (Diep et al., 2008b; Joshi et al., 2011), è espresso prevalentemente in ceppi MRSA (Shore et al., 2011). Analogamente, la leucocidina pantene-valentina pantenica (PVL), che mostra effetti tossici sui neutrofili (Genestier et al., 2005) è presente praticamente in tutti i ceppi di CA-MRSA e in alcuni ceppi di MSSA (Diep et al., 2008a; Wirtz et al., 2009). Sia il knock out del locus ACME in USA300 substrain SF8300 che la lisogenializzazione di PVL-negativo Newman con phage ΦSa2MW, che porta il locus del gene PVL lukF/S-PV (Bubeck Wardenburg et al., 2007), non hanno avuto alcun effetto sulla generazione di MDSC rispetto ai rispettivi ceppi parentali.
Il ceppo RN6390 è un membro altamente mutato della stirpe NCTC 8325 dei ceppi di S. aureus. Il suo ceppo parentale NCTC 8325 (noto anche come RN1) è un ceppo di laboratorio consolidato comunemente usato per gli studi genetici, per cui numerosi ceppi derivati sono stati ingegnerizzati mediante procedure mutagene e la ricostruzione di distinti loci genici mutagenizzati (Herbert et al., 2010; Baek et al., 2013). Per verificare se uno qualsiasi dei geni mutati ha avuto un impatto sulla formazione di MDSC, abbiamo usato supernatanti del ceppo parentale NCTC 8325 e dei suoi ceppi derivati RN6390, 8325-4, RN4220, HG003, e SA113 per la stimolazione (Figura 1D). Il ceppo parentale NCTC 8325 ha agr funzionale, sarA, e sae regolatori di virulenza globale, tuttavia, è caratterizzato da una carenza di acitività del sigma importante regolatore virulenza SigB fattore Sigma, che è dovuto ad una delezione nel gene rsbU, e nel tcaR gene regolatore, un attivatore della proteina A trascrizione, entrambi i quali sono stati ricostituiti nel ceppo HG003. Inoltre, è carente nella produzione di beta-emolisina a causa dell’inserimento del profago ϕ13 nel gene hlb. ϕ13, tuttavia, è la codifica per le IgG, C3b e la stafilocchinasi degradante della fibrina. I ceppi RN6390 e 8325-4 sono stati curati da ϕ13 e da altri profagi. RN4220 mostra un’ulteriore carenza nella produzione di alfa funzionale e delta-emolisine e una parziale inattivazione del regolatore globale della virulenza regione agr, mentre il ceppo SA113 è un altro mutante agr derivato direttamente da NCTC 8325. Nel nostro sistema di generazione MDSC in vitro, tutti i membri della famiglia 8325 indotti MDSC indotti dalla famiglia 8325 sono stati testati in misura simile (Figura 1D). Presi nel loro insieme, i nostri risultati suggeriscono che i fattori di virulenza sopra citati non sono necessari per la modulazione dei livelli di MDSC.
Il genoma del ceppo parentale NCTC 8325, ma non quello del ceppo RN6390, è stato sequenziato e annotato (GenBank accession n. NC_07795.1) e sono disponibili database di proteine (UniProt Taxon ID 93061). Abbiamo quindi effettuato ulteriori analisi comparative funzionali, genomiche e proteomiche utilizzando NCTC 8325 e gli isolati clinici strettamente correlati dal punto di vista filogenetico (Baba et al., 2008) e ben caratterizzati USA300, Newman e COL.
La modulazione dei livelli di PMN-like MDSC da parte di S. aureus supernatanti è dose-dipendente
Utilizzando ampie gamme di concentrazione di supernatanti di S. aureus, abbiamo dimostrato che sia l’inibizione di MDSC che le attività che inducono MDSC esistono nella maggior parte dei ceppi di S. aureus. Concentrazioni più basse indotte da PMN-MDSC (livelli di picco a 0,01 per USA300, 0,003 per Newman e 0,001% per il COL), mentre concentrazioni più elevate hanno portato ad una marcata soppressione del PMN-MDSC quando si utilizzano supernatanti di questi ceppi (Figure 2A,B). Al contrario, i supernatanti del ceppo NCTC 8325 non hanno mostrato praticamente nessuna attività di espansione del MDSC al di sotto dello 0,01% di concentrazione del supernatante, i livelli massimi di PMN-MDSC non sono stati rilevati fino al 10%, e non è stata rilevata alcuna soppressione anche con concentrazioni di supernatanti fino al 30%.

Figura 2.S. aureus supernatanti dose-dipendentemente modulano la formazione di PMN-like MDSC, la proliferazione e gli effetti citotossici in PBMC. PBMC sono stati trattati con supernatanti da ceppi di S. a ureus USA300, NCTC 8325, Newman e COL in un ampio intervallo di concentrazione. (A,B)PMN-like MDSC sono stati quantificati con la citometria a flusso. (C,D) La proliferazione del PBMC totale è stata valutata mediante colorazione blu di trypan. (E,F) La morte delle cellule nelle colture PBMC totali è stata misurata mediante colorazione PI e successiva citometria a flusso. I dati sono presentati come mezzi ± SEM. Le differenze tra le stimolazioni e i controlli del mezzo sono state analizzate con un t-test a un campione e sono rappresentate con colori che si riferiscono alle rispettive curve. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001.

Figura 2.S. aureus supernatanti dose-dipendente modulano la formazione di PMN-come MDSC, la proliferazione e gli effetti citotossici in PBMC. PBMC sono stati trattati con supernatanti da ceppi di S. a ureus USA300, NCTC 8325, Newman e COL in un ampio intervallo di concentrazione. (A,B)PMN-like MDSC sono stati quantificati con la citometria a flusso. (C,D) La proliferazione del PBMC totale è stata valutata mediante colorazione blu di trypan. (E,F) La morte delle cellule nelle colture PBMC totali è stata misurata mediante colorazione PI e successiva citometria a flusso. I dati sono presentati come mezzi ± SEM. Le differenze tra le stimolazioni e i controlli del mezzo sono state analizzate con un t-test a un campione e sono rappresentate con colori che si riferiscono alle rispettive curve. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001.
La modulazione PMN-like MDSC è correlata sia con la proliferazione PBMC che con la citotossicità
Con l’aumento delle concentrazioni dei sopranatanti abbiamo osservato una concomitante proliferazione del PBMC in cultura. Pertanto, non abbiamo solo determinato i numeri assoluti di PMN-like MDSC, ma abbiamo anche quantificato i numeri totali di PBMC e discriminato le cellule vive e morte per esclusione del blu di tripan e colorazione PI (Figura 2). Durante una prima fase i numeri di PMN-like MDSC sono aumentati, mentre i numeri totali PBMC sono aumentati simultaneamente e si è verificata solo una morte cellulare moderata. Durante una seconda fase i numeri di PMN-like MDSC sono diminuiti, mentre il totale di PBMC si è ancora accumulato sostanzialmente nonostante l’aumento della citotossicità. Durante una terza fase tutte le cellule mieloidi sono state uccise prevalentemente quando la tossicità ha superato un certo livello. Con l’eccezione dei supernatanti del ceppo NCTC 8325, queste fasi dose-dipendenti sono state applicate alle stimolazioni con tutti i supernatanti di S. aureus testati. Al fine di determinare se l’effetto abrogante sull’MDSC è inibendo la differenziazione in MDSC o uccidendo effettivamente l’MDSC, abbiamo eseguito la colorazione Annexin/PI su MDSC gated. I risultati nella Figura Supplementare 2A indicano effetti citotossici su MDSC simili al PMN piuttosto che un arresto della differenziazione in MDSC. Se questo effetto di uccisione è diretto o indiretto attraverso altri tipi di cellule non può essere chiarito all’interno dello studio attuale, perché la maggior parte delle cellule CD33+, se isolate, sono morte entro tre giorni anche nel controllo del mezzo (Figura supplementare 2B). Il meccanismo esatto per la cessazione dell’MDSC rimane un argomento per le indagini future.
L’inibizione dell’MDSC supera l’induzione da parte di diversi surnatanti di S. aureus
Quando si mescolavano diversi surnatanti di S. aureus con effetti divergenti, le attività induttive erano superate dalle attività soppressive (Figura 3A). Il ceppo NCTC 8325 ha aumentato significativamente i livelli di PMN-like MDSC solo ad alte concentrazioni. Questo NCTC 8325-mediata MDSC induzione MDSC è stato soppresso da concentrazioni inibitorie di supernatanti USA300 (Figura 3B). Abbiamo concluso che, in termini di modulazione del PMN-like MDSC, alte concentrazioni di supernatanti NCTC 8325 corrispondono a concentrazioni più basse di supernatanti derivati da altri ceppi di S. aureus, e che ci potrebbe essere un fattore universale tra i diversi ceppi di S. aureus per la modulazione dose-dipendente di MDSC.
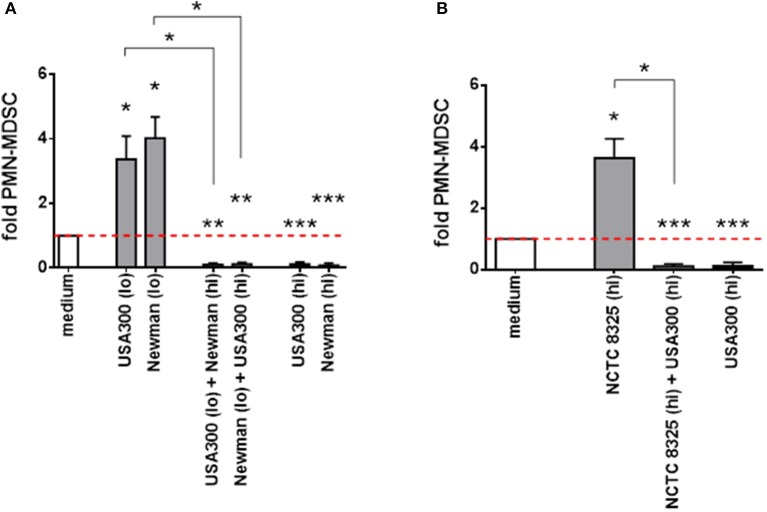
Figura 3.Figura 3. L’inibizione di MDSCsimile al PMN supera l’induzione da parte di diversi supernatanti di S . aureus. PBMC sono stati stimolati contemporaneamente con alte e basse dosi di supernatanti presi da diversi ceppi di S. aureus. L’induzione di PMN-like MDSC mediata da supernatanti provenienti da ceppi di S. a ureus USA300 o Newman (A) e dal ceppo meno attivo NCTC 8325 (B) è soppressa da supernatanti derivati da altri ceppi a concentrazioni precedentemente dimostrate come inibitorie. Per l’induzione di PMN-like MDSC (mostrato come barre grigie chiare), sono state utilizzate basse concentrazioni (0,01%) da USA300 o Newman e alte concentrazioni (10%) da NCTC 8325. Per la soppressione (mostrata come barre nere), sono state utilizzate alte concentrazioni (3%) da USA300 o Newman. Le stimolazioni miste sono illustrate sotto forma di barre grigie scure. I set di dati sono rappresentati come mezzi ± SEM. Le differenze tra le stimolazioni e i controlli del mezzo sono state analizzate con un t-test di un campione. Le differenze tra stimolazioni singole e miste sono state analizzate con un test Mann-Whitney. *p < 0,05; **p < 0,01; ***p < 0, 001.

Figura 3.Figura 3.PMN-come l’inibizione MDSC supera l’induzione da diversi sopranatanti S. aureus. PBMC sono stati stimolati contemporaneamente con alte e basse dosi di supernatanti presi da diversi ceppi di S. a ureus. L’induzione di PMN-like MDSC mediata da supernatanti provenienti da ceppi di S. a ureus USA300 o Newman (A) e dal ceppo meno attivo NCTC 8325 (B) è soppressa da supernatanti derivati da altri ceppi a concentrazioni precedentemente dimostrate come inibitorie. Per l’induzione di PMN-like MDSC (mostrato come barre grigie chiare), sono state utilizzate basse concentrazioni (0,01%) da USA300 o Newman e alte concentrazioni (10%) da NCTC 8325. Per la soppressione (mostrata come barre nere), sono state utilizzate alte concentrazioni (3%) da USA300 o Newman. Le stimolazioni miste sono illustrate sotto forma di barre grigie scure. I set di dati sono rappresentati come mezzi ± SEM. Le differenze tra le stimolazioni e i controlli del mezzo sono state analizzate con un t-test di un campione. Le differenze tra stimolazioni singole e miste sono state analizzate con un test Mann-Whitney. *p < 0,05; **p < 0,01; ***p < 0, 001.
S. aureus modula il PMN-come MDSC attraverso proteine secrete e termostabili
Per meglio definire la natura del fattore modulante MDSC, i supernatanti di S. aureus sono stati pretrattati in diverse condizioni prima di utilizzarli per la stimolazione di PBMC. Dopo la digestione con la proteinasi K, sia l’induzione MDSC che le attività inibitorie MDSC sono state abrogate, come mostrato per USA300 (Figura 4A). Il trattamento termico dei surnatanti non ha influito sui risultati (Figura 4B). Esperimenti sulle frazioni di dimensione utilizzando colonne di spin con i rispettivi pori tagliati MWCO hanno rivelato che le attività modulanti MDSC risiedono in frazioni di dimensione che vanno da 10 a 50 kDa. Attività di stimolazione residua sono stati osservati irregolarmente nella frazione di molecole più grandi, ma senza significato statistico (Figura 4C).
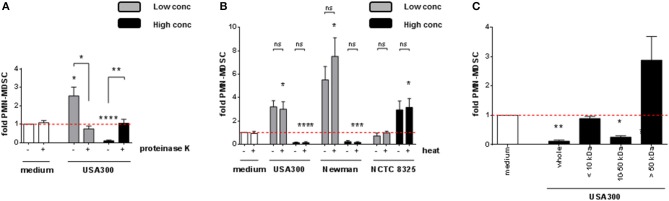
Figura 4.I livelli diPMN-like MDSC sono modulati dalle proteine di S. aureus termostabili di dimensioni da 10 a 50 kDa. S. aureus supernatanti sono stati pretrattati prima della stimolazione di PBMC. (A) Pretrattamento con 20 μg/ml di proteinasi K a 37°C per 16 h, (B) trattamento termico a 80°C per 20 min, (C) filtrazione attraverso colonne di spin con pori cut-off MWCO come indicato. 3% (USA300 o Newman) o 10% (NCTC 8325) di supernatanti sono stati utilizzati come alte concentrazioni (barre nere), 0,01% (tutti i ceppi testati) sono stati utilizzati come basse concentrazioni (barre grigie). I dati sono presentati come mezzi ± SEM. Le differenze tra i campioni stimolati e i controlli del mezzo sono stati analizzati con un t-test di un campione. Le differenze tra le diverse condizioni di pretrattamento (A,B) sono state analizzate con un test di Mann-Whitney. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.
I nostri risultati hanno indicato che i principali fattori modulanti MDSC di S. aureus sono molecole secrete, termostabili, proteiche con massa molecolare compresa tra 10 e 50 kDa e sono prodotte da stafilococchi patogeni piuttosto che da stafilococchi apatogeni. Inoltre, per tutti i ceppi tranne che per la famiglia NCTC 8325, abbiamo osservato un calo di PMN-like MDSC a concentrazioni crescenti di supernatanti, mentre il numero totale di PBMC era ancora in crescita. Con l’immunofenotipizzazione questa proliferazione è stata attribuita, tra l’altro, ad una notevole crescita delle cellule T CD3+. L’osservazione che fattori proteici stabili al calore, di dimensioni comprese tra 10 e 50 kDa, sono responsabili della modulazione dei livelli di PMN-like MDSC, e che una considerevole crescita delle cellule T si è verificata in concomitanza con il declino dei numeri di PMN-like MDSC, ci ha portato a indagare se le enterotossine stafilococciche superantigeniche possono essere il fattore chiave coinvolto nella modulazione di S. aureus-mediata di MDSC .

Figura 4.I livelli diPMN-like MDSC sono modulati da proteine di S. aureus termostabili di dimensioni da 10 a 50 kDa. S. a ureus supernatanti sono stati pretrattati prima della stimolazione di PBMC. (A) Pretrattamento con 20 μg/ml di proteinasi K a 37°C per 16 h, (B) trattamento termico a 80°C per 20 min, (C) filtrazione attraverso colonne di spin con pori cut-off MWCO come indicato. 3% (USA300 o Newman) o 10% (NCTC 8325) di supernatanti sono stati utilizzati come alte concentrazioni (barre nere), 0,01% (tutti i ceppi testati) sono stati utilizzati come basse concentrazioni (barre grigie). I dati sono presentati come mezzi ± SEM. Le differenze tra i campioni stimolati e i controlli del mezzo sono stati analizzati con un t-test di un campione. Le differenze tra le diverse condizioni di pretrattamento (A,B) sono state analizzate con un test di Mann-Whitney. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.
S. enterotossine enterotossine S. aureus modulano in modo dose-dipendente PMN-come MDSC
Coerente con i nostri risultati ottenuti per i supernatanti completi di S. aureus, enterotossina A (SEA) stafilococcica e enterotossina B (SEB) modulata in funzione della dose di PMN-like MDSC. Le potenze modulanti MDSC di SEA e SEB erano simili (Figura 5A). Inoltre, entrambe le enterotossine esercitavano effetti citotossici profondi e proliferativi sulle PBMC, con numeri di PMN-like MDSC in diminuzione e numeri totali di PBMC in aumento a concentrazioni più elevate. I modelli di variazioni dose-dipendenti dei numeri MDSC e PBMC totali mediate dalle enterotossine assomigliavano a quelli ottenuti da stimolazioni con l’uso di supernatanti completi di S. aureus (Figure 5B,C). Basse concentrazioni di SEA (0,1 ng/ml) indotte funzionali, PMN-MDSC soppressivo delle cellule T, come mostrato dai saggi di proliferazione CFSE (Figura 5D). Inoltre, se applicate contemporaneamente, le concentrazioni indotte da MDSC sono state superate dalle concentrazioni soppressive di S. aureus supernatanti e enterotossine stafilococciche, rispettivamente (Figura 5E). Gli effetti modulanti MDSC delle enterotossine erano termostabili, come mostrato per la SEA (Figura 5F).
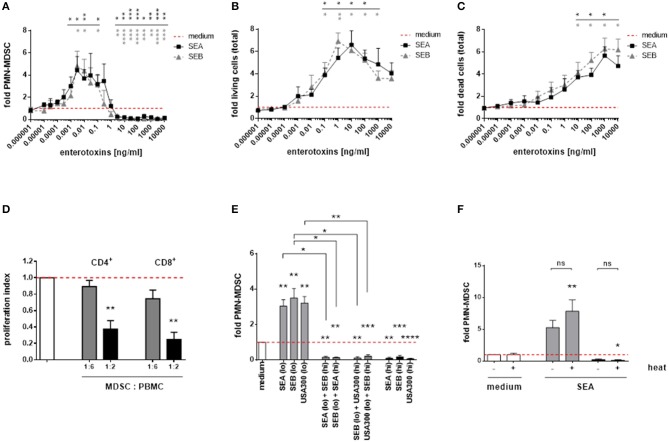
Figura 5.Le enterotossine stafilococciche modulano in modo dose-dipendente la formazione di PMN-come MDSC, la proliferazione e gli effetti citotossici in PBMC. (A)PMN-like MDSC sono stati quantificati dalla citometria a flusso. (B) La proliferazione del PBMC totale è stata valutata mediante colorazione blu di tripan. (C) Gli effetti citotossici nelle colture PBMC totali sono stati misurati mediante colorazione PI e successiva citometria a flusso. (D) Soppressione delle cellule T. PMN-come MDSC sono stati indotti dalla stimolazione di PBMC con SEA (0,1 ng / ml) per 7 giorni, e il test di proliferazione CFSE è stato eseguito come descritto in Figura 1. Le barre rappresentano l’indice di proliferazione. (E) Cross-inibizione della formazione di MDSC da enterotossine. Elevate concentrazioni di enterotossine hanno inibito l’induzione di PMN-like MDSC mediata dai surnatanti di S. aureus o da altre enterotossine. Le PBMC sono state stimolate con alte concentrazioni (3% di supernatanti USA300 o Newman, 5 ng/ml di SEA, o SEB, indicati come barre nere) e basse concentrazioni (0,01% di supernatanti USA300 o Newman, 0,1 ng/ml di SEA o SEB, barre grigie chiare), rispettivamente. Le stimolazioni miste sono indicate come barre grigio scuro. (F) Natura termostabile della MARE. Sia l’induzione mediata da SEA che la soppressione di PMN-come MDSC non sono state influenzate dal trattamento termico. SEA è stato riscaldato a 80 ° C per 20 minuti prima della stimolazione a 0,1 ng / ml (barre grigie) o a 10 ng / ml (barre nere). I dati sono presentati come mezzi ± SEM. Le differenze tra i campioni stimolati e controlli del mezzo sono stati analizzati da un t-test di un campione. Le differenze tra stimolazioni singole e miste (E) o tra campioni trattati termicamente e non trattati (F) sono state analizzate con un test Mann-Whitney. I risultati statistici sono mostrati a colori con riferimento alle rispettive curve. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.

Figura 5.Figura 5. Enterotossine stafilococciche dose-dipendentemente modulano la formazione di PMN-come MDSC, la proliferazione e gli effetti citotossici in PBMC. (A)PMN-like MDSC sono stati quantificati dalla citometria a flusso. (B) La proliferazione del PBMC totale è stata valutata mediante colorazione blu di tripan. (C) Gli effetti citotossici nelle colture PBMC totali sono stati misurati mediante colorazione PI e successiva citometria a flusso. (D) Soppressione delle cellule T. PMN-come MDSC sono stati indotti dalla stimolazione di PBMC con SEA (0,1 ng / ml) per 7 giorni, e il test di proliferazione CFSE è stato eseguito come descritto in Figura 1. Le barre rappresentano l’indice di proliferazione. (E) Cross-inibizione della formazione di MDSC da enterotossine. Elevate concentrazioni di enterotossine hanno inibito l’induzione di PMN-like MDSC mediata dai surnatanti di S. aureus o da altre enterotossine. Le PBMC sono state stimolate con alte concentrazioni (3% di supernatanti USA300 o Newman, 5 ng/ml di SEA, o SEB, indicati come barre nere) e basse concentrazioni (0,01% di supernatanti USA300 o Newman, 0,1 ng/ml di SEA o SEB, barre grigie chiare), rispettivamente. Le stimolazioni miste sono indicate come barre grigio scuro. (F) Natura termostabile della MARE. Sia l’induzione mediata da SEA che la soppressione di PMN-come MDSC non sono state influenzate dal trattamento termico. SEA è stato riscaldato a 80 ° C per 20 minuti prima della stimolazione a 0,1 ng / ml (barre grigie) o a 10 ng / ml (barre nere). I dati sono presentati come mezzi ± SEM. Le differenze tra i campioni stimolati e controlli del mezzo sono stati analizzati da un t-test di un campione. Le differenze tra stimolazioni singole e miste (E) o tra campioni trattati termicamente e non trattati (F) sono state analizzate con un test Mann-Whitney. I risultati statistici sono mostrati a colori con riferimento alle rispettive curve. *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.
La mutazione dei geni delle enterotossine diminuisce le capacità modulanti MDSC dei surnatanti di S. aureus
Per chiarire ulteriormente il ruolo delle enterotossine nella formazione di PMN-like MDSC, abbiamo usato mutanti transposon derivati dal ceppo S. aureus JE2, un derivato di USA300 LAC che era stato stabilito dalla polimerizzazione del plasmide. Il ceppo JE2 e i suoi mutanti sono stati presi dalla libreria di mutanti del Nebraska di ceppi di S. a ureus, che comprende circa 2.000 mutanti, ognuno dei quali ospita un inserto di transposon in un singolo gene distinto (Fey et al., 2013). Il genoma JE2 comprende i geni sek e seq e un gene selX-homolog, che codifica l’enterotossina stafilococcica K (SEK), l’enterotossina stafilococcica Q (SEQ) e la proteina SElX simile all’enterotossina. Contiene anche un gene che codifica una proteina con omologia ad altre enterotossine, denominata “enterotossina putativo di tipo A”. Quando viene utilizzato per la stimolazione di PBMC, il gene seq inattivato ha portato ad una potenza 100 volte inferiore di S. aureus supernatanti per modulare MDSC, mentre sek inattivato ha portato ad una potenza ~ 10 volte inferiore (Figura 6A). Al contrario, la mutagenesi transposon dei geni che codificano SElX o la presunta enterotossina di “tipo A”, che non è un omologo di SEA come determinato da analisi blastp, non ha influenzato i livelli di MDSC rispetto al wild-type (Figura 6B).
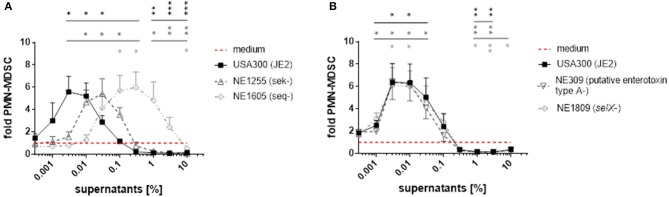
Figura 6.Le mutazioni in sek e seq influenzano le attività modulanti di PMN-like MDSC dei supernatanti di S. aureus. Per la stimolazione di PBMC, sono stati utilizzati supernatanti da ceppi JE2 che ospitano mutazioni del transposon nei geni sek, seq, selX e in un gene presunto di “tipo A” enterotossina. (A)PMN-come MDSCche induce e inibisce le capacità dei supernatanti JE2 sono stati colpiti da mutazioni nei geni sek e seq. (B) Le mutazioni nel selX e nel gene che codifica l’enterotossina “tipo A” non hanno avuto alcun effetto sui livelli di PMN-like MDSC. I dati rappresentano significa ± SEM. *p < 0,05; **p < 0,01; ***p < 0, 001.
I nostri risultati suggeriscono che le enterotossine stafilococciche sono componenti cruciali nei supernatanti S. aureus per la modulazione dei livelli MDSC, con l’enterotossina stafilococcica Q che è la più forte enterotossina modulante MDSC nei supernatanti USA300.

Figura 6.Figura 6. Le mutazioni in sek e seq influenzano le attività modulanti MDSC simili al PMN dei supernatanti di S. a ureus. Per la stimolazione di PBMC, sono stati utilizzati supernatanti da ceppi JE2 che ospitano mutazioni del transposon nei geni sek, seq, selX e in un presunto gene di enterotossina di tipo A. (A)PMN-come MDSCche induce e inibisce le capacità dei supernatanti JE2 sono stati colpiti da mutazioni nei geni sek e seq. (B) Le mutazioni nel selX e nel gene che codifica l’enterotossina “tipo A” non hanno avuto alcun effetto sui livelli di PMN-like MDSC. I dati rappresentano significa ± SEM. *p < 0,05; **p < 0,01; ***p < 0, 001.
La modulazione di PMN-like MDSC correla con l’espressione dell’enterotossina
Abbiamo esaminato il contenuto di enterotossine in modo semi-quantitativo nei supernatanti di USA300, Newman, COL e NCTC 8325 utilizzando la spettrometria di massa. Dopo la digestione della tripsina delle proteine, i peptidi risultanti sono stati allineati con le sequenze proteiche derivate da tutti i ceppi di S. aureus disponibili nel database UniProt. Nella Tabella 2 sono elencate tutte le proteine rilevate nei supernatanti e identificate come enterotossine stafilococciche (SEs) o proteine simili alle enterotossine (SEls). I peptidi derivati da otto SE e SEls possono essere identificati nei supernatanti di coltura del ceppo COL, dove il SEB si è rivelato essere l’enterotossina dominante. A Newman, si sono potuti individuare peptidi derivati da quattro SE o SEls, con SEA e SelX che sono le enterotossine più prominenti. Si è scoperto che USA300 esprimeva solo tre tipi di SE o SEls, rispettivamente. Il SEQ è apparso essere l’enterotossina principale in USA300, seguito dal SEK, in linea con la capacità di induzione di PMN-MDSC sostanzialmente ridotta del seq- mutante del substantialrain JE2 di USA300 (Figura 6A). Solo basse quantità di peptidi derivati da enterotossine sono state ottenute da supernatanti NCTC 8325.
| Intensità di LPQ | ||||||||
|---|---|---|---|---|---|---|---|---|
| Targhetta di localizzazione | Gene | Proteina | aa | kDa | USA300 | NCTC 8325 | COL | Newman |
| NWMN_0400 | mare (entA) | MARE | 257 | 29.7 | 0 | 0 | 0 | 1,259,600,000 |
| SACOL0907 | seb (entB) | SEB | 266 | 31.4 | 0 | 0 | 49,282,000,000 | 0 |
| G8RCC1 [b] | [a] | SEB-come [a,d] | [a] | [a] | 0 | 1,328,100 | 0 | 7,862,500 |
| SAUSA300_0800, SACOL0886 | sek | SEK | 242 | 27.8 | 118,420,000 | 0 | 4,856,160,000 | 7,460,700 |
| Q6G7U0 [b] | sek2 | SEK2 [e] | 242 | 27.8 | 0 | 0 | 7,080,700 | 0 |
| SAUSA300_0801, SACOL0887 | seq, sei | SEQ, SEI [f] | 242 | 28.2 | 849,400,000 | 0 | 2,840,600,000 | 0 |
| SAZ172_0832 [c] | sel | SEL | 256 | 29.8 | 0 | 0 | 741,110,000 | 0 |
| HMPREF0769_11854 [c] | seu | SEU | 261 | 30.5 | 0 | 0 | 2,741,200,000 | 0 |
| CH52_09990 [c] | yent1 | Yent1 [g] | 131 | 15.3 | 0 | 0 | 42,015,000 | 0 |
| SAUSA300_0370, SACOL0442, NWMN_0362 | selX | SElX | 203 | 23.2 | 16,568,000 | 0 | 26,516,000 | 3,726,600,000 |
| Totale LFQ | 984,388,000 | 1,328,100 | 60,536,681,700 | 5,001,523,200 | ||||
I nostri risultati mostrano che i ceppi di S. aureus con la più alta capacità funzionale di modulazione MDSC hanno mostrato il più alto contenuto totale di enterotossine determinato dalla spettrometria di massa. Un’intensità complessiva di LFQ di circa 60,5 miliardi di unità è stata determinata per le enterotossine nei supernatanti del COL che hanno indotto livelli massimi di PMN-like MDSC a concentrazioni dello 0,001% (vedi Figura 2), seguito da Newman (~5,0 miliardi di unità; induzione massima allo 0,003%), USA300 (~1,0 miliardi di unità; massimo allo 0,01%), e NCTC 8325 (~0,01 miliardi di unità; 10% di supernatante necessario per l’induzione massima MDSC). Si deve tener conto del fatto che per l’analisi in spettrometria di massa è stato necessario passare da RPMI a TSB per le colture stafilococciche, al fine di ottenere una densità batterica sufficientemente elevata per l’analisi delle proteine. Pertanto il contenuto di enterotossine non è direttamente correlato tra queste due condizioni di crescita, poiché il tasso di crescita di S. aureus influenza il fattore di virulenza/produzione di enterotossine (Derzelle et al., 2009).
Le enterotossine stafilococciche interferiscono sostanzialmente con i segnali che inducono MDSC
MDSC sono indotti da vari fattori di crescita endogeni, citochine e stimolanti microbici. GM-CSF è un importante fattore di crescita indotto da MDSC, che ha portato ad un aumento di circa 7 volte il numero di PMN-MDSC nei nostri esperimenti. La forte capacità di induzione di MDSC di GM-CSF è dose-dipendente soppressa dai surnatanti derivati da S. aureuse dalle enterotossine (Figure 7A,B). Inoltre, il PMN-MDSC che sopprime le concentrazioni di supernatanti e enterotossine di S. aureus ha anche abolito le notevoli capacità di induzione di MDSC dell’IL-2 e dei preparati derivati da diversi microbi, ad esempio i supernatanti di Pseudomonas aeruginosa o gli Aspergillus fumigatus lisati (Figure 7C-E). Pertanto proponiamo che le enterotossine di S. aureus abbiano un effetto dominante su una serie diversificata di fattori che inducono MDSC.
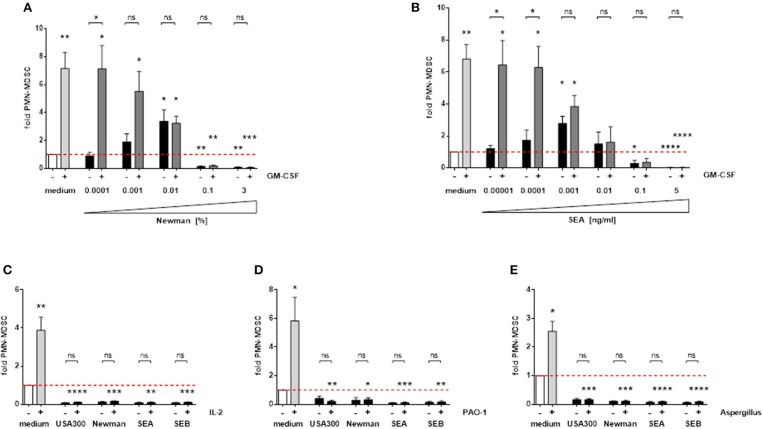
Figura 7.I surnatanti diS. aureus e le enterotossine interferiscono con altre sostanze che inducono MDSC. Le PBMC sono state stimolate simultaneamente con 10 ng/ml GM-CSF e concentrazioni crescenti di sopranatanti Newman (A) o SEA (B). Alte concentrazioni di S. aureus supernatanti (3%) o enterotossine (5 ng/ml) sono stati utilizzati per la co-stimolazione con IL-2 (100 U/ml) (C), P. aeruginosa PAO-1 supernatanti (1%) ( D) o Aspergillus fumigatus lisati (10 μg/ml) (E ). I dati rappresentano i mezzi ± SEM. Le differenze tra i controlli stimolati PBMC e i controlli medi (barre bianche) sono stati analizzati con un t-test di un campione. Le differenze tra stimolazioni singole e miste sono state analizzate da un test Mann-Whitney *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.

Figura 7.I surnatanti diS. aureus e le enterotossine interferiscono con altre sostanze che inducono MDSC. PBMC sono stati stimolati contemporaneamente con 10 ng/ml GM-CSF e aumentando le concentrazioni di sopranatanti Newman (A) o SEA (B). Alte concentrazioni di S. aureus supernatanti (3%) o enterotossine (5 ng/ml) sono stati utilizzati per la co-stimolazione con IL-2 (100 U/ml) (C), P. aeruginosa PAO-1 supernatanti (1%) ( D) o Aspergillus fumigatus lisati (10 μg/ml) (E ). I dati rappresentano i mezzi ± SEM. Le differenze tra i controlli stimolati PBMC e i controlli medi (barre bianche) sono stati analizzati con un t-test di un campione. Le differenze tra stimolazioni singole e miste sono state analizzate da un test Mann-Whitney *p < 0,05; **p < 0,01; ***p < 0,001; ****p < 0, 0001; ns-non significativo.
Discussione
In questo studio abbiamo analizzato sistematicamente la generazione di MDSC su stimolazione con ceppi multipli di S. aureus e i loro principali fattori di virulenza. Abbiamo dimostrato per la prima volta che le enterotossine stafilococciche modulano in modo dose-dipendente la generazione di PMN-like MDSC. Concentrazioni più basse di enterotossine indotte da PMN-like MDSC, mentre concentrazioni più elevate inibiscono la generazione di PMN-like MDSC e aboliscono in modo impressionante l’induzione di MDSC da GM-CSF, P. aeruginosa, A. fumigatus e IL-2.
Questi risultati dipendenti dalla concentrazione di enterotossine stafilococciche sono eccezionali se confrontati con altre interazioni patogeno-MDSC. Finora, tutti gli agenti patogeni riportati e i fattori di virulenza associati hanno portato ad un’induzione o inibizione di MDSC (Ost et al., 2016). Esempi di induzione includono, ma non sono limitati a, S. aureus, Pseudomonas aeruginosa/flagellin, Klebsiella pneumonia, o funghi patogeni (Poe et al., 2013; Rieber et al., 2013, 2015; Heim et al., 2014; Skabytska et al., 2014; Tebartz et al., 2015; Szabo et al., 2016). Per l’agonista TLR3 Poly (I:C) (Ho et al., 2015) è stato osservato un effetto inibitorio come si è visto con le enterotossine stafilococciche a concentrazioni maggiori. La stimolazione di TLR9 con oligonucleotidi CpG ha indotto la maturazione di M-MDSC e ha portato ad una perdita della loro funzione immunosoppressiva (Zoglmeier et al., 2011; Shirota et al., 2012). Una combinazione di TLR7, 8 e 9 ligandi ha migliorato le risposte antitumorali delle cellule NK e delle cellule T citotossiche e ha ridotto la frequenza delle MDSC (Zhao et al., 2014).
Come interagiscono le enterotossine stafilococciche con l’MDSC a livello molecolare? Esiste un meccanismo citotossico specifico per l’MDSC? È stato dimostrato che le enterotossine stafilococciche interagiscono principalmente con le cellule che presentano l’antigene professionale tramite MHC di classe II, portando alla loro attivazione e alla produzione di citochine e chemochine proinfiammatorie (Pinchuk et al., 2010). A nostra conoscenza, non è stato ancora identificato alcun meccanismo citotossico diretto di enterotossine stafilococciche. Tuttavia, MDSC potrebbe presentare recettori unici o una suscettibilità altrimenti specificata alle enterotossine stafilococciche citotossiche rispetto ad altre cellule mieloidi e linfociti. Un’altra possibile modalità d’azione di questi super-antigeni sarebbe indiretta, ad esempio, da una massiccia secrezione di citochine nell’ambiente cellulare che circonda l’MDSC, uno scenario a volte chiamato tempesta di citochine, che potrebbe portare ad un’apoptosi/necroptosi accelerata dell’MDSC. Con l’aumento degli effetti tossici, il PMN-come MDSC è diminuito, mentre il numero totale di PBMC inizialmente è aumentato ulteriormente. Tuttavia, a causa della regione di gating utilizzata nei nostri esperimenti per la valutazione dei numeri di PMN-like MDSC, riteniamo improbabile che la diminuzione di MDSC sia semplicemente causata da un aumento concomitante di altri tipi di cellule e può quindi essere solo una diminuzione relativa. L’unico rapporto precedente sull’interazione tra enterotossine stafilococciche e MDSC di Szabo et al. ha descritto un rapido afflusso di PMN-MDSC nel fegato del topo dopo l’inoculazione di enterotossina stafilococcica B. Gli autori hanno concluso che questo rapido afflusso non era dovuto alla proliferazione o alla generazione di MDSC, ma piuttosto a segnali di homing al fegato dal midollo osseo. Le vie esatte, tuttavia, sono una questione di indagine futura (Szabo et al., 2016). Un effetto citotossico delle enterotossine sull’MDSC non è stato descritto in quello studio.
I superantigeni di S. aureus innescano una proliferazione esaustiva delle cellule T policlonali nell’organismo infetto. I nostri risultati indicano un percorso di rinforzo che supporta questo meccanismo abrogando le cellule T soppressive MDSC se la densità delle cellule batteriche è abbastanza alta e le enterotossine sono secrete in concentrazioni considerevoli. Questo meccanismo potrebbe ricordare il ben definito sistema di rilevamento del quorum di S. aureus, cioè la regolazione del gene dipendente dalla densità cellulare mediata dal sistema accessorio di regolazione del gene (Agr) (Peschel e Otto, 2013). Nel nostro studio non abbiamo analizzato i percorsi di segnalazione che sono coinvolti nell’interazione tra enterotossine e MDSC. Tuttavia, questo non rientrava nell’ambito di questo studio iniziale, ma è attualmente oggetto di indagine all’interno del nostro gruppo. Inoltre, la valutazione dei nostri risultati in modelli in vivo e la differenziazione dell’interazione tra S. aureus e MDSC tra diversi compartimenti d’organo sarà di particolare importanza per gli studi futuri.
Le cellule T CD4 attivate hanno dimostrato di promuovere la patogenicità di S. a ureus (Parker et al., 2015). Pertanto, proponiamo che l’inibizione della generazione di MDSC rappresenti un meccanismo patogeno di S . aureus di nuova definizione. Bloccare le enterotossine nelle infezioni invasive di S. a ureus o il trasferimento adottivo di MDSC potrebbe migliorare le reazioni iperinfiammatorie a S. aureus. D’altra parte, le enterotossine potrebbero essere di interesse terapeutico per prevenire la formazione di MDSC. Soprattutto nei pazienti affetti da tumore, dove le MDSC sono costantemente indotte e indeboliscono la risposta immunitaria innata e adattiva anti-tumorale, l’inibizione delle MDSC sarebbe vantaggiosa. La messa a punto del rapporto bifasico tra S. aureus enterotossine e MDSC in vivo sarà impegnativo e richiederà studi approfonditi in modelli animali per prevenire gli effetti dannosi. Un effetto piuttosto dominante delle enterotossine sui segnali che inducono MDSC è stato dimostrato nei nostri esperimenti di co-cultura con le molecole che inducono MDSC associate al tumore GM-CSF (Lechner et al., 2010), IL-2 (Rodriguez et al., 2009) e Pseudomonas- (Rieber et al., 2013) e Aspergillus- (Rieber et al., 2015). Tuttavia, per gli approcci terapeutici sarebbe fondamentale definire meglio il dominio responsabile delle enterotossine per questo effetto abrogante MDSC e biochimicamente ingegnerizzare queste molecole per eliminare gli effetti collaterali gravi esistenti di queste tossine. L’interazione tra l’abrogazione di S. aureus enterotossine correlate a S. a ureus e l’induzione di MDSC correlata a Pseudomonas (Rieber et al., 2013) evidenzia ancora una volta le condizioni fisiopatologiche specifiche per le frequenti co-infezioni con diversi agenti patogeni in malattie sfaccettate come la fibrosi cistica.
Presi insieme, abbiamo identificato le enterotossine stafilococciche come principali modulatori della generazione di MDSC. L’interazione delle enterotossine stafilococciche con le cellule soppressori di derivazione mieloide potrebbe giocare un ruolo importante nella reazione infiammatoria di sovrashooting spesso vista nelle infezioni sistemiche di S. aureus e potrebbe rappresentare un nuovo bersaglio terapeutico nelle infezioni di S. aureus e oltre.
Dichiarazione di etica
Questo studio è stato condotto secondo le raccomandazioni del comitato etico dell’Università di Tubinga. Tutti i soggetti hanno dato il loro consenso informato per iscritto secondo la Dichiarazione di Helsinki e il comitato etico dell’Università di Tubinga ha approvato il protocollo di studio.
Contributi degli autori
HS ha progettato ed eseguito esperimenti, ha analizzato i dati e ha co-scritto il manoscritto. MO e RM hanno condotto esperimenti e analizzato i dati. AS ha eseguito e supervisionato gli esperimenti, ha discusso i dati e ha revisionato il manoscritto. DN ha discusso i dati e ha aiutato con gli esperimenti. È stato aiutato con gli esperimenti. AV e BM hanno eseguito l’analisi delle proteine. DK e CW hanno generato specifici supernatanti di coltura e purificato i prodotti di S. aureus e hanno discusso i dati. AH, RH, FG e AP hanno fornito una guida allo studio e hanno rivisto il manoscritto. DH ha co-progettato lo studio, ha discusso i dati e ha rivisto il manoscritto. NR ha co-progettato lo studio, ha supervisionato gli esperimenti, ha analizzato e discusso i dati e ha scritto il manoscritto.
Dichiarazione di conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che possano essere interpretate come un potenziale conflitto di interessi. Il revisore JM e l’editor di gestione hanno dichiarato la loro comune affiliazione.
References
- Baba T., Bae T., Schneewind O., Takeuchi F., Hiramatsu K.. Genome sequence of Staphylococcus aureus strain Newman and comparative analysis of staphylococcal genomes: polymorphism and evolution of two major pathogenicity islands. J. Bacteriol.. 2008; 190:300-310. DOI | PubMed
- Baba T., Takeuchi F., Kuroda M., Yuzawa H., Aoki K., Oguchi A.. Genome and virulence determinants of high virulence community-acquired MRSA. Lancet. 2002; 359:1819-1827. DOI | PubMed
- Baek K. T., Frees D., Renzoni A., Barras C., Rodriguez N., Manzano C.. Genetic variation in the Staphylococcus aureus 8325 strain lineage revealed by whole-genome sequencing. PLoS ONE. 2013; 8:e77122. DOI | PubMed
- Bronte V., Brandau S., Chen S. H., Colombo M. P., Frey A. B., Greten T. F.. Recommendations for myeloid-derived suppressor cell nomenclature and characterization standards. Nat. Commun.. 2016; 7:12150. DOI | PubMed
- Bubeck Wardenburg J., Bae T., Otto M., Deleo F. R., Schneewind O.. Poring over pores: alpha-hemolysin and Panton-Valentine leukocidin in Staphylococcus aureus pneumonia. Nat. Med.. 2007; 13:1405-1406. DOI | PubMed
- Burian M., Velic A., Matic K., Günther S., Kraft B., Gonser L.. Quantitative proteomics of the human skin secretome reveal a reduction in immune defense mediators in ectodermal dysplasia patients. J. Invest. Dermatol.. 2015; 135:759-767. DOI | PubMed
- Cheung G. Y., Wang R., Khan B. A., Sturdevant D. E., Otto M.. Role of the accessory gene regulator agr in community-associated methicillin-resistant Staphylococcus aureus pathogenesis. Infect. Immun.. 2011; 79:1927-1935. DOI | PubMed
- Cox J., Mann M.. MaxQuant enables high peptide identification rates, individualized p.p.b.-range mass accuracies and proteome-wide protein quantification. Nat. Biotechnol.. 2008; 26:1367-1372. DOI | PubMed
- Derzelle S., Dilasser F., Duquenne M., Deperrois V.. Differential temporal expression of the staphylococcal enterotoxins genes during cell growth. Food Microbiol.. 2009; 26:896-904. DOI | PubMed
- Diep B. A., Gill S. R., Chang R. F., Phan T. H., Chen J. H., Davidson M. G., Perdreau-Remington. Complete genome sequence of USA300, an epidemic clone of community-acquired meticillin-resistant Staphylococcus aureus. Lancet. 2006; 367:731-739. DOI | PubMed
- Diep B. A., Palazzolo-Ballance A. M., Tattevin P., Basuino L., Braughton K. R., Whitney A. R.. Contribution of Panton-Valentine leukocidin in community-associated methicillin-resistant Staphylococcus aureus pathogenesis. PLoS ONE. 2008a; 3:e3198. DOI | PubMed
- Diep B. A., Stone G. G., Basuino L., Graber C. J., Miller A., des Etages S. A.. The arginine catabolic mobile element and staphylococcal chromosomal cassette mec linkage: convergence of virulence and resistance in the USA300 clone of methicillin-resistant Staphylococcus aureus. J. Infect. Dis.. 2008b; 197:1523-1530. DOI | PubMed
- du Plessis N., Loebenberg L., Kriel M., von Groote-Bidlingmaier Ribechini E., Loxton A. G.. Increased frequency of myeloid-derived suppressor cells during active tuberculosis and after recent mycobacterium tuberculosis infection suppresses T-cell function. Am. J. Respir. Crit. Care Med.. 2013; 188:724-732. DOI | PubMed
- Dumitru C. A., Moses K., Trellakis S., Lang S., Brandau S.. Neutrophils and granulocytic myeloid-derived suppressor cells: immunophenotyping, cell biology and clinical relevance in human oncology. Cancer Immunol. Immunother.. 2012; 61:1155-1167. DOI | PubMed
- Fey P. D., Endres J. L., Yajjala V. K., Widhelm T. J., Boissy R. J., Bose J. L.. A genetic resource for rapid and comprehensive phenotype screening of nonessential Staphylococcus aureus genes. MBio. 2013; 4:e00537-12. DOI | PubMed
- Gabrilovich D. I., Nagaraj S.. Myeloid-derived suppressor cells as regulators of the immune system. Nat. Rev. Immunol.. 2009; 9:162-174. DOI | PubMed
- Genestier A. L., Michallet M. C., Prévost G., Bellot G., Chalabreysse L., Peyrol S.. Staphylococcus aureus Panton-Valentine leukocidin directly targets mitochondria and induces Bax-independent apoptosis of human neutrophils. J. Clin. Invest.. 2005; 115:3117-3127. DOI | PubMed
- Gill S. R., Fouts D. E., Archer G. L., Mongodin E. F., Deboy R. T., Ravel J.. Insights on evolution of virulence and resistance from the complete genome analysis of an early methicillin-resistant Staphylococcus aureus strain and a biofilm-producing methicillin-resistant Staphylococcus epidermidis strain. J. Bacteriol.. 2005; 187:2426-2438. DOI | PubMed
- Green E. R., Mecsas J.. Bacterial secretion systems: an overview. Microbiol. Spectr.. 2016; 4:215-239. DOI | PubMed
- Heim C. E., Vidlak D., Scherr T. D., Kozel J. A., Holzapfel M., Muirhead D. E.. Myeloid-derived suppressor cells contribute to Staphylococcus aureus orthopedic biofilm infection. J. Immunol.. 2014; 192:3778-3792. DOI | PubMed
- Herbert S., Ziebandt A. K., Ohlsen K., Schäfer T., Hecker M., Albrecht D.. Repair of global regulators in Staphylococcus aureus 8325 and comparative analysis with other clinical isolates. Infect. Immun.. 2010; 78:2877-2889. DOI | PubMed
- Ho V., Lim T. S., Lee J., Steinberg J., Szmyd R., Tham M.. TLR3 agonist and Sorafenib combinatorial therapy promotes immune activation and controls hepatocellular carcinoma progression. Oncotarget. 2015; 6:27252-27266. DOI | PubMed
- Iordanescu S., Surdeanu M.. Two restriction and modification systems in Staphylococcus aureus NCTC8325. J. Gen. Microbiol.. 1976; 96:277-281. DOI | PubMed
- Joshi G. S., Spontak J. S., Klapper D. G., Richardson A. R.. Arginine catabolic mobile element encoded speG abrogates the unique hypersensitivity of Staphylococcus aureus to exogenous polyamines. Mol. Microbiol.. 2011; 82:9-20. DOI | PubMed
- Kreiswirth B. N., Löfdahl S., Betley M. J., O’Reilly M., Schlievert P. M., Bergdoll M. S.. The toxic shock syndrome exotoxin structural gene is not detectably transmitted by a prophage. Nature. 1983; 305:709-712. DOI | PubMed
- Kuroda M., Ohta T., Uchiyama I., Baba T., Yuzawa H., Kobayashi I.. Whole genome sequencing of meticillin-resistant Staphylococcus aureus. Lancet. 2001; 357:1225-1240. DOI | PubMed
- Lechner M. G., Liebertz D. J., Epstein A. L.. Characterization of cytokine-induced myeloid-derived suppressor cells from normal human peripheral blood mononuclear cells. J. Immunol.. 2010; 185:2273-2284. DOI | PubMed
- Lowy F. D.. Staphylococcus aureus infections. N. Engl. J. Med.. 1998; 339:520-532. DOI | PubMed
- Luber C. A., Cox J., Lauterbach H., Fancke B., Selbach M., Tschopp J.. Quantitative proteomics reveals subset-specific viral recognition in dendritic cells. Immunity. 2010; 32:279-289. DOI | PubMed
- Marigo I., Dolcetti L., Serafini P., Zanovello P., Bronte V.. Tumor-induced tolerance and immune suppression by myeloid derived suppressor cells. Immunol. Rev.. 2008; 222:162-179. DOI | PubMed
- McDougal L. K., Steward C. D., Killgore G. E., Chaitram J. M., McAllister S. K., Tenover F. C.. Pulsed-field gel electrophoresis typing of oxacillin-resistant Staphylococcus aureus isolates from the United States: establishing a national database. J. Clin. Microbiol.. 2003; 41:5113-5120. DOI | PubMed
- Novick R.. Properties of a cryptic high-frequency transducing phage in Staphylococcus aureus. Virology. 1967; 33:155-166. DOI | PubMed
- Novick R. P.. Autoinduction and signal transduction in the regulation of staphylococcal virulence. Mol. Microbiol.. 2003; 48:1429-1449. DOI | PubMed
- Olsen J. V., de Godoy L. M., Li G., Macek B., Mortensen P., Pesch R.. Parts per million mass accuracy on an Orbitrap mass spectrometer via lock mass injection into a C-trap. Mol. Cell. Proteomics MCP. 2005; 4:2010-2021. DOI | PubMed
- Ost M., Singh A., Peschel A., Mehling R., Rieber N., Hartl D.. Myeloid-derived suppressor cells in bacterial infections. Front. Cell. Infect. Microbiol.. 2016; 6:37. DOI | PubMed
- Parker D., Ryan C. L., Alonzo F., Torres V. J., Planet P. J., Prince A. S.. CD4+ T cells promote the pathogenesis of Staphylococcus aureus pneumonia. J. Infect. Dis.. 2015; 211:835-845. DOI | PubMed
- Peng H. L., Novick R. P., Kreiswirth B., Kornblum J., Schlievert P.. Cloning, characterization, and sequencing of an accessory gene regulator (agr) in Staphylococcus aureus. J. Bacteriol.. 1988; 170:4365-4372. DOI | PubMed
- Peng K. T., Hsieh C. C., Huang T. Y., Chen P. C., Shih H. N., Lee M. S.. Staphylococcus aureus biofilm elicits the expansion, activation and polarization of myeloid-derived suppressor cells in vivo and in vitro. PLoS ONE. 2017; 12:e0183271. DOI | PubMed
- Peschel A., Otto M.. Phenol-soluble modulins and staphylococcal infection. Nat. Rev. Microbiol.. 2013; 11:667-673. DOI | PubMed
- Pinchuk I. V., Beswick E. J., Reyes V. E.. Staphylococcal enterotoxins. Toxins. 2010; 2:2177-2197. DOI | PubMed
- Poe S. L., Arora M., Oriss T. B., Yarlagadda M., Isse K., Khare A.. STAT1-regulated lung MDSC-like cells produce IL-10 and efferocytose apoptotic neutrophils with relevance in resolution of bacterial pneumonia. Mucosal Immunol.. 2013; 6:189-199. DOI | PubMed
- Rieber N., Brand A., Hector A., Graepler-Mainka U., Ost M., Schäfer I.. Flagellin induces myeloid-derived suppressor cells: implications for Pseudomonas aeruginosa infection in cystic fibrosis lung disease. J. Immunol.. 2013; 190:1276-1284. DOI | PubMed
- Rieber N., Singh A., Öz H., Carevic M., Bouzani M., Amich J.. Pathogenic fungi regulate immunity by inducing neutrophilic myeloid-derived suppressor cells. Cell Host Microbe. 2015; 17:507-514. DOI | PubMed
- Rodriguez P. C., Ernstoff M. S., Hernandez C., Atkins M., Zabaleta J., Sierra R.. Arginase I-producing myeloid-derived suppressor cells in renal cell carcinoma are a subpopulation of activated granulocytes. Cancer Res.. 2009; 69:1553-1560. DOI | PubMed
- Rosenstein R., Nerz C., Biswas L., Resch A., Raddatz G., Schuster S. C.. Genome analysis of the meat starter culture bacterium Staphylococcus carnosus TM300. Appl. Environ. Microbiol.. 2009; 75:811-822. DOI | PubMed
- Schmielau J., Finn O. J.. Activated granulocytes and granulocyte-derived hydrogen peroxide are the underlying mechanism of suppression of t-cell function in advanced cancer patients. Cancer Res.. 2001; 61:4756-4760. PubMed
- Schreiner J., Kretschmer D., Klenk J., Otto M., Bühring H. J., Stevanovic S.. Staphylococcus aureus phenol-soluble modulin peptides modulate dendritic cell functions and increase in vitro priming of regulatory T cells. J. Immunol.. 2013; 190:3417-3426. DOI | PubMed
- Shirota Y., Shirota H., Klinman D. M.. Intratumoral injection of CpG oligonucleotides induces the differentiation and reduces the immunosuppressive activity of myeloid-derived suppressor cells. J. Immunol.. 2012; 188:1592-1599. DOI | PubMed
- Shore A. C., Rossney A. S., Brennan O. M., Kinnevey P. M., Humphreys H., Sullivan D. J.. Characterization of a novel arginine catabolic mobile element (ACME) and staphylococcal chromosomal cassette mec composite island with significant homology to Staphylococcus epidermidis ACME type II in methicillin-resistant Staphylococcus aureus genotype ST22-MRSA-IV. Antimicrob. Agents Chemother.. 2011; 55:1896-1905. DOI | PubMed
- Skabytska Y., Wölbing F., Günther C., Koberle M., Kaesler S., Chen K. M.. Cutaneous innate immune sensing of Toll-like receptor 2-6 ligands suppresses T cell immunity by inducing myeloid-derived suppressor cells. Immunity. 2014; 41:762-775. DOI | PubMed
- Stover C. K., Pham X. Q., Erwin A. L., Mizoguchi S. D., Warrener P., Hickey M. J.. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen. Nature. 2000; 406:959-964. DOI | PubMed
- Szabo P. A., Goswami A., Memarnejadian A., Mallett C. L., Foster P. J., McCormick J. K.. Swift intrahepatic accumulation of granulocytic myeloid-derived suppressor cells in a humanized mouse model of toxic shock syndrome. J. Infect. Dis.. 2016; 213:1990-1995. DOI | PubMed
- Tebartz C., Horst S. A., Sparwasser T., Huehn J., Beineke A., Peters G.. A major role for myeloid-derived suppressor cells and a minor role for regulatory T cells in immunosuppression during Staphylococcus aureus infection. J. Immunol.. 2015; 194:1100-1111. DOI | PubMed
- Travassos L. H., Girardin S. E., Philpott D. J., Blanot D., Nahori M. A., Werts C.. Toll-like receptor 2-dependent bacterial sensing does not occur via peptidoglycan recognition. EMBO Rep.. 2004; 5:1000-1006. DOI | PubMed
- Wang R., Braughton K. R., Kretschmer D., Bach T. H., Queck S. Y., Li M.. Identification of novel cytolytic peptides as key virulence determinants for community-associated MRSA. Nat. Med.. 2007; 13:1510-1514. DOI | PubMed
- Winstel V., Liang C., Sanchez-Carballo P., Steglich M., Munar M., Bröker B. M.. Wall teichoic acid structure governs horizontal gene transfer between major bacterial pathogens. Nat. Commun.. 2013; 4:2345. DOI | PubMed
- Wirtz C., Witte W., Wolz C., Goerke C.. Transcription of the phage-encoded Panton-Valentine leukocidin of Staphylococcus aureus is dependent on the phage life-cycle and on the host background. Microbiology. 2009; 155:3491-3499. DOI | PubMed
- Zhao B. G., Vasilakos J. P., Tross D., Smirnov D., Klinman D. M.. Combination therapy targeting toll like receptors 7, 8 and 9 eliminates large established tumors. J. Immunother. Cancer. 2014; 2:12. DOI | PubMed
- Zoglmeier C., Bauer H., Nörenberg D., Wedekind G., Bittner P., Sandholzer N.. CpG blocks immunosuppression by myeloid-derived suppressor cells in tumor-bearing mice. Clin. Cancer Res.. 2011; 17:1765-1775. DOI | PubMed
Fonte
Stoll H, Ost M, Singh A, Mehling R, Neri D, et al. (2018) Staphylococcal Enterotoxins Dose-Dependently Modulate the Generation of Myeloid-Derived Suppressor Cells. Frontiers in Cellular and Infection Microbiology 8321. https://doi.org/10.3389/fcimb.2018.00321





