Abstract
Introduzione
Si stima che ci siano1031 particelle simili a virus che abitano il nostro pianeta, in numero superiore a tutte le forme di vita cellulari(Suttle, 2005; Wigington et al., 2016). Nonostante la loro presenza in numero sorprendente e il loro impatto sulle dinamiche della popolazione e sulle traiettorie evolutive dei loro ospiti, la nostra conoscenza quantitativa delle tendenze delle proprietà genomiche dei virus rimane in gran parte limitata, con molte delle quantità chiave utilizzate per caratterizzare questi genomi sparse in tutta la letteratura o non disponibili del tutto. Questo è in contrasto con la crescente capacità esibita in risorse come il database BioNumbers(Milo et al., 2010) di assemblare in un’unica raccolta curata i numeri chiave che caratterizzano le forme di vita cellulari. Il nostro obiettivo è stato quello di integrare questi database di numeri chiave della biologia cellulare(Milo et al., 2010; Phillips et al., 2012; Milo e Phillips, 2015; Phillips e Milo , 2009) con i dati corrispondenti dei virus. Con l’avvento delle tecnologie di sequenziamento ad alta velocità, recenti studi hanno permesso di effettuare indagini genomiche e metagenomiche di numerosi habitat naturali, slegandoci dagli organismi che conosciamo e amiamo e dandoci accesso a un mare di dati genomici provenienti da nuovi organismi(Paez-Espino et al., 2016). Tali progressi ci permettono di apprezzare la diversità genomica che caratterizza i genomi virali(Paez-Espino et al., 2016; Edwards e Rohwer, 2005; Rohwer e Thurber, 2009; Simmonds et al., 2017; Simmonds, 2015; Mokili et al., 2012) e permettono ora di assemblare alcuni dei numeri chiave della virologia.
A differenza dei genomi cellulari, che sono universalmente codificati nel linguaggio del DNA a doppio filamento (dsDNA), i genomi dei virus sono straordinariamente versatili. I genomi virali possono essere trovati come versioni a singolo o doppio filamento di DNA e RNA, confezionati in segmenti o in un unico pezzo, e presenti sia in forma lineare che circolare. Inoltre, in base ai loro rapidi cicli infettivi, alle grandi dimensioni delle esplosioni e alla replicazione spesso altamente soggetta a errori, i virus rilevano collettivamente un ampio spazio di sequenza genomica e comprendono una grande porzione della diversità genomica totale ospitata dal nostro pianeta(Kristensen et al., 2010; Hendrix, 2003). Recentemente, attraverso un ampio studio delle sequenze metagenomiche, lo spazio di sequenza virale conosciuto è stato aumentato di un ordine di grandezza(Paez-Espino et al., 2016), e molto più della “materia oscura” virale rimane probabilmente inesplorata (Youle etal., 2012).
Nell’analizzare uno spettro crescente di dati di sequenza, ci troviamo di fronte a una sfida considerevole che è unica per i virus, vale a dire, come trovare quelle caratteristiche all’interno dei genomi virali che potrebbero rivelare aspetti nascosti della loro storia evolutiva. Per mettere questa sfida in prospettiva, quando si analizzano dati non virali, si utilizzano marcatori universali dell’RNA ribosomiale come le sequenze 16S per classificare gli organismi appena scoperti e per localizzarli sull’albero evolutivo della vita(Hug et al., 2016). I genomi dei virus, invece, sono altamente divergenti e non possiedono tali sequenze universalmente condivise (Kristensenet al., 2011).
In assenza di marcatori genomici universali, i virus sono stati storicamente classificati sulla base di una varietà di attributi, forse in particolare di caratteristiche morfologiche, proposti nel 1962 dall’International Committee on Taxonomy of Viruses o ICTV(King et al., 2011), o sulla base delle diverse modalità con cui producono mRNA, proposte da David Baltimore nel 1971(Baltimore, 1971;Figura 1). L’ICTV classifica i virus in sette ordini: Herpesvirales, virus a DNA a doppio filamento eucariotico di grandi dimensioni; Caudovirales, virus a DNA a doppio filamento a coda coda tipicamente infettanti i batteri; Ligamenvirales, virus lineari a doppio filamento che infettano gli arcaici; Mononegavirales, virus a RNA a filamento singolo non segmentato negativo (o antisenso) a filamento singolo di piante e animali; Nidovirales, virus RNA a filamento singolo a filamento singolo positivo (o sensitivo) a filamento singolo dei vertebrati; Picornavirales, virus RNA a filamento singolo a filamento piccolo positivo che infettano piante, insetti e animali; e infine, i Tymovirales, virus RNA a filamento singolo positivo monopartito delle piante. Oltre a questi ordini, ci sono famiglie ICTV, alcune delle quali non sono state assegnate a un ordine ICTV. Vengono esplorate solo quelle famiglie virali ICTV con più di pochi membri presenti nel nostro dataset.
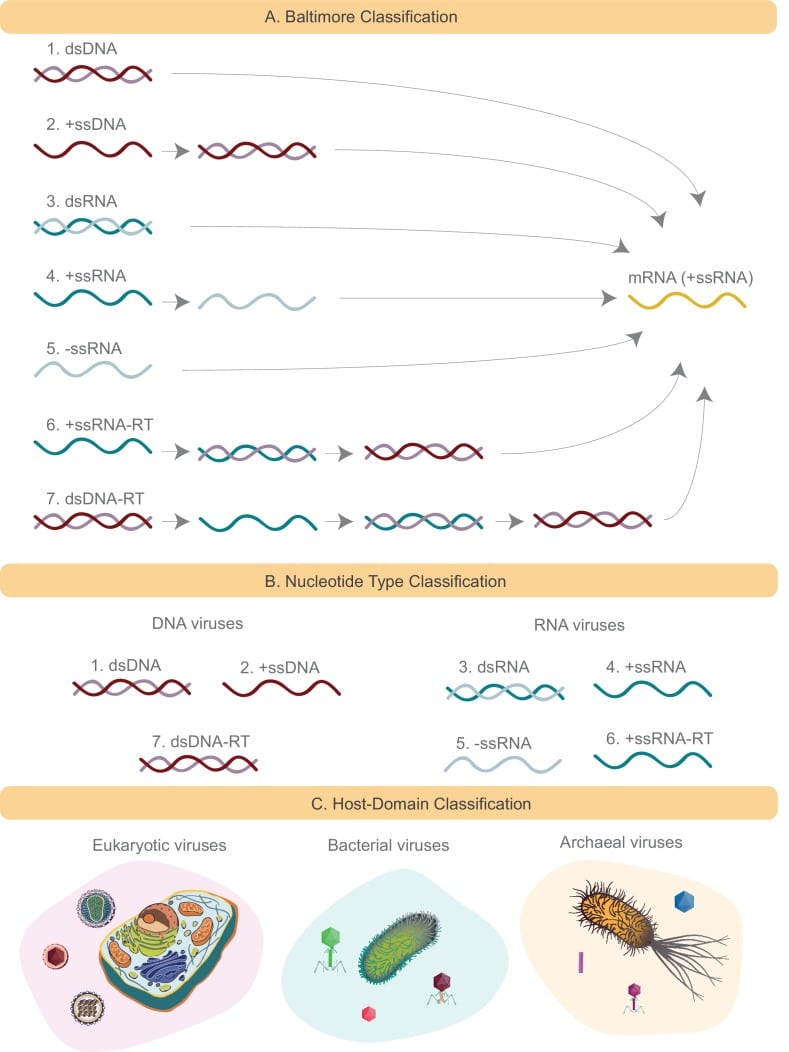
Figura 1.Schemi di diversi sistemi di classificazione virale esplorati in questo studio.(A) La classificazione di Baltimora divide tutti i virus in sette gruppi in base a come viene prodotto l’mRNA virale. I filamenti di DNA sono indicati in rosso (+ssDNA in una tonalità più scura di rosso rispetto a -ssDNA). Allo stesso modo i filamenti di RNA sono indicati in verde (+ssRNA in una tonalità più scura di verde rispetto a -ssRNA). Nel caso di Baltimora gruppi 1,2,6, e 7, il genoma è o è convertito in dsDNA, che viene poi convertito in mRNA attraverso l’azione di DNA-dipendente RNA polimerasi RNA. Nel caso di Baltimora gruppi 3, 4 e 5, il genoma è o è convertito in +ssRNA, che è mRNA, che è mRNA, attraverso l’azione di RNA polimerasi RNA-dipendente.(B) La classificazione del tipo di nucleotide divide i virus in base al loro materiale genomico in DNA e virus RNA. Baltimora gruppi virali 1, 2 e 7 sono tutti considerati virus del DNA, e i restanti gruppi virali sono considerati virus RNA.(C) La classificazione del dominio host raggruppa i virus in base al dominio host che infettano. Si formano tre gruppi: virus eucarioti, batterici e arcaici.
La classificazione di Baltimora raggruppa i virus in sette categorie(Figura 1): virus DNA a doppio filamento (Gruppo I); virus DNA a singolo filamento (Gruppo II); virus RNA a doppio filamento (Gruppo III); virus RNA positivi a singolo filamento (Gruppo IV); virus RNA negativi a singolo filamento (Gruppo V); virus RNA positivi a singolo filamento con intermedi del DNA (Gruppo VI), comunemente noti come retrovirus; e, i retrovirus a doppio filamento (Gruppo VII).
Data la prevalenza di questi sistemi di classificazione virale nella categorizzazione dei virus oggi, vale la pena ricordare che il loro inizio è anteriore al sequenziamento del primo genoma nel 1976. Con i tassi di sequenziamento più veloci ed economici disponibili fino ad oggi, viviamo in un momento opportuno per esplorare le proprietà genomiche virali e valutare questi sistemi di classificazione esistenti alla luce del crescente corpo di informazioni sulla sequenza.
Oltre alle classificazioni ICTV e Baltimora abbiamo utilizzato un semplice sistema di classificazione basato sulle informazioni del dominio host, e abbiamo diviso i virus in virus batterici, archeologici ed eucariotici(Figura 1). La motivazione alla base di questo tipo di classificazione è l’ipotesi della rivoluzione(Mahy e Van Regenmortel, 2010; Forterre, 2010). I virus sono organismi obbligati che non sono in grado di sopravvivere senza il loro ospite, e come corollario si ipotizza che essi abbiano coevoluto con i loro ospiti mentre gli ospiti si sono allontanati per miliardi di anni per formare i tre domini della vita(Mahy e Van Regenmortel, 2010; Forterre, 2010). Una possibile prova a sostegno di questa ipotesi è che finora non sono state osservate infezioni di host di un dominio da parte di virus di un altro dominio. Abbiamo anche esplorato un sistema di classificazione minimo che divide il mondo dei virus in due gruppi in base al loro tipo di nucleotide (RNA e DNA), qui chiamato “Nucleotide Type” (Figura 1). Questa classificazione viene introdotta come una versione semplificata del sistema di classificazione di Baltimora. In pratica, abbiamo assegnato i gruppi di Baltimora 1, 2 e 7 alla categoria virale del DNA, e i restanti gruppi di Baltimora alla categoria virale dell’RNA.
Sebbene molti virus non siano caratterizzati, al momento dell’analisi dei dati qui presentati, c’erano 4.378 genomi completi disponibili presso la risorsa di genomi virali della NCBI(Brister et al., 2015) (dati acquisiti nell’agosto 2015). Tuttavia, le analisi su larga scala delle proprietà genomiche di questi virus non sono generalmente disponibili. Ciò è in netto contrasto con le analisi approfondite effettuate su genomi virali parzialmente assemblati o contigui virali derivati da studi metagenomici(Paez-Espino et al., 2016; Roux et al., 2016). Anche se questi studi hanno scoperto molti aspetti importanti dell’ecologia virale con relativamente poca parzialità nel campionamento, essi sono limitati dal fatto che gli studi metagenomici in genere non portano all’assemblaggio completo dei genomi. Un esempio interessante che illustra la difficoltà dell’assemblaggio completo del genoma dagli studi metagenomici è il genoma crAssfago, che nonostante abbia preso frazioni prominenti di letture attraverso vari set di dati metagenomici, era passato inosservato ed è rimasto non assemblato(Dutilh et al., 2014). Tuttavia, i recenti metodi per contrastare queste limitazioni forniscono un futuro promettente per l’uso di set di dati metagenomici nell’acquisizione di genomi completi da ambienti complessi(Marbouty et al., 2017; Nielsen et al., 2014).
Senza genomi virali completi, sarebbe difficile sviluppare una comprensione sistematica degli aspetti chiave dell’architettura genomica virale. Per affrontare almeno in parte questo problema, ci siamo proposti di fornire un’analisi su larga scala di varie metriche genomiche misurate a partire da genomi virali completi già esistenti. Per eseguire un’analisi completa, abbiamo prima esplorato la diversità dei virus conosciuti e dei loro ospiti all’interno del database NCBI (vedi Materiali e metodi). Abbiamo poi creato distribuzioni su una serie di metriche, vale a dire la lunghezza del genoma, la lunghezza del gene, la densità del gene, la percentuale di DNA non codificante (o RNA), l’abbondanza delle categorie di geni funzionali e l’ordine dei geni. Abbiamo fornito brevi introduzioni a queste metriche nelle seguenti sottosezioni.

Figura 1.Schemi di diversi sistemi di classificazione virale esplorati in questo studio.(A) La classificazione di Baltimora divide tutti i virus in sette gruppi in base a come viene prodotto l’mRNA virale. I filamenti di DNA sono indicati in rosso (+ssDNA in una tonalità più scura di rosso rispetto a -ssDNA). Allo stesso modo i filamenti di RNA sono indicati in verde (+ssRNA in una tonalità più scura di verde rispetto a -ssRNA). Nel caso di Baltimora gruppi 1,2,6, e 7, il genoma è o è convertito in dsDNA, che viene poi convertito in mRNA attraverso l’azione di DNA-dipendente RNA polimerasi RNA. Nel caso di Baltimora gruppi 3, 4 e 5, il genoma è o è convertito in +ssRNA, che è mRNA, che è mRNA, attraverso l’azione di RNA polimerasi RNA-dipendente.(B) La classificazione del tipo di nucleotide divide i virus in base al loro materiale genomico in DNA e virus RNA. Baltimora gruppi virali 1, 2 e 7 sono tutti considerati virus del DNA, e i restanti gruppi virali sono considerati virus RNA.(C) La classificazione del dominio host raggruppa i virus in base al dominio host che infettano. Si formano tre gruppi: virus eucarioti, batterici e arcaici.
Lunghezza del genoma virale, lunghezza del gene e densità del gene
I genomi sono pieni di informazioni sul passato e sul presente di un organismo. Un’informazione centrale e rivelatrice è la lunghezza del genoma. Man mano che si sono resi disponibili genomi sempre più completi, abbiamo imparato che le lunghezze dei genomi degli organismi cellulari variano in modo piuttosto esteso, in particolare di sei ordini di grandezza(Phillips et al., 2012; Alberts et al., 2002). Poiché questi studi si sono concentrati sugli organismi cellulari, e poiché le informazioni sulla lunghezza del genoma sono generalmente inaccessibili attraverso studi metagenomici, mancano analisi su larga scala che catturino sistematicamente le distribuzioni di lunghezza del genoma virale alla luce dei diversi sistemi di classificazione e in relazione ad altri parametri genomici. Uno di questi parametri genomici è il numero di geni codificati per genoma, chiamato anche densità genica(Keller e Feuillet, 2000; Hou et al., 2012). Un’altra serie di distribuzioni mancanti riguarda le lunghezze dei geni, e anche in questo caso è importante vedere come variano tra le diverse categorie di classificazione virale.
Le percentuali non codificanti dei genomi virali
Una delle scoperte più sorprendenti degli ultimi decenni è stata la ricca ed enorme diversità del DNA non codificante nel genoma umano(Elgar e Vavouri, 2008). Anche se originariamente considerate come “DNA spazzatura”, le regioni non codificanti dei nostri genomi si sono poi dimostrate di grande importanza funzionale. DNA non codificante è un termine ombrello per elementi molto diversi, per esempio RNA funzionali come micro RNA (miRNA), elementi regolatori come promotori e potenziatori, così come transposoni e pseudogeni.
Inoltre, i genomi variano molto nelle loro percentuali non codificanti. Mentre i genomi eucariotici multicellulari come piante e vertebrati hanno il 50% o più dei loro genomi pieni di regioni non codificanti, i genomi eucariotici monocellulari hanno il 25-50% dei loro genomi presenti come regioni non codificanti e i genomi procariotici hanno percentuali ancora più basse di DNA non codificante, generalmente dal 15 al 20%(Mattick e Makunin, 2006; Morris, 2012; Mattick, 2004). Quindi, si pensa che la percentuale non codificante del genoma sia correlata alla complessità fenotipica dell’organismo e, di conseguenza, gran parte dell’indagine sulle frazioni non codificanti dei genomi si è concentrata sugli eucarioti superiori. Tuttavia, la scoperta dell’immunità batterica contro i fagi e altre fonti di DNA estraneo, altrimenti noto come sistema CRISPR/Cas (Clustered Regularly Interspaced Short Palindromic Repeats), così come la scoperta di una nuova classe di antibiotici che prendono di mira il DNA batterico non codificante(Howe et al., 2015) dimostrano il livello di impatto biotecnologico e la comprensione scientifica che lo studio degli elementi non codificanti nei batteri può fornire. Ancora meno si sa sulla frazione non codificante dei genomi virali.
La letteratura sul DNA o l’RNA virale non codificante è relativamente scarsa ma molto intrigante. I primi RNA virali non codificanti sono stati scoperti in adenovirus, virus dsDNA che infettano gli esseri umani, ed erano ~ 160 coppie di base lunghe(Reich et al., 1966; Tycowski et al., 2015; Steitz et al., 2011). Queste sequenze sono state dimostrate responsabili dell’evasione virale dell’immunità dell’ospite attraverso l’inibizione della proteina chinasi R, una proteina cellulare responsabile dell’inattivazione della sintesi proteica virale(Mathews e Shenk, 1991). Nell’herpesvirus ovino, i miRNA hanno dimostrato di mantenere la latenza virale(Riaz et al., 2014). Questi sono solo alcuni esempi in cui è stato dimostrato che gli elementi non codificanti virali permettono la fuga virale dall’immunità dell’ospite, oltre a regolare il ciclo di vita virale e la persistenza virale(Tycowski et al., 2015). Nonostante molti studi interessanti che esplorano il tema del DNA non codificante cellulare(Mattick e Makunin, 2006; Morris, 2012; Mattick , 2004), non ci sono studi, a nostra conoscenza, che rivelino le statistiche della percentuale di non codifica dei genomi virali.
Categorie di geni funzionali virali
Esistono studi dettagliati sui conteggi dei geni cellulari appartenenti a ciascuna categoria funzionale ampia(Molina e van Nimwegen, 2009; Grilli et al., 2012). Questi studi ci hanno aiutato a capire meglio la scalabilità delle categorie funzionali tra i diversi involucri di organismi. Infatti c’è stata un’intrigante conclusione che per i genomi procariotici esiste un’organizzazione universale che governa il numero relativo di geni in ogni categoria(Molina e van Nimwegen, 2009). Tali rappresentazioni dei genomi virali, tuttavia, sono in gran parte carenti. Pertanto, ci proponiamo di capire meglio come i geni virali siano distribuiti in diverse categorie funzionali e come queste distribuzioni possano differire tra i vari gruppi virali.
Organizzazione del genoma virale
L’organizzazione del genoma virale è un argomento che ha una grande profondità ma un’ampiezza limitata. Esistono diagrammi molto dettagliati a livello di genoma che illustrano la posizione, la direzione e la funzione prevista dei geni virali, che vengono poi confrontati con illustrazioni simili di un piccolo numero di genomi virali(Labonté et al., 2015; Casjens et al. , 2005; Marinelli et al., 2012; Brüssow e Hendrix, 2002). Mentre questo approccio altamente dettagliato è indispensabile per lo studio dei singoli virus, un’illustrazione semplificata dell’organizzazione del genoma è un requisito di qualsiasi visualizzazione ad alto rendimento e confronto dei genomi. Quest’ultimo approccio potrebbe aiutarci a scoprire le regole generali che regolano l’organizzazione genomica, nello stesso modo in cui la sintesi, o l’ordine dei geni conservati, è stato utilizzato per confrontare i genomi animali(Telford e Copley, 2011; Jaillon et al., 2004) e i genomi dei virus RNA che infettano gli invertebrati(Shi et al., 2016).
Risultati
Esplorare la banca dati virale delle BCN
Abbiamo utilizzato il più grande dataset di genomi virali completi disponibili presso la risorsa di genomi virali del National Center for Biotechnology Information (NCBI)(Brister et al., 2015), contenente un totale di 4.378 genomi virali completi al momento dell’acquisizione dei dati (agosto 2015). Dopo aver implementato diverse fasi manuali e programmate per la curatela dei dati, un totale di 2.399 virus (esclusi i virus satellitari) potrebbero essere associati a un host utilizzando la documentazione del NCBI (vedi Materiali e metodi). Questi virus sono stati inclusi per ulteriori analisi e, salvo diversa indicazione, costituiranno il nostro set di dati in questo studio. Esaminando questi virus attraverso diverse classificazioni(Figura 2), è chiaro che si tratta in gran parte di virus DNA(Figura 2B4) e, più specificamente, si tratta principalmente di virus DNA a doppio filamento (dsDNA)(Figura 2C4). Ciò è in contrasto con i virus RNA di questo database, che sono per lo più a singolo filamento(Figura 2B4 e Figura 2C4).
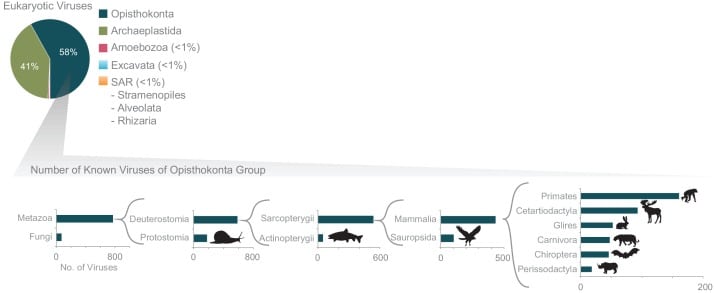
Figura 2-figure supplement 1.Un censimento di tutti i virus con genomi completi segnalati alle BCN che sono stati abbinati ad un host (N= 2399).Ulteriore esplorazione della più grande frazione del viro eucariotico: i virus del supergruppo Opisthokonta (animali).(A) Percentuale di virus che infettano gli host dei tre domini della vita. 1) Viromi eucarioti, 2) batteri e 3) viromi archeologici sono ulteriormente classificati secondo il(B) Nucleotide Type,(C) Baltimora, e D) sistemi di classificazione ICTV.(E) Le distribuzioni di phyla ospite (o supergruppi) infettati dai virus (1) eucarioti, (2) batterici e (3) archeali sono mostrati. Come nel caso del pannello F, l’identificazione tassonomica dell’host deriva dalla banca dati tassonomica della BCN (cfr. Materiali e metodi).(F) Istogrammi del numero di virus noti che infettano le specie ospiti. Il numero medio e mediano di virus che infettano una specie ospite è fornito in ogni area permanente. La gamma completa dei valori x per gli istogrammi batterici ed eucariotici si estende oltre n=20 (vedi virusHostHistograms.ipynb nel nostro repository GitHub[Mahmoudabadi, 2018]). Un’ulteriore esplorazione della più grande frazione del viromo eucariotico (cioè i virus animali) è mostrata nella figura 2-figure supplement 1.L’asse delle x corrisponde al numero di virus che infettano ogni gruppo ospite. 2. In modo ricorsivo, il gruppo ospite con il maggior numero di virus conosciuti viene ulteriormente ingrandito (i gruppi ospiti infettati da pochi virus conosciuti non vengono mostrati). La classificazione dell’host è stata ottenuta dalla banca dati tassonomica della BCN.
Abbiamo inoltre osservato che gli eucarioti ospitano quasi un numero uguale di virus del DNA e dell’RNA(Figura 2B1). A differenza dei procarioti, che ospitano prevalentemente virus con genomi a doppio filamento, gli eucarioti ospitano un numero maggiore di virus con genomi a filamento singolo. Perché i virus del DNA a doppio filamento, nonostante la loro elevata prevalenza nel mondo batterico e archeologico, sono solo il terzo gruppo di virus che infettano gli eucarioti in questo database? Una spiegazione proposta è la separazione fisica dei processi trascrizionali dal citoplasma attraverso il nucleo eucariota(Koonin et al., 2015). Si ritiene che questa separazione fisica imponga un’ulteriore barriera per i virus del DNA nell’accesso all’ambiente trascrizionale dell’ospite.
Più della metà dei virus con genomi completi non sono stati assegnati ad alcun ordine virale secondo la classificazione ICTV(Figura 2D4). Circa un terzo di tutti i virus conosciuti sono assegnati all’ordine Caudovirales, mentre gli altri ordini sono in minoranza. La grande maggioranza dei virus batterici è classificata come parte dell’ordine Caudovirales(Figura 2D2), ma la maggior parte dei virus arcaici ed eucarioti non è stata assegnata a nessun ordine.
Prima di ogni ulteriore esplorazione di questo set di dati, abbiamo cercato di valutare la sua diversità e le possibili fonti di bias(Figura 2E-F). È stato subito chiaro, ad esempio, che i virus archeologici erano fortemente sottocampionati. Al contrario, i virus batterici infettano gli ospiti da una serie diversificata di phyla batterici(Figura 2E2). Tuttavia, anche per i virus batterici, ci sono dei phyla ospiti i cui virus sono completamente assenti dal database, ad esempio Synergistes e Acidobacteria, i cui membri sono tipicamente batteri del suolo incolturabili. Dato che l’isolamento e la caratterizzazione dei virus archeologici e batterici dipende tradizionalmente dalla coltura dei loro ospiti, la maggior parte dei virus con ospiti incolturabili rimane inesplorata. Inoltre, i virus eucarioti presenti nel database infettano gli ospiti principalmente dai Viridiplantae o dai supergruppi Opisthokonta(Figura 2E1). Tra i Viridiplantae, la maggior parte degli host appartengono al gruppo Streptophytina (piante terrestri), e all’interno del supergruppo Opisthokonta, la maggior parte dei virus sono metazoici. Esaminiamo ulteriormente la distribuzione dei virus del supergruppo Opisthokonta nella figura 2-figure supplement 1.
Abbiamo continuato ad esplorare la diversità dell’ospite ad una risoluzione più fine e mappato il numero di virus che infettano ogni specie ospite(Figura 2F). Come previsto, organismi come Staphylococcus aureus, Escherichia coli e Solanum lycopersicum, che sono specie ospiti con rilevanza medica, di ricerca o agricola, hanno molti virus conosciuti e sono outlier nelle distribuzioni asimmetriche mostrate in Figura 2F. Tuttavia, il numero mediano di virus noti per infettare una specie ospite eucariotica o procariotica è approssimativamente 1(Figura 2F). Ciò significa che anche per le specie di ospiti già rappresentate nella nostra collezione, il numero di virus noti è probabilmente una sottostima, considerando il maggior numero di virus noti per infettare le specie di ospiti più studiate.

Figura 2-figure supplement 1.Un censimento di tutti i virus con genomi completi segnalati alle BCN che sono stati abbinati ad un ospite (N= 2399).Ulteriore esplorazione della più grande frazione del viro eucariotico: i virus del supergruppo Opisthokonta (animali).(A) Percentuale di virus che infettano gli host dei tre domini della vita. 1) Viromi eucarioti, 2) batteri e 3) viromi archeologici sono ulteriormente classificati secondo il(B) Nucleotide Type,(C) Baltimora, e D) sistemi di classificazione ICTV.(E) Le distribuzioni di phyla ospite (o supergruppi) infettati dai virus (1) eucarioti, (2) batterici e (3) archeali sono mostrati. Come nel caso del pannello F, l’identificazione tassonomica dell’host deriva dalla banca dati tassonomica della BCN (cfr. Materiali e metodi).(F) Istogrammi del numero di virus noti che infettano le specie ospiti. Il numero medio e mediano di virus che infettano una specie ospite è fornito in ogni area permanente. La gamma completa dei valori x per gli istogrammi batterici ed eucariotici si estende oltre n=20 (vedi virusHostHistograms.ipynb nel nostro repository GitHub[Mahmoudabadi, 2018]). Un’ulteriore esplorazione della frazione più grande del viromo eucariotico (cioè i virus animali) è mostrata nella figura 2-figure supplement 1.L’asse delle x corrisponde al numero di virus che infettano ogni gruppo ospite. 2. In modo ricorsivo, il gruppo ospite con il maggior numero di virus conosciuti viene ulteriormente ingrandito (i gruppi ospiti infettati da pochi virus conosciuti non vengono mostrati). La classificazione dell’host è stata ottenuta dalla banca dati tassonomica della BCN.

Figura 2-figure supplement 1.Ulteriore esplorazione della più grande frazione del viro eucariotico: i virus del supergruppo Opisthokonta (animali).L’asse delle ascisse corrisponde al numero di virus che infettano ciascun gruppo ospite. In modo ricorsivo, il gruppo ospite con il maggior numero di virus conosciuti viene ulteriormente ingrandito (i gruppi ospiti infettati da pochi virus conosciuti non vengono mostrati). La classificazione dell’host è stata ottenuta dalla banca dati tassonomica della BCN.
Lunghezza del genoma virale, lunghezza dei geni, densità dei geni
Le lunghezze dei genomi per tutti i genomi virali completamente sequenziati variavano ampiamente di tre ordini di grandezza(Figura 3A, Tabella 1). Secondo la classificazione del dominio host, i virus procariotici tendono ad avere genomi più lunghi dei virus eucariotici(Figura 3 – dati fonte 1, Figura 3 – supplemento 1). Tuttavia, questa differenza può essere meglio spiegata dalla classificazione del tipo di nucleotide, poiché la lunghezza mediana del genoma del virus RNA è quattro volte più corta della lunghezza mediana del genoma del virus del DNA. Pertanto, il confronto tra le lunghezze del genoma virale procariotico ed eucariotico è confuso dal fatto che il viromo procariotico, come rappresentato da questo database, è composto principalmente da virus del DNA, mentre il viro eucariotico è composto solo per metà da virus del DNA(Figura 2C4).
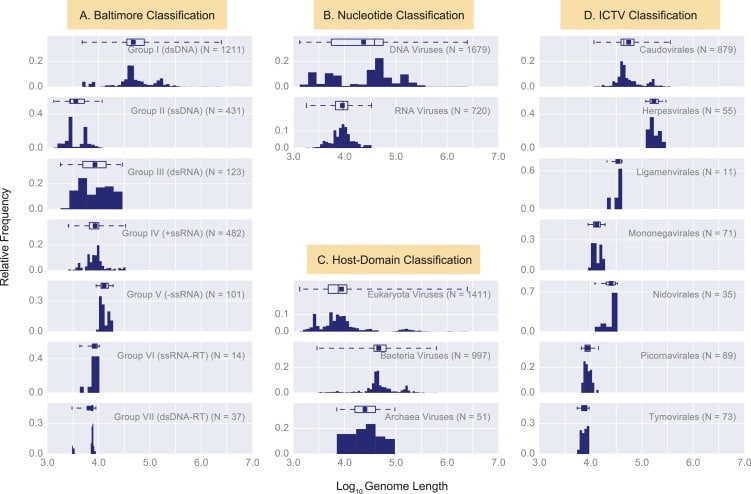
Figura 3-figure supplement 1.Figura 3—supplemento alla figura 1. Descrizione dei genomi virali attraverso la distribuzione della lunghezza del genoma, della lunghezza del gene e della densità genica.statistiche della lunghezza del genoma per gruppi virali attraverso diversi sistemi di classificazione (arrotondate alla kilobase più vicina).statistiche della lunghezza del genoma per gruppi virali attraverso diversi sistemi di classificazione (arrotondate alla kilobase più vicina).istogrammi della lunghezza del genoma (Log10) attraverso tutti i genomi virali completi associati a un ospite.(A) Box plot di lunghezza del genoma (Log10) su tutti i virus inclusi nel nostro dataset (in alto), ulteriormente partizionato in base alle categorie di classificazione di Baltimora (in basso). Il numero di virus inclusi in ogni gruppo è indicato da N. (B) Un esame più attento delle lunghezze del genoma virale dsDNA e ssDNA attraverso la sovrapposizione di Host Domain e sistemi di classificazione ICTV. Le distribuzioni delle lunghezze del genoma associate ai virus eucarioti, batterici e arcaici sono mostrate rispettivamente in salmone, blu e alzavola. Le famiglie virali ICTV con pochi membri sono omesse. Le distribuzioni delle lunghezze del genoma tra i diversi sistemi di classificazione insieme a varie statistiche sono mostrate nella figura 3-figure supplement 1. e nella figura 3-source data 1 . Si noti che la distribuzione bimodale dei virus eucariotici ssDNA, che appare anche nella figura seguente, deriva dai Begomovirus, che sono virus vegetali con genomi monopartiti e bipartiti circolari(Melgarejo et al., 2013).(C) La lunghezza del gene mediano è tracciata in base al numero di geni per ogni genoma per tutti i genomi del nostro dataset, codificati per colore secondo diversi sistemi di classificazione.(D) Numero di geni per lunghezza genomica (densità genica) per i virus dsDNA in base alla sovrapposizione delle categorie di classificazione del dominio host (in basso) e della famiglia ICTV (in alto) (le correlazioni di Pearson e la loro rilevanza statistica, i valori P del test t-test a due code, sono indicati).10.7554/eLife.31955.007Figure 3-source data 1.Genome length statistics for viral groups across different classification systems (arrotondato al kilobase più vicino).Gli istogrammi sono raggruppati secondo quattro sistemi di classificazione virale:(A) classificazione di Baltimora,(B) classificazione del tipo di nucleotide,(C) classificazione del dominio host, e D) classificazione ICTV. Invece di mostrare i conteggi virali assoluti sull’asse y, i conteggi sono normalizzati dal numero totale di virus in ogni categoria virale (i conteggi totali dei virus in ogni categoria sono indicati come N all’interno delle trame). La media di ogni distribuzione è indicata come un punto sulle boxplot. Le statistiche rilevanti per ogni distribuzione sono fornite nella figura 3 – dati fonte 1. In ogni istogramma il numero di bins e la loro larghezza è impostato dalla regola di Freedman-Diaconis(Reich et al., 1966).
| Classificazione | N | Lunghezza del genoma (kb) | Percentuale non codifica (DNA/RNA) | Lunghezza del gene mediano (basi) | |
|---|---|---|---|---|---|
| Dominio Host | Virus eucarioti | 1384 | 8 | 10 | 1055 |
| Virus di batteri | 969 | 43 | 9 | 408 | |
| Virus di Archaea | 46 | 24 | 10 | 400 | |
| Baltimora | Gruppo I (dsDNA) | 1211 | 44 | 9 | 429 |
| Gruppo II (ssDNA) | 431 | 3 | 14 | 588 | |
| Gruppo III (dsRNA) | 123 | 8 | 8 | 2291 | |
| Gruppo IV (+ssRNA) | 482 | 9 | 5 | 2366 | |
| Gruppo V (-ssRNA) | 101 | 12 | 7 | 1353 | |
| Gruppo VI (ssRNA-RT) | 14 | 8 | 16 | 1799 | |
| Gruppo VII (dsDNA-RT) | 37 | 8 | 11 | 558 | |
| Tipo di nucleotide | Virus del DNA | 1679 | 38 | 10 | 444 |
| Virus RNA | 720 | 9 | 6 | 2072 | |
| ICTV (ordini) | Caudovirales | 879 | 44 | 9 | 408 |
| Herpesvirales | 55 | 159 | 19 | 1107 | |
| Ligamenvirales | 11 | 37 | 12 | 372 | |
| Mononegavirales | 71 | 12 | 8 | 1266 | |
| Nidovirales | 35 | 27 | 3 | 672 | |
| Picornavirales | 89 | 8 | 11 | 7056 | |
| Tymovirales | 73 | 8 | 4 | 693 | |
| Combinazioni di diverse classificazioni | Tutti i virus dsDNA eucarioti | 271 | 33 | 11 | 990 |
| Tutti i virus dsDNA batterici | 899 | 44 | 9 | 408 | |
| Tutti i virus Archaeal dsDNA | 41 | 28 | 10 | 396 | |
| Tutti i virus ssDNA eucariotici | 375 | 3 | 14 | 732 | |
| Tutti i virus ssDNA batterici | 51 | 7 | 14 | 348 | |
Per quanto riguarda la lunghezza del genoma virale, la classificazione di Baltimora sembra offrire il potere più esplicativo. Sapere se un genoma virale è basato sul DNA o sull’RNA fornisce già una forte indicazione sulla lunghezza del genoma virale, specialmente per i virus RNA in cui la deviazione standard è di pochi kilobasi(Figura 3 – dati fonte 1). Tuttavia, distinguendo tra virus ssDNA, dsDNA e dsDNA-RT, la classificazione di Baltimore offre una visione più completa delle distribuzioni della lunghezza del genoma rispetto alla classificazione binaria del tipo di nucleotide(Figura 3A). In tutti i gruppi di Baltimora, i virus dsDNA hanno lunghezze genomiche che hanno la maggiore deviazione standard, tuttavia, considerando la limitata gamma di lunghezze genomiche associate ad altri gruppi di Baltimora, è molto probabile che un genoma virale più grande sia composto da dsDNA(Figura 3A). Forniamo una visione più dettagliata delle distribuzioni di lunghezza del genoma attraverso la stratificazione di diversi sistemi di classificazione, applicando prima la classificazione di Baltimora, seguita dalle classificazioni del dominio host e della famiglia ICTV(Figura 3B, Figura 3-dati sorgente 1). Infine, vale la pena notare che la dimensione capsid, sorprendentemente, non sembra correlare con la dimensione del genoma virale e, in gradi diversi, molti virus mostrano di sottoutilizzare il volume capsid(Brandes e Linial, 2016).
Osservando la relazione tra la lunghezza mediana del gene e il numero di geni per genoma virale(Figura 3C), diventano evidenti due diverse strategie di codifica. Vale a dire, rispetto ai virus del DNA, i virus RNA mostrano una vasta gamma di lunghezze geniche. Questa tendenza riflette almeno in parte le sfide che i virus RNA si trovano ad affrontare quando incontrano i requisiti della macchina traslazionale del loro ospite (Firthe Brierley, 2012). Per esempio, molti dei genomi RNA che abbiamo esaminato contenevano geni che codificano le poliproteine, lo slippage ribosomiale (frame-shifting) o la lettura del codone attraverso eventi, tra gli altri meccanismi translazionali non canonici.
Come nel caso della lunghezza del genoma, esaminando solo le classificazioni dell’ICTV o del Dominio Host sarebbe difficile trarre conclusioni significative sui modelli osservati, e nel caso della classificazione del Dominio Host, le nostre conclusioni sarebbero confuse dal rapporto sproporzionato tra RNA e virus del DNA che sono noti per infettare ogni dominio host in questo database. Tuttavia, la stratificazione di questi sistemi di classificazione offre nuovi spunti di riflessione, di cui parleremo nei paragrafi seguenti.
Ne seguiamo altri(Keller e Feuillet, 2000; Hou et al., 2012) nel definire la densità genica di un genoma come il numero di geni diviso per la lunghezza del genoma(Figura 3D). Abbiamo ulteriormente partizionato i virus dsDNA secondo il dominio host e successivamente le classificazioni ICTV (famiglia). Abbiamo osservato una forte correlazione lineare tra le lunghezze dei genomi virali dsDNA e il numero di geni codificati da questi genomi(Figura 3D). Le densità medie (e mediane) dei geni per i genomi virali dsDNA batterici, arcaici ed eucariotici sono approssimativamente 1,4, 1,6 e 0,9 geni per chilo di paia di basi. Come illustrato dalle pendenze delle linee di regressione, così come attraverso un test statistico non parametrico eseguito sulle densità dei geni virali dsDNA eucariotici e batterici (test Mann-Whitney U unilaterale, P<10-5), i virus dsDNA batterici hanno densità geniche significativamente più elevate rispetto alle loro controparti eucariotiche in questo database.
Un esame più attento delle lunghezze geniche mediane rivela più chiaramente le lunghezze geniche significativamente più lunghe dei virus RNA rispetto ai virus del DNA (test Mann-Whitney U unilaterale, P<10-5)(Figura 4, Tabella 1). Concentrandoci sui virus del DNA e dividendo ulteriormente questi virus in base alle classificazioni di Baltimora, Host Domain e ICTV (famiglia), arriviamo a una tendenza interessante. Vale a dire, i virus eucariotici, siano essi dsDNA o ssDNA, hanno lunghezze geniche significativamente più lunghe rispetto ai virus batterici della stessa categoria di classificazione di Baltimora(Figura 4, Figura 4, dati fonte 1) (test Mann-Whitney U unilaterale, P<10-5). Questa tendenza segue quello che vediamo nei genomi cellulari, dato che i geni e le proteine procariotiche sono significativamente più corti di quelli eucariotici(Milo e Phillips, 2015; Brocchieri e Karlin, 2005).
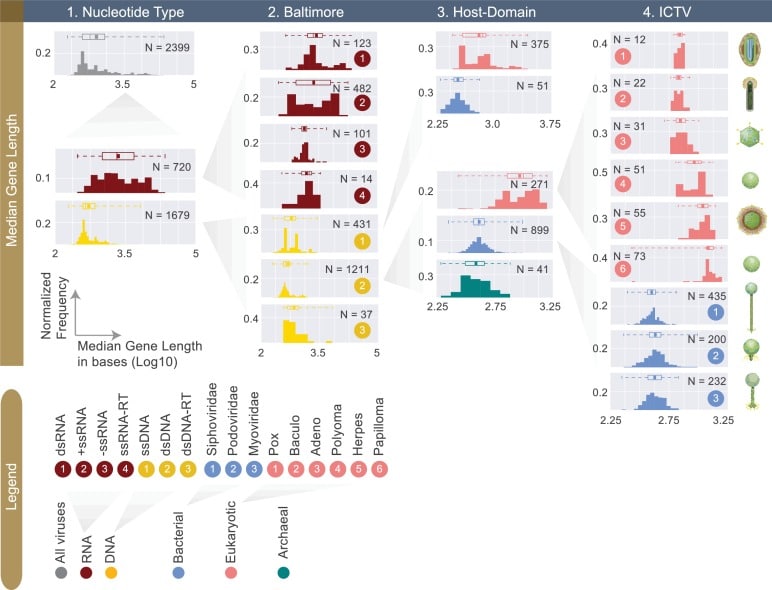
Figura 4 dati fonte 1.Figura 4—dati fonte 1. Istogrammi normalizzati delle lunghezze mediane dei geni (log10) su tutti i genomi virali completi associati a un host.statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).Invece di mostrare i conteggi virali assoluti sugli assi y, i conteggi sono normalizzati dal numero totale di virus in ogni categoria virale (indicato come N all’interno di ogni grafico). La media di ogni distribuzione è indicata come un punto sul boxplot. Per tutti gli istogrammi, i numeri di bin e le larghezze dei bin sono sistematicamente decisi dalla regola di Freedman-Diaconis(Reich et al., 1966). Gli schemi virali a destra della figura sono modificati da ViralZone(Hulo et al., 2011). Le statistiche chiave che descrivono queste distribuzioni si trovano nella Tabella 1 e nella Figura 4 dati fonte 1.10.7554/eLife.31955.010Figure 4-source data 1.Median gene length statistics for viral groups across different classification systems (rounded to the nearest base).è importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze dei geni mediani.È importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze mediane dei geni.È importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze dei geni mediani.

Figura 3-figure supplemento 1.Figura 3—supplemento alla figura 1. Descrizione dei genomi virali attraverso le distribuzioni di lunghezza del genoma, lunghezza del gene e densità del gene.statistiche di lunghezza del genoma per gruppi virali attraverso diversi sistemi di classificazione (arrotondate alla kilobase più vicina).statistiche di lunghezza del genoma per gruppi virali attraverso diversi sistemi di classificazione (arrotondate alla kilobase più vicina).istogrammi di lunghezza del genoma (Log10) attraverso tutti i genomi virali completi associati a un ospite.(A) Box plot di lunghezza del genoma (Log10) su tutti i virus inclusi nel nostro dataset (in alto), ulteriormente partizionato in base alle categorie di classificazione di Baltimora (in basso). Il numero di virus inclusi in ogni gruppo è indicato da N. (B) Un esame più attento delle lunghezze del genoma virale dsDNA e ssDNA attraverso la sovrapposizione di Host Domain e sistemi di classificazione ICTV. Le distribuzioni delle lunghezze del genoma associate ai virus eucarioti, batterici e arcaici sono mostrate rispettivamente in salmone, blu e alzavola. Le famiglie virali ICTV con pochi membri sono omesse. Le distribuzioni delle lunghezze del genoma tra i diversi sistemi di classificazione insieme a varie statistiche sono mostrate nella Figura 3-figure supplement 1. e nella Figura 3-source data 1 . Si noti che la distribuzione bimodale dei virus eucariotici ssDNA, che appare anche nella figura seguente, deriva dai Begomovirus, che sono virus vegetali con genomi monopartiti e bipartiti circolari(Melgarejo et al., 2013).(C) La lunghezza del gene mediano è tracciata in base al numero di geni per ogni genoma per tutti i genomi del nostro dataset, codificati per colore secondo diversi sistemi di classificazione.(D) Numero di geni per lunghezza genomica (densità genica) per i virus dsDNA basati sulla sovrapposizione delle categorie di classificazione di Host Domain (in basso) e della famiglia ICTV (in alto) (le correlazioni di Pearson e la loro significatività statistica, i valori P del test t-test a due code, sono indicati).10.7554/eLife.31955.007Figure 3-source data 1.Genome length statistics for viral groups across different classification systems (arrotondato al kilobase più vicino).Gli istogrammi sono raggruppati secondo quattro sistemi di classificazione virale:(A) classificazione di Baltimora,(B) classificazione del tipo di nucleotide,(C) classificazione del dominio host, e D) classificazione ICTV. Invece di mostrare i conteggi virali assoluti sull’asse y, i conteggi sono normalizzati dal numero totale di virus in ogni categoria virale (i conteggi totali dei virus in ogni categoria sono indicati come N all’interno delle trame). La media di ogni distribuzione è indicata come un punto sulle boxplot. Le statistiche rilevanti per ogni distribuzione sono fornite nella figura 3 – dati fonte 1. In ogni istogramma il numero di bins e la loro larghezza è impostato dalla regola di Freedman-Diaconis(Reich et al., 1966).

Figura 3-figure supplement 1.2. Istogrammi della lunghezza del genoma (Log10) su tutti i genomi virali completi associati ad un ospite.Gli istogrammi sono raggruppati secondo quattro sistemi di classificazione virale:(A) classificazione di Baltimora,(B) classificazione del tipo di nucleotide,(C) classificazione del dominio host e (D) classificazione ICTV. Invece di mostrare i conteggi virali assoluti sull’asse y, i conteggi sono normalizzati dal numero totale di virus in ogni categoria virale (i conteggi totali dei virus in ogni categoria sono indicati come N all’interno delle trame). La media di ogni distribuzione è indicata come un punto sulle boxplot. Le statistiche rilevanti per ogni distribuzione sono fornite nella figura 3 – dati fonte 1. In ogni istogramma il numero di bins e la loro larghezza è impostato dalla regola di Freedman-Diaconis(Reich et al., 1966).

Figura 4 dati fonte 1.Figura 4—dati sorgente 1. Istogrammi normalizzati delle lunghezze mediane dei geni (log10) su tutti i genomi virali completi associati ad un host.statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).statistiche delle lunghezze mediane dei geni per gruppi virali su diversi sistemi di classificazione (arrotondate alla base più vicina).Invece di mostrare i conteggi virali assoluti sugli assi y, i conteggi sono normalizzati dal numero totale di virus in ogni categoria virale (indicato come N all’interno di ogni grafico). La media di ogni distribuzione è indicata come un punto sul boxplot. Per tutti gli istogrammi, i numeri di bin e le larghezze dei bin sono sistematicamente decisi dalla regola di Freedman-Diaconis(Reich et al., 1966). Gli schemi virali a destra della figura sono modificati da ViralZone(Hulo et al., 2011). Le statistiche chiave che descrivono queste distribuzioni si trovano nella Tabella 1 e nella Figura 4 dati fonte 1.10.7554/eLife.31955.010Figure 4-source data 1.Median gene length statistics for viral groups across different classification systems (rounded to the nearest base).è importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze dei geni mediani.È importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze mediane dei geni.È importante chiarire che i valori mediani in questa tabella rappresentano la mediana delle lunghezze dei geni mediani.
Percentuali non codificanti dei genomi virali
Finora ci siamo concentrati principalmente sulle frazioni di codifica dei genomi virali. Così, abbiamo creato distribuzioni di percentuali non codificanti dei genomi virali (vedi Materiali e metodi, Figura 5, Tabella 1, Figura 5 – dati fonte 1). In generale, i genomi virali del DNA contengono circa il 10% di regioni non codificanti, che è anche inferiore alla percentuale non codificante dei genomi batterici(Mattick e Makunin, 2006; Morris, 2012). Con una percentuale mediana di non codifica di appena il 6%, i genomi virali dell’RNA hanno una percentuale di non codifica significativamente più bassa rispetto ai virus del DNA in questo database (test a U di Mann-Whitney, P<10-5). Una notevole eccezione al gruppo virale RNA è lo ssRNA-RT con una percentuale mediana di non codifica del 16%. È interessante notare che entrambi i gruppi retrovirali avevano percentuali relativamente elevate di DNA non codificante. Questo è probabilmente dovuto alla presenza di geni retrovirali defunti. Ad esempio, il retrovirus endogeno Xenopus laevis (NCBI taxon ID 204873) appartenente al gruppo ssRNA-RT ha una percentuale non codificante del 93%. Questa elevata percentuale di non codifica può essere spiegata dal fatto che questo genoma del virus contiene tre pseudogeni precedentemente codificati per le proteine env, pol e gag.
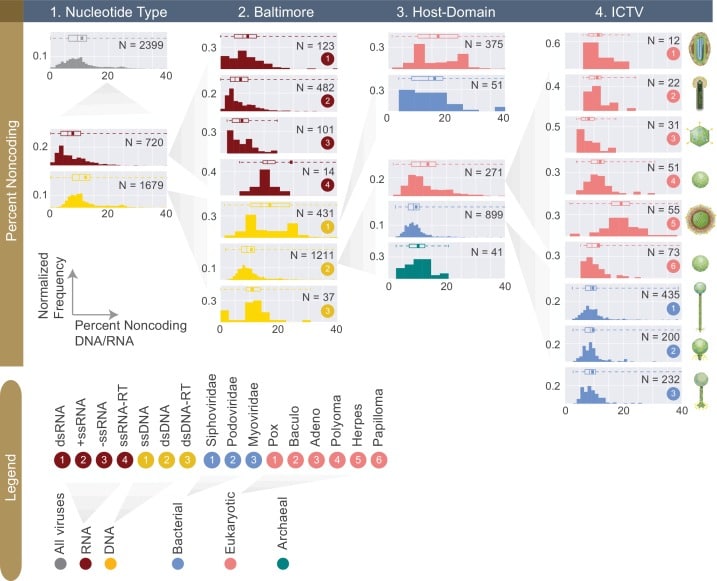
Figura 5 – Dati fonte 1.Figura 5—dati fonte 1. Istogrammi normalizzati della percentuale di DNA/RNA non codificante per tutti i genomi virali completi associati ad un host.Percentuale di DNA (o RNA) non codificante per gruppi virali in diversi sistemi di classificazione (arrotondata alla percentuale più vicina).Percentuale di DNA (o RNA) non codificante per gruppi virali in diversi sistemi di classificazione (arrotondata alla percentuale più vicina).I conteggi dei virus sono normalizzati in base al numero totale di virus in ogni categoria virale (indicato come N all’interno di ogni grafico). La media di ogni distribuzione è indicata come un punto sul boxplot. Per tutti gli istogrammi, i numeri di bin e le larghezze dei bin sono sistematicamente decisi dalla regola di Freedman-Diaconis(Reich et al., 1966). Gli schemi virali sono modificati da ViralZone(Hulo et al., 2011). Le statistiche chiave che descrivono queste distribuzioni si trovano nella Tabella 1 e nella Figura 5 – dati fonte 1.10.7554/eLife.31955.012Figure 5-source data 1.Percentuale di DNA (o RNA) non codificante per gruppi virali attraverso diversi sistemi di classificazione (arrotondata alla percentuale più vicina).

Figura 5 – dati fonte 1.Figura 5—dati fonte 1. Istogrammi normalizzati della percentuale di DNA/RNA non codificante per tutti i genomi virali completi associati ad un host.Percentuale di DNA (o RNA) non codificante per gruppi virali in diversi sistemi di classificazione (arrotondata alla percentuale più vicina).Percentuale di DNA (o RNA) non codificante per gruppi virali in diversi sistemi di classificazione (arrotondata alla percentuale più vicina).I conteggi dei virus sono normalizzati in base al numero totale di virus in ogni categoria virale (indicato come N all’interno di ogni grafico). La media di ogni distribuzione è indicata come un punto sul boxplot. Per tutti gli istogrammi, i numeri di bin e le larghezze dei bin sono sistematicamente decisi dalla regola di Freedman-Diaconis(Reich et al., 1966). Gli schemi virali sono modificati da ViralZone(Hulo et al., 2011). Le statistiche chiave che descrivono queste distribuzioni si trovano nella Tabella 1 e nella Figura 5 – dati fonte 1.10.7554/eLife.31955.012Figure 5-source data 1.Percentuale di DNA (o RNA) non codificante per gruppi virali attraverso diversi sistemi di classificazione (arrotondata alla percentuale più vicina).
Categorie di geni funzionali virali
Abbiamo classificato i geni virali secondo diverse categorie funzionali principali, tra cui i geni strutturali come i geni del capside e della coda, i geni metabolici, i geni informativi, che definiamo come quelli coinvolti nella replicazione, trascrizione o traduzione del codice genetico virale, tra le altre categorie(Figura 6, vedi Materiali e metodi). Oltre alla frazione di geni virali che siamo stati in grado di assegnare a queste categorie funzionali, rimane ancora quella che definiremo una frazione “senza etichetta” che è composta da ipotetici geni o geni con scarsa annotazione (vedi Materiali e metodi). Quando riportiamo l’abbondanza relativa di diverse categorie di geni funzionali, normalizzeremo il numero di geni appartenenti a ciascuna categoria funzionale per il numero totale di geni etichettati.
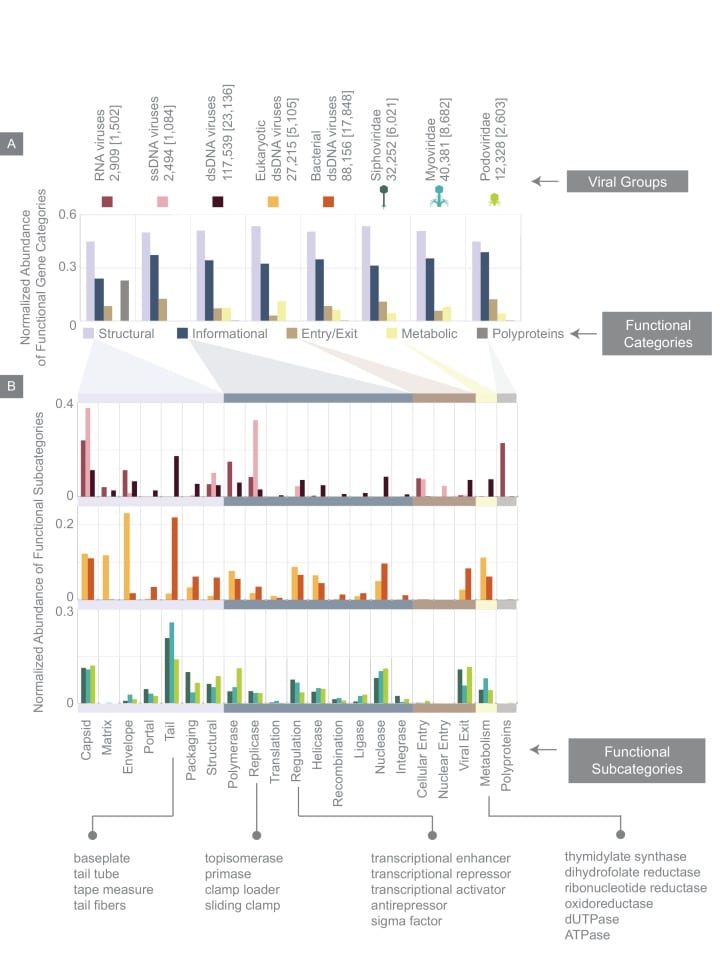
Figura 6.Abbondanza normalizzata delle categorie di geni funzionali tra i diversi gruppi virali.(A) Abbondanze di categorie di geni funzionali tra 8 gruppi virali normalizzati per il numero di geni etichettati in ogni gruppo virale (il numero totale di geni in ogni gruppo virale è mostrato sopra il pannello e tra parentesi è il numero di geni etichettati per ogni gruppo virale).(B) Abbondanze di sottocategorie di geni funzionali in 8 gruppi virali: RNA, ssDNA, e gruppi virali dsDNA (trama in alto); gruppi virali eucarioti e batterici dsDNA (in mezzo); gruppi virali Siphoviridae, Myoviridae, e Podoviridae (in basso). Sono forniti alcuni esempi dei tipi di geni contenuti come parte di ogni sottocategoria funzionale.
I virus RNA, dsDNA e ssDNA, nonostante le differenze nella categorizzazione dettagliata dei loro geni(Figura 6B) hanno caratteristiche generali simili(Figura 6A). Ad esempio, in tutti e tre i gruppi virali, circa la metà dei geni sono strutturali. Allo stesso modo, i virus dsDNA di eucarioti e batteri presenti in questo database, in contrasto con le diverse proprietà genomiche e morfologie, hanno sorprendentemente una distribuzione molto simile delle abbondanze delle categorie funzionali dei geni e delle sottocategorie. La differenza principale tra questi due gruppi virali, come ci si aspetta dalla nostra conoscenza delle morfologie virali, è che una porzione più grande di geni virali eucarioti dsDNA virali sono i geni dell’involucro e della matrice, mentre una porzione più grande di geni batterici dsDNA sono geni portale e geni associati alla coda. Con un ulteriore zoom sui virus dsDNA batterici, è ancora una volta interessante vedere che i gruppi virali Myoviridae, Siphoviridae e Podoviridae, con le loro diverse morfologie e la vasta gamma di ospiti, hanno abbondanze di categoria genica funzionale molto simili anche a livello di sottocategorie.

Figura 6.Abbondanza normalizzata delle categorie di geni funzionali tra i diversi gruppi virali.(A) Abbondanze di categorie di geni funzionali tra 8 gruppi virali normalizzate al numero di geni etichettati in ogni gruppo virale (il numero totale di geni in ogni gruppo virale è mostrato sopra il pannello, e tra parentesi il numero di geni etichettati per ogni gruppo virale).(B) Abbondanze di sottocategorie di geni funzionali in 8 gruppi virali: RNA, ssDNA, e gruppi virali dsDNA (trama in alto); gruppi virali eucarioti e batterici dsDNA (in mezzo); gruppi virali Siphoviridae, Myoviridae, e Podoviridae (in basso). Sono forniti alcuni esempi dei tipi di geni contenuti come parte di ogni sottocategoria funzionale.
Organizzazione del genoma virale
Per esplorare l’organizzazione del genoma virale abbiamo sviluppato un metodo a grana grossa per visualizzare un gran numero di genomi in un’unica istantanea. Per prima cosa abbiamo definito l’organizzazione del genoma come l’ordine in cui i geni appaiono attraverso un genoma. Abbiamo poi simboleggiato ogni gene con una lettera, indifferente alla lunghezza del gene o al suo orientamento sul genoma. I geni con funzioni simili sono raggruppati e sono rappresentati dalla stessa lettera(Figura 7). Pertanto ogni genoma virale, analogo ad una sequenza nucleotidica, è descritto in modo compatto da una sequenza di lettere che rappresentano il suo ordine genico(Figura 7), a cui faremo riferimento come sequenza di ordine genico. Poiché volevamo studiare le sequenze di ordine genico attraverso diversi gruppi virali, ci siamo concentrati sui geni le cui funzioni sono universalmente richieste, cioè i geni strutturali. textFile-1.txt (vedi il nostro repository GitHub) fornisce le sequenze di ordine genico strutturale per tutti i virus (vedi Materiali e metodi per i filtri applicati), anche se lo script sviluppato può essere modificato per visualizzare il posizionamento di qualsiasi numero di geni o gruppi di geni definiti dall’utente.
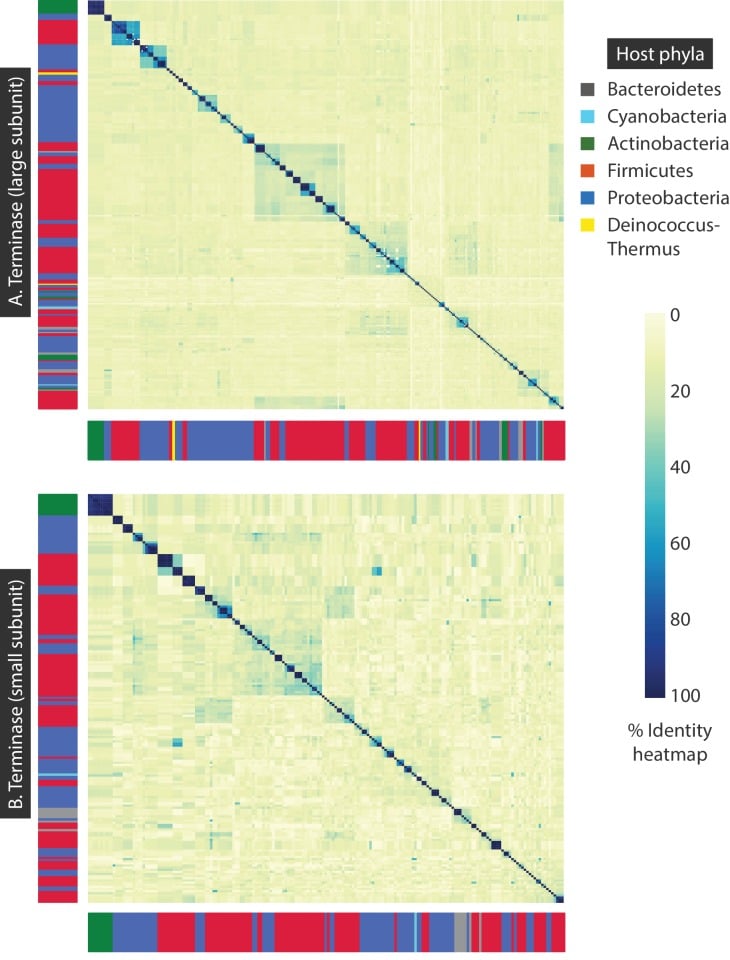
Figura 7-figure supplement 1.Allineamento dei modelli di ordine dei geni più comuni per i virus batterici dsDNA.mappe di calore di identità percentuale di A) 320 sequenze di aminoacidi della terminasi (sottounità grande) e B) 191 sequenze di aminoacidi della terminasi (sottounità piccola) dei batteriofagi dsDNA.Ogni genoma è riassunto da una sequenza di lettere, con ogni lettera corrispondente a un gene, posizionata nell’ordine in cui appare sul genoma. A titolo di esempio, viene mostrata la sequenza dell’ordine dei geni per la Salmonella phage FSL SP-004. Si noti che le lettere mostrate servono solo a denotare i geni con funzioni simili. Ai geni strutturali sono assegnati dei colori, mentre gli altri geni sono indicati in nero. In tutti e tre i pannelli, ogni riga corrisponde alla sequenza di ordine dei geni per un determinato virus, e quindi, la lunghezza della sequenza denota il numero di geni all’interno di un determinato genoma. Le due colonne a sinistra che accompagnano ogni pannello forniscono ulteriori informazioni sugli ospiti e sulle morfologie virali. I pannelli A, B e C rappresentano rispettivamente i modelli di ordine genico A, B e C. L’allineamento globale genetico(Steitz et al., 2011) è stato utilizzato per allineare le sequenze di ordine genico (vedi Materiali e metodi). Fare riferimento alla Figura 7-figure supplemento 1 per vedere le mappe di calore di identità percentuale di identità delle terminazioni (sottounità grandi e piccole) attraverso i virus batterici dsDNA.Le barre laterali indicano il phylum ospite per ogni sequenza di batteriofago.
Inoltre, concentrandoci sui virus batterici dsDNA presenti nel database virale NCBI, siamo stati in grado di identificare i modelli di ordine genico più comuni attraverso questo viromo (vedi Materiali e metodi). Un particolare modello di ordine genico e le sue variazioni esistono tra i vari tipi di virus batterici dsDNA. Ci riferiremo ad esso come schema di ordine genico A(Figura 7A). Nel modello A, i geni legati al confezionamento dei geni, al portale e al capside sono per lo più strettamente raggruppati e sono seguiti da geni associati alla coda. È interessante notare che questo modello si verifica all’inizio del genoma per alcuni virus e per altri sembra essere stato spostato più in basso sul genoma. Modello A si verifica attraverso i virus da cinque diversi phyla ospite. Gli altri due modelli di ordine genico più comuni (modelli B e C) si verificano tra virus con una gamma di ospiti e morfologie più limitate.
Al di là del loro ordine nel genoma, ci siamo chiesti fino a che punto le proteine batteriofagiche di ospiti tassonomicamente simili sono simili l’uno all’altro in sequenza? Nel tentativo di rispondere a questa domanda, abbiamo analizzato le sequenze da due proteine strutturali in batteriofagi dsDNA, vale a dire la sottounità terminase grande e la sottounità piccola, che sono utilizzati nell’imballaggio del DNA all’interno di capsidi e rappresentano alcune delle proteine batteriofagiche più chiaramente annotati. Le sequenze di aminoacidi sono state allineate utilizzando Clustal-Omega(Arndt et al., 2016) e le percentuali di somiglianza delle sequenze sono mostrate come mappe di calore(Figura 7-figure supplement 1). L’informazione del phylum ospite è il codice colore. Come si può vedere da questa figura, i batteriofagi che infettano gli ospiti dallo stesso phylum non hanno necessariamente sequenze di terminazioni più simili. Nei casi in cui vi è una somiglianza tra le sequenze di termiti, è principalmente da batteriofagi che infettano la stessa specie ospite.
Per fornire più informazioni sull’organizzazione genomica dei batteriofagi del dsDNA, abbiamo esaminato le posizioni del sito di attaccamento, le distribuzioni di lunghezza e la diversità di sequenza. I siti di attaccamento sono luoghi di ricombinazione sito-specifica che i fagi lisogeni usano per inserire il loro DNA nel genoma dell’ospite (vedere Materiali e metodi). Tra le diverse centinaia di batteriofagi dsDNA che sono stati inclusi in questa analisi, abbiamo trovato circa un quarto dei siti di attacco putativo. Abbiamo trovato che la lunghezza mediana del sito di attacco è di 13 coppie di basi(Figura 8A). La posizione iniziale dell’attacco sinistro nel genoma si trova a ~ 2 kb (questa è la mediana delle posizioni iniziali del sito dell’attacco sinistro in tutti i genomi analizzati). La posizione mediana del sito di attacco destro si trova a ~40 kb.(Figura 8B). La Figura 8C mostra gli stessi dati ma normalizzati dalla lunghezza del genoma.
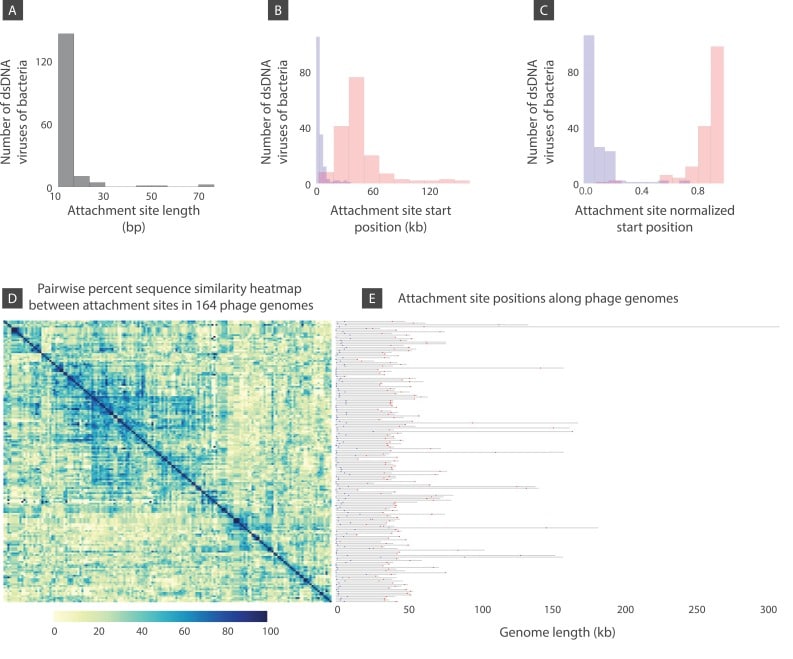
Figura 8-dati sorgente 1.Figura 8—dati di origine 1. Lunghezza del sito di attacco, posizione e diversità della sequenza per virus batterici 164 dsDNA.diversi batteriofagi della Figura 8D con sequenze del sito di attacco simili o identiche.diversi batteriofagi della Figura 8D con sequenze del sito di attacco simili o identiche.(A) Istogramma della lunghezza del sito di attacco.(B) Istogramma delle posizioni iniziali del sito di attacco (allegato sinistro: blu, allegato destro: rosso). (C) Istogramma delle posizioni iniziali del sito di attacco normalizzate dalla lunghezza del genoma.(D) Matrice di somiglianza della sequenza percentuale tra i siti di attacco. (E) Posizioni del sito di attacco lungo i genomi virali (allegato sinistro: blu, allegato destro: rosso). Figura 8-di dati fonte 1 mostra diversi batteriofagi mostrati nel pannello E con sequenze di siti di attaccamento simili o identici.10.7554/eLife.31955.017Figure 8-source data 1.Several bacteriophages from Figure 8D with similar or identical attachment site sequences.
Per esaminare la diversità delle sequenze dei siti di attaccamento, abbiamo usato Clustal-Omega(Sievers et al., 2011) per creare un allineamento delle sequenze. La Figura 8D è una mappa termica dei punteggi di somiglianza percentuale della sequenza. La Figura 8E mostra i siti di attaccamento a sinistra (blu) e a destra (rosso) nei genomi dei fago. Nota, i genomi sono mostrati secondo il loro ordine nella Figura 8D. Mentre la stragrande maggioranza dei siti di attaccamento sono molto diversi in sequenza, come mostrato dalle regioni di bassa somiglianza nella mappa termica, ci sono un certo numero di virus che hanno sequenze di siti di attaccamento identiche(Figura 8-dati fonte 1, Materiali e metodi). Forse non sorprende che questi fagi siano in gran parte quelli che infettano ceppi diversi della stessa specie ospite. I fagi che infettano gli host al di fuori della stessa specie sembrano avere più probabilità di avere sequenze di siti di attacco diverse.

Figura 7-figure supplement 1.Allineamento dei modelli di ordine genico più comuni per i virus batterici dsDNA.mappe di calore di identità percentuale di A) 320 sequenze di aminoacidi della terminasi (grande sottounità) e B) 191 sequenze di aminoacidi della terminasi (piccola sottounità) dei batteriofagi dsDNA.Ogni genoma è riassunto da una sequenza di lettere, con ogni lettera corrispondente a un gene, posizionata nell’ordine in cui appare sul genoma. A titolo di esempio, viene mostrata la sequenza dell’ordine dei geni per la Salmonella phage FSL SP-004. Si noti che le lettere mostrate servono solo a denotare i geni con funzioni simili. Ai geni strutturali sono assegnati dei colori, mentre gli altri geni sono indicati in nero. In tutti e tre i pannelli, ogni riga corrisponde alla sequenza di ordine dei geni per un determinato virus, e quindi, la lunghezza della sequenza denota il numero di geni all’interno di un determinato genoma. Le due colonne a sinistra che accompagnano ogni pannello forniscono ulteriori informazioni sugli ospiti e sulle morfologie virali. I pannelli A, B e C rappresentano rispettivamente i modelli di ordine genico A, B e C. L’allineamento globale genetico(Steitz et al., 2011) è stato utilizzato per allineare le sequenze di ordine genico (vedi Materiali e metodi). Fare riferimento alla Figura 7-figure supplemento 1 per vedere le mappe di calore di identità percentuale di identità delle terminazioni (sottounità grandi e piccole) attraverso i virus batterici dsDNA.Le barre laterali indicano il phylum ospite per ogni sequenza di batteriofago.

Figura 7-figure supplement 1.Figura 7—supplemento alla figura 1. Mappe di calore dell’identità percentuale di A) 320 sequenze di amminoacidi della termalasi (sottounità grande) e B) 191 sequenze di amminoacidi della termalasi (sottounità piccola) dei batteriofagi dsDNA.Le barre laterali indicano il phylum ospite per ogni sequenza di batteriofagi.

Figura 8 – Dati di origine 1.Figura 8—dati di origine 1. Lunghezza del sito di attacco, posizione e diversità della sequenza per i virus batterici 164 dsDNA.diversi batteriofagi della Figura 8 D con sequenze del sito di attacco simili o identiche.diversi batteriofagi della Figura 8D con sequenze del sito di attacco simili o identiche.(A) Istogramma della lunghezza del sito di attacco.(B) Istogramma delle posizioni iniziali del sito di attacco (allegato sinistro: blu, allegato destro: rosso). (C) Istogramma delle posizioni iniziali del sito di attacco normalizzate dalla lunghezza del genoma.(D) Matrice di somiglianza della sequenza percentuale tra i siti di attacco. (E) Posizioni del sito di attacco lungo i genomi virali (allegato sinistro: blu, allegato destro: rosso). Figura 8-di dati fonte 1 mostra diversi batteriofagi mostrati nel pannello E con sequenze di siti di attaccamento simili o identici.10.7554/eLife.31955.017Figure 8-source data 1.Several bacteriophages from Figure 8D with similar or identical attachment site sequences.
Fare un po’ di luce sulle proteine virali “ipotetiche”.
Come dimostrato nelle sezioni precedenti, le proteine annotate come forma ipotetica o presunta più della metà di tutte le proteine associate ai batteriofagi dsDNA. Nel tentativo di saperne di più su queste proteine, abbiamo usato BLASTP per interrogare tutte le ~88.000 proteine dei batteriofagi dsDNA contro il database di proteine NCBI Refseq (limitato ai batteri) (Vedi Materiali e metodi). Lo scopo di questo esercizio è stato quello di utilizzare le annotazioni degli omologhi batterici alle proteine virali per ottenere una migliore comprensione di ciò che la funzione di ogni ipotetica proteina batteriofago potrebbe essere.
Una relazione omologa è stata definita come una corrispondenza con il punteggio del valore E del BLASTP < 10-10. L’omologo batterico più vicino ad ogni proteina batteriofaga (cioè la partita con il più basso valore E) è stato raccolto. Non tutte le proteine batteriofagiche avevano un omologo batterico, almeno non uno che si trova attualmente nella banca dati NCBI. Tuttavia, un numero sorprendentemente grande ha avuto omologhi batterici, e abbiamo raccolto queste proteine insieme ad altre informazioni utili in textFile-2.txt (vedi il nostro repository GitHub). Questo set di dati è in parte visualizzato nella Figura 9.
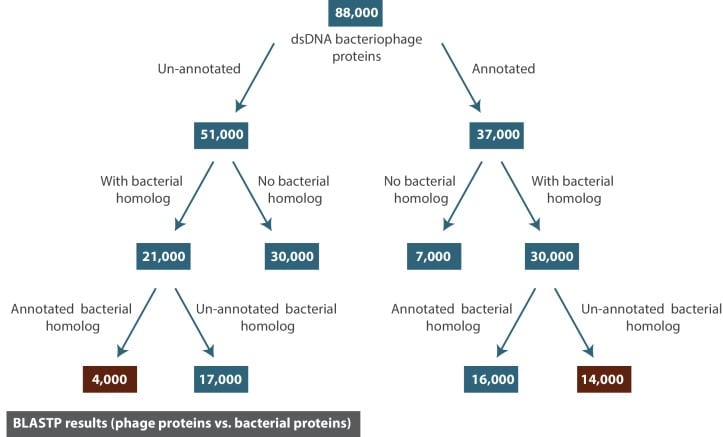
Figura 9.Il risultato del BLASTP per tutte le proteine batteriofagiche dsDNA contro il database delle proteine NCBI Refseq (limitato alle proteine batteriche).I numeri riportati corrispondono al numero di proteine batteriofagiche dsDNA (arrotondato al migliaio più vicino).
La maggior parte degli omologhi batterici di ipotetiche proteine fagiche sono stati annotati come proteine ipotetiche. Tuttavia, alcune migliaia di ipotetiche proteine fagiche potrebbero essere assegnate all’annotazione presunta in base all’annotazione dei loro omologhi batterici (vedere Materiali e metodi). È interessante notare che siamo stati in grado di abbinare ancora più proteine ipotetiche batteriche a un’annotazione presunta basata sulle annotazioni dei loro omologhi delle proteine batteriofagiche. Anche se, questo metodo può certamente essere utile per colmare alcune delle lacune nelle annotazioni delle proteine, è solo buono come le annotazioni e la convenzione che stabiliamo per descrivere le proteine. Purtroppo, un numero considerevole di annotazioni è attualmente troppo specializzato o troppo vago per essere utile.

Figura 9.Il risultato del BLASTP per tutte le proteine batteriofagiche dsDNA contro il database delle proteine NCBI Refseq (limitato alle proteine batteriche).I numeri riportati corrispondono al numero di proteine batteriofagiche dsDNA (arrotondato al migliaio più vicino).
L’entità della sovrapposizione tra pool di geni virali e cellulari
Una delle caratteristiche che definiscono i virus è la loro dipendenza dagli organismi ospitanti. È noto che le interazioni tra virus e cellule spesso portano allo scambio di informazioni genetiche. Per esplorare la misura in cui i pool di geni virali e cellulari si sovrappongono, abbiamo usato BLASTP per cercare proteine batteriche omologhe alle proteine batteriofagiche dsDNA (vedi Materiali e metodi). Nel complesso, ciascuno dei ~900 genomi batteriofagi dsDNA che abbiamo esaminato ha codificato almeno una proteina omologa ad una proteina batterica.
Per esaminare sistematicamente l’estensione dell’omologia tra batteriofago e proteine batteriche, abbiamo calcolato il numero di proteine per genoma batteriofago con omologia ad una proteina batterica e abbiamo diviso questo numero per il numero totale di proteine codificate dal genoma batteriofago. Nella Figura 10-figure supplement 1 ( a sinistra), dimostriamo l’istogramma della frazione di proteine omologhe per genoma batteriofago. Sulla base della frazione mediana delle proteine omologhe, possiamo concludere che 7 proteine batteriofagiche su 10 presentano omologia ad una proteina batterica. Ciò suggerisce che c’è una sovrapposizione significativa tra i due pool genici.
Ci sono molteplici meccanismi attraverso i quali una proteina batterica e una proteina batteriofaga potrebbero mostrare omologia. Il più banale, concettualmente, è quando la stessa proteina è registrata come parte sia di un genoma batterico che di un genoma batteriofago, come sarebbe per una proteina di un profago. Nel caso dei proffi, ci aspetteremmo di vedere un’alta frazione di proteine batteriofagiche per genoma che sono omologhe alle proteine batteriche poiché i loro genomi dovrebbero a un certo punto essere incorporati nei genomi dei loro ospiti.
Così, per esaminare il contributo dei profagi, abbiamo implementato diversi filtri per identificare i probabili genomi dei profagi (vedi Materiali e metodi). Sulla base di questi filtri, sono stati identificati 173 genomi. Questi genomi stavano principalmente contribuendo al grande picco nell’istogramma di sinistra in Figura 10-figure supplement 1. Per valutare questi filtri, abbiamo effettuato una ricerca in letteratura per i primi 20 genomi batteriofagi della lista e abbiamo scoperto che la maggior parte sono stati, infatti, sperimentalmente identificati come fago temperato. Poiché non siamo riusciti a trovare un database che contenesse un elenco di tutti i profagi verificati sperimentalmente e dei loro parenti litici con cui confrontare le nostre previsioni, non abbiamo escluso questi genomi da ulteriori analisi.
Un meccanismo non banale con cui i batteriofagi e i batteri possono esibire proteine omologhe è attraverso scambi di geni su scala temporale evolutiva. È interessante notare che l’omologo più vicino a una proteina batteriofaga non si trova sempre nel genoma del suo ospite. Infatti ci può essere una grande distanza tassonomica(Figura 10) tra l’ospite e il batterio che contiene l’omologo più vicino. Rappresentiamo questa distanza categorizzando le proteine batteriofagiche in base all’organismo in cui è stato trovato il loro omologo più vicino (vedi cerchi inscritti nella Figura 10). Se sono state trovate nella stessa specie di batteri dell’ospite, allora queste proteine sono collocate nel cerchio più interno, mentre se sono state trovate nello stesso phylum, le proteine sono collocate nel cerchio più esterno.
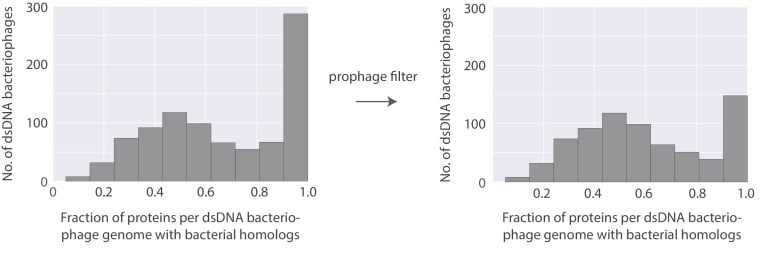
Figura 10-figure supplemento 1.Una rappresentazione della distanza tassonomica tra l’organismo ospite batteriofago e il batterio contenente l’omologo più vicino ad un batteriofago protein.Histogram della frazione di proteine per genoma batteriofago con omologhi batterici (a sinistra) e lo stesso istogramma con un filtro aggiuntivo per identificare i possibili profagi e i loro parenti litici (a destra).Tutti i cerchi sono disegnati in scala rispetto al numero di proteine (N) che ciascuno di essi rappresenta. Si noti che il numero di proteine indicate in ogni strato tassonomico comprende le proteine negli strati tassonomici inferiori. Ad esempio, la cifra di 20.000 indicate nello strato del genere comprende già le 11.000 proteine indicate nello strato della specie. I valori N sono arrotondati alle migliaia più vicine. Gli istogrammi della frazione di proteine con omologhi batterici per genoma batteriofago sono mostrati in Figura 10-figure supplement 1.
Possiamo vedere dalla Figura 10 che c’è una probabilità del 26% che l’omologo più vicino ad una proteina batteriofaga compaia in un membro della sua specie ospite. Questa possibilità è aumentata all’84% quando si suppone più ampiamente che l’omologo apparirà in un batterio che è almeno nello stesso phylum dell’ospite (Figura 10). Il valore di probabilità è calcolato dividendo il numero di proteine in un dato strato tassonomico per il numero totale di proteine nell’analisi.
Inoltre, un aspetto interessante di questo dataset diventa evidente quando esaminiamo la qualità della corrispondenza tra una proteina batteriofaga e il suo omologo batterico più vicino in funzione della distanza tassonomica tra l’ospite batteriofago e il batterio che contiene l’omologo. Abbiamo usato il bit score come misura della qualità della partita. Il bit score è un’uscita BLAST e una misura di somiglianza indipendente dalla dimensione del database o dalla lunghezza della sequenza di interrogazione. Identifica la dimensione di un database necessaria per trovare casualmente la stessa corrispondenza di qualità. Naturalmente, più alto è il bit score, migliore è la corrispondenza.
Possiamo vedere che c’è una significativa diminuzione del bit score mediano quando si passa dallo strato “stessa specie” allo strato “stesso genere” e infine allo strato “stesso phylum” (Figura 11). Così, più l’ospite è vicino (tassonomicamente) al batterio contenente l’omologo, migliore è la corrispondenza tra la proteina del batteriofago e il suo omologo batterico. Pensiamo che ci siano interessanti implicazioni co-evolutive faga-ospite che si possono concludere da questa analisi dei dati e metodo di visualizzazione dei dati, e speriamo di fare ulteriore luce su queste ipotesi in futuro.
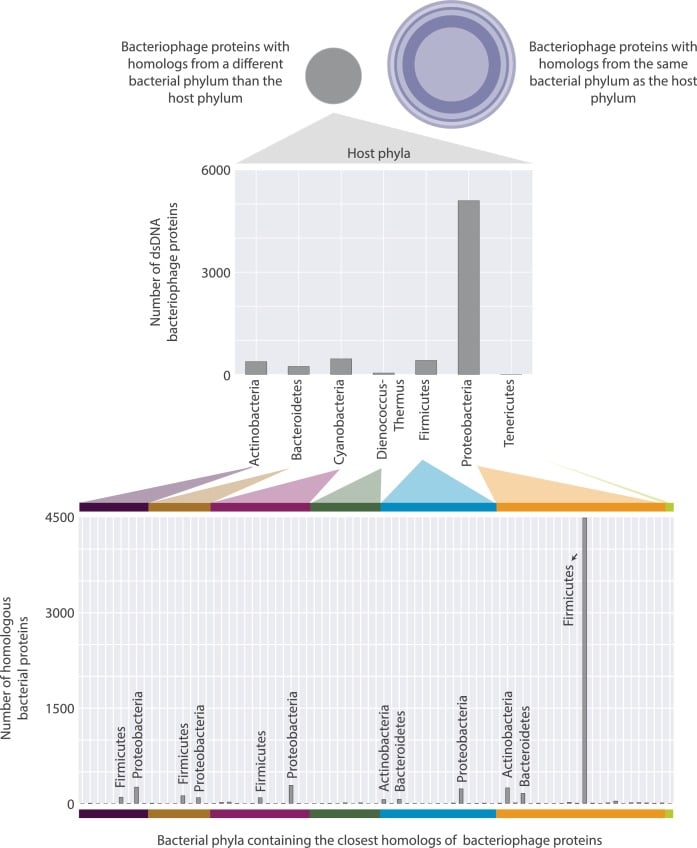
Figura 11-figure supplement 1.Figura 11— figura 1. Istogrammi dei punteggi dei bit che descrivono la corrispondenza tra ogni proteina batteriofaga e il suo omologo batterico più vicino.Gli istogrammi vengono creati in base alle proteine appartenenti a tre diversi strati corrispondenti ad una crescente distanza tassonomica tra l’organismo ospite e il batterio che contiene l’omologo più vicino.(A) Quando l’ospite e il batterio contenente l’omologo appartengono alla stessa specie, il punteggio medio del bit è significativamente più alto (test di Mann-Whitney U unilaterale, P<0,001) rispetto a quelli che sono solo parte dello stesso genere.(B) Analogamente, quando si confrontano le proteine dello strato “stessa specie” con lo strato “stesso phylum”, il punteggio medio del bit è significativamente più alto per lo strato “stessa specie” (test di Mann-Whitney U su un lato, P<0.001). Si noti che per ogni strato, quando si confrontano gli strati “stessa specie” e “stesso genere”, si confrontano le 11.000 proteine dello strato “stessa specie” con le 9.000 proteine dello strato “stesso genere” che non appartengono allo stesso strato “stessa specie”. Lo stesso principio si applica quando si confronta lo strato “stessa specie” con lo strato “stesso phylum”. Le distribuzioni di proteine batteriofagiche con omologhi di un phylum diverso dal loro phylum ospite sono mostrati in Figura 11-figure supplement 1.Queste proteine sono classificate in base al phylum del loro ospite (in alto), e poi in base al phylum dove appare il loro omologo più vicino (in basso). Ci sono 26 diversi phyla che gli omologhi batterici appaiono in, tuttavia, solo quelli che contengono il maggior numero di omologhi sono annotati per chiarezza visiva.
Mentre la maggior parte degli omologhi appartengono a membri dello stesso phylum come l’ospite, c’è ancora una probabilità del 16% che l’omologo batterico più vicino a una proteina batteriofago in realtà appare in un batterio da un phylum diverso rispetto all’ospite. Per esaminare ulteriormente queste associazioni di crossphyla, mappiamo la distribuzione delle proteine batteriofagiche in funzione del phylum dell’ospite. Poi, ingrandiamo il phyla batterico contenente gli omologhi(Figura 11-figure supplement 1). Di gran lunga, il maggior numero di omologhi di crossphyla sono condivisi tra i batteriofagi che infettano i Proteobatteri e i batteri del phylum Firmicutes. Sarebbe interessante esplorare in futuro la causa alla base del numero relativamente elevato di omologhi che esistono tra i membri microbici dei Firmicutes e dei Proteobatteri phyla.

Figura 10-figure supplemento 1.Una rappresentazione della distanza tassonomica tra l’organismo ospite batteriofago e il batterio contenente l’omologo più vicino ad un batteriofago protein.Histogram della frazione di proteine per genoma batteriofago con omologhi batterici (a sinistra) e lo stesso istogramma con un filtro aggiuntivo per identificare i possibili profagi e i loro parenti litici (a destra).Tutti i cerchi sono disegnati in scala rispetto al numero di proteine (N) che ciascuno di essi rappresenta. Si noti che il numero di proteine indicate in ogni strato tassonomico comprende le proteine negli strati tassonomici inferiori. Ad esempio, la cifra di 20.000 indicate nello strato del genere comprende già le 11.000 proteine indicate nello strato della specie. I valori N sono arrotondati alle migliaia più vicine. Gli istogrammi della frazione di proteine con omologhi batterici per genoma batteriofago sono mostrati in Figura 10-figure supplement 1.

Figura 10-figure supplement 1.2. Istogramma della frazione di proteine per genoma batteriofago con omologhi batterici (a sinistra) e lo stesso istogramma con un filtro aggiuntivo per identificare eventuali profagi e i loro parenti litici (a destra).

Figura 11-figure supplement 1.Figura 11— figura 1. Istogrammi dei punteggi dei bit che descrivono la corrispondenza tra ciascuna proteina batteriofaga e il suo omologo batterico più vicino.Gli istogrammi vengono creati in base alle proteine appartenenti a tre diversi strati corrispondenti ad una crescente distanza tassonomica tra l’organismo ospite e il batterio che contiene l’omologo più vicino.(A) Quando l’ospite e il batterio contenente l’omologo appartengono alla stessa specie, il punteggio medio del bit è significativamente più alto (test di Mann-Whitney U unilaterale, P<0,001) rispetto a quelli che sono solo parte dello stesso genere.(B) Analogamente, quando si confrontano le proteine dello strato “stessa specie” con lo strato “stesso phylum”, il punteggio medio del bit è significativamente più alto per lo strato “stessa specie” (test di Mann-Whitney U su un lato, P<0.001). Si noti che per ogni strato, quando si confrontano gli strati “stessa specie” e “stesso genere”, si confrontano le 11.000 proteine dello strato “stessa specie” con le 9.000 proteine dello strato “stesso genere” che non appartengono allo stesso strato “stessa specie”. Lo stesso principio si applica quando si confronta lo strato “stessa specie” con lo strato “stesso phylum”. Le distribuzioni di proteine batteriofagiche con omologhi di un phylum diverso dal loro phylum ospite sono mostrati in Figura 11-figure supplement 1.Queste proteine sono classificate in base al phylum del loro ospite (in alto), e poi in base al phylum dove appare il loro omologo più vicino (in basso). Ci sono 26 diversi phyla che gli omologhi batterici appaiono in, tuttavia, solo quelli che contengono il maggior numero di omologhi sono annotati per chiarezza visiva.

Figura 11-figure supplemento 1.1. Distribuzioni di proteine batteriofagiche con un omologo in un batterio da un phylum diverso dal loro phylum ospite.Queste proteine sono classificate in base al phylum del loro ospite (in alto), e poi in base al phylum dove appare il loro omologo più vicino (in basso). Ci sono 26 diversi phyla che gli omologhi batterici appaiono in, tuttavia, solo quelli che contengono il maggior numero di omologhi sono annotati per chiarezza visiva.
Discussione
La nostra motivazione principale per condurre uno studio su larga scala dei genomi virali è stata quella di fornire le distribuzioni dei numeri chiave che caratterizzano i genomi virali. Tuttavia, è importante notare che, mentre il database virale dell’NCBI rappresenta un’ampia collezione di genomi virali completi, esso rappresenta ancora una piccola frazione della diversità virale totale in natura. Alla luce delle sorprendenti tendenze genomiche osservate nei diversi gruppi virali, sono necessari studi futuri per riesaminare queste tendenze, dato che i nostri database crescono di dimensioni con una maggiore attenzione a diversi gruppi sottorappresentati, come i virus archeologici e i virus RNA batterici. A questo proposito, dopo aver riesaminato il database virale del NCBI nel 2018, siamo rimasti sorpresi nel constatare che, anche se il database è quasi raddoppiato, l’aumento ha favorito in modo sproporzionato i gruppi virali già ben rappresentati, per cui i gruppi sottorappresentati continuano ad essere sottorappresentati.
La nostra seconda motivazione per condurre questo studio è stata quella di confrontare diversi sistemi di classificazione virale. Poiché i sistemi di classificazione virale sono stati costruiti prima della comparsa del sequenziamento, eravamo interessati a vedere quanto bene possono descrivere le tendenze genomiche. Sulla base di un confronto dei sistemi di classificazione attraverso varie metriche genomiche, la classificazione di Baltimora e in alcuni casi la sua forma più minimale (Nucleotide Type classification) sembrano fornire la spiegazione più chiara per le tendenze osservate. Sospettiamo che ciò sia dovuto al discernimento della classificazione di Baltimora dei genomi RNA, ssDNA e dsDNA, che presentano differenze fisiche sorprendenti.
La maggiore stabilità del dsDNA rispetto all’RNA(Lindahl, 1993) e ssDNA è pensato per essere un fattore importante nelle variazioni osservate nelle lunghezze del genoma. Il gruppo 2′-idrossile in RNA lo rende più suscettibile agli eventi di idrolisi e la scissione della spina dorsale rispetto al DNA. È stato dimostrato che per batteri e virus, il tasso di mutazione e la lunghezza del genoma sono inversamente correlati(Drake, 1991; Sanjuán et al., 2010), ed è quindi ipotizzato che la mancanza di meccanismi di correzione nella replicazione dell’RNA e i conseguenti tassi di mutazione più elevati rispetto alla replicazione del DNA (Sanjuánet al., 2010) impone limiti di lunghezza ai genomi virali dell’RNA. A sostegno del sospetto legame tra tassi di mutazione e lunghezza del genoma, è stato dimostrato che i virus RNA lunghi (oltre 20 kb) contengono 3′-5′ esonucleasi, che è un omologo degli enzimi di correzione del DNA (Lauberet al., 2013).
Allo stesso modo, l’idrolisi della citosina in uracile avviene due ordini di grandezza più velocemente nei genomi ssDNA che nei genomi dsDNA(Frederico et al., 1990). Questo può spiegare gli elevati tassi di mutazione dei virus ssDNA, che rientrano nell’intervallo dei tassi di mutazione virale dell’RNA, nonostante l’utilizzo di polimerasi dell’ospite che correggono gli errori per replicarsi. In contrasto con la lunghezza del genoma in cui i virus ssDNA e RNA hanno distribuzioni simili, è stato interessante vedere che i virus ssDNA sono in realtà più simili ai virus dsDNA in termini di lunghezza del gene e percentuali non codificanti.
Mentre la classificazione di Baltimora serve come un significativo sistema di classificazione a grana grossa, è storicamente incentrata sui virus animali e trarrà vantaggio dall’essere ampliata per includere sottocategorie di virus batterici e archeologici. Come mostrato dalle distribuzioni della lunghezza dei geni (figura 4), lo strato aggiuntivo di categorizzazione fornito dalla classificazione del dominio host offre nuove prospettive. Ad esempio, i virus dsDNA e ssDNA degli eucarioti hanno una lunghezza genica molto più lunga rispetto ai loro omologhi procarioti – un’osservazione che può far pensare alla coevoluzione dei genomi e dei proteomi dell’ospite e virali, poiché i geni e le proteine eucarioti sono anche significativamente più lunghi di quelli procarioti(Brocchieri e Karlin, 2005; Zhang, 2000; Tiessen et al., 2012). È noto che alcuni genomi virali eucariotici, simili a quelli dei loro ospiti, contengono geni con introni (Himmelspachet al., 1995; Barksdale e Baker, 1995; Ge e Manley , 1990), il che può spiegare la lunghezza mediana più lunga dei geni dei virus eucariotici. Infatti lo splicing dell’mRNA è stato scoperto per la prima volta in uno studio sull’espressione dell’mRNA dell’adenovirus(Flint et al., 2000). È stato dimostrato che i proteomi dei virus sono anche sintonizzati con i proteomi dei loro ospiti, avendo un uso simile del codone e preferenze aminoacidiche (Bahiret al., 2009). Tuttavia, sono necessari studi futuri per accertare ulteriormente i meccanismi responsabili delle differenze nelle lunghezze dei geni virali eucarioti e procarioti.
La classificazione ICTV, che viene utilizzata forse più di qualsiasi altro sistema di classificazione per descrivere i virus batterici e archeologici, offre alcuni dati di supporto (ad esempio la morfologia virale o, in alcuni casi, le informazioni sull’ospite), forse come strato finale della classificazione. Tuttavia, è limitato dal fatto che lascia molti virus non classificati e, soprattutto, che manca di criteri di classificazione veramente sistematici. Man mano che la nostra esplorazione dei virus sposta la sua base dalla coltura dei virus al sequenziamento dei virus dal loro habitat naturale, i dati morfologici sono destinati a diventare sempre più scarsi. Di conseguenza, l’ICTV dovrà adattare il suo sistema di classificazione per operare esclusivamente sui dati genomici, un punto di vista ampiamente condiviso da molti esperti del settore(Simmonds et al., 2017).
In questo lavoro abbiamo descritto il nostro tentativo di fornire una visione completa e quantitativa dei genomi virali completamente sequenziati. Analogamente ai precedenti lavori sulla numerazione biologica, come esemplificato dal database BioNumbers(Milo et al., 2010), abbiamo identificato una serie di interessanti tendenze associate ai genomi virali che saranno utili per ottenere un’ampia panoramica di gruppi virali molto diversi tra loro e delle loro interazioni con i loro ospiti.
Materiali e metodi
Acquisizione dati, cura dei dati e analisi statistica
Tutti i dati genomici sono stati recuperati dal server FTP del Genoma del NCBI (recuperato nell’agosto 2015) (Bristeret al., 2015). La corrispondenza dei virus ai loro host è stata effettuata analizzando i file ASN dal server FTP NCBI Genome mentre si cercava il termine “nat-host”. Tutti gli altri dati tassonomici, compresi i lignaggi host e virali, sono stati recuperati dal database tassonomico del NCBI utilizzando la classe Taxa del NCBI Taxa dell’ETE Toolkit (Huerta-Cepaset al., 2010). Una volta che abbiamo avuto il nome “nat-host” degli organismi in inglese, abbiamo recuperato i loro taxidi usando l’ETE Toolkit. Questi sono stati a loro volta utilizzati per identificare il lignaggio tassonomico dell’ospite. I virus con genomi completi sono stati identificati cercando nei rapporti di assemblaggio del server FTP del Genoma NCBI Genome per gli assemblaggi etichettati come “Genoma completo”, quindi utilizzando l’indirizzo FTP associato per scaricare i file _assembly_stats.txt e _protein.faa. Per ulteriori analisi sono stati inclusi solo i virus che potevano essere abbinati a un host. Inoltre, sono stati effettuati manualmente diversi controlli di qualità per garantire che i virus con annotazioni non corrette fossero esclusi da ulteriori analisi. Ad esempio, abbiamo trovato virus e host con informazioni tassonomiche incomplete o errate ed abbiamo escluso questi virus da ulteriori analisi. L’elenco dei virus esclusi si trova nel nostro codice (vedi sottocapitolo successivo). Gli outlier non sono esclusi dalla nostra analisi. Cerchiamo di attenuarne l’effetto concentrandoci sui valori mediani piuttosto che sulla media(tabella 1). Data la presenza di alcune distribuzioni oblique, abbiamo utilizzato principalmente il test di Mann-Whitney U per l’analisi statistica, in modo da evitare l’ipotesi di normalità.
Disponibilità dei dati
Abbiamo compilato tutti i dati di input, i file di output e gli script (notebook Jupyter) utilizzati per scrivere questo manoscritto in un repository GitHub(https://github.com/gitamahm/VirologyByTheNumbers)(Mahmoudabadi, 2018). viromePieChartsVF.ipynb e virusHostHistogramsVF.ipynb sono stati utilizzati per creare la Figura 2. Gli script da Figura 3 a Figura 5 si trovano rispettivamente in genomeLengthsVF.ipynb, geneLengthsVF.ipynb e percentNoncodingVF.ipynb. Il codice per la Figura 6 e la Figura 7 è fornito in geneOrderAndGeneAbundanceVF.ipynb. viralAttachmentSites.ipynb è utilizzato per creare la Figura 8 e viralBacterialBlast.ipynb è utilizzato per creare(Figure 9- 11e le loro figure supplementari).
Tutti i file di testo supplementari si trovano anche in questo repository. Il file di testo supplementareFile-1.txt mostra le sequenze di ordine dei geni per tutti i virus i cui genomi contengono almeno il 15% di geni etichettati. Le lettere I, C, E e Q corrispondono rispettivamente ai geni relativi a capsid-related, portal-related, tail-related e genome packaging-related. Tutti gli altri geni sono indicati dalla lettera A. Testo supplementareFile-2.txt contiene l’elenco delle migliori corrispondenze BLASTP per le proteine batteriofagiche che avevano omologhi di proteine batteriche (la corrispondenza superiore è considerata come la corrispondenza con il valore E più basso). Il testo supplementare textFile-3.txt fornisce le annotazioni degli omologhi batterici delle proteine batteriofagiche ipotetiche. Il testo supplementare textFile-4.txt contiene le annotazioni di omologhi batteriofagi di ipotetiche proteine batteriche.
Lunghezza del genoma e densità del gene
Le lunghezze del genoma sono state estratte da file .ptt e da file _assembly_stats.txt per i virus. I file .ptt sono stati analizzati per trovare “genoma completo – 1.” che è seguito dalla lunghezza del genoma. Per i genomi segmentati, la lunghezza totale dei segmenti è riportata come lunghezza del genoma. Il numero di geni che codificano le proteine, che è stato utilizzato nel calcolo delle densità geniche, è stato trovato analizzando i file .faa. Per gli istogrammi della lunghezza dei geni, abbiamo prima ottenuto le lunghezze dei geni per ogni virus, e poi abbiamo creato un istogramma basato sulla lunghezza mediana dei geni associata a ciascun virus. Per determinare sistematicamente il numero di bins necessari per ogni istogramma, abbiamo utilizzato la regola di Freedman-Diaconis (Freedmane Diaconis, 1981).
Percentuali di DNA/RNA non codificanti
Per estrarre la percentuale del genoma che non è codificante, non potremmo semplicemente sottrarre le lunghezze dei geni dalla lunghezza del genoma, perché questo non terrebbe conto dei geni che si sovrappongono. Al contrario, abbiamo usato i file .ptt per identificare dove ogni gene iniziava e finiva nel genoma, quindi abbiamo aggiunto tutti gli indici tra i geni che codificano le proteine a un insieme. Abbiamo quindi potuto sottrarre la dimensione di questo insieme dalla lunghezza del genoma per arrivare al numero di basi non codificanti, che viene poi trasformato in una percentuale.
Decomposizione dei geni virali in categorie funzionali
Per ottenere l’abbondanza di varie categorie funzionali dei geni, abbiamo raccolto le annotazioni dei prodotti COG(Tatusov et al., 2000) che accompagnano ogni gene da file .ptt forniti per ogni virus. Sulla base dei nomi dei prodotti COG più frequenti, abbiamo costruito un dizionario dei termini di ricerca per interrogare i geni virali e misurare l’abbondanza di varie categorie funzionali (misurando l’abbondanza, ci riferiamo al numero di geni che appartengono a una data categoria funzionale). Per determinare i termini di ricerca più comuni, abbiamo ricavato l’insieme unico di annotazioni dei prodotti COG per i diversi viromi. Abbiamo utilizzato le annotazioni condivise tra i viromi per escludere termini di ricerca problematici con significati multipli. Come risultato abbiamo evitato termini di ricerca con associazioni funzionali multiple come “gp41”, che nel contesto dell’HIV significa una glicoproteina transmembrana, e nel contesto del Mycobacterium phage Bxb1 denota un’esonucleasi 3′-5” coinvolta nella replicazione del DNA.
Mentre il dizionario costruito contiene molte parole chiave che catturano categorie funzionali essenziali del gene comuni a molti virus, non tiene conto delle annotazioni COG che non sono descrittive (ad esempio “proteina fago” o “proteina Z”). Inoltre, c’è tipicamente un gran numero di geni che codificano per “proteine ipotetiche”. Insieme, queste due frazioni costituiscono la componente senza etichetta, che non includiamo per ulteriori analisi. Nonostante le limitazioni introdotte da questi geni senza etichetta, ci sono ancora un gran numero di geni (~105) che sono inclusi nella nostra analisi. Nel costruire le abbondanze relative delle diverse categorie funzionali dei geni(Figura 6), dividiamo l’abbondanza di una categoria funzionale genica per il numero totale di geni etichettati (indicato in cima alla Figura 6.A per ogni gruppo virale).
Ordine dei geni
Nella visualizzazione dell’ordine dei geni abbiamo utilizzato una strategia di ricerca simile a quella spiegata nella sezione precedente. Per individuare i modelli potenzialmente conservati in ordine genico in genomi virali molto diversi, abbiamo cercato solo i geni strutturali, in quanto sono essenziali per qualsiasi virus. Abbiamo usato i file .ptt per determinare l’ordine dei geni in quanto contengono gli indici di inizio e fine dei geni. Il codice sviluppato utilizza i file .ptt come input e produce una stringa di caratteri per genoma virale, che abbiamo chiamato sequenza di ordine genico. Ogni carattere rappresenta un gene virale nell’ordine in cui appare sul genoma (senza distinguere il filamento di DNA su cui si trova il gene). Tutti i geni appartenenti alla stessa categoria funzionale, ad esempio tutti i geni legati alla coda, sono rappresentati dallo stesso carattere. Tutti i geni non etichettati (cioè i geni non strutturali, ipotetici o scarsamente annotati) sono anch’essi rappresentati dallo stesso carattere. Ogni sequenza di ordine genico, analoga a una sequenza nucleotidica, può essere allineata con altre sequenze di ordine genico mediante un software di allineamento esistente.
Anche se sarebbe ideale calcolare una matrice di distanza a coppie tra le sequenze di ordine genico e definire quantitativamente un modello di ordine genico basato sulla somiglianza delle sequenze di ordine genico (simile alla definizione di un’unità tassonomica operativa), questo sforzo richiederebbe lo sviluppo di algoritmi di allineamento appropriati e metodi di inferenza adatti ad elaborare le sequenze di ordine genico. Nel frattempo, abbiamo usato il software di allineamento esistente come guida e abbiamo raggruppato le sequenze di ordine dei geni sulla base di caratteristiche generalmente condivise.
Abbiamo usato il software Geneious(Kearse et al., 2012) per allineare le sequenze di ordine dei geni utilizzando l’allineamento globale con i gap di estremità libera e la matrice dei costi di identità (con gap di default aperti e penalità di estensione). Utilizzando l’allineamento globale di Geneious come guida, abbiamo ulteriormente migliorato manualmente l’allineamento allineando un sottomodello ampiamente condiviso, per esempio i geni del portale/collo nel modello A o i caratteri capsid e tail nel modello C, senza introdurre alcun gap. Questo passo è stato necessario perché qualsiasi algoritmo di allineamento mira a massimizzare l’allineamento tra i geni non etichettati, incapaci di distinguere tra questi caratteri e i caratteri più significativi corrispondenti ai geni strutturali etichettati. Inoltre, a causa dell’elevata frazione di geni che hanno un’annotazione COG di “ipotetica proteina”, abbiamo dovuto imporre filtri per estrarre sequenze di ordine genico che non sono interamente composte da geni non etichettati. Per generare gli allineamenti mostrati in figura 7, abbiamo imposto che almeno il 15% dei caratteri in una sequenza di ordine genico deve corrispondere a geni etichettati, e che la sequenza di ordine genico deve essere lunga almeno 40 caratteri. Per le sequenze di ordine genico mostrate in textFile-1.txt (vedere il repository GitHub) il limite di lunghezza dell’ordine di sequenza non è stato imposto.
Siti di attaccamento dei batteriofagi
Per esplorare i siti di attaccamento dei batteriofagi, abbiamo utilizzato il programma PHASTER per ottenere siti di attaccamento putativo(Arndt et al., 2016). Utilizzando i numeri di accesso al genoma del fago e l’API dell’URL PHASTER, abbiamo ottenuto informazioni sulla sequenza e la posizione del sito dell’allegato. Abbiamo analizzato e visualizzato questi dati utilizzando il nostro set di script, che si trovano nel notebook attachmentSites.ipynb. Quando abbiamo confrontato le sequenze dei siti allegati, abbiamo selezionato coppie di pagine con una somiglianza del 100% nel loro allineamento. Abbiamo anche imposto che la lunghezza di allineamento fosse di almeno 8 bp (che è più della metà della lunghezza mediana del sito dell’allegato). La Figura 8 – dati fonte 1 mostra i faggi che soddisfano questi criteri.
BLASSAGGIO delle proteine batteriofagiche del dsDNA contro le proteine batteriche
Il BLASTP multithreaded su questo database è stato un processo intensivo dal punto di vista dei calcoli, che ha richiesto oltre 8900 ore di lavoro. Utilizzando script personalizzati in R, i risultati taxID di ogni proteina interrogata sono stati collegati a lignaggi completi utilizzando NCBI taxdump e NCBItax2lin (disponibile su https://github.com/zyxue/ncbitax2lin). Un elenco di ipotetiche proteine batteriofagiche e i loro omologhi batterici più vicini sono forniti in textFile-3.txt (vedi il nostro repository GitHub). Analogamente, le ipotetiche proteine batteriche e le annotazioni dei loro omologhi batteriofagi più vicini sono fornite in textFile-4.txt. Gli omologhi più vicini sono determinati in base alla partita con il valore E più basso. Una partita è stata presa in considerazione solo se aveva un valore E < 10-10.
Identificazione dei genomi dei profagi putativi
Abbiamo sospettato che le proteine del profago avrebbero avuto un’alta percentuale di identità ai loro omologhi batterici, e quindi, abbiamo prima filtrato le proteine con meno del 50% di identità ai loro omologhi batterici. Allora abbiamo selezionato soltanto le proteine con gli omologhi batterici se il batterio che contiene l’omologo era la stessa specie dell’ospite del batteriofago. Infine, abbiamo richiesto che almeno la metà delle proteine per genoma batteriofago soddisfacesse le condizioni sopra descritte per l’identificazione del batteriofago come potenziale profago. La ragione per cui non abbiamo imposto filtri più rigorosi è stata quella di poter identificare anche eventuali parenti litici dei profagi, in quanto le loro proteine sarebbero state percepite anche omologhe alle proteine batteriche, ma solo a causa della loro omologia alle proteine del batteriofago.
References
- Alberts B, Johnson A, Lewis J, Walter P, Raff M, Roberts K. Molecular Biology of the Cell 4th Edition: International Student Edition. Routledge; 2002.
- Arndt D, Grant JR, Marcu A, Sajed T, Pon A, Liang Y, Wishart DS. PHASTER: a better, faster version of the PHAST phage search tool. Nucleic Acids Research. 2016; 44:W16-W21. DOI | PubMed
- Bahir I, Fromer M, Prat Y, Linial M. Viral adaptation to host: a proteome-based analysis of codon usage and amino acid preferences. Molecular Systems Biology. 2009; 5DOI | PubMed
- Baltimore D. Expression of animal virus genomes. Bacteriological Reviews. 1971; 35:235-241. PubMed
- Barksdale S, Baker CC. Differentiation-specific alternative splicing of bovine papillomavirus late mRNAs. Journal of Virology. 1995; 69:6553-6556. PubMed
- Brandes N, Linial M. Gene overlapping and size constraints in the viral world. Biology Direct. 2016; 11DOI | PubMed
- Brister JR, Ako-Adjei D, Bao Y, Blinkova O. NCBI viral genomes resource. Nucleic Acids Research. 2015; 43:D571-D577. DOI | PubMed
- Brocchieri L, Karlin S. Protein length in eukaryotic and prokaryotic proteomes. Nucleic Acids Research. 2005; 33:3390-3400. DOI | PubMed
- Brüssow H, Hendrix RW. Phage genomics: small is beautiful. Cell. 2002; 108:13-16. DOI | PubMed
- Casjens SR, Gilcrease EB, Winn-Stapley DA, Schicklmaier P, Schmieger H, Pedulla ML, Ford ME, Houtz JM, Hatfull GF, Hendrix RW. The generalized transducing Salmonella bacteriophage ES18: complete genome sequence and DNA packaging strategy. Journal of Bacteriology. 2005; 187:1091-1104. DOI | PubMed
- Drake JW. A constant rate of spontaneous mutation in DNA-based microbes. PNAS. 1991; 88:7160-7164. DOI | PubMed
- Dutilh BE, Cassman N, McNair K, Sanchez SE, Silva GG, Boling L, Barr JJ, Speth DR, Seguritan V, Aziz RK, Felts B, Dinsdale EA, Mokili JL, Edwards RA. A highly abundant bacteriophage discovered in the unknown sequences of human faecal metagenomes. Nature Communications. 2014; 5DOI | PubMed
- Edwards RA, Rohwer F. Viral metagenomics. Nature Reviews Microbiology. 2005; 3:504-510. DOI | PubMed
- Elgar G, Vavouri T. Tuning in to the signals: noncoding sequence conservation in vertebrate genomes. Trends in Genetics. 2008; 24:344-352. DOI | PubMed
- Firth AE, Brierley I. Non-canonical translation in RNA viruses. Journal of General Virology. 2012; 93:1385-1409. DOI | PubMed
- Flint SJ, Enquist LW, Racaniello VR, Skalka AM. Principles of Virology: Molecular Biology, Pathogenesis, and Control of Animal Viruses. ASM Press: Washington, DC; 2000.
- Forterre P. Defining life: the virus viewpoint. Origins of Life and Evolution of Biospheres. 2010; 40:151-160. DOI | PubMed
- Frederico LA, Kunkel TA, Shaw BR. A sensitive genetic assay for the detection of cytosine deamination: determination of rate constants and the activation energy. Biochemistry. 1990; 29:2532-2537. DOI | PubMed
- Freedman D, Diaconis P. On the histogram as a density estimator: L2 theory. Zeitschrift für Wahrscheinlichkeitstheorie und Verwandte Gebiete. 1981; 57:453-476. DOI
- Ge H, Manley JL. A protein factor, ASF, controls cell-specific alternative splicing of SV40 early pre-mRNA in vitro. Cell. 1990; 62:25-34. DOI | PubMed
- Grilli J, Bassetti B, Maslov S, Cosentino Lagomarsino M. Joint scaling laws in functional and evolutionary categories in prokaryotic genomes. Nucleic Acids Research. 2012; 40:530-540. DOI | PubMed
- Hendrix RW. Bacteriophage genomics. Current Opinion in Microbiology. 2003; 6:506-511. DOI | PubMed
- Himmelspach M, Cavaloc Y, Chebli K, Stévenin J, Gattoni R. Titration of serine/arginine (SR) splicing factors during adenoviral infection modulates E1A pre-mRNA alternative splicing. RNA. 1995; 1:794-806. PubMed
- Hou C, Li L, Qin ZS, Corces VG. Gene density, transcription, and insulators contribute to the partition of the Drosophila genome into physical domains. Molecular Cell. 2012; 48:471-484. DOI | PubMed
- Howe JA, Wang H, Fischmann TO, Balibar CJ, Xiao L, Galgoci AM, Malinverni JC, Mayhood T, Villafania A, Nahvi A, Murgolo N, Barbieri CM, Mann PA, Carr D, Xia E, Zuck P, Riley D, Painter RE, Walker SS, Sherborne B, de Jesus R, Pan W, Plotkin MA, Wu J, Rindgen D, Cummings J, Garlisi CG, Zhang R, Sheth PR, Gill CJ, Tang H, Roemer T. Selective small-molecule inhibition of an RNA structural element. Nature. 2015; 526:672-677. DOI | PubMed
- Huerta-Cepas J, Dopazo J, Gabaldón T. ETE: a python environment for tree exploration. BMC Bioinformatics. 2010; 11DOI | PubMed
- Hug LA, Baker BJ, Anantharaman K, Brown CT, Probst AJ, Castelle CJ, Butterfield CN, Hernsdorf AW, Amano Y, Ise K, Suzuki Y, Dudek N, Relman DA, Finstad KM, Amundson R, Thomas BC, Banfield JF. A new view of the tree of life. Nature Microbiology. 2016; 1DOI | PubMed
- Hulo C, de Castro E, Masson P, Bougueleret L, Bairoch A, Xenarios I, Le Mercier P. ViralZone: a knowledge resource to understand virus diversity. Nucleic Acids Research. 2011; 39:D576-D582. DOI | PubMed
- Jaillon O, Aury JM, Brunet F, Petit JL, Stange-Thomann N, Mauceli E, Bouneau L, Fischer C, Ozouf-Costaz C, Bernot A, Nicaud S, Jaffe D, Fisher S, Lutfalla G, Dossat C, Segurens B, Dasilva C, Salanoubat M, Levy M, Boudet N, Castellano S, Anthouard V, Jubin C, Castelli V, Katinka M, Vacherie B, Biémont C, Skalli Z, Cattolico L, Poulain J, De Berardinis V, Cruaud C, Duprat S, Brottier P, Coutanceau JP, Gouzy J, Parra G, Lardier G, Chapple C, McKernan KJ, McEwan P, Bosak S, Kellis M, Volff JN, Guigó R, Zody MC, Mesirov J, Lindblad-Toh K, Birren B, Nusbaum C, Kahn D, Robinson-Rechavi M, Laudet V, Schachter V, Quétier F, Saurin W, Scarpelli C, Wincker P, Lander ES, Weissenbach J, Roest Crollius H. Genome duplication in the teleost fish Tetraodon nigroviridis reveals the early vertebrate proto-karyotype. Nature. 2004; 431:946-957. DOI | PubMed
- Kearse M, Moir R, Wilson A, Stones-Havas S, Cheung M, Sturrock S, Buxton S, Cooper A, Markowitz S, Duran C, Thierer T, Ashton B, Meintjes P, Drummond A. Geneious basic: an integrated and extendable desktop software platform for the organization and analysis of sequence data. Bioinformatics. 2012; 28:1647-1649. DOI | PubMed
- Keller B, Feuillet C. Colinearity and gene density in grass genomes. Trends in Plant Science. 2000; 5:246-251. DOI | PubMed
- King AMQ, Lefkowitz E, Adams MJ, Casrstens EB. Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier; 2011.
- Koonin EV, Dolja VV, Krupovic M. Origins and evolution of viruses of eukaryotes: the ultimate modularity. Virology. 2015; 479-480:2-25. DOI | PubMed
- Kristensen DM, Mushegian AR, Dolja VV, Koonin EV. New dimensions of the virus world discovered through metagenomics. Trends in Microbiology. 2010; 18:11-19. DOI | PubMed
- Kristensen DM, Cai X, Mushegian A. Evolutionarily conserved orthologous families in phages are relatively rare in their prokaryotic hosts. Journal of Bacteriology. 2011; 193:1806-1814. DOI | PubMed
- Labonté JM, Swan BK, Poulos B, Luo H, Koren S, Hallam SJ, Sullivan MB, Woyke T, Wommack KE, Stepanauskas R. Single-cell genomics-based analysis of virus-host interactions in marine surface bacterioplankton. The ISME Journal. 2015; 9:2386-2399. DOI | PubMed
- Lauber C, Goeman JJ, Parquet MC, Nga PT, Snijder EJ, Morita K, Gorbalenya AE. The footprint of genome architecture in the largest genome expansion in RNA viruses. PLoS Pathogens. 2013; 9DOI | PubMed
- Lindahl T. Instability and decay of the primary structure of DNA. Nature. 1993; 362:709-715. DOI | PubMed
- Mahmoudabadi G. GitHub. 2018. Publisher Full Text
- Mahy BWJ, Van Regenmortel MHV. Desk Encyclopedia of Human and Medical Virology. Academic Press; 2010.
- Marbouty M, Baudry L, Cournac A, Koszul R. Scaffolding bacterial genomes and probing host-virus interactions in gut microbiome by proximity ligation (chromosome capture) assay. Science Advances. 2017; 3DOI | PubMed
- Marinelli LJ, Fitz-Gibbon S, Hayes C, Bowman C, Inkeles M, Loncaric A, Russell DA, Jacobs-Sera D, Cokus S, Pellegrini M, Kim J, Miller JF, Hatfull GF, Modlin RL. Propionibacterium acnes bacteriophages display limited genetic diversity and broad killing activity against bacterial skin isolates. mBio. 2012; 3DOI
- Mathews MB, Shenk T. Adenovirus virus-associated RNA and translation control. Journal of Virology. 1991; 65PubMed
- Mattick JS. RNA regulation: a new genetics?. Nature Reviews Genetics. 2004; 5:316-323. DOI | PubMed
- Mattick JS, Makunin IV. Non-coding RNA. Human Molecular Genetics. 2006; 15:R17-R29. DOI | PubMed
- Melgarejo TA, Kon T, Rojas MR, Paz-Carrasco L, Zerbini FM, Gilbertson RL. Characterization of a new world monopartite begomovirus causing leaf curl disease of tomato in Ecuador and Peru reveals a new direction in geminivirus evolution. Journal of Virology. 2013; 87:5397-5413. DOI | PubMed
- Milo R, Jorgensen P, Moran U, Weber G, Springer M. BioNumbers–the database of key numbers in molecular and cell biology. Nucleic Acids Research. 2010; 38:D750-D753. DOI | PubMed
- Milo R, Phillips R. Cell Biology by the Numbers. Garland Science; 2015.
- Mokili JL, Rohwer F, Dutilh BE. Metagenomics and future perspectives in virus discovery. Current Opinion in Virology. 2012; 2:63-77. DOI | PubMed
- Molina N, van Nimwegen E. Scaling laws in functional genome content across prokaryotic clades and lifestyles. Trends in Genetics. 2009; 25:243-247. DOI | PubMed
- Morris KV. Non-Coding RNAs and Epigenetic Regulation of Gene Expression: Drivers of Natural Selection. Horizon Scientific Press; 2012.
- Nielsen HB, Almeida M, Juncker AS, Rasmussen S, Li J, Sunagawa S, Plichta DR, Gautier L, Pedersen AG, Le Chatelier E, Pelletier E, Bonde I, Nielsen T, Manichanh C, Arumugam M, Batto JM, Quintanilha Dos Santos MB, Blom N, Borruel N, Burgdorf KS, Boumezbeur F, Casellas F, Doré J, Dworzynski P, Guarner F, Hansen T, Hildebrand F, Kaas RS, Kennedy S, Kristiansen K, Kultima JR, Léonard P, Levenez F, Lund O, Moumen B, Le Paslier D, Pons N, Pedersen O, Prifti E, Qin J, Raes J, Sørensen S, Tap J, Tims S, Ussery DW, Yamada T, Renault P, Sicheritz-Ponten T, Bork P, Wang J, Brunak S, Ehrlich SD, MetaHIT Consortium. Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nature Biotechnology. 2014; 32:822-828. DOI | PubMed
- Paez-Espino D, Eloe-Fadrosh EA, Pavlopoulos GA, Thomas AD, Huntemann M, Mikhailova N, Rubin E, Ivanova NN, Kyrpides NC. Uncovering Earth’s virome. Nature. 2016; 536:425-430. DOI | PubMed
- Phillips R, Milo R. A feeling for the numbers in biology. PNAS. 2009; 106:21465-21471. DOI | PubMed
- Phillips R, Kondev J, Theriot J, Garcia H. Physical Biology of the Cell. Garland Science; 2012.
- Reich PR, Forget BG, Weissman SM, Rose JA. RNA of low molecular weight in KB cells infected with adenovirus type 2. Journal of Molecular Biology. 1966; 17:428-439. DOI | PubMed
- Riaz A, Dry I, Levy CS, Hopkins J, Grey F, Shaw DJ, Dalziel RG. Ovine herpesvirus-2-encoded microRNAs target virus genes involved in virus latency. Journal of General Virology. 2014; 95:472-480. DOI | PubMed
- Rohwer F, Thurber RV. Viruses manipulate the marine environment. Nature. 2009; 459:207-212. DOI | PubMed
- Roux S, Brum JR, Dutilh BE, Sunagawa S, Duhaime MB, Loy A, Poulos BT, Solonenko N, Lara E, Poulain J, Pesant S, Kandels-Lewis S, Dimier C, Picheral M, Searson S, Cruaud C, Alberti A, Duarte CM, Gasol JM, Vaqué D, Bork P, Acinas SG, Wincker P, Sullivan MB, Tara Oceans Coordinators. Ecogenomics and potential biogeochemical impacts of globally abundant ocean viruses. Nature. 2016; 537:689-693. DOI | PubMed
- Sanjuán R, Nebot MR, Chirico N, Mansky LM, Belshaw R. Viral mutation rates. Journal of Virology. 2010; 84:9733-9748. DOI | PubMed
- Shi M, Lin XD, Tian JH, Chen LJ, Chen X, Li CX, Qin XC, Li J, Cao JP, Eden JS, Buchmann J, Wang W, Xu J, Holmes EC, Zhang YZ. Redefining the invertebrate RNA virosphere. Nature. 2016; 540:539-543. DOI | PubMed
- Sievers F, Wilm A, Dineen D, Gibson TJ, Karplus K, Li W, Lopez R, McWilliam H, Remmert M, Söding J, Thompson JD, Higgins DG. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega. Molecular Systems Biology. 2011; 7DOI | PubMed
- Simmonds P. Methods for virus classification and the challenge of incorporating metagenomic sequence data. Journal of General Virology. 2015; 96:1193-1206. DOI | PubMed
- Simmonds P, Adams MJ, Benkő M, Breitbart M, Brister JR, Carstens EB, Davison AJ, Delwart E, Gorbalenya AE, Harrach B, Hull R, King AM, Koonin EV, Krupovic M, Kuhn JH, Lefkowitz EJ, Nibert ML, Orton R, Roossinck MJ, Sabanadzovic S, Sullivan MB, Suttle CA, Tesh RB, van der Vlugt RA, Varsani A, Zerbini FM. Consensus statement: virus taxonomy in the age of metagenomics. Nature Reviews Microbiology. 2017; 15:161-168. DOI | PubMed
- Steitz J, Borah S, Cazalla D, Fok V, Lytle R, Mitton-Fry R, Riley K, Samji T. Noncoding RNPs of viral origin. Cold Spring Harbor Perspectives in Biology. 2011; 3DOI | PubMed
- Suttle CA. Viruses in the sea. Nature. 2005; 437:356-361. DOI | PubMed
- Tatusov RL, Galperin MY, Natale DA, Koonin EV. The COG database: a tool for genome-scale analysis of protein functions and evolution. Nucleic Acids Research. 2000; 28:33-36. DOI | PubMed
- Telford MJ, Copley RR. Improving animal phylogenies with genomic data. Trends in Genetics. 2011; 27:186-195. DOI | PubMed
- Tiessen A, Pérez-Rodríguez P, Delaye-Arredondo LJ. Mathematical modeling and comparison of protein size distribution in different plant, animal, fungal and microbial species reveals a negative correlation between protein size and protein number, thus providing insight into the evolution of proteomes. BMC Research Notes. 2012; 5DOI | PubMed
- Tycowski KT, Guo YE, Lee N, Moss WN, Vallery TK, Xie M, Steitz JA. Viral noncoding RNAs: more surprises. Genes & Development. 2015; 29:567-584. DOI | PubMed
- Wigington CH, Sonderegger D, Brussaard CP, Buchan A, Finke JF, Fuhrman JA, Lennon JT, Middelboe M, Suttle CA, Stock C, Wilson WH, Wommack KE, Wilhelm SW, Weitz JS. Re-examination of the relationship between marine virus and microbial cell abundances. Nature Microbiology. 2016; 1DOI | PubMed
- Youle M, Haynes M, Rohwer F. Viruses: Essential Agents of Life. Springer; 2012.
- Zhang J. Protein-length distributions for the three domains of life. Trends in Genetics. 2000; 16:107-109. DOI | PubMed
Fonte
Mahmoudabadi G, Phillips R, Chakraborty AK () A comprehensive and quantitative exploration of thousands of viral genomes. eLife 7e31955. https://doi.org/10.7554/eLife.31955





