Abstract
Introduzione
L’habitat sotterraneo è stato colonizzato da numerose specie animali per il suo riparo e per le sue uniche fonti di cibo(Andersen, 1987; Nevo, 1979). Le specie fossili obbligatorie, in particolare, hanno adottato il sottosuolo come una casa dedicata, ma le intense esigenze della vita sotterranea richiedono spesso specializzazioni uniche. Per esempio, l’aria presente nelle gallerie è spesso povera di ossigeno (ipossica) e ad alto contenuto di anidride carbonica (ipercapnica) (Nevo,1979). Questo ambiente buio richiede anche lo sviluppo di sensi potenziati per compensare la perdita della vista. Queste ed altre specializzazioni sotterranee sono state segnalate in molti lignaggi evolutivi indipendenti di insetti, anfibi, rettili e mammiferi(Leys et al., 2003; Lacey et al., 2000; Albert et al., 2007; Wilkinson, 2012). All’interno dei soli mammiferi, ci sono diverse specie sotterranee non correlate, tra cui le talpe vere e proprie (famiglia Talpidae), le talpe dorate africane (Chrysochloridae) e le talpe marsupiali (Notoryctidae). Ci sono anche almeno tre lignaggi non correlati di roditori sotterranei: il topo talpa nudo(Heterocephalus glaber), il topo talpa cieco (Spalacidae) e il topo tascabile (Geomyidae).
I mammiferi fossili hanno sviluppato una serie di tratti morfologici e fisiologici che sono considerati adattamenti alla vita sotterranea, influenzando il modo in cui percepiscono il loro ambiente, come si muovono attraverso di esso, o come si occupano delle sue esigenze fisiologiche. Per esempio, le specie che scavano con gli arti anteriori, come la talpa dal naso a stella, hanno arti anteriori ingrossati e artigli che permettono loro di scavare un tunnel attraverso il substrato, mentre le specie che scavano con i denti, come il topo talpa nudo, hanno arti ridotti e incisivi in continua crescita(Dubost, 1968; Ellerman, 1956). In assenza di luce, sono stati elaborati sistemi sensoriali non visivi, come le vibrisse sensibili (cioè i peli sensoriali) del neo-tratto nudo e il muso grande ed elaborato del neo dal naso stellato, che è diventato un notevole “occhio tattile” (Eloff, 1951; Hillet al., 2009; Catania, 1999). Anche i mammiferi fossili hanno sviluppato una capacità di resistenza alle condizioni ipossiche che rivaleggia con quella delle specie che vivono in alta quota (Nevo, 1979; Ar et al., 1977). Ad esempio, alcune specie hanno un elevato numero di eritrociti e un elevato numero di mioglobina scheletro-muscolare per facilitare lo scambio di ossigeno. Alcune di queste strategie di adattamento sono condivise da diversi lignaggi fossili e come tali rappresentano esempi di evoluzione convergente(Nevo, 1979). Tuttavia, alcune delle trasformazioni convergenti più eclatanti nei mammiferi sotterranei sono le riduzioni e le perdite di tratti condivisi tra i mammiferi in superficie, di cui l’esempio più evidente è la riduzione dell’occhio(Dubost, 1968; Hill et al., 2009; Cei, 1946a, 1946b).
La vista in molti mammiferi sotterranei è limitata, e il grado di limitazione in ogni specie è legato all’estensione della sua abitazione sotterranea(Nemec et al., 2008; Quilliam, 2009; Sanyal et al., 1990). Ad esempio, le talpe dal naso stellato(Condylura cristata) che condividono il loro tempo fuori terra e nel sottosuolo possiedono occhi minuscoli con palpebre spesse(Catania, 1999), mentre il topo talpa nudo, che passa quasi tutto il suo tempo sottoterra, ha occhi minuscoli che raramente vengono aperti (Hetlinget al., 2005). Ancora più estremi sono gli occhi completamente sottocutanei della talpa dorata del mantello(Chrysochloris asiatica) e del topo cieco (genere Nannospalax), che si pensa riflettano il loro stile di vita strettamente sotterraneo(Sanyal et al., 1990; Sweet, 1909). Mentre un certo grado di regressione visiva è condiviso tra i mammiferi sotterranei, non tutte le strutture visive e i percorsi genetici sono regrediti allo stesso grado. Ad esempio, gli occhi dei veri nei e dei veri topi di talpa mostrano una regressione anatomica e mostrano sempre un occhio piccolo, ma conservano l’architettura oculare, suggerendo che i programmi di base dello sviluppo oculare devono essere in gran parte intatti in questi animali (Carmonaet al., 2008, 2010; Quilliam , 1966). La perdita convergente della vista e delle strutture visive nei mammiferi sotterranei ci permette di chiederci quali regioni genetiche – codificanti o meno – abbiano contribuito alla regressione in queste specie e quali siano state conservate.
Le cause genetiche di queste malformazioni sono state indagate attraverso studi sui pesci cavernicoli ciechi e l’analisi evolutiva dei geni della retina nei mammiferi sotterranei(Jeffery, 2009; Emerling e Springer, 2014). Il lavoro pionieristico di Hendriks et al. ha trovato il tasso evolutivo del cristallino e della proteina della retina αA-cristallina essere notevolmente accelerato nel topo cieco mediorientale (Spalaxehrenbergi), come ci si aspetterebbe sotto costrizione rilassata (Hendrikset al., 1987). Inoltre, Emerling e Springer (2014) hanno rivelato che i cambiamenti genetici regressivi delle proteine della retina sono distribuiti in modo disomogeneo su diversi percorsi visivi e tessuti oculari. Studi precedenti hanno posto maggiore enfasi sulle componenti retiniche della visione e sulle connessioni alla corteccia visiva perché sono queste componenti che percepiscono la luce e trasmettono immagini al cervello per la visione(Emerling e Springer, 2014; Cooper et al., 1993). Meno enfasi è stata posta sui geni che contribuiscono ad altri tessuti oculari, come la cornea.
I genomi di quattro mammiferi sotterranei sono stati sequenziati e studiati per i cambiamenti che si sono verificati in risposta al loro ambiente unico. Il genoma del topo nudo ha rivelato cambiamenti genetici nei geni chiave coinvolti nella termogenesi e nel ritmo circadiano, così come la perdita di geni e la disattivazione delle mutazioni nei geni della percezione visiva di base(Kim et al., 2011). Il genoma del topo talpa cieco (Nannospalax gailili) ha anche dato diverse intuizioni sui suoi adattamenti sotterranei, come un cambiamento di impatto alla proteina P53 che permette alle cellule di sfuggire all’apoptosi indotta dall’ipossia, così come l’upregulation di percorsi specifici coinvolti nella risposta all’ipossia e all’ipercapnia (Fang et al., 2014). Inoltre, l’evoluzione parallela è stata vista nella disattivazione dei geni della percezione visiva nel topo cieco e nel topo nudo. La convergenza di tali cambiamenti fornisce la prova che si sono verificati in risposta all’ambiente sotterraneo piuttosto che come risultato di condizioni specie-specifiche non correlate o processi neutri, evidenziando una potenziale strategia per scoprire ulteriori regioni genetiche che mostrano una risposta simile (Losos, 2011;Stern, 2013; Rosenblum et al ., 2014).
Studi precedenti hanno utilizzato l’evoluzione convergente per rivelare cambiamenti genetici che sono legati a cambiamenti ambientali senza aspettative a priori su quali regioni potrebbero rispondere. Una strategia è stata quella di cercare sostituzioni convergenti di aminoacidi in siti proteici specifici(Foote et al., 2015; Liu et al., 2010; Dobler et al., 2012). Una strategia complementare è quella di cercare cambiamenti convergenti nella pressione selettiva su regioni funzionali più grandi, come i geni o le sequenze regolatorie, perché l’evoluzione a diversi nucleotidi all’interno di un gene potrebbe comunque portare a effetti fenotipici convergenti. In pratica, i cambiamenti convergenti nella pressione selettiva sono dedotti dallo studio dei tassi evolutivi, perché il vincolo selettivo rallenta l’evoluzione, mentre la mancanza di vincolo e l’adattamento la velocizzano. I metodi computazionali che utilizzano questa strategia cercano elementi funzionali i cui tassi evolutivi sono cambiati nei rami che mostrano il cambiamento ambientale convergente(Marcovitz et al., 2016; Hiller et al., 2012; Chikina et al., 2016; Lartillot e Poujol, 2011). Una dimostrazione di questo approccio da parte del nostro gruppo ha identificato i geni che hanno risposto in modo convergente quando i lignaggi di mammiferi si sono spostati da un ambiente terrestre a uno marino(Chikina et al., 2016). Un altro recente studio di Prudent et al. (2016) ha dimostrato che le regioni che mostravano un’accelerazione di velocità convergente nell’ambiente sotterraneo erano arricchite di geni della percezione visiva e contenevano anche geni del ritmo circadiano. Insieme, questi studi mostrano la promessa di tassi convergenti per rivelare i geni alla base di importanti cambiamenti nella morfologia e nella fisiologia che sono correlati a drastici cambiamenti ambientali.
Per indagare le richieste poste alle specie sotterranee dal loro ambiente estremo, abbiamo cercato geni che mostrassero cambiamenti di ritmo convergenti in quattro mammiferi sotterranei. Riportiamo un ampio insieme di geni che mostrano un marcato rilassamento dei vincoli nelle specie sotterranee, che sono stati altamente arricchiti per le funzioni visive. Questo set conteneva anche molti geni di funzione indeterminata, che potevano essere geni causali non riconosciuti nelle malattie correlate agli occhi. Infine, abbiamo individuato gli stimolatori trascrizionali specifici per gli occhi nella regione del gene Pax6 utilizzando una nuova variante del nostro metodo e abbiamo dimostrato il potenziale di rilevare nuovi stimolatori oculari specifici per i geni chiave dello sviluppo.
Risultati
Molti geni hanno alterato i tassi evolutivi in particolare nei mammiferi sotterranei
Per prima cosa abbiamo cercato di identificare i geni che rispondevano alle condizioni dell’ambiente sotterraneo. Di conseguenza, abbiamo usato metodi di tasso relativo evolutivo (RER) per identificare i geni che codificano le proteine che si sono evoluti ad un tasso più rapido in particolare sui rami sotterranei dell’albero filogenetico dei mammiferi. I rami sotterranei erano costituiti da quelli che portano alla talpa dal naso stellato(Condylura cristata), la talpa aurea mantello(Chrysochloris asiatica), il topo talpa nudo(Heterocephalus glaber) e il topo talpa cieco (Nannospalaxgalili). Ognuna di queste specie rappresenta una stirpe che ha colonizzato in modo indipendente l’habitat sotterraneo, in quanto ciascuna di esse è più strettamente legata ai mammiferi in superficie che non agli altri(Figura 1A). Pertanto, simili cambiamenti fenotipici all’interno di queste specie sono considerati tratti convergenti. Per dimostrare i nostri metodi RER, presentiamo innanzitutto il caso del gene oculare specifico LIM2, che codifica la proteina di membrana intrinseca della lente 2. In primo luogo, la quantità di divergenza aminoacidica in LIM2 su ogni ramo di mammifero è stata quantificata utilizzando sequenze di 39 specie e modelli evolutivi standard(Figura 1B) (vedi Materiali e metodi). L’albero LIM2 risultante è marcatamente diverso dall’albero medio a livello genomico della Figura 1A, e rivela quantità decisamente elevate di divergenza nel LIM2 per le quattro specie sotterranee. Questa rapida divergenza è probabilmente dovuta alla perdita del vincolo selettivo nell’ambiente sotterraneo buio. Per quantificare questa accelerazione di velocità nell’albero LIM2, abbiamo normalizzato tutte le lunghezze dei rami per la quantità di variazione prevista, come definito dalla divergenza media a livello genomico per ogni ramo. Questa media, dopo la scalatura (vedi Materiali e metodi), dovrebbe riflettere sia i tempi di speciazione sottostanti nella filogenesi dei mammiferi, sia i cambiamenti dei fattori demografici che influenzano i tassi di sostituzione. I valori RER risultanti per ogni ramo sono riportati nella Figura 1C. Un RER pari a zero indica che il LIM2 si è evoluto esattamente al tasso previsto su quel ramo, mentre i valori positivi e negativi riflettono rispettivamente tassi più veloci e più lenti. Esaminando i RER diventa chiaro che il LIM2 è cambiato a tassi anormalmente rapidi nei quattro mammiferi sotterranei; i tassi per tutte e quattro le specie sotterranee sono più rapidi di tutte le specie in superficie, e questa differenza è supportata statisticamente (p=0,00084, test Mann-Whitney U). Così, estendendo i calcoli RER a tutti gli altri geni, possiamo distinguere le funzioni che hanno risposto durante l’adattamento all’ambiente sotterraneo. È importante notare che la convergenza di queste specie ci permette di dedurre con sicurezza i geni che hanno risposto in modo specifico alla vita sotterranea, perché i tassi più veloci in tutte e quattro le specie non sono probabilmente dovuti a fluttuazioni casuali, come riflette il basso valore P per il LIM2.
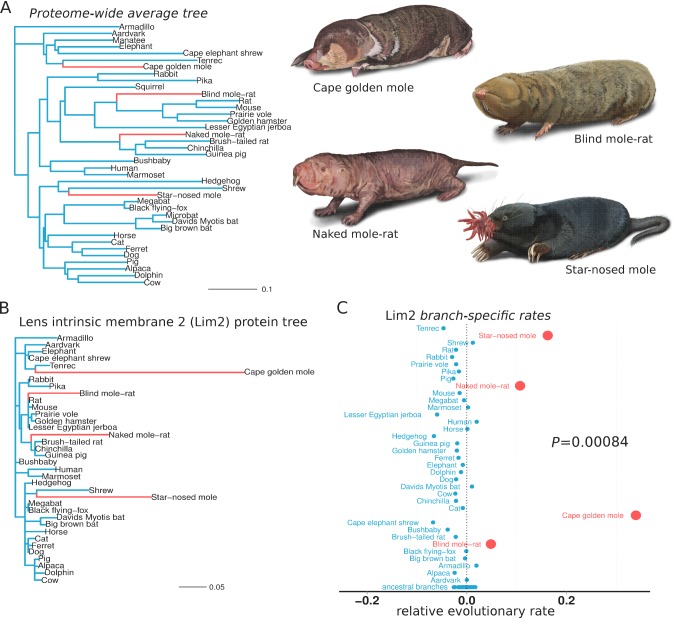
Figura 1.Figura 1. Tassi evolutivi della proteina di membrana intrinseca della lente 2 (LIM2) tra le specie.(A) Mammiferi transizioni in un ambiente sotterraneo si è verificato in quattro lignaggi mostrato in rosso. Le lunghezze dei rami sull’albero di mammifero riflettono il tasso evolutivo medio attraverso 18.980 geni che codificano le proteine.(B) LIM2 protein-coding sequenza mostra tassi accelerati di evoluzione sui rami sotterranei rispetto a quelli del proteoma a livello di albero medio.(C) Tassi di evoluzione relativa di LIM2 ha mostrato la più forte accelerazione sui rami sotterranei tra tutti i geni studiati. Illustrazioni di Michelle Leveille (Artifact Graphics).
Abbiamo eseguito la stessa analisi RER su 18.980 geni codificanti le proteine per determinare quali si sono spostati a tassi evolutivi più veloci o più lenti in particolare nelle specie sotterranee. Di seguito faremo riferimento a tali geni come ‘mole-accelerato’ e ‘mole-decelerato’, rispettivamente (vedi Materiali e metodi). Con un tasso di falsificazione (FDR) del 15%, abbiamo identificato 55 geni accelerati da talpa. Ci aspettiamo che i geni accelerati da talpa risultino sia dalla selezione per i cambiamenti di aminoacidi (cioè, selezione darwiniana positiva) o, in alternativa, da una riduzione della selezione purificante, come suggerito per la proteina LIM2. All’altro estremo, abbiamo identificato 1306 geni decelerati dalla muffa allo stesso FDR. Ci aspettiamo che i geni mostrino una decelerazione del tasso se c’è una più forte selezione purificante sulla funzione di quel gene nell’ambiente sotterraneo, forse come risultato di una maggiore importanza per il fitness.

Figura 1.Figura 1. Tassi evolutivi della proteina di membrana intrinseca della lente 2 (LIM2) tra le specie.(A) Le transizioni dei mammiferi in un ambiente sotterraneo si è verificato in quattro lignaggi mostrato in rosso. Le lunghezze dei rami sull’albero di mammifero riflettono il tasso evolutivo medio attraverso 18.980 geni che codificano le proteine.(B) LIM2 protein-coding sequenza mostra tassi accelerati di evoluzione sui rami sotterranei rispetto a quelli del proteoma a livello di albero medio.(C) Tassi di evoluzione relativa di LIM2 ha mostrato la più forte accelerazione sui rami sotterranei tra tutti i geni studiati. Illustrazioni di Michelle Leveille (Artifact Graphics).
Le funzioni legate alla visione sono altamente arricchite tra i geni accelerati dalla talpa
I geni con la più forte evidenza di accelerazione del neo sono stati costantemente associati con la funzione in due organi, occhio e pelle. Per illustrare, 17 dei primi 30 geni con accelerazione molare sono espressi esclusivamente nei tessuti oculari o sono associati a disturbi correlati agli occhi, mentre tre geni accelerati sono associati a pelle, capelli e unghie(Tabella 1). Tra i geni che mostrano segnali molto forti di accelerazione della talpa, troviamo proteine che sono specificamente espresse nei tessuti dell’occhio, come le proteine specifiche della retina ROM1 e GNAT1 (Figura 2). L’elenco completo dei 55 geni accelerati da talpa contiene allo stesso modo una grande proporzione che sono legati alla visione e tessuti esterni (file supplementare1), e sono stati altamente arricchiti per le annotazioni funzionali tra cui la morfologia dell’occhio, fotorecettori, trasduzione del segnale visivo, e fenotipi mutanti correlati all’occhio (Tabella 2, filesupplementare 2). La forza di questo arricchimento è chiaramente illustrata esaminando tutti i geni annotato al termine Gene Ontologia (GO) ‘percezione visiva’, perché una grande frazione dei geni che hanno questa annotazione sono classificati molto in alto nella lista dei geni accelerati da talpa (Figura 3A‘sotterranea ‘). Inoltre, se dovessimo utilizzare l’accelerazione delle talpe come unico predittore della funzione visiva, una ricerca identificherebbe correttamente molti geni noti della percezione visiva con alta precisione, anche quando si cerca l’intero genoma(Figura 3B). Questo forte arricchimento ci permette di formulare ipotesi specifiche in sezioni successive su quali tessuti e percorsi genetici sono stati alterati durante l’evoluzione regressiva dell’occhio.
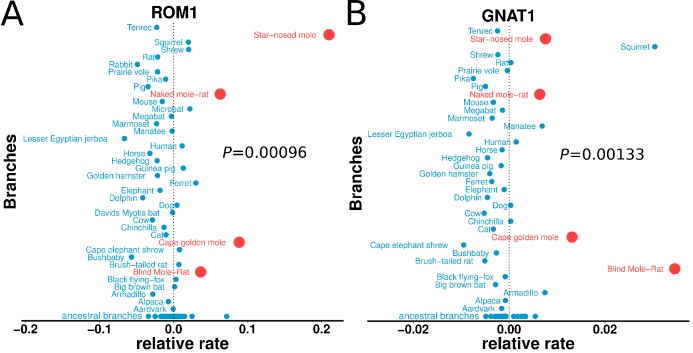
Figura 2.Figura 2. Tassi evolutivi relativi di due proteine della retina tra le specie.Tassi evolutivi relativi di due proteine della retina, (A) retinica segmento esterno membrana proteina 1 (ROM1) e (B) Rod proteina G cellulare specifico G, subunità alfa (GNAT1), mostrano una forte accelerazione nei mammiferi sotterranei (segnato in rosso).
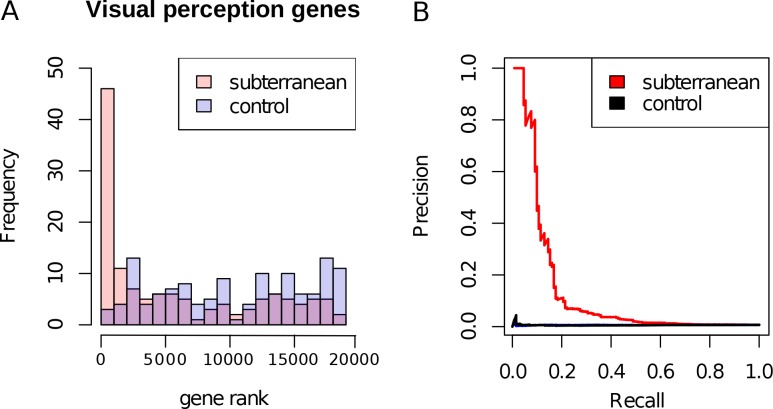
Figura 3.Arricchimento dei geni della percezione visiva.(A) Istogramma della classifica dei 189 geni di percezione visiva in base alla loro accelerazione molare. Vediamo un chiaro arricchimento dei geni con numeri di rango basso, che riflette il forte segnale di accelerazione della talpa nei geni di percezione visiva. Come controllo, usiamo quattro specie non sotterranee e, come previsto, i geni coinvolti nella visione non mostrano un’accelerazione convergente.(B) L’accelerazione della talpa può servire come un predittore per la funzione nella percezione visiva. Il grafico mostra i valori di Precisione-Richiamo a soglie di valore p variabili che riflettono la frazione dei geni della percezione visiva significativa ad una particolare soglia (Precisione) e la frazione dei geni della percezione visiva recuperati alla stessa soglia (Richiamo). Vediamo che l’accelerazione della talpa identifica specificamente i geni della percezione visiva con alta precisione rispetto all’accelerazione in due serie di quattro specie di controllo non sotterranee.
| Gene | Valore P | Tessuti | Descrizione |
|---|---|---|---|
| LIM2* | 0.00084 | Lente | Proteina di membrana intrinseca della lente 2 |
| CRYBB3* | 0.00087 | Lente | Cristallina specifica della lente, beta B3 |
| R0M1* | 0.00096 | Retina | Proteina di membrana del segmento esterno retinico 1 |
| CRYBA1* | 0.00098 | Lente | Cristallina specifica della lente, beta Al |
| CRYGC* | 0.00119 | Lente | Cristallina specifica della lente, gamma C |
| CRYBB2* | 0.00128 | Lente | Cristallina specifica della lente, beta B2 |
| GPR89B | 0.00130 | Ubiquitaria | Recettore 89B accoppiato alle proteine G, mediatore di pH in Golgi |
| GNAT1* | 0.00133 | Retina | Proteina G specifica delle cellule dell’asta, sottounità alfa |
| GPRS9A | 0.00134 | Ubiquitaria | Recettore 89A accoppiato alle proteine G, mediatore di pH in Golgi |
| NRL* | 0.00138 | Retina | Cerniera leucina della retina neurale responsabile dell’espressione della rodopsina |
| CRICCHI* | 0.00146 | Lente | Cristallina specifica della lente, gamma S |
| GRM6* | 0.00150 | Retina | Recettore 6 del glutammato metabotropico, necessario per la visione normale |
| GBX2 | 0.00165 | Embrione | Gastrulazione cervello homeobox 2, fattore di trascrizione dello sviluppo |
| LGSN* | 0.00171 | Lente | Lengsin, proteina della lente con dominio della glutammina sintetasi |
| CRYBB1* | 0.00183 | Lente | Cristallina specifica della lente, beta Bl |
| KLHDC3 | 0.00186 | Ubiquitaria | Kelch-domain-contenuto 3, alta espressione nel cervello |
| KRT81# | 0.00186 | Capelli e unghie | Cheratina 81, principalmente nella corteccia dei capelli |
| WDFY1 | 0.00192 | Ubiquitaria | Ripetizione WD e FYVE-dominio contenente 1, proteina endosomica |
| KRT9# | 0.00195 | Skin | Cheratina 9, specifica per i palmi delle mani e le piante dei piedi |
| POMP# | 0.00199 | Ubiquitaria | Proteasoma proteico di maturazione, associato a un raro disturbo della pelle |
| RRH* | 0.00201 | Retina | Pigmento retinico epitelio-pigmento derivato dalla rodopsina omologato |
| DPCD* | 0.00201 | Celle ciliate | Soppressa nella discinesia ciliare primaria; mantenimento delle cellule ciliate |
| RAD54L | 0.00217 | Ubiquitaria | Tipo RAD54: Riparazione della rottura del DNA a doppio filamento |
| TATDN1 | 0.00235 | Ubiquitaria | TatD DNase-dominio contenente 1 |
| ITLN2 | 0.00244 | Intestino tenue | Intelectin 2, può avere un ruolo nella difesa contro gli agenti patogeni |
| STX3* | 0.00245 | Ubiquitaria | Sintassi 3, associata a cataratta congenita e disabilità intellettiva |
| SKJV2L* | 0.00254 | Ubiquitaria | DEAD box protein, lievito SKI2 omologato, implicato nella degenerazione maculare |
| DPY19L1 | 0.00254 | Ubiquitaria | dpy-19-come 1(Caenorhabditis elegans), probabile C-mannosiltransferasi |
| TFPT | 0.00266 | Ubiquitaria | TCF3 (E2A) partner di fusione (nella leucemia infantile) |
| RSI* | 0.00275 | Retina | Retinoschisina 1, proteina extracellulare coinvolta nell’organizzazione della retina |
| Annotazione funzionale | Arricchimento della piega | p-valore | Valore FDR q |
|---|---|---|---|
| Percezione visiva | 23.16 | 6.84E-16 | 1.02E-11 |
| Percezione sensoriale dello stimolo luminoso | 22.69 | 9,12E-L6 | 6.82E-12 |
| Percezione sensoriale | 8.47 | 5.83E-10 | 2.91E-06 |
| Processo del sistema neurologico | 5.39 | 1.75E-07 | 6.53E-O4 |
| Rilevamento dello stimolo luminoso | 29.57 | 7.04E-07 | 2.10E-03 |
| Rilevamento dello stimolo luminoso coinvolto nella percezione sensoriale | 56.35 | 1.92E-05 | 4.77E 02 |
| Rilevamento dello stimolo luminoso coinvolto nella percezione visiva | 56.35 | 1.92E-05 | 4.09E-02 |
| Rilevamento di stimoli esterni | 14.38 | 2 49E-05 | 4.66E-02 |
Abbiamo effettuato un’analisi di controllo per dimostrare che questi arricchimenti funzionali sono unici per le specie sotterranee. Abbiamo scelto quattro specie fuori terra (specie di controllo) per le quali non c’è motivo di aspettarsi una convergenza fenotipica e le cui lunghezze dei rami sono simili alle talpe – pika, cavia, scoiattolo e mucca. Mentre i geni accelerati dalle talpe sono stati arricchiti in 15 categorie GO a un FDR del 15%, i geni accelerati dal controllo non avevano categorie arricchite nello stesso FDR (File supplementare2). Inoltre, queste specie di controllo non hanno mostrato alcun arricchimento dei geni della percezione visiva in particolare(Figura 3).
C’erano anche 1306 geni decelerati dalla muffa che si sono evoluti a tassi significativamente più lenti nelle specie sotterranee rispetto ad altri mammiferi (file supplementare 3). Anche se i geni decelerati dalle talpe sono individualmente significativi, solo una categoria GO ha mostrato un significativo arricchimento funzionale – GO Biological Process: Attività del fattore di trascrizione dell’acido nucleico che lega l’acido nucleico – con un FDR del 15% (File supplementare4). Un’analisi di controllo simile ha mostrato 626 geni come significativamente decelerato a un FDR del 15%, e questi geni decelerati dal controllo sono stati arricchiti in 24 categorie GO. Pertanto, nonostante ci siano molti più geni decelerati dalla talpa che non i geni accelerati dalla talpa, i geni decelerati dalla talpa come gruppo non mostrano un forte arricchimento funzionale. Questo risultato è in netto contrasto con il forte arricchimento visto nei geni accelerati dalla talpa.

Figura 2.Figura 2. Tassi evolutivi relativi di due proteine della retina tra le specie.Tassi evolutivi relativi di due proteine della retina, (A) retinica segmento esterno membrana proteina 1 (ROM1) e (B) Rod proteina G cellulare specifico G, subunità alfa (GNAT1), mostrano una forte accelerazione nei mammiferi sotterranei (segnato in rosso).

Figura 3.Arricchimento dei geni della percezione visiva.(A) Istogramma della classifica dei 189 geni di percezione visiva in base alla loro accelerazione molare. Vediamo un chiaro arricchimento dei geni con numeri di rango basso, che riflette il forte segnale di accelerazione della talpa nei geni di percezione visiva. Come controllo, usiamo quattro specie non sotterranee e, come previsto, i geni coinvolti nella visione non mostrano un’accelerazione convergente.(B) L’accelerazione della talpa può servire come un predittore per la funzione nella percezione visiva. Il grafico mostra i valori di Precisione-Richiamo a soglie di valore p variabili che riflettono la frazione dei geni della percezione visiva significativa ad una particolare soglia (Precisione) e la frazione dei geni della percezione visiva recuperati alla stessa soglia (Richiamo). Vediamo che l’accelerazione della talpa identifica specificamente i geni della percezione visiva con alta precisione rispetto all’accelerazione in due serie di quattro specie di controllo non sotterranee.
La maggior parte dei geni ad accelerazione di talpa sono sotto una costrizione rilassata
Tassi accelerati avrebbero potuto derivare da un’evoluzione adattiva o, in alternativa, da un allentamento dei vincoli. Abbiamo distinto questi scenari utilizzando modelli evolutivi basati su codici per rilevare le firme dell’evoluzione adattiva. Abbiamo testato se il rapporto di tasso da non sinonimico a sinonimo(dN/dS) era significativamente maggiore di 1 – l’aspettativa di selezione positiva – per qualsiasi porzione del gene specificamente sui rami delle specie sotterranee, e anche più in generale sull’intera filogenesi dei mammiferi (Yang,2007). Dei 55 geni top 55 accelerati da talpa, solo un gene ha rifiutato un modello neutro che non permetteva rapporti dN/dSsuperiori a 1 in favore di un modello che permetteva una selezione positiva(dN/dS> 1) sui rami sotterranei (file supplementare5). Questo gene è coinvolto nel tessuto connettivo e nella struttura dei capelli (KRTAP17-1).
Gli altri geni accelerati non hanno mostrato prove di evoluzione adattiva e quindi sono probabilmente sotto una limitazione rilassata. Quasi tutti i geni accelerati hanno rifiutato un modello che richiedeva loro di avere vincoli identici in tutti i mammiferi (modello M1) a favore di un modello che consentisse un rilassamento del vincolo specifico sotterraneo (modello BS1)(File supplementare 5). Alcuni di questi geni sembrano aver perso tutti i vincoli funzionali perché mostrano lesioni genetiche come i codoni di arresto e i frame shift in alcune specie sotterranee(file supplementare 6). Questa prova di rilassamento del vincolo è coerente con l’aspettativa che alcuni geni legati alla visione abbiano subito un’evoluzione regressiva.
I geni legati alla pelle sono stati accelerati forse in risposta alle richieste di tunneling
Lo stile di vita fossile delle specie sotterranee è stato selezionato per tratti legati allo scavo e alla locomozione nel sottosuolo(Nevo, 1979). Forse a causa di questa pressione selettiva, molti dei geni superiori accelerati dalla talpa codificano proteine che sono componenti strutturali della pelle, dei capelli e dei tessuti connettivi epiteliali. Le ragioni della loro accelerazione sono il risultato del rilassamento della costrizione della loro sequenza di codifica. I geni che codificano le proteine della cheratina 9, 12 e 81 (KRT9, KRT12, KRT81) sono stati studiati utilizzando modelli di codoni, e i risultati hanno indicato che hanno sperimentato una costrizione rilassata nelle specie sotterranee, ma non una selezione positiva per la diversificazione degli aminoacidi(file supplementare 5). Essi contengono codoni di arresto precoce in più specie sotterranee, il che è coerente con la completa perdita di vincolo(File supplementare 6).
L’accelerazione convergente e la pseudogenializzazione di KRT9 è particolarmente interessante in relazione alla scavatura(Figura 4). Nei topi, l’espressione di KRT9 è limitata alle pedane, e i Krt9-/-mutanti nulli sviluppano calli delle pedane a causa dell’iperproliferazione della pelle (Fu et al., 2014). Nell’uomo, KRT9 si esprime esclusivamente sui palmi delle mani e sulle piante dei piedi, e le mutazioni portano a un disturbo della pelle caratterizzato da ipercheratosi (ispessimento) delle superfici dei palmi e delle piante dei piedi – cheratoderma epidermolitico palmoplantare epidermolitico (Hennieset al., 1995). Per estensione, la perdita di KRT9 in specie sotterranee può anche aver portato a iperproliferazione di pedane, che potrebbe portare benefici per il tunneling. Ad esempio, la talpa dal naso a stella scava con le zampe anteriori e i topi talpa nudi raccolgono e rimuovono lo sporco con i piedi(Jarvis and Sale, 2010; Hamilton, 1931). Tali attività abrasive potrebbero porre elevate esigenze alle superfici delle pedane. Inoltre, anche l’accelerazione delle talpe del gene POMP potrebbe essere stata causata dalle esigenze delle pedane. Una mutazione umana nella POMP è associata alla sindrome di KLICK, una malattia della pelle caratterizzata anche da iperproliferazione e ispessimento di palme e pedane (Dahlqvistet al., 2010).
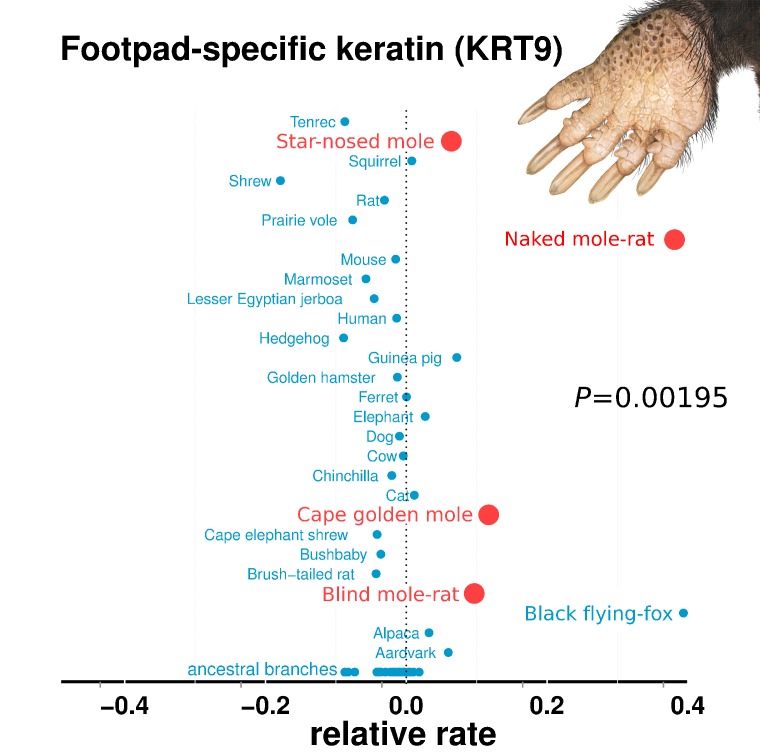
Figura 4.Figura 4. Tassi relativi di cheratina 9 (KRT9).KRT9 mostra una forte accelerazione sui rami sotterranei. L’immagine mostrata è la pedana della talpa dal naso a stella, che mostra l’ipercheratosi caratteristica. Mutazioni della cheratina 9 portano anche a ipercheratosi nei modelli di topo e nell’uomo. Illustrazioni di Michelle Leveille (Artifact Graphics).
Inoltre, abbiamo scoperto geni legati alla pelle e ai capelli che mostrano prove di selezione positiva piuttosto che di perdita di funzione nelle specie sotterranee. Anche se questi geni non sono stati significativamente accelerati da talpa ad un FDR del 15%, essi riflettono potenzialmente i cambiamenti funzionali in risposta all’adattamento sotterraneo. Uno di questi geni, COL4A4, un gene che codifica una sottounità di collagene di tipo IV, è stato fortemente accelerato, non conteneva lesioni genetiche, e ha mostrato prove di selezione positiva nelle specie sotterranee(file supplementare 1,5,6). Il collagene di tipo IV è il principale componente strutturale della lamina basale in molti tessuti, compreso l’epitelio cutaneo, ed è composto da sei sottounità, tre delle quali (COL4A4, COL4A5 e COL4A3) sono state notevolmente accelerate da talpa. In media, le sei sottounità sono state più accelerate del 71% di tutti gli altri geni, il che rappresenta una differenza significativa (p=0,0342, test di Mann-Whitney U). Mentre il collagene di tipo IV sembra aver risposto all’ambiente sotterraneo, altri componenti principali della lamina basale, le proteine laminiche (ad esempio, LAMA1), non sono state notevolmente accelerate.

Figura 4.Tassi relativi di cheratina 9 (KRT9).KRT9 mostra una forte accelerazione sui rami sotterranei. L’immagine mostrata è la pedana della talpa dal naso a stella, mostrando ipercheratosi caratteristica. Mutazioni della cheratina 9 portano anche a ipercheratosi nei modelli di topo e nell’uomo. Illustrazioni di Michelle Leveille (Artifact Graphics).
L’evoluzione regressiva è limitata al cristallino, alla retina e ai geni dello sviluppo specifico dell’occhio
Al fine di confrontare l’evoluzione di specifici tessuti oculari in specie sotterranee, abbiamo compilato per la prima volta set di geni specifici per i tessuti utilizzando i dati di espressione di 91 tessuti di topo(Su et al., 2004). Abbiamo identificato i geni tessuti-specifici per cornea, iride, lenti e retina selezionando quei geni con un’espressione differenziale significativa nel tessuto di interesse ma non in altri tessuti. Utilizzando la letteratura, abbiamo anche compilato una serie di 71 importanti geni dello sviluppo dell’occhio(file supplementare 7). Per prima cosa abbiamo chiesto se esiste una relazione tra il grado di specificità del tessuto e il grado di accelerazione della talpa misurata come differenza in dN/dStra le specie sotterranee e quelle in superficie(Figura 5A). Abbiamo trovato una chiara correlazione positiva tra la specificità del tessuto oculare e l’accelerazione della talpa, che è coerente con un maggiore rilassamento del vincolo sui geni con pochi o nessun ruolo al di fuori dell’occhio. Successivamente, abbiamo chiesto quali geni con espressione specifica del tessuto oculare hanno mostrato accelerazione e abbiamo trovato che i geni che sono espressi specificamente nella cornea (un tessuto protettivo dell’occhio esterno) e l’iride non sono stati accelerati nelle specie sotterranee se confrontati con un insieme di geni scelti a caso (sfondo) (Figura 5B e C). Al contrario, molti geni specifici della lente e della retina sono accelerati. In media, i geni delle lenti sono più accelerati dell’84% dei geni di fondo e i geni della retina sono più accelerati dell’82% (p=9,07×10-6 e p=6, 10×10-10 per lenti e retina, rispettivamente, test Mann-Whitney U). Il contrasto tra il fronte e l’interno dell’occhio suggerisce che le funzioni sensoriali dell’occhio interno, come la fototrasduzione e il ciclo visivo, sono sotto un vincolo di rilassamento, mentre la funzione protettiva della cornea non lo è. Infatti, due di queste specie sotterranee hanno occhi aperti sull’ambiente, cosicché la cornea può continuare a fungere da barriera agli agenti patogeni e ai detriti.
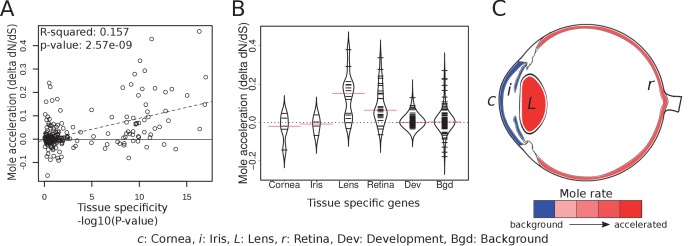
Figura 5.I geni della retina e del cristallino specifici dei tessuti sono altamente accelerati nelle specie sotterranee.(A) I geni oculari che sono più specifici per i tessuti mostrano una maggiore accelerazione nelle specie sotterranee “talpe”. L’asse y rappresenta il cambiamento nel tasso di evoluzione sui rami che si spostano in un ambiente sotterraneo.(B) Pannelli di tessuti-geni specifici sono stati testati per le loro accelerazioni relative nei mammiferi sotterranei. Cento geni “di fondo” scelti a caso non erano in media più veloci o più lenti, e forniscono una stima della varianza prevista per i geni casuali. I geni retina e lente-specifici mostrano molti casi di accelerazione nell’ambiente sotterraneo, e le loro distribuzioni sono significativamente elevate rispetto allo sfondo (p = 1,4 × 10-5 e 3,2 × 10-4, rispettivamente).(C) Rappresentazione dell’accelerazione media del neo per i geni specificamente espressi in quattro diversi tessuti dell’occhio.
I geni dello sviluppo dell’occhio nel suo complesso non sono stati accelerati rispetto allo sfondo, che può riflettere il fatto che la maggior parte di loro, comeSonic Hedgehog(Shh), sono importanti nello sviluppo di tessuti non oculari. Tuttavia, cinque geni oculari specifici dello sviluppo erano presenti in particolare in cima alla lista accelerata(Vax2, Nrl, Foxe3, Crx eAldh3A1), mentre nessun gene specifico dell’occhio è stato trovato più in basso nella lista (file supplementare7). Ciò è coerente con la relazione positiva tra la specificità oculare e il rilassamento del vincolo(Figura 5A).

Figura 5.I geni della retina e delle lenti specifiche dei tessuti sono altamente accelerati nelle specie sotterranee.(A) I geni oculari che sono più specifici per i tessuti mostrano una maggiore accelerazione nelle specie sotterranee “talpe”. L’asse y rappresenta il cambiamento nel tasso di evoluzione sui rami che si spostano in un ambiente sotterraneo.(B) Pannelli di tessuti-geni specifici sono stati testati per le loro accelerazioni relative nei mammiferi sotterranei. Cento geni “di fondo” scelti a caso non erano in media più veloci o più lenti, e forniscono una stima della varianza prevista per i geni casuali. I geni retina e lente-specifici mostrano molti casi di accelerazione nell’ambiente sotterraneo, e le loro distribuzioni sono significativamente elevate rispetto allo sfondo (p = 1,4 × 10-5 e 3,2 × 10-4, rispettivamente).(C) Rappresentazione dell’accelerazione media del neo per i geni specificamente espressi in quattro diversi tessuti dell’occhio.
Gli stimolatori specifici dell’occhio di PAX6 mostrano un’accelerazione convergente nei mammiferi sotterranei
Anche se osserviamo casi specifici in cui i geni dello sviluppo dell’occhio mostrano tassi accelerati nei mammiferi sotterranei, non c’è una tendenza globale significativa. Ciò è comprensibile, dato che la maggior parte di questi fattori di trascrizione dello sviluppo hanno un ruolo importante nello sviluppo di tessuti non correlati all’occhio. Ad esempio, Pax6 è importante nello sviluppo del pancreas e del cervello oltre che dell’occhio(Kleinjan et al., 2006; Kammandel et al., 1999; Xu et al., 1999). Da qui le sequenze di codifica delle proteine dei fattori di trascrizione codificati da questi geni subiscono una pressione selettiva contro le mutazioni deleterie. Tuttavia, le regioni di regolazione che controllano l’espressione di questi geni dello sviluppo nell’occhio potrebbero essere sotto una limitazione rilassata nei mammiferi sotterranei, dato il rilassamento della necessità di mantenere la funzionalità dei percorsi visivi. Ipotizziamo che questi elementi cis-regolatori specifici dell’occhio (CREs) mostrerebbero quindi tassi di evoluzione accelerati nei mammiferi sotterranei.
Abbiamo testato questa ipotesi applicando il nostro metodo evolutivo per identificare gli elementi regolatori specifici dell’occhio che controllano l’espressione del fattore di trascrizione dello sviluppo PAX6. Abbiamo scelto il sistema PAX6 perché ci siamo impegnati a fondo per caratterizzare la regolazione spazio-temporale della sua espressione(Kleinjan et al., 2006; Kammandel et al., 1999; Xu et al., 1999; Kleinjan et al., 2001; Dimanlig et al., 2001; Griffin et al., 2002), ed esiste un’annotazione completa diCRE che controlla l’espressione di PAX6 in vari tessuti, compreso l’occhio. Sulla base della letteratura esistente sulla regolazione trascrizionale dell’espressione di Pax6, abbiamo identificato una finestra di 500 kb contenente Pax6 e il suo gene vicino Elp4 come la nostra finestra genomica di interesse (Kleinjanet al., 2006). Esperimenti che coinvolgono topi transgenici hanno rivelato che vari stimolatori specifici per i tessuti in una regione di 200 kb all’interno di questa finestra genomica sono importanti per l’espressione di Pax6. Abbiamo successivamente identificato 150 elementi non codificanti altamente conservati in questa finestra genomica e abbiamo stimato i loro tassi evolutivi su ogni ramo di mammifero. Abbiamo poi calcolato i tassi relativi dei rami utilizzando lo stesso metodo dell’operatore di proiezione utilizzato per gli alberi del gene della codifica delle proteine. Abbiamo poi utilizzato il quadro di prova delle ipotesi di Mann-Whitney U per identificare gli elementi non codificanti che si sono evoluti ad un tasso accelerato in particolare sui rami sotterranei (Materiali e metodi).
I risultati delle nostre analisi mostrano che le tre regioni che mostrano i segnali più forti di accelerazione convergente nei mammiferi sotterranei si sovrappongono ampiamente alle regioni precedentemente annotate per essere potenziatori importanti per la regolazione nei tessuti specifici degli occhi(Figura 6A, B).(i) ‘cre149’ è una regione di 558-basepair (bp) contenente la regione di 530-bp annotata come enhancer ‘alfa, introne 4 retinico’ (Kammandel et al., 1999). ii) “cre21” è una regione di 552 pb situata all’interno del frammento contenente HS2 e HS3 della regione regolatoria distale, un potenziatore specifico della retinica di PAX6 (Kleinjanet al., 2001). iii) “cre86” è una regione di 429 bp contenente il “potenziatore ectodermico” lungo 341 bp, che si è dimostrato importante per guidare l’espressione del PAX6 nella lente di sviluppo (Dimanlig et al., 2001). Le regioni che si sovrappongono ad un elemento potenziatore che ha dimostrato di regolare l’espressione del PAX6 nella lente, nel cervelletto e nel diencefalo (l’elemento potenziatore ‘EI’) non mostrano un’accelerazione significativa della velocità nei nei nei (Kleinjanet al., 2001). Questo è in accordo con la nostra aspettativa che solo gli elementi specifici dell’occhio mostrano un’accelerazione convergente, e quindi le regioni che si sovrappongono all’esaltatore EI non mostrano accelerazione data la loro importanza per l’espressione del PAX6 nei tessuti non oculari. Allo stesso modo, una regione di 120 bp che si sovrappone all’intensificatore del pancreas non mostra un’accelerazione significativa nei nei nei, come previsto(Kleinjan et al., 2006; Xu et al., 1999). Oltre agli elementi specifici del potenziatore oculare, osserviamo altre regioni che mostrano un’accelerazione di velocità comparabile nei nei nei non ancora caratterizzati(file supplementare 8). Queste regioni sono candidate CRE per l’espressione PAX6 nell’occhio. Questo studio preliminare del modulo di regolazione della trascrizione di Pax6 serve a confermare la nostra ipotesi che gli elementi di regolazione specifici dell’occhio sono sotto un rilassato vincolo e quindi mostrano tassi di evoluzione accelerati nei mammiferi sotterranei.
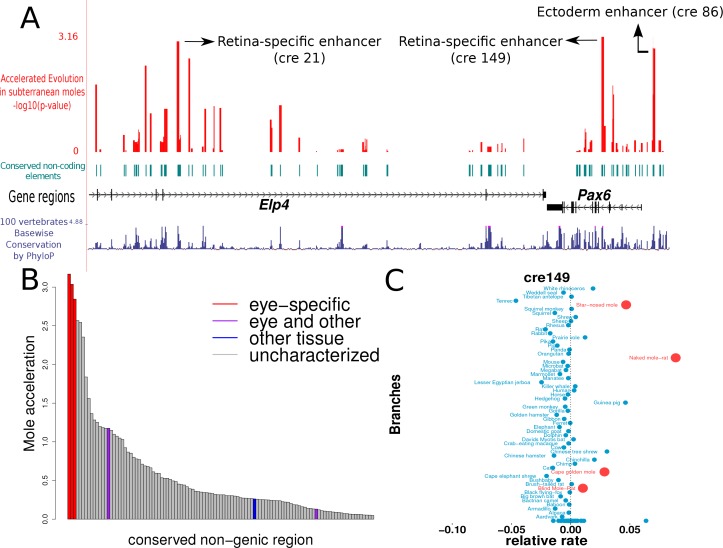
Figura 6.Figura 6. Accelerazione molare degli stimolatori oculari specifici nella regione del gene Pax6.(A) Regione genomica che abbraccia Pax6 e il suo vicino Elp4. Gli esoni e gli introni dei due geni sono rappresentati rispettivamente da blocchi e linee nere, mentre le regioni non codificanti conservate analizzate sono rappresentate in azzurro. Il segnale di conservazione come dato dai 100 vertebrati Anche la conservazione è mostrata in blu scuro. I punteggi di accelerazione delle talpe per queste regioni sono rappresentati in rosso. Le tre regioni più accelerate non codificanti identificate in questa analisi sono coerenti con gli stimolatori oculari specifici che regolano l’espressione di Pax6 nell’occhio.(B) I punteggi di accelerazione molare per i tre stimolatori oculari specifici di Pax6 sono i più alti tra le 150 regioni analizzate, compresi gli stimolatori di altri tessuti e le regioni non codificanti non caratterizzate.(C) I tassi relativi in ogni specie per la regione più accelerata “cre149”.

Figura 6.Figura 6. Mole-accelerazione degli stimolatori specifici per gli occhi nella regione del gene Pax6.(A) Regione genomica che abbraccia Pax6 e il suo vicino Elp4. Gli esoni e gli introni dei due geni sono rappresentati rispettivamente da blocchi e linee nere, mentre le regioni non codificanti conservate analizzate sono rappresentate in azzurro. Il segnale di conservazione come dato dai 100 vertebrati Anche la conservazione è mostrata in blu scuro. I punteggi di accelerazione delle talpe per queste regioni sono rappresentati in rosso. Le tre regioni più accelerate non codificanti identificate in questa analisi sono coerenti con gli stimolatori oculari specifici che regolano l’espressione di Pax6 nell’occhio.(B) I punteggi di accelerazione molare per i tre stimolatori oculari specifici di Pax6 sono i più alti tra le 150 regioni analizzate, compresi gli stimolatori di altri tessuti e le regioni non codificanti non caratterizzate.(C) I tassi relativi in ogni specie per la regione più accelerata “cre149”.
Mole-accelerato elementi non codificanti sono fortemente arricchiti vicino ai geni del fattore di trascrizione che guidano lo sviluppo dell’occhio
Espandendoci dalla nostra analisi di Pax6, abbiamo eseguito una scansione su larga scala di elementi non codificanti accelerati in modo convergente in prossimità dei geni del fattore di trascrizione nel genoma dei mammiferi. Abbiamo compilato due set di geni del fattore di trascrizione – uno composto da 20 geni noti per essere importanti nello sviluppo dell’occhio (il set Eye), come Pax6, Pax2, Otx2, e un altro set composto da un numero uguale di fattori di trascrizione specifici del tessutogeni che sono espressi in altri tessuti e senza alcuna prova di espressione nell’occhio (l’Altro set), che comprende Hoxa9, Pax8 e Sox13 (file supplementare 9). Abbiamo identificato 200 elementi non codificanti conservati vicino a ciascun gene in entrambi i set, per un totale di 8000 elementi divisi equamente tra i due set di geni (vedi Materiali e metodi). Successivamente abbiamo applicato il nostro metodo e calcolato l’accelerazione molare di ogni elemento. Questa scansione su larga scala ha rivelato un totale di 17 elementi come convergentemente accelerato ad un FDR del 10%(Figura 7A, file supplementare 10). Quattordici dei 17 elementi si trovano vicino a geni appartenenti all’insieme dell’occhio, che riflette un arricchimento significativo di elementi accelerati dalla talpa vicino ai geni del fattore di trascrizione che guidano lo sviluppo dell’occhio (test ipergeometrico, p-valore=0,001). Abbiamo successivamente controllato le posizioni genomiche di questi elementi accelerati dalla talpa per garantire che non siano raggruppati nello stesso locus, ad esempio. Questi 17 elementi si trovano vicino a 14 geni unici, con 11 geni unici appartenenti all’insieme dell’occhio, e tre geni appartenenti all’altro insieme, dimostrando ulteriormente il forte arricchimento dei geni del fattore di trascrizione dello sviluppo oculare unico vicino agli elementi accelerati dalla talpa (test ipergeometrico p-valore p = 0,0016).
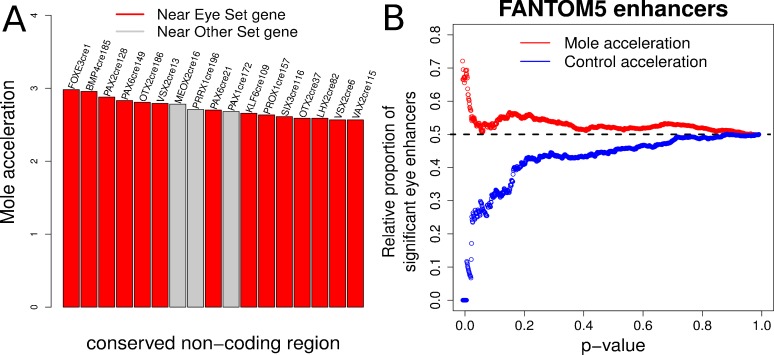
Figura 7.Prove di accelerazione della talpa in candidati potenziatori oculari specifici.(A) Arricchimento di elementi accelerati da talpa vicino a geni di fattore di trascrizione dello sviluppo dell’occhio. La trama bar mostra il 17 mole-accelerato conservato non codifica 17 elementi non codificanti identificati. Quattordici dei 17 elementi sono presenti vicino ai geni del fattore di trascrizione nell’insieme dell’occhio, indicati in rosso.(B) FANTOM5 Eye enhancer mostrano una forte accelerazione della talpa. La trama mostra la proporzione relativa degli stimolatori oculari FANTOM5 identificati tra tutti gli stimolatori significativi alla corrispondente soglia di p-valore. Vediamo un forte arricchimento degli stimolatori oftalmici identificati a bassa accelerazione del neo (punti rossi), mentre non si osserva alcun arricchimento di questo tipo utilizzando i valori p di controllo dell’accelerazione della specie (punti blu).

Figura 7.Prove di accelerazione della talpa in candidati potenziatori oculari specifici.(A) Arricchimento di elementi accelerati da talpa vicino a geni fattore di trascrizione dello sviluppo dell’occhio. La trama bar mostra il 17 mole-accelerato conservato non codifica 17 elementi non codificanti identificati. Quattordici dei 17 elementi sono presenti vicino ai geni del fattore di trascrizione nell’insieme dell’occhio, indicati in rosso.(B) FANTOM5 Eye enhancer mostrano una forte accelerazione della talpa. La trama mostra la proporzione relativa degli stimolatori oculari FANTOM5 identificati tra tutti gli stimolatori significativi alla corrispondente soglia di p-valore. Vediamo un forte arricchimento degli stimolatori oftalmici identificati a bassa accelerazione del neo (punti rossi), mentre non si osserva alcun arricchimento di questo tipo utilizzando i valori p di controllo dell’accelerazione della specie (punti blu).
Gli stimolatori oftalmici FANTOM5 mostrano una forte accelerazione convergente nei mammiferi sotterranei
Il consorzio FANTOM5 ha identificato siti di putativi potenziatori nel genoma umano e nel genoma del topo sulla base della trascrizione bidirezionale dei potenziatori attraverso i tessuti e in diversi punti temporali di sviluppo (Anderssonet al., 2014). Questi siti di putativo potenziatore includono regioni genomiche che vengono trascritte nel bulbo oculare degli embrioni di topo in quattro punti temporali di sviluppo. Sulla base di questa risorsa, abbiamo compilato due set di siti di valorizzazione FANTOM5 (vedi Materiali e metodi) – un set composto da 900 regioni genomiche con espressione non zero nel bulbo oculare attraverso quattro punti temporali di sviluppo (“Eye” enhancer), e un altro set composto da 6000 regioni con espressione zero attraverso gli stessi campioni (“Other” enhancer). Successivamente abbiamo calcolato l’accelerazione convergente di questi elementi genomici nei quattro mammiferi sotterranei e abbiamo confrontato l’accelerazione osservata per gli stimolatori “Occhio” con quella degli stimolatori “Altro”. La nostra analisi ha rivelato un forte arricchimento degli stimolatori FANTOM5 ‘Eye’ enhancer che mostrano un’accelerazione convergente nelle quattro specie sotterranee rispetto alle quattro specie di controllo (Figura 7B). Osserviamo 62 potenziatori FANTOM5 in totale che hanno mostrato un’accelerazione molare significativa ad un FDR del 15% (file supplementare11). Quindici di questi corrispondono all’insieme degli stimolatori oculare FANTOM5, che riflettono un arricchimento significativo degli stimolatori oculare FANTOM5 rilevati utilizzando l’accelerazione della talpa (test ipergeometrico p-valore p=0,006).
Alcune specie in superficie mostrano un’accelerazione genica indicativa delle loro capacità visive alterate
Per comprendere le differenze nelle capacità visive dei mammiferi in modo sistematico, abbiamo studiato i tassi relativi complessivi di evoluzione dei geni visivi di tutti i mammiferi. Il nostro set di geni di interesse (189 geni in totale) era composto da tutti i geni con l’annotazione del termine GO “percezione visiva”, esclusi i fattori di trascrizione dello sviluppo. Per ogni specie, abbiamo poi calcolato il tasso relativo medio di tutti i geni (Figura 8). Abbiamo osservato i quattro mammiferi sotterranei per essere tra le specie accelerate (con media >0), come era la nostra aspettativa. Tuttavia, abbiamo anche osservato specie fuori terra con accelerazioni di tasso globale paragonabili a quelle delle talpe, come l’armadillo, lo scoiattolo di terra a tredici linee, il grande pipistrello marrone, il pipistrello miytis di David e un toporagno. In particolare, tutti questi mammiferi mostrano vari tipi di regressione visiva: l’armadillo ha una scarsa vista caratterizzata dalla mancanza di cellule coniche nella retina (McDonoughe Loughry, 2013), e i toporagni hanno anche una scarsa vista e occhi piccoli, che in alcune specie sono nascosti nella pelliccia (Nowak, 1999). Il grande pipistrello bruno notturno e il pipistrello miytis di David hanno occhi ridotti e si affidano all’ecolocazione per la navigazione (Koayet al., 1998). Lo scoiattolo di terra a tredici linee mostra un raro tratto visivo: la regione centrale della sua retina è dominata da fotorecettori a cono in contrasto con la retina della maggior parte dei mammiferi (Kimet al., 2016). Questi scenari potrebbero avere importanti implicazioni perché lo scoiattolo di terra è usato come modello per la ricerca sulla visione(Li et al., 2010; Chen e Li, 2012).
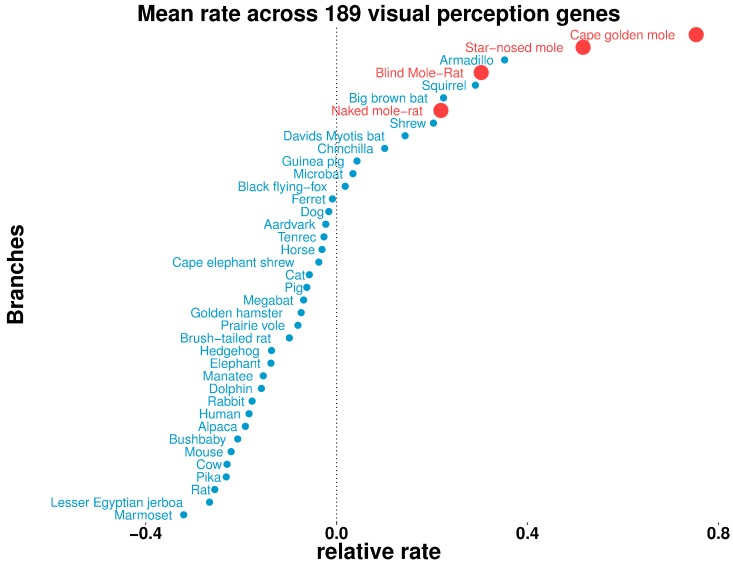
Figura 8.Alcune specie in superficie mostrano tassi accelerati di cambiamento evolutivo dei geni della percezione visiva.Sulla base dei tassi evolutivi relativi di tutte le specie per 189 geni con l’annotazione GO “percezione visiva”, abbiamo calcolato il tasso relativo relativo medio dei geni in base alla specie. Le nostre precedenti osservazioni sull’accelerazione delle talpe nei geni della percezione visiva sono qui ricapitolate – i quattro mammiferi sotterranei sono tra le specie che mostrano un tasso accelerato su questi geni. È interessante notare che troviamo altre specie non sotterranee che mostrano un’accelerazione paragonabile a quella dei mammiferi sotterranei, indicando adattamenti nei sistemi visivi.

Figura 8.Alcune specie fuori terra mostrano tassi accelerati di cambiamento evolutivo nei geni della percezione visiva.Sulla base dei tassi evolutivi relativi di tutte le specie per 189 geni con l’annotazione GO “percezione visiva”, abbiamo calcolato il tasso relativo relativo medio dei geni in base alla specie. Le nostre precedenti osservazioni sull’accelerazione delle talpe nei geni della percezione visiva sono qui ricapitolate – i quattro mammiferi sotterranei sono tra le specie che mostrano un tasso accelerato su questi geni. È interessante notare che troviamo altre specie non sotterranee che mostrano un’accelerazione paragonabile a quella dei mammiferi sotterranei, indicando adattamenti nei sistemi visivi.
Discussione
Le transizioni indipendenti di quattro mammiferi in un ambiente sotterraneo sono state accompagnate da cambiamenti fenotipici convergenti che sono emersi come risultato dell’adattamento a nuovi stress ambientali nell’ecotopo sotterraneo (Nevo,1979; Leys et al., 2003; Lacey et al., 2000; Albert et al., 2007; Wilkinson, 2012). Qui riportiamo uno sforzo a livello genomico che comprende sia la codifica che le regioni di regolazione per identificare i cambiamenti del genotipo che hanno accompagnato questo adattamento fenotipico studiando i cambiamenti nei loro tassi evolutivi. Il nostro studio rivela che i geni che mostrano un’accelerazione convergente nelle specie sotterranee sono altamente arricchiti per la funzione nei percorsi visivi. La diminuzione della pressione selettiva sui percorsi visivi nell’ambiente sotterraneo poco illuminato porta ad un rilassamento della costrizione degli elementi genetici coinvolti in vari fenotipi correlati agli occhi, tra cui la morfologia oculare, la fotoringeccezione e la trasduzione visiva. Oltre al cambiamento accelerato dei geni nei percorsi visivi, osserviamo un tasso di evoluzione accelerato di molti geni coinvolti nei fenotipi legati alla pelle nei mammiferi sotterranei. Mentre vediamo un cambiamento accelerato nei geni visivi principalmente come risultato del rilassamento delle costrizioni, vediamo che alcuni geni legati alla pelle mostrano anche un cambiamento accelerato dovuto alla selezione positiva, forse come risultato della selezione di tratti che contribuiscono a uno stile di vita fossile. A parte questi due fenotipi, non osserviamo un arricchimento comparabilmente forte per i geni coinvolti nelle altre sfide ambientali associate a uno stile di vita sotterraneo, come l’ipossia, l’ipercapnia e l’alta infettività. È possibile che i mammiferi sotterranei possano mostrare adattamenti specie-specifici a queste sollecitazioni, mentre la nostra analisi da una prospettiva evolutiva convergente riflette i cambiamenti comuni a tutte le specie.
Un esame più attento dei geni accelerati che sono arricchiti per i percorsi legati alla visione rivela che i geni accelerati tendono ad essere lenti o retinici specifici. D’altra parte, i geni che codificano specificamente per la struttura oculare esterna, la cornea, non mostrano un’accelerazione significativa, indicando la conservazione di programmi di sviluppo importanti per l’architettura oculare. In due dei quattro nei con occhi non sottocutanei, la cornea può entrare in contatto diretto con l’ambiente esterno, rendendo forse necessario il corretto sviluppo della struttura nella nicchia sotterranea altamente infettiva. I geni specifici della lente e della retina che sono coinvolti nei processi di fotorilevamento e fototrasduzione sarebbero sottoposti a un maggiore rilassamento, dato l’ambiente poco illuminato, e subirebbero mutazioni dannose ad un ritmo molto più elevato. Le nostre analisi rivelano anche che i geni associati alle malattie oculari congenite sono accelerati nei quattro mammiferi sotterranei. Per il cristallino, che è in gran parte costituito da cristallini, troviamo molti geni cristallini(Crybb3, Cryba1, Crybb1, Crygc, Crygs, ecc.) nel nostro insieme accelerato di geni che contribuiscono a varie forme di cataratta(Graw, 2009). Allo stesso modo, troviamo che più geni coinvolti nelle ciliopatie siano accelerati, tra cui “cancellati nella discinesia ciliare primaria” (Dpcd), Iqcb1 (un componente delle ciglia primarie) e il fattore neurotrofico ciliare (Cntf). Un’ulteriore ispezione della lista accelerata dei geni potrebbe potenzialmente rivelare nuovi geni candidati che sono importanti per le malattie oculari congenite.
I geni che sono coinvolti nello sviluppo embrionale degli occhi non mostrano un’accelerazione globale significativa, potenzialmente dovuta alla loro natura pleiotropica: questi fattori di trascrizione dello sviluppo tendono ad avere importanti ruoli di regolazione in percorsi non legati all’occhio che non sono sotto vincolo rilassato. Abbiamo testato con successo la nostra ipotesi che gli elementi regolatori specifici per gli occhi di tali geni siano sotto una limitazione rilassata nei nei, utilizzando una nuova variante del nostro approccio che calcola un’accelerazione convergente a livello non codificante. Sebbene la forte accelerazione del tasso di accelerazione nei tre potenziatori oculari specifici del PAX6 suggerisca un rilassamento del vincolo nei mammiferi sotterranei, in assenza di test funzionali non possiamo essere sicuri che l’attività oculare specifica sia veramente persa. Inoltre, abbiamo trovato un arricchimento di tali regioni convergentemente accelerate non codificanti preferibilmente vicino ai geni del fattore di trascrizione dello sviluppo oculare, identificando i potenziali elementi potenziatori che guidano l’espressione di questi geni specificamente nell’occhio. Come approccio di validazione su larga scala, abbiamo dimostrato che l’accelerazione del tasso nei mammiferi sotterranei si sovrappone fortemente alle regioni identificate dal consorzio FANTOM5 come stimolatori dell’occhio. Queste analisi di prova di principio servono a illustrare il potere degli strumenti basati sull’evoluzione dei convertitori per l’identificazione degli elementi regolatori specifici dell’occhio. Nonostante l’apparente rapido tasso di evoluzione degli stimolatori nei mammiferi, i nostri metodi e quelli dei nostri colleghi dimostrano l’utilità di applicare approcci basati sull’evoluzione alle regioni conservate non codificate nell’identificazione degli elementi normativi alla base di importanti funzioni di sviluppo(Marcovitz et al., 2016; Villar et al., 2015). Questi metodi rappresentano un’opportunità unica per eseguire scansioni a livello genomico per gli elementi regolatori specifici dell’occhio e di altri tessuti, e potenzialmente servono come approcci complementari ai saggi a livello genomico nell’identificazione degli elementi attivi di potenziamento del genoma. Poiché un numero maggiore di genomi è sequenziato, ci aspettiamo che questi metodi diventino più potenti nel rivelare i cambiamenti di regolazione genica alla base dei fenotipi convergenti.
Nel complesso, i nostri risultati suggeriscono che i geni e le regioni non codificanti che sono coinvolti nei percorsi della visione stanno accumulando mutazioni deleterie attraverso processi neutri, dato il rilassamento della costrizione su questi percorsi nell’ambiente sotterraneo. Tuttavia, ciò non esclude la possibilità che le mutazioni iniziali inattivanti in questi percorsi siano di natura adattiva. L’arresto iniziale dello sviluppo dell’occhio può essere stato causato da cambiamenti selezionati positivamente, seguiti da una continua regressione dei geni strutturali e fisiologici dell’occhio attraverso processi neutri. In effetti, vi sono prove di una tale progressione degli eventi durante la regressione oculare nei pesci cavernicoli ciechi(Jeffery, 2005). Le forze adattive per la riduzione degli occhi possono essere state guidate dai costi energetici per il mantenimento di occhi funzionanti e dal rischio di ingresso di agenti patogeni attraverso l’occhio(Moran et al., 2015). Notiamo che la nostra analisi basata sui tassi rileva le firme di divergenza di sequenza che si basano su quanto osservato alla fine di questi processi e non fa luce sulla natura dei cambiamenti iniziali di inattivazione. Inoltre, i nostri metodi rilevano cambiamenti convergenti nei tassi di evoluzione dei geni e quindi non sono progettati per rilevare cambiamenti specie-specifici che potrebbero contribuire all’adattamento sotterraneo.
I nostri risultati mostrano l’accelerazione dei tassi di evoluzione convergente dei geni visivi e supportano fortemente i precedenti rapporti di regressione visiva nell’habitat sotterraneo. Emerling e Springer (2014) hanno studiato la regressione dei geni della retina in tre di queste quattro specie sotterranee e hanno mostrato che una diminuzione della quantità di luce che entra nella retina è associata a una maggiore incidenza di mutazioni inattivanti nei geni della retina. Hanno trovato un numero significativamente più alto di pseudogeni retinici nelle talpe rispetto a quello delle specie subaeree strettamente correlate, un’osservazione concordante con i nostri risultati basati sull’accelerazione del tasso. Gli sforzi di sequenziamento del genoma per il topo nudo e il topo cieco hanno anche mostrato un forte arricchimento degli pseudogeni nei percorsi visivi che sono associati al degrado della vista in queste specie (Cooper etal., 1993; Kim et al., 2011). Uno studio a livello genomico condotto da Prudent et al. (2016) ha rilevato significative differenze genomiche nei geni coinvolti nei percorsi visivi, come lo sviluppo degli occhi e la percezione della luce in due di questi quattro mammiferi sotterranei, ovvero la talpa dorata del mantello e il topo cieco. Utilizzando la nostra struttura basata sui tassi, abbiamo eseguito un’indagine rigorosa dei geni in evoluzione convergente in un ampio insieme di quattro specie sotterranee, e abbiamo chiarito la specificità dei tessuti e le ragioni alla base delle loro variazioni convergenti dei tassi. In una prima dimostrazione di questo tipo a livello non codificante, abbiamo applicato con successo i nostri metodi per rilevare gli stimolatori oculari specifici che mostrano un’evoluzione accelerata nei mammiferi sotterranei.
La regressione visiva non è limitata a queste quattro specie di talpe, e i mammiferi mostrano tipi specifici di regressione e altre differenze generali nelle capacità visive. La nostra analisi dei tassi genetici visivi di altre specie ha rivelato modelli e tendenze interessanti, in cui alcune specie in superficie con sistemi visivi scadenti o rimodellati hanno mostrato un’accelerazione media paragonabile a quella dei mammiferi sotterranei(Figura 8). Ciò offre l’opportunità di sondare ulteriormente le differenze specifiche nello sviluppo e nella funzione dei sistemi visivi in termini di percorsi specifici che sono rilassati o sotto costrizione tra le specie. Inoltre, l’integrazione di queste altre specie nella nostra struttura basata sul tasso può aiutare a mettere a punto il potere predittivo degli approcci basati sull’evoluzione. Una selezione deliberata di rami in primo piano basata su combinazioni specifiche tra questi mammiferi ipovedenti potrebbe migliorare notevolmente il potere dei metodi nel rilevare i cambiamenti convergenti, specialmente a livello non codificante. A questo proposito, la disponibilità di ricche e diverse annotazioni fenotipiche tra i mammiferi pone ulteriormente le basi per lo sviluppo di approcci basati sull’evoluzione nelle annotazioni funzionali e fenotipiche delle regioni non codificanti(Marcovitz et al., 2016; O’Leary et al., 2011; O‘Leary et al., 2013).
Materiali e metodi
Aggiunta di ortologi Nannospalax galili all’allineamento
Data l’assenza di Nannospalax galili (topo talpa cieco o BMR) negli allineamenti di 100 specie messi a disposizione dal browser del genoma UCSC, abbiamo impiegato un approccio personalizzato per aggiungere la corretta sequenza ortologica BMR in base al suo parente più prossimo sulla filogenesi delle specie di mammiferi, il topo. Utilizzando i modelli del gene BMR disponibili al pubblico(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCF_000622305.1_S.galili_v1.0/), abbiamo prima eseguito l’esplosione reciproca a coppie di tutte le sequenze di cDNA del gene BMR e le corrispondenti sequenze di cDNA di tutti i geni del genoma del topo mm9. Per ogni sequenza del gene mm9, successivamente identifichiamo l’ortologia BMR corretta usando il programma InParanoid come segue: il programma raggruppa coppie di sequenze dei due genomi interrogati in gruppi di ortologia, e la sequenza BMR che forma la coppia di ortologia principale (coppie con il miglior risultato reciproco) in ogni gruppo è stata identificata come l’ortologia corretta (Remmet al., 2001). Abbiamo poi eseguito un allineamento del profilo utilizzando il programma Muscle apertamente disponibile per aggiungere l’ortologia BMR identificata all’allineamento multispecie del gene (Edgar, 2004). Per tutte le analisi che coinvolgono regioni non codificanti, abbiamo utilizzato un approccio più semplice per identificare la regione ortologia BMR. Per ogni regione non codificata di interesse, abbiamo eseguito blastn con la sequenza ortologia mm9 come query contro il genoma assemblato BMR, con il valore di default Expect (E) di 10(NCBI Resource Coordinators, 2016). L’eventuale miglior punteggio di blastn risultante nel genoma BMR è stato aggiunto all’allineamento multispecie della regione non codificata (ottenuto dal browser del genoma UCSC) utilizzando l’utilità di allineamento del profilo del programma Muscle (Edgar, 2004).
Calcolo delle correlazioni geniche con l’ambiente sotterraneo
Utilizzando gli allineamenti degli aminoacidi di 100 specie di vertebrati a 100 vie dall’allineamento multiz disponibile presso il browser del genoma UCSC(Blanchette et al., 2004; Harris, 2007), sono stati selezionati per lo studio quegli allineamenti con un minimo di 10 specie che sono presenti anche in almeno due specie sotterranee. Abbiamo potato ogni allineamento per includere solo le 39 specie di interesse rappresentate nell’albero medio a livello proteomico(Figura 1A), dopo aver aggiunto l’ortologia BMR della sequenza genica corrispondente a questo allineamento come descritto nella sezione precedente. Per ogni allineamento di aminoacidi risultante, abbiamo stimato la lunghezza dei rami utilizzando il programma ‘aaml’ dell’analisi filogenetica con il pacchetto di massima probabilità (PAML) (Yang, 2007). Le lunghezze dei rami sono state stimate secondo un modello empirico di tassi di sostituzione degli aminoacidi con variabilità di tasso tra siti modellati come una distribuzione gamma approssimata con quattro classi discrete (per l’efficienza computazionale) e una classe aggiuntiva per i siti invariabili (modello aaml ‘Empirical + F’) (Whelan e Goldman, 2001; Yang, 1996). Le lunghezze dei rami sono state stimate sulla topologia di un albero di specie di mammiferi pubblicato(Murphy et al., 2004), modificato per includere Nannospalax galili la cui posizione nell’albero è stata dedotta in base alla letteratura esistente sulla sua ascendenza (Fanget al., 2014). Per le analisi che coinvolgono elementi non codificanti conservati, abbiamo identificato gli elementi di interesse sulla base della traccia phastCons umana generata dall’allineamento multiz a 100 vie dei vertebrati, eliminando ogni regione di sovrapposizione con la traccia degli mRNA umani. Per ognuno di questi elementi, abbiamo ottenuto un allineamento delle regioni ortologhe della nostra specie di interesse, potando dall’allineamento multiz a 100 vie dell’UCSC. La sezione precedente descrive ulteriormente la procedura utilizzata per aggiungere la regione ortologia BMR. Abbiamo successivamente stimato le lunghezze delle ramificazioni utilizzando il programma di base del pacchetto PAML sotto il modello del processo generale reversibile (REV) per i tassi di sostituzione dei nucleotidi, con variabilità di tasso tra i siti modellati come distribuzione gamma approssimata con quattro classi discrete e una classe aggiuntiva per i siti invariabili(Blanchette et al., 2004; Rodríguez et al., 1990).
Dopo aver ricostruito gli alberi di massima probabilità utilizzando PAML, filtriamo gli alberi di elementi genetici che hanno lunghezze di rami pari a zero in almeno l’80% delle specie presenti nell’albero. Le lunghezze dei rami grezzi degli alberi mantenuti dopo questa fase di filtraggio sono state trasformate in tassi relativi con il metodo dell’operatore di proiezione(Sato et al., 2005). Questi tassi relativi specifici per ramo sono stati poi utilizzati per eseguire un test di Mann-Whitney U e un’analisi di correlazione sulla variabile binaria dei rami ‘sotterranei’ o ‘fuori terra’ (cioè non sotterranei) (Figura 1A). I rami sotterranei sono quelli che portano alla talpa dal naso stellato(Condylura cristata), talpa d’oro a capo(Chrysochloris asiatica), topo nudo(Heterocephalus glaber) e topo cieco(Nannospalax galili).
Set di dati di elementi non codificanti “Occhio” contro “Altro” conservati e potenziatori FANTOM5
A. Elementi non codificanti conservati in prossimità dei geni del fattore di trascrizione
Abbiamo identificato 20 fattori di trascrizione dello sviluppo che hanno importanti ruoli di sviluppo nella formazione dei tessuti oculari (set “Eye”) sulla base di una ricerca bibliografica. I ruoli funzionali dettagliati di questi fattori di trascrizione e i tessuti oculari specifici dove sono rilevanti sono forniti nel file supplementare 9. La seconda parte del set di dati comprende 20 fattori di trascrizione identificati come appartenenti al set “Altro”. Questi fattori di trascrizione non hanno un ruolo noto nello sviluppo dell’occhio e le loro funzioni specifiche per i tessuti sono state identificate da un censimento dei fattori di trascrizione umani[File supplementare 9](Vaquerizas et al., 2009). Successivamente, abbiamo usato la traccia UCSC phastConsElements100way per identificare gli elementi non codificanti conservati vicino a ciascun gene dei fattori di trascrizione. Per ogni gene, identifichiamo 200 elementi che espandono la finestra di ricerca dal centro del gene lungo entrambe le direzioni. Limitiamo il numero di elementi a 200 per evitare che si verifichino distorsioni derivanti dal numero totale di elementi studiati vicino a un particolare gene. Questo porta ad un totale di 8000 elementi divisi equamente tra l’insieme “Occhio” e “Altro”.
B. FANTOM5 potenziatori
Abbiamo scaricato un set di dati di 44.460 potenziatori putativi identificati dal consorzio FANTOM5, incluse le loro coordinate mm9 e la quantificazione dell’espressione su 1190 campioni di tessuto(Andersson et al., 2014). Questi includono 1217 stimolatori con livello di espressione non zero nel bulbo oculare dei topi (‘Occhio’) attraverso quattro punti temporali di sviluppo e 13.100 stimolatori con espressione zero nel bulbo oculare (‘Altro’). Abbiamo ottenuto le coordinate hg19 corrispondenti di questi potenziatori utilizzando l’utility UCSC liftOver(Kent et al., 2002). Sulla base di questo, siamo stati in grado di mappare correttamente 995 potenziatori appartenenti al set “Eye” e 7695 potenziatori appartenenti al set “Other”. Tra questi potenziatori, siamo stati in grado di calcolare con fiducia la correlazione del tasso di evoluzione con l’ambiente sotterraneo per 946 potenziatori FANTOM5 “Eye” e 6.331 potenziatori FANTOM5 “Other”, dopo aver filtrato gli potenziatori che erano mal conservati nel nostro set di specie o i cui alberi erano dominati da rami di lunghezza zero.
Analisi di arricchimento funzionale
Abbiamo eseguito un’analisi di arricchimento funzionale utilizzando lo strumento GOrilla, cercando i termini GO arricchiti nell’insieme dei geni in primo piano rispetto all’insieme completo dei geni testati per la convergenza delle talpe(Eden et al., 2009). Inoltre, le informazioni funzionali per i geni associati al sottosuolo sono state estratte dai database Uniprot e RefSeq e dalla letteratura citata direttamente (UniProtConsortium, 2007; Pruitt et al., 2007). L’analisi di arricchimento è stata effettuata utilizzando il test ipergeometrico con il set di geni di fondo limitato ai geni che sono stati testati per la convergenza delle talpe e che avevano almeno un’annotazione nel file di annotazione corrispondente. La correzione per i test multipli è stata eseguita utilizzando i valori q del tasso di falsificazione(Storey, 2002).
Correzione di test per ipotesi multiple
L’analisi del False Discovery Rate è stata effettuata sulle probabilità risultanti dal test di Mann-Whitney U. Abbiamo impiegato un calcolo FDR basato sulla permutazione empirica, spesso l’approccio standard nell’analisi del genoma. Abbiamo generato 10.000 set di dati nulli, ottenendo ogni set di dati con la permutazione casuale delle etichette delle specie dei tassi relativi. Questo processo equivale a calcolare il test di Mann-Whitney U e l’analisi di correlazione su quattro rami casuali rispetto al resto invece che sulla variabile binaria dei rami ‘sotterranei’ rispetto a quelli ‘fuori terra’. I successivi set di dati permutati sono stati utilizzati per stimare i valori q di FDR per ogni p-valore nella nostra analisi di correlazione sotterranea. I geni che mostrano una correlazione maggiore o uguale a 0,5 nel test di Mann-Whitney U e significativa ad un FDR del 15% sono stati considerati geni con accelerazione molare, e il valore p che riflette la forza di questa accelerazione è indicato come accelerazione molare. Allo stesso modo, i geni che mostrano una correlazione inferiore a 0,5 e significativa a un FDR del 15% sono considerati geni decelerati dalla talpa.
Analisi genica specifica del tessuto
Al fine di determinare come specifici tessuti oculari si sono evoluti attraverso le specie sotterranee, abbiamo identificato per la prima volta set di geni specifici per i tessuti utilizzando i dati di espressione microarray da 91 tessuti di topo(Su et al., 2004). Abbiamo isolato i geni tessuti-specifici per cornea, iride, lenti e retina (compreso l’epitelio pigmentato della retina). Questi insiemi sono stati definiti come quelli con espressione differenziale significativa solo nel tessuto di interesse rispetto a tutti gli altri tessuti ad un alfa di 0,05 (T-test).
Modelli filogenetici di pressione selettiva
I geni ad accelerazione sotterranea sono stati sottoposti a modelli filogenetici dell’evoluzione del codone per verificare la presenza di prove significative di rilassamento del vincolo o di selezione positiva sui rami sotterranei dei mammiferi. Usando PAML, abbiamo eseguito codeml utilizzando cinque diversi modelli: il modello neutro di ramo-sito (BS Neutro), il modello di selezione di ramo-sito (BS Alt Mod), il modello neutro di sito (M1), il modello di selezione positiva (M8) e il suo modello nullo (M8A) (Yang, 2007). Per valutare la significatività del rilassamento del vincolo sui rami dei mammiferi sotterranei, abbiamo eseguito test di likelihood ratio (LRT) tra BS Neutral e il suo nullo modello M1. Le LRT tra il BS Alt Mod e il suo BS Neutro nullo sono state utilizzate per dedurre una selezione positiva sui rami dei mammiferi sotterranei. Le probabilità sono state assegnate per ciascuna di queste due LRT utilizzando la distribuzione chi-quadrata con 1 grado di libertà. La selezione positiva a livello di mammifero è stata dedotta utilizzando i modelli M8 vs M8A e le rispettive LRT, utilizzando la distribuzione chi quadrata con 1 grado di libertà per valutare la significatività delle LRT. Per calcolare la correlazione tra l’accelerazione delle talpe e il grado di specificità tissutale dei geni, abbiamo stimato l’accelerazione delle talpe di ciascun gene come segue: utilizzando un modello di selezione del sito del ramo (BS Alt Mod) abbiamo stimato due diversi valori di ω (dN/dS) –unoper i quattro rami sotterranei e uno per il resto dei rami sull’albero. L’accelerazione della talpa è stata calcolata come differenza tra i due valori di ω che sono stati stimati.
References
- Albert EM, Zardoya R, García-París M. Phylogeographical and speciation patterns in subterranean worm lizards of the genus Blanus (Amphisbaenia: Blanidae). Molecular Ecology. 2007; 16:1519-1531. DOI | PubMed
- Andersen DC. Below-ground herbivory in natural communities: A review emphasizing fossorial animals. The Quarterly Review of Biology. 1987; 62:261-286. DOI
- Andersson R, Gebhard C, Miguel-Escalada I, Hoof I, Bornholdt J, Boyd M, Chen Y, Zhao X, Schmidl C, Suzuki T, Ntini E, Arner E, Valen E, Li K, Schwarzfischer L, Glatz D, Raithel J, Lilje B, Rapin N, Bagger FO, Jørgensen M, Andersen PR, Bertin N, Rackham O, Burroughs AM, Baillie JK, Ishizu Y, Shimizu Y, Furuhata E, Maeda S, Negishi Y, Mungall CJ, Meehan TF, Lassmann T, Itoh M, Kawaji H, Kondo N, Kawai J, Lennartsson A, Daub CO, Heutink P, Hume DA, Jensen TH, Suzuki H, Hayashizaki Y, Müller F, Forrest ARR, Carninci P, Rehli M, Sandelin A. An atlas of active enhancers across human cell types and tissues. Nature. 2014; 507:455-461. DOI | PubMed
- Ar A, Arieli R, Shkolnik A. Blood-gas properties and function in the fossorial mole rat under normal and hypoxic-hypercapnic atmospheric conditions. Respiration Physiology. 1977; 30:201-218. DOI | PubMed
- Kent WJ, Sugnet CW, Furey TS, Roskin KM, Pringle TH, Zahler AM, Haussler D. The human genome browser at UCSC. Genome Research. 2002; 12:996-1006. DOI | PubMed
- Blanchette M, Kent WJ, Riemer C, Elnitski L, Smit AF, Roskin KM, Baertsch R, Rosenbloom K, Clawson H, Green ED, Haussler D, Miller W. Aligning multiple genomic sequences with the threaded blockset aligner. Genome Research. 2004; 14:708-715. DOI | PubMed
- Carmona FD, Jiménez R, Collinson JM. The molecular basis of defective lens development in the Iberian mole. BMC Biology. 2008; 6DOI | PubMed
- Carmona FD, Ou J, Jiménez R, Collinson JM. Development of the cornea of true moles (Talpidae): morphogenesis and expression of PAX6 and cytokeratins. Journal of Anatomy. 2010; 217:488-500. DOI | PubMed
- Catania KC. A nose that looks like a hand and acts like an eye: the unusual mechanosensory system of the star-nosed mole. Journal of Comparative Physiology A: Sensory, Neural, and Behavioral Physiology. 1999; 185:367-372. DOI
- Cei G. Ortogenesi parallela e degradazione degli organi della vista negli Spalacidi. Monit Zool Ital, Firenze. 1946a; 55:69-88.
- Cei G. L’occhio di “Heterocephalus glaber” Rupp. Note anatomodes -crittive e istologiche. Monit Zool Ital, Firenze. 1946b; 55:89-96.
- Chen S, Li W. A color-coding amacrine cell may provide a blue-off signal in a mammalian retina. Nature Neuroscience. 2012; 15:954-956. DOI | PubMed
- Chikina M, Robinson JD, Clark NL. Hundreds of Genes Experienced Convergent Shifts in Selective Pressure in Marine Mammals. Molecular Biology and Evolution. 2016; 33:2182-2192. DOI | PubMed
- Cooper HM, Herbin M, Nevo E. Visual system of a naturally microphthalmic mammal: the blind mole rat, Spalax ehrenbergi. The Journal of Comparative Neurology. 1993; 328:313-350. DOI | PubMed
- Dahlqvist J, Klar J, Tiwari N, Schuster J, Törmä H, Badhai J, Pujol R, van Steensel MA, Brinkhuizen T, Brinkhuijzen T, Gijezen L, Chaves A, Tadini G, Vahlquist A, Dahl N. A single-nucleotide deletion in the POMP 5′ UTR causes a transcriptional switch and altered epidermal proteasome distribution in KLICK genodermatosis. The American Journal of Human Genetics. 2010; 86:596-603. DOI | PubMed
- Dimanlig PV, Faber SC, Auerbach W, Makarenkova HP, Lang RA, a LR. The upstream ectoderm enhancer in Pax6 has an important role in lens induction. Development. 2001; 128:4415-4424. PubMed
- Dobler S, Dalla S, Wagschal V, Agrawal AA. Community-wide convergent evolution in insect adaptation to toxic cardenolides by substitutions in the Na,K-ATPase. PNAS. 2012; 109:13040-13045. DOI | PubMed
- Dubost G. Les mammiferes souterrains. Rev Ecol Biol Sol. 1968; 5:99-133.
- Eden E, Navon R, Steinfeld I, Lipson D, Yakhini Z. GOrilla: a tool for discovery and visualization of enriched GO terms in ranked gene lists. BMC Bioinformatics. 2009; 10DOI | PubMed
- Edgar RC. MUSCLE: multiple sequence alignment with high accuracy and high throughput. Nucleic Acids Research. 2004; 32:1792-1797. DOI | PubMed
- Ellerman JR. The subterranean mammals of the world. Transactions of the Royal Society of South Africa. 1956; 35:11-20. DOI
- Eloff G. Orientation in the mole-rat cryptomys. British Journal of Psychology. General Section. 1951; 42:134-145. DOI
- Emerling CA, Springer MS. Eyes underground: regression of visual protein networks in subterranean mammals. Molecular Phylogenetics and Evolution. 2014; 78:260-270. DOI | PubMed
- Fang X, Nevo E, Han L, Levanon EY, Zhao J, Avivi A, Larkin D, Jiang X, Feranchuk S, Zhu Y, Fishman A, Feng Y, Sher N, Xiong Z, Hankeln T, Huang Z, Gorbunova V, Zhang L, Zhao W, Wildman DE, Xiong Y, Gudkov A, Zheng Q, Rechavi G, Liu S, Bazak L, Chen J, Knisbacher BA, Lu Y, Shams I, Gajda K, Farré M, Kim J, Lewin HA, Ma J, Band M, Bicker A, Kranz A, Mattheus T, Schmidt H, Seluanov A, Azpurua J, McGowen MR, Ben Jacob E, Li K, Peng S, Zhu X, Liao X, Li S, Krogh A, Zhou X, Brodsky L, Wang J. Genome-wide adaptive complexes to underground stresses in blind mole rats Spalax. Nature Communications. 2014; 5:1039-1052. DOI | PubMed
- Foote AD, Liu Y, Thomas GW, Vinař T, Alföldi J, Deng J, Dugan S, van Elk CE, Hunter ME, Joshi V, Khan Z, Kovar C, Lee SL, Lindblad-Toh K, Mancia A, Nielsen R, Qin X, Qu J, Raney BJ, Vijay N, Wolf JB, Hahn MW, Muzny DM, Worley KC, Gilbert MT, Gibbs RA. Convergent evolution of the genomes of marine mammals. Nature Genetics. 2015; 47:272-275. DOI | PubMed
- Fu DJ, Thomson C, Lunny DP, Dopping-Hepenstal PJ, McGrath JA, Smith FJ, McLean WH, Pedrioli DM. Keratin 9 is required for the structural integrity and terminal differentiation of the palmoplantar epidermis. The Journal of investigative dermatology. 2014; 134:754-763. DOI | PubMed
- Graw J. Genetics of crystallins: cataract and beyond. Experimental Eye Research. 2009; 88:173-189. DOI | PubMed
- Griffin C, Kleinjan DA, Doe B, van Heyningen V, Heyningen van. New 3′ elements control Pax6 expression in the developing pretectum, neural retina and olfactory region. Mechanisms of Development. 2002; 112:89-100. DOI | PubMed
- Hamilton WJ. Habits of the Star-Nosed Mole, Condylura cristata. Journal of Mammalogy. 1931; 12DOI
- Harris RS. Improved pairwise alignment of genomic DNA. ProQuest. 2007.
- Hendriks W, Leunissen J, Nevo E, Bloemendal H, de Jong WW. The lens protein alpha A-crystallin of the blind mole rat, Spalax ehrenbergi: evolutionary change and functional constraints. PNAS. 1987; 84:5320-5324. DOI | PubMed
- Hennies HC, Küster W, Mischke D, Reis A. Localization of a locus for the striated form of palmoplantar keratoderma to chromosome 18q near the desmosomal cadherin gene cluster. Human Molecular Genetics. 1995; 4:1015-1020. DOI | PubMed
- Hetling JR, Baig-Silva MS, Comer CM, Pardue MT, Samaan DY, Qtaishat NM, Pepperberg DR, Park TJ. Features of visual function in the naked mole-rat Heterocephalus glaber. Journal of Comparative Physiology A. 2005; 191:317-330. DOI | PubMed
- Hill WCO, Porter A, Bloom RT, Seago J, Southwick MD. Field and laboratory studies on the naked mole rat, heterocephalus glaber. Proceedings of the Zoological Society of London. 2009; 128:455-514. DOI
- Hiller M, Schaar BT, Indjeian VB, Kingsley DM, Hagey LR, Bejerano G. A "forward genomics" approach links genotype to phenotype using independent phenotypic losses among related species. Cell Reports. 2012; 2:817-823. DOI | PubMed
- Jarvis JUM, Sale JB. Burrowing and burrow patterns of East African mole-rats Tachyoryctes, Heliophobius and Heterocephalus. Journal of Zoology. 2010; 163:451-479. DOI
- Jeffery WR. Adaptive evolution of eye degeneration in the Mexican blind cavefish. Journal of Heredity. 2005; 96:185-196. DOI | PubMed
- Jeffery WR. Regressive evolution in Astyanax cavefish. Annual Review of Genetics. 2009; 43:25-47. DOI | PubMed
- Kammandel B, Chowdhury K, Stoykova A, Aparicio S, Brenner S, Gruss P. Distinct cis-essential modules direct the time-space pattern of the Pax6 gene activity. Developmental Biology. 1999; 205:79-97. DOI | PubMed
- Kim EB, Fang X, Fushan AA, Huang Z, Lobanov AV, Han L, Marino SM, Sun X, Turanov AA, Yang P, Yim SH, Zhao X, Kasaikina MV, Stoletzki N, Peng C, Polak P, Xiong Z, Kiezun A, Zhu Y, Chen Y, Kryukov GV, Zhang Q, Peshkin L, Yang L, Bronson RT, Buffenstein R, Wang B, Han C, Li Q, Chen L, Zhao W, Sunyaev SR, Park TJ, Zhang G, Wang J, Gladyshev VN. Genome sequencing reveals insights into physiology and longevity of the naked mole rat. Nature. 2011; 479:223-227. DOI | PubMed
- Kim JW, Yang HJ, Oel AP, Brooks MJ, Jia L, Plachetzki DC, Li W, Allison WT, Swaroop A. Recruitment of rod photoreceptors from short-wavelength-sensitive cones during the evolution of nocturnal vision in mammals. Developmental Cell. 2016; 37:520-532. DOI | PubMed
- Kleinjan DA, Seawright A, Mella S, Carr CB, Tyas DA, Simpson TI, Mason JO, Price DJ, van Heyningen V. Long-range downstream enhancers are essential for Pax6 expression. Developmental Biology. 2006; 299:563-581. DOI | PubMed
- Kleinjan DA, Seawright A, Schedl A, Quinlan RA, Danes S, van Heyningen V. Aniridia-associated translocations, DNase hypersensitivity, sequence comparison and transgenic analysis redefine the functional domain of PAX6. Human Molecular Genetics. 2001; 10:2049-2059. DOI | PubMed
- Koay G, Kearns D, Heffner HE, Heffner RS. Passive sound-localization ability of the big brown bat (Eptesicus fuscus). Hearing Research. 1998; 119:37-48. DOI | PubMed
- Lacey EA, Patton JL, Cameron GN. Life Underground: The Biology of Subterranean Rodents. University of Chicago Press: Chicago; 2000.
- Lartillot N, Poujol R. A phylogenetic model for investigating correlated evolution of substitution rates and continuous phenotypic characters. Molecular Biology and Evolution. 2011; 28:729-744. DOI | PubMed
- Leys R, Watts CHS, Cooper SJB, Humphreys WF. Evolution of subterranean diving beetles (Coleoptera: Dytiscidae hydroporini, Bidessini) in the arid zone of Australia. Evolution. 2003; 57:2819-2834. DOI | PubMed
- Li W, Chen S, DeVries SH. A fast rod photoreceptor signaling pathway in the mammalian retina. Nature Neuroscience. 2010; 13:414-416. DOI | PubMed
- Liu Y, Cotton JA, Shen B, Han X, Rossiter SJ, Zhang S. Convergent sequence evolution between echolocating bats and dolphins. Current Biology. 2010; 20:R53-R54. DOI | PubMed
- Losos JB. Convergence, adaptation, and constraint. Evolution. 2011; 65:1827-1840. DOI | PubMed
- Marcovitz A, Jia R, Bejerano G. "Reverse Genomics" Predicts Function of Human Conserved Noncoding Elements. Molecular Biology and Evolution. 2016; 33:1358-1369. DOI | PubMed
- McDonough CM, Loughry WJ. The Nine-Banded Armadillo: A Natural History. University of Oklahoma Press; 2013.
- Moran D, Softley R, Warrant EJ. The energetic cost of vision and the evolution of eyeless Mexican cavefish. Science Advances. 2015; 1:e1500363. DOI | PubMed
- Murphy WJ, Pevzner PA, O’Brien SJ. Mammalian phylogenomics comes of age. Trends in Genetics. 2004; 20:631-639. DOI | PubMed
- NCBI Resource Coordinators. Database resources of the National Center for Biotechnology Information. Nucleic Acids Research. 2016; 44:D7-D19. DOI | PubMed
- Nevo E. Adaptive Convergence and Divergence of Subterranean Mammals. Annual Review of Ecology and Systematics. 1979; 10:269-308. DOI
- Nowak RM. Walker’s Mammals of the World. Johns Hopkins University Press; 1999.
- Nemec P, Cveková P, Benada O, Wielkopolska E, Olkowicz S, Turlejski K, Burda H, Bennett NC, Peichl L. The visual system in subterranean African mole-rats (Rodentia, Bathyergidae): retina, subcortical visual nuclei and primary visual cortex. Brain Research Bulletin. 2008; 75:356-364. DOI | PubMed
- O’Leary MA, Bloch JI, Flynn JJ, Gaudin TJ, Giallombardo A, Giannini NP, Goldberg SL, Kraatz BP, Luo ZX, Meng J, Ni X, Novacek MJ, Perini FA, Randall ZS, Rougier GW, Sargis EJ, Silcox MT, Simmons NB, Spaulding M, Velazco PM, Weksler M, Wible JR, Cirranello AL. The placental mammal ancestor and the post-K-Pg radiation of placentals. Science. 2013; 339:662-667. DOI | PubMed
- O’Leary MA, Kaufman S. MorphoBank: phylogenomics in the cloud. Cladistics : The International Journal of the Willi Hennig Society. 2011; 27:529-537. DOI
- Prudent X, Parra G, Schwede P, Roscito JG, Hiller M. Controlling for Phylogenetic Relatedness and Evolutionary Rates Improves the Discovery of Associations Between Species’ Phenotypic and Genomic Differences. Molecular Biology and Evolution. 2016; 33:2135-2150. DOI | PubMed
- Pruitt KD, Tatusova T, Maglott DR. NCBI reference sequences (RefSeq): a curated non-redundant sequence database of genomes, transcripts and proteins. Nucleic Acids Research. 2007; 35:D61-D65. DOI | PubMed
- Quilliam TA. The mole’s sensory apparatus. Journal of Zoology. 1966; 149:76-88. DOI
- Quilliam TA. The mole’s sensory apparatus. Journal of Zoology. 2009; 149:76-88. DOI
- Remm M, Storm CE, Sonnhammer EL. Automatic clustering of orthologs and in-paralogs from pairwise species comparisons. Journal of Molecular Biology. 2001; 314:1041-1052. DOI | PubMed
- Rodríguez F, Oliver JL, Marín A, Medina JR. The general stochastic model of nucleotide substitution. Journal of Theoretical Biology. 1990; 142:485-501. DOI | PubMed
- Rosenblum EB, Parent CE, Brandt EE. The molecular basis of phenotypic convergence. Annual Review of Ecology, Evolution, and Systematics. 2014; 45:203-226. DOI
- Sanyal S, Jansen HG, de Grip WJ, Nevo E, de Jong WW, de GWJ, de JWW. The eye of the blind mole rat, Spalax ehrenbergi. Rudiment with hidden function?. Investigative ophthalmology & visual science. 1990; 31:1398-1404. PubMed
- Sato T, Yamanishi Y, Kanehisa M, Toh H. The inference of protein-protein interactions by co-evolutionary analysis is improved by excluding the information about the phylogenetic relationships. Bioinformatics. 2005; 21:3482-3489. DOI | PubMed
- Stern DL. The genetic causes of convergent evolution. Nature Reviews Genetics. 2013; 14:751-764. DOI | PubMed
- Storey JD. A direct approach to false discovery rates. Journal of the Royal Statistical Society: Series B. 2002; 64:479-498. DOI
- Su AI, Wiltshire T, Batalov S, Lapp H, Ching KA, Block D, Zhang J, Soden R, Hayakawa M, Kreiman G, Cooke MP, Walker JR, Hogenesch JB. A gene atlas of the mouse and human protein-encoding transcriptomes. PNAS. 2004; 101:6062-6067. DOI | PubMed
- Swanson WJ, Nielsen R, Yang Q. Pervasive adaptive evolution in mammalian fertilization proteins. Molecular Biology and Evolution. 2003; 20:18-20. DOI | PubMed
- Sweet G. The eyes of Chrysochloris hottentota and C. asiatica. Journal of Cell Science. 1909; 2:328-338.
- UniProt Consortium. The Universal Protein Resource (UniProt). Nucleic acids research. 2007; 35:D193-197. DOI | PubMed
- Vaquerizas JM, Kummerfeld SK, Teichmann SA, Luscombe NM. A census of human transcription factors: function, expression and evolution. Nature Reviews Genetics. 2009; 10:252-263. DOI | PubMed
- Villar D, Berthelot C, Aldridge S, Rayner TF, Lukk M, Pignatelli M, Park TJ, Deaville R, Erichsen JT, Jasinska AJ, Turner JM, Bertelsen MF, Murchison EP, Flicek P, Odom DT. Enhancer evolution across 20 mammalian species. Cell. 2015; 160:554-566. DOI | PubMed
- Whelan S, Goldman N. A general empirical model of protein evolution derived from multiple protein families using a maximum-likelihood approach. Molecular Biology and Evolution. 2001; 18:691-699. DOI | PubMed
- Wilkinson M. Caecilians. Current Biology. 2012; 22:R668-R669. DOI | PubMed
- Xu PX, Zhang X, Heaney S, Yoon A, Michelson AM, Maas RL. Regulation of Pax6 expression is conserved between mice and flies. Development. 1999; 126:383-395. PubMed
- Yang Z. Among-site rate variation and its impact on phylogenetic analyses. Trends in Ecology & Evolution. 1996; 11:367-372. DOI | PubMed
- Yang Z. PAML 4: phylogenetic analysis by maximum likelihood. Molecular Biology and Evolution. 2007; 24:1586-1591. DOI | PubMed
- Zhang J, Nielsen R, Yang Z. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level. Molecular Biology and Evolution. 2005; 22:2472-2479. DOI | PubMed
Fonte
Partha R, Chauhan BK, Ferreira Z, Robinson JD, Lathrop K, et al. () Subterranean mammals show convergent regression in ocular genes and enhancers, along with adaptation to tunneling. eLife 6e25884. https://doi.org/10.7554/eLife.25884





