Il cancro al seno è una malattia ad alta prevalenza, che colpisce molte donne in tutto il mondo ad un certo punto della loro vita. 1 Attualmente, la chirurgia conservativa del seno (BCS) è una buona opzione per le donne con tumore al seno in stadio precoce e può essere combinata con la radioterapia post-operatoria per ridurre il rischio di recidiva del tumore al seno ipsilaterale (IBTR). Una meta-analisi eseguita dal Gruppo di Collaborazione dei Tumori del Seno in fase precoce (EBCTCG) ha dimostrato che la radioterapia post-operatoria riduce significativamente l’IBTR e la mortalità per tumore al seno a 15 anni in seguito a BCS. 2 Tuttavia, anche i margini chirurgici sono importanti. Nel 2014, la Society of Surgical Oncology e l’American Society for Radiation Oncology hanno pubblicato una “consensus guideline” che sottolinea l’importanza dei margini di gestione per la BCS nei pazienti in fase I e II stadio. Questa linea guida si basa su una meta-analisi di 28162 pazienti di 33 studi che hanno rilevato che i margini positivi, definiti sul carcinoma duttale invasivo (IDC) o sul carcinoma duttale in situ (DCIS), che sono stati analizzati con metodi patologici postoperatori, sono associati ad un aumento almeno doppio dell’IBTR. Così, i margini negativi sono associati a bassi tassi di IBTR e hanno il potenziale di diminuire i tassi di ri-escrezione. 3 Tuttavia, non esiste ancora uno standard globale stabilito per la gestione dei margini intraoperatori in tempo reale in BCS.
I metodi patologici intraoperatori che includono l’analisi di sezioni congelate e la citologia dell’impronta hanno il potenziale di ridurre le percentuali di margini positivi. 4 Ma questi metodi non possono essere generalmente applicati agli attuali BCS negli ospedali di tutto il mondo, a causa dell’aumento del tempo operativo derivante dalla procedura patologica e dell’aumento del carico di lavoro per i patologi durante la realizzazione di sezioni congelate (figura1A).
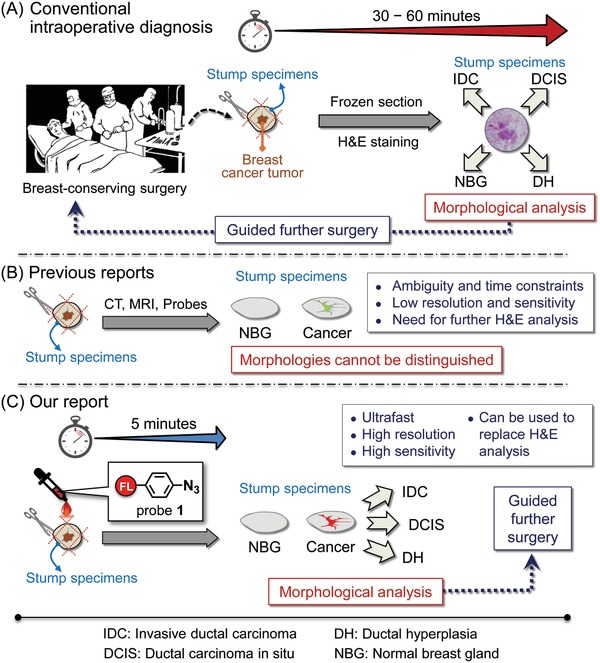
Figura 1.Presentazione schematica della diagnosi intraoperatoria. A) Il metodo basato sulla patologia attualmente utilizzato in BCS. I campioni congelati di monconi di cancro al seno sono analizzati al microscopio con colorazione H&E. Ulteriori interventi chirurgici sono guidati in base all’analisi morfologica, cioè, a seconda delle regioni IDC, DCIS, DH, e NBG. Diverse procedure patologiche richiedono circa 1 h. B) Le tecniche recentemente emergenti utilizzano la TAC, la risonanza magnetica e le sonde chimiche. I metodi possono visualizzare i tessuti tumorali vivi, ma hanno ancora problemi associati alla sensibilità e alla selettività. È importante notare che questi metodi non sono in grado di visualizzare la morfologia dei vari stadi tumorali e richiedono ancora una patologia per confermare la loro diagnosi (quindi è improbabile che sostituiscano il metodo convenzionale H&E come descritto in (A)). C) Il metodo “click-to-sense” descritto in questo articolo permette di diagnosticare la morfologia del cancro con elevata sensibilità e selettività semplicemente trattando i tessuti vivi con la sonda chimica 1 per 5 minuti e poi con la microscopia a fluorescenza. Il nostro nuovo metodo potrebbe sostituire la diagnosi patologica convenzionale.
In tali circostanze, sono stati ricercati nuovi metodi o tecniche e recentemente ne sono emersi diversi per la gestione intraoperatoria in tempo reale dei margini del seno con l’utilizzo di tessuti vivi. Questi metodi includono MarginProbe, che rileva le onde elettromagnetiche riflesse dalle regioni del cancro al seno,5 tomografia microcalcolata (TC),6 e risonanza magnetica ex vivo (RM),7 (Figura 1B). Questi metodi comportano l’imaging delle macrodimensioni delle lesioni tumorali, ma non è possibile valutare la morfologia del cancro e la localizzazione anche a livello cellulare nei tessuti vivi. Infatti, il tasso di falsi positivi utilizzando il dispositivo MarginProbe era più alto (53,6%) rispetto a quello del braccio di controllo (16,6%) in uno studio prospettico randomizzato. 5 È stata anche riportata una sonda fluorescente basata sulla chimica che viene attivata selettivamente in presenza di γ-glutamil transferasi, che è sovraespressa nelle lesioni tumorali (Figura 1B). 8 Questo metodo si basa sull’aumento della fluorescenza in funzione del tempo, per cui la riproducibilità da campione a campione (cioè la selettività del cancro dovuta all’elevata fluorescenza di fondo e alla diffusione della fluorescenza) ha reso i chirurghi che si occupano di tumore al seno esitanti ad utilizzarla in BCS reali. Più criticamente, questo metodo chimico non può diagnosticare la morfologia del cancro. A volte si presume che questi metodi siano utili per la chirurgia conservativa del seno, ma poiché non possono essere utilizzati per determinare la morfologia del cancro nei tessuti vivi, è improbabile che sostituiscano i metodi patologici che utilizzano tessuti congelati. Infatti, nessuno dei nuovi metodi emergenti è stato applicato con successo alla gestione dei margini intraoperatori in tempo reale nella BCS. Quindi, c’è ancora bisogno di nuovi metodi per consentire la diagnosi rapida, selettiva e sensibile di varie morfologie tumorali utilizzando tessuti vivi.
L’acroleina, un’aldeide altamente tossica α,β-insaturizzata,9 è stata a lungo conosciuta come un biomarcatore associato a una serie di disturbi legati allo stress ossidativo, tra cui i tumori. È prodotta attraverso l’ossidazione enzimatica della treonina o delle poliammine10 ed è anche generata durante l’ossidazione reattiva delle specie di ossigeno (ROS)-mediata dall’ossidazione di lipidi altamente insaturi. 11 A volte viene generato su una scala di pochi 100 × 10-6m nelle cellule stressate dall’ossidazione12 ed è più tossico per le cellule rispetto al ROS come il perossido di idrogeno (H2O2) o i radicali idrossilici (-OH), i principali fattori di stress ossidativo associati a una varietà di disturbi. 13 Infatti, l’analisi dello spettro di massa degli estratti in fase liquida dei lisati cellulari ha dimostrato che le cellule tumorali producono acroleina. 14
Abbiamo recentemente sviluppato una sonda 1 “click-to-sense” per il rilevamento dell’acroleina, basata su una reazione “clic” acroleina/azide (Figura 1C). 15 A differenza dei precedenti metodi per il rilevamento dell’acroleina, come la derivatizzazione/HPLC,16 analisi degli spettri di massa,14 o il rilevamento mAb degli addotti dell’acroleina-lisina N-(3-formil-3,4-deidropiperidino)lisina (FDP-lisina),17 il nostro metodo è in grado di rilevare in modo sensibile la presenza di acroleina utilizzando la fluorescenza, in modo che anche l’acroleina presente a livello di nm possa essere rilevata direttamente all’interno delle cellule vive. Così, abbiamo scoperto che la funzionalità dell’azoturo nella sonda 1 ha partecipato senza problemi alla reazione di ciclocaricodi 1,3-dipolare precedentemente inesplorata con l’acroleina generata dalle cellule, producendo derivati della 1,2,3-triazolina (figura2A). I derivati della 1,2,3-triazolina si sono poi decomposti nei corrispondenti composti diazoelettrici all’interno o sulla superficie delle cellule, e che questi composti si sono immediatamente e non discriminatamente coniugati con i costituenti cellulari per ancorare la fluorescenza all’interno delle cellule. Il nuovo metodo è degno di nota per la sua semplicità: le cellule stressate ossidativamente vengono trattate con la sonda 1, e i coniugati acroleina/prova 1 “cliccati” ancorati all’interno delle cellule possono essere misurati direttamente dalla lettura della fluorescenza. L’intensità della fluorescenza è proporzionale alla concentrazione di acroleina generata dalle cellule.
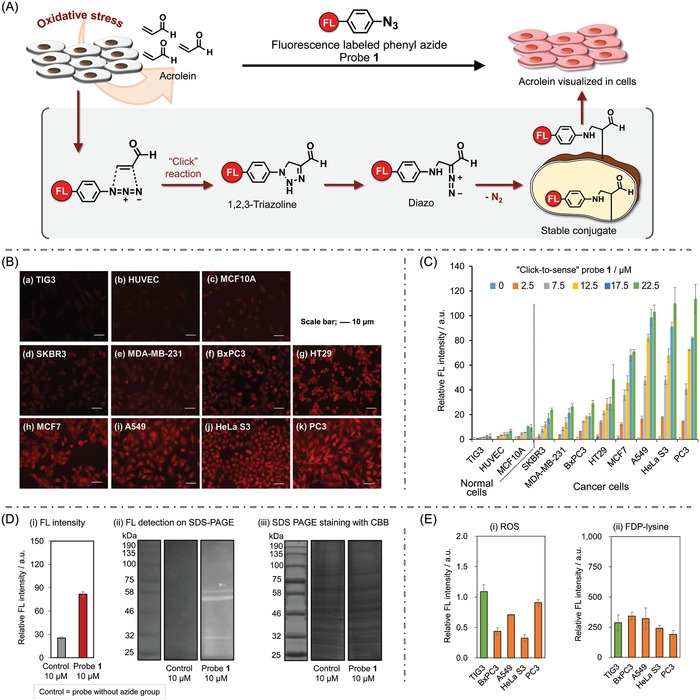
Figura 2.A) “Click-to-sense” il cancro: metodo e meccanismo. L’azoturo 1 etichettato in fluorescenza reagisce senza problemi con l’acroleina generata dalle cellule tumorali attraverso una reazione di ciclocarico 1,3-dipolare (reazione di “click” dell’acroleina/azide). 1,2,3-triazolo come il prodotto “cliccato” si decompone in composti diazoici, che reagiscono con i costituenti delle cellule per ancorare l’etichetta di fluorescenza all’interno delle cellule. La concentrazione di acroleina è analizzata in modo semplice dalla lettura di fluorescenza a livello di cellule intere. B) Discriminazione delle cellule tumorali dalle cellule normali mediante la sonda “click-to-sense” 1. Undici linee cellulari sono state trattate con la nostra sonda a 37 °C per 30 min. Le cellule sono state fissate e la fluorescenza è stata registrata da SpectraMax M2e, Dispositivi Molecolari. Immagini di fluorescenza delle cellule dopo l’incubazione con 22,5 × 10-6m di sonda 1 (vedi Figura S1, Informazioni di supporto, per le immagini dopo il trattamento con altre concentrazioni di 1). (a) TIG3; (b) HUVEC; (c) MCF10A; (d) SKBR3; (e) MDA-MB-231; (f) BxPC3; (g) HT29; (h) MCF7; (i) A549; (j) HeLa S3; e (k) PC3. La barra della scala indica 10 µm. C) Intensità di fluorescenza osservata per ogni linea cellulare nel campo di concentrazione di 0-22,5 × 10-6m della sonda 1. Da sinistra a destra: TIG3, HUVEC, MCF10A, SKBR3, MDA-MB-231, BxPC3, HT29, MCF7, A549, HeLa S3 e PC3. L’intensità della fluorescenza è stata normalizzata come quella emessa da 10 000 cellule. D-i) Intensità totale di fluorescenza dei lisati delle cellule HeLa S3 (vedi metodo in Informazioni di supporto); (ii) le immagini SDS-PAGE per il rilevamento di fluorescenza dei lisati delle cellule HeLa S3 trattati con 10 × 10-6m di sonda 1 e sonda di controllo senza gruppo azide (vedi anche Figura S2, Informazioni di supporto). I costituenti delle cellule sono etichettati dalla sonda 1 in base al meccanismo descritto in (A); (iii) la SDS-PAGE da colorazione coomassiale (vedi metodo in Informazioni di supporto). E) (i) ROS e (ii) analisi FDP-lisina dei lisati delle linee cellulari selezionate (da sinistra a destra: TIG3, BxPC3, A549, HeLa S3, e PC3). Questi metodi (vedi Informazioni di supporto) non sono in grado di rilevare selettivamente il cancro.
Considerando che l’acroleina è prodotta in quantità significative nel cancro da cellule sottoposte a stress ossidativo, abbiamo ipotizzato che la nostra sonda 1 “click-to-sense” potrebbe permetterci di visualizzare in modo selettivo e sensibile le cellule tumorali nei tessuti vivi. Ci siamo resi conto che se la selettività poteva essere garantita, il metodo poteva essere utilizzato come metodo discriminante, economico e facile da eseguire per il rilevamento del cancro durante l’intervento chirurgico.
In questo articolo riportiamo, utilizzando il nostro metodo, il primo successo in assoluto della valutazione intraoperatoria “in tempo reale” con l’utilizzo di monconi di resezione da tessuti vivi. La nostra sonda supera molte delle limitazioni di altre tecniche finora riportate (Figura 1C). A differenza di altri metodi, può discriminare, in modo chiaro, IDC e DCIS dalla ghiandola mammaria normale (NBG) e dall’iperplasia duttale (DH), e visualizzare in modo sensibile la morfologia e la localizzazione del cancro (anche a livello cellulare) nel moncone di resezione durante l’intervento. La nostra sonda potrebbe essere utilizzata insieme agli attuali interventi chirurgici di conservazione del seno negli ospedali di tutto il mondo, eliminando la necessità di procedure patologiche laboriose, costose e dispendiose in termini di tempo con l’utilizzo di tessuti congelati.
“Sonda “Click-to-Sense 1 Immagini chiaramente l’acroleina generata dalle cellule tumorali in vitro: Abbiamo iniziato ad indagare se la nostra sonda 1 “click-to-sense” potesse rappresentare selettivamente le cellule tumorali in vitro. In questo studio, otto linee di cellule tumorali come PC3 (cellule tumorali della prostata umana), HeLa S3 (cellule tumorali della cervice uterina umana), A549 (cellule epiteliali alveolari alveolari adenocarcinomiche umane), MCF7 (cellule tumorali della mammella umana), HT29 (cellule tumorali del colon umano), BxPC3 (cellule tumorali del pancreas umano), MDA-MB-231 (cellule tumorali umane del seno) e SKBR3 (cellule tumorali umane del seno), così come tre linee cellulari normali come MCF10A (cellule mammarie umane normali), HUVEC (cellule endoteliali della vena ombelicale umana), e TIG3 (cellule diploidi umane normali) come controllo sono stati utilizzati. A seguito di procedure stabilite,15 cellule sono state trattate con cinque concentrazioni di sonda 1 (2,5 × 10-6, 7,5 × 10-6, 12,5 × 10-6, 17,5 × 10-6 e 22,5 × 10-6m) a 37 °C per 30 minuti, e l’intensità della fluorescenza è stata registrata da spettrofluorimetro (SpectraMax M2e; Dispositivi Molecolari). L’intensità della fluorescenza è stata normalizzata per ogni linea cellulare dal numero di 10000 cellule (Figura 2B,C e Figura S1, Informazioni di supporto).
Mentre le cellule MCF10A, HUVEC e TIG3 hanno mostrato solo una fluorescenza trascurabile nel range di concentrazione di 0-22,5 × 10-6m della nostra sonda, è stato osservato un notevole aumento della fluorescenza dipendente dalla dose per tutti i tipi di cellule tumorali. La più alta intensità di fluorescenza è stata osservata in PC3, HeLa S3, A549 e MCF7, seguita dalle serie di cellule HT29, BxPC3, MDA-MB-231 e SKBR3 (Figura 2C). Così, diverse linee cellulari tumorali mostrano diverse intensità di fluorescenza (il che implica diversi livelli di aumento della produzione di acroleina) al trattamento con la nostra sonda. Coerente con il meccanismo proposto di come il “click-to-sense” sonda 1 rileva l’acroleina generata da cellule cancellate (vedi Figura 2A), costituenti intracellulari e di membrana (ad esempio, proteine o lipidi) delle cellule tumorali rappresentative sono stati etichettati dalla sonda 1, in base alla filtrazione ripartita per dimensioni (Figura 2D-i) e l’analisi SDS-PAGE dei lisati delle cellule HeLa S3 trattate con la sonda 1 (Figura 2D-ii,iii, e Figura S2, Informazioni di supporto).
Va notato che queste cellule tumorali non possono essere rilevate in modo selettivo da una sonda ROS (ROS-ID Total ROS detection kit, Enzo) o da una sonda di acroleina convenzionale (che rileva la FDP-lisina prodotta sulle proteine cellulari)17, 18 (Figura 2E). Pertanto, i dati della Figura 2 mostrano che la nostra sonda 1 “click-to-sense” può discriminare sensibilmente il cancro dalle cellule normali sulla base dell’acroleina prodotta in situ in o su cellule vive.
“Sonda “Click-to-Sense1
e colorazione Hoechst per la differenziazione tra tumore al seno vivo, NBG e tessuti DH: Per studiare la capacità della sonda di essere utilizzata in vivo, abbiamo eseguito analisi su 30 tessuti tumorali (20 IDC e 10 DCIS), 30 tessuti NBG e 5 tessuti DH di 30 pazienti affette da tumore al seno che hanno subito un intervento chirurgico al seno nel periodo dal marzo 2017 al marzo 2018 presso l’ospedale universitario di Osaka, Osaka, Giappone, utilizzando un metodo di colorazione a doppia fluorescenza con entrambi i nostri tessuti”.sonda “click-to-sense” 1 e Hoechst 33 342 + 33 258. I tessuti vivi sono stati tagliati dalla camera a matrice di tessuto per creare una superficie piana, immersi in una soluzione di 5 × 10-6, 10 × 10-6, o 20 × 10-6m della sonda 1 “click-to-sense” per 5 minuti, e risciacquati con tampone (Figura3A). I tessuti risultanti sono stati poi analizzati direttamente dalla Keyence BZ-X710 dotata del sistema di algoritmo di sezionamento ottico per ottenere sia immagini grossolane che immagini di colorazione a doppia fluorescenza (Figura 3B). L’algoritmo di sezionamento Keyence permette all’utente di ottenere immagini chiare senza sfocatura di fluorescenza, in un modo paragonabile a quelle catturate su un microscopio confocale laser, ma in una frazione del tempo e senza gli effetti dannosi di un laser. Nel sezionamento ottico la luce di eccitazione viene fatta passare attraverso un modulatore elettronico di luce e viene proiettata in un modello a griglia solo sulle aree focalizzate del campione. Queste sezioni focalizzate vengono poi combinate per creare un’immagine chiara e priva di fluorescenza fuori fuoco (Figura 3C). Poiché questo non richiede l’uso di laser ad alta intensità di illuminazione, il danno al campione è ridotto al minimo. 19
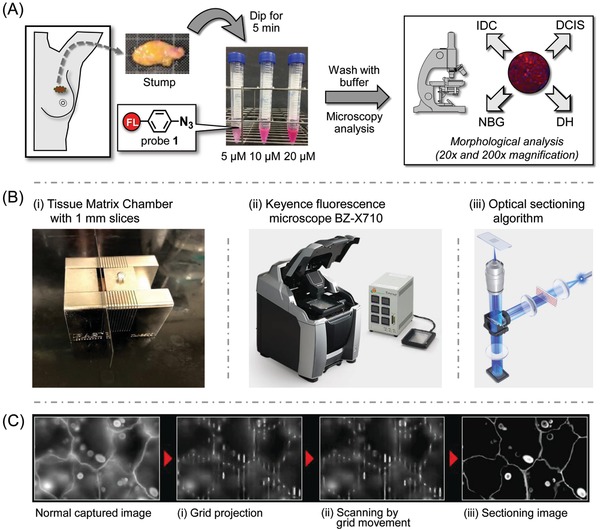
Figura 3.A) Procedura schematica per l’etichettatura dei tessuti vivi (monconi di cancro al seno) con la sonda 1. B) (i) Una camera a matrice di tessuto è stata utilizzata per tagliare il tessuto fresco; (ii) Microscopio a fluorescenza Keyence BZ-X710; (iii) Sistema di algoritmo di sezionamento ottico. C) Il BZ-X710 utilizza una luce di eccitazione strutturata per scansionare il campione, rendendo possibile l’acquisizione di immagini chiare senza sfocatura di fluorescenza. (i) La luce di eccitazione di una lampada a ioduri metallici viene fatta passare attraverso un elemento di proiezione elettronico, che proietta la luce sul campione secondo un modello a griglia. La griglia viene proiettata solo sulle aree focalizzate del campione. (ii) Lo spostamento della griglia fa sì che il campione venga scansionato, consentendo la cattura di più immagini del campione. (iii) L’estrazione delle sole aree su cui il reticolo viene proiettato dalle immagini multiple catturate evita l’effetto di sfocatura della fluorescenza in direzione verticale. Ciò produce immagini chiare in cui viene estratto solo il segnale dalla superficie focalizzata. Riprodotto con il permesso. Copyright 2018, KEYENCE CORPORATION.
Le immagini rappresentative sono mostrate in figura4A-i (IDC e NBG) e Figura 4A-ii (DCIS e NBG). Sia nelle immagini grossolane che in quelle fluorescenti, i tessuti del cancro al seno (IDC o DCIS) sono stati più macchiati dalla nostra sonda rispetto ai tessuti NBG. Il significato statistico della selettività del tumore è mostrato nella Figura 4B. L’intensità media di fluorescenza dei tumori al seno (IDC o DCIS, con la loro identificazione basata sull’analisi morfologica come discusso di seguito) era significativamente più alta di quella del tessuto NBG in modo statisticamente significativo in funzione della dose, a concentrazioni della sonda di 10 × 10-6 e 20 × 10-6m. Quando abbiamo ottimizzato il valore di soglia divisibile dell’intensità di fluorescenza, la sensibilità e la specificità erano del 90% e del 97% alla concentrazione della sonda di 10 × 10-6m; ed erano del 97% e del 97% alla concentrazione della sonda di 20 × 10-6m, per la classificazione binaria tra tumori del seno e NBG, rispettivamente (Figura 4B). Questa sensibilità e specificità non sono mai state raggiunte con altri metodi. È importante notare che la nostra sonda 1 “click-to-sense” ha etichettato i tessuti del cancro al seno con un’intensità di fluorescenza media simile, indipendentemente dai sottotipi IDC come lo stato del recettore degli estrogeni (ER) o del recettore 2 del fattore di crescita epidermico umano (HER2), così come dai sottotipi DCIS per diversi stati ER (Figura S4, Informazioni di supporto).
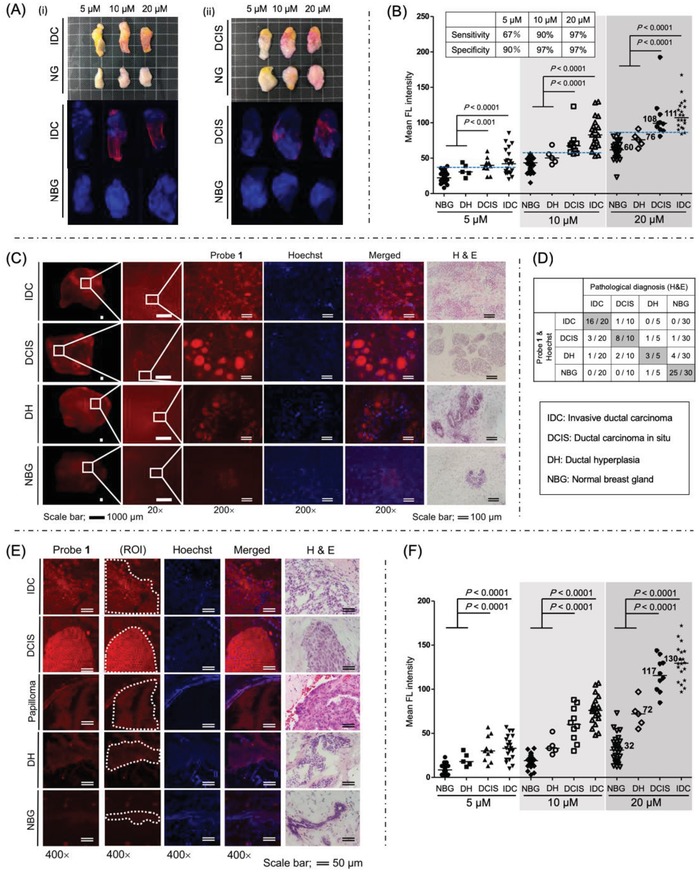
Figura 4.Figura 4. Immagini microscopiche di tumore al seno vivo (IDC, DCIS, DH) e dei tessuti della ghiandola mammaria normale (NBG) etichettati con la sonda “click-to-sense” 1 [per 5 minuti a 5 × 10-6, 10 × 10-6, e 20 × 10-6m (rosso)] e Hoechst (blu). A) Immagini e immagini microscopiche a doppia colorazione di (i) IDC e NBG, (ii) DCIS e NBG. Abbiamo definito il ROI come mostrato nella Figura S3 (Informazioni di supporto). B) Analisi statistica della sensibilità e della selettività del cancro tra IDC, DCIS, DH e NBG. Test statistico: Valori P, Mann-Whitney U-test; Bars, mediana. C) Espansione delle immagini; la morfologia di IDC, DCIS, DH e NBG può essere chiaramente rilevata da 20× e 200× (immagini fluorescenti ad alto ingrandimento) essendo etichettata dalla sonda 1 a 20 × 10-6m. Il rilevamento della morfologia del cancro è stato coerente con le immagini di colorazione H&E delle sezioni congelate preparate a partire dagli stessi campioni di tessuto anonimi. D) Analisi di 30 tessuti tumorali vivi (20 IDC e 10 DCIS), 30 tessuti NBG vivi adiacenti e 5 tessuti DH vivi adiacenti di 30 pazienti affette da cancro al seno. Confronto dell’analisi morfologica tra il metodo “click-to-sense” (mediante imaging diretto dei tessuti vivi contrassegnati con 1 etichetta per microscopia (20×) e (200×)) con il metodo patologico convenzionale H&E, eseguito dal patologo in forma anonima. L’analisi H&E è stata eseguita preparando le sezioni congelate dagli stessi campioni di tessuto. E) Immagini microscopiche confocali (400×) delle sezioni congelate degli stessi campioni preparati in (C). Per questi esperimenti, abbiamo fatto alcune fette dello stesso blocco tumorale, e abbiamo fatto un’immagine delle cellule tumorali usando una fetta per i nostri metodi “click-to-sense” e un’altra per i metodi H&E. Sembrano leggermente diverse dalle diverse sezioni, cioè la differenza causata dallo “spessore” delle sezioni (ciascuna presa consecutivamente dal blocco tumorale di 6 µm), ma sono clinicamente considerate come la stessa colorazione della sezione. Abbiamo definito il ROI per l’analisi. F) Analisi statistica della sensibilità e della selettività delle cellule tumorali tra IDC, DCIS, DH e NBG. Test statistico: Valori P, Mann-Whitney U-test; Bars, mediana.
Degno di nota è che ingrandendo le immagini IDC e DCIS marcate a fluorescenza di 20 volte, la morfologia di IDC e DCIS potrebbe essere chiaramente immaginata. Per consentire un’immagine ancora più chiara della morfologia del cancro, abbiamo ottenuto immagini di tessuti vivi di pazienti affette da cancro al seno macchiati da 20 × 10-6m di sonda 1 “click-to-sense” in campo ad alta potenza (200×). I patologi hanno diagnosticato e confrontato le immagini di colorazione a doppia fluorescenza (“click-to-sense” sonda 1 e Hoechst) con le immagini di colorazione dell’ematossilina e dell’eosina (H&E) degli stessi campioni di tessuto in forma anonima. Le immagini sono mostrate nella Figura 4C (IDC, DCIS, DH e NBG) e nelle Figure S5-S8 (Informazioni di supporto). Colorando i tessuti del cancro al seno a un livello di fluorescenza più elevato rispetto all’NBG, la fluorescenza TAMRA derivata dalla nostra sonda “click-to-sense” 1 (rosso) e le immagini di colorazione Hoechst (blu) hanno mostrato chiaramente le caratteristiche morfologiche dei tessuti e i confini tra IDC, DCIS, DH e NBG. Va notato che le immagini fluorescenti dei tumori sono in buon accordo con quelle della colorazione H&E (Figura 4C e Figure S5-S8, Informazioni di supporto). Quindi, questo segna il primo successo dell’imaging della morfologia tumorale a livello di tessuto vivo al di fuori dei metodi convenzionali di patologia.
Al fine di confrontare l’accuratezza della nostra chimica “click-to-sense” con la patologia convenzionale (colorazione H&E delle sezioni permanenti), abbiamo analizzato 30 tessuti tumorali vivi (20 IDC e 10 DCIS), 30 tessuti NBG vivi adiacenti e 5 tessuti DH vivi adiacenti di 30 pazienti con cancro al seno. In seguito agli esperimenti della Figura 4C, le immagini a fluorescenza di tutti i tessuti vivi sono state ampliate (di 20× e di 200×) per rappresentare la morfologia patologica dei tumori. A questo ingrandimento, la chimica “click-to-sense” ha discriminato molto chiaramente IDC, DCIS, DH e NBG, con i dati che mostrano un eccellente accordo con la colorazione H&E delle sezioni permanenti come metodo patologico convenzionale (Figura 4D). Va notato che la diagnosi e la discriminazione dei tessuti tumorali vivi solo attraverso l’analisi dell’intensità della fluorescenza sarebbe difficile per il DH perché una parte significativa della fluorescenza DH (media di 76 a 20 × 10-6m della sonda 1, Figura 4B) si sovrapponeva all’NBG (media di 60). Tuttavia, sulla base dell’analisi morfologica, diventa possibile diagnosticare correttamente 3 su 5 (60%) DH, e 4 su 5 (80%) lesioni non maligne (Figura 4D). La diagnosi può essere eseguita più rapidamente rispetto al metodo convenzionale utilizzando l’analisi della sezione congelata, che richiede molto tempo.
Per confermare che la colorazione fluorescente era basata sull’etichettatura a livello delle cellule tumorali, l’analisi fluorescente delle sezioni congelate (spessore 6 µm) è stata ulteriormente eseguita con un microscopio confocale Zeiss LSM710 in un campo ad alta potenza (400×), e quindi le stesse sezioni congelate sono state anche colorate con H&E (Figura 4E). Abbiamo trovato che le cellule del cancro al seno (IDC o DCIS) sono state colorate in modo fluorescente a livelli molto più alti rispetto alle cellule dei tessuti NBG in modo statisticamente significativo dose-dipendente (Figura 4F). In buon accordo con l’analisi dei tessuti nella Figura 4B, l’intensità media di fluorescenza delle cellule DH era di 72 (Figura 4F), che è tra quelle delle cellule DCIS e NBG. Questo valore è allo stesso livello del papilloma intraduttale, una condizione benigna del seno, (valore di 79 nella Figura 4E) quando sono state colorate da 20 × 10-6m della sonda 1 (Figura 4E). Tuttavia, l’analisi morfologica (Figura S9, Informazioni di supporto) potrebbe discriminare il papilloma intraduttale dal DCIS, evidenziando il vantaggio della nostra chimica “click-to-sense” in termini di capacità di visualizzare la morfologia della lesione.
Allo stesso modo, il nostro metodo poteva anche distinguere la morfologia del carcinoma lobulare invasivo (ILC) (Figura S10, Informazioni di supporto). Così, vari carcinomi residui possono essere accuratamente identificati utilizzando la sonda “click-to-sense” 1. Questo risultato dimostra che la morfologia dei tessuti vivi nelle pazienti con cancro al seno può essere facilmente identificata, fornendo un supporto per la BCS.
In questo articolo abbiamo dimostrato, utilizzando sia studi in vitro che in vivo, che la nostra sonda “click-to-sense” è in grado di discriminare rapidamente tra cellule tumorali e cellule normali, richiedendo solo la colorazione dei tessuti vivi per 5 minuti, e che può visualizzare chiaramente la morfologia e la localizzazione del cancro in un modo quasi equivalente alla sezione congelata. Il nostro metodo non è stato influenzato dalle condizioni del campione, dagli sfondi di fluorescenza, o dalla diffusione della fluorescenza, e non dipende dal tempo delle reazioni enzimatiche o dell’espressione enzimatica da parte delle cellule tumorali, in quanto si concentra specificamente sulla sovraespressione dell’acroleina generata endogena in varie cellule tumorali, con la chimica in-cellula “click-to-sense” che si ancorano con successo all’etichetta di fluorescenza nei costituenti delle cellule tumorali. Si può pensare che la colorazione in immunofluorescenza potrebbe essere l’ideale. Ma in questa ricerca, dovremmo macchiare i vari tumori vivi “eterogenei” (e tutti) delle pazienti affette da cancro al seno, ma non quelli della cellula e dei modelli animali, dove vengono espressi gli antigeni specifici associati al tumore. Le precedenti sperimentazioni di noi e degli altri non sono riuscite per questo motivo ad immunocolorare i campioni clinici di tumore. Per questo motivo il nostro nuovo metodo “click-to-sense” è estremamente significativo e importante, e dovrebbe essere messo in evidenza.
Gli esperimenti descritti in questo articolo indicano che il nostro metodo potrebbe essere utilizzato come un nuovo metodo per la gestione dei margini dei tessuti vivi. La potenziale utilità del nostro metodo richiede studi clinici prospettici per la valutazione intraoperatoria in tempo reale con l’utilizzo di monconi di resezione di pazienti con cancro al seno. L’importanza clinica della nostra sonda per le caratteristiche morfologiche e patologiche merita un ulteriore studio negli ospedali di tutto il mondo. Si ipotizza che la nostra chimica “click-to-sense” potrebbe essere utile anche per la gestione dei margini in tempo reale nel cancro gastrico, nel cancro al colon, nel cancro ai polmoni e in altri tumori dei tessuti vivi durante l’intervento chirurgico, così come per la gestione dei margini in tempo reale della polipectomia endoscopica. Sarebbe anche utile valutare il nostro metodo nella diagnosi di metastasi linfonodali ascellari del cancro al seno durante l’intervento chirurgico. Un altro potenziale utilizzo della chimica “click-to-sense” potrebbe essere quello di identificare le cellule tumorali circolanti (CTC).
Sarebbe anche utile considerare l’uso dell’intelligenza artificiale (IA) accanto al nostro metodo. L’IA ha dimostrato una potenziale utilità come strumento diagnostico in clinica. In un recente studio, l’IA ha superato i patologi nella diagnosi di piccole quantità di tumore che si era diffuso ai linfonodi nelle donne con tumore al seno. 20 Poiché la nostra sonda 1 “click-to-sense” potrebbe essere utilizzata per diagnosticare i tessuti tumorali durante il BCS più rapidamente rispetto ai metodi convenzionali di H&E, prevediamo che l’IA possa essere utilizzata per analizzare i tessuti vivi macchiati dalla sonda 1, aumentando l’efficienza della valutazione intraoperatoria in tempo reale.
In conclusione, siamo stati in grado di dimostrare che la nostra sonda 1 “click-to-sense” ha chiaramente e rapidamente discriminato i tumori al seno da NBG nei tessuti vivi delle pazienti, non solo immaginando la presenza di lesioni tumorali, ma anche mostrando le loro caratteristiche morfologiche. Il nostro nuovo metodo ha il potenziale per diventare un nuovo metodo di gestione dei margini dei tessuti vivi con elevata selettività, e potrebbe essere utilizzato come metodo discriminante, a basso costo e facile da eseguire per il rilevamento del cancro durante l’intervento chirurgico. Il metodo sarà confermato in uno studio clinico prospettico che comprenderà la valutazione intraoperatoria dei monconi di resezione nelle pazienti affette da cancro al seno.
Sezione sperimentale
Rilevamento dell’acroleina nelle cellule tumorali e nelle cellule normali: Undici linee di cellule (TIG3, HUVEC, MCF10A, SKBR3, MDA-MB-231, BxPC3, HT29, MCF7, A549, HeLa S3 e PC3) sono state seminate su una piastra a 96 pozzetti (2 × 104 cellule per pozzetto) e lasciate attaccare per 24 ore a 37 °C. Le cellule sono state poi trattate con 100 µL di varie concentrazioni di sonda 1 soluzione (2,5 × 10-6, 7,5 × 10-6, 12,5 × 10-6, 17,5 × 10-6, 22,5 × 10-6m) nel terreno di coltura, e incubato per 30 minuti a temperatura ambiente. Dopo questa incubazione, le cellule sono state risciacquate due volte con soluzione salina tamponata con fosfato (PBS) e risospese in 100 µL PBS. L’intensità della fluorescenza è stata poi misurata utilizzando uno spettrofluorimetro (SpectraMax M2e, Dispositivi Molecolari). L’intensità della fluorescenza è stata normalizzata per ogni linea cellulare dal numero di 10 000 cellule. Le cellule sono state poi fissate utilizzando il 4% di paraformaldeide in PBS per 10 minuti a temperatura ambiente, risciacquate con PBS due volte, e risospese in 100 µL PBS. Le immagini delle cellule fisse sono state registrate con il microscopio a fluorescenza invertito (IX71, Olympus).
Cancro e tessuti della ghiandola mammaria normale da pazienti con cancro al seno: Tutti i campioni di tessuto sono stati prelevati da resezione chirurgica e il consenso informato è stato ottenuto da ogni paziente. I soggetti reclutati per questo studio comprendevano 30 pazienti con cancro al seno primario (20 pazienti IDC e 10 pazienti DCIS; età media: 49,4 anni; range: 36–67 anni) che hanno subito un intervento chirurgico al seno nel periodo dal marzo 2017 al marzo 2018 presso l’ospedale universitario di Osaka, Osaka, Giappone. Questo studio riguarda anche il DH di cinque pazienti e il papilloma intraduttale di due pazienti.
Microscopia a fluorescenza Analisi della sonda “Click-to-Sense 1 e colorazione Hoechst: I tessuti vivi sono stati tagliati dalla camera a matrice di tessuto, immersi in una soluzione di 5 × 10-6, 10 × 10-6, o 20 × 10-6m della sonda “click-to-sense” 1 in tubi conici da 15 mL per 5 minuti a temperatura ambiente, e poi risciacquato con PBS tre volte. I nuclei dei tessuti risultanti sono stati macchiati con Hoechst 33 342 + 33 258 (Dojindo, Kumamoto, Giappone). I campioni di tessuto sono stati poi trasferiti in un piatto con fondo di vetro (Thermo Scientific Nunc) e inseriti nel microscopio a fluorescenza (Keyence BZ-X710, Osaka, Giappone) dotato di sistema di algoritmo di sezionamento ottico (per ottenere immagini chiare senza sfocatura fluorescenza). Le immagini a fluorescenza di tutto il tessuto sono state scattate in campo a bassa potenza. Per valutare l’intensità media di fluorescenza della sonda 1 (colorata di rosso), è stata impostata una regione di interesse (ROI) al centro delle lesioni rappresentative (Figura S3, Informazioni di supporto). L’intensità media di fluorescenza di ogni ROI è stata calcolata e analizzata con il software Image J (National Institute of Health, Rockville, MD, USA). I campioni di tessuto sono stati collocati nella stessa luce di eccitazione e tempo di esposizione per l’analisi sia della sonda 1 (TAMRA) che di Hoechst. Le immagini a fluorescenza di tessuti vivi colorati da 20 × 10-6m della sonda 1 sono state prese in campo ad alta potenza (200×) per l’osservazione morfologica dei campioni di tessuto.
Gli stessi tessuti vivi sono stati poi incorporati nel composto della temperatura di taglio ottimale (OCT), congelare, e tagliare per l’analisi con il microscopio confocale (Zeiss LSM710, Göttingen, Germania) in campo ad alta potenza (400×). Le immagini sono state prese da almeno tre diverse e scelte a caso di ogni campione. Per valutare l’intensità media di fluorescenza della sonda 1 (colorata di rosso), il ROI è stato impostato al centro delle lesioni rappresentative (Figura 4E). L’intensità media di fluorescenza di ogni ROI è stata calcolata e analizzata con il software Image J. Gli stessi tessuti sono stati poi fissati in paraformaldeide al 4% per la colorazione immunoistochimica. La valutazione patologica è stata effettuata mediante analisi al microscopio ottico delle sezioni colorate H&E.
Analisi statistica: Per tutte le analisi statistiche è stato utilizzato il programma statistico R (http://cran.r-project.org-project.org). L’associazione dell’intensità media di fluorescenza della sonda 1 nei tessuti chirurgici è stata valutata con il test a U di Mann-Whitney. La significatività statistica è stata assunta per P < 0,05.
Conflitto di interessi
Gli autori non dichiarano alcun conflitto di interessi.
Informazioni di supporto
References
- Cancers. 2015; 7:908. PubMed
- Early Breast Cancer Trialists’ Collaborative Group (EBCTCG). Lancet. 2011; 378:1707. PubMed
- Ann. Surg. Oncol.. 2014; 21:717. PubMed
- Ann. Surg.. 2017; 265:300. PubMed
- Ann. Surg. Oncol.. 2014; 21:1589. PubMed
- Br. J. Radiol.. 2016; 89:20150581. PubMed
- Breast J.. 2013; 19:659. PubMed
- Sci. Rep.. 2015; 5:12080. PubMed
- Toxicol. Sci.. 2000; 57:6. PubMed
- Biochim. Biophys. Acta, Nucleic Acids Protein Synth.. 1971; 228:223. PubMed
- Trends Cardiovasc. Med.. 1999; 9:109. PubMed
- Mol. Nutr. Food Res.. 2011; 55:1320. PubMed
- Biochem. Biophys. Res. Commun.. 2009; 378:313. PubMed
- Anal. Biochem.. 2002; 305:251. PubMed
- ACS Sens.. 2016; 1:623.
- J. Chromatogr. B: Biomed. Sci. Appl.. 1986; 375:159. PubMed
- J. Biol. Chem.. 1998; 273:16058. PubMed
- Sci. Rep.. 2016; 6:35872. PubMed
- Keyence Corporation. 2018. Publisher Full Text
- The Camelyon Consortium. J. Am. Med. Assoc.. 2017; 318:2199.
Fonte
Tanei T, Pradipta AR, Morimoto K, Fujii M, Arata M, et al. (2018) Cascade Reaction in Human Live Tissue Allows Clinically Applicable Diagnosis of Breast Cancer Morphology. Advanced Science 6(2): 1801479. https://doi.org/10.1002/advs.201801479





