Abstract
Introduzione
ATP è universale nelle cellule. È usato come blocco di costruzione metabolica e come cofattore per accoppiare reazioni esergoniche ed endergoniche, rendendo l’ATP fondamentale per la vita come i gradienti protonici e il codice genetico. Una funzione aggiuntiva come idrotropo biologico per mantenere le proteine solubili è stato ulteriormente suggerito (Mandl et al., 1952; Patel et al., 2017). De-fosforilazione ad ADP e AMP, e ri-fosforilazione ad ATP consentono flussi ad alta energia in base alle dimensioni relativamente piccole del pool nella cellula (Rich, 2003). I principali siti di sintesi di ATP sono le sintasi ATP guidate dalla forza motrice protonica stabilita attraverso la membrana interna dei mitocondri, e i plastidiotidi tilakoidi nelle piante. L’ATP prodotto alimenta tipicamente la vasta gamma di processi che richiedono energia, come la motilità, il trasporto e l’espressione genica, in altre parti della cellula, anche se i plastidi consumano anche notevoli quantità di ATP nel ciclo di Calvin-Benson, in una misura che varia sostanzialmente con il ciclo chiaro/scuro. L’accoppiamento di queste interazioni richiede lo scambio di ATP/ADP tra gli organuli e il citosol. Così, la regolazione dei livelli di ATP citosolico e organellare e il trasporto di ATP/ADP attraverso l’involucro mitocondriale e plastidico, danno luogo a dinamiche di ATP particolarmente complesse nelle cellule vegetali, che dipendono in modo critico dal tipo di tessuto e dalle condizioni esterne (Neuhaus et al., 1997; Flügge, 1998; Reiser et al., 2004; Haferkamp et al., 2011). L’esatta natura dell’interazione tra mitocondri e cloroplasti nel mantenimento dell’omeostasi citosolica e nucleare dell’ATP, soprattutto in condizioni mutevoli, come i cicli luce-buio o la variazione dello stato di O2/CO2, è stata studiata per decenni, utilizzando prevalentemente tecniche biochimiche o NMR in vivo (Bailleul et al., 2015; Gardeström e Igamberdiev, 2016). Ad esempio, la comprensione dei pool nucleotidici subcellulari di adenina è stata possibile attraverso il frazionamento rapido dei protoplasti fogliari basato su filtri a membrana, rivelando un’interazione complessa e dinamica tra i tre compartimenti cellulari (Lilley et al., 1982; Stitt et al., 1982; Gardeström e Wigge, 1988; Krömer e Heldt, 1991; Krömer et al., 1993). L’attuale consenso è che l’ATP citosolico è fornito principalmente dai mitocondri, sia al buio, ma anche alla luce, quando la fotorespirazione può essere il principale motore della sintesi dell’ATP (Igamberdiev et al., 2001). Poco si sa, tuttavia, sulle differenze tra organi, tessuti e cellule e sulle caratteristiche delle loro specifiche risposte nel tempo.
Sfide simili si applicano ad altri parametri fisiologici e metabolici, come il pH, il Ca2+ libero, i potenziali delle coppie tiolo redox e le concentrazioni di piccole molecole, compresi i regolatori di crescita delle piante. Tuttavia, lo sviluppo di reporter in situ ha fornito una comprensione sempre più sofisticata del loro comportamento in vivo (De Michele et al., 2014; Uslu e Grossmann, 2016). Ad esempio, approfondimenti dettagliati sui gradienti di pH subcellulari e le loro dinamiche si sono rivelati fondamentali per il trasporto delle membrane, la degradazione delle proteine, l’energia e l’omeostasi ionica (Schwarzländer et al., 2012; Luo et al., 2015). Allo stesso modo, le caratteristiche spazio-temporali dei transitori liberi di Ca2+ sono fondamentali per la segnalazione delle risposte di stress e delle interazioni pianta-microbo (Choi et al., 2014b; Keinath et al., 2015). La capacità di monitorare separatamente i potenziali redox delle piscine subcellulari di glutatione ha rivelato un paesaggio redox citosolico molto più ridotto di quanto precedentemente previsto e ha portato a nuovi concetti di regolazione e segnalazione del redox (Marty et al., 2009; Morgan et al., 2013; Schwarzländer et al., 2016).
Recentemente, sono state ingegnerizzate diverse diverse proteine fluorescenti per l’ATP (Berg et al., 2009; Imamura et al., 2009; Kotera et al., 2010; Nakano et al., 2011; Tantama et al., 2013; Yoshida et al., 2016). ‘Perceval’ si basa su una singola proteina mVenus permuta circolare fusa alla proteina batterica regolatrice GlnK1 di Methanococcus jannaschii. Il legame competitivo di ATP e ADP a GlnK1 porta a cambiamenti inversi di due massimi di eccitazione per fornire una lettura raziometrica di ATP:ADP (Berg et al., 2009). La variante ‘PercevalHR’ è stata ottenuta per mutagenesi, ed ha un range dinamico migliorato di circa 4 (Tantama et al., 2013). Tuttavia, entrambe le varianti sono fortemente sensibili al pH, e richiedono la misurazione del pH e la correzione per misurazioni significative in vivo. Al contrario, la famiglia di sensori raziometrici ATeam è stata introdotta come molto meno sensibile al pH. I sensori ATeam condividono il loro design complessivo con i sensori a trasferimento di energia a risonanza di Förster (FRET) della famiglia Cameleon, ampiamente utilizzati, utilizzando il frammento ε-subunità di sintasi ATP di Bacillus sp. PS3 per il legame ATP reversibile (Imamura et al., 2009; Kotera et al., 2010). Il legame ATP alla sottounità ε induce un cambiamento conformazionale nella struttura del sensore modificando l’orientamento relativo del donatore N e C-terminale e dei fluorofori accettanti (proteina fluorescente monomerica super-enhanced cyan (mseCFP); Venere monomerica permuta circolare (cp173-mVenus), una variante della proteina fluorescente gialla), aumentando l’efficienza FRET. Entrambe le classi di sensori hanno fornito informazioni sulla dinamica subcellulare dell’ATP negli animali (Ando et al., 2012; Tarasov et al., 2012; Li et al., 2015; Merrins et al., 2016). Tuttavia, ad oggi, esiste un solo rapporto sul loro uso nelle piante (Hatsugai et al., 2012), e l’istituzione di un affidabile monitoraggio ATP basato sulla fluorescenza nelle piante è stato carente. Questo nonostante il ruolo preminente dell’ATP nella rete fisiologica delle piante, compresi due organelli produttori di ATP e le condizioni ambientali spesso fluttuanti che determinano lo sviluppo del loro piano corporeo flessibile.
In questo lavoro ci siamo prefissati di stabilire protocolli per il rilevamento ATP negli impianti. In primo luogo, generiamo linee di Arabidopsis che esprimono il biosensore ATeam1.03-nD/nA nel citosol, nella matrice mitocondriale o nello stroma plastidio, e dimostriamo che le piante che ospitano la sonda nel citosol o nei plastidi sono stabili e non mostrano alcun cambiamento fenotipico. Al contrario, le linee che esprimono i sensori mitocondriali sono nane, ma ancora vitali. In secondo luogo, convalidiamo le caratteristiche biochimiche del sensore ATeam 1.03-nD/nA in vitro in condizioni tipiche dei sistemi vegetali. In terzo luogo, sviluppiamo un test ex situ per i mitocondri isolati per sondare il trasporto e la sintesi dell’ATP. In quarto luogo, mappiamo le differenze tissutali e gradienti delle concentrazioni citosoliche MgATP2- di MgATP2- nelle piantine viventi, compresa la variazione da cellula a cellula nei peli delle radici che è inversamente correlata con il tasso di crescita. Infine, dimostriamo come l’ipossia progressiva porta a cambiamenti caratteristici dipendenti dal tempo nella dinamica di MgATP2-.
Risultati
Generazione di linee di Arabidopsis per il rilevamento di ATP nel citosol, cloroplasti e mitocondri
Per stabilire le misure ATP nelle piante viventi, abbiamo generato linee di Arabidopsis che esprimono ATeam1.03-nD/nA nel citosol, nello stroma cloroplastico e nella matrice mitocondriale. Abbiamo selezionato almeno tre linee indipendenti per ogni compartimento in base all’espressione del fluoroforo, due delle quali sono state propagate all’omozigosi. Nonostante l’espressione sia stata guidata da un promotore del CaMV 35S, non abbiamo osservato un forte silenziamento del sensore nelle generazioni successive, contrariamente a quanto osservato frequentemente per altri sensori (Pei et al., 2000; Deuschle et al., 2006; Chaudhuri et al., 2008; Yang et al., 2010; Jones et al., 2014; Behera et al., 2015; Loro et al., 2016; Schwarzländer et al., 2016). La fluorescenza nel citoplasma periferico, così come nei filamenti trans-vacuolari e nel nucleoplasma, ha dimostrato l’espressione citosolica, mentre la co-localizzazione con l’auto-fluorescenza clorofilliana ha confermato l’espressione cloroplastica (Figura 1A). Inoltre, tutte le linee indipendenti per l’espressione citosolica o plastidica hanno mostrato un fenotipo wild-type-like a livello dell’intera pianta (Figura 1B), che è stato convalidato da una dettagliata fenotipizzazione che quantifica la lunghezza delle radici, la dimensione della rosetta, l’altezza dell’infiorescenza e il numero di silique (Figura 1-figure supplements 2 e 3). Al contrario, i trasformanti per l’espressione mitocondriale hanno mostrato una fluorescenza costantemente più debole e un forte fenotipo di sviluppo (Figura 1A,B; Figura 1-figure supplement 2). La fluorescenza del sensore co-localizzato con il marcatore di matrice mitocondriale MitoTracker, ma l’organizzazione degli organuli etichettati è stato perturbato, suggerendo anomalo un certo grado di anomalia mitocondriale. Ciononostante, i trasformanti fiorirono dopo 14 settimane e fecondarono il seme, permettendo la loro propagazione. Nonostante queste osservazioni, abbiamo registrato la fluorescenza di Venere e PCP in piantine di cinque giorni (Figura 1-figure supplement 1) e abbiamo trovato rapporti nettamente inferiori di Venere / PCP nei mitocondri, mentre il citosol e cloroplasti erano simili (Figura 1C; Figura 1-figure supplement 1).10.7554/eLife.26770.002Figure 1.ATeam espressione 1.ATeam in linee stabili di Arabidopsis.ATeam1.03-nD/nA è stata espressa sotto il controllo di un promotore 35S come proteina non fusa per la localizzazione nel citosol, come fusione con il peptide bersaglio transketolasi (TkTp) per il targeting plastidico o fusa alla Nicotiana plumbaginifolia β-ATPase per il targeting mitocondriale. (A) Piantine di cinque giorni, cresciute verticalmente sulla metà della forza MS +1% (w / v) piastre di saccarosio medio sono stati utilizzati per CLSM. Venere (verde) fluorescenza è stato registrato accanto alla fluorescenza clorofilla in cellule di cotelydon o il mitocondriale MitoTracker Orange marcatore nelle cellule dell’ipocotile. L’immagine di fusione mostra entrambi i canali di fluorescenza proiettati sulla rispettiva immagine del campo luminoso. (B) Sono stati confrontati fenotipi di più linee indipendenti per costruzione e un’immagine rappresentativa viene mostrata dopo la crescita per cinque settimane sul terreno. (C) Per un’analisi raziometrica, la fluorescenza di Venere e PCP è stata valutata in cellule ipocotiliche di piantine di cinque giorni, cresciute come in (A) con potenza del laser 458 nm impostato al 10% (citosolico e plastidico) e 30% (mitocondriale) di massima potenza. Le regioni di interesse (ROI) di dimensioni simili, indicate da linee tratteggiate, sono state definite per calcolare il rapporto Venus/CFP mostrato nel grafico. n = 36 (citosolo/plastide) o 105 (mitocondri) ROI in 12 (citosolo/plastide) o 22 (mitocondri) immagini da 4 (citosolo/plastide) o 6 (mitocondri) singole piante; barre di errore = SD. ns: p>0,05, ***p≤0,001 (test t). Barra di scala (tutti i pannelli) = 10 µm. DOI:http://dx.doi.org/10.7554/eLife.26770.00210.7554/eLife.26770.003Figure 1-figure supplement 1.Ratiometric immagine 1.Ratiometric di ATeam in compartimenti cellulari di piantine di Arabidopsis.5-day-old piantine cresciute verticalmente su mezza forza MS +1% (w / v) saccarosio sono stati ripresi da CLSM. La fluorescenza di Venere (verde) e PCP (magenta) è stata valutata in cellule ipocotiliche utilizzando le stesse impostazioni, ad eccezione della potenza del laser a 458 nm, che è stata impostata al 10% della potenza massima per la linea nucleare-citosolica e plastidica e al 30% per la linea mitocondriale. Barra di scala = 10 µm. L’analisi raziometrica delle immagini è mostrata nella Figura 1C. DOI:http://dx.doi.org/10.7554/eLife.26770.00310.7554/eLife.26770.004Figure 1-figure supplement 2.Whole pianta fenotipizzazione fenotipizzazione di linee omozigote ATeam. (A) Piante omozigote esprimendo ATeam nel citosol (cyt), plastidi (cp) o mitocondri (mt) sono stati cresciuti verticalmente per 5 giorni su solidificato metà resistenza MS medio fianco a fianco con wild-type Col-0. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. La lunghezza delle radici primarie di 35 piante di ATeam e 35 piante di Col-0 per linea di sensore indipendente è stata quantificata 5 giorni dopo la stratificazione e la differenza statistica da Col-0 è stata testata con un ANOVA unidirezionale seguito dal test di Dunnett. ns: p>0,05, ***p≤0,001; barre di errore = SD. (B) 20 (Col-0, cyt, cp) e 12 (mt) piante selezionate a caso mostrate in (A) sono state trasferite nel terreno e coltivate in camere di crescita a lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita per ogni linea del sensore sono state tracciate contro lo stesso set di piante di Col-0 e le differenze statistiche sono state valutate per i singoli punti temporali separatamente con un ANOVA a senso unico, seguito dal test di Dunnett. *p≤0,05, ***p≤0,001; barre di errore = SD. (C) L’altezza dell’infiorescenza primaria delle piante di 46-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 61-d sono state contate manualmente. n = 20; barre di errore = SD; ns: p>0.05 (ANOVA unidirezionale con test Dunnett per confrontare le linee del sensore con Col-0). Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 61 e quindi non sono state incluse nell’analisi. DOI:http://dx.doi.org/10.7554/eLife.26770.00410.7554/eLife.26770.005Figure 1-figure supplement 3.Whole plant phenotyping di linee di ATeam eterozigote. (A) Le piante che esprimono ATeam eterozigote nel citosol (cyt) o plastidi (cp) sono stati cresciuti verticalmente per 5 giorni su mezzo di MS solidificato a mezza resistenza. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Gli individui fluorescenti sono contrassegnati da un asterisco rosso. La lunghezza della radice primaria delle piante fluorescenti e non fluorescenti in un rapporto 3:1 è stata quantificata 4 giorni dopo la stratificazione e la differenza statistica è stata testata con un ANOVA unidirezionale seguito dal test Tukey. ns: p>0,05; barre di errore = SD. (B) 25 piante fluorescenti e 15 piante non fluorescenti per linea indipendente sono state selezionate in modo casuale, trasferite al suolo e coltivate in camere di crescita di lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita degli individui fluorescenti e non fluorescenti sono state tracciate per ogni linea e non sono state rilevate differenze statistiche in qualsiasi momento con un ANOVA a senso unico, seguito dal test Tukey. (C) L’altezza dell’infiorescenza primaria delle piante di 43-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 56-d sono state contate manualmente. ns: p>0,05 (ANOVA unidirezionale con il test di Tukey); barre di errore = SD. Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 56 e quindi non sono state incluse nell’analisi. DOI:http://dx.doi.org/10.7554/eLife.26770.00510.7554/eLife.26770.006Figure supplemento a 1 cifra 4.Phenotyping di Arabidopsis che esprime le proteine del sensore mitocondriale delle piante che esprimono omozigotemente ATeam mitocondriale, YC3.6 e roGFP2-Grx1 (n = 5 per ogni linea indipendente) costitutivamente sotto un promotore CaMV 35S sono stati cresciuti verticalmente per 5 giorni su un mezzo MS solidificato a mezza resistenza, trasferiti al suolo e cresciuti in condizioni di lunga giornata. La dimensione totale della rosetta è stata quantificata 35 giorni dopo la stratificazione e confrontata con lo stesso set di piante wild-type Col-0 come mostrato in Figura 1-figure supplement 2 (n = 20). Tre piante rappresentative di ciascuna linea sono state selezionate per la fotografia inferiore mostrata 46 giorni dopo la stratificazione, che è stata inoltre utilizzata per quantificare l’area della rosetta (grafico inferiore). La differenza statistica rispetto a Col-0 è stata testata con un ANOVA a senso unico seguito dal test di Dunnett. **p≤0,01, ***p≤0,001; barre di errore = SD. Barra di scala = 5 cm. DOI:http://dx.doi.org/10.7554/eLife.26770.006
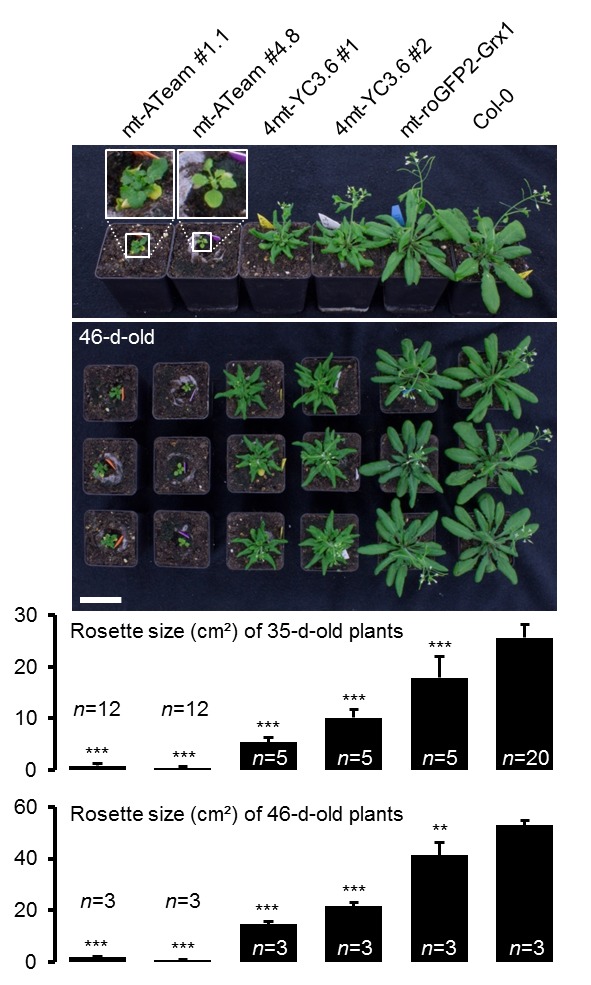
Figura 1-figure supplement 4.Figura 1. Espressione di ATeam in linee stabili di Arabidopsis.immagine raziometrica di ATeam in compartimenti cellulari di piantine di Arabidopsis.fenotipizzazione di linee omozigote di ATeam.fenotipizzazione di linee eterozigote di ATeam.fenotipizzazione di linee eterozigote di ATeam.fenotipizzazione di Arabidopsis che esprimono proteine mitocondriali del sensore.ATeam1.03-nD/nA è stato espresso sotto il controllo di un promotore 35S come proteina non fusa per la localizzazione nel citosol, come fusione con il peptide bersaglio transketolasi (TkTp) per il targeting plastid o fusa al plumbaginifolia Nicotiana β-ATPase per il targeting mitocondriale. (A) Piantine di cinque giorni, cresciute verticalmente sulla metà della forza MS +1% (w / v) piastre di saccarosio medio sono stati utilizzati per CLSM. Venere (verde) fluorescenza è stato registrato accanto alla fluorescenza clorofilla in cellule di cotelydon o il mitocondriale MitoTracker Orange marcatore nelle cellule dell’ipocotile. L’immagine di fusione mostra entrambi i canali di fluorescenza proiettati sulla rispettiva immagine del campo luminoso. (B) Sono stati confrontati fenotipi di più linee indipendenti per costruzione e un’immagine rappresentativa viene mostrata dopo la crescita per cinque settimane sul terreno. (C) Per un’analisi raziometrica, la fluorescenza di Venere e PCP è stata valutata in cellule ipocotiliche di piantine di cinque giorni, cresciute come in (A) con potenza del laser 458 nm impostato al 10% (citosolico e plastidico) e 30% (mitocondriale) di massima potenza. Le regioni di interesse (ROI) di dimensioni simili, indicate da linee tratteggiate, sono state definite per calcolare il rapporto Venus/CFP mostrato nel grafico. n = 36 (citosolo/plastide) o 105 (mitocondri) ROI in 12 (citosolo/plastide) o 22 (mitocondri) immagini da 4 (citosolo/plastide) o 6 (mitocondri) singole piante; barre di errore = SD. ns: p>0,05, ***p≤0,001 (test t). Barra di scala (tutti i pannelli) = 10 µm.DOI:
http://dx.doi.org/10.7554/eLife.26770.002Piantine di cinque giorni, cresciute verticalmente su MS a mezza forza +1% (w/v) di saccarosio, sono state riprese dal CLSM. La fluorescenza di Venere (verde) e PCP (magenta) è stata valutata in cellule ipocotiliche utilizzando le stesse impostazioni ad eccezione della potenza del laser a 458 nm, che è stato impostato al 10% della potenza massima per il nucleare-citosolico e plastidico e il 30% per la linea mitocondriale. Barra di scala = 10 µm. L’analisi raziometrica delle immagini è mostrata nella Figura 1C.DOI:
http://dx.doi.org/10.7554/eLife.26770.003(A) Le piante omozigotizzanti che esprimono ATeam nel citosol (cyt), plastidi (cp) o mitocondri (mt) sono state coltivate verticalmente per 5 giorni sul mezzo di MS solidificato a mezza resistenza fianco a fianco con il wild-type Col-0. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. La lunghezza delle radici primarie di 35 piante di ATeam e 35 piante di Col-0 per linea di sensore indipendente è stata quantificata 5 giorni dopo la stratificazione e la differenza statistica da Col-0 è stata testata con un ANOVA unidirezionale seguito dal test di Dunnett. ns: p>0,05, ***p≤0,001; barre di errore = SD. (B) 20 (Col-0, cyt, cp) e 12 (mt) piante selezionate a caso mostrate in (A) sono state trasferite nel terreno e coltivate in camere di crescita a lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita per ogni linea del sensore sono state tracciate contro lo stesso set di piante di Col-0 e le differenze statistiche sono state valutate per i singoli punti temporali separatamente con un ANOVA a senso unico, seguito dal test di Dunnett. *p≤0,05, ***p≤0,001; barre di errore = SD. (C) L’altezza dell’infiorescenza primaria delle piante di 46-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 61-d sono state contate manualmente. n = 20; barre di errore = SD; ns: p>0.05 (ANOVA unidirezionale con test Dunnett per confrontare le linee del sensore con Col-0). Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 61 e quindi non sono state incluse nell’analisi.DOI:
http://dx.doi.org/10.7554/eLife.26770.004(A) Le piante che esprimono eterozigotemente l’ATeam nel citosol (cyt) o nei plastidi (cp) sono state coltivate verticalmente per 5 giorni su substrato MS solidificato a mezza resistenza. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Gli individui fluorescenti sono contrassegnati da un asterisco rosso. La lunghezza della radice primaria delle piante fluorescenti e non fluorescenti in un rapporto 3:1 è stata quantificata 4 giorni dopo la stratificazione e la differenza statistica è stata testata con un ANOVA unidirezionale seguito dal test Tukey. ns: p>0,05; barre di errore = SD. (B) 25 piante fluorescenti e 15 piante non fluorescenti per linea indipendente sono state selezionate in modo casuale, trasferite al suolo e coltivate in camere di crescita di lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita degli individui fluorescenti e non fluorescenti sono state tracciate per ogni linea e non sono state rilevate differenze statistiche in qualsiasi momento con un ANOVA a senso unico, seguito dal test Tukey. (C) L’altezza dell’infiorescenza primaria delle piante di 43-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 56-d sono state contate manualmente. ns: p>0,05 (ANOVA unidirezionale con il test di Tukey); barre di errore = SD. Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 56 e quindi non sono state incluse nell’analisi.DOI:
http://dx.doi.org/10.7554/eLife.26770.005Le piante omozigotizzanti che esprimono omozigoti ATeam mitocondriale, YC3.6 e roGFP2-Grx1 (n = 5 per ogni linea indipendente) costitutivamente sotto un promotore CaMV 35S sono state coltivate in verticale per 5 giorni su un mezzo MS solidificato a mezza resistenza, trasferite al suolo e cresciute in condizioni di lunga giornata. La dimensione totale della rosetta è stata quantificata 35 giorni dopo la stratificazione e confrontata con lo stesso set di piante wild-type Col-0 come mostrato in Figura 1-figure supplement 2 (n = 20). Tre piante rappresentative di ciascuna linea sono state selezionate per la fotografia inferiore mostrata 46 giorni dopo la stratificazione, che è stata inoltre utilizzata per quantificare l’area della rosetta (grafico inferiore). La differenza statistica rispetto a Col-0 è stata testata con un ANOVA a senso unico seguito dal test di Dunnett. **p≤0,01, ***p≤0,001; barre di errore = SD. Barra di scala = 5 cm.DOI:
http://dx.doi.org/10.7554/eLife.26770.006
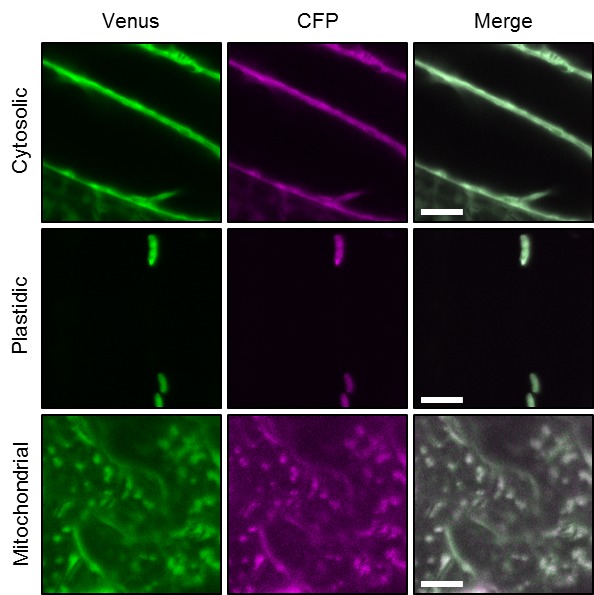
Figura 1-figure supplement 1.Immagini raziometriche di ATeam in compartimenti cellulari di piantine di Arabidopsis.Piantine di cinque giorni, cresciute verticalmente su mezza forza MS +1% (w / v) saccarosio sono stati ripresi dal CLSM. La fluorescenza di Venere (verde) e PCP (magenta) è stata valutata in cellule ipocotiliche utilizzando le stesse impostazioni, ad eccezione della potenza del laser a 458 nm, che è stato impostato al 10% della potenza massima per il nucleare-citosolico e plastidico e il 30% per la linea mitocondriale. Barra di scala = 10 µm. L’analisi raziometrica delle immagini è mostrata nella Figura 1C.DOI:
http://dx.doi.org/10.7554/eLife.26770.003
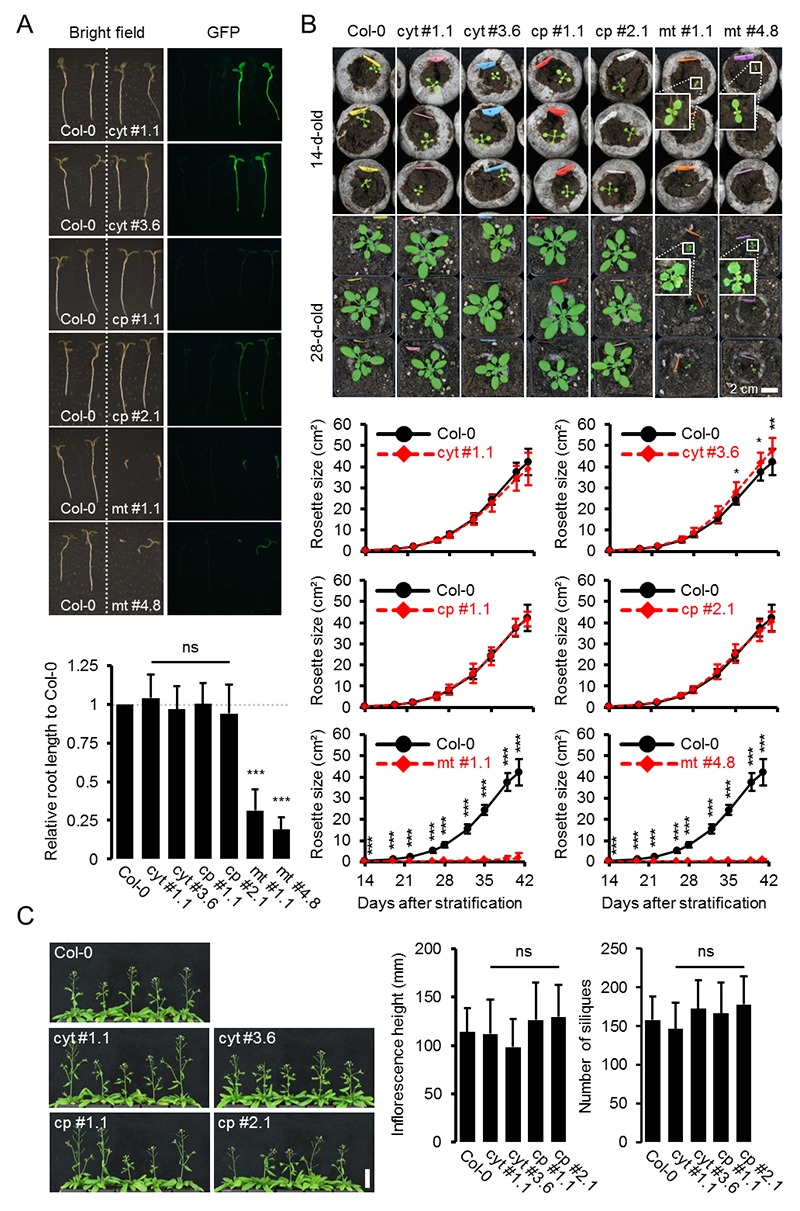
Figura 1-figure supplemento 2.Figura 1—supplemento figura 2. Fenotipizzazione dell’intero impianto di linee omozigote ATeam.(A) Piante omozigoti che esprimono ATeam omozigote nel citosol (cyt), plastidi (cp) o mitocondri (mt) sono stati cresciuti verticalmente per 5 giorni su solidificato metà resistenza MS medio fianco a fianco con wild-type Col-0. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. La lunghezza delle radici primarie di 35 piante di ATeam e 35 piante di Col-0 per linea di sensore indipendente è stata quantificata 5 giorni dopo la stratificazione e la differenza statistica da Col-0 è stata testata con un ANOVA unidirezionale seguito dal test di Dunnett. ns: p>0,05, ***p≤0,001; barre di errore = SD. (B) 20 (Col-0, cyt, cp) e 12 (mt) piante selezionate a caso mostrate in (A) sono state trasferite nel terreno e coltivate in camere di crescita a lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita per ogni linea del sensore sono state tracciate contro lo stesso set di piante di Col-0 e le differenze statistiche sono state valutate per i singoli punti temporali separatamente con un ANOVA a senso unico, seguito dal test di Dunnett. *p≤0,05, ***p≤0,001; barre di errore = SD. (C) L’altezza dell’infiorescenza primaria delle piante di 46-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 61-d sono state contate manualmente. n = 20; barre di errore = SD; ns: p>0.05 (ANOVA unidirezionale con test Dunnett per confrontare le linee del sensore con Col-0). Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 61 e quindi non sono state incluse nell’analisi.DOI:
http://dx.doi.org/10.7554/eLife.26770.004
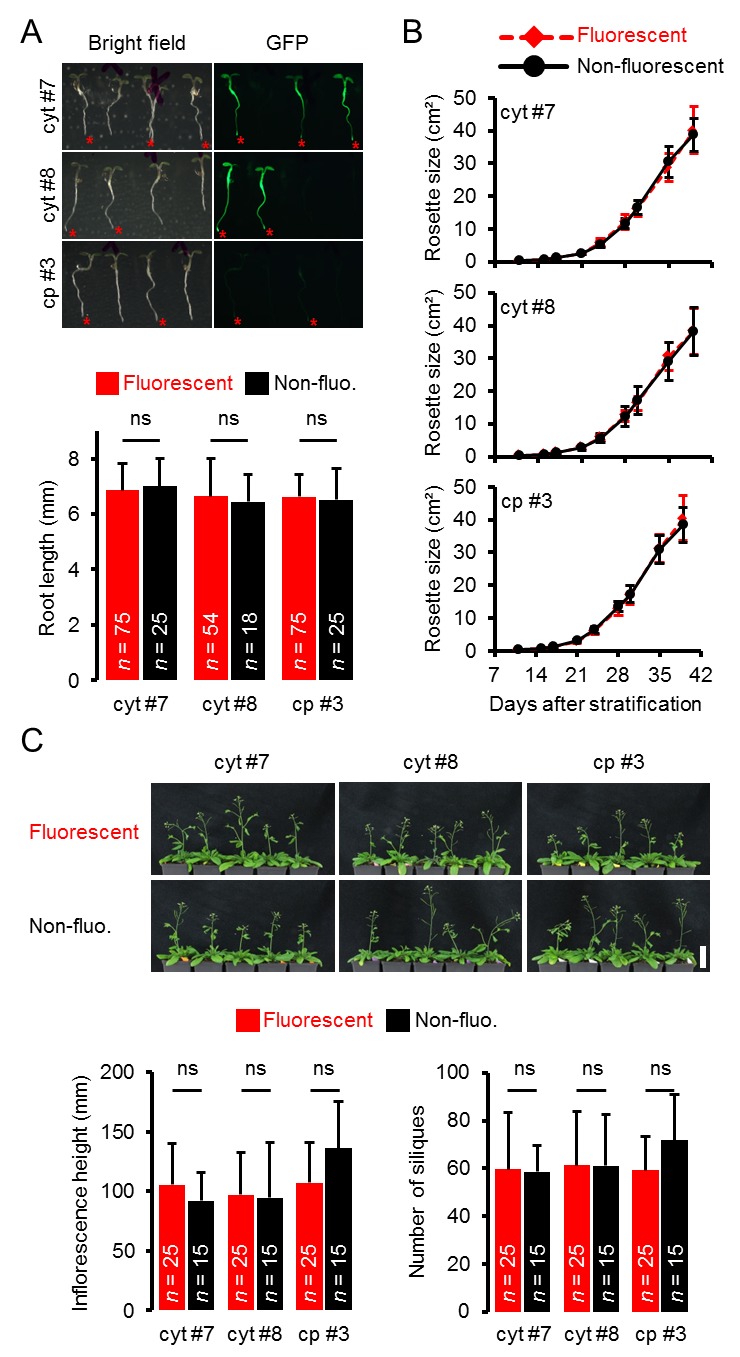
Figura 1-figure supplement 3.Fenotipizzazione di piante intere di linee di ATeam eterozigoti.(A) Le piante che esprimono eterozigotemente l’ATeam nel citosol (cyt) o nei plastidi (cp) sono state coltivate verticalmente per 5 giorni su substrato MS solidificato a mezza resistenza. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Gli individui fluorescenti sono contrassegnati da un asterisco rosso. La lunghezza della radice primaria delle piante fluorescenti e non fluorescenti in un rapporto 3:1 è stata quantificata 4 giorni dopo la stratificazione e la differenza statistica è stata testata con un ANOVA unidirezionale seguito dal test Tukey. ns: p>0,05; barre di errore = SD. (B) 25 piante fluorescenti e 15 piante non fluorescenti per linea indipendente sono state selezionate in modo casuale, trasferite al suolo e coltivate in camere di crescita di lunga durata. Lo sviluppo della rosetta è stato documentato fotograficamente e l’area della rosetta fogliare è stata analizzata fino a quando la maggior parte delle piante ha sviluppato i primi fiori aperti. Le curve di crescita degli individui fluorescenti e non fluorescenti sono state tracciate per ogni linea e non sono state rilevate differenze statistiche in qualsiasi momento con un ANOVA a senso unico, seguito dal test Tukey. (C) L’altezza dell’infiorescenza primaria delle piante di 43-d vecchie piante è stata catturata fotograficamente e quantificata. Silique di piante vecchie di 56-d sono state contate manualmente. ns: p>0,05 (ANOVA unidirezionale con il test di Tukey); barre di errore = SD. Barra di scala = 50 mm. Le piante che esprimono l’ATeam mitocondriale non sono state bullonate fino al giorno 56 e quindi non sono state incluse nell’analisi.DOI:
http://dx.doi.org/10.7554/eLife.26770.005

Figura 1-figure supplement 4.Fenotipizzazione dell’Arabidopsis che esprime le proteine dei sensori mitocondriali.Le piante omozigotizzanti che esprimono ATeam mitocondriale, YC3.6 e roGFP2-Grx1 (n = 5 per ogni linea indipendente) costitutivamente sotto un promotore CaMV 35S sono state coltivate in verticale per 5 giorni su un mezzo MS solidificato a mezza resistenza, trasferite al suolo e cresciute in condizioni di lunga giornata. La dimensione totale della rosetta è stata quantificata 35 giorni dopo la stratificazione e confrontata con lo stesso set di piante wild-type Col-0 come mostrato in Figura 1-figure supplement 2 (n = 20). Tre piante rappresentative di ciascuna linea sono state selezionate per la fotografia inferiore mostrata 46 giorni dopo la stratificazione, che è stata inoltre utilizzata per quantificare l’area della rosetta (grafico inferiore). La differenza statistica rispetto a Col-0 è stata testata con un ANOVA a senso unico seguito dal test di Dunnett. **p≤0,01, ***p≤0,001; barre di errore = SD. Barra di scala = 5 cm.DOI:
http://dx.doi.org/10.7554/eLife.26770.006
Caratterizzazione in vitro della proteina del sensore purificato che rivela la specificità per MgATP2-
Per interpretare le misure in vivo nelle linee del sensore Arabidopsis, abbiamo mirato ad una comprensione approfondita delle caratteristiche chiave del sensore. Il Kd(ATP), la specificità dei nucleotidi e la sensibilità del pH delle varianti originali della famiglia ATeam sono stati caratterizzati per l’uso in cellule animali a 37°C (Imamura et al., 2009). Mentre la più recente variante ATeam1.03-nD/nA mostra un migliore range dinamico (Kotera et al., 2010), le altre sue proprietà non sono state caratterizzate in dettaglio, in particolare alle condizioni di pH e temperatura che si possono sperimentare nel citosol vegetale, nella matrice mitocondriale o nello stroma plastidico. Abbiamo quindi caratterizzato purificato ATeam1.03-nD/nA proteina purificata in vitro (Figura 2A). Lo spettro di emissione del sensore ha mostrato uno spostamento raziometrico ben definito in risposta a ATP. Il picco mseCFP (475 nm) è diminuito e il picco cp173-mVenus (527 nm) è aumentato, con l’aumento delle concentrazioni di ATP nel campo micromolare alto / basso millimetrico (Figura 2B). Il punto isosbico era a 512 nm. La proteina purificata era stabile a -86°C e manteneva lo stesso range dinamico del sensore appena purificato. Tuttavia, la conservazione non congelata ha causato un declino della reattività del sensore nel tempo, derivante dalla degradazione del sensore, probabilmente per proteolisi, spiegando la diminuzione del range dinamico per separazione dei due cromofori (Figura 2-figure supplement 1A). Quindi, aliquote di proteine purificate sono state congelate immediatamente dopo la purificazione e conservate a -86°C. La risposta ATP del sensore, determinata a 25 ° C, è stata sigmoidale con un range dinamico spettroscopico di 4,0, superiore a quello precedentemente riportato a 3,2 (Kotera et al., 2010). Il Kd (ATP) era 0,74 mM con un coefficiente di Hill di 1,02, compatibile con un singolo sito di legame ATP nella sintasi ATP sintasi ε-subunità (Yagi et al., 2007) (Figura 2C). Il sensore non ha mostrato alcuna risposta all’ADP e all’AMP, in accordo con i precedenti rapporti che utilizzavano altri nucleotidi, tra cui GTP (Imamura et al., 2009), e conferma la selettività per l’ATP. Mg2+ titolazione sotto ATP saturando ha mostrato una forte dipendenza Mg2+, indicando che il sensore risponde selettivamente a MgATP2-, e non ad ATP4- (Figura 2D,E). In assenza di ATP, i segnali del sensore erano stabili da pH 6,5 a 8,5 (Figura 2F), in linea con l’assenza di effetti diretti del pH sui fluorofori. L’insensibilità al pH è stata osservata anche a ATP parzialmente saturi e saturi da pH 7,5 a 8,5, anche se il range di risposta del sensore spettroscopico è stato diminuito al di sotto di pH 7,0. Questa diminuzione del rapporto è stata dovuta ad una risposta FRET in buona fede (segnale del donatore mseCFP in aumento, cp173-mVenus acceptor segnale decrescente; Figura 2-figure supplement 1B), e correlato con la diminuzione della specie MgATP2- come il pH è stato abbassato. Si deduce che la risposta del sensore riporta la concentrazione di MgATP2-, che a sua volta dipende dalla concentrazione ambiente di Mg2+ e dal pH (O’Sullivan e Perrin, 1961; Storer e Cornish-Bowden, 1976; Adolfsen e Moudrianakis, 1978). Poiché MgATP2- è la specie che agisce come cofattore per la grande maggioranza delle proteine ATP-dipendenti, il sensore fornisce una lettura della proporzione fisiologicamente significativa del pool di ATP che è visibile per quelle proteine. Il MgATP2- La risposta del sensore sarà indicata come risposta ATP per semplicità nelle sezioni di questo lavoro.10.7554/eLife.26770.007Cifra 2.Caratteristiche di ATeam1.03-nD/nA purificato.(A) Una proteina fluorescente ciano (CFP, PDB: 2WSN) e una variante della proteina fluorescente giallo (Venere, PDB: 3EKJ) sono stati collegati manualmente dalla ε-subunità di Bacillus subtilis ATP sintasi nel MgATP2–bound (PDB: 2E5Y) e MgATP2-libero (PDB: 4XD7) stato per generare un ipotetico modello strutturale di ATeam. L’efficienza FRET in assenza o presenza di MgATP2- è indicata da una freccia grigia. (B) Spettri di emissione di ATeam normalizzati (eccitazione a 435 ± 5 nm) in presenza di concentrazioni di ATP crescenti e di un eccesso in Mg2+ di 2 mM. (C) ATeam è stato eccitato a 435 ± 5 nm e il rapporto di emissione a 527 nm (cp173-Venus) e 475 nm (mseCFP) a 25°C in presenza di nucleotidi di adenina. La funzione di Boltzmann è stata utilizzata per adattare i dati MgATP2–binding. (D) Struttura di MgATP2-. La sua stabilità dipende dal pH e dalla concentrazione di Mg2+ libero. (E) ATeam Venere / PCP rapporti in 4 mM ATP (punti neri) e 0 mM ATP (punti grigi) titolato con concentrazioni crescenti di MgCl2. Il triangolo grigio mostra il rapporto Venus/CFP in presenza di 4 mM ATP e 20 mM NaCl. (F) ATeam rapporti Venus/CFP a pH diverso e in presenza di 0 (grigio chiaro), 0,5 (grigio scuro) e 4 mM (nero) MgATP. I dati in (B), (C) e (F) sono mediati da quattro repliche tecniche e le barre di errore sono rappresentate come SD, ma troppo piccole per essere visualizzate. DOI:http://dx.doi.org/10.7554/eLife.26770.00710.7554/eLife.26770.008Figure Supplemento a 2 cifre 1.Caratteristiche di ATeam1.03-nD/nA.(A) ATeam appena purificato è stato conservato per un giorno (1d) o una settimana (1w) a temperature comprese tra -86°C e 37°C. Il range dinamico (DR) della proteina immagazzinata rappresenta il rapporto di Venus/CFP a zero e ATP saturante (4 mM in presenza di Mg2+ in eccesso) ed è stato normalizzato alla proteina appena purificata. n (repliche tecniche) = 4; barre di errore = SD. La degradazione delle proteine in questi campioni è stata analizzata da SDS-PAGE. La massa molecolare attesa di 6×His-ATeam1.03-nD/nA intatto è di 70,1 kDa. (B) Intensità di emissione di fluorescenza (eccitazione a 435 ± 5 nm) delle singole proteine fluorescenti mseCFP e cp173-Venus a pH variabile e 0 o 4 (saturazione) mM ATP. I dati sono mediati da quattro repliche tecniche e le barre di errore rappresentano SD ma sono troppo piccole per essere visualizzate. DOI:http://dx.doi.org/10.7554/eLife.26770.008

Figura 2-figure supplement 1.Caratteristiche di ATeam1.03-nD/nA.Caratteristiche di ATeam1.03-nD/nA purificato.(A) Una proteina fluorescente ciano (CFP, PDB: 2WSN) e una variante della proteina fluorescente giallo (Venere, PDB: 3EKJ) sono stati collegati manualmente dalla ε-subunità di Bacillus subtilis ATP sintasi nel MgATP2–bound (PDB: 2E5Y) e MgATP2-libero (PDB: 4XD7) stato per generare un ipotetico modello strutturale di ATeam. L’efficienza FRET in assenza o presenza di MgATP2- è indicata da una freccia grigia. (B) Spettri di emissione di ATeam normalizzati (eccitazione a 435 ± 5 nm) in presenza di concentrazioni di ATP crescenti e di un eccesso in Mg2+ di 2 mM. (C) ATeam è stato eccitato a 435 ± 5 nm e il rapporto di emissione a 527 nm (cp173-Venus) e 475 nm (mseCFP) a 25°C in presenza di nucleotidi di adenina. La funzione di Boltzmann è stata utilizzata per adattare i dati MgATP2–binding. (D) Struttura di MgATP2-. La sua stabilità dipende dal pH e dalla concentrazione di Mg2+ libero. (E) ATeam Venere / PCP rapporti in 4 mM ATP (punti neri) e 0 mM ATP (punti grigi) titolato con concentrazioni crescenti di MgCl2. Il triangolo grigio mostra il rapporto Venus/CFP in presenza di 4 mM ATP e 20 mM NaCl. (F) ATeam rapporti Venus/CFP a pH diverso e in presenza di 0 (grigio chiaro), 0,5 (grigio scuro) e 4 mM (nero) MgATP. I dati in (B), (C) e (F) sono mediati da quattro repliche tecniche e le barre di errore sono rappresentate come SD, ma troppo piccole per essere visualizzate.DOI:
http://dx.doi.org/10.7554/eLife.26770.007(A) L’ATeam appena purificata è stata conservata per un giorno (1d) o una settimana (1w) a temperature comprese tra -86°C e 37°C. Il range dinamico (DR) della proteina immagazzinata rappresenta il rapporto di Venere / PCP a zero e ATP saturazione (4 mM in presenza di Mg2 + in eccesso) ed è stato normalizzato alla proteina appena purificata. n (repliche tecniche) = 4; barre di errore = SD. La degradazione delle proteine in questi campioni è stata analizzata da SDS-PAGE. La massa molecolare attesa di 6×His-ATeam1.03-nD/nA intatto è di 70,1 kDa. (B) Intensità di emissione di fluorescenza (eccitazione a 435 ± 5 nm) delle singole proteine fluorescenti mseCFP e cp173-Venus a pH variabile e 0 o 4 (saturazione) mM ATP. I dati sono mediati da quattro repliche tecniche e le barre di errore rappresentano SD ma sono troppo piccole per essere visualizzate.DOI:
http://dx.doi.org/10.7554/eLife.26770.008

Figura 2-figure supplement 1.Caratteristiche di ATeam1.03-nD/nA purificato.(A) L’ATeam appena purificato è stato conservato per un giorno (1d) o una settimana (1w) a temperature comprese tra -86°C e 37°C. Il range dinamico (DR) della proteina immagazzinata rappresenta il rapporto di Venus/CFP a zero e ATP saturante (4 mM in presenza di Mg2+ in eccesso) ed è stato normalizzato alla proteina appena purificata. n (repliche tecniche) = 4; barre di errore = SD. La degradazione delle proteine in questi campioni è stata analizzata da SDS-PAGE. La massa molecolare attesa di 6×His-ATeam1.03-nD/nA intatto è di 70,1 kDa. (B) Intensità di emissione di fluorescenza (eccitazione a 435 ± 5 nm) delle singole proteine fluorescenti mseCFP e cp173-Venus a pH variabile e 0 o 4 (saturazione) mM ATP. I dati sono mediati da quattro repliche tecniche e le barre di errore rappresentano SD ma sono troppo piccole per essere visualizzate.DOI:
http://dx.doi.org/10.7554/eLife.26770.008
Stabilire un test ex situ per monitorare le dinamiche ATP mitocondriali
Le dinamiche ATP in vivo nella cella sono regolate dalla produzione e dal consumo. Sul lato della produzione, i mitocondri esportano l’ATP al citosol (e il nucleo a sua volta per diffusione), facilitato dalla fosforilazione ossidativa sulla superficie della matrice della membrana mitocondriale interna e dall’estrusione di ATP a gradiente di membrana da parte di un sistema di trasporto ADP/ATP molto attivo (AAC) (Haferkamp et al., 2011; Gout et al., 2014). Abbiamo ipotizzato che l’integrazione dei mitocondri purificati e funzionali con la proteina esterna del sensore permetterebbe di monitorare i flussi di trasporto dell’ATP in entrata e in uscita dai mitocondri, e anche di sezionare il ruolo dell’adenilato chinasi (AK), che si pensa sia localizzato nello spazio intermembrana (Figura 3A). Un tale sistema eliminerebbe l’influenza di altri processi cellulari che hanno un impatto sulla concentrazione di MgATP2- nel citosol, tra cui l’idrolisi ATP, il trasporto attraverso le membrane di altri compartimenti cellulari, così come le variazioni di pH e Mg2 + concentrazioni.10.7554/eLife.26770.009Figure 3.ATP flussi di flusso in isolato mitocondri Arabidopsis. (A) mitocondri puri intatti isolati da due settimane piantine di Arabidopsis sono stati mescolati con terreno di incubazione di base e purificato ATeam in 96 pozzetti microtiter piastre. I mitocondri sono stati alimentati con succinato come substrato respiratorio per alimentare il ciclo dell’acido tricarbossilico (TCA) e la catena di trasporto degli elettroni (ETC). ATP, generato chimicamente da ATP sintasi (complesso V) nella matrice, viene scambiato con ADP attraverso il vettore ADP / AAC, mentre ATP generato da adenilato chinasi (AK) nello spazio intermembrana non richiede il trasporto attraverso la membrana mitocondriale interna. L’ATP extracondriale esportato dai mitocondri viene rilevato da ATeam nel mezzo come MgATP2-. (B, C) ATeam è stato utilizzato come mostrato in (A) e 10 mM succinato è stato aggiunto prima (B) o dopo (C) ADP a concentrazioni tra 0 e 4 mM. ATP è stato aggiunto a 4 mM come riferimento al punto temporale indicato (tracce rosse). n (repliche tecniche) = 4; barre di errore = SD. I pannelli inferiori mostrano i saggi polarografici del consumo di ossigeno eseguiti con un elettrodo di tipo Clark in parallelo. I mitocondri, il succinato e l’ADP sono stati aggiunti al mezzo di incubazione di base come indicato. Una traccia rappresentativa di un singolo esperimento è mostrato e tassi di consumo di ossigeno (nmol min-1 mg-1 proteina) per ogni stato respiratorio sono indicati come media ± SD da tre repliche tecniche. DOI:http://dx.doi.org/10.7554/eLife.26770.009
Abbiamo integrato i mitocondri delle piantine di Arabidopsis appena isolate con la proteina ricombinante purificata del sensore ATeam, utilizzando lo stesso mezzo utilizzato per la caratterizzazione del sensore in vitro (Figura 3B). Il terreno conteneva Mg2+ e Pi in eccesso, ed è stato impostato a pH 7,5 per assomigliare al pH citosolico (Ratcliffe, 1997; Schulte et al., 2006). L’esatta concentrazione del sensore non è critica, perché la lettura raziometrica del FRET è auto-normalizzante, anche se sono state evitate concentrazioni molto basse o molto alte del sensore, per evitare un basso segnale-rumore, o ATP-buffering da parte del sensore stesso quando è vicino al suo Kd (ATP).
Il rapporto FRET non è cambiato con l’aggiunta di succinato come substrato respiratorio in assenza di ADP, ma la successiva aggiunta di ADP ha portato ad un aumento del FRET che si è stabilizzato ad un valore di stato stazionario a seconda della concentrazione di ADP (Figura 3B). Saggi polarografici sul consumo di ossigeno eseguiti in parallelo hanno confermato le risposte dell’attività respiratoria e l’accoppiamento dei mitocondri con un coefficiente di controllo respiratorio di circa 2 (Figura 3B), e hanno confermato che il sensore ha risposto rapidamente all’ATP generato dai mitocondri respiratori e scambiato dall’AAC. Tuttavia, l’aggiunta di ADP prima del succinato ha rivelato che l’ADP da solo era sufficiente a far sì che il sensore rispondesse in modo dose-dipendente, anche se con piccoli aumenti di FRET (Figura 3C). Considerando che il sensore non risponde all’ADP (vedi Figura 2C), la risposta è indicativa della produzione di ATP in assenza di respirazione attiva. I rapporti FRET sono aumentati ulteriormente dopo la successiva aggiunta di succinato, raggiungendo un valore di plateau simile a prima. La produzione di ATP da solo ADP in assenza di un substrato respiratorio esclude l’attività della sintasi ATP, ma sarebbe coerente con l’attività AK che porta alla conversione di ADP in ATP e AMP (Busch e Ninnemann, 1996). Ciò implica che il saggio fornisce una lettura integrata delle attività combinate di ATP sintasi, AK e AAC.
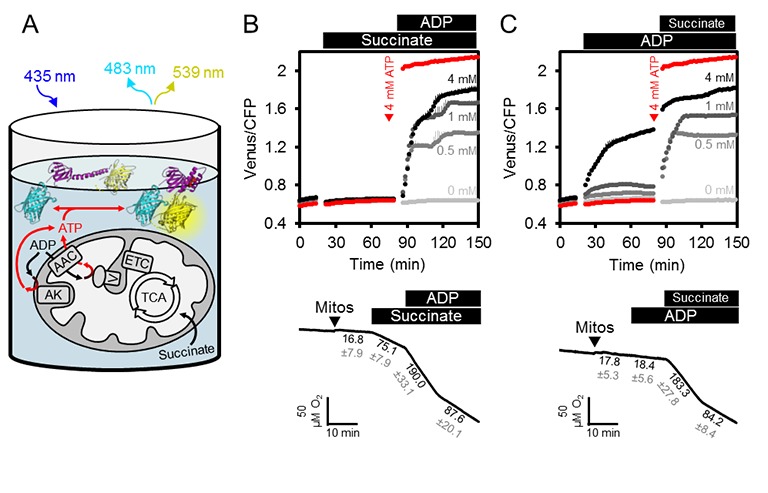
Figura 3.Flussi di ATP in mitocondri isolati Arabidopsis.(A) mitocondri puri e intatti isolati da due settimane di piantine di Arabidopsis sono stati mescolati con terreno di incubazione di base e purificato ATeam in piastre di microtitolazione a 96 pozzetti. I mitocondri sono stati alimentati con succinato come substrato respiratorio per alimentare il ciclo dell’acido tricarbossilico (TCA) e la catena di trasporto degli elettroni (ETC). ATP, generato chimicamente da ATP sintasi (complesso V) nella matrice, viene scambiato con ADP attraverso il vettore ADP / AAC, mentre ATP generato da adenilato chinasi (AK) nello spazio intermembrana non richiede il trasporto attraverso la membrana mitocondriale interna. L’ATP extracondriale esportato dai mitocondri viene rilevato da ATeam nel mezzo come MgATP2-. (B, C) ATeam è stato utilizzato come mostrato in (A) e 10 mM succinato è stato aggiunto prima (B) o dopo (C) ADP a concentrazioni tra 0 e 4 mM. ATP è stato aggiunto a 4 mM come riferimento al punto temporale indicato (tracce rosse). n (repliche tecniche) = 4; barre di errore = SD. I pannelli inferiori mostrano i saggi polarografici del consumo di ossigeno eseguiti con un elettrodo di tipo Clark in parallelo. I mitocondri, il succinato e l’ADP sono stati aggiunti al mezzo di incubazione di base come indicato. Una traccia rappresentativa di un singolo esperimento è mostrato e tassi di consumo di ossigeno (nmol min-1 mg-1 proteina) per ogni stato respiratorio sono indicati come media ± SD da tre repliche tecniche.DOI:
http://dx.doi.org/10.7554/eLife.26770.009
Dissezione della produzione di ATP mitocondriale, scambio ATP/ADP e attività adenilato chinasi
Il saggio mitocondriale permette di indagare il ruolo di AK e AAC nel controllo delle dinamiche mitocondriali ATP. Per verificare se l’AK è coinvolto nella produzione di ATP in assenza di trasporto di elettroni respiratori, abbiamo previsto che la produzione di ATP sarebbe stata sensibile alla presenza di AMP guidata dall’azione di massa, in una reazione completamente reversibile catalizzata dall’AK (Figura 4A). Coerentemente con questa visione, l’aggiunta di AMP a seguito dell’aumento del rapporto FRET innescato dall’ADP, ha portato ad una graduale diminuzione del FRET dipendente dalla dose (Figura 4B). Allo stesso modo, la presenza di AMP prima dell’aggiunta di ADP, ha inibito la generazione di ATP indotta da ADP (Figura 4C).10.7554/eLife.26770.010Figure 4.Modulating the ATP production in isolato Arabidopsis mitocondri.(A) Reazione catalizzata da adenilato chinasi (AK). (B, C) ATeam è stato utilizzato in una configurazione come mostrato in Figura 3A. Mitocondri isolati e purificati ATeam sono stati mescolati con AMP tra 0 (grigio chiaro) e 32 (nero) mM dopo (B) o prima (C) l’aggiunta di 4 mM ADP e l’ATeam Venere / PCP rapporto è stato registrato. n (repliche tecniche) = 4. (D) Rappresentazione della catena di trasporto degli elettroni mitocondriali (complessi I-IV) che genera una forza motrice protonica (Δp) utilizzata dalla sintasi ATP (complesso V) per produrre ATP. Il vettore ADP/ATP (AAC) trasporta ATP dalla matrice mitocondriale allo spazio intermembrana (IMS) in cambio di ADP. Sono indicati i trattamenti che riducono la produzione o il trasporto di ATP mitocondriale e il loro sito di azione. (E) I mitocondri non trattati (simboli neri) o mitocondri trattati con 10 µM cAT (simboli rossi) sono stati alimentati con ADP a 0,5 mM (cerchi) o 4 mM (triangoli) seguiti da 10 mM succinato. (F) L’effetto inibitorio dei trattamenti riassunti in (F) sulla produzione di ATP mitocondriale è stato calcolato attraverso l’aumento Venus / CFP dopo l’aggiunta di ADP (grigio), ADP e succinato (nero) o ADP e succinato sotto inibizione (rosso). cAT, carboxyatractyloside; CCCP, cianuro di carbonile m-chlorophenyl hydrazone; KCN, cianuro di potassio. E e F mostrano la media di tre repliche tecniche; barre di errore = SD. DOI:http://dx.doi.org/10.7554/eLife.26770.010
Per testare il ruolo di AAC-mediata ADP / ADP / ATP scambio attraverso la membrana interna (Figura 4D), abbiamo aggiunto carboxyatractyloside (cAT) per bloccare l’AAC (Figura 4E). L’aggiunta di 4 mM ADP in assenza di attività respiratoria ha portato ad un simile aumento FRET sia nel controllo e mitocondri trattati con cAT, suggerendo che AK-derivato ATP non si basava su AAC-mediata ADP o trasporto ATP. L’energizzazione successiva da succinato ha portato ad un leggero ma riproducibile ulteriore aumento del controllo, che era assente per i mitocondri trattati con cAT. Questa differenza mostra che la sintasi ATP esposta a matrice non può contribuire in presenza di cAT e convalida l’effettiva inibizione AAC. A concentrazioni più basse di ADP (0,5 mM), quando la produzione di ATP da AK è vicino al limite di rilevazione (vedi Figura 3C), succinato ha dato un aumento di MgATP2 rilevabile in assenza di cAT, ma questo è stato quasi completamente abolito in presenza di cAT (Figura 4E). Ciò è coerente con la localizzazione di AK al di fuori della membrana mitocondriale interna e supporta la localizzazione nello spazio intermembrana mitocondriale, come riportato in precedenza per altre specie vegetali (Day et al., 1979; Birkenhead et al., 1982; Stitt et al., 1982; Roberts et al., 1997; Zancani et al., 2001), ma contrasta la situazione nei mammiferi dove le isoforme AK3 sono presenti anche nella matrice (Schulz, 1987).
Sfruttando la sensibilità differenziale di AK e il sistema di sintasi AAC/ATP ad ADP, è possibile ridurre al minimo il contributo di AK alla produzione di ATP, e utilizzare basse concentrazioni di ADP (0,5 mM) per monitorare selettivamente l’ATP prodotto da sintasi ATP e l’attività AAC (Figura 4C). La sintesi di ATP dipende quindi strettamente dall’importazione di ADP, dalla funzionalità della sintasi ATP, dalla forza motrice dei protoni e dal trasporto funzionale degli elettroni (Figura 4F). Il trattamento con inibitori specifici che coprono questi quattro livelli funzionali ha impedito costantemente l’aumento di FRET in risposta all’energizzazione mitocondriale da succinato (Figura 4D-F), convalidando il saggio come mezzo per monitorare i cambiamenti funzionali in fasi specifiche dei percorsi bioenergetici.
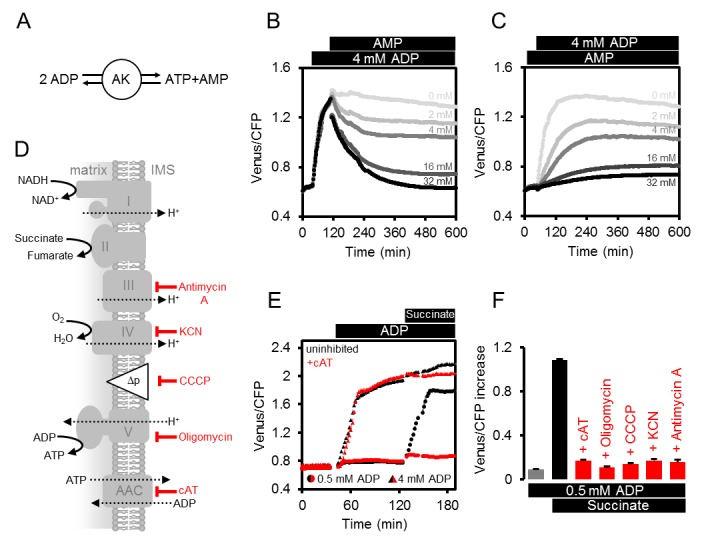
Figura 4.Figura 4. Modulazione della produzione di ATP in mitocondri isolati di Arabidopsis.(A) Reazione catalizzata da adenilato chinasi (AK). (B, C) ATeam è stato utilizzato in un setup come mostrato in Figura 3A. Mitocondri isolati e purificati ATeam sono stati mescolati con AMP tra 0 (grigio chiaro) e 32 (nero) mM dopo (B) o prima (C) l’aggiunta di 4 mM ADP e l’ATeam Venere / PCP rapporto è stato registrato. n (repliche tecniche) = 4. (D) Rappresentazione della catena di trasporto degli elettroni mitocondriali (complessi I-IV) che genera una forza motrice protonica (Δp) utilizzata dalla sintasi ATP (complesso V) per produrre ATP. Il vettore ADP/ATP (AAC) trasporta ATP dalla matrice mitocondriale allo spazio intermembrana (IMS) in cambio di ADP. Sono indicati i trattamenti che riducono la produzione o il trasporto di ATP mitocondriale e il loro sito di azione. (E) I mitocondri non trattati (simboli neri) o mitocondri trattati con 10 µM cAT (simboli rossi) sono stati alimentati con ADP a 0,5 mM (cerchi) o 4 mM (triangoli) seguiti da 10 mM succinato. (F) L’effetto inibitorio dei trattamenti riassunti in (F) sulla produzione di ATP mitocondriale è stato calcolato attraverso l’aumento Venus / CFP dopo l’aggiunta di ADP (grigio), ADP e succinato (nero) o ADP e succinato sotto inibizione (rosso). cAT, carboxyatractyloside; CCCP, cianuro di carbonile m-chlorophenyl hydrazone; KCN, cianuro di potassio. E e F mostrano la media di tre repliche tecniche; barre di errore = SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.010
Misure ad alta produttività di MgATP2- in piantagione mediante fluorimetria
Per consentire misurazioni ad alto rendimento dei livelli di MgATP2- livelli nelle piantagioni, abbiamo ottimizzato una configurazione fluorimetrica a piastra microtiter per i tessuti vegetali e le piantine intere. L’analisi raziometrica rende le misurazioni indipendenti dal livello di espressione del sensore, così come la quantità e la forma del tessuto, a condizione che ci sia sufficiente segnale-rumore e poca interferenza da auto-fluorescenza del tessuto. Gli spettri di emissione sono stati registrati con eccitazione a 435 nm da piantine di Arabidopsis intatte di sette giorni (Sweetlove et al., 2007), e dischi di foglie di piante di quattro settimane che esprimono ATeam citosolico (Figura 5A,B). Entrambi i tipi di campione hanno mostrato spettri di fluorescenza che erano praticamente identici con la proteina purificata del sensore, mentre l’auto-fluorescenza era bassa al confronto. La fluorescenza della clorofilla è stata efficacemente separata e non ha causato alcuna interferenza significativa. Ciò è stato confermato indipendentemente anche per il sensore cloroplastico da microscopia confocale a livello di cloroplasti individuali (Figura 5-figure supplemento 2).10.7554/eLife.26770.011Figure 5.Fluorimetry setup per monitorare MgATP2- dinamiche in piantagione. (A, B) Sette giorni piantine di Arabidopsis (due per pozzo, A) o dischi di foglie di singole piante di quattro settimane (B) sono stati immersi in mezzo di imaging su 96 pozzetti microtiter piastre. La fluorescenza del materiale vegetale che esprime stabilmente il citosolico ATeam è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP e confrontato con il wild-type Col-0. Gli spettri di emissione in fluorescenza tra 460 e 600 nm sono stati registrati utilizzando un lettore di piastre e una lunghezza d’onda di eccitazione di 435 ± 10 nm. n = 5; barre di errore = SD. (C-E) Per ogni pozzetto, due piantine di Arabidopsis di sette giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono state eccitate a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. Sono stati aggiunti CCCP (100 µM), antimicina A (100 µM) o adenina (10 mM) dove indicato (punti dati neri) mentre gli impianti di controllo sono stati lasciati non trattati (punti dati grigi). Le emissioni nei pozzi con piante di Col-0 sono state mediate e sottratte da quelle delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 3-4 pozzi e le barre di errore sono SD. Gli inserti mostrano l’intensità di emissione di fluorescenza (I) di Venere e PCP nei singoli pozzi rappresentativi. DOI:http://dx.doi.org/10.7554/eLife.26770.01110.7554/eLife.26770.012Figure 5-figure supplement 1.Fluorometric letture 1.Fluorometric di MgATP2- dynamics in Arabidopsis semenzali.Independent ripetizione di esperimenti mostrati in Figura 5C-E. Per bene, due semenzali di sette giorni di Arabidopsis che non esprimono alcun sensore (Col-0) o citosolico ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. Sono stati aggiunti CCCP (100 µM), antimicina A (50 µM) o adenina (10 mM) dove indicato (punti dati neri) mentre gli impianti di controllo sono stati lasciati non trattati (punti dati grigi). Le emissioni nei pozzi con piante di Col-0 sono state mediate e sottratte da quelle delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 3-4 pozzi e le barre di errore sono SD. DOI:http://dx.doi.org/10.7554/eLife.26770.01210.7554/eLife.26770.013Figure Supplemento a 5 cifre 2.Impatto della fluorescenza della clorofilla sul segnale di ATeam.Ipocotile di piantine di Arabidopsis vecchie di cinque giorni che esprimono ATeam-TkTp plastidico immaginato dal CLSM. Segnale Venere, verde; fluorescenza clorofilliana, magenta; segnale CFP, bianco; rapporto Venus/CFP, scala raziometrica a falsi colori. Sono state definite le regioni di interesse per i centri di cloroplasti in cui era presente il segnale della clorofilla (cerchio nero) e le stromole in cui non è stato rilevato alcun segnale di clorofilla (cerchio rosso). Il grafico a barre mostra i rapporti Venus/CFP per i centri di cloroplasti (nero) e stromoli (rosso). n = 11 plastidi di tre singole piante; barre di errore = SD. ns: p>0,5 (t test). Barra di scala = 2 µm. DOI:http://dx.doi.org/10.7554/eLife.26770.01310.7554/eLife.26770.014Figure 5-figure supplement 3.The effect of CCCP and antimycin A treatments on the ATeam response at the different medium pH.Due piantine di Arabidopsis di sette giorni per pozzo, che non esprimono alcun sensore (Col-0) o citosolico ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata nel mezzo di imaging a valori di pH compresi tra 6,0 (rosso) e 8,5 (blu). DMSO come controllo (A), 100 µM CCCP (B) o 100 µM antimicina A (C) sono stati aggiunti ai punti temporali indicati. L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione di fondo sono la media di tre pozzi. DOI:http://dx.doi.org/10.7554/eLife.26770.014
Per valutare la velocità e la gamma della risposta del sensore in vivo, abbiamo monitorato il rapporto FRET di piantine in piastre microtiter nel tempo in presenza e assenza di carbonile cianuro m-clorofenilidrazone (CCCP), antimicina A, e adenina, per modificare le concentrazioni di MgATP2- endogeno. CCCP dissipa i gradienti protonici sulle membrane cellulari, inibendo la produzione di ATP e aumentando il consumo di ATP; l’antimicina A inibisce il trasporto degli elettroni mitocondriali al complesso III (Figura 4D), mentre l’adenina è stato dimostrato che porta ad un aumento della concentrazione di MgATP2- cellulare agendo come substrato per la sintesi di ATP (Loef et al., 2001; Gout et al., 2014). Ci siamo concentrati sul citosol come spazio di integrazione per i flussi di ATP da e verso altre sedi subcellulari, dove la concentrazione di MgATP2- stazionaria è impostata dall’interazione di numerosi processi di sintesi, idrolisi e trasporto.
CCCP e antimicina A hanno entrambi innescato una rapida e pronunciata diminuzione del FRET, mentre l’adenina ha portato ad un modesto, ma riproducibile aumento (Figura 5C,D,E; Figura 5-figure supplement 1). Variando il pH medio tra 6,0 e 8,5 ha mostrato che la diminuzione del FRET dopo il trattamento con antimicina A era indipendente dal pH. Anche dopo l’aggiunta di CCCP, solo una piccola frazione della risposta era dovuta all’acidificazione citosolica, e la destabilizzazione di MgATP2- a sua volta (Figura 5-figure supplement 3). Il range massimo di risposta spettroscopica è stato di circa 3, leggermente inferiore al range in vitro, che è parzialmente rappresentato dalla lunghezza d’onda di eccitazione e dalla larghezza di banda di emissione utilizzata. Si deduce che il sensore era intatto e funzionale in vivo. Alto, ma non completamente saturo FRET, allo stato stazionario ha permesso una stima delle concentrazioni citosoliche di MgATP2- MgATP2- nell’intervallo di circa 2 mM, in media sui tessuti delle piantine. Ciò è coerente con le stime precedenti e con i valori da manuale (Taiz et al., 2015), ma superiore a quanto riportato in altri casi (Gotta et al., 2014).
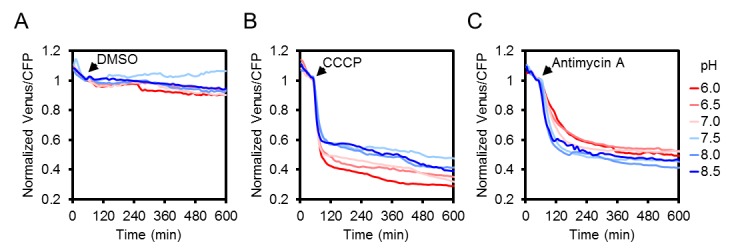
Figura 5-figure supplement 3.Figura 5— supplemento alla figura 3. Impostazione della fluorimetria per monitorare la dinamica di MgATP2 nelle piantagioni. Impatto della fluorescenza della clorofilla sul segnale di ATeam, l’effetto dei trattamenti CCCP e antimicina A sulla risposta di ATeam a diversi pH medi.(A,B) Piantine di Arabidopsis di sette giorni (due per pozzo; A) o dischi di foglie di singole piante di quattro settimane (B) sono state immerse nel mezzo di imaging su piastre microtiter a 96 pozzetti. La fluorescenza del materiale vegetale che esprime stabilmente il citosolico ATeam è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP e confrontato con il wild-type Col-0. Gli spettri di emissione in fluorescenza tra 460 e 600 nm sono stati registrati utilizzando un lettore di piastre e una lunghezza d’onda di eccitazione di 435 ± 10 nm. n = 5; barre di errore = SD. (C-E) Per ogni pozzetto, due piantine di Arabidopsis di sette giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono state eccitate a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. Sono stati aggiunti CCCP (100 µM), antimicina A (100 µM) o adenina (10 mM) dove indicato (punti dati neri) mentre gli impianti di controllo sono stati lasciati non trattati (punti dati grigi). Le emissioni nei pozzi con piante di Col-0 sono state mediate e sottratte da quelle delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 3-4 pozzi e le barre di errore sono SD. Gli inserti mostrano l’intensità di emissione di fluorescenza (I) di Venere e PCP nei singoli pozzi rappresentativi.DOI:
http://dx.doi.org/10.7554/eLife.26770.011Ripetizione indipendente degli esperimenti mostrati in Figura 5C-E. Per bene, due piantine di Arabidopsis di sette giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. Sono stati aggiunti CCCP (100 µM), antimicina A (50 µM) o adenina (10 mM) dove indicato (punti dati neri) mentre gli impianti di controllo sono stati lasciati non trattati (punti dati grigi). Le emissioni nei pozzi con piante di Col-0 sono state mediate e sottratte da quelle delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 3-4 pozzi e le barre di errore sono SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.012Ipotile di piantine di Arabidopsis di cinque giorni che esprimono l’ATeam-TkTp plastidico immaginato dal CLSM. Segnale Venere, verde; fluorescenza clorofilliana, magenta; segnale CFP, bianco; rapporto Venere/PCP, scala raziometrica a falsi colori. Sono state definite le regioni di interesse per i centri di cloroplasti in cui era presente il segnale della clorofilla (cerchio nero) e le stromole in cui non è stato rilevato alcun segnale di clorofilla (cerchio rosso). Il grafico a barre mostra i rapporti Venus/CFP per i centri di cloroplasti (nero) e stromoli (rosso). n = 11 plastidi di tre singole piante; barre di errore = SD. ns: p>0,5 (t test). Barra di scala = 2 µm.DOI:
http://dx.doi.org/10.7554/eLife.26770.013Due piantine di Arabidopsis di sette giorni per pozzo, che non esprimono alcun sensore (Col-0) o ATeam citosolico, sono state eccitate a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata nel mezzo di imaging a valori di pH compresi tra 6,0 (rosso) e 8,5 (blu). DMSO come controllo (A), 100 µM CCCP (B) o 100 µM antimicina A (C) sono stati aggiunti ai punti temporali indicati. L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione di fondo sono la media di tre pozzi.DOI:
http://dx.doi.org/10.7554/eLife.26770.014
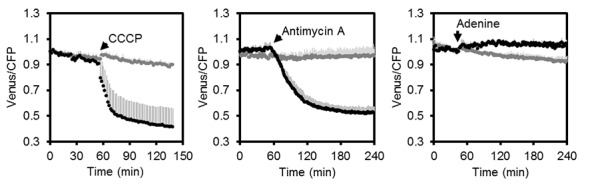
Figura 5-figure supplement 1.Letture fluorometriche di MgATP2- dinamica in piantine di Arabidopsis.2. Ripetizione indipendente degli esperimenti mostrati in Figura 5C-E. Per bene, due piantine di Arabidopsis di sette giorni che non esprimono alcun sensore (Col-0) o citosolico ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. Sono stati aggiunti CCCP (100 µM), antimicina A (50 µM) o adenina (10 mM) dove indicato (punti dati neri) mentre gli impianti di controllo sono stati lasciati non trattati (punti dati grigi). Le emissioni nei pozzi con piante di Col-0 sono state mediate e sottratte da quelle delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 3-4 pozzi e le barre di errore sono SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.012
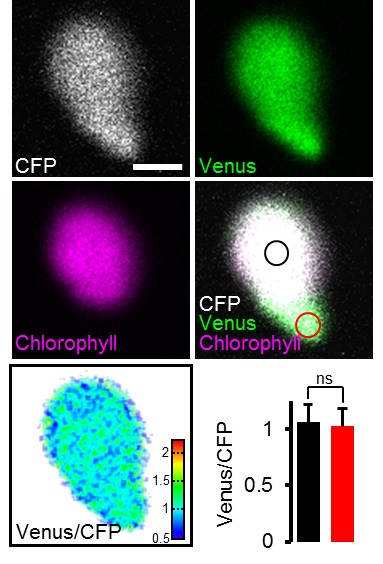
Figura 5-figure supplement 2.Impatto della fluorescenza della clorofilla sul segnale di ATeam.Ipotile di piantine di Arabidopsis di cinque giorni che esprimono l’ATeam-TkTp plastidico ripreso dal CLSM. Segnale Venere, verde; fluorescenza clorofilliana, magenta; segnale CFP, bianco; rapporto Venus/CFP, scala raziometrica a falsi colori. Sono state definite le regioni di interesse per i centri di cloroplasti in cui era presente il segnale della clorofilla (cerchio nero) e le stromole in cui non è stato rilevato alcun segnale di clorofilla (cerchio rosso). Il grafico a barre mostra i rapporti Venus/CFP per i centri di cloroplasti (nero) e stromoli (rosso). n = 11 plastidi di tre singole piante; barre di errore = SD. ns: p>0,5 (t test). Barra di scala = 2 µm.DOI:
http://dx.doi.org/10.7554/eLife.26770.013

Figura 5-figure supplement 3.L’effetto dei trattamenti con CCCP e antimicina A sulla risposta di ATeam a diversi pH medi.Due semenzali di sette giorni di Arabidopsis per pozzo, esprimendo nessun sensore (Col-0) o citosolico ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato in mezzo di imaging a valori di pH tra 6,0 (rosso) e 8,5 (blu). DMSO come controllo (A), 100 µM CCCP (B) o 100 µM antimicina A (C) sono stati aggiunti ai punti temporali indicati. L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione di fondo sono la media di tre pozzi.DOI:
http://dx.doi.org/10.7554/eLife.26770.014
Una mappa MgATP2- mappa della piantina di Arabidopsis
L’eterogeneità e i gradienti tra tessuti e cellule sono stati di grande interesse nella segnalazione e nello sviluppo degli ormoni vegetali, e recentemente sono stati introdotti sensori fluorescenti per l’acido ascissico e l’auxina (Brunoud et al., 2012; Wend et al., 2013; Jones et al., 2014; Waadt et al., 2014). Analoghe intuizioni sono largamente carenti per i metaboliti e i cofattori, nonostante il fatto che il metabolismo sia alla base dello sviluppo (Sweetlove et al., 2017) e che i fattori abiotici, come l’ipossia, agiscano come segnali (Considine et al., 2017). Per misurare le differenze di potenziale nelle concentrazioni di MgATP2- citosolico tra tessuti e cellule in vivo, abbiamo eseguito un’analisi al microscopio confocale di piantine di Arabidopsis intatte di cinque giorni (Figura 6A). Le piantine sono state tenute al buio per 30 minuti prima dell’acquisizione dell’immagine per evitare potenziali effetti della fotosintesi attiva. Una panoramica di FRET attraverso le piantine ha rivelato grandi differenze tissutali. I cotiledoni hanno mostrato valori elevati, che erano più bassi nell’ipocotile e sono scesi bruscamente alla transizione germoglio-radice. La radice ha mostrato valori bassi che poi sono aumentati nella punta della radice. Le differenze relative sono state osservate in modo riproducibile tra le singole piantine, e coerenti in linee di sensore indipendenti (# 1,1, # 3,6; Figura 6B; Figura 6-figure supplement 1). L’area intorno all’apice del germoglio ha mostrato bassi rapporti, paragonabili a quelli della radice, in molti ma non in tutti gli individui. Al contrario, le differenze tra i tessuti erano meno pronunciate nelle piantine eziolate di cinque giorni in condizioni identiche (Figura 6C,D), con un FRET relativamente più basso nei germogli e un FRET più alto nelle radici. Il ripido gradiente alla transizione germoglio-radice era anche assente, indicando che la foto-morfogenesi è necessaria per la sua formazione.10.7554/eLife.26770.015Figure 6.A MgATP2- mappa della piantina di Arabidopsis. (A) Per mappare il MgATP2- livelli nei tessuti di piantine di Arabidopsis, tre giorni (piantina intera) o di cinque giorni (primi piani) piante che esprimono ATeam citosolico sono stati analizzati da CLSM. È stata registrata la fluorescenza di Venere (verde) e CFP (magenta) e il rapporto è stato tracciato come immagine a falsi colori dove alti valori di Venere/CFP (rosso) corrispondono ad alti livelli di MgATP2-. Nei primi piani, i rapporti Venus/PCP sono stati analizzati nelle regioni di interesse indicate. Barra di scala = 500 µm. (B) I grafici rappresentano i dati di due linee indipendenti. n (per linea) = 6; barre di errore = SD. I dati di entrambe le linee sono stati raggruppati per un’analisi statistica con un ANOVA unidirezionale seguito dal test Tukey (p≤0,05) e lettere diverse indicano differenze significative. L’esperimento è stato ripetuto due volte con risultati coerenti. (C, D) analisi CLSM di piantine di cinque giorni, sia coltivate alla luce o eziolate al buio. SRT, shoot-root transition. n (piantine per condizione) = 11; barre di errore = SD. ns: p>0.05, ***p≤0.001 (ANOVA bidirezionale seguito dal test Tukey). DOI:http://dx.doi.org/10.7554/eLife.26770.01510.7554/eLife.26770.016Figure supplemento a 6 cifre 1.A MgATP2- mappa della piantina di Arabidopsis.ripetizione indipendente degli esperimenti mostrati in Figura 6A,B. (A) Le piantine di arabidopsis di cinque giorni che esprimono l’ATeam citosolico (linea #1.1) sono state analizzate dal CLSM. È stata registrata la fluorescenza di Venere (verde) e CFP (magenta), e il rapporto è tracciato come un’immagine a falsi colori dove alti valori di Venere / CFP (rosso) corrispondono ad alti livelli di ATP. I rapporti Venus/PCP sono stati analizzati nelle regioni di interesse indicate. Barra di scala = 200 µm. (B) I grafici rappresentano i dati di 24 singoli impianti; barre di errore = SD. Lettere diverse indicano differenze significative in un ANOVA unidirezionale seguito dal test Tukey (p≤0,05). DOI:http://dx.doi.org/10.7554/eLife.26770.01610.7554/eLife.26770.017Figure Supplemento a 6 cifre 2.L’effetto del CCCP sulla risposta di ATeam in diversi tessuti di piantine di Arabidopsis.(A) Piantine di Arabidopsis di cinque giorni che esprimono ATeam citosolico sono state immerse in 100 µM CCCP a pH 7,5 e analizzate dal CLSM. Fluorescenza di Venere (verde) e CFP (magenta) è stato registrato e il rapporto è tracciato come un falso colore immagine dove alti valori di Venere / CFP (rosso) corrispondono ad alti livelli di MgATP2-. (B) I rapporti Venus/CFP in ogni punto temporale dopo il trattamento CCCP sono stati quantificati nei cotiledoni, nell’ipocotile e nella radice superiore. n = 3 singole piantine; barre di errore = SD. ns: p>0,05, ***p≤0,001 (ANOVA bidirezionale seguito dal test Tukey). (C) Le piantine di dieci giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono state tagliate con un bisturi per separare i germogli e le radici che sono state immerse individualmente nel mezzo di imaging su piastre microtiter a 96 pozzetti. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Spettri di emissione in fluorescenza tra 460 e 600 nm sono stati registrati utilizzando un lettore di piastre e una lunghezza d’onda di eccitazione di 435 ± 10 nm. I grafici mostrano gli spettri delle parti della pianta che esprimono l’ATeam citosolico dopo la sottrazione della fluorescenza Col-0 in due esperimenti indipendenti (Exp #1/2). n = 5 pozzi ciascuno; barre di errore = SD. (D) I germogli (raggruppati da tre piantine per pozzo) e le radici (raggruppati da 10 a 15 piantine per pozzo) sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. CCCP a 100 µM è stato aggiunto al punto temporale indicato e i dati sono stati tracciati dopo aver sottratto la fluorescenza delle parti di impianto di Col-0 che sono stati ugualmente trattati. n = 5 e 3 pozzetti per tipo di tessuto nell’esperimento indipendente Exp # 1 ed Exp # 2, rispettivamente; barre di errore = SD. DOI:http://dx.doi.org/10.7554/eLife.26770.017
Per verificare se i rapporti osservati potevano essere attribuiti a concentrazioni di MgATP2- piuttosto che ad artefatti ottici provenienti dalle diverse geometrie dei tessuti, o ad altre modifiche biochimiche dei sensori, il CCCP è stato utilizzato per esaurire MgATP2-. Inoltre, il pH del tampone medio è stato impostato a 7,5 per evitare la destabilizzazione di MgATP2- come risultato dell’acidificazione citosolica. In presenza di CCCP, i rapporti FRET sono diminuiti verso un valore minimo simile in tutti i tessuti e l’eterogeneità dei tessuti è stato gradualmente abolito (Figura 6-figure supplement 2A,B). Cinetica specifica del tessuto che potrebbe essere risolta dal CLSM può riflettere la capacità differenziale dei tessuti di mantenere le concentrazioni di MgATP2- o semplicemente la penetrazione differenziale da CCCP. Per convalidare in modo indipendente la differenza complessiva tra germoglio e radice, abbiamo utilizzato l’approccio fluorimetrico del pozzetto del microtitolo per misurare separatamente i campioni di germoglio e radice, come tentato in precedenza negli estratti di tessuto (Mustroph et al., 2006). Gli spettri completi indicavano differenze FRET bona fide, che sono state abolite dal trattamento CCCP (Figura 6-figure supplement 2C,D). È importante notare che le variazioni relative e normalizzate del FRET, ma non i valori assoluti del FRET, possono essere confrontati tra diverse configurazioni fluorimetriche/microscopiche a causa delle diverse configurazioni dei rivelatori di eccitazione e di emissione.
La conclusione che l’eterogeneità FRET in vivo ha registrato in modo affidabile l’eterogeneità MgATP2, ci ha spinto a risolvere ulteriormente le differenze a livello cellulare. Confrontando le cellule di pavimentazione e di guardia dell’epidermide abassiale di cotiledone da una regione di interesse (ROI) l’analisi non ha rivelato alcuna differenza nelle nostre mani (Figura 7A). Un precedente rapporto di foglie più vecchie e vere aveva mostrato concentrazioni più elevate di MgATP2- nelle cellule di guardia rispetto alle cellule di pavimentazione, suggerendo che le differenze possono essere indotte a seconda delle condizioni di sviluppo e ambientali (Hatsugai et al., 2012). L’analisi di sette strati cellulari della zona di transizione germoglio-radice ha rivelato un gradiente continuo, indicando che le cellule vicine possono mantenere diversi, ma stabili, gradienti di MgATP2- (Figura 7B). Alla punta della radice, le cellule del tappo ha mostrato un basso MgATP2-. Allo stesso modo, bassi valori di FRET sono stati osservati nei siti di ferimento (Figura 7C). Un hotspot di alti livelli di MgATP2- è stato localizzato nella columella appena sotto il centro quiescente (Figura 7D).10.7554/eLife.26770.018Figure 7.Local MgATP2- eterogeneità MgATP2- eterogeneità nelle cellule di piantine di Arabidopsis e tessuti.Fluorescenza di Venere (verde) e PCP (magenta) da ATeam citosolico è stato registrato dal CLSM in cinque giorni piantine di Arabidopsis. Il rapporto è tracciato come una falsa immagine a colori dove alti valori di Venere / PCP (rosso) corrispondono ad alti livelli di MgATP2-. (A) I rapporti Venus/CFP sono stati valutati in regioni di interesse indicato per confrontare le cellule di guardia e le cellule di pavimentazione dell’epidermide cotiledone. n = 24 coppie di marciapiede e cellula di guardia da cinque singole piante; barre di errore = SD. ns: p>0,05 (t test). Barra di scala = 20 µm. (B) I rapporti Venere / PCP sono stati valutati in strati cellulari indicati nella transizione germoglio-radice. L’analisi della regione di interesse degli strati cellulari successivi è indicata in scala di grigi. n = 11; barre di errore = SD. Lettere diverse indicano differenze statistiche in un ANOVA unidirezionale seguito dal test Tukey (p≤0,05). Barra di scala = 100 µm. (C) I cotiledoni sono stati feriti con un ago e i rapporti Venus/CFP sono stati valutati nelle regioni di interesse indicate che rappresentano tessuti feriti o non feriti. n = 5; barre di errore = SD, ***p≤0,001 (test t). Barra di scala = 500 µm. (D) Il rapporto Venus/CFP è stato valutato in due regioni di interesse alla radice. n = 12; barre di errore = SD, ***p≤0,001 (t test). Barra di scala = 100 µm. DOI:http://dx.doi.org/10.7554/eLife.26770.018
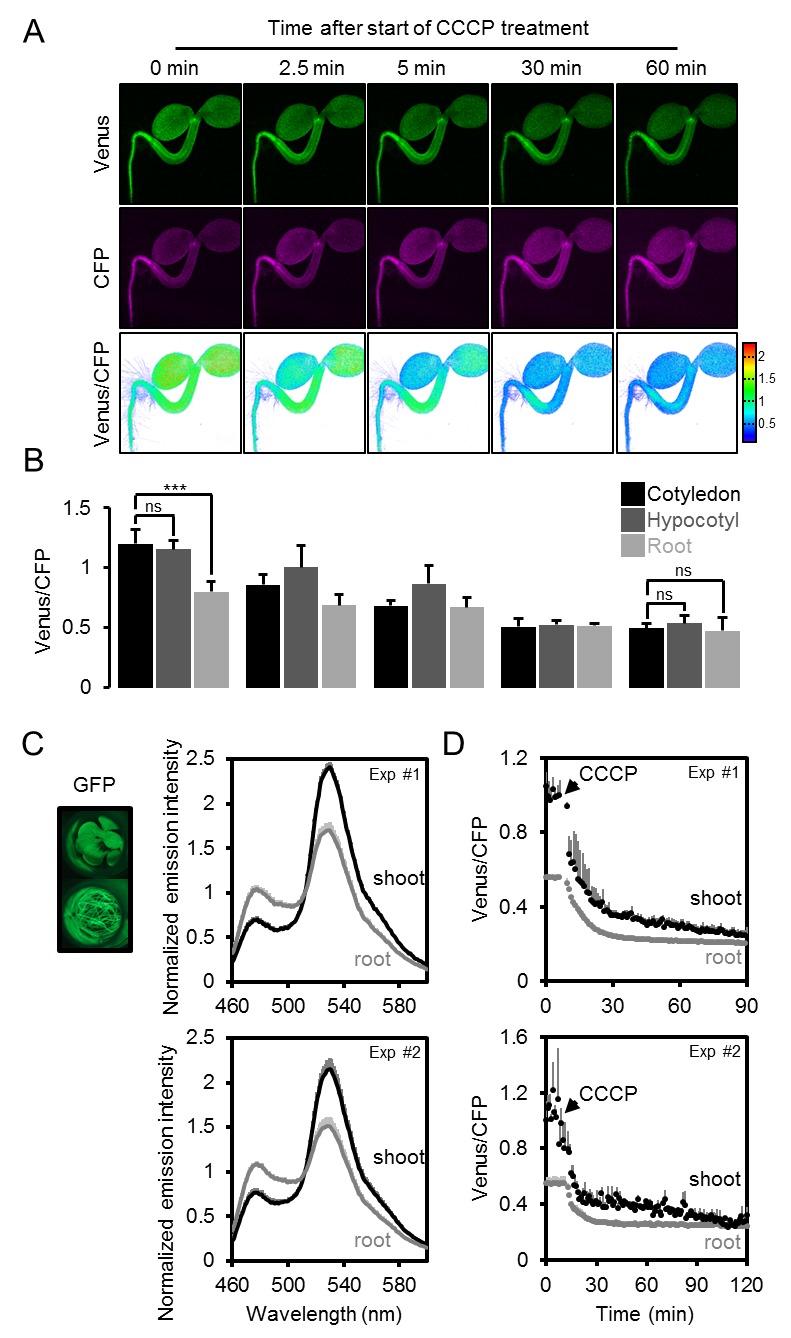
Figura 6-figure supplement 2.Una mappa MgATP2- mappa della piantina di Arabidopsis.A MgATP2- mappa della piantina di Arabidopsis.l’effetto del CCCP sulla risposta di ATeam in diversi tessuti di piantine di Arabidopsis.(A) Per mappare i livelli di MgATP2- nei tessuti di piantine di Arabidopsis, sono state analizzate dal CLSM le piante di tre giorni (piantine intere) o di cinque giorni (primi piani) che esprimono l’ATeam citosolico. È stata registrata la fluorescenza di Venere (verde) e CFP (magenta) e il rapporto è stato tracciato come immagine a falsi colori dove alti valori di Venere/CFP (rosso) corrispondono ad alti livelli di MgATP2-. Nei primi piani, i rapporti Venus/PCP sono stati analizzati nelle regioni di interesse indicate. Barra di scala = 500 µm. (B) I grafici rappresentano i dati di due linee indipendenti. n (per linea) = 6; barre di errore = SD. I dati di entrambe le linee sono stati raggruppati per un’analisi statistica con un ANOVA unidirezionale seguito dal test Tukey (p≤0,05) e lettere diverse indicano differenze significative. L’esperimento è stato ripetuto due volte con risultati coerenti. (C, D) analisi CLSM di piantine di cinque giorni, sia coltivate alla luce o eziolate al buio. SRT, shoot-root transition. n (piantine per condizione) = 11; barre di errore = SD. ns: p>0.05, ***p≤0.001 (ANOVA bidirezionale seguito dal test Tukey).DOI:
http://dx.doi.org/10.7554/eLife.26770.015Ripetizione indipendente degli esperimenti mostrati in Figura 6A,B. (A) Le piantine di Arabidopsis di cinque giorni che esprimono l’ATeam citosolico (linea #1.1) sono state analizzate dal CLSM. La fluorescenza di Venere (verde) e CFP (magenta) è stata registrata, e il rapporto è tracciato come un’immagine a falsi colori dove alti valori di Venere / CFP (rosso) corrispondono ad alti livelli di ATP. I rapporti Venus/PCP sono stati analizzati nelle regioni di interesse indicate. Barra di scala = 200 µm. (B) I grafici rappresentano i dati di 24 singoli impianti; barre di errore = SD. Lettere diverse indicano differenze significative in un ANOVA unidirezionale seguito dal test Tukey (p≤0,05).DOI:
http://dx.doi.org/10.7554/eLife.26770.016(A) Le piantine di Arabidopsis di cinque giorni che esprimono l’ATeam citosolico sono state immerse in 100 µM CCCP a pH 7,5 e analizzate dal CLSM. La fluorescenza di Venere (verde) e CFP (magenta) è stata registrata e il rapporto è stato tracciato come un’immagine a falsi colori dove alti valori di Venere / CFP (rosso) corrispondono ad alti livelli di MgATP2-. (B) I rapporti Venus/CFP in ogni punto temporale dopo il trattamento CCCP sono stati quantificati nei cotiledoni, nell’ipocotile e nella radice superiore. n = 3 singole piantine; barre di errore = SD. ns: p>0,05, ***p≤0,001 (ANOVA bidirezionale seguito dal test Tukey). (C) Le piantine di dieci giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono state tagliate con un bisturi per separare i germogli e le radici che sono state immerse individualmente nel mezzo di imaging su piastre microtiter a 96 pozzetti. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Spettri di emissione in fluorescenza tra 460 e 600 nm sono stati registrati utilizzando un lettore di piastre e una lunghezza d’onda di eccitazione di 435 ± 10 nm. I grafici mostrano gli spettri delle parti della pianta che esprimono l’ATeam citosolico dopo la sottrazione della fluorescenza Col-0 in due esperimenti indipendenti (Exp #1/2). n = 5 pozzi ciascuno; barre di errore = SD. (D) I germogli (raggruppati da tre piantine per pozzo) e le radici (raggruppati da 10 a 15 piantine per pozzo) sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. CCCP a 100 µM è stato aggiunto al punto temporale indicato e i dati sono stati tracciati dopo aver sottratto la fluorescenza delle parti di impianto di Col-0 che sono stati ugualmente trattati. n = 5 e 3 pozzetti per tipo di tessuto nell’esperimento indipendente Exp # 1 ed Exp # 2, rispettivamente; barre di errore = SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.017
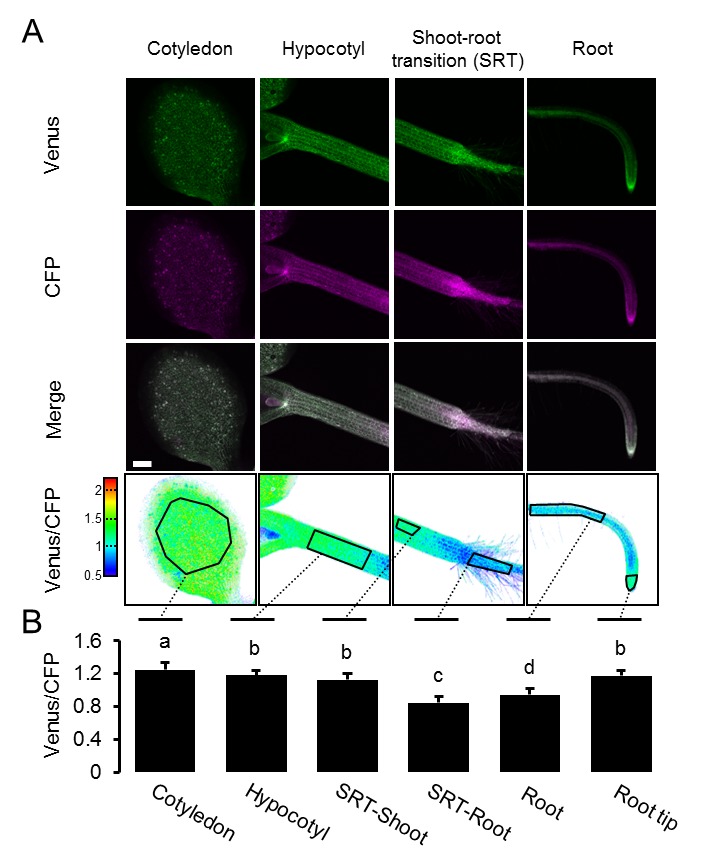
Figura 6-figure supplement 1.Una mappa MgATP2- mappa della piantina di Arabidopsis.2. Ripetizione indipendente degli esperimenti mostrati in Figura 6A,B. (A) Le piantine di arabidopsis di cinque giorni che esprimono l’ATeam citosolico (linea #1.1) sono state analizzate dal CLSM. È stata registrata la fluorescenza di Venere (verde) e CFP (magenta), e il rapporto è stato tracciato come immagine a falsi colori dove alti valori di Venere/CFP (rosso) corrispondono ad alti livelli di ATP. I rapporti Venus/PCP sono stati analizzati nelle regioni di interesse indicate. Barra di scala = 200 µm. (B) I grafici rappresentano i dati di 24 singoli impianti; barre di errore = SD. Lettere diverse indicano differenze significative in un ANOVA unidirezionale seguito dal test Tukey (p≤0,05).DOI:
http://dx.doi.org/10.7554/eLife.26770.016

Figura 6-figure supplement 2.L’effetto del CCCP sulla risposta di ATeam in diversi tessuti di piantine di Arabidopsis.(A) Piantine di Arabidopsis di cinque giorni che esprimono l’ATeam citosolico sono state immerse in 100 µM CCCP a pH 7,5 e analizzate dal CLSM. La fluorescenza di Venere (verde) e CFP (magenta) è stata registrata e il rapporto è tracciato come un’immagine a falsi colori dove alti valori di Venere / CFP (rosso) corrispondono ad alti livelli di MgATP2-. (B) I rapporti Venus/CFP in ogni punto temporale dopo il trattamento CCCP sono stati quantificati nei cotiledoni, nell’ipocotile e nella radice superiore. n = 3 singole piantine; barre di errore = SD. ns: p>0,05, ***p≤0,001 (ANOVA bidirezionale seguito dal test Tukey). (C) Le piantine di dieci giorni che non esprimono alcun sensore (Col-0) o ATeam citosolico sono state tagliate con un bisturi per separare i germogli e le radici che sono state immerse individualmente nel mezzo di imaging su piastre microtiter a 96 pozzetti. La fluorescenza del materiale vegetale è stata controllata con un microscopio a epifluorescenza dotato di un filtro GFP. Spettri di emissione in fluorescenza tra 460 e 600 nm sono stati registrati utilizzando un lettore di piastre e una lunghezza d’onda di eccitazione di 435 ± 10 nm. I grafici mostrano gli spettri delle parti della pianta che esprimono l’ATeam citosolico dopo la sottrazione della fluorescenza Col-0 in due esperimenti indipendenti (Exp #1/2). n = 5 pozzi ciascuno; barre di errore = SD. (D) I germogli (raggruppati da tre piantine per pozzo) e le radici (raggruppati da 10 a 15 piantine per pozzo) sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stata registrata. CCCP a 100 µM è stato aggiunto al punto temporale indicato e i dati sono stati tracciati dopo aver sottratto la fluorescenza delle parti di impianto di Col-0 che sono stati ugualmente trattati. n = 5 e 3 pozzetti per tipo di tessuto nell’esperimento indipendente Exp # 1 ed Exp # 2, rispettivamente; barre di errore = SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.017
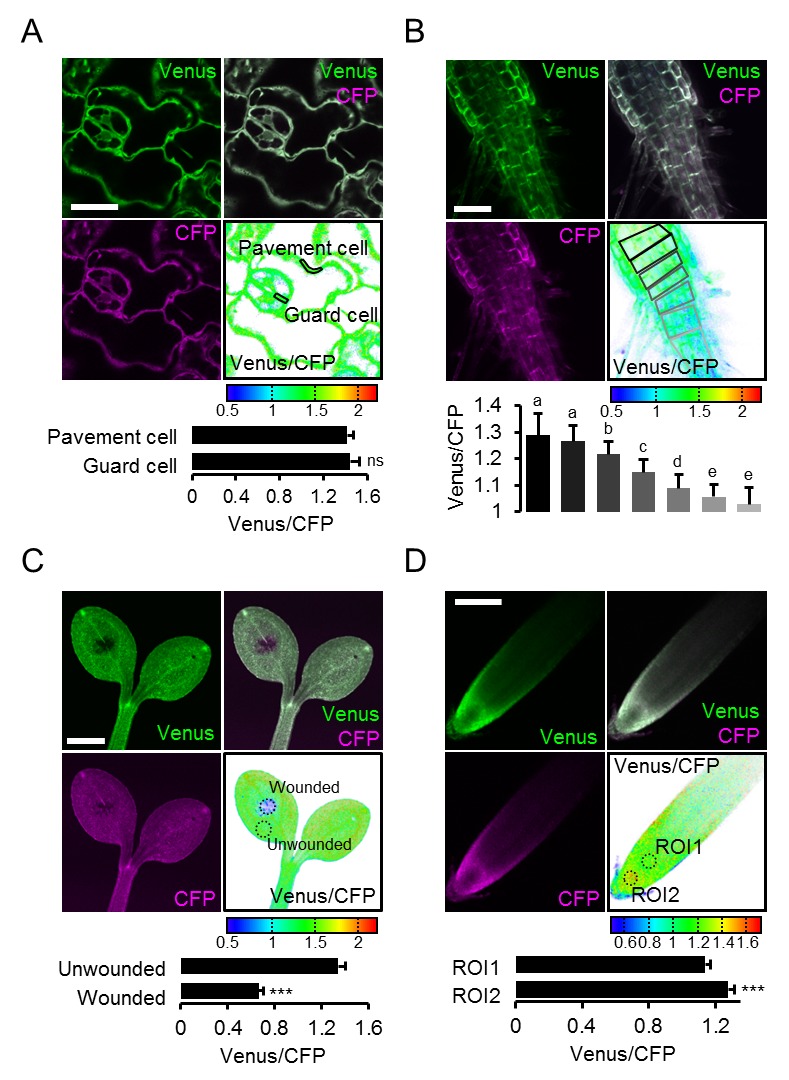
Figura 7.Figura 7. MgATP2- eterogeneità locale MgATP2- eterogeneità nelle cellule e nei tessuti delle piantine di Arabidopsis.La fluorescenza di Venere (verde) e PCP (magenta) da ATeam citosolico è stata registrata dal CLSM in piantine di Arabidopsis di cinque giorni. Il rapporto è tracciato come un’immagine a falsi colori in cui alti valori di Venere / PCP (rosso) corrispondono ad alti livelli di MgATP2-. (A) I rapporti Venus/CFP sono stati valutati in regioni di interesse indicato per confrontare le cellule di guardia e le cellule di pavimentazione dell’epidermide cotiledone. n = 24 coppie di marciapiede e cellula di guardia da cinque singole piante; barre di errore = SD. ns: p>0,05 (t test). Barra di scala = 20 µm. (B) I rapporti Venere / PCP sono stati valutati in strati cellulari indicati nella transizione germoglio-radice. L’analisi della regione di interesse degli strati cellulari successivi è indicata in scala di grigi. n = 11; barre di errore = SD. Lettere diverse indicano differenze statistiche in un ANOVA unidirezionale seguito dal test Tukey (p≤0,05). Barra di scala = 100 µm. (C) I cotiledoni sono stati feriti con un ago e i rapporti Venus/CFP sono stati valutati nelle regioni di interesse indicate che rappresentano tessuti feriti o non feriti. n = 5; barre di errore = SD, ***p≤0,001 (test t). Barra di scala = 500 µm. (D) Il rapporto Venus/CFP è stato valutato in due regioni di interesse alla radice. n = 12; barre di errore = SD, ***p≤0,001 (t test). Barra di scala = 100 µm.DOI:
http://dx.doi.org/10.7554/eLife.26770.018
La velocità di crescita dei peli della radice è correlata con i livelli di MgATP2- come indicato dalla microscopia a fluorescenza a foglio luminoso
Durante la crescita del tubo pollinico è stata riscontrata un’elevata respirazione (Dickinson, 1965) e lo stesso è probabile per altre cellule a rapida crescita in punta, come i peli delle radici. Da un lato, alti tassi di respirazione possono dare origine a livelli elevati di ATP; dall’altro, una domanda elevata durante la crescita rapida può comunque causare l’esaurimento dell’ATP. Affrontare la questione della relazione tra i livelli di ATP e la crescita delle singole cellule è stato in precedenza tecnicamente impossibile. Abbiamo impiegato la microscopia a fluorescenza a foglio luminoso (LSFM) per l’imaging 4D della radice di Arabidopsis in crescita per quantificare i tassi di crescita e FRET dei singoli peli della radice (Figura 8A). Abbiamo misurato sia la velocità di crescita e il rapporto FRET delle singole cellule ciliate (Figura 8B). Mentre il 20% delle cellule ciliate che crescono più rapidamente si è allungato otto volte più velocemente del 20% più lento, quest’ultimo ha mostrato un aumento del loro rapporto FRET indicando che la loro concentrazione citosolica MgATP2- è stata aumentata (Figura 8C,D). Infatti, la velocità media di crescita è stata inversamente correlata con il contenuto di MgATP2- citosolico e l’abbassamento di MgATP2- è stato rilevato esclusivamente nelle cellule ciliate in rapida crescita (Figura 8E). È interessante notare che non vi era alcuna evidenza di oscillazioni in MgATP2- livelli simili a quelli osservati per Ca2 + (Hepler et al., 2001; Monshausen et al., 2008; Candeo et al., 2017), come rapporti FRET è rimasto stabile e non ha mostrato alcuna oscillazione periodica nell’ordine dei secondi, come convalidato da analisi di Fourier (Figura 9).10.7554/eLife.26770.019Figure 8.Light foglio microscopia a fluorescenza (LSFM) analisi dei livelli di MgATP2- livelli di cellule ciliate radicali in crescita.la crescita delle cellule ciliate sulle radici da piantine di sei giorni, che esprimono ATeam citosolico è stata seguita per 10 minuti da LSFM. (A) Massima proiezione del segnale di Venere nella zona di allungamento e di maturazione inferiore generato da 4D dati di imaging in vivo della radice di Arabidopsis in crescita. Barra di scala = 100 µm. (B) Rappresentante zona radice con le cellule ciliate in diverse fasi di sviluppo. Fluorescenza di Venere (verde) e PCP (magenta) è stato registrato e il rapporto è tracciato come immagini a colori falsi su tre punti di tempo. Barre di scala = 100 µm. Sezioni di immagini ingrandite cinque volte, esemplificano (1) una cellula ciliata a crescita lenta e (2) una cellula ciliata a crescita rapida. (C) Cellule ciliate nell’intervallo quantile superiore e inferiore del 20% della velocità di crescita e (D) il corrispondente rapporto citosolico ATeam FRET misurato nella punta delle stesse cellule. n = 48 cellule ciliate da sei radici; barre di errore = SD, *p≤0,05, ***p≤0,001 (t test). (E) Velocità di crescita delle singole cellule ciliate in funzione del loro rapporto citosolico ATeam FRET. I valori delle cellule a crescita più lenta e più rapida, come indicato in (C) e (D), sono indicati in rosso e blu; la linea indica la regressione lineare. Sono forniti la significatività della correlazione, espressa come valore p determinato da un test F, e il coefficiente di determinazione (R²) basato su tutti i punti dei dati. DOI:http://dx.doi.org/10.7554/eLife.26770.01910.7554/eLife.26770.020Figure 9.Analisi delle oscillazioni nelle cellule ciliate radicali.FRET analisi nelle punte dei capelli radicali di piantine di sei giorni che esprimono (A) NES-Yellow Cameleon 3.6 e (B) cytosolic ATeam1.03-nD/nA da LSFM. Le oscillazioni di tre peli radicali rappresentativi sono mostrate come rapporti normalizzati Venus/CFP e spettri di densità spettrale di potenza (PSD) dall’analisi di Fourier. Gli spettri medi di PSD rappresentano 22 peli radicali ciascuno. Barre di errore = SEM. Si noti la presenza di tipiche oscillazioni di Ca2+ intorno a 0,05 Hz nel set di dati Cameleon, che erano assenti nel set di dati ATeam. DOI:http://dx.doi.org/10.7554/eLife.26770.020
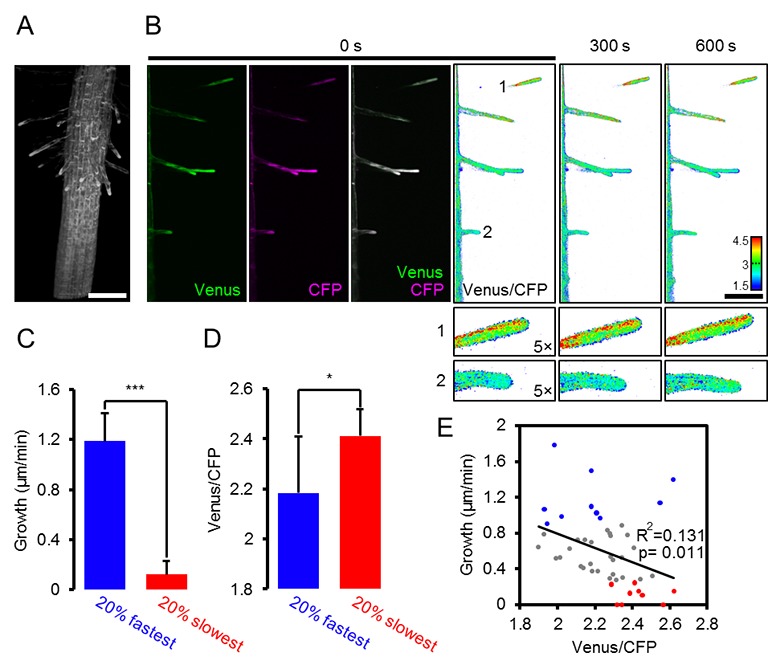
Figura 8.Figura 8. Analisi al microscopio a fluorescenza a foglio luminoso (LSFM) dei livelli di MgATP2 nelle cellule ciliate radicali in crescita.La crescita delle cellule ciliate sulle radici da piantine di sei giorni che esprimono ATeam citosolico è stato seguito per 10 minuti da LSFM. (A) Massima proiezione del segnale di Venere nella zona di allungamento e di maturazione inferiore generato da 4D dati di imaging in vivo della radice di Arabidopsis in crescita. Barra di scala = 100 µm. (B) Rappresentante zona radice con le cellule ciliate in diverse fasi di sviluppo. Fluorescenza di Venere (verde) e PCP (magenta) è stato registrato e il rapporto è tracciato come immagini a colori falsi su tre punti di tempo. Barre di scala = 100 µm. Sezioni di immagini ingrandite cinque volte, esemplificano (1) una cellula ciliata a crescita lenta e (2) una cellula ciliata a crescita rapida. (C) Cellule ciliate nell’intervallo quantile superiore e inferiore del 20% della velocità di crescita e (D) il corrispondente rapporto citosolico ATeam FRET misurato nella punta delle stesse cellule. n = 48 cellule ciliate da sei radici; barre di errore = SD, *p≤0,05, ***p≤0,001 (t test). (E) Velocità di crescita delle singole cellule ciliate in funzione del loro rapporto citosolico ATeam FRET. I valori delle cellule a crescita più lenta e più rapida, come indicato in (C) e (D), sono indicati in rosso e blu; la linea indica la regressione lineare. Sono forniti la significatività della correlazione, espressa come valore p determinato da un test F, e il coefficiente di determinazione (R²) basato su tutti i punti dei dati.DOI:
http://dx.doi.org/10.7554/eLife.26770.019
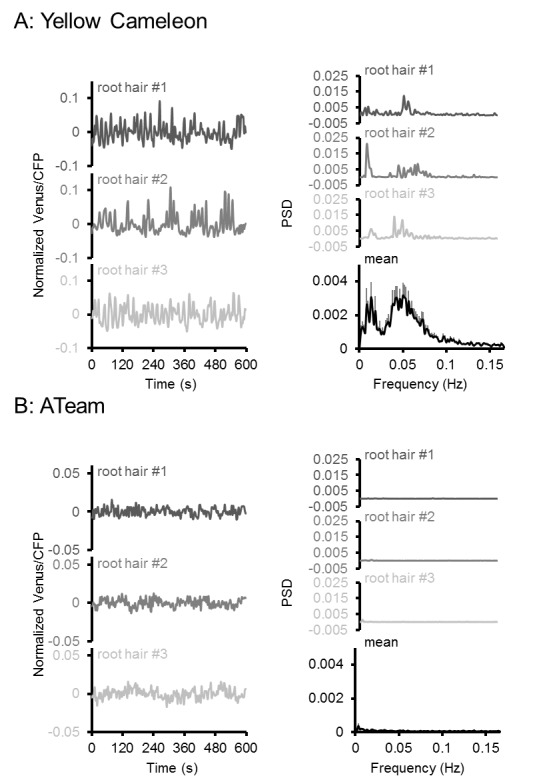
Figura 9.Analisi delle oscillazioni nelle cellule ciliate radicali.Analisi FRET nelle punte dei capelli radice di sei giorni di piantine che esprimono (A) NES-giallo Cameleon 3.6 e (B) citosolico ATeam1.03-nD/nA da LSFM. Le oscillazioni di tre peli radicali rappresentativi sono mostrate come rapporti normalizzati Venus/CFP e spettri di densità spettrale di potenza (PSD) dall’analisi di Fourier. Gli spettri medi di PSD rappresentano 22 peli radicali ciascuno. Barre di errore = SEM. Si noti la presenza di tipiche oscillazioni di Ca2+ intorno a 0,05 Hz nel set di dati Cameleon, che erano assenti nel set di dati ATeam.DOI:
http://dx.doi.org/10.7554/eLife.26770.020
Monitoraggio in vivo del calo dei livelli di MgATP2- citosolico durante l’ipossia
Diverse condizioni di stress sono state correlate ad una crisi energetica cellulare. L’ipossia ha un impatto diretto sulla produzione di ATP mitocondriale, poiché la mancanza di ossigeno come accettore finale di elettroni inibisce la catena respiratoria. Tuttavia, risposte metaboliche flessibili sono state descritte in risposta all’ipossia per prolungare il mantenimento della fornitura di energia cellulare (Geigenberger et al., 2000; Geigenberger, 2003; van Dongen et al., 2009; Zabalza et al., 2009; van Dongen e Licausi, 2015). Per monitorare la dinamica MgATP2- durante la progressione dell’ipossia, abbiamo usato un nastro trasparente a prova di ossigeno per sigillare i pozzetti mediamente riempiti contenenti singole piantine cresciute su piastre di agar e in coltura liquida, senza lasciare spazio residuo di aria (Figura 10; Figura 10-figure supplement 1). Gli esperimenti sono stati condotti al buio (ad eccezione dei flash di eccitazione per le letture fluorimetriche) per evitare l’evoluzione dell’ossigeno tramite la fotosintesi. Le piantine in pozzi non sigillati servivano come controlli. Tutti i rapporti FRET hanno mostrato una diminuzione iniziale subito dopo l’immersione. Questo è stato seguito da una fase di diminuzione costante per le piantine sigillate, mentre le piantine non sigillate hanno mantenuto i rapporti FRET intorno ai valori iniziali. La riossigenazione mediante rimozione del sigillo ha permesso il pieno recupero dei valori di controllo, mentre il mantenimento del sigillo ha portato ad una fase di netta diminuzione e di appiattimento dei rapporti FRET a valori bassi, simili a quelli osservati per il trattamento CCCP. Mentre le caratteristiche di risposta erano riproducibili, l’insorgenza e i tassi delle diverse fasi variavano tra le singole piantine, probabilmente come risultato di differenze nella biomassa, nel tasso di respirazione e nel tasso di diminuzione dell’ossigeno a sua volta. Il modello di ipossia dimostra la possibilità di monitorare in modo affidabile la dinamica subcellulare MgATP2- dinamiche in diretta durante l’insulto da stress, e può essere adattato in modo flessibile ad altre condizioni esterne, tessuti e background genetici.10.7554/eLife.26770.021Figure 10.Arabidopsis MgATP2- dinamiche sotto basso ossigeno.10-day-old piantine di Arabidopsis, cresciute verticalmente su piastre, sono stati immersi in mezzo di imaging su 96 pozzetti microtiter piastre. Per pozzo, tre piantine che non esprimono alcun sensore (Col-0) o ATeam citosolico sono stati eccitati a 435 ± 10 nm, e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. I pozzi sono stati lasciati aperti (grigio), sigillati con una pellicola qPCR trasparente a prova di ossigeno (nero) o sigillati per 15,5 ore prima che la pellicola venisse rimossa per riossi i campioni (rosso). L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 9-12 pozzi e le barre di errore sono SD. DOI:http://dx.doi.org/10.7554/eLife.26770.02110.7554/eLife.26770.022Figure 10-figure supplemento supplemento 1.Arabidopsis MgATP2- dinamiche sotto ossigeno basso. ripetizione indipendente di esperimenti mostrati in figura 10, ma utilizzando sette giorni, piantine di Arabidopsis, coltivate in coltura idroponica. Piantine sono stati immersi in mezzo di imaging su 96 pozzetti microtiter piastre. Per pozzo, due piantine che non esprimono alcun sensore (Col-0) o cytosolic ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. I pozzi sono stati lasciati aperti (grigio), sigillati con una pellicola qPCR trasparente a prova di ossigeno (nero) o sigillati per 17,5 ore prima che la pellicola venisse rimossa per riossi i campioni (rosso). L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 6-10 pozzi e le barre di errore sono SD. DOI:http://dx.doi.org/10.7554/eLife.26770.022
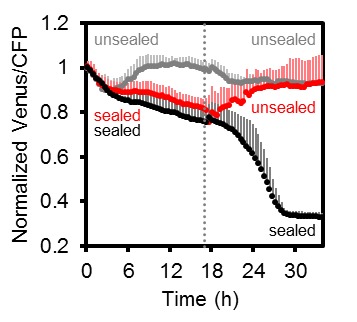
Figura 10-figure supplement 1.Arabidopsis MgATP2- dinamica in condizioni di basso ossigeno.Arabidopsis MgATP2- dinamica in condizioni di basso ossigeno.Dieci giorni piantine di Arabidopsis, cresciute verticalmente su piastre, sono state immerse nel mezzo di imaging su piastre microtiter a 96 pozzetti. Per pozzo, tre piantine che non esprimono alcun sensore (Col-0) o cytosolic ATeam sono stati eccitati a 435 ± 10 nm, e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. I pozzetti sono stati lasciati aperti (grigio), sigillati con una pellicola qPCR trasparente a prova di ossigeno (nero) o sigillati per 15,5 ore prima che la pellicola venisse rimossa per riossi i campioni (rosso). L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 9-12 pozzi e le barre di errore sono SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.021Ripetizione indipendente di esperimenti mostrati in Figura 10, ma utilizzando piantine di Arabidopsis di sette giorni, coltivate in coltura idroponica. Le piantine sono state immerse nel mezzo di imaging su piastre microtiter a 96 pozzetti. Per pozzo, due piantine che non esprimono alcun sensore (Col-0) o cytosolic ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. I pozzi sono stati lasciati aperti (grigio), sigillati con una pellicola qPCR trasparente a prova di ossigeno (nero) o sigillati per 17,5 ore prima che la pellicola venisse rimossa per riossi i campioni (rosso). L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 6-10 pozzi e le barre di errore sono SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.022

Figura 10-figure supplement 1.Arabidopsis MgATP2- dinamica sotto basso ossigeno.Ripetizione indipendente di esperimenti mostrati in Figura 10, ma utilizzando piantine di Arabidopsis di sette giorni, coltivate in coltura idroponica. Piantine sono state immerse in mezzo di imaging su piastre microtiter a 96 pozzetti. Per pozzo, due piantine che non esprimono alcun sensore (Col-0) o cytosolic ATeam sono stati eccitati a 435 ± 10 nm e l’emissione a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-Venus) è stato registrato. I pozzi sono stati lasciati aperti (grigio), sigillati con una pellicola qPCR trasparente a prova di ossigeno (nero) o sigillati per 17,5 ore prima che la pellicola venisse rimossa per riossi i campioni (rosso). L’emissione nei pozzi con piante di Col-0 è stata mediata e sottratta da quella delle piante di ATeam-expressing per correggere la fluorescenza di fondo. I dati mostrati e utilizzati per la sottrazione dello sfondo sono la media di 6-10 pozzi e le barre di errore sono SD.DOI:
http://dx.doi.org/10.7554/eLife.26770.022
Discussione
Monitoraggio in fluorescenza per comprendere le dinamiche ATP
I sensori ATP fluorescenti sono stati inizialmente progettati per cellule e tessuti di mammiferi (Berg et al., 2009; Imamura et al., 2009; Kotera et al., 2010; Nakano et al., 2011; Tantama et al., 2013), dove il loro uso ha permesso nuove intuizioni, come i gradienti di concentrazione di ATP subcellulare tra i compartimenti in situ, la visualizzazione delle dinamiche energetiche indotte dagli stimoli nei neuroni, la reattività dell’attività del canale K+ sensibile all’ATP nelle singole cellule e la sincronizzazione delle dinamiche di Ca2+ e ATP nelle cellule HeLa con la stimolazione dell’istamina. Con l’eccezione di un rapporto sulla morte cellulare indotta da risposta ipersensibile (Hatsugai et al., 2012), le misure di fluorescenza ATP sono state carenti nella ricerca sulle piante. Il monitoraggio della fluorescenza a cellule vive integra approcci standard diffusi, come le determinazioni ATP luminescenti o basate su HPLC in estratti biologici, o tecniche basate su radioisotopi per saggi di trasporto a membrana (Manfredi et al., 2002; Khlyntseva et al., 2009; Lorenz et al., 2015; Monné et al., 2015). Mentre queste tecniche offrono un’elevata sensibilità e accuratezza nella quantificazione dell’ATP, e di altri nucleotidi, in estratti, forniscono misure di endpoint dopo la rimozione dal sistema biologico funzionale e hanno un uso limitato per la risoluzione della concentrazione di ATP nel tempo a livello (sub)cellulare e tissutale. Tuttavia, elevati tassi di flusso, rapide fluttuazioni e gradienti (sub)cellulari sono caratteristiche fondamentali della fisiologia dell’energia cellulare. La misurazione non distruttiva in tempo reale dell’ATP è stata resa disponibile dalla spettroscopia 31P-NMR, che offre l’ulteriore vantaggio di misurare anche l’ADP e altri di- e tri-nucleotidi (Gout et al., 2014). Tuttavia, 31P-NMR è limitata nella risoluzione spaziale e nella sensibilità. La sensibilità superiore di fluorescenza a base di MgATP2- monitoraggio è dimostrato negli esperimenti con isolata Arabidopsis mitocondri (Figura 3), dove i cambiamenti nella concentrazione di MgATP2- sono stati monitorati con risoluzione temporale nell’ordine di secondi su solo 20 µg di proteina mitocondriale. La risoluzione spaziale del rilevamento ATP fluorescente è evidenziata dalla sua capacità di rilevare i livelli di MgATP2- in singole cellule (Figura 7) e cloroplasti singoli (Figura 1; Figura 5-figure supplemento 2), rendendo le sonde ATP-binding sonde una tecnica particolarmente versatile nella fisiologia dei tessuti e delle cellule.
Linee di Arabidopsis per il monitoraggio di MgATP2- nel citosol, cloroplasti e mitocondri
La governance dei livelli di ATP citosolico da parte di due organelli bioenergetici nelle cellule vegetali verdi solleva importanti questioni sulla base regolamentare del controllo subcellulare dell’ATP (Gardeström e Igamberdiev, 2016). Le linee del sensore citosolico non hanno indicato differenze nel rapporto tra citosol e nucleoplasma, indicative di una rapida diffusione tra le due sedi e in linea con le precedenti osservazioni per le concentrazioni di Ca2+ libero e il potenziale redox del glutatione (Loro et al., 2012; Schwarzländer et al., 2016). Le linee del sensore dei cloroplasti forniscono un buon segnale FRET, anche in presenza di clorofilla (Figura 5-figure supplement 2). Queste linee saranno un valido strumento per sezionare l’impatto della fotosintesi sull’ATP subcellulare, e le indagini sono attualmente in corso nei nostri laboratori. Mentre non è stato osservato alcun fenotipo di sviluppo per le linee del sensore citosolico e plastidico (Figura 1; Figura 1-figure supplement 2 e 3), il fenotipo forte che sistematicamente è risultato dal sensore mirato alla matrice mitocondriale (Figura 1C; Figura 1-figure supplement 2) richiede cautela. Non si può escludere che l’omeostasi ATP è generalmente perturbato in quelle linee di sensori, anche se le misurazioni basate su sensori in cellule di mammifero hanno anche indicato ATP abbassato nella matrice, che può essere rappresentato da membrana potenziale guidato AAC-mediata ATP esportazione ATP (Imamura et al., 2009). Il motivo del fenotipo stentato non è attualmente chiaro. Interferenze con l’omeostasi della matrice ATP da buffering ATP sembra improbabile, soprattutto perché fenotipi simili di gravità variabile può essere osservato anche per altri sensori mitocondrialmente mirati (Figura 1-figure supplemento 4); problemi simili sono stati osservati anche nel lievito (Schwarzländer et al., 2016). L’intasamento del macchinario TIM/TOM mediante l’arresto dell’importazione della costruzione del sensore sintetico può spiegare gli effetti osservati.
Caratteristiche e limitazioni del sensore rivelate in vitro
ATeam1.03-nD/nA purificato è stato selettivo per MgATP2- (Figura 2), affinando le precedenti caratterizzazioni del sensore padre ATeam1.03 (Imamura et al., 2009). Il sensore non riporta la carica energetica impostata dal rapporto ATP:ADP direttamente (Pradet e Raymond, 1983), senza un’ulteriore ipotesi che il pool totale di adenosina sia costante. In vivo, è improbabile che la sintesi e la degradazione de novo cambino il pool totale di adenosina così rapidamente come il ciclo ATP/ADP/AMP mediante (de-)fosforilazione. Quindi, i cambiamenti MgATP2- cambiamenti indotti da trattamenti inibitori e durante l’ipossia sono suscettibili di riflettere anche la diminuzione della carica energetica. Al contrario, le differenze nei livelli di ATP allo stato stazionario tra cellule e tessuti e organi possono riflettere differenze costitutive nelle dimensioni totali del pool di adenilato. Analisi combinatorie con sensori che rispondono direttamente alla carica energetica, come le varianti di Perceval (Tantama et al., 2013), saranno auspicabili in futuro. Tuttavia, i sensori ATP:ADP attualmente disponibili soffrono di una grave sensibilità al pH, che aggiunge un livello di complessità all’interpretazione significativa nelle piantagioni, dove possono verificarsi pronunciate fluttuazioni del pH.
Un test dinamico per sezionare i flussi di ATP dei mitocondri isolati
Un’innovazione chiave del rilevamento a base di proteine fluorescenti è stata la capacità di monitorare la biochimica come avviene nel sistema biologico intatto. Tuttavia, le conclusioni meccanicistiche sono state spesso problematiche sulla base dei tentativi di collegare il riduzionismo in vitro con la complessità in vivo, e le sfide nel portare i sistemi intatti sotto controllo sperimentale. I saggi ATP che stabiliamo per i mitocondri isolati introducono un utile intermedio (Figure 3 e 4). Nonostante l’uso di interi organuli come unità multifunzionali complesse, i diversi passi della dinamica ATP possono essere monitorati e interpretati meccanicamente. L’alimentazione del substrato e i trattamenti inibitori permettono uno stretto controllo sullo stato funzionale dell’organulo e la manipolazione controllata della fisiologia dell’ATP. Ad esempio, siamo stati in grado di distinguere le attività di fosforilazione di ATP sintasi e AK, e monitorare entrambe le attività sfruttando le loro caratteristiche differenziali, compresa la loro esposizione sub mitocondriale alla matrice e lo spazio intermembrana, rispettivamente. Perturbazione della funzione mitocondriale è stata osservata con elevata sensibilità e risolta cineticamente a seguito di inibizione dei giocatori definiti nella produzione di ATP mitocondriale. I substrati e gli inibitori sono adatti per modulare l’attività respiratoria, la capacità e l’efficienza, e per indurre transizioni nelle dinamiche ATP. Essi possono essere applicati per sezionare i riarrangiamenti bioenergetici nei mutanti respiratori in futuro.
La comprensione attuale della bioenergetica cellulare è stata guidata da misurazioni dinamiche in mitocondri isolati e cloroplasti, ad esempio del consumo di ossigeno o del trasporto Ca2+. Tuttavia, il monitoraggio delle dinamiche ATP, come prodotto centrale, si è tipicamente basato sull’inferenza indiretta, o sulla ricostruzione di dati risolti nel tempo da singoli campioni (Attucci et al., 1991). Il concetto del saggio ATP basato su biosensori fluorescenti introduce un metodo complementare per monitorare in modo continuo e dinamico i flussi di trasporto dell’ATP da e verso le cellule e gli organuli isolati, come dimostrato dall’uso dei mitocondri delle piante. Poiché la proteina del sensore viene aggiunta esternamente ai campioni e non richiede l’espressione del sensore transgenico da parte dei campioni, i componenti cellulari di qualsiasi specie possono essere studiati per la generazione di ATP, il consumo e il trasporto di membrana. I saggi possono essere ulteriormente ampliati per studiare la dinamica del trasporto di membrana. Ad esempio, la risposta del trasporto di ATP attraverso la busta dei cloroplasti ai cambiamenti nell’attività fotosintetica dovrebbe essere facilmente rilevabile, rendendo possibile affrontare questioni ancora irrisolte sull’efficienza del trasporto, la direzionalità e i percorsi, in condizioni esterne controllate (Haferkamp et al., 2011; Gardeström e Igamberdiev, 2016). Il principio del test è adattabile ad altre strutture subcellulari, e potenzialmente anche ai sistemi liposomici, cellulari e tissutali. Può anche facilitare il miglioramento dei saggi di enzimi accoppiati con ATP. Il monitoraggio dell’esportazione e dell’assorbimento dell’ATP da parte delle cellule può essere di specifico interesse per affrontare le attuali domande sui flussi intercellulari di ATP e la funzione di segnalazione dell’ATP extracellulare (Kim et al., 2006; Tanaka et al., 2010; Choi et al., 2014a). Sono state ingegnerizzate varianti di ATeam con affinità più elevate per abbinare intervalli di concentrazione di ATP inferiori, e sono probabilmente specifiche anche per MgATP2- (Imamura et al., 2009).
Monitoraggio dell’ATP in piantagione con tre approcci di rilevamento dei sensori
Abbiamo eseguito il rilevamento dal vivo dell’ATP in piantagione con tre distinte tecniche in vivo. La fluorimetria a micropiastre (figure 3-5 e 10), il CLSM (figure 1, 6 e 7) e l’LSFM (figure 8 e 9) ne completano un’altra per studiare la dinamica e la distribuzione dell’ATP nei tessuti vegetali viventi. Mentre il CLSM è stato ampiamente utilizzato in combinazione con altri sensori di proteine fluorescenti nelle piante, l’LSFM è stato regolato solo recentemente per consentire misurazioni Ca2+ basate su FRET nelle radici di Arabidopsis (Costa et al., 2013; Candeo et al., 2017). La fluorimetria a base di micropiastre è stata ampiamente utilizzata per le cellule animali in coltura e i lieviti (Birk et al., 2013; Morgan et al., 2016), ma raramente nei tessuti vegetali (Rosenwasser et al., 2010, 2011), e mancava una rigorosa convalida tecnica. La nostra analisi mostra che un’attenta regolazione delle impostazioni fluorimetriche, con le proprietà del sensore e le caratteristiche del tessuto può rendere le misurazioni basate su lettori di lastre un approccio robusto per il monitoraggio continuo dei sensori di proteine fluorescenti per lunghi periodi di tempo. Mentre l’eterogeneità tra cellule e tessuti è mediata, è possibile valutare in parallelo grandi numeri di campioni, consentendo controlli robusti e tenendo conto della variabilità biologica. Il puntamento genetico del sensore ad una specifica posizione subcellulare dove risponde ai livelli locali di ATP significa che la risoluzione della fisiologia subcellulare viene mantenuta. Inoltre, le misurazioni fianco a fianco di diversi sensori in diversi tessuti o compartimenti cellulari possono permettere di monitorare e sezionare l’interazione di diverse risposte fisiologiche allo stesso stimolo. Ad esempio, si può valutare l’impatto dell’ipossia su ATP citonucleare, pH e Ca2+, o l’impatto dell’illuminazione sulla concentrazione di ATP nel citosol e nello stroma cloroplastico. Il monitoraggio parallelo affiancato fornisce un’alternativa al multiplexing di più sensori co-espressi nello stesso impianto. La robustezza del test può anche essere adatta per schermi biologici genetici o chimici (Dejonghe e Russinova, 2017) basati su sensori fluorescenti in futuro.
Rilevamento e manipolazione di MgATP2- in vivo
La diminuzione e l’aumento dei livelli di MgATP2- cytosolic MgATP2- livelli in vivo da trattamenti chimici ha portato ad una risposta affidabile che ha coperto quasi l’intera gamma dinamica teorica del sensore (Figura 5). Si deduce che il sensore è funzionale in vivo, e non è influenzato dalla scissione proteolitica. La diminuzione effettiva di MgATP2- dopo l’inibizione con antimicina A ha convalidato il ruolo dei mitocondri come principali fornitori di ATP citosolico, in quanto la perdita di ATP derivato dalla respirazione non poteva essere sostituito. Tuttavia, la cinetica di risposta ai trattamenti chimici sembrava essere dominata dal tasso di assorbimento dei tessuti. Il monitoraggio dei trattamenti da parte del CLSM ha indicato tassi di risposta variabili a seconda del tipo di tessuto (Figura 6-figure supplement 2). Tuttavia, tutti i tessuti hanno gradualmente adottato un rapporto FRET altrettanto basso del sensore privo di MgATP2. La capacità di guidare il sensore ai suoi estremi in vivo permette la conversione dei rapporti FRET in vivo in concentrazioni assolute di MgATP2-, come di routine nel rilevamento Ca2+ (Palmer e Tsien, 2006), e nel rilevamento del tiolo redox (Schwarzländer et al., 2008). Poiché la conversione richiede ulteriori ipotesi, e diventa imprecisa verso il campo di risposta non lineare del sensore, è stato criticato un approccio completamente quantitativo. In questo lavoro utilizziamo i valori FRET come rappresentazione diretta della dinamica in vivo e delle differenze relative (Wagner et al., 2015b). Quando si desidera la conversione in concentrazioni assolute, esse devono essere considerate come una stima. Qui circa 2 mM MgATP2- possono essere stimati per il citosol allo stato stazionario al buio (Figure 2 e 5). Questo valore di concentrazione integra le determinazioni di ATP normalizzate al contenuto di clorofilla mediante frazionamento dei protoplasti (Stitt et al., 1982; Gardeström e Wigge, 1988) e mediante NMR (Gout et al., 2014).
Verso una comprensione integrata della fisiologia energetica nello sviluppo e sotto stress
CLSM risolto le differenze di organi, tessuti e cellule per dare un dettagliato MgATP2- mappa MgATP2- della piantina di Arabidopsis (Figure 6 e 7). Mappe simili sono state generate per la distribuzione ormonale (ad esempio auxina e acido ascissico Brunoud et al., 2012; Jones et al., 2014) per studiare lo sviluppo delle piante. Per i metaboliti centrali l’analisi trascrizionale ad alta risoluzione ha mirato a generare mappe tissutali (Chaudhuri et al., 2008), ma non è stata intrapresa una mappatura diretta in vivo tra tessuti e organi. Le differenze nella concentrazione di MgATP2- tra tessuti e cellule non sono state precedentemente distinguibili con una risoluzione ragionevole, e la mappa MgATP2- rivela quindi un quadro sorprendentemente eterogeneo e dinamico. I tessuti verdi hanno mostrato una concentrazione complessiva di MgATP2- alta, che sono improbabili a causa dell’attività fotosintetica, perché le piantine erano scure-adattate. Eziolamento non solo è diminuito MgATP2- nei tessuti verdi, ma anche aumentato i livelli nelle radici. Il significato biologico di tessuto-specifico MgATP2- eterogeneità merita una dissezione approfondita nel contesto del metabolismo e lo sviluppo in futuro. Poiché la mappa rappresenta MgATP2-, come la principale forma biodisponibile di ATP, le differenze possono, in linea di principio, essere dovute alle dimensioni della piscina ATP, pH e / o Mg2 +. Molte proteine legano l’MgATP2-, ma la misura in cui questo influisce sul buffering della piscina e sulla concentrazione libera, che è disponibile per il legame da parte del sensore, è attualmente sconosciuta.
Con la dovuta cautela, la mappa fornisce una visione della distribuzione di MgATP2- a livello di sistema e il concetto stabilisce un punto di riferimento per l’espansione in diverse dimensioni, compreso il tempo, il compartimento subcellulare, e altri parametri genetici, biochimici o fisiologici. Il time-lapse imaging può risolvere i cambiamenti dinamici in un tessuto, sia durante i processi di sviluppo che in risposta a stimoli esterni. La nostra osservazione che la concentrazione di MgATP2- aumenta con la diminuzione della crescita dei peli della radice esemplifica il verificarsi di tali dinamiche e fornisce la prova della complessa relazione tra lo stato energetico cellulare e la crescita. Analoghe mappe possono essere generate per altri compartimenti subcellulari, come i plastidi, per ottenere una mappa tissutale risolta dell’eterogeneità subcellulare MgATP2- eterogeneità. Altri sensori, ad esempio per pH, Ca2+ o potenziale redox di glutatione, possono essere utilizzati e sovrapposti per costruire gradualmente una rappresentazione completa dello stato fisiologico cellulare dell’intera pianta. La sovrapposizione con mappe trascrittomiche e proteomiche (ad esempio, Chaudhuri et al., 2008; Li et al., 2016) può anche consentire una correlazione tra i livelli organizzativi della cellula. Tale mappatura sistemica multidimensionale in vivo fornirà un nuovo fondamento ai tentativi di modellazione del metabolismo di piante intere e alla comprensione di come il metabolismo sia alla base dello sviluppo delle piante.
L’interruzione della fornitura di ossigeno fresco alle piantine di Arabidopsis ha portato ad una graduale diminuzione della concentrazione citosolica MgATP2- (Figura 6), seguendo le aspettative dei precedenti lavori sugli estratti vegetali e utilizzando NMR (Xia e Saglio, 1992; Geigenberger et al., 2000; Gout et al., 2001; van Dongen et al., 2003). La diminuzione è avvenuta in quattro fasi ed è stata completamente reversibile mediante riossigenazione. I rapporti FRET sono inizialmente diminuiti lentamente, tuttavia, dopo un prolungato blocco dell’apporto di ossigeno, si è verificata una forte diminuzione della concentrazione di MgATP2- la concentrazione di MgATP2- ad un nuovo livello di plateau, che può rappresentare il limite inferiore di sensibilità della sonda. La cinetica esatta dipendeva dalla configurazione sperimentale e il tipo di tessuto, ma la risposta complessiva era riproducibile (Figura 10; Figura 10-figure supplemento 1). La risposta rapida e la graduale diminuzione insieme sostengono le ragioni che il pool citosolico MgATP2- dipende strettamente dalla fosforilazione ossidativa, che il buffering basato su AK può al massimo ritardare l’esaurimento di MgATP2-, ma che un forte esaurimento di MgATP2- può essere evitato, probabilmente attraverso la ristrutturazione dei flussi metabolici (Geigenberger, 2003; Zabalza et al., 2009). L’impostazione sperimentale non ha generato un’improvvisa diminuzione della disponibilità di ossigeno, ma si è basata sulla respirazione oscura per un graduale esaurimento dell’ossigeno. Le risposte osservate rappresentano quindi gli effetti integrati della progressione dinamica dell’ipossia e dei cambiamenti metabolici di compensazione, in quanto possono verificarsi in una situazione di improvvisa (profonda) registrazione dell’acqua. Ipossia-associata ipossia acidificazione intracellulare può anche influenzare la risposta del sensore influenzando MgATP2- dissociazione. Tuttavia, questi cambiamenti indotti dal pH non sono artefatti e hanno un significato fisiologico, poiché una diminuzione della concentrazione di MgATP2- non solo influenza il sensore, ma anche le proteine endogene MgATP2-dipendenti. Inoltre, il pH controllato MgATP2- trattamenti di esaurimento MgATP2- indicano solo un contributo minore di pH (Figura 5-figure supplemento 3) e il lavoro precedente ha suggerito che il pH citosolico difficilmente diminuisce sotto 7, anche sotto anossia (Gout et al., 2001; Schulte et al., 2006). Come tale, i saggi di ipossia dimostrano che ATeam1.03-nD/nA permette il monitoraggio continuo dei pool subcellulari di MgATP2 in risposta allo stress. Poiché l’esatta cinetica ATP è modellata dal macchinario di risposta allo stress cellulare, essi forniscono una lettura sensibile ed integrata per difetti o modifiche nei mutanti.
Conclusioni e prospettive
Abbiamo studiato la bioenergetica mitocondriale, le risposte all’ipossia delle piante e il contenuto di MgATP2 nei tessuti vegetali a seconda delle condizioni di crescita e di sviluppo. Questi studi esemplificano la versatilità del rilevamento ATP fluorescente per aprire nuove porte nella biologia vegetale. Il loro seguito sistematico e l’estrapolazione saranno necessari per una visione sistemica dell’ATP in futuro. Anche se la biochimica dell’ATP è stata ampiamente studiata nel secolo scorso, rimangono lacune sorprendentemente grandi nella nostra comprensione delle sue dinamiche all’interno delle cellule e delle piante intere. Il rilevamento di MgATP2- in modo dinamico e con risoluzione (sub)cellulare aggiunge una nuova profondità allo studio del metabolismo delle piante, dello sviluppo, della segnalazione e delle risposte allo stress. La nostra comprensione delle interazioni dei microbi vegetali, dove la biochimica alla base delle risposte localizzate e dinamiche è stata notoriamente difficile da catturare, può trarre beneficio in particolare. Altre potenziali applicazioni includono l’identificazione e la validazione in vivo dei sistemi di trasporto ATP, una migliore comprensione del coordinamento tra plastidi e mitocondri nella produzione di ATP, una valutazione dell’impatto dei sistemi di disaccoppiamento, la visualizzazione del parassitismo energetico nelle diatomee, l’estensione dei saggi enzimatici in situ e la mappatura di MgATP2- ad altre uscite dei sensori e gradienti di ossigeno nei tessuti. L’ottimizzazione dei sensori ATP ad alta affinità per l’apoplasto potrebbe supportare le indagini sulla segnalazione ATP extracellulare. Ulteriori sensori per il rapporto AMP, ADP e ATP:ADP possono essere integrati nel quadro metodologico qui introdotto. Ogni volta che un biosensore per un nuovo aspetto della fisiologia cellulare, ad esempio per il Ca2+ libero o il potenziale redox del glutatione (Allen et al., 1999; Pei et al., 2000; Meyer et al., 2007; Schwarzländer et al., 2008; Krebs et al., 2012; Loro et al., 2012; Wagner et al., 2015b), è stato introdotto nella ricerca sulle piante, ciò ha prodotto un’esplosione di scoperte. Ci aspettiamo che il rilevamento ATP non faccia eccezione.
Materiali e metodi
Clonazione dei costrutti dei sensori e generazione di linee di impianto
La sequenza ATeam1.03-nD/nA è stata amplificata in PCR da pENTR1A:ATeam1.03-nD/nA. La sequenza leader da Nicotiana plumbaginifolia ß-ATPase (Logan e Leaver, 2000) per l’importazione mitocondriale è stata fusa al N-termino per estensione PCR. Per l’espressione costitutiva dell’impianto sotto un promotore CaMV 35S, questa fusione e la sequenza non fusa per il targeting citonucleare sono state subclonate in pDONR207 (Invitrogen Ltd, Carlsbad, CA) e infine pB7WG2 e pH2GW7, rispettivamente (Karimi et al., 2002). Per il targeting dello stroma plastid, la sequenza leader di Nicotiana tabacum transketolase (Wirtz and Hell, 2003; Schwarzländer et al., 2008) e la sequenza ATeam1.03-nD/nA sono state subclonate in pENTR/D-TOPO (Invitrogen Ltd) tramite i siti di restrizione NdeI/PstI e BamHI/XbaI, rispettivamente. La fusione è stata clonata in pEarleyGate100 (Earley et al., 2006) per l’espressione 35S-driven. Le sequenze di primer sono dettagliate nel file supplementare 1. La sequenza non fusa è stata anche inserita in pETG10A per l’espressione della proteina nelle cellule di Escherichia coli. 2. Agrobacterium-mediata trasformazione di Arabidopsis thaliana (L.) Heynh. (adesione Columbia, Col-0) è stata eseguita da immersione floreale (Clough e Bent, 1998). I trasformatori e le linee omozigoti sono stati selezionati in base alla resistenza chimica e all’intensità della fluorescenza. La generazione della linea NES-YC3.6 Cameleon è stata descritta in precedenza (Krebs et al., 2012).
Prodotti chimici
I prodotti chimici sono stati acquistati da Sigma-Aldrich (Taufkirchen, Germania). Soluzioni stock di ATP, ADP e AMP sono state appena integrate con concentrazioni equimolari di MgCl2 ad eccezione della titolazione di Mg2+. Tutte le soluzioni madre sono state regolate per il dosaggio del pH prima dell’uso.
Coltura delle piante
Dove non diversamente indicato, le piantine di Arabidopsis sono state coltivate da semi sterilizzati in superficie su piastre verticali contenenti Murashige e Skoog a mezza resistenza (MS) medium (Murashige e Skoog, 1962) con 1% (p/v) di saccarosio e 0.8% (p/v) Phytagel in condizioni di lunga giornata (16 ore 80-120 µmol fotoni m-2 s-1 a 22°C, 8 ore di buio a 18°C) dopo stratificazione a 4°C al buio. Per la preparazione della linea dei dischi fogliari, le piante sono state germinate e cresciute sul terreno in condizioni di lunga giornata (17°C, 16 ore 50-75 µmol fotoni m-2 s-1, 8 ore di buio) dopo la stratificazione a 4°C al buio.
Fenotipizzazione delle piante
I semi sono stati stratificati a 4°C al buio per 2 d su mezzo MS a mezza forza +1% (w/v) saccarosio +1% (w/v) MES +1% (w/v) Phytagel, pH 5.8 e piantine sono state coltivate sulle piastre verticalmente affiancate con i relativi controlli per 5 d in condizioni di lunga giornata (16 ore a 22°C e 75-100 µmol fotoni m-2 s-1, 8 ore a 18°C e buio). La lunghezza della radice primaria è stata documentata e quantificata utilizzando ImageJ. Le piante sono state poi trasferite individualmente in vasi Jiffy, distribuite in modo casuale su piani standard in serra e coltivate in camere di crescita a lunga giornata (16 ore a 19°C e 60-80 µE m-2 s-1, 8 ore a 17°C e buio). Lo sviluppo della rosetta fogliare è stato documentato fotograficamente e le dimensioni della rosetta sono state analizzate con lo strumento personalizzato Leaf Lab (Versione 1.41) come descritto in precedenza (Wagner et al., 2015b). L’altezza dell’infiorescenza primaria è stata sistematicamente catturata con una macchina fotografica e quantificata con ImageJ. Le silique sono state contate manualmente quando le prime silique sono diventate gialle, ma non si erano ancora frantumate.
Purificazione di ATeam1.03-nD/nA
Il ceppo E. coli Rosetta 2 (DE3) che porta il pETG10A-ATeam1.03-nD/nA è stato coltivato in brodo di lisogenia (LB; Bertani, 1951) medio a 37°C fino ad un OD600 di 0,2. La coltura è stata trasferita a 20 ° C fino a quando le cellule hanno raggiunto un OD600 di 0,6-0,8. Espressione di 6×His-ATeam1.03-nD/nA è stata indotta da isopropil β-D-1-tiogalattopiranoside ad una concentrazione finale di 0,2 mM durante la notte a 20 ° C. Le cellule sono state raccolte per centrifugazione a 4000 g per 10 minuti a 4°C e il pellet è stato risospeso in tampone di lisi (100 mM Tris-HCl, pH 8,0, 200 mM NaCl, 10 mM imidazolo) integrato con 1 mg / ml lisozima, 0,1 mg / ml DNasiI (Roche, Mannheim, Germania) e cOmplete proteasi inibitore cocktail (Roche). Dopo 30 minuti di incubazione su ghiaccio, le cellule sono state sonicate (3 × 2 min, 40% di potenza, 50% duty cycle). Il lisato è stato centrifugato a 40.000 g per 40 minuti a 4°C e il supernatante è stato caricato su una colonna Ni-NTA HisTrap (GE Healthcare, Freiburg, Germania) e le proteine sono state eluite con un gradiente di imidazolo (10-200 mM in 100 mM Tris-HCl, pH 8,0, 200 mM NaCl) utilizzando un sistema di cromatografia ÄKTA Prime Plus (GE Healthcare). Le frazioni contenenti ATeam1.03-nD/nA sono state messe in pool, concentrate mediante ultrafiltrazione e applicate ad una colonna HiLoad 16/600 Superdex 200 (GE Healthcare) pre-equilibrata con 20 mM Tris-HCl, pH 8.0, 150 mM NaCl. Le frazioni contenenti ATeam1.03-nD/nA intatto sono state messe in pool, concentrate mediante ultrafiltrazione, integrate con il 20% (v/v) di glicerolo e conservate a -86°C.
Caratterizzazione di ATeam1.03-nD/nA in vitro
La concentrazione di ATeam1.03-nD/nA purificato è stata quantificata secondo Bradford (1976). Proteine ad una concentrazione finale di 1 µM è stato miscelato con terreno di incubazione di base (0,3 M saccarosio, 5 mM KH2PO4, 50 mM TES-KOH, pH 7,5, 10 mM NaCl, 2 mM MgSO4, 0,1% (w / v) BSA) per tutti i saggi in vitro, ad eccezione delle titolazioni Mg2 +, in cui MgSO4 è stato omesso dal terreno di incubazione di base. Uno spettrofluorimetro FP-8300 (Jasco, Gross-Umstadt, Germania) a 25°C è stato utilizzato per eccitare mseCFP a 435 ± 5 nm e sono stati registrati spettri di emissione tra 450 e 600 nm con una larghezza di banda di 5 nm. I rapporti Venus/CFP sono stati calcolati come misura dell’efficienza FRET dalle intensità di emissione di fluorescenza a 527 nm (cp173-Venus) e 475 (mseCFP) nm dopo la correzione del bianco.
Isolamento mitocondriale e saggi di consumo di ossigeno
Le piantine di Arabidopsis sono state coltivate in vasi idroponici per 14 giorni come descritto da Sweetlove et al. (2007) in condizioni di lunga giornata (16 ore 50-75 µmol fotoni m-2s-1 a 22°C; 8 ore di buio a 18°C). I mitocondri delle piantine sono stati isolati come descritto da Sweetlove et al. (2007) e Schwarzländer et al. (2011). Il consumo di ossigeno è stato analizzato come descritto da Sweetlove et al. (2002) e Schwarzländer et al. (2009) utilizzando due elettrodi di tipo Clark (Oxytherm, Hansatech, Norfolk, UK).
Fluorimetria basata su lettori di piastre multipozzetto
ATeam1.03-nD/nA è stato eccitato con luce monocromatica ad una lunghezza d’onda di 435 ± 10 nm in un lettore di lastre CLARIOstar (BMG Labtech, Ortenberg, Germania). L’emissione è stata registrata a 483 ± 9 nm (mseCFP) e 539 ± 6,5 nm (cp173-mVenus) utilizzando piastre trasparenti a 96 pozzetti (Sarstedt, Nümbrecht, Germania). La temperatura interna è stata mantenuta a 25°C, e la piastra è stata agitata orbitalmente a 400 rpm per 10 s dopo ogni ciclo. Per i saggi con mitocondri isolati, 20 µg di proteine totali in mezzo di incubazione di base (0,3 M saccarosio, 5 mM KH2PO4, 50 mM TES-KOH, pH 7,5, 10 mM NaCl, 2 mM MgSO4, 0,1% (w / v) BSA) sono stati integrati con 1 µM purificato ATeam1.03-nD / nA in un volume totale di 200 µL per pozzo. Sfondo fluorescente del mezzo di incubazione di base è stato registrato e sottratto da tutti i dati prima dell’analisi. Per esperimenti in vivo con piantine di Arabidopsis e dischi di foglie, il materiale vegetale è stato immerso in 10 mM MES, pH 5,8, 5 mM KCl, 10 mM MgCl2, 10 mM CaCl2. Le piastre sono state tenute al buio per almeno 30 minuti prima di registrare per ridurre al minimo i potenziali effetti della fotosintesi attiva. Il tampone TES è stato sostituito con Bis-Tris-HCl (che copre valori di pH compresi tra 6,0 e 7,0) o Tris-HCl (che copre valori di pH compresi tra 7,5 e 8,5) dove indicato. Per limitare l’apporto di ossigeno, le piastre sono state sigillate con pellicole ultra trasparenti per qPCR (VWR, Langenfeld, Germania).
Microscopia e analisi a scansione laser confocale
L’imaging confocale è stato eseguito a 20°C utilizzando un microscopio Zeiss LSM780 e una lente ×5 (EC Plan-Neofluar, 0,16 N.A.),×10 (Plan-Apochromat, 0,3 N.A.) o ×40 (C-Apochromat, 1,20 N.A., immersione in acqua) utilizzando la procedura descritta in precedenza (Wagner et al., 2015a). ATeam1.03-nD/nA è stato eccitato a 458 nm e la fluorescenza di mseCFP e cp173-mVenus sono stati misurati a 465-500 nm e 526-561 nm, rispettivamente, con il foro stenopeico impostato su tre unità ariose. La fluorescenza della clorofilla è stata raccolta a 650-695 nm. MitoTracker Orange è stato eccitato a 543 nm e l’emissione è stata registrata a 570-619 nm. Le piante sono state oscurate per almeno 30 minuti prima dell’acquisizione dell’immagine. Le immagini di un singolo piano sono state elaborate con un software personalizzato basato su MATLAB (Fricker, 2016) utilizzando il filtraggio del rumore x,y e la sottrazione di fondo a fluorescenza.
Microscopia a fluorescenza a foglio luminoso e analisi
La crescita delle cellule ciliate sulle radici di piantine di sei giorni è stata seguita per 10 minuti con immagini acquisite ogni 3 s. Ogni immagine è stata generata come proiezione di massima intensità (MIP) di 15 pile spaziate di 3 µm ed il tempo di acquisizione MIP è stato di 1 s, basato sull’esposizione di 50 ms per pila e sul tempo di trasferimento dati. Il LSFM utilizzato è stato specificamente progettato per studiare le radici delle piante (Costa et al., 2013; Candeo et al., 2017). La sorgente era un laser monomodale a fibra accoppiata che emetteva radiazioni a 442 nm (MDL-III-442, CNI) per eccitare ATeam mseCFP. Una lente cilindrica in combinazione con un obiettivo per microscopio ad immersione ad acqua 10× (Olympus UMPLFLN 10×W, 0,3 N.A.) ha creato un sottile foglio di luce laser sul campione (5-µm di spessore e 800-µm di altezza in direzione verticale). L’unità di rilevamento consisteva in un obiettivo da microscopio ad immersione 20× acqua (Olympus UMPLFLN 20×W, 0,5 N.A.), tenuto ortogonalmente all’asse di eccitazione. Uno splitter di immagine (filtro dicroico a 505 nm, DMLP505, Thorlabs, Newton, NY; filtri passa banda, MF479-40 e MF535-22, Thorlabs, Newton, NY) e una fotocamera veloce (Neo 5.5 sCMOS, ANDOR, Belfast, UK) hanno permesso l’acquisizione simultanea a largo campo di due immagini spettralmente diverse, come richiesto per un indicatore FRET. Il sistema ha fornito un sezionamento ottico intrinseco con esposizione minima della luce del campione, consentendo una rapida acquisizione volumetrica e l’acquisizione di immagini a lungo termine su un ampio campo visivo con dettagli a singola cellula. Le piantine sono state coltivate verticalmente in tubi di etilene propilene fluorurato riempito con mezzo MS a mezza resistenza, 0,1% (w / v) saccarosio; 0,05% (w / v) MES, pH 5,8 (Tris), solidificato da 0,5% (w / v) Phytagel come descritto in Candeo et al. (2017) e conservati in una camera di imaging riempito con mezzo liquido a base di MS privo di saccarosio (mezzo MS a mezza resistenza; 0,05% (w / v) MES; pH 5,8 (Tris)).
Il rapporto Venus/CFP sulla punta dei singoli peli della radice è stato estratto con FIJI (https://fiji.sc/) dopo la registrazione dell’immagine utilizzando il plugin delle Fiji ‘Template Matching’ (https://sites.google.com/site/qingzongtseng/template-matching-ij-plugin). Infine, la trasformata di Matlab Fast Fourier è stata utilizzata per calcolare le densità spettrali di potenza dai corsi normalizzati di Venus/CFP (Candeo et al., 2017). Per quantificare la velocità media di crescita dei peli delle radici abbiamo misurato il loro allungamento durante l’intera acquisizione e diviso per il tempo (10 min).
References
- Adolfsen R, Moudrianakis EN. Control of complex metal ion equilibria in biochemical reaction systems. Intrinsic and apparent stability constants of metal-adenine nucleotide complexes. The Journal of Biological Chemistry. 1978; 253:4378-4379. PubMed
- Allen GJ, Kwak JM, Chu SP, Llopis J, Tsien RY, Harper JF, Schroeder JI. Cameleon calcium indicator reports cytoplasmic calcium dynamics in Arabidopsis guard cells. The Plant Journal. 1999; 19:735-747. DOI | PubMed
- Ando T, Imamura H, Suzuki R, Aizaki H, Watanabe T, Wakita T, Suzuki T. Visualization and measurement of ATP levels in living cells replicating hepatitis C virus genome RNA. PLoS Pathogens. 2012; 8DOI | PubMed
- Attucci S, Carde JP, Raymond P, Saint-Gès V, Spiteri A, Pradet A. Oxidative phosphorylation by mitochondria extracted from dry sunflower seeds. Plant Physiology. 1991; 95:390-398. DOI | PubMed
- Bailleul B, Berne N, Murik O, Petroutsos D, Prihoda J, Tanaka A, Villanova V, Bligny R, Flori S, Falconet D, Krieger-Liszkay A, Santabarbara S, Rappaport F, Joliot P, Tirichine L, Falkowski PG, Cardol P, Bowler C, Finazzi G. Energetic coupling between plastids and mitochondria drives CO2 assimilation in diatoms. Nature. 2015; 524:366-369. DOI | PubMed
- Behera S, Wang N, Zhang C, Schmitz-Thom I, Strohkamp S, Schültke S, Hashimoto K, Xiong L, Kudla J. Analyses of Ca2+ dynamics using a ubiquitin-10 promoter-driven Yellow Cameleon 3.6 indicator reveal reliable transgene expression and differences in cytoplasmic Ca2+ responses in Arabidopsis and rice (Oryza sativa) roots. New Phytologist. 2015; 206:751-760. DOI | PubMed
- Berg J, Hung YP, Yellen G. A genetically encoded fluorescent reporter of ATP:ADP ratio. Nature Methods. 2009; 6:161-166. DOI | PubMed
- Bertani G. Studies on Lysogenesis. I. the mode of phage liberation by lysogenic Escherichia coli. Journal of Bacteriology. 1951; 62:293-300. PubMed
- Birk J, Ramming T, Odermatt A, Appenzeller-Herzog C. Green fluorescent protein-based monitoring of endoplasmic reticulum redox poise. Frontiers in Genetics. 2013; 4DOI | PubMed
- Birkenhead K, Walker D, Foyer C. The intracellular distribution of adenylate kinase in the leaves of spinach, wheat and barley. Planta. 1982; 156:171-175. DOI | PubMed
- Bradford MM. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 1976; 72:248-254. DOI | PubMed
- Brunoud G, Wells DM, Oliva M, Larrieu A, Mirabet V, Burrow AH, Beeckman T, Kepinski S, Traas J, Bennett MJ, Vernoux T. A novel sensor to map auxin response and distribution at high spatio-temporal resolution. Nature. 2012; 482:103-106. DOI | PubMed
- Busch K, Ninnemann H. Characterization of adenylate kinase in intact mitochondria of fertile and male sterile potato (Solanum tuberosum L.). Plant Science. 1996; 116:1-8. DOI
- Candeo A, Doccula FG, Valentini G, Bassi A, Costa A. Light sheet fluorescence microscopy quantifies calcium oscillations in root hairs of Arabidopsis thaliana. Plant and Cell Physiology. 2017. DOI | PubMed
- Chaudhuri B, Hörmann F, Lalonde S, Brady SM, Orlando DA, Benfey P, Frommer WB. Protonophore- and pH-insensitive glucose and sucrose accumulation detected by FRET nanosensors in Arabidopsis root tips. The Plant Journal. 2008; 56:948-962. DOI | PubMed
- Choi J, Tanaka K, Cao Y, Qi Y, Qiu J, Liang Y, Lee SY, Stacey G. Identification of a plant receptor for extracellular ATP. Science. 2014a; 343:290-294. DOI | PubMed
- Choi WG, Toyota M, Kim SH, Hilleary R, Gilroy S. Salt stress-induced Ca2+ waves are associated with rapid, long-distance root-to-shoot signaling in plants. PNAS. 2014b; 111:6497-6502. DOI | PubMed
- Clough SJ, Bent AF. Floral dip: a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana. The Plant Journal. 1998; 16:735-743. DOI | PubMed
- Considine MJ, Diaz-Vivancos P, Kerchev P, Signorelli S, Agudelo-Romero P, Gibbs DJ, Foyer CH. Learning to breathe: developmental phase transitions in oxygen status. Trends in Plant Science. 2017; 22:140-153. DOI | PubMed
- Costa A, Candeo A, Fieramonti L, Valentini G, Bassi A. Calcium dynamics in root cells of Arabidopsis thaliana visualized with selective plane illumination microscopy. PLoS One. 2013; 8DOI | PubMed
- Day DA, Arron GP, Laties GG. Enzyme distribution in potato mitochondria. Journal of Experimental Botany. 1979; 30:539-549. DOI
- De Michele R, Carimi F, Frommer WB. Mitochondrial biosensors. The International Journal of Biochemistry & Cell Biology. 2014; 48:39-44. DOI | PubMed
- Dejonghe W, Russinova E. Plant chemical genetics: from phenotype-based screens to synthetic biology. Plant Physiology. 2017; 174:5-20. DOI | PubMed
- Deuschle K, Chaudhuri B, Okumoto S, Lager I, Lalonde S, Frommer WB. Rapid metabolism of glucose detected with FRET glucose nanosensors in epidermal cells and intact roots of Arabidopsis RNA-silencing mutants. The Plant Cell Online. 2006; 18:2314-2325. DOI | PubMed
- Dickinson DB. Germination of lily pollen: respiration and tube growth. Science. 1965; 150:1818-1819. DOI | PubMed
- Earley KW, Haag JR, Pontes O, Opper K, Juehne T, Song K, Pikaard CS. Gateway-compatible vectors for plant functional genomics and proteomics. The Plant Journal. 2006; 45:616-629. DOI | PubMed
- Flügge UI. Metabolite transporters in plastids. Current Opinion in Plant Biology. 1998; 1:201-206. DOI | PubMed
- Fricker MD. Quantitative redox imaging software. Antioxidants & Redox Signaling. 2016; 24:752-762. DOI | PubMed
- Gardeström P, Igamberdiev AU. The origin of cytosolic ATP in photosynthetic cells. Physiologia Plantarum. 2016; 157:367-379. DOI | PubMed
- Gardeström P, Wigge B. Influence of Photorespiration on ATP/ADP ratios in the chloroplasts, mitochondria, and cytosol, studied by rapid fractionation of barley (Hordeum vulgare) Protoplasts. Plant Physiology. 1988; 88:69-76. DOI | PubMed
- Geigenberger P, Fernie AR, Gibon Y, Christ M, Stitt M. Metabolic activity decreases as an adaptive response to low internal oxygen in growing potato tubers. Biological Chemistry. 2000; 381:723-740. DOI | PubMed
- Geigenberger P. Response of plant metabolism to too little oxygen. Current Opinion in Plant Biology. 2003; 6:247-256. DOI | PubMed
- Gout E, Boisson A, Aubert S, Douce R, Bligny R. Origin of the cytoplasmic pH changes during anaerobic stress in higher plant cells. Carbon-13 and phosphorous-31 nuclear magnetic resonance studies. Plant Physiology. 2001; 125:912-925. DOI | PubMed
- Gout E, Rébeillé F, Douce R, Bligny R. Interplay of Mg2+, ADP, and ATP in the cytosol and mitochondria: unravelling the role of Mg2+ in cell respiration. PNAS. 2014; 111:E4560-E4567. DOI | PubMed
- Haferkamp I, Fernie AR, Neuhaus HE. Adenine nucleotide transport in plants: much more than a mitochondrial issue. Trends in Plant Science. 2011; 16:507-515. DOI | PubMed
- Hatsugai N, Perez Koldenkova V, Imamura H, Noji H, Nagai T. Changes in cytosolic ATP levels and intracellular morphology during bacteria-induced hypersensitive cell death as revealed by real-time fluorescence microscopy imaging. Plant and Cell Physiology. 2012; 53:1768-1775. DOI | PubMed
- Hepler PK, Vidali L, Cheung AY. Polarized cell growth in higher plants. Annual Review of Cell and Developmental Biology. 2001; 17:159-187. DOI | PubMed
- Igamberdiev AU, Bykova NV, Lea PJ, Gardeström P. The role of photorespiration in redox and energy balance of photosynthetic plant cells: a study with a barley mutant deficient in glycine decarboxylase. Physiologia Plantarum. 2001; 111:427-438. DOI | PubMed
- Imamura H, Nhat KP, Togawa H, Saito K, Iino R, Kato-Yamada Y, Nagai T, Noji H. Visualization of ATP levels inside single living cells with fluorescence resonance energy transfer-based genetically encoded indicators. PNAS. 2009; 106:15651-15656. DOI | PubMed
- Jones AM, Danielson JA, Manojkumar SN, Lanquar V, Grossmann G, Frommer WB. Abscisic acid dynamics in roots detected with genetically encoded FRET sensors. eLife. 2014; 3DOI | PubMed
- Karimi M, Inzé D, Depicker A. GATEWAY vectors for agrobacterium-mediated plant transformation. Trends in Plant Science. 2002; 7:193-195. DOI | PubMed
- Keinath NF, Waadt R, Brugman R, Schroeder JI, Grossmann G, Schumacher K, Krebs M. Live cell imaging with R-GECO1 sheds light on flg22- and chitin-Induced transient [Ca2+]cyt patterns in Arabidopsis. Molecular Plant. 2015; 8:1188-1200. DOI | PubMed
- Khlyntseva SV, Bazel’ YR, Vishnikin AB, Andruch V. Methods for the determination of adenosine triphosphate and other adenine nucleotides. Journal of Analytical Chemistry. 2009; 64:657-673. DOI
- Kim SY, Sivaguru M, Stacey G. Extracellular ATP in plants. Visualization, localization, and analysis of physiological significance in growth and signaling. Plant Physiology. 2006; 142:984-992. DOI | PubMed
- Kotera I, Iwasaki T, Imamura H, Noji H, Nagai T. Reversible dimerization of Aequorea victoria fluorescent proteins increases the dynamic range of FRET-based indicators. ACS Chemical Biology. 2010; 5:215-222. DOI | PubMed
- Krebs M, Held K, Binder A, Hashimoto K, Den Herder G, Parniske M, Kudla J, Schumacher K. FRET-based genetically encoded sensors allow high-resolution live cell imaging of Ca²⁺ dynamics. The Plant Journal. 2012; 69:181-192. DOI | PubMed
- Krömer S, Heldt HW. On the role of mitochondrial oxidative phosphorylation in photosynthesis metabolism as studied by the effect of oligomycin on photosynthesis in protoplasts and leaves of barley (Hordeum vulgare). Plant Physiology. 1991; 95:1270-1276. DOI | PubMed
- Krömer S, Malmberg G, Gardeström P. Mitochondrial contribution to photosynthetic metabolism (A study with barley (Hordeum vulgare L.) leaf protoplasts at different light intensities and CO2 concentrations). Plant Physiology. 1993; 102:947-955. DOI | PubMed
- Li J, Yu Q, Ahooghalandari P, Gribble FM, Reimann F, Tengholm A, Gylfe E. Submembrane ATP and Ca2+ kinetics in α-cells: unexpected signaling for glucagon secretion. The FASEB Journal. 2015; 29:3379-3388. DOI | PubMed
- Li S, Yamada M, Han X, Ohler U, Benfey PN. High-resolution expression map of the Arabidopsis root reveals alternative splicing and lincRNA regulation. Developmental Cell. 2016; 39:508-522. DOI | PubMed
- Lilley RM, Stitt M, Mader G, Heldt HW. Rapid fractionation of wheat leaf protoplasts using membrane filtration : the determination of metabolite levels in the chloroplasts, cytosol, and mitochondria. Plant Physiology. 1982; 70:965-970. DOI | PubMed
- Loef I, Stitt M, Geigenberger P. Increased levels of adenine nucleotides modify the interaction between starch synthesis and respiration when adenine is supplied to discs from growing potato tubers. Planta. 2001; 212:782-791. DOI | PubMed
- Logan DC, Leaver CJ. Mitochondria-targeted GFP highlights the heterogeneity of mitochondrial shape, size and movement within living plant cells. Journal of Experimental Botany. 2000; 51:865-871. DOI | PubMed
- Lorenz A, Lorenz M, Vothknecht UC, Niopek-Witz S, Neuhaus HE, Haferkamp I. In vitro analyses of mitochondrial ATP/phosphate carriers from Arabidopsis thaliana revealed unexpected Ca2+-effects. BMC Plant Biology. 2015; 15DOI | PubMed
- Loro G, Drago I, Pozzan T, Schiavo FL, Zottini M, Costa A. Targeting of Cameleons to various subcellular compartments reveals a strict cytoplasmic/mitochondrial Ca²⁺ handling relationship in plant cells. The Plant Journal. 2012; 71:1-13. DOI | PubMed
- Loro G, Wagner S, Doccula FG, Behera S, Weinl S, Kudla J, Schwarzländer M, Costa A, Zottini M. Chloroplast-specific in vivo Ca2+ imaging using Yellow Cameleon fluorescent protein sensors reveals organelle-autonomous Ca2+ signatures in the stroma. Plant Physiology. 2016; 171:2317-2330. DOI | PubMed
- Luo Y, Scholl S, Doering A, Zhang Y, Irani NG, Di Rubbo S, Neumetzler L, Krishnamoorthy P, Van Houtte I, Mylle E, Bischoff V, Vernhettes S, Winne J, Friml J, Stierhof YD, Schumacher K, Persson S, Russinova E. V-ATPase activity in the TGN/EE is required for exocytosis and recycling in Arabidopsis. Nature Plants. 2015; 1:15094. DOI | PubMed
- Mandl I, Grauer A, Neuberg C. Solubilization of insoluble matter in nature. Biochimica Et Biophysica Acta. 1952; 8:654-663. DOI | PubMed
- Manfredi G, Yang L, Gajewski CD, Mattiazzi M. Measurements of ATP in mammalian cells. Methods. 2002; 26:317-326. DOI | PubMed
- Marty L, Siala W, Schwarzländer M, Fricker MD, Wirtz M, Sweetlove LJ, Meyer Y, Meyer AJ, Reichheld JP, Hell R. The NADPH-dependent thioredoxin system constitutes a functional backup for cytosolic glutathione reductase in Arabidopsis. PNAS. 2009; 106:9109-9114. DOI | PubMed
- Merrins MJ, Poudel C, McKenna JP, Ha J, Sherman A, Bertram R, Satin LS. Phase analysis of metabolic oscillations and membrane potential in pancreatic islet β-Cells. Biophysical Journal. 2016; 110:691-699. DOI | PubMed
- Meyer AJ, Brach T, Marty L, Kreye S, Rouhier N, Jacquot JP, Hell R. Redox-sensitive GFP in Arabidopsis thaliana is a quantitative biosensor for the redox potential of the cellular glutathione redox buffer. The Plant Journal. 2007; 52:973-986. DOI | PubMed
- Monné M, Miniero DV, Obata T, Daddabbo L, Palmieri L, Vozza A, Nicolardi MC, Fernie AR, Palmieri F. Functional characterization and organ distribution of three mitochondrial ATP-Mg/Pi carriers in Arabidopsis thaliana. Biochimica Et Biophysica Acta (BBA) – Bioenergetics. 2015; 1847:1220-1230. DOI | PubMed
- Monshausen GB, Messerli MA, Gilroy S. Imaging of the Yellow Cameleon 3.6 indicator reveals that elevations in cytosolic Ca2+ follow oscillating increases in growth in root hairs of Arabidopsis. Plant Physiology. 2008; 147:1690-1698. DOI | PubMed
- Morgan B, Ezeriņa D, Amoako TN, Riemer J, Seedorf M, Dick TP. Multiple glutathione disulfide removal pathways mediate cytosolic redox homeostasis. Nature Chemical Biology. 2013; 9:119-125. DOI | PubMed
- Morgan B, Van Laer K, Owusu TN, Ezeriņa D, Pastor-Flores D, Amponsah PS, Tursch A, Dick TP. Real-time monitoring of basal H2O2 levels with peroxiredoxin-based probes. Nature Chemical Biology. 2016; 12:437-443. DOI | PubMed
- Murashige T, Skoog F. A revised medium for rapid growth and bioassays with tobacco tissue cultures. Physiologia Plantarum. 1962; 15:473-497. DOI
- Mustroph A, Boamfa EI, Laarhoven LJ, Harren FJ, Albrecht G, Grimm B. Organ-specific analysis of the anaerobic primary metabolism in rice and wheat seedlings. I: Dark ethanol production is dominated by the shoots. Planta. 2006; 225:103-114. DOI | PubMed
- Nakano M, Imamura H, Nagai T, Noji H. Ca²⁺ regulation of mitochondrial ATP synthesis visualized at the single cell level. ACS Chemical Biology. 2011; 6:709-715. DOI | PubMed
- Neuhaus HE, Thom E, Möhlmann T, Steup M, Kampfenkel K. Characterization of a novel eukaryotic ATP/ADP translocator located in the plastid envelope of Arabidopsis thaliana L. The Plant Journal. 1997; 11:73-82. DOI | PubMed
- O’Sullivan WJ, Perrin DD. The stability constants of MgATP2-. Biochimica Et Biophysica Acta. 1961; 52:612-614. DOI | PubMed
- Palmer AE, Tsien RY. Measuring calcium signaling using genetically targetable fluorescent indicators. Nature Protocols. 2006; 1:1057-1065. DOI | PubMed
- Patel A, Malinovska L, Saha S, Wang J, Alberti S, Krishnan Y, Hyman AA. ATP as a biological hydrotrope. Science. 2017; 356:753-756. DOI | PubMed
- Pei ZM, Murata Y, Benning G, Thomine S, Klüsener B, Allen GJ, Grill E, Schroeder JI. Calcium channels activated by hydrogen peroxide mediate abscisic acid signalling in guard cells. Nature. 2000; 406:731-734. DOI | PubMed
- Pradet A, Raymond P. Adenine nucleotide ratios and adenylate energy charge in energy metabolism. Annual Review of Plant Physiology. 1983; 34:199-224. DOI
- Ratcliffe RG. In vivo NMR studies of the metabolic response of plant tissues to anoxia. Annals of Botany. 1997; 79:39-48. DOI
- Reiser J, Linka N, Lemke L, Jeblick W, Neuhaus HE. Molecular physiological analysis of the two plastidic ATP/ADP transporters from Arabidopsis. Plant Physiology. 2004; 136:3524-3536. DOI | PubMed
- Rich P. Chemiosmotic coupling: the cost of living. Nature. 2003; 421DOI | PubMed
- Roberts J, Aubert S, Gout E, Bligny R, Douce R. Cooperation and competition between adenylate kinase, nucleoside diphosphokinase, electron transport, and atp synthase in plant mitochondria studied by 31P-nuclear magnetic resonance. Plant Physiology. 1997; 113:191-199. DOI | PubMed
- Rosenwasser S, Rot I, Meyer AJ, Feldman L, Jiang K, Friedman H. A fluorometer-based method for monitoring oxidation of redox-sensitive GFP (roGFP) during development and extended dark stress. Physiologia Plantarum. 2010; 138:493-502. DOI | PubMed
- Rosenwasser S, Rot I, Sollner E, Meyer AJ, Smith Y, Leviatan N, Fluhr R, Friedman H. Organelles contribute differentially to reactive oxygen species-related events during extended darkness. Plant Physiology. 2011; 156:185-201. DOI | PubMed
- Schulte A, Lorenzen I, Böttcher M, Plieth C. A novel fluorescent pH probe for expression in plants. Plant Methods. 2006; 2DOI | PubMed
- Schulz GE. Structural and functional relationships in the adenylate kinase family. Cold Spring Harbor Symposia on Quantitative Biology. 1987; 52:429-439. DOI | PubMed
- Schwarzländer M, Dick TP, Meyer AJ, Morgan B. Dissecting redox biology using fluorescent protein sensors. Antioxidants & Redox Signaling. 2016; 24:680-712. DOI | PubMed
- Schwarzländer M, Fricker MD, Müller C, Marty L, Brach T, Novak J, Sweetlove LJ, Hell R, Meyer AJ. Confocal imaging of glutathione redox potential in living plant cells. Journal of Microscopy. 2008; 231:299-316. DOI | PubMed
- Schwarzländer M, Fricker MD, Sweetlove LJ. Monitoring the in vivo redox state of plant mitochondria: effect of respiratory inhibitors, abiotic stress and assessment of recovery from oxidative challenge. Biochimica Et Biophysica Acta (BBA) – Bioenergetics. 2009; 1787:468-475. DOI | PubMed
- Schwarzländer M, Logan DC, Fricker MD, Sweetlove LJ. The circularly permuted yellow fluorescent protein cpYFP that has been used as a superoxide probe is highly responsive to pH but not superoxide in mitochondria: implications for the existence of superoxide ‘flashes’. Biochemical Journal. 2011; 437:381-387. DOI | PubMed
- Schwarzländer M, Logan DC, Johnston IG, Jones NS, Meyer AJ, Fricker MD, Sweetlove LJ. Pulsing of membrane potential in individual mitochondria: a stress-induced mechanism to regulate respiratory bioenergetics in Arabidopsis. The Plant Cell. 2012; 24:1188-1201. DOI | PubMed
- Stitt M, Lilley RM, Heldt HW. Adenine nucleotide levels in the cytosol, chloroplasts, and mitochondria of wheat leaf protoplasts. Plant Physiology. 1982; 70:971-977. DOI | PubMed
- Storer AC, Cornish-Bowden A. Concentration of MgATP2- and other ions in solution. Calculation of the true concentrations of species present in mixtures of associating ions. Biochemical Journal. 1976; 159:1-5. DOI | PubMed
- Sweetlove LJ, Heazlewood JL, Herald V, Holtzapffel R, Day DA, Leaver CJ, Millar AH. The impact of oxidative stress on Arabidopsis mitochondria. The Plant Journal. 2002; 32:891-904. DOI | PubMed
- Sweetlove LJ, Nielsen J, Fernie AR. Engineering central metabolism – a grand challenge for plant biologists. The Plant Journal. 2017; 90:749-763. DOI | PubMed
- Sweetlove LJ, Taylor NL, Leaver CJ. Isolation of intact, functional mitochondria from the model plant Arabidopsis thaliana. Methods in Molecular Biology. 2007; 372:125-136. DOI | PubMed
- Taiz L, Zeiger E, Møller IM, Murphy A. Plant Physiology and Development. Sunderland, CT: Sinauer Associates; 2015.
- Tanaka K, Gilroy S, Jones AM, Stacey G. Extracellular ATP signaling in plants. Trends in Cell Biology. 2010; 20:601-608. DOI | PubMed
- Tantama M, Martínez-François JR, Mongeon R, Yellen G. Imaging energy status in live cells with a fluorescent biosensor of the intracellular ATP-to-ADP ratio. Nature Communications. 2013; 4DOI | PubMed
- Tarasov AI, Semplici F, Ravier MA, Bellomo EA, Pullen TJ, Gilon P, Sekler I, Rizzuto R, Rutter GA. The mitochondrial Ca2+ uniporter MCU is essential for glucose-induced ATP increases in pancreatic β-cells. PLoS One. 2012; 7DOI | PubMed
- Uslu VV, Grossmann G. The biosensor toolbox for plant developmental biology. Current Opinion in Plant Biology. 2016; 29:138-147. DOI | PubMed
- van Dongen JT, Fröhlich A, Ramírez-Aguilar SJ, Schauer N, Fernie AR, Erban A, Kopka J, Clark J, Langer A, Geigenberger P. Transcript and metabolite profiling of the adaptive response to mild decreases in oxygen concentration in the roots of Arabidopsis plants. Annals of Botany. 2009; 103:269-280. DOI | PubMed
- van Dongen JT, Licausi F. Oxygen sensing and signaling. Annual Review of Plant Biology. 2015; 66:345-367. DOI | PubMed
- van Dongen JT, Schurr U, Pfister M, Geigenberger P. Phloem metabolism and function have to cope with low internal oxygen. Plant Physiology. 2003; 131:1529-1543. DOI | PubMed
- Waadt R, Hitomi K, Nishimura N, Hitomi C, Adams SR, Getzoff ED, Schroeder JI. FRET-based reporters for the direct visualization of abscisic acid concentration changes and distribution in Arabidopsis. eLife. 2014; 3DOI | PubMed
- Wagner S, Behera S, De Bortoli S, Logan DC, Fuchs P, Carraretto L, Teardo E, Cendron L, Nietzel T, Füßl M, Doccula FG, Navazio L, Fricker MD, Van Aken O, Finkemeier I, Meyer AJ, Szabò I, Costa A, Schwarzländer M. The EF-hand Ca2+ binding protein MICU choreographs mitochondrial Ca2+dynamics in Arabidopsis. The Plant Cell. 2015b; 27:3190-3212. DOI | PubMed
- Wagner S, Nietzel T, Aller I, Costa A, Fricker MD, Meyer AJ, Schwarzländer M. Analysis of plant mitochondrial function using fluorescent protein sensors. Methods in Molecular Biology. 2015a; 1305:241-252. DOI | PubMed
- Wend S, Dal Bosco C, Kämpf MM, Ren F, Palme K, Weber W, Dovzhenko A, Zurbriggen MD. A quantitative ratiometric sensor for time-resolved analysis of auxin dynamics. Scientific Reports. 2013; 3DOI | PubMed
- Wirtz M, Hell R. Production of cysteine for bacterial and plant biotechnology: application of cysteine feedback-insensitive isoforms of serine acetyltransferase. Amino Acids. 2003; 24:195-203. DOI | PubMed
- Xia JH, Saglio PH. Lactic acid efflux as a mechanism of hypoxic acclimation of maize root tips to Anoxia. Plant Physiology. 1992; 100:40-46. DOI | PubMed
- Yagi H, Kajiwara N, Tanaka H, Tsukihara T, Kato-Yamada Y, Yoshida M, Akutsu H. Structures of the thermophilic F1-ATPase epsilon subunit suggesting ATP-regulated arm motion of its C-terminal domain in F1. PNAS. 2007; 104:11233-11238. DOI | PubMed
- Yang H, Bogner M, Stierhof YD, Ludewig U. H-independent glutamine transport in plant root tips. PLoS One. 2010; 5DOI | PubMed
- Yoshida T, Kakizuka A, Imamura H. BTeam, a novel BRET-based biosensor for the accurate quantification of ATP concentration within living cells. Scientific Reports. 2016; 6DOI | PubMed
- Zabalza A, van Dongen JT, Froehlich A, Oliver SN, Faix B, Gupta KJ, Schmälzlin E, Igal M, Orcaray L, Royuela M, Geigenberger P. Regulation of respiration and fermentation to control the plant internal oxygen concentration. Plant Physiology. 2009; 149:1087-1098. DOI | PubMed
- Zancani M, Casolo V, Vianello A, Macrì F. Involvement of apyrase in the regulation of the adenylate pool by adenylate kinase in plant mitochondria. Plant Science. 2001; 161:927-933. DOI
Fonte
De Col V, Fuchs P, Nietzel T, Elsässer M, Voon CP, et al. () ATP sensing in living plant cells reveals tissue gradients and stress dynamics of energy physiology. eLife 6e26770. https://doi.org/10.7554/eLife.26770





