Abstract
I fenotipi biologici non sono fenomeni a singolo gene, ma sono invece funzioni di complesse interazioni tra molteplici loci, l’ambiente e il caso. Inoltre, i fenotipi, anche se governati da singoli loci ad effetto maggiore, sono generalmente sensibili al background genetico, cioè alla variazione molecolare attraverso il genoma (Ayroleset al.2009; Chandleret al.2013; Chari e Dworkin 2013). Inoltre, i diversi background genetici variano nella loro capacità di tamponare le perturbazioni genetiche e ambientali, contribuendo ulteriormente alla variazione di tratti complessi (Chandleret al.2013; Chari e Dworkin 2013). I loci che agiscono come modificatori di tratti complessi lo fanno attraverso l’interazione con la variazione nel resto del genoma; di conseguenza, reti biologiche più complesse, che coinvolgono numerosi geni, sono probabilmente più suscettibili di effetti genetici di fondo rispetto a reti più semplici (Spenceret al.2003; Chandleret al.2013; Chari e Dworkin 2013; Binget al.2014).
Molti tratti complessi mostrano anche dimorfismo sessuale, o variazione specifica del sesso nei fenotipi, comprese le differenze nella morfologia, fisiologia, biochimica, comportamento o strategia di storia della vita (Ranzet al.2003; Fairbairn e Roff 2006; Giordaniaet al.2007; Yamamotoet al.2009; Assiset al.2012). Inoltre, il dimorfismo sessuale è una componente della determinazione del sesso, la gerarchia degli eventi che porta allo sviluppo delle caratteristiche sessuali (Clough e Oliver 2012). Nella Drosophila melanogaster, la determinazione del sesso è controllata da una cascata di geni e la regolazione differenziale dei loci durante la differenziazione sessuale potrebbe contribuire alla variazione indotta dal background genetico (Cloughet al.2014).
La comprensione della traduzione da genotipo a fenotipo si basa sulla comprensione delle complesse interazioni che avvengono all’interno e tra i loci. Questa comprensione è limitata e compromessa quando gli studi utilizzano un background singolo, isogenico o un singolo sesso (Phillipset al.1989; Radyuket al.2004; Bernardoet al.2011). Detto questo, può essere difficile definire e quantificare accuratamente i fenotipi appropriati con cui valutare il background genetico e gli effetti sul sesso. La comprensione attuale delle interazioni genetiche si basa in gran parte sull’esame degli effetti degli alleli mutanti sui fenotipi [ad esempio, SOD; Parkeset al.(1998a,b) e Bernardet al.(2011)], in ambienti e contesti genetici controllati. Questo approccio semplifica il modo in cui vengono analizzati gli effetti mutazionali, ma anche il modo in cui vengono interpretati gli effetti allelici [recensione in Chandleret al.(2013)]. È invece importante rendersi conto che la variazione del background genetico può contribuire alla variazione fenotipica, anche quando si esamina l’influenza di un singolo allele mutante [ad esempio, Rzezniczak e Merritt (2012) e Binget al.(2014)].
Nella maggior parte degli organismi, la SOD, come componente della rete di difesa degli enzimi antiossidanti (Phillipset al.1989; Michielset al.1994), è il principale spazzino delle specie reattive dell’ossigeno (ROS) superossido (O2-; McCord e Fridovich 1969). I ROS sono una classe biologicamente importante di molecole prodotte sia come sottoprodotti metabolici che in modo coordinato per la regolazione metabolica essenziale (Ozcan e Ogun 2015), e il bilanciamento dei ROS è fondamentale per la funzione cellulare. Nel metabolismo del ROS, la SOD converte il superossido in perossido di idrogeno (H2O2), che la catalisi e una varietà di perossidasi riducono ulteriormente in acqua (Phillipset al.1989; Michielset al.1994). Come l’anione superossido è in gran parte membrana impermeabile, produce principalmente effetti locali (Bafanaet al.2011). Esistono quindi diverse isoforme di SOD anche localizzate nella cellula, che si differenziano per i meccanismi catalitici, i nuclei ionici metallici e la funzione fisiologica, ognuna delle quali si trova a scavare il superossido di scavenge in compartimenti cellulari distinti (Bafanaet al.2011). cSOD, la più abbondante isoforma della SOD eucariotica, si trova nel citosol, nello spazio intermembrana mitocondriale, nei lisosomi e nel nucleo (Zelkoet al.2002; Bafanaet al.2011). Una seconda isoforma SOD, la SOD mitocondriale (manganese) (MnSOD), è localizzata nella matrice mitocondriale e funziona indipendentemente dalla cSOD (Duttaroyet al.1997; Zelkoet al.2002).
D. melanogaster privo di attività cSOD (cSOD-nullo omozigoti) non sono in grado di metabolizzare il superossido, e quindi esistono in uno stato di stress ossidativo cronico (Bernardet al.2011), dando luogo a una serie di condizioni patologiche note collettivamente come sindrome cSOD-nulla (Phillipset al.1989; Parkeset al.1998a). Questa sindrome è caratterizzata da una serie di fenotipi che vanno da differenze comportamentali e di storia della vita [ad esempio, cambiamenti nella locomozione, Martinet al.(2009) e Jones e Grotewiel (2011); redditività, Sole e torre (1999); e longevità, Phillipset al.(1989), e Parkeset al.(1998b)], alle differenze biochimiche [ad es.et al.(2013) e l’attività dell’enzima NADP(H), Bernardet al.(2011)]. Sebbene la funzione cSOD sia spesso studiata quantificando i cambiamenti fenotipici, prevalentemente nei maschi con background isogenico (Parkeset al.1998a; Bernardoet al.2011; Ginocchioet al.2013), i fenotipi della sindrome suggeriscono che il sistema è meglio studiato come tratto complesso. La vitalità e la longevità, i fenotipi della storia della vita e la geotassi negativa, un fenotipo locomotorio, sono depressi nelle mosche cSOD-nulle (Phillipset al.1989; Parkeset al.1998b; Sole e Torre 1999; Martinet al.2009). Allo stesso modo, gli enzimi NADP(H), l’enzima malico (MEN), l’isocitrato deidrogenasi (IDH), e il glucosio-6-fosfato deidrogenasi (G6PD), hanno ridotto l’attività nelle mosche maschio cSOD-nulle (Bernardet al.2011). Questi enzimi riducono il NADP+ a NADP(H), un cofattore chiave utilizzato dalla catalisi e dagli antiossidanti dipendenti dalla glutatione nel perossido di idrogeno prodotto dalla SOD (Kanzoket al.2001). Inoltre, molti fenotipi cSOD, tra cui la longevità (Spenceret al.2003), locomozione (Giordaniaet al.2007; Yamamotoet al.2009), e la biochimica (Ranzet al.2003; Merrittet al.2009), sono noti per avere una componente sessuale specifica. Riconoscere i limiti dell’esame delle interazioni all’interno di sistemi genetici relativamente semplici ha portato alla realizzazione della necessità di migliorare la nostra comprensione del funzionamento dei geni in sistemi genetici complessi e biologicamente rilevanti.
In questo articolo, esaminiamo l’influenza del sesso e del background genetico su un insieme di storia della vita, locomotore e fenotipi biochimici associati al cSOD in una serie di attività cSOD: 0-80% di tipo selvaggio (WT). Abbiamo ipotizzato che le differenze nello stato ROS tra le attività cSOD si manifesterebbero come variazioni in risposta al sesso e al background genetico, con background genetici diversi che potrebbero migliorare o potenziare gli effetti fenotipici associati alla sindrome cSOD-nulla. I nostri risultati mostrano che le differenze su larga scala nell’attività cSOD portano a cambiamenti fenotipici pervasivi, e che questi cambiamenti sono significativamente modificati, sia potenziati che soppressi, dalle differenze di sesso e di background genetico. Livelli più elevati di attività cSOD in generale hanno portato a fenotipi più vicini al WT, che erano più suscettibili di modificazione per sesso e background genetico.
Materiali e metodi
Stock e linee di volo
Le linee di sostituzione del secondo cromosoma sono state generate utilizzando l’introgressione assistita da marker e un sottoinsieme strategicamente selezionato di otto linee isofemminali del D. melanogaster Genetic Reference Panel (DGRP) stabilito dal Mackay Lab (Mackayet al.2012): 304, 307, 313, 324, 335, 517, 705 e 820. Il sottoinsieme è stato scelto utilizzando i dati disponibili del fenotipo DGRP (Mackayet al.2012), per includere una linea di alta e una di bassa magnitudine ciascuna per quattro fenotipi legati al cSOD: longevità, fitness, sensibilità al paraquat e risposta all’avvio. Abbiamo inserito questi otto cromosomi del secondo cromosoma DGRP in un background genetico comune: w1118DGRPi/CyO; VT83, con ” i” che è il 2° cromosoma di una delle linee DGRP e VT83 che è un 3° cromosoma isolato dall’ambiente naturale (Merrittet al.2006).
In D. melanogaster, il locus cSOD è sul 3° cromosoma, e l’enzima cSOD attivo è un omodimero (Campbellet al.1986). Abbiamo usato un set di alleli cSOD-null e WT cSOD, precedentemente descritti da Campbellet al.(1986), Phillipset al.(1989), e Parkeset al.(1998a,b). In breve, la linea cSOD-nulla (T0) possiede il cSodn108allele rosso nullo sul 3° cromosoma (Parkeset al.1998a,b), mentre la linea di controllo parallela (T5) possiede questo stesso allele nullo sul 3° cromosoma in combinazione con un transgene cSod sul 2° cromosoma, sotto il controllo del promotore cSod nativo, creando un intero organismo transgenico linea di soccorso (Parkeset al.1998a,b). Le linee T0 e T5 si differenziano solo per l’assenza o la presenza del transgene. Quando è omozigote, il transgene ripristina le prospettive per il 2% dell’attività del WT cSOD (Parkeset al.1998a,b) e le mosche T5 sono state fenotipicamente indistinguibili dalle mosche WT cSOD negli studi precedenti (Parkeset al.1998b; Bernardoet al.2011).
Per quantificare gli effetti dell’attività cSOD, del sesso e del background genetico sui fenotipi, abbiamo incrociato le linee T0 e T5 con le linee del cromosoma iso 2º cromosoma sostituite in background per generare due intervalli di attività cSOD: un intervallo basso (0 e 50% dell’attività WT vola) e un intervallo medio (30 e 80% dell’attività WT vola). Gli schemi distinti sono stati progettati per estrarre sia il 2° che il 3° cromosoma dalle linee T0 (bassa attività cSOD; Figura 1, A-C) e T5 (moderata attività cSOD; Figura 1, D-F) perché gli elementi che contribuiscono all’attività cSOD si trovano in entrambi i cromosomi (2° per il transgene e 3° per il locus nativo). Lo Schema 1 incrocia la progenie creata con lo 0 o il 50% dell’attività cSOD del WT (w+T0/DGRPi; csodn108,rosso/ csodn108rosso e w+T0/DGRPi; csodn108, rosso/VT83, rispettivamente; Figura 1, Ci e Cii), mentre lo Schema 2 incrocia la progenie creata con il 30 o l’80% dell’attività del WT cSOD (w+T5/DGRPi; csodn108rosso /csodn108rosso e w+T5/DGRPi; csodn108rosso/VT83, rispettivamente; Figura 1, Fi e Fii). Per ogni test fenotipico, abbiamo generato la progenie dei genotipi appropriati attraverso cinque incroci replicati di ogni schema di incrocio e background genetico (due schemi, otto background e cinque replicati = 80 incroci separati). Tutte le mosche sperimentali sono state assunte come non-virgin in quanto sono state raccolte 48 ore dopo l’eclosione. I ceppi sono disponibili su richiesta.
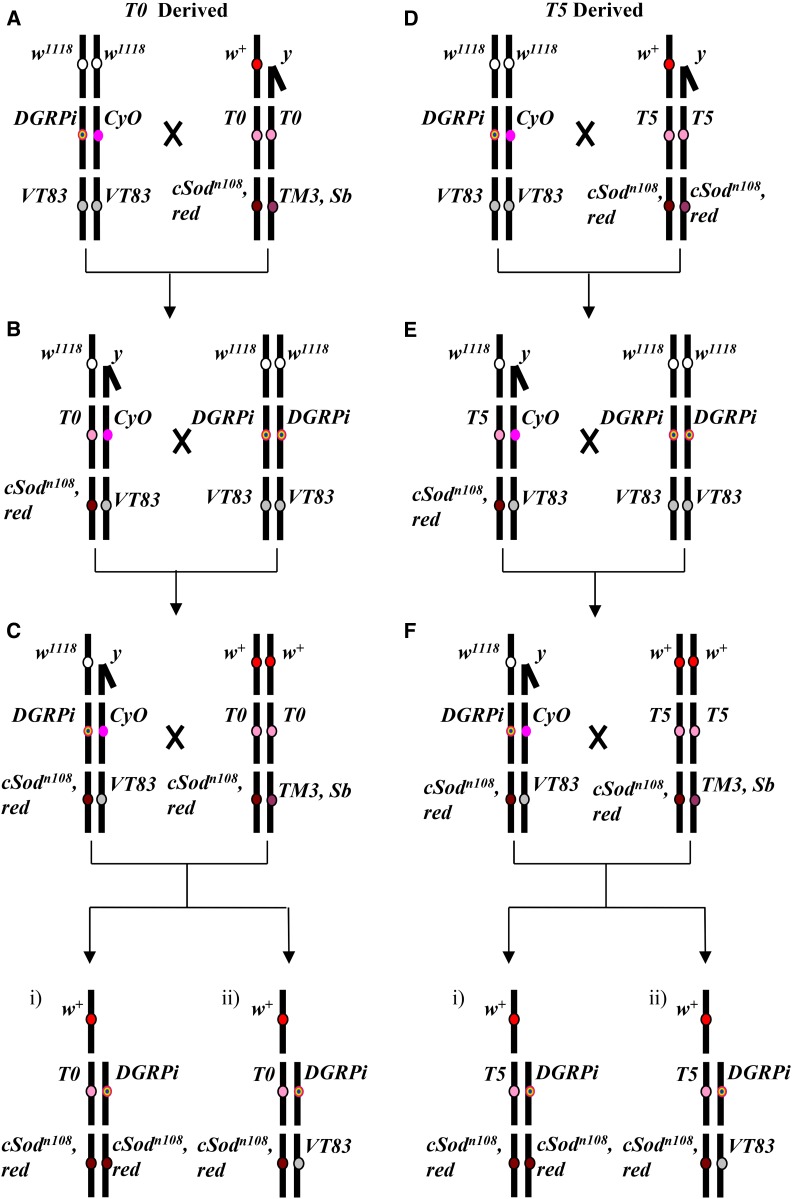
Figura 1.Schemi di attraversamento utilizzati per generare mosche cSOD 0 e 50% (A-C) e mosche cSOD 30 e 80% (D-F). In tutti gli incroci gli adulti sono stati collocati su supporti di mais freschi, sciroppo di mais, sciroppo di mais e lievito di agar e sono stati autorizzati ad accoppiarsi e a deporre le uova per 1 settimana, spostati su supporti freschi per 1 settimana e poi scartati. Gli incroci (A, D) e (B, E) sono stati allestiti utilizzando due mosche maschio e due mosche femmina vergini. Maschi singoli sono stati incrociati a due femmine vergini in croce (C, F) come progenie maschio da croce (B, E) che esprime il fenotipo dell’ala arricciata potrebbe aver posseduto uno dei due genotipi, solo uno dei quali è stato voluto per croce (C, F). L’uso di maschi singoli ha impedito di mescolare maschi con genotipi diversi e ha permesso di scartare gli incroci che erano stati impostati con il genotipo sbagliato. Incrocio (C) generato (i) w+;T0/DGRPi;csodn108rosso/csodn108rosso (0% attività cSOD di tipo selvaggio) e (ii) w+T0/DGRPi;csodn108rosso/VT83 (50% attività cSOD di tipo selvaggio). La croce (C) produce anche w+;T0/CyO;csodn108rosso/csodn108, rosso, w+;T0/CyO; csodn108, rosso/VT83, w+;T0/CyO; csodn108,rosso/TM3, ser, w+;T0/CyO; VT83/TM3, ser, w+;T0/DGRPicsodn108, rosso/TM3, ser, e w+;T0/DGRPiVT83/TM3, sierotipi. Croce (F) generata (i) w+;T5/DGRPi;csodn108rosso/csodn108rosso (30% attività cSOD di tipo selvaggio) e (ii) w+T5/DGRPi;csodn108rosso/VT83 (80% attività cSOD di tipo selvaggio). La croce (F) produce anche w+;T5/CyO;csodn108rosso/csodn108rosso, rosso, e w+;T5/CyO; csodn108rosso / VT83 progenie. Attraverso i genotipi, le mosche femmine erano eterozigoti al cromosoma X per w+/w1118, mentre i maschi erano emizygous per la w+ allele. I quattro genotipi che portano il TM3, Sb 3° equilibratore cromosomico, prodotto da (C), si distinguono per un fenotipo di setole di stoppia e sono stati rimossi dall’analisi in quanto i quattro genotipi comprendono due fenotipi che sono visivamente indistinguibili e possiedono diversi livelli di attività cSOD. I genotipi che portano il cromosoma CyO, prodotto in (C) e (F), sono stati esclusi dalle analisi in quanto i risultati preliminari suggerivano che il cromosoma equilibratore stava falsamente guidando le differenze genetiche di fondo. cSOD, rame citosolico / zinco superossido dismutasi.

Figura 1.Schemi di attraversamento utilizzati per generare mosche cSOD 0 e 50% (A-C) e mosche cSOD 30 e 80% (D-F). In tutti gli incroci gli adulti sono stati collocati su supporti di mais freschi, sciroppo di mais, sciroppo di mais e lievito di agar e sono stati autorizzati ad accoppiarsi e a deporre le uova per 1 settimana, spostati su supporti freschi per 1 settimana e poi scartati. Gli incroci (A, D) e (B, E) sono stati allestiti utilizzando due mosche maschio e due mosche femmina vergini. Maschi singoli sono stati incrociati a due femmine vergini in croce (C, F) come progenie maschio da croce (B, E) che esprime il fenotipo dell’ala arricciata potrebbe aver posseduto uno dei due genotipi, solo uno dei quali è stato voluto per croce (C, F). L’uso di maschi singoli ha impedito di mescolare maschi con genotipi diversi e ha permesso di scartare gli incroci che erano stati impostati con il genotipo sbagliato. Incrocio (C) generato (i) w+;T0/DGRPi;csodn108rosso/csodn108rosso (0% attività cSOD di tipo selvaggio) e (ii) w+T0/DGRPi;csodn108rosso/VT83 (50% attività cSOD di tipo selvaggio). La croce (C) produce anche w+;T0/CyO;csodn108rosso/csodn108, rosso, w+;T0/CyO; csodn108, rosso/VT83, w+;T0/CyO; csodn108,rosso/TM3, ser, w+;T0/CyO; VT83/TM3, ser, w+;T0/DGRPicsodn108, rosso/TM3, ser, e w+;T0/DGRPiVT83/TM3, sierotipi. Croce (F) generata (i) w+;T5/DGRPi;csodn108rosso/csodn108rosso (30% attività cSOD di tipo selvaggio) e (ii) w+T5/DGRPi;csodn108rosso/VT83 (80% attività cSOD di tipo selvaggio). La croce (F) produce anche w+;T5/CyO;csodn108rosso/csodn108rosso, rosso, e w+;T5/CyO; csodn108rosso / VT83 progenie. Attraverso i genotipi, le mosche femmine erano eterozigoti al cromosoma X per w+/w1118, mentre i maschi erano emizygous per la w+ allele. I quattro genotipi che portano il TM3, Sb 3° equilibratore cromosomico, prodotto da (C), si distinguono per un fenotipo di setole di stoppia e sono stati rimossi dall’analisi in quanto i quattro genotipi comprendono due fenotipi che sono visivamente indistinguibili e possiedono diversi livelli di attività cSOD. I genotipi che portano il cromosoma CyO, prodotto in (C) e (F), sono stati esclusi dalle analisi in quanto i risultati preliminari suggerivano che il cromosoma equilibratore stava falsamente guidando le differenze genetiche di fondo. cSOD, rame citosolico / zinco superossido dismutasi.
Saggio di vitalità genotipica
La vitalità genotipica è stata valutata come descritto da Merrittet al.(2006) utilizzando le mosche generate dalle croci mostrate in Figura 1, C e F). Le mosche adulte di ogni incrocio sono state contate per 1 wk (7 d) dalla prima eclosione, e le frequenze di occorrenza di ogni genotipo sono state calcolate sul numero totale di mosche raccolte per ogni incrocio. A causa della variazione di successo tra le linee, il numero totale di animali analizzati variava tra 2660 mosche e 1928 mosche, per il confronto 0 e 50% e il confronto 30 e 80%, rispettivamente.
Saggio di longevità
La longevità, basata sulla mortalità del genotipo, è stata misurata come descritto da Parkeset al.(1998b). Mosche adulte di ogni genotipo sono state raccolte 48 ore dopo l’eclosione e trasferite in fiale standard da 25 × 95 mm con un massimo di 20 mosche per fiala. La longevità è stata misurata in fiale mono-sesso, mono-genotipo, mantenute a 25°, 12 ore di luce: ciclo scuro – e la mortalità è stata registrata ogni 2 d. Le mosche sono state trasferite su supporti freschi ogni 4 d fino a quando non sono rimaste mosche vive. A causa della variazione di successo tra le linee, il numero totale di animali analizzati variava tra 2260 mosche e 1705 mosche, per il confronto 0 e 50% e il confronto 30 e 80%, rispettivamente.
Saggio di geotassi negativa
La geotassi negativa per adulti (di seguito semplicemente “geotassi”) è stata misurata utilizzando un test modificato da Patel e Tamanoi (2006) e Sofolaet al.(2010). In breve, cinque repliche di 15 mosche di ogni genotipo sono state raccolte 48 ore dopo l’eclosione, e invecchiate per 3 d in fiale monosessuali, monotipo, monotipo. In seguito all’invecchiamento, i gruppi di mosche sono stati trasferiti in fiale vuote di 25 × 95 mm con un’altezza di 5 cm e hanno permesso di recuperare 30 secondi. Dopo il recupero, le mosche sono state picchiettate fino al fondo della fiala e hanno permesso di salire per 10 secondi. Dopo 10 secondi, il numero di mosche sopra e sotto i 5 cm è stato contato ad occhio. Per ogni fiala, sono state eseguite tre prove di picchiettio verso il basso, ciascuna a intervalli di 1 min. Un indice di prestazione (PI), una stima della probabilità che una mosca si arrampichi, è stato calcolato per ogni genotipo seguendo Sofolaet al.(2010). I valori PI vanno da zero a uno; valori vicini a uno indicano che le mosche hanno una risposta geotassica elevata, mentre valori vicini a zero indicano che le mosche hanno una scarsa risposta geotassica (Sofolaet al.2010). PI è stato calcolato come PI = 1/2 (nTOTAL+ nTOP – nBOTTOM / nTOTAL), dove nTOTAL è il numero totale di voli, nTOP è il numero di voli sopra la linea dei 5 cm e nBOTTOM è il numero di voli sotto la linea dei 5 cm (Sofolaet al.2010). A causa della variazione di successo tra le linee, il numero totale di animali analizzati variava tra 2168 mosche e 1923 mosche, rispettivamente per il confronto 0 e 50% e il confronto 30 e 80%.
Poiché le mosche cSOD-nulle sono sorprendentemente povere di arrampicatori, abbiamo modificato il saggio di geotassi utilizzando una distanza di arrampicata più breve di quella tipicamente impiegata (Patel e Tamanoi 2006; Sofolaet al.2010). Tuttavia, questa modifica potrebbe ridurre la capacità del saggio di rilevare piccole differenze geotassiche nelle mosche ad alte prestazioni (ad esempio, animali con un’attività di 30, 50 e 80% di cSOD). Una tale riduzione del potere risolutivo potrebbe portare a non rilevare alcuna differenza tra gli sfondi, anche se esistono piccole differenze. Pertanto, abbiamo condotto un secondo test locomotorio, il test di arrampicata in controcorrente (Petersenet al.2013), per valutare più da vicino le potenziali differenze di performance tra i genotipi.
Saggio di arrampicata in controcorrente
La capacità di arrampicata degli adulti è stata misurata utilizzando un test di arrampicata in controcorrente (di seguito semplicemente “controcorrente”), come descritto da Petersenet al.(2013). L’apparecchio per controcorrente è costituito da due serie di quattro fiale a conchiglia nastrate insieme, con il set inferiore etichettato 1-4 e il set superiore etichettato 5-8 (Materiale supplementare, Figura S1 nel file S1; Petersenet al.2013). Cinque repliche di ∼15 mosche adulte di ogni genotipo e sesso sono state raccolte 48 ore dopo l’eclosione, combinate e invecchiate per 3 d in fiale standard da 25 × 95 mm in fiale monosessuali, misto-genotipo, in fiale. Per ogni fiala di test, è stato contato il numero di mosche di ogni genotipo, fuori dal gruppo di genotipi misti, post saggio (Benzer 1967). Le mosche invecchiate sono state caricate nella fiala 1 e la fiala 8 è stata invertita sulla fiala 1 (Figura S1B nel file S1), le fiale sono state toccate verso il basso, e le mosche hanno avuto 1 minuto per salire. Dopo 1 minuto, il set superiore di fiale è stato spostato sopra, le fiale sono state picchiettate verso il basso, e le mosche hanno avuto 1 minuto per salire (Figura S1B nel file S1). Questo processo è stato ripetuto un totale di sette volte, con fiale non contrastate da altre fiale tappate per evitare che le mosche fuoriescano (Figura S1B nel File S1). Le mosche sono state classificate in base alla capacità di arrampicarsi in base alle fiale in cui si trovavano alla fine della prova: scarsa = fiala 1, moderata = fiale 2-4, o buona = fiale 5-8 (Figura S1B nel File S1; Petersenet al.2013). Per ogni genotipo è stato calcolato un coefficiente di partizione (CF), una stima della probabilità che una mosca esca dalla sua fiala di partenza ad ogni prova, per ogni genotipo (Kamikouchiet al.2009). I valori di CF vanno da zero a uno; valori vicini a uno indicano che le mosche tendono a salire, mentre valori vicini a zero indicano che le mosche tendono a non salire (Kamikouchiet al.2009). CF è stato calcolato come CF = ƩNk(k-1)/(n-1)ƩNk, dove n = il numero di classi di arrampicata, Nk è il numero di mosche nella classe di arrampicata kth, e alle classi di arrampicata sono stati assegnati i valori k poveri (k = 1), moderati (k = 2) e buoni (k = 3; Kamikouchiet al.2009). Le analisi sono state ripetute utilizzando la fiala che ogni mosca è finita in una classe (otto classi potenziali) e i risultati sono stati coerenti con le analisi binned (discusse di seguito). Di conseguenza, mostriamo solo i dati e i risultati delle analisi binned. A causa della variazione di successo tra le linee, il numero totale di animali analizzati variava tra 2106 mosche e 1962 mosche, rispettivamente per il confronto 0 e 50% e il confronto 30 e 80%.
Saggi di attività enzimatica
Per tutti i test enzimatici, le mosche adulte sono state raccolte 48 ore dopo l’eclosione, invecchiate per 3 d, e congelate a -80° in gruppi di quattro mosche di un solo sesso e di un solo genotipo. Prima dell’omogeneizzazione, i campioni sono stati pesati con una microbilancia con un’approssimazione di 0,01 mg (MX5 Balance, Mettler Toledo AG, Greifensee, Svizzera). Tutte le reazioni enzimatiche sono state eseguite in una piastra microtiter standard a 96 pozzetti e l’assorbanza è stata misurata con uno spettrofotometro a micropiastra (SpectraMax Plus 384, Dispositivi Molecolari, Sunnyvale, CA).
Saggi di attività enzimatica MEN, IDH e G6PD:
I saggi degli enzimi NADP(H) sono stati eseguiti come descritto da Merrittet al.(2006) e Bernardet al.(2011). In breve, i campioni di mosca sono stati omogeneizzati in 100 μl di tampone omogeneizzante per mosca (0,1 M TRIS-HCl pH 7,4 e 0,01 M NADP +), centrifugato a 13.000 giri al minuto per 12 minuti a 4 °, e il supernatante raccolto. All’interno di ogni pozzetto micropiastra, la miscela di reazione consisteva di 10 microlitri di campione supernatante e 100 microlitri della soluzione di analisi (MEN: 0,1 M TRIS-HCl pH 7,4, 10 mM malato, 5 mM MnCl2, e 0.34 mM NADP+; IDH: 0,1 M TRIS-HCl pH 8,6, 1,37 mM isocitrato, 0,84 mM MgSO4, e 0,1 mM NADP+; e G6PD: 20 mM TRIS-HCl pH 7,4, 3,5 mM G6P, e 0,2 mM NADP+). NADP(H) prodotto è stato quantificato come un aumento dell’assorbanza misurata a 340 nm. Per le reazioni MEN e IDH, l’assorbanza è stata misurata a 25° ogni 9 sec per 3 min. Per la reazione G6PD, l’assorbanza è stata misurata a 25° ogni 9 sec per 5 min. Le attività enzimatiche per ogni campione di genotipo sono state calcolate in base all’attività enzimatica media di tre repliche tecniche. Il numero totale di animali analizzati è stato di 1184 mosche e 1248 mosche, per il confronto 0 e 50% e il confronto 30 e 80%, rispettivamente. I geni per MEN e IDH in D. melanogaster si trovano sul 3° cromosoma, mentre il gene per G6PD si trova sul cromosoma X. Pertanto, le differenze nel background genetico del 2° cromosoma non cambieranno gli alleli MEN, IDH e G6PD presenti nelle mosche sperimentali.
test di attività enzimatica cSOD:
L’attività cSOD è stata quantificata utilizzando un kit di dosaggio commerciale (Cayman Chemical Superoxide Dismutase Assay Kit, Ann Arbor, MI, numero di prodotto: 706002) seguendo il protocollo suggerito dal produttore con modifiche minime. In breve, i campioni di mosca sono stati omogeneizzati in freddo 20 mM HEPES tampone HEPES (pH 7,4, 1 mM EDTA, 210 mM mannitolo, e 70 mM saccarosio) ad un rapporto di 100 μl / fly e poi diluito ad un rapporto finale di 400 μl / fly. Gli omogeneizzati di mosca sono stati centrifugati a 4° per 10 minuti a 3000 rpm e il supernatante raccolto [i campioni sono centrifugati a un numero di giri inferiore a quello dei campioni NADP(H) per evitare la pellettatura della proteina MnSOD]. Prima del saggio, il supernatante è stato diluito in un rapporto di 1:4 supernatante al tampone del campione del saggio. Per ogni campione, sono state eseguite due reazioni distinte per determinare l’attività cSOD: una per l’attività totale di SOD e una per l’attività MnSOD. L’attività cSOD viene poi calcolata come differenza tra le due. L’attività cSOD viene calcolata in questo modo perché il saggio cSOD non può distinguere tra le due isoforme cSOD, quindi è necessario un inibitore per determinare l’attività di una singola isoforma. Per l’attività totale di SOD (MnSOD e cSOD), la miscela di reazione consisteva di 10 μl del surnatante campione diluito, 200 μl di rivelatore di radicali e 20 μl di xantina ossidasi. Per l’attività MnSOD, ogni reazione consisteva di 10 μl del supernatante campione diluito, 190 μl di rivelatore di radicali, 20 μl di xantina ossidasi, e 10 μl di 5 mM di cianuro di sodio (che inattiva la cSOD attraverso la destabilizzazione del complesso Cu/Zn). Tutte le reazioni sono state incubate a temperatura ambiente per 30 minuti e poi l’assorbanza del punto finale è stata misurata a 25 ° e 450 nm con uno spettrofotometro micropiastra. Ogni campione è stato saggiato due volte per ogni reazione (attività totale SOD e attività MnSOD) e le attività SOD campione sono stati calcolati utilizzando l’equazione generata dallo standard SOD (eritrociti bovini Cu / Zn SOD) curva. Il numero totale di animali analizzati è stato di 636 mosche e 624 mosche, rispettivamente per il confronto 0 e 50% e il confronto 30 e 80%.
Concentrazione proteica totale
La concentrazione totale di proteine è stata quantificata con un dosaggio di acido bicinconinico utilizzando un kit commerciale (Pierce, Thermo Scientific, Rockford, IL, numero di prodotto 23225) con modifiche a seguito di Rzezniczak e Merritt (2012). All’interno di ogni pozzetto della micropiastra, la reazione consisteva di 10 μl di supernatante del campione e 100 μl di reagente del test. Le micropiastre sono state incubate a 37° per 20 minuti e lasciate raffreddare prima delle letture di assorbanza. Assorbanza punto finale è stata misurata a 562 nm e a 25 ° con uno spettrofotometro micropiastra. Concentrazioni proteiche totali sono state calcolate per confronto con una curva standard generata utilizzando 1200 μg/ml, 800 μg/ml, 400 μg/ml, e 100 μg/ml di albumina di siero bovino. Ogni campione è stato analizzato tre volte e le concentrazioni medie delle proteine del campione sono state utilizzate per standardizzare le attività enzimatiche.
Analisi statistica
ANOVA è stato usato per dividere la variazione fenotipica tra l’attività cSOD, il sesso, il background genetico, le loro interazioni, e la varianza di errore [y = cSOD + sesso + background genetico + cSOD*sesso + cSOD* background genetico + sesso* background genetico + cSOD*sesso* background genetico + errore; Leips e Mackay (2000), e Ayroleset al.(2009)]. I confronti post hoc sono stati effettuati utilizzando il test HSD di Tukey. Tutte le analisi sono state eseguite utilizzando il software statistico JMP 12. Sono stati effettuati confronti separati sui dati dell’incrocio Scheme 1 (0 e 50% di attività cSOD) e dell’incrocio Scheme 2 (30 e 80% di attività cSOD) in quanto sono stati condotti in modo indipendente e i genotipi ottenuti da questi incroci differivano (a causa della presenza o dell’assenza del transgene cSOD sul 2° cromosoma nelle linee T0 e T5). Tuttavia, sono state rilevate tendenze generali in tutti e quattro i livelli di attività e i due confronti.
La dimensione dell’effetto misura l’eta parziale al quadrato (η 2p) è stata utilizzata per stimare la grandezza della variazione attribuibile a ciascun fattore e interazione. η 2p misura la proporzione della variazione attribuibile a un particolare fattore, eliminando la variazione spiegata da altre variabili predittive. η 2p è stato determinato utilizzando l’equazione η 2p = SSFactor / SSFactor + SSError (Pierceet al.2004) per ogni fattore, e l’interazione utilizzando la Somma dei valori dei quadrati prodotti nell’uscita ANOVA.
Disponibilità dei dati
Tutti i ceppi sono disponibili su richiesta. Gli autori dichiarano che tutti i dati necessari per confermare le conclusioni presentate nell’articolo sono rappresentati integralmente all’interno dell’articolo.
Risultati
Qui quantifichiamo gli effetti del background genetico e del sesso nel modificare l’effetto dell’attività cSOD su una serie di fenotipi sensibili al cSOD. Esaminando più background genetici tra i sessi, siamo stati in grado di suddividere la quantità di variazione attribuibile all’attività cSOD, al sesso, al background genetico e alle loro interazioni, per ogni fenotipo. Abbiamo usato la dimensione dell’effetto η 2p per quantificare la proporzione di variazione attribuibile a ciascun fattore, e tutti η 2p e le relative statistiche si trovano nella Tabella S1, Tabella S2, Tabella S3, Tabella S4, Tabella S5, Tabella S6 e Tabella S7 nel File S1. Le tabelle riassuntive degli effetti massimi e minimi delle righe, i valori medi del genotipo e i valori massimi e minimi delle righe sono illustrati rispettivamente nella tabella S8, nella tabella S9 e nella tabella S10 del file S1.
L’attività cSOD è controllata dal genotipo cSOD, ma non dai modificatori del secondo cromosoma dominante
Abbiamo generato genotipi con una gamma di livelli di attività cSOD, attraverso gli stessi otto sfondi del 2° cromosoma (DGRPi), utilizzando due schemi di incrocio separati (Figura 1). L’attività cSOD per ogni genotipo è stata inizialmente stimata in base al genotipo cSOD complessivo; tuttavia, altri studi che utilizzano linee di mosca isofemminile hanno dimostrato che il background genetico del 2° cromosoma può influenzare l’attività cSOD (Graf e Ayala 1986). Per quantificare la potenziale variazione di background nell’attività cSOD risultante dai distinti cromosomi del 2° cromosoma DGRP, abbiamo misurato l’attività cSOD in tutti i genotipi e non abbiamo trovato alcuna influenza significativa del background genetico sull’attività cSOD (Figura S2 e Figura S3 nel File S1). Le differenze su larga scala nell’attività cSOD sono quindi una funzione del genotipo complessivo cSOD con solo piccole, statisticamente insignificanti variazioni nel background genetico. Di seguito, ci riferiamo ai diversi genotipi per le loro attività cSOD previste (e osservate): 0, 50, 30 e 80% di attività cSOD. È interessante notare che nel confronto 30-80% c’è stato un effetto significativo del sesso sull’attività cSOD, con un’attività significativamente più bassa nei maschi rispetto alle femmine, anche se il dimorfismo sessuale nell’espressione cSOD non è osservato nel locus cSOD nativo (Gnad e Parsch 2006; discusso di seguito).
I fenotipi della storia della vita sono maggiormente influenzati dall’attività cSOD con una sensibilità variabile al sesso e al background genetico
Come previsto, i punteggi dei fenotipi della storia della vita sono stati più bassi allo 0% di attività cSOD e più alti a una moderata attività cSOD (Figura 2 e Figura 3). I punteggi erano simili tra il 50 e l’80% dei gruppi di attività cSOD, suggerendo che solo il 50% di attività cSOD era necessario per recuperare i fenotipi a livelli WT. L’attività cSOD ha avuto un effetto maggiore rispetto al sesso o al background genetico sia sulla vitalità che sulla longevità nel confronto 0-50%. Infatti, la vitalità era influenzata solo dall’attività cSOD, mentre la longevità era anche sensibile al sesso e al background genetico (Figura 4 e Figura 5). Le mosche maschio e femmina con il 50% di attività cSOD hanno vissuto più a lungo dei cSOD-nulli (Figura 2). Inoltre, le femmine con il 50% di attività cSOD hanno vissuto più a lungo dei maschi, anche se l’entità del dimorfismo sessuale variava a seconda del background genetico (Figura 4). In particolare, mentre una tendenza comparabile di femmine più longeve è stata osservata anche nelle mosche a 0% di attività cSOD, le analisi post hoc hanno indicato che non c’era una differenza significativa nella longevità dei cSOD-nulli a seconda del sesso o del background genetico (Figura S4 nel File S1), che potrebbe riflettere la gravità della condizione di knockout cSOD. Tuttavia, la mancanza di effetti di modifica sui cSOD-nulli è specifica del fenotipo in quanto vi erano effetti significativi sul sesso e sul background genetico in altri fenotipi (sotto).
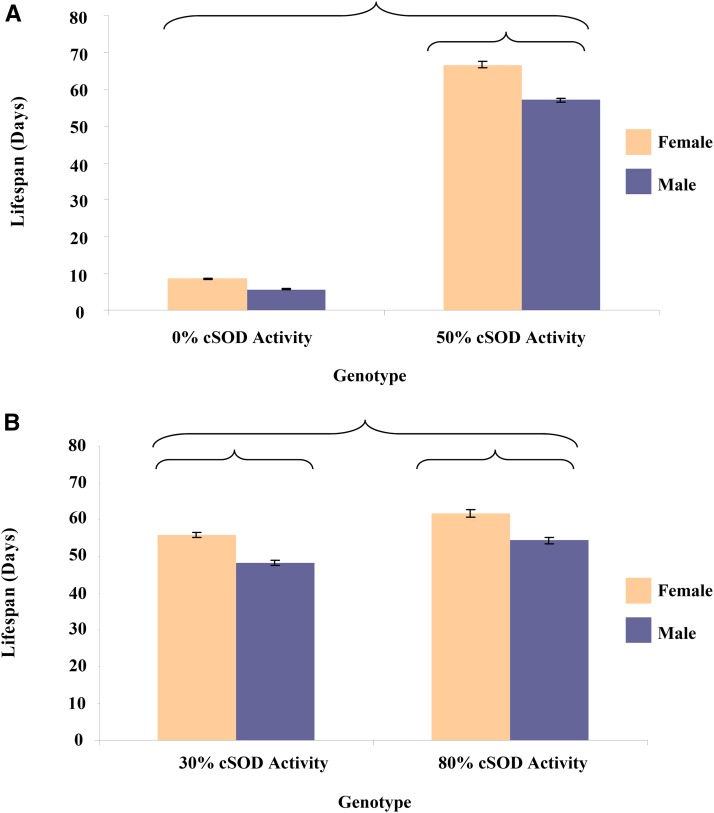
Figura 2.Longevità media ± SEM (d) per le mosche adulte, maschio e femmina, raggruppate negli otto background genetici DGRPi all’interno di ciascun 3° genotipo del cromosoma. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) Sesso per attività di CSOD: F1,2227 = 35,2, P < 0,0001, ηp2 = 0.016. (B) attività cSOD: F1,1673 = 64,9, P < 0,0001, ηp2 = 0,038; Sesso: F1,1673 = 66,1, P < 0,0001, ηp2 = 0.038. Le staffe racchiudono gruppi significativamente diversi. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.
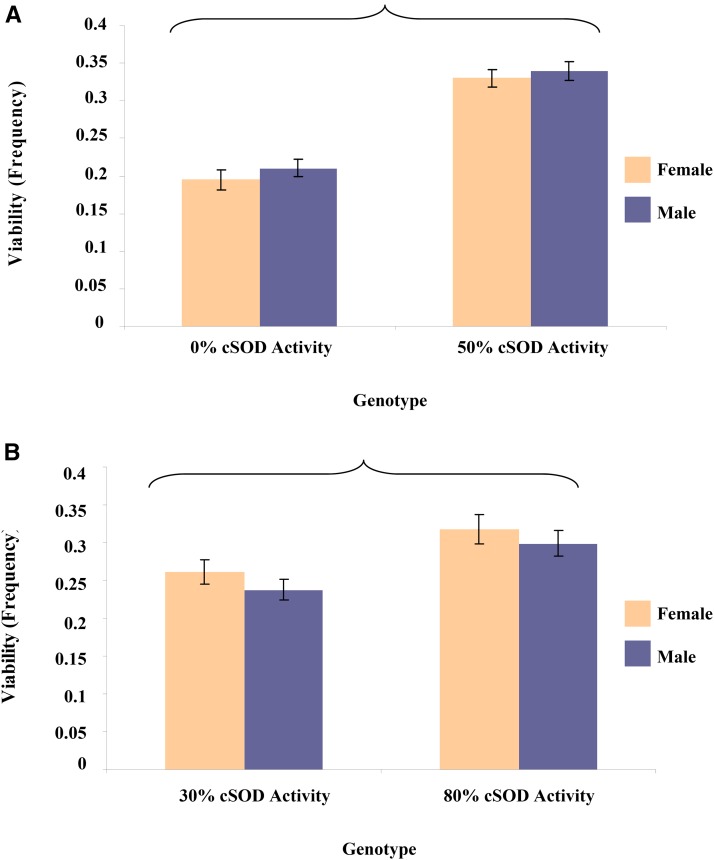
Figura 3.Media ± SEM di vitalità (frequenza) per mosche adulte messe in comune attraverso gli otto background genetici DGRPi all’interno di ogni 3 ° genotipo cromosomico. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) attività cSOD: F1,143 = 106,7, P < 0,0001, ηp2 = 0.43. (B) attività cSOD: F1,148 = 11,2, P = 0,001, ηp2 = 0.070. Le staffe racchiudono gruppi significativamente diversi. cSOD, rame citosolico / zinco superossido dismutasi.
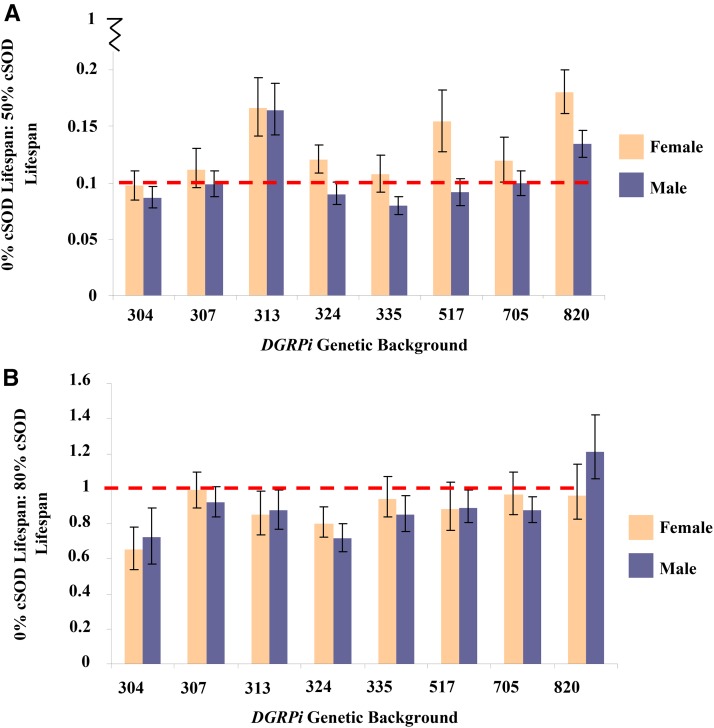
Figura 4.Media ± SEM del rapporto di bassa cSOD: alta cSOD longevità di attività cSOD per le mosche adulte maschili e femminili attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano longevità più simili ai livelli di attività cSOD messi a confronto, e i C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEM. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 304, 0,0846 < 0,0972 < 0,111; rapporto più alto 820, 0,162 < 0,180 < 0,201; Maschio: rapporto più basso 335, 0,0715 < 0,0793 < 0,0873, rapporto più alto 313, 0,143 < 0,165 < 0,188 (B) 30% attività cSOD: 80% attività cSOD: Femmina: rapporto più basso 304, 0,535 < 0,650 < 0,780; rapporto più alto 307, 0,889 < 0,990 < 1,10; Maschio: rapporto più basso 324, 0,642 < 0,720 < 0,803; rapporto più alto 820, 1,05 < 1,21 < 1,42. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ciascun fattore. (A) Sesso per attività di background genetico, F7,2227 = 4,38, P < 0,0001, ηp2 = 0.0136. (B) Sfondo sessuale per ogni attività genetica, F7,1673 = 2,91, P = 0,0049, ηp2 = 0,0121; background genetico per attività del CSOD; F7,1673 = 6,03, P < 0,0001, ηp2 = 0,0246. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.
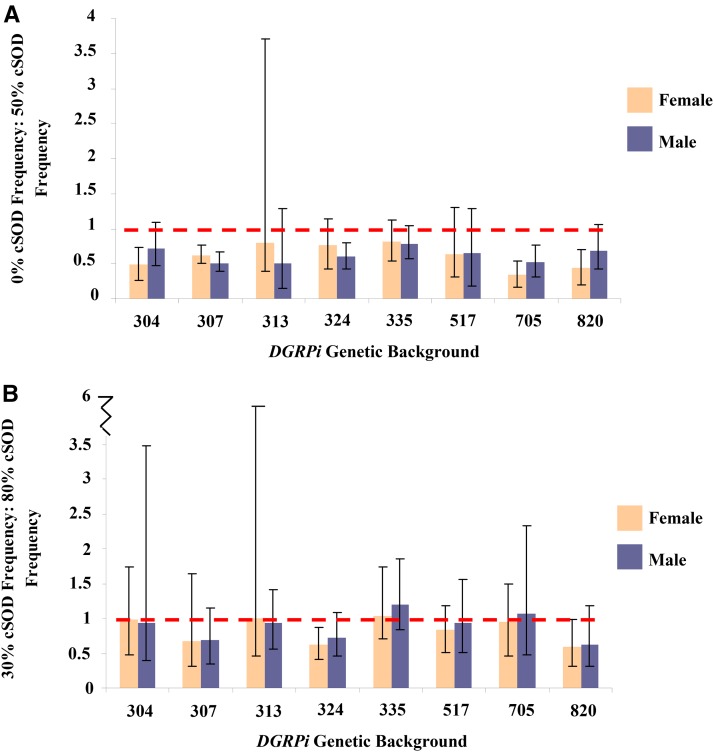
Figura 5.Media ± SEM del rapporto di bassa cSOD: alta vitalità di attività cSOD (frequenza) per adulti maschi e femmine vola attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano una viabilità più simile ai livelli di attività cSOD da confrontare; C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEMs. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 705, 0,168 < 0,344 < 0,532; rapporto più alto 335, 0,538 < 0,819 < 1,12: Maschio: rapporto più basso 313, 0,147 < 0,498 < 1,28; rapporto più alto 335, 0.575 < 0,781 < 1,05 (B) 30% attività cSOD: 80% attività cSOD, Femmina: rapporto più basso 820, 0,309 < 0,586 < 0,979; rapporto più alto 335, 0,699 < 1,04 < 1,74: Maschio: rapporto più basso 304, 0,0162 < 0,492 < 0,974; rapporto più alto 335, 0,720 < 0,902 < 1,08. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) attività cSOD, F1,143 = 106,7, P < 0,0001, ηp2 = 0.427. (B) attività cSOD, F1,148 = 11,2, P = 0,001, ηp2 = 0,0704. cSOD, cSOD, rame citosolico / zinco superossido dismutasi.
Nel confronto 30-80%, l’attività cSOD ha avuto ancora un grande effetto sulla vitalità e sulla longevità, con punteggi per entrambi i fenotipi inferiori al 30% di cSOD rispetto all’80% di cSOD (Figura 2 e Figura 3). Coerentemente con il basso confronto cSOD (sopra), la vitalità è stata influenzata dall’attività cSOD solo nel confronto cSOD moderato, mentre la longevità è stata anche sensibile al sesso e al background genetico (Figura S4 nel File S1). L’80% delle mosche con attività cSOD maschili e femminili ha vissuto più del 30% di mosche con attività cSOD. Come nel confronto a basso contenuto di cSOD, le femmine hanno vissuto più a lungo dei maschi attraverso le attività cSOD, con l’entità del dimorfismo sessuale nella longevità che varia a seconda del background genetico (Figura 4). Anche l’effetto del background genetico sulla longevità variava tra le attività cSOD. È interessante notare che, mentre l’attività cSOD ha avuto un effetto significativo sui fenotipi della storia della vita attraverso i confronti, la dimensione dell’effetto del cSOD sui fenotipi era più piccola quando era presente una maggiore attività cSOD (Figura 6).
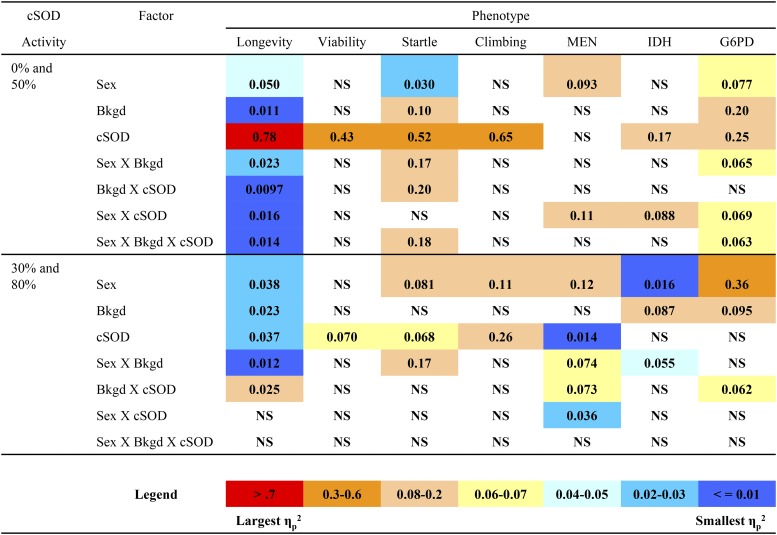
Figura 6.Eta parziale al quadrato (ηp2) valori per longevità, vitalità, geotassi negativa, controcorrente (arrampicata), enzima malico (MEN), isocitrato deidrogenasi (IDH), e glucosio-6-fosfato deidrogenasi (G6PD), calcolati per ogni fattore in condizioni di attività cSOD (rame citosolico / superossido di zinco dismutasi). Valori più grandi rappresentano effetti più grandi (arance), e valori più piccoli rappresentano effetti più piccoli (blues). NS, fattori o interazioni non significativi.

Figura 2.Media ± longevità SEM (d) per le mosche adulte maschio e femmina messe in comune attraverso gli otto background genetici DGRPi all’interno di ogni genotipo del 3° cromosoma. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) Sesso per attività di CSOD: F1,2227 = 35,2, P < 0,0001, ηp2 = 0.016. (B) attività cSOD: F1,1673 = 64,9, P < 0,0001, ηp2 = 0,038; Sesso: F1,1673 = 66,1, P < 0,0001, ηp2 = 0.038. Le staffe racchiudono gruppi significativamente diversi. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.

Figura 3.Media ± SEM di vitalità (frequenza) per mosche adulte messe in comune attraverso gli otto background genetici DGRPi all’interno di ogni 3 ° genotipo cromosomico. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) attività cSOD: F1,143 = 106,7, P < 0,0001, ηp2 = 0.43. (B) attività cSOD: F1,148 = 11,2, P = 0,001, ηp2 = 0.070. Le staffe racchiudono gruppi significativamente diversi. cSOD, rame citosolico / zinco superossido dismutasi.

Figura 4.Media ± SEM del rapporto di bassa cSOD: alta cSOD longevità di attività cSOD per le mosche adulte maschili e femminili attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano longevità più simili ai livelli di attività cSOD messi a confronto, e i C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEM. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 304, 0,0846 < 0,0972 < 0,111; rapporto più alto 820, 0,162 < 0,180 < 0,201; Maschio: rapporto più basso 335, 0,0715 < 0,0793 < 0,0873, rapporto più alto 313, 0,143 < 0,165 < 0,188 (B) 30% attività cSOD: 80% attività cSOD: Femmina: rapporto più basso 304, 0,535 < 0,650 < 0,780; rapporto più alto 307, 0,889 < 0,990 < 1,10; Maschio: rapporto più basso 324, 0,642 < 0,720 < 0,803; rapporto più alto 820, 1,05 < 1,21 < 1,42. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ciascun fattore. (A) Sesso per attività di background genetico, F7,2227 = 4,38, P < 0,0001, ηp2 = 0.0136. (B) Sfondo sessuale per ogni attività genetica, F7,1673 = 2,91, P = 0,0049, ηp2 = 0,0121; background genetico per attività del CSOD; F7,1673 = 6,03, P < 0,0001, ηp2 = 0,0246. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.

Figura 5.Media ± SEM del rapporto di bassa cSOD: alta vitalità di attività cSOD (frequenza) per adulti maschi e femmine vola attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano una viabilità più simile ai livelli di attività cSOD da confrontare; C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEMs. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 705, 0,168 < 0,344 < 0,532; rapporto più alto 335, 0,538 < 0,819 < 1,12: Maschio: rapporto più basso 313, 0,147 < 0,498 < 1,28; rapporto più alto 335, 0.575 < 0,781 < 1,05 (B) 30% attività cSOD: 80% attività cSOD, Femmina: rapporto più basso 820, 0,309 < 0,586 < 0,979; rapporto più alto 335, 0,699 < 1,04 < 1,74: Maschio: rapporto più basso 304, 0,0162 < 0,492 < 0,974; rapporto più alto 335, 0,720 < 0,902 < 1,08. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) attività cSOD, F1,143 = 106,7, P < 0,0001, ηp2 = 0.427. (B) attività cSOD, F1,148 = 11,2, P = 0,001, ηp2 = 0,0704. cSOD, cSOD, rame citosolico / zinco superossido dismutasi.

Figura 6.Eta parziale al quadrato (ηp2) valori per longevità, vitalità, geotassi negativa, controcorrente (arrampicata), enzima malico (MEN), isocitrato deidrogenasi (IDH), e glucosio-6-fosfato deidrogenasi (G6PD), calcolati per ogni fattore in condizioni di attività cSOD (rame citosolico / superossido di zinco dismutasi). Valori più grandi rappresentano effetti più grandi (arance), e valori più piccoli rappresentano effetti più piccoli (blues). NS, fattori o interazioni non significativi.
I fenotipi locomotori sono maggiormente influenzati dall’attività cSOD e dal sesso, ma variano in termini di sensibilità al background genetico
Come per i fenotipi della storia di vita, le prestazioni in entrambi i saggi locomotori sono state minime allo 0% di attività cSOD e superiori a una moderata attività cSOD (Figura 7). La capacità locomotoria è stata simile tra il 50 e l’80% di attività cSOD, indicando ancora una volta che solo il 50% di attività cSOD è stato necessario per recuperare i fenotipi a livelli indistinguibili dal WT. Inoltre, coerentemente con i fenotipi della storia della vita, l’attività cSOD ha avuto un effetto maggiore rispetto al sesso o al background genetico sia sulla geotassi che sulla locomozione controcorrente nel confronto 0-50% (Figura S5 e Figura S6 nel File S1). Le mosche maschio e femmina con il 50% di attività cSOD hanno avuto una migliore capacità locomotoria rispetto ai cSOD-nulli, attraverso entrambi i fenotipi (Figura S7 nel File S1). È interessante notare che i maschi avevano una migliore capacità geotassica rispetto alle femmine in entrambe le attività cSOD, anche se l’entità del dimorfismo sessuale variava a seconda del background genetico (Figura S5 nel File S1). Mentre non c’era un effetto significativo del sesso sulla locomozione controcorrente nel confronto a basso cSOD, abbiamo notato una tendenza simile a quella osservata nella geotassi; i maschi avevano una migliore capacità di arrampicarsi rispetto alle femmine (Figura S6 nel File S1).
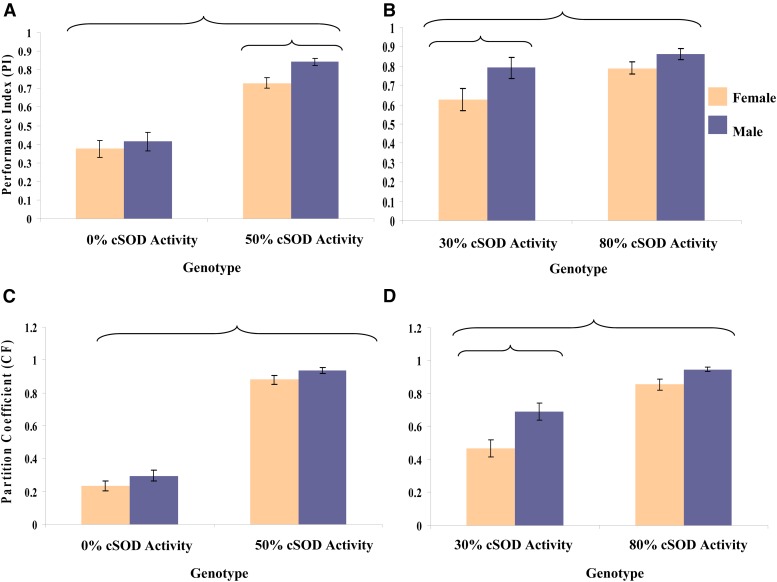
Figura 7.(A e B) Media ± SEM dell’indice negativo di prestazione della geotassi (PI), e (C e D) media ± SEM del coefficiente di ripartizione rampicante (CF), per mosche adulte maschio e femmina in pool attraverso gli otto background genetici DGRPi all’interno di ogni genotipo. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) cSOD Attività, F1,156 = 169,6, P < 0,0001, ηp2 = 0,52; sesso, F1,156 = 4,83, P = 0,0294, ηp2 = 0.030. (B) cSOD Attività, F1,100 = 7,33, P = 0,008, ηp2 = 0,0683; sesso – F1,100 = 8,80, P = 0,0038, ηp2 = 0.0809. (C) attività cSOD, F1,269 = 501,0, P < 0,0001, ηp2 = 0.651. (D) attività cSOD, F1,175 = 61,6, P < 0,0001, ηp2 = 0,260; sesso – F1,175 = 20,8, P < 0,0001, ηp2 = 0.106. Le staffe racchiudono gruppi significativamente diversi. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.
Nel confronto 30-80%, l’attività cSOD ha avuto un grande effetto sulla geotassi e sulla locomozione in controcorrente, dove la capacità locomotoria complessiva in entrambi i fenotipi al 30% di cSOD era inferiore all’80% di cSOD (Figura 7). Tuttavia, anche il sesso ha avuto un effetto significativo; i maschi hanno avuto una capacità locomotoria migliore delle femmine in entrambi i fenotipi, in entrambe le attività cSOD (Figura S7 nel File S1). Questa differenza è coerente con gli studi precedenti (Long and Rice 2007) e riflette potenzialmente le differenze nella strategia della storia della vita tra i sessi. Nella geotassi, ma non in controcorrente, l’entità del dimorfismo sessuale nell’abilità locomotoria variava a seconda del background genetico (Figura S5 e Figura S6 nel File S1). È interessante notare che il background genetico ha avuto un effetto maggiore sulla geotassi nel confronto 0-50% rispetto al confronto 30-80% (tendenza discussa di seguito; Figura 6 e Figura S7 nel File S1). Al contrario, la tendenza opposta è stata osservata nella storia della vita e nella locomozione controcorrente (anche se l’effetto non era significativo).
Nel calcolo della CF per la locomozione in controcorrente, abbiamo raggruppato le mosche in tre classi: cattivi, moderati e buoni arrampicatori, raggruppando essenzialmente le mosche con prestazioni “simili”. Per determinare se questo binning stava mascherando effetti sessuali o genetici di fondo, abbiamo ricalcolato i valori di FC usando le fiale come singole classi (otto classi potenziali invece di tre). Anche questa analisi unbinned non ha indicato alcun effetto di fondo genetico significativo. L’analisi unbinned ha indicato un effetto significativo del sesso nel confronto 0-50%, e in entrambe le analisi è stata osservata la tendenza dei maschi che hanno una migliore capacità di arrampicarsi rispetto alle femmine. Nel complesso, il binning sembrava avere un effetto limitato sulle analisi.

Figura 7.(A e B) Media ± SEM dell’indice negativo di prestazione della geotassi (PI), e (C e D) media ± SEM del coefficiente di ripartizione rampicante (CF), per mosche adulte maschio e femmina messe in comune attraverso gli otto sfondi genetici DGRPi all’interno di ogni genotipo. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) cSOD Attività, F1,156 = 169,6, P < 0,0001, ηp2 = 0,52; sesso, F1,156 = 4,83, P = 0,0294, ηp2 = 0.030. (B) cSOD Attività, F1,100 = 7,33, P = 0,008, ηp2 = 0,0683; sesso – F1,100 = 8,80, P = 0,0038, ηp2 = 0.0809. (C) attività cSOD, F1,269 = 501,0, P < 0,0001, ηp2 = 0.651. (D) attività cSOD, F1,175 = 61,6, P < 0,0001, ηp2 = 0,260; sesso – F1,175 = 20,8, P < 0,0001, ηp2 = 0.106. Le staffe racchiudono gruppi significativamente diversi. cSOD, superossido dismutasi citosolica rame/zinco dismutasi.
Gli enzimi NADP(H) hanno risposte variabili all’attività cSOD, al sesso e al background genetico
Anche gli enzimi NADP(H) hanno risposto ai livelli di attività cSOD, ma a differenza della storia di vita e dei fenotipi locomotori, che richiedono il 50% di attività cSOD per recuperare i fenotipi WT, il 30% di attività cSOD è stato sufficiente per ripristinare i fenotipi biochimici alle attività WT (Figura 8). Coerentemente con la storia della vita e i fenotipi locomotori, l’entità del dimorfismo sessuale nelle attività degli enzimi NADP(H) variava a 0 e 50% di attività cSOD. Tuttavia, a differenza dei fenotipi precedenti, lo schema di cambiamento variava a seconda del sesso (Figura 6). Nei maschi, le attività degli enzimi NADP(H) erano tutte inferiori allo 0% di attività cSOD rispetto al 50% di attività cSOD, mentre nelle femmine il modello variava tra gli enzimi NADP(H) (Figura 9; Figura S8 e Figura S9 nel File S1). I maschi tendevano ad avere un’attività enzimatica NADP(H) più elevata rispetto alle femmine, purché fosse presente una certa attività cSOD (Figura S10 nel File S1). Sorprendentemente, solo l’attività G6PD è stata modificata dal background genetico nel confronto 0-50%, con l’effetto di un background genetico variabile tra maschi e femmine attraverso le attività cSOD (Figura S9 nel File S1).
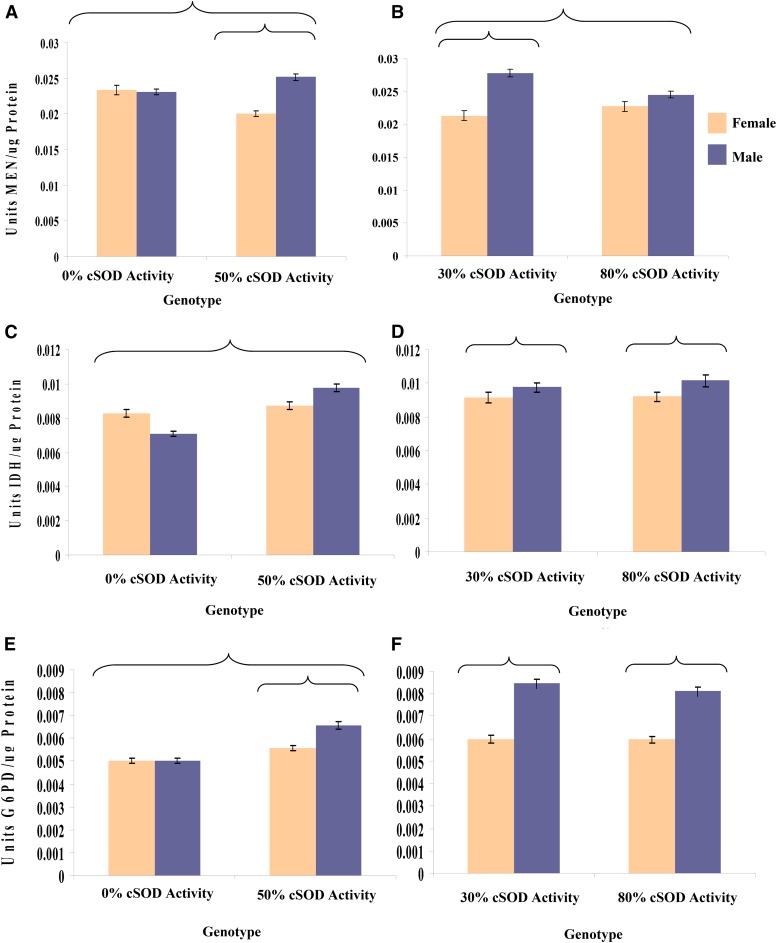
Figura 8.(A e B) Media ± SEM dell’attività MEN; (C e D) media ± SEM dell’attività IDH; e (E e F) media ± SEM dell’attività G6PD, standardizzata dalla concentrazione di proteine (microgrammi per millilitro) per le mosche adulte maschio e femmina messe in comune attraverso gli otto background genetici DGRPi all’interno di ogni genotipo del 3° cromosoma. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) Sesso per attività cSOD, F1,265 = 31,8, P < 0,0001, ηp2 = 0.110. (B) Attività sesso per sesso, F1,280 = 10,5, P = 0,0013, ηp2 = 0.0363. (C) Attività sesso per sesso, F1,265 = 25,5, P < 0,0001, ηp2 = 0.0879. (D) Sesso, F1,280 = 4,58, P = 0,0333, ηp2 = 0.0161. (E) Attività sesso per sesso, F1,265 = 19,6, P < 0,0001, ηp2 = 0.0690. (F) Sesso, F1,280 = 155,2, P < 0,0001, ηp2 = 0.357. Staffe racchiudono gruppi significativamente diversi. cSOD, rame citosolico / zinco superossido dismutasi; DGRPi, ; G6PD, glucosio-6-fosfato deidrogenasi; IDH, isocitrato deidrogenasi; MEN, enzima malico.
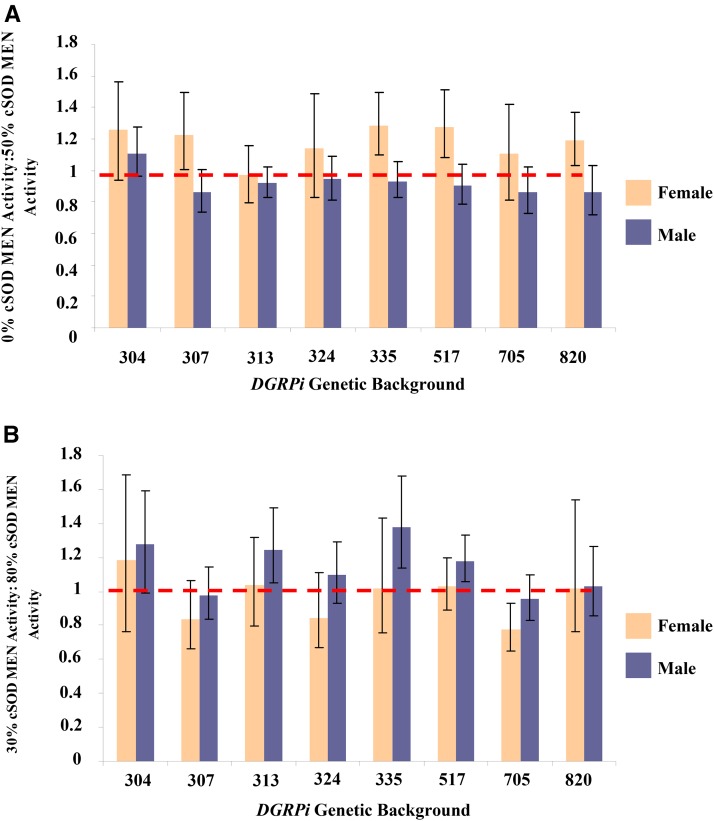
Figura 9.Media ± SEM del rapporto di bassa cSOD: alta attività cSOD attività MEN standardizzata dalla concentrazione di proteine (microgrammi per millilitro) per le mosche maschio e femmina adulte attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano attività enzimatiche più simili ai livelli di attività cSOD messi a confronto, e i C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEM. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 313, 0,796 < 0,970 < 1,16; rapporto più alto 335, 1,09 < 1,28 < 1,50; Maschio: rapporto più basso 705, 0,730 < 0,863 < 1,02; rapporto più alto 304 – 0,963 < 1,11 < 1,28. (B) 30% attività cSOD: 80% attività cSOD: Femmina: rapporto più basso 705, 0,646 < 0,777 < 0,929, rapporto più alto 304, 0,762 < 1,18 < 1,68; Maschio: rapporto più basso 705, 0,827 < 0,956 < 1,10; rapporto più alto 335, 1,14 < 1,38 < 1,68. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ciascun fattore. (A) Sesso per attività di CSOD, F1,265 = 32,8, P < 0,0001, ηp2 = 0.110. (B) Sesso per attività genetica, F7.280 = 3,18, P = 0,003, ηp2 = 0,0736; Sfondo genetico per attività del CSOD, F7.280 = 3,16, P = 0,0031, ηp2 = 0,0733; attività sesso-per-cSOD, F1,280 = 10,5, P = 0,0013, ηp2 = 0,0364. cSOD, superossido dismutasi citosolica rame/zinco dismutasi; DGRPi, ; MEN, enzima malico.
Inoltre, e ancora una volta paragonabile alla storia della vita e fenotipi locomotori, l’effetto dell’attività cSOD sui fenotipi biochimici tendeva ad essere più piccolo nel confronto 30-80% rispetto al confronto 0-50% (Figura 6). Coerentemente con il confronto 0-50% (sopra), i maschi tendevano ad avere un’attività più elevata rispetto alle femmine finché c’era una certa attività cSOD (Figura 8). Questo modello coerente suggerisce una sensibilità specifica del sesso alla variazione del genotipo dell’enzima NADP(H) in cui i maschi sono più sensibili alle differenze dei genotipi dell’enzima NADP(H) rispetto alle femmine (Merrittet al.2009). È interessante notare che l’esame dei dati di espressione genica per cSOD, MEN, IDH e G6PD, utilizzando la funzione modMine di ModENCODE, non ha mostrato alcuna indicazione di espressione specifica del sesso a livello dell’intero organismo (Contrinoet al., 2012) nelle linee disponibili tramite FlyBase (dati non mostrati). Nel complesso, i fenotipi biochimici variano nella loro risposta all’attività cSOD e al background genetico nel confronto 30-80% (Figura 9; Figura S8 e Figura S9 nel File S1). Per determinare se i risultati biochimici erano semplicemente una funzione della covariata utilizzata (concentrazione proteica), abbiamo ripetuto le analisi biochimiche utilizzando la massa della mosca e la concentrazione proteica come covariate e abbiamo trovato risultati essenzialmente identici a quelli che utilizzano la concentrazione proteica da sola (dati non mostrati).

Figura 8.(A e B) Media ± SEM dell’attività MEN; (C e D) media ± SEM dell’attività IDH; e (E e F) media ± SEM dell’attività G6PD, standardizzata dalla concentrazione di proteine (microgrammi per millilitro) per mosche adulte maschili e femminili messe in pool attraverso gli otto background genetici DGRPi all’interno di ogni genotipo del 3° cromosoma. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ogni fattore. (A) Sesso per attività di CSOD, F1,265 = 31,8, P < 0,0001, ηp2 = 0.110. (B) Attività sesso per sesso, F1,280 = 10,5, P = 0,0013, ηp2 = 0.0363. (C) Attività sesso per sesso, F1,265 = 25,5, P < 0,0001, ηp2 = 0.0879. (D) Sesso, F1,280 = 4,58, P = 0,0333, ηp2 = 0.0161. (E) Attività sesso per sesso, F1,265 = 19,6, P < 0,0001, ηp2 = 0.0690. (F) Sesso, F1,280 = 155,2, P < 0,0001, ηp2 = 0.357. Staffe racchiudono gruppi significativamente diversi. cSOD, rame citosolico / zinco superossido dismutasi; DGRPi, ; G6PD, glucosio-6-fosfato deidrogenasi; IDH, isocitrato deidrogenasi; MEN, enzima malico.

Figura 9.Media ± SEM del rapporto di bassa cSOD: alta attività cSOD attività MEN standardizzata dalla concentrazione di proteine (microgrammi per millilitro) per le mosche maschio e femmina adulte attraverso gli otto background genetici DGRPi. Valori più vicini a 1 indicano attività enzimatiche più simili ai livelli di attività cSOD messi a confronto, e i C.I.S. sono al 95% e rappresentano il rapporto tra la bassa attività cSOD e l’alta attività cSOD SEM. (A) 0% di attività cSOD: 50% di attività cSOD: Femminile: rapporto più basso 313, 0,796 < 0,970 < 1,16; rapporto più alto 335, 1,09 < 1,28 < 1,50; Maschio: rapporto più basso 705, 0,730 < 0,863 < 1,02; rapporto più alto 304 – 0,963 < 1,11 < 1,28. (B) 30% attività cSOD: 80% attività cSOD: Femmina: rapporto più basso 705, 0,646 < 0,777 < 0,929, rapporto più alto 304, 0,762 < 1,18 < 1,68; Maschio: rapporto più basso 705, 0,827 < 0,956 < 1,10; rapporto più alto 335, 1,14 < 1,38 < 1,68. ANOVA è stato utilizzato per testare gli effetti del fattore con un α di 0,05. Eta parziale al quadrato (ηp2) è stato calcolato per quantificare l’effetto di ciascun fattore. (A) Sesso per attività di CSOD, F1,265 = 32,8, P < 0,0001, ηp2 = 0.110. (B) Sesso per attività genetica, F7.280 = 3,18, P = 0,003, ηp2 = 0,0736; Sfondo genetico per attività del CSOD, F7.280 = 3,16, P = 0,0031, ηp2 = 0,0733; attività sesso-per-cSOD, F1,280 = 10,5, P = 0,0013, ηp2 = 0,0364. cSOD, superossido dismutasi citosolica rame/zinco dismutasi; DGRPi, ; MEN, enzima malico.
Discussione
Le grandezze delle risposte fenotipiche variano a seconda delle attività cSOD, dei sessi e del background genetico
Le differenze nell’attività del cSOD hanno portato a cambiamenti sostanziali nella storia della vita, nel locomotore e nei fenotipi biochimici, e queste differenze su larga scala sono state significativamente modificate dal background genetico e dal sesso. Ricerche precedenti in D. melanogaster hanno dimostrato che la perdita completa dell’attività cSOD modifica una vasta gamma di fenotipi, ma che solo il 50% della normale attività cSOD è generalmente sufficiente per ripristinare completamente i fenotipi WT (Phillipset al.1989; Parkeset al.1998b; Sole e Torre 1999; Pasyukovaet al.2000; Woodruffet al.2004; Bernardoet al.2011; Jones e Grotewiel 2011; Ginocchioet al.2013). Tuttavia, questi studi hanno utilizzato prevalentemente sfondi singoli, isogenici, anche se i fenotipi complessi sono generalmente influenzati da più geni (Laurie-Ahlberget al.1982; Leips e Mackay 2000; Spenceret al.2003; Giordaniaet al.2007; Rzezniczak e Merritt 2012). Il sesso e il background genetico modificano entrambi in modo significativo gli effetti del cSOD che dimostriamo, ma è interessante notare che queste modifiche sono relativamente piccole rispetto all’effetto del cSOD. Negli otto background che abbiamo analizzato, nessuno dei fenotipi cSOD ha eliminato, o addirittura fortemente migliorato o esagerato, nessuno dei fenotipi cSOD.
Ci aspettavamo che la storia della vita e i fenotipi locomotori fossero una funzione di un numero maggiore di interazioni genetiche e ambientali rispetto ai fenotipi biochimici; pertanto, ci aspettavamo di vedere una maggiore sensibilità nella storia della vita e nei fenotipi locomotori ai cambiamenti nell’attività cSOD, nel sesso e nel background genetico. Invece, tutti i fenotipi hanno mostrato una sensibilità simile a tutti e tre i fattori. Tuttavia, l’anamnesi della vita e i fenotipi locomotori erano più sensibili a differenze minori nell’attività cSOD rispetto ai fenotipi biochimici, probabilmente a sostegno della nostra aspettativa che questi fenotipi siano più complessi delle attività biochimiche. È interessante notare che è stata richiesta una soglia inferiore di attività cSOD per recuperare i fenotipi biochimici a WT, anche se non è chiaro se questa differenza rifletta una differenza fondamentale tra le classi di fenotipi.
L’assenza di attività cSOD ha il maggiore effetto su tutti i fenotipi esaminati
Ci aspettavamo anche che l’attività cSOD fosse il più importante fattore di controllo dei fenotipi a bassa attività cSOD, che riflette lo stress ossidativo e i cambiamenti metabolici di ampia portata causati dalla perdita di funzione di un enzima chiave di difesa antiossidante. Coerentemente con le nostre aspettative, i fenotipi sono stati maggiormente alterati in cSOD-nulli, e la perdita di attività cSOD ha avuto un effetto maggiore rispetto al sesso o a qualsiasi altro sfondo. Coerente con studi precedenti (ad esempio, Setoet al.1990) abbiamo trovato che i fenotipi sono stati recuperati a livelli di WT di circa il 50% di attività cSOD, sostenendo l’idea che questa quantità di attività cSOD è generalmente sufficiente a superare la tossicità del paraquat. Inoltre, mentre l’entità delle risposte fenotipiche era diversa, nel confronto tra 0-50% rispetto al confronto tra il 30-80%, l’attività cSOD era attribuibile a una maggiore variazione.
La storia di vita e i fenotipi locomotori sono stati significativamente compromessi nelle mosche senza attività cSOD, con differenze fenotipiche che riflettono la quantità di attività cSOD (Figura S4 nel File S1), coerente con altri studi (Parkeset al.1998b; Sun e Tower 1999; Jones e Grotewiel 2011). È interessante notare che gli enzimi NADP(H) hanno risposto ciascuno in modo diverso ai cambiamenti nell’attività del cSOD. Queste risposte uniche sono in contrasto con il nostro studio precedente (Bernardet al.2011), in cui abbiamo riscontrato riduzioni simili delle attività enzimatiche, che riflettono forse i diversi background utilizzati in questi due studi.
La sensibilità dei fenotipi biochimici ai cambiamenti nell’attività cSOD, nel sesso e nel background genetico, suggerisce che questi “semplici” fenotipi sono più complicati di quanto ci aspettassimo o almeno lo è la loro connessione con il cSOD. Originariamente, abbiamo classificato i fenotipi biochimici come “prossimali” (fenotipi vicini alla mutazione), e la storia della vita e fenotipi locomotori come “distali” (fenotipi lontani dalla mutazione). Poiché i fenotipi distali sono più a valle della mutazione, ci aspettavamo che fossero una funzione di un numero maggiore di interazioni genetiche e ambientali rispetto ai fenotipi prossimali. Tuttavia, le interazioni tra MEN, IDH e G6PD (Merrittet al.2009; Rzezniczak e Merritt 2012), e tra questi enzimi NADP(H) e cSOD [ad esempio, in Bernardet al.(2011) e i risultati di questo studio], indicano che i legami tra cSOD e gli enzimi NADP(H) sono complessi e influenzati da molteplici geni. Chiaramente, questi fenotipi biochimici non sono così prossimali al cSOD come abbiamo immaginato e le semplici classificazioni “prossimali” e “distali” originariamente assegnate non riflettono accuratamente la natura dei fenotipi misurati. Tuttavia, il fatto che i fenotipi biochimici siano recuperati a WT da una soglia inferiore di cSOD rispetto alla storia della vita e ai fenotipi locomotori riflette probabilmente le differenze nell’influenza relativa di cSOD su questi tipi di fenotipi. Non è chiaro se i percorsi di interazione che collegano cSOD, i loci di modificazione e i fenotipi biochimici siano più o meno complessi rispetto a quelli coinvolti nella modificazione della storia della vita e dei tratti locomotori, e ulteriori ricerche che includono enzimi più direttamente collegati con cSOD, come la catalasi (Michielset al.1994), è necessario.
La forza di interazione tra cSOD e ciascun fenotipo riflette probabilmente il grado di connessione tra cSOD e i meccanismi molecolari di ciascun fenotipo, presumibilmente indirettamente attraverso il ruolo di cSOD e NADP(H) nella rete di difesa antiossidante (Phillipset al.1989; Bernardoet al.2011). Quanto più i fenotipi sono legati al metabolismo del ROS, tanto maggiori sono gli effetti delle differenze nell’attività del cSOD sui fenotipi. Inoltre, diversi gradi di connessione dovrebbero risultare o essere una componente di diverse soglie di attività cSOD necessarie per recuperare i fenotipi WT. Queste interazioni potrebbero essere dovute al consumo di NADP(H) o all’accumulo di stress ossidativo superossidante. In entrambi gli scenari, la perdita completa dell’attività cSOD dovrebbe avere il massimo effetto, con questa condizione estrema che limita gli effetti di modificazione del sesso o del background genetico, in linea con i nostri risultati.
Il sesso ha un effetto maggiore sui fenotipi a maggiore attività cSOD
Il cSOD e i fenotipi associati sono stati studiati principalmente nei maschi (Parkeset al.1998a; Bernardoet al.2011; Ginocchioet al.2013), ad eccezione di alcuni fenotipi specifici del sesso (ad esempio, fertilità maschile e femminile; Parkeset al.1998b). Questa esclusione riflette un maggiore pregiudizio contro l’inclusione di soggetti di sesso femminile negli studi biologici (Miller 2012; Kleinet al.2015; Parikh 2015). L’uso predominante di soggetti maschi riflette la percezione che le femmine sono soggetti di ricerca più variabili, e quindi più poveri, rispetto ai maschi. I nostri risultati non supportano questa percezione. È possibile che l’aumento del carico metabolico imposto dallo sforzo riproduttivo nelle femmine, rispetto ai maschi, possa aumentare la variabilità dei fenotipi della storia della vita (ad esempio, la vitalità) tra le femmine, confondendo potenzialmente l’analisi di tali fenotipi. Tuttavia, la Figura 2 illustra la variabilità relativa della longevità tra maschi e femmine osservata in questo studio e mostra chiaramente che le femmine non sono sostanzialmente più variabili dei maschi. Esprimendo i SEM per la longevità in entrambi i sessi come percentuale della media stessa, i dati sulla longevità relativa rivelano che la variabilità è molto simile tra i sessi: 1,2-2,4% della media nelle femmine e 0,9-1,9% nei maschi. Gli altri sei fenotipi hanno mostrato uno schema simile; le femmine non erano sostanzialmente più variabili dei maschi. È importante notare che i saggi in questo studio sono stati eseguiti su mosche accoppiate, quindi qualsiasi aumento della variabilità nelle femmine derivante dalla variabilità dello sforzo riproduttivo dovrebbe essere evidente, ma non ci sono prove nei nostri dati a sostegno della nozione che le femmine sono più fenotipicamente variabili rispetto ai maschi. Date le differenze specifiche del sesso che abbiamo documentato, e l’importanza di comprendere gli effetti fenotipici completi, non semplicemente gli effetti specifici del maschio, è fondamentale che i tratti siano studiati sia nei maschi che nelle femmine.
Differenze fenotipiche specifiche del sesso sono state documentate in D. melanogaster e in molti altri organismi, comprese le differenze in alcuni dei fenotipi che abbiamo quantificato (ad esempio, Pasyukovaet al.2000; Spenceret al.2003; Giordaniaet al.2007; Merrittet al.2009). Abbiamo osservato un significativo dimorfismo sessuale nella longevità, nella geotassi, nella locomozione controcorrente e nelle attività degli enzimi NADP(H), indicando che tali differenze sono diffuse. Una nota qualificante: le mosche maschio e femmina in questo esperimento differivano per il genotipo al cromosoma X; i maschi hanno solo il w+ Cromosoma X, le femmine hanno il w+ X e la w1118 X. È possibile, quindi, che alcuni effetti dominanti della w1118 cromosoma potrebbe influenzare i nostri risultati. Tuttavia, il fatto che il dimorfismo sessuale che abbiamo osservato è coerente con quello documentato in letteratura (Spenceret al.2003; Giordaniaet al.2007; Merrittet al.2009) suggerisce che le differenze osservate non sono solo una funzione delle differenze nel genotipo del cromosoma X.
Abbiamo scoperto che l’attività cSOD stessa era sessualmente dimorfica, anche se solo nel confronto 30-80%. Il dimorfismo sessuale nella longevità è stato riportato in mosche che esprimono in modo eccessivo il cSOD, possibilmente riflettendo il background genetico o l’attività sessuale specifica dell’attivatore Gal4 e/o del transgene cSOD impiegato in questi studi (Spenceret al.2003). La disparità sessuale specifica nell’attività cSOD saggiata che osserviamo può riflettere in modo simile il dimorfismo sessuale nell’espressione del transgene cSOD T5. Poiché non esiste alcun dimorfismo sessuale nell’attività cSOD o nell’espressione genica nel locus nativo D. melanogaster cSod (Koppet al.2003; Gnad e Parsch 2006), sembra probabile che il dimorfismo sessuale nell’attività cSOD nel confronto 30-80% rifletta qualche caratteristica del transgene, possibilmente riflettendo l’ambiente genomico locale, e non è una caratteristica generale del cSOD.
Ci sono state differenze sostanziali nella sensibilità al background genetico tra i fenotipi esaminati
Le differenze nei fenotipi tra i diversi background genetici, gli “effetti di linea”, sono comuni, dovuti alla variazione nel modificare i loci del genoma. La ricerca precedente ha dimostrato che alcuni tratti della storia della vita differiscono tra i diversi background genetici (ad esempio, Fryet al.1998; Spenceret al.2003), mentre altri non lo fanno (de Visseret al.2003; Fry 2008). Nel nostro studio, la longevità e la vitalità differiscono per la sensibilità al background genetico, e solo la longevità è significativamente influenzata dalla perturbazione del background genetico (Figura S4 nel File S1). Fitness e longevità variano tra le linee parentali DGRP (Mackayet al.2012) e ci aspettavamo variazioni nelle linee che abbiamo costruito, ma probabilmente quantità un po’ inferiori che riflettono l’inclusione della sola variazione al 2° cromosoma nel nostro studio. Il fatto che abbiamo trovato che la longevità, ma non la vitalità, sia sensibile al background genetico, suggerisce che non solo le reti genetiche per la vitalità e la longevità sono indipendenti, ma che la rete per la vitalità è più robusta della longevità.
Sono stati eseguiti due saggi locomotori complementari per rilevare piccole differenze nell’effetto del background genetico sulla locomozione: la geotassi negativa e la locomozione in controcorrente. Mentre gli effetti del background genetico sono stati rilevati nella geotassi attraverso i confronti cSOD, gli effetti sono stati minori nel confronto 30-80% rispetto al confronto 0-50% (Figura 6), coerentemente con la riduzione della potenza di risoluzione che abbiamo ipotizzato potrebbe risultare dalla modifica dell’altezza di salita nel saggio di geotassi (vedi Materiali e Metodi). È interessante notare che la locomozione in controcorrente era meno sensibile al background genetico o al sesso rispetto alla geotassi (Figura S7 nel File S1). Il saggio controcorrente è più un saggio di resistenza, piuttosto che reattivo, rispetto al saggio di geotassi (Jones e Grotewiel 2011), e gli effetti minori del background possono riflettere un particolare deficit nella resistenza locomotoria nelle mosche cSOD-nulle. In generale, i due saggi locomotori hanno risposto in modo diverso alle variazioni del background genetico, suggerendo che questi fenotipi locomotori hanno meccanismi genetici diversi, in linea con le ricerche precedenti (Jordanet al.2007, 2012).
MEN, IDH e G6PD fanno parte di una rete NADP(H) interconnessa, in cui i cambiamenti di attività di un enzima causano cambiamenti nelle attività degli altri enzimi, probabilmente attraverso i pool del cofattore NADP(H) condiviso (Merrittet al.2009; Bernardoet al.2011; Rzezniczak e Merritt 2012; Rzezniczaket al.2012). In precedenza, risposte simili in MEN, IDH e G6PD a differenze nell’attività cSOD sono state osservate in un unico background genetico (Bernardet al.2011). Dimostriamo qui che queste interazioni sono, di fatto, sensibili al background genetico, e che ogni enzima differisce in questa sensibilità (Figura S10 nel File S1). Mentre tutti questi enzimi condividono il cofattore NADP(H), ogni componente di questa rete risponde in modo diverso ai cambiamenti genetici, metabolici e ambientali, e le interazioni attraverso la rete sono complesse e non semplicemente compensative (Rzezniczak e Merritt 2012; Merrittet al.2009).
Nessuno dei background genetici ha dimostrato modifiche coerenti dei fenotipi cSOD
Abbiamo selezionato le linee DGRP per le estrazioni dei secondi cromosomi specificamente per massimizzare le differenze nei nostri fenotipi, selezionando linee alte e basse per ogni fenotipo, ma abbiamo osservato solo modifiche relativamente piccole degli effetti cSOD per sfondo. Tuttavia, i fenotipi complessi sono controllati da più geni con differenze fenotipiche probabilmente dovute a variazioni nel genoma (nessuno dei fenotipi è stato completamente mappato), e le linee che abbiamo creato riveleranno solo modificatori dominanti al 2° loci del cromosoma. L’estrazione dei cromosomi di linea “alti” e “bassi” dalle linee parentali ha alterato il loro contesto genetico che può aver modificato le interazioni che hanno portato alla loro classificazione di linea alta o bassa. Inoltre, siamo stati costretti a saggiare solo il secondo cromosoma eterozigote, impedendoci di osservare le interazioni recessive. Una conclusione generale su tutti i fenotipi è che gli effetti genetici di fondo erano più piccoli nei cSOD-nulli, suggerendo che questa condizione estrema limita questi effetti, ma altri [esaminati in Chari e Dworkin (2013)] hanno dimostrato effetti di fondo in tutti i tipi di mutazioni. Questo contrasto suggerisce che la presenza e l’entità degli effetti di fondo è sia gene-specifica che fenotipica.
Anche con l’avvertenza della restrizione ai modificatori dominanti, siamo stati sorpresi che nessun background abbia sostanzialmente soppresso o migliorato i fenotipi cSOD-null. La variazione genetica al 2° cromosoma di fondo ha modificato, ma non ha cambiato in modo sostanziale, la variazione fenotipica che osserviamo. Il fatto che le linee parentali DGRP mostrano una variazione sostanziale per ogni fenotipo indica che la variazione genetica presente è in grado di modificare i fenotipi. Quindi, la mancanza di variazione su larga scala delle nostre linee derivate riflette probabilmente una complessa architettura genomica negli effetti di fondo, suggerendo che questi effetti sono di per sé complessi e comportano interazioni dominanti e recessive tra i loci del genoma.
Conclusioni
La sindrome cSOD-nulla è guidata dall’assenza di attività cSOD, ma è sensibile e modificata dal sesso e dal background genetico. Anche se di entità minore rispetto agli effetti cSOD, il background e il sesso hanno influito in modo significativo sui fenotipi, compresi i notevoli effetti specifici del sesso, l’entità degli effetti del background genetico varia in modo sorprendente con il livello di attività cSOD e il fenotipo. Coerentemente con questa sensibilità specifica del fenotipo, anche la quantità di soglia di attività cSOD necessaria per recuperare un fenotipo a WT è diversa, con la storia della vita e i fenotipi locomotori che hanno una soglia di attività cSOD più alta rispetto ai fenotipi biochimici. Sorprendentemente, non è stato trovato alcun background genetico che abbia portato al miglioramento o alla soppressione su larga scala dei fenotipi cSOD-dipendenti. Infine, i nostri risultati indicano che le mosche maschio e femmina differiscono sostanzialmente nelle loro risposte fenotipiche ad alcuni fattori, rafforzando il punto che sia i soggetti maschi che femmine, di qualsiasi specie, hanno bisogno di essere analizzati per comprendere i processi biochimici e fisiologici.
Materiale supplementare
Il materiale supplementare è disponibile online all’indirizzo www.g3journal.org/lookup/suppl/doi:10.1534/g3.117.043836/-/DC1.
References
- Assis R., Zhou Q., Bachtrog D.. Sex-biased transcriptome evolution in Drosophila. Genome Biol. Evol.. 2012; 4(11):1189-1200. PubMed
- Ayroles J. F., Carbone M. A., Stone E. A., Jordan K. W., Lyman R. F.. Systems genetics of complex traits in Drosophila melanogaster. Nat. Genet.. 2009; 41(3):299-307. PubMed
- Bafana A., Dutt S., Kumar A., Kumar S., Ahuja P. S.. The basic and applied aspects of superoxide dismutase. J. Mol. Catal., B Enzym.. 2011; 68:129-138.
- Benzer S.. Behavioural mutants of Drosophila isolated by countercurrent distribution. Genetics. 1967; 58:1112-1119.
- Bernard K. E., Parkes T. L., Merritt T. J.. A model of oxidative stress management: moderation of carbohydrate metabolizing enzymes in SOD1-null Drosophila melanogaster.. PLoS One.. 2011; 6(9):e24518. PubMed
- Bing X., Rzezniczak T. Z., Bateman J. R., Merritt T. J.. Transvection-based gene regulation in Drosophila is a complex and plastic trait.. G3 (Bethesda). 2014; 4:2175-2187. PubMed
- Campbell S. D., Hilliker A. J., Phillips J. P.. Cytogenetic analysis of the cSOD microregion in Drosophila melanogaster. Genetics. 1986; 112:205-215. PubMed
- Chandler C. H., Chari S., Dworkin I.. Does your gene need a background check? How genetic background impacts the analysis of mutations, genes, and evolution. Trends Genet.. 2013; 29(6):358-366. PubMed
- Chari S., Dworkin I.. The conditional nature of genetic interactions: the consequences of wild-type backgrounds on mutational interactions in a genome-wide modifier screen. PLoS. 2013; 9(8):e1003661.
- Clough E., Oliver B.. Genomics of sex determination in Drosophila. Brief. Funct. Genomics. 2012; 11(5):387-394. PubMed
- Clough E., Jimenez E., Kim Y. A., Whitworth C., Neville M. C.. Sex- and tissue-specific functions of Drosophila doublesex transcription factor target genes. Dev. Cell. 2014; 31:761-773. PubMed
- Contrino S., Smith R. N., Butano D., Carr A., Hu F.. modMine: flexible access to modENCODE data.. Nucleic Acids Res.. 2012; 40(D1):D1082-D1088. PubMed
- de Visser J. A. G. M., Hermisson J., Wagner G. P., Meyers L. A., Bagheri-Chaichian H.. Perspective: evolution and detection of genetic robustness. Evolution. 2003; 57(9):1959-1972. PubMed
- Duttaroy A., Parkes T., Emtage P., Kirby K., Boulianne G. L.. The manganese superoxide dismutase gene of Drosophila: structure, expression, and evidence for regulation by MAP kinase. DNA Cell Biol.. 1997; 16(4):391-399. PubMed
- Fairbairn D. J., Roff D. A.. The quantitative genetics of sexual dimorphism: assessing the importance of sex-linkage. Heredity. 2006; 97:319-328. PubMed
- Fry J. D.. Genotype-environment interaction for total fitness in Drosophila. J. Genet.. 2008; 87:355-362. PubMed
- Fry J. D., Nuzhdin S. V., Pasyukova E. G., Mackay T. F. C.. QTL mapping of genotype-environment interaction for fitness in Drosophila melanogaster. Genet. Res.. 1998; 71:133-141. PubMed
- Gnad F., Parsch J.. Sebida: a database for the functional and evolutionary analysis of genes with sex-biased expression. Bioinformatics. 2006; 22:2577-2579. PubMed
- Graf J. D., Ayala F. J.. Genetic variation for superoxide dismutase level in Drosophila melanogaster. Biochem. Genet.. 1986; 24(3/4):153-168. PubMed
- Jones M. A., Grotewiel M.. Drosophila as a model for age-related impairment in locomotor and other behaviours. Exp. Gerontol.. 2011; 46:320-325. PubMed
- Jordan K. W., Carbone M. A., Yamamoto A., Morgan T. J., Mackay T. F. C.. Quantitative genomics of locomotor behaviour in Drosophila melanogaster. Genome Biol.. 2007; 8:R172. PubMed
- Jordan K. W., Craver K. L., Magwire M. M., Cubilla C. E., Mackay T. F. C.. Genome-wide association for sensitivity to chronic oxidative stress in Drosophila melanogaster. PLoS One. 2012; 7(6):e38722. PubMed
- Kamikouchi A., Inagaki H. K., Effertz T., Hendrich O., Fiala A.. The neural basis of Drosophila gravity-sensing and hearing. Nature. 2009; 458:165-172. PubMed
- Kanzok S. M., Fechner A., Bauer H., Ulschmid J. K., Muller H. M.. Substitution of the thioredoxin system for glutathione reductase in Drosophila melanogaster. Science. 2001; 291:643-646. PubMed
- Klein S. L., Schiebinger L., Stefanick M. L., Cahill L., Danska J.. Opinion: sex inclusion in basic research drives discovery. Proc. Natl. Acad. Sci. USA. 2015; 12(17):S257-S258.
- Knee J. M., Rzezniczak T. Z., Barsch A., Guo K. Z., Merritt T. J. S.. A novel ion pairing LC/MS metabolomics protocol for study of a variety of biologically relevant polar metabolites. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci.. 2013; 936:63-73.
- Kopp A., Graze R. M., Xu S., Carroll S. B., Nuzhdin S. Z.. Quantitative trait loci responsible for variation in sexually dimorphic traits in Drosophila melanogaster. Genetics. 2003; 163:771-787. PubMed
- Laurie-Ahlberg C. C., Wilton A. N., Curtsinger J. W., Emigh T. H.. Naturally occurring enzyme activity variation in Drosophila melanogaster. I. Sources of variation for 23 enzymes. Genetics. 1982; 102:191-206. PubMed
- Leips J., Mackay T. F. C.. Quantitative trait loci for life span in Drosophila melanogaster: interactions with genetic background and larval density. Genetics. 2000; 155:1773-1788. PubMed
- Long T. A. F., Rice W. R.. Adult locomotor activity mediates intralocus sexual conflict in a laboratory-adapted population of Drosophila melanogaster. Proc. Biol. Sci.. 2007; 274:3105-3112. PubMed
- Mackay T. F. C., Richards S., Stone E. A., Barbadilla A., Ayroles J. F.. The Drosophila melanogaster genetic reference panel. Nature. 2012; 428:173-178.
- Martin I., Jones M. A., Grotewiel M.. Manipulation of Sod1 expression ubiquitously, but not in the nervous system or muscle, impacts age-related parameters in Drosophila. FEBS Lett.. 2009; 583:2308-2314. PubMed
- McCord J. M., Fridovich I.. Superoxide dismutase: an enzymatic function for erythrocuprein (hemocuprein). J. Biol. Chem.. 1969; 244(22):6049-6055. PubMed
- Merritt T. J. S., Sezgin E., Zhu C. T., Eanes W. F.. Triglyceride flight pools, flight and activity variation at the Gpdh locus in Drosophila melanogaster. Genetics. 2006; 172:293-304. PubMed
- Merritt T. J. S., Kuczynski C., Sezgin E., Zhu C. T., Kumagai S.. Quantifying interactions within the NADP(H) enzyme network in Drosophila melanogaster. Genetics. 2009; 182:565-574. PubMed
- Michiels C., Raes M., Toussaint O., Remacle J.. Importance of Se-glutathione peroxidase, catalase, and Cu/Zn-SOD for cell survival against oxidative stress. Free Radic. Biol. Med.. 1994; 17(3):235-248. PubMed
- Miller V. M.. In pursuit of scientific excellence: sex matters. Physiol. Genomics. 2012; 44:485-486. PubMed
- Publisher Full Text
- Parikh M.. Subject selection bias in animal studies. Curr. Sci.. 2015; 109(4):678-679.
- Parkes T. L., Elia A. J., Dickenson D., Hilliker A. J., Phillips J. P.. Extension of Drosophila lifespan by overexpression of human SOD1 in motorneurons. Nat. Genet.. 1998; 19(2):171-174. PubMed
- Parkes T. L., Kirby K., Phillips J. P., Hilliker A. J.. Transgenic analysis of the cSOD-null phenotypic syndrome in Drosophila. Genome. 1998; 41:642-651. PubMed
- Pasyukova E. G., Viera C., Mackay T. F. C.. Deficiency mapping of quantitative trait loci affecting longevity in Drosophila melanogaster. Genetics. 2000; 156:1129-1146. PubMed
- Patel P. H., Tamanoi F.. Increased Rheb-TOR signalling enhances sensitivity of the whole organism to oxidative stress. J. Cell Sci.. 2006; 119(20):4285-4292. PubMed
- Petersen A. J., Katzenberger R. J., Wassarman D. A.. The innate immune response transcription factor relish is necessary for neurodegeneration in a Drosophila model of ataxia-telangiectasia. Genetics. 2013; 194:133-142. PubMed
- Phillips J. P., Campbell S. D., Michaud D., Charbonneau M., Hilliker A. J.. Null mutation of copper/zinc superoxide dismutase in Drosophila confers hypersensitivity to paraquat and reduced longevity. Proc. Natl. Acad. Sci. USA. 1989; 88:2761-2765.
- Pierce C. A., Block R. A., Aguinis H.. Cautionary note on reporting eta-squared values from multifactor ANOVA designs. Educ. Psychol. Meas.. 2004; 64(6):916-924.
- Radyuk S. N., Klichko V. I., Orr W. C.. Profiling Cu,Zn-superoxide dismutase expression in Drosophila melanogaster – a critical regulatory role for intron/exon sequence within the coding domain. Gene. 2004; 328:37-48. PubMed
- Ranz J. M., Castillo-Davis C. I., Meiklejohn C. D., Hartl D. L.. Sex-dependent gene expression and evolution of the Drosophila transcriptome. Science. 2003; 300:1742-1745. PubMed
- Rzezniczak T. Z., Merritt T. J. S.. Interactions of NADP-reducing enzymes across varying environmental conditions: a model of biological complexity.. G3 (Bethesda). 2012; 2:1613-1623. PubMed
- Rzezniczak T. Z., Lum T. E., Harniman R., Merritt T. J. S.. A combination of structural and Cis-regulatory factors drives biochemical differences in Drosophila melanogaster. Biochem. Genet.. 2012; 50:823-837. PubMed
- Seto N. O. L., Hayashi S., Tener G. M.. Overexpression of Cu-Zn superoxide dismutase in Drosophila does not affect life-span. Proc. Natl. Acad. Sci. USA. 1990; 87:4270-4274. PubMed
- Sofola O., Kerr F., Rogers I., Killick R., Augustin H.. Inhibition of GSK-3 ameliorates Aβ pathology in an adult-onset Drosophila model of Alzheimer’s disease. PLoS Genet.. 2010; 6(9):e1001087. PubMed
- Spencer C. C., Howell C. E., Wright A. R., Promislow D. E. L.. Testing an ‘aging gene’ in long-lived Drosophila strains: increased longevity depends on sex and genetic background. Aging Cell. 2003; 2:123-130. PubMed
- Sun J., Tower J.. FLP recombinase-mediated induction of Cu/Zn-superoxide dismutase transgene expression can extend the life span of adult Drosophila melanogaster flies. Mol. Cell. Biol.. 1999; 19:216-228. PubMed
- Woodruff R. C., Phillips J. P., Hilliker A. J.. Increased spontaneous DNA damage in Cu/Zn superoxide dismutase (SOD1) deficient Drosophila. Genome. 2004; 47:1029-1035. PubMed
- Yamamoto A., Anholt R. R. H., Mackay T. F. C.. Epistatic interactions attenuate mutations affecting startle behaviour in Drosophila melanogaster. Genet. Res.. 2009; 91:373-382.
- Zelko I. N., Mariani T. J., Folz R. J.. Superoxide dismutase multigene family: a comparison of the CuZn-SOD (SOD1), Mn-SOD (SOD2), and EC-SOD (SOD3) gene structures, evolution, and expression. Free Radic. Biol. Med.. 2002; 33:337-349. PubMed
Fonte
Lessel CE, Parkes TL, Dickinson J, Merritt TJS (2017) Sex and Genetic Background Influence Superoxide Dismutase (cSOD)-Related Phenotypic Variation in . G3: Genes|Genomes|Genetics 7(8): . https://doi.org/10.1534/g3.117.043836





