Abstract
Introduzione
Le simbiosi sono interazioni persistenti e intime tra gli organismi. Nelle interazioni patogene, un partner ne trae vantaggio a scapito dell’altro. Nelle simbiosi mutualistiche, i partner specifici interagiscono e ne traggono reciproco beneficio. Le simbiosi associative sono una variante del mutualismo in cui vi è una minore specificità tra i partner che interagiscono(Drogue et al., 2012). Nei sistemi agricoli, le pratiche sono utilizzate per limitare gli agenti patogeni, per introdurre rizobie mutualistiche che fissano l’azoto e per ripristinare i simbiotici associativi come i batteri promotori della crescita delle piante (PGPB). Il PGPB può promuovere direttamente la crescita delle piante e proteggere dagli agenti patogeni (Bareaet al., 2005; Pieterse et al., 2014).
I risultati positivi o parassitari delle simbiosi, in particolare quelli che coinvolgono partner acquisiti in campo ambientale, spesso non sono garantiti. La salute dell’ospite, l’ubicazione del simbionte sull’ospite o la proliferazione non regolamentata del simbionte possono portare a risultati alternativi(Lin e Koskella, 2015). Anche il genotipo del simbionte è un fattore critico, in quanto il trasferimento genico orizzontale (HGT) può portare all’acquisizione di nuovi geni che innovano i genomi, guidando le transizioni evolutive e stabilendo nuovi lignaggi di simbiotici benefici o patogeni(Soucy et al., 2015). In alcuni simbiotici patogeni, tuttavia, l’HGT non conferisce al genoma funzioni innovative, né questi genomi presentano cambiamenti sostanziali. Piuttosto, i pochi geni acquisiti orizzontalmente codificano prodotti che riprogrammano i geni del nucleo, cooptando così il genoma per la virulenza(Letek et al., 2010).
Ilrodococco è un genere di batteri Gram-positivi con membri che persistono in una varietà di ecosistemi terrestri e acquatici(de Carvalho et al., 2014; Larkin et al., 2005). Il rodococco comprende gruppi tassonomici con membri che sono stati ripetutamente recuperati da tessuti fogliari e radicali di varie specie di piante(Bai et al., 2015; Bodenhausen et al., 2013; Bulgarelli et al., 2012; Honget al., 2015, 2016; Lebeis et al., 2015;Lundberg et al., 2012; Qin et al., 2009, 2011; Salam et al., 2017). È stato suggerito che gli ospiti arricchiscano i membri del Rodococco a causa dei tratti benefici del batterio(Hong et al.,2015, 2016).
Le specie di rodococco associate alle piante sono meglio conosciute come agenti patogeni(Putnam e Miller, 2007). Due cladi di Rhodococcus includono membri che possono causare malattie in oltre 100 generi di piante(Creason et al., 2014a2014a; Putnam e Miller, 2007). Le piante erbacee sono le più comunemente colpite, mentre le piante legnose sono meno frequentemente infette. I sintomi della malattia includono le ghiandole fogliari, i funghi delle streghe e altre crescite sfiguranti. Gli isolati patogeni di rodococco, caratterizzati dall’isolato più studiato D188, richiedono tre loci di virulenza che si trovano più frequentemente raggruppati su plasmidi di virulenza(Crespi et al., 1992; Stes et al. , 2011). Questi plasmidi hanno una lunghezza di circa 200 kb e sono repliconi lineari(Francis et al., 2012; Creason et al., 2014b). Alcuni dei geni fas(fasciazione) sono necessari per la malattia e codificano le proteine che sintetizzano e modificano le citochinine, che si prevede siano degli effettori secreti(Crespi et al., 1994; Pertry et al., 2009). Il gene fasR, che si prevede sia un regolatore trascrizionale, è necessario anche per la patogenicità(Temmerman et al., 2000). I mutanti att(attenuazione), secondo quanto riportato, sono attenuati nella malattia e quindi implicati nella virulenza (Crespi et al.,1992; Maes et al., 2001). Il gene vicA, che codifica la malato sintasi, un enzima del ciclo del gliossilato, è l’unico locus codificato sul cromosoma implicato nella virulenza (Vereeckeet al., 2002).
I rodococchi patogeni sono particolarmente problematici in ambienti agricoli che producono piante per il loro valore estetico. Una varietà di stress biotici e abiotici, alcuni indotti da pratiche antropogeniche, causano sintomi che vengono confusi con quelli causati dal rodococco(Putnam e Miller, 2007). Inoltre, gli isolati di rodococco privi di geni virulenti sono spesso coltivati da tessuti sintomatici (Creasonet al., 2014b; Nikolaeva et al., 2009). Più test vengono utilizzati per confermare che una pianta è infettata da rodococco patogeno. I batteri devono presentare la morfologia corretta su terreni selettivi ed essere tassonomicamente assegnati al rodococco. I batteri devono avere geni virulenti e devono causare sintomi di malattia nelle piante indicatori sensibili. Poiché il gene fasR e alcuni deigeni fas sono necessari per la patogenicità dei batteri, il loro rilevamento è sufficiente a confermare la patogenicità degli isolati di Rhodococcus.
Nel 2011, le popolazioni di pistacchio micropropagato UCB-1(Pistacia atlantica × Pistacia integerrima) piantate in campi commerciali hanno iniziato a mostrare un fenotipo insolito (Stamler et al.,2015a, 2015b). I fenotipi aerei della “sindrome della cima a cespuglio di pistacchio” includono internodi accorciati, perdita di dominanza apicale, galle del gambo e ridotto successo dell’innesto. Le stime suggeriscono che più di 1 milione di alberi cresciuti su 25.000-30.000 acri sono stati colpiti. Gli isolati di rodococco sono stati coltivati da piante sintomatiche e, quando sono stati inoculati su UCB-1, hanno causato cambiamenti morfologici agli ospiti (Stamleret al., 2015b). Questa è stata la prima segnalazione di pistacchio suscettibile al rodococco. Il successivo rilascio delle sequenze genomiche per PBTS1 e PBTS2, i ceppi di focolai segnalati, ha indicato che mancano di loci di virulenza(Stamler et al., 2016). Il rilevamento di vicA è stato utilizzato come prova per i batteri patogeni del rodococco e per guidare le pratiche di gestione, la più estrema e costosa delle quali è la rimozione di interi frutteti. Una seconda incidenza della sindrome della cima a cespuglio di pistacchio si è verificata nel 2016, con la conseguente distruzione di 1,5 milioni e mezzo di alberi da vivaio.
Abbiamo determinato e analizzato le sequenze genomiche di oltre 80 isolati di rodococco, per lo più raccolti da piante erbacee sintomatiche coltivate in ambienti di produzione. L’analisi delle sequenze cromosomiche mostra che le piante ospitano più lignaggi di rodococco. Gli isolati che mancano di plasmidi virulenti possono promuovere cambiamenti nell’architettura delle radici, ma se viene acquisito un plasmide virulento, gli isolati passano ad essere patogeni. L’analisi delle sequenze cromosomiche degli agenti patogeni ha rivelato il potenziale per le infezioni multiple e le popolazioni serbatoio nei siti di vivaio, così come per i focolai di origine puntuale. Tuttavia, i modelli di distribuzione dei plasmidi della virulenza hanno suggerito che i sistemi agricoli possono essere luoghi che promuovono le transizioni evolutive e la rapida generazione di nuovi lignaggi di agenti patogeni, fornendo una via alternativa per la diffusione degli agenti patogeni. Infine, i nostri risultati mettono in discussione le precedenti conclusioni secondo cui gli isolati di rodococco privi di geni della virulenza sono agenti causali della sindrome del cespuglio del pistacchio, suggerendo che la sindrome del pistacchio è stata probabilmente diagnosticata in modo errato (Stamler et al., 2015a,2015b).
Risultati
Collegamenti epidemiologici nei vivai
Abbiamo usato un approccio epidemiologico genomico per studiare i modelli di trasmissione del rodococco in un sistema agricolo vegetale. Sessanta isolati sono stati raccolti gerarchicamente attraverso lo spazio e il tempo. Più isolati sono stati coltivati dagli stessi tessuti sintomatici o da diverse piante coltivate nello stesso sito di produzione(file supplementare 1A). Sono stati inclusi gli isolati precedentemente sequenziati, molti dei quali raccolti anche da siti di produzione(file supplementare 1A; Creason et al., 2014b). Le fonti degli isolati sono state rese anonime.
L’analisi filogenetica ha collocato i 60 isolati all’interno delle Clade I e II del rodococco, che includevano anche i 15 isolati patogeni precedentemente confermati (Figura 1; Creason et al., 2014b). Le due clade sono sorelle delle Clade III e IV. La Clade III è costituita da isolati che sono stati coltivati da microbiota della pianta modello Arabidopsisthaliana(Bai et al., 2015; Bodenhausen et al., 2013; Bulgarelli et al., 2012; Lebeis et al., 2015; Lundberg et al., 2012). Nessun membro delle Cladi III o IV ha geni di virulenza. I quattro cladi sono distinti, e separati da un lungo ramo, dalle altre specie di Rhodococcus(Figura 1-figure supplement 1). Gli isolati nei quattro clades sono stati operativamente classificati in 17 specie, indicate con una lettera minuscola, di cui 14 hanno almeno un isolato coltivato da una pianta(file supplementare 1B).
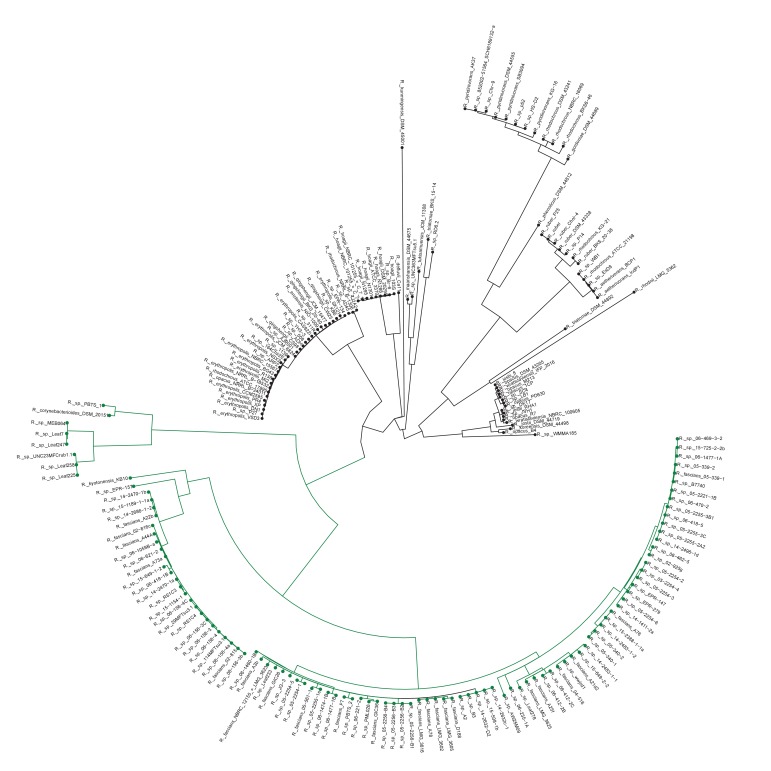
Figura 1-figura supplemento 1.Gli isolati di rodococco associati alle piante formano quattro cladi gemelli.i rodocochi associati alle piante formano un clado distinto.2. Analisi della sequenza multiloco albero di massima probabilità di isolati di rodococco associati alle piante. Le sequenze tradotte per ftsY, infB, rpoB, rsmA, secY, tsaD e ychF da 104 membri di Rodococco sono state identificate utilizzando TBLASTN, allineate e utilizzate per generare un albero di massima probabilità multiloco. Le designazioni delle clade si basano sull’analisi dell’identità nucleotidica media(file supplementare 1B). Le colonne indicano le caratteristiche del corrispondente isolato. Le barre grigie indicano rispettivamente non testato, assente, sconosciuto o altro per fenotipo, plasmide lineare, loci di virulenza e colonne host. La metà sinistra della colonna corrispondente al fenotipo indica un fenotipo confermato, mentre la metà destra indica un’inferenza basata sulla presenza o assenza di geni di virulenza.Analisi della sequenza multiloco albero di massima probabilità di isolati del genere Rhodococcus. Le sequenze tradotte per ftsY, infB, rpoB, rpoB, rsmA, secY, tsaD e ychF di 199 membri di Rhodococcus sono state identificate utilizzando TBLASTN, allineate e utilizzate per generare un albero di massima probabilità multiloco.
Cinquantuno degli isolati appena sequenziati codificano i geni della virulenza. Abbiamo ispezionato i loro gruppi genomici così come quelli degli isolati patogeni precedentemente sequenziati(Creason et al., 2014b). Tutti i 66 gruppi genomici, tranne quattro, avevano att, fasR e fas sullo stesso contig di pFi_009. Il fas locus è presente in una regione che è conservata in pFiD188, il plasmide della virulenza dell’isolato D188, suggerito come necessario per la replicazione e il mantenimento del plasmide (Francis etal., 2012). Si prevede che il gene pFi_009 codifichi una proteina associata al telomero, ipotizzata come necessaria per la replicazione del plasmide della virulenza lineare. Gli assemblaggi genomici degli isolati 06-469-3-2 e 05-2254-6 avevano interruzioni di contig che hanno interrotto i legami tra virulenza e loci associati al plasmide, ma questi contig erano comunque simili nella composizione e sono co-lineari alla sequenza plasmidica di riferimento. La copertura media di sequenziamento dei plasmidi rispetto a quella dei cromosomi corrispondenti è stata di 1,89 ± 0,56. Solo tre gruppi avevano una copertura inferiore a 1,0, ma tutti e tre avevano loci di virulenza sullo stesso contog di pFi_009. A21d2 e A25f, precedentemente sequenziati, sono eccezionali perché i loci della virulenza sono codificati nei loro cromosomi(Creason et al., 2014b). Pertanto, dei 66 isolati confermati o desunti come patogeni, 64 sono portatori di un plasmide della virulenza. Gli isolati patogeni sono stati assegnati a otto specie diverse(Figura 1).
Abbiamo identificato polimorfismi nucleotidici singoli (SNPs) per 82 isolati e abbiamo utilizzato questi SNPs per definire i genotipi e per assemblare due reti a distanza minima specifica del clade (file supplementari1C e 1D; Figura 2). I genotipi mostrano differenze a coppie tra 220 e 11.714 SNPs. Abbiamo mappato le informazioni di nursery sulla rete, che hanno fornito informazioni sui potenziali schemi di trasmissione (Figura 2). In questa figura, l’identificazione ‘a’ è associata a vivai in cui abbiamo identificato prove di infezioni multiple e indipendenti. Le piante del vivaio N15, indicate con ‘a1’, sono state infettate da cinque genotipi appartenenti alla Clade I e due appartenenti alla Clade II. Anche il vivaio N8 (‘a2’) e diversi altri erano associati a genotipi multipli. Tuttavia, il vivaio N8 aveva una singola pianta ospite che era infettata da almeno tre genotipi che rappresentavano sei degli isolati coltivati. I tre isolati all’interno di uno di questi genotipi differiscono fino a 20 SNP a coppie, mentre i due isolati in uno degli altri genotipi non presentano differenze (file supplementare1C). Il terzo genotipo è separato dagli altri due da 231 e 1414 SNPs a coppie(Figura 2E). Il collegamento epidemiologico “b” è stato rilevato nel vivaio N15. Il genotipo corrispondente comprende sette isolati da Clade IIa, campionati quattro anni a parte le piante di Campanula(file supplementare 1A). Questi isolati sono separati da 0-2 SNPs a coppie. Anche i collegamenti epidemiologici designati come “c” sono stati effettuati tra gli isolati raccolti da vivai geograficamente separati. I vivai N1, N12 e N13 (‘c1’) avevano isolati del genotipo associato (Clade IIb) che sono separati da 1-4 SNPs a coppie e sono stati raccolti fino a 13 anni a parte le piante di Leucanthemum e Geranio. I vivai N7, N11 e N14 (‘c2’) avevano isolati del genotipo associato (Clade Ia) che erano separati da 12-20 SNPs a coppie. Gli isolati sono stati raccolti a nove anni di distanza l’uno dall’altro, e da piante di Veronica.
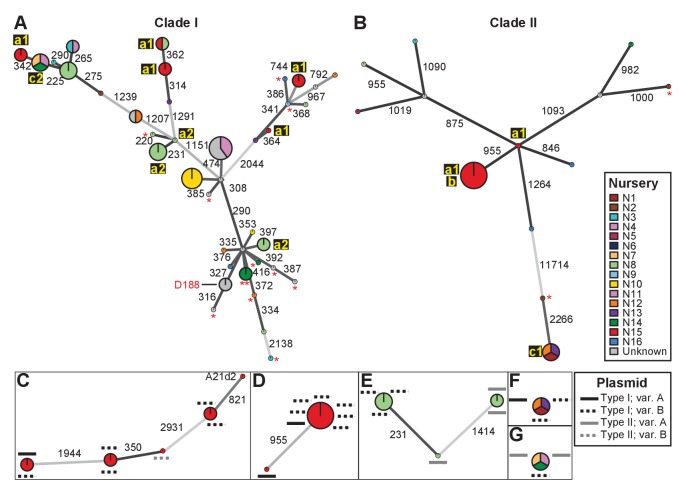
Figura 2.L’analisi di SNPs rivela tre modelli di trasmissione di rodococco patogeno.Minime reti di distribuzione degli isolati di(A) Clade I e(B) Clade II. Ogni genotipo viene visualizzato come un cerchio, con dimensioni scalate per rappresentare il numero di isolati associati (più piccolo = 1 isolato). I colori rappresentano la sorgente degli isolati (vedi chiave), con colorazione proporzionale al rapporto degli isolati di ogni sorgente. Le lettere minuscole e i numeri (a1, a2, b, b, c1 e c2) evidenziano i potenziali schemi di trasmissione; vedi pannelli C-G). Asterischi = mancanza di geni di virulenza. Viene indicato il genotipo che include D188.(C, D) Reti a raggio minimo di isolati patogeni appartenenti alla Clade I (C;‘a1 ‘) e alla Clade II (D; ‘a1’e ‘b’) del vivaio N15. A21d2 manca di un plasmide virulento e i suoi loci virulenti sono presenti nel cromosoma.(E) La rete minima di copertura per gli isolati di isolati patogeni dal vivaio N8 (‘a2’).(F) Il collegamento epidemiologico ‘c1’ tra gli isolati da vivai N1, N12, e N13.(G) Il collegamento epidemiologico ‘c2’ tra gli isolati dai vivai N7, N11, e N14. I tipi di plasmide e le loro varianti sono mappati su ciascuno dei nodi (vedi chiave). I numeri adiacenti alle linee di collegamento indicano il numero di SNP che separano ciascun genotipo. Le lunghezze delle linee di connessione sono arbitrarie; le linee grigie indicano distanze che superano una soglia arbitraria.

Figura 1-figure supplement 1.Gli isolati di rodococco associati alle piante formano quattro clade sorelle.i rodocochi associati alle piante formano un clade distinto.2. Analisi della sequenza multiloco albero di massima probabilità di isolati di rodococco associati alle piante. Le sequenze tradotte per ftsY, infB, rpoB, rsmA, secY, tsaD e ychF da 104 membri di Rodococco sono state identificate utilizzando TBLASTN, allineate e utilizzate per generare un albero di massima probabilità multiloco. Le designazioni delle clade si basano sull’analisi dell’identità nucleotidica media(file supplementare 1B). Le colonne indicano le caratteristiche del corrispondente isolato. Le barre grigie indicano rispettivamente non testato, assente, sconosciuto o altro per fenotipo, plasmide lineare, loci di virulenza e colonne host. La metà sinistra della colonna corrispondente al fenotipo indica un fenotipo confermato, mentre la metà destra indica un’inferenza basata sulla presenza o assenza di geni di virulenza.Analisi della sequenza multiloco albero di massima probabilità di isolati del genere Rhodococcus. Le sequenze tradotte per ftsY, infB, rpoB, rpoB, rsmA, secY, tsaD e ychF di 199 membri di Rhodococcus sono state identificate utilizzando TBLASTN, allineate e utilizzate per generare un albero di massima probabilità multiloco.

Figura 1-figure supplement 1.I rodocochi associati alle piante formano un clade distinto.2. Analisi della sequenza multiloco albero di massima probabilità di isolati del genere Rhodococcus. Le sequenze tradotte per ftsY, infB, rpoB, rpoB, rsmA, secY, tsaD e ychF da 199 membri del Rhodococcus sono stati identificati utilizzando TBLASTN, allineati, e utilizzati per generare un albero di massima probabilità multiloco.

Figura 2.L’analisi degli SNP rivela tre modelli di trasmissione di rodococco patogeno.Minime reti di spaziatura delle reti di isolati di(A) Clade I e(B) Clade II. Ogni genotipo viene visualizzato come un cerchio, con dimensioni scalate per rappresentare il numero di isolati associati (più piccolo = 1 isolato). I colori rappresentano la sorgente degli isolati (vedi chiave), con colorazione proporzionale al rapporto degli isolati di ogni sorgente. Le lettere minuscole e i numeri (a1, a2, b, b, c1 e c2) evidenziano i potenziali schemi di trasmissione; vedi pannelli C-G). Asterischi = mancanza di geni di virulenza. Viene indicato il genotipo che include D188.(C, D) Reti a raggio minimo di isolati patogeni appartenenti alla Clade I (C;‘a1 ‘) e alla Clade II (D; ‘a1’e ‘b’) del vivaio N15. A21d2 manca di un plasmide virulento e i suoi loci virulenti sono presenti nel cromosoma.(E) La rete minima di copertura per gli isolati di isolati patogeni dal vivaio N8 (‘a2’).(F) Il collegamento epidemiologico ‘c1’ tra gli isolati da vivai N1, N12, e N13.(G) Il collegamento epidemiologico ‘c2’ tra gli isolati dai vivai N7, N11, e N14. I tipi di plasmide e le loro varianti sono mappati su ciascuno dei nodi (vedi chiave). I numeri adiacenti alle linee di collegamento indicano il numero di SNP che separano ciascun genotipo. Le lunghezze delle linee di collegamento sono arbitrarie; le linee grigie indicano distanze che superano una soglia arbitraria.
I modelli di distribuzione multipla dei plasmidi si verificano in isolati coltivati in ambiente agricolo
I plasmidi della virulenza di 64 isolati di Rhodococcus patogeni per piante sono stati classificati sulla base dell’analisi filogenetica di 123 geni che sono presenti in almeno il 95% dei plasmidi della virulenza, e sotto-categorizzati sulla base dei modelli di presenza/assenza del gene (Figura 3; Figura 3-figure supplement 1; File supplementare 1E). La filogenesi ha un forte supporto per due tipi principali di plasmidi, così come un unico tipo portato solo da isolato LMG3616 (Figura 3-figure supplement1). Le sequenze di plasmidi all’interno dei plasmidi principali sono conservate e forniscono pochi polimorfismi nucleotidici informativi per la sotto-categorizzazione dei plasmidi. Il maggior contributo alla diversità dei plasmidi è il guadagno e la perdita di geni, e il relativo raggruppamento dei plasmidi basato sulla presenza/assenza di geni permette il sottogruppo in varianti INDEL, come indicato con lettere maiuscole nella Figura 3. A livello di tipo plasmidico, la categorizzazione della presenza/assenza era identica alla filogenesi(Figura 3-figure supplement 1). I geni che definiscono ogni variante INDEL sono spesso presenti in regioni in gran parte contigue nel plasmide, e potrebbero essere stati acquisiti come blocchi(Francis et al., 2012).
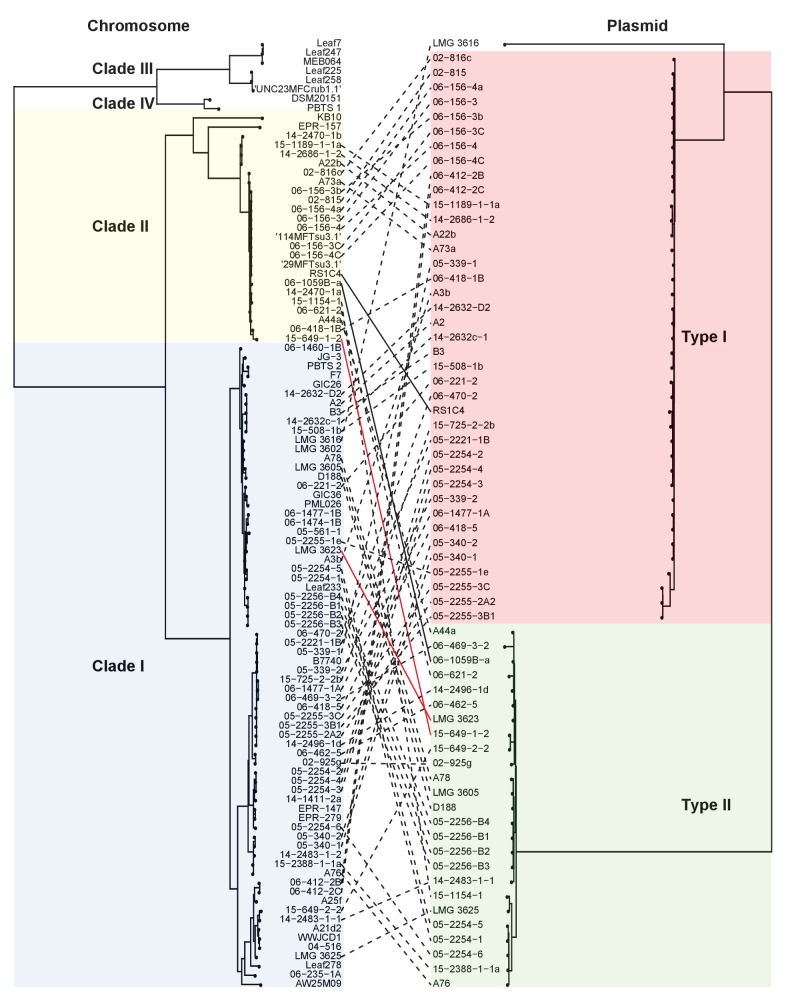
Figura 3-figure supplement 2.L’analisi della variazione del plasmide rivela modelli multipli nella distribuzione.Presenza/assenza-cladogramma e filogenesi supportano lo schema di classificazione per il plasmide della virulenza.entrambi i tipi di plasmidi della virulenza sono presenti in entrambi i cladi di rodococco patogeni .Le righe indicano i geni presenti in (blu) o assenti da (bianco) i plasmidi degli isolati (elencati a destra; D188 è etichettato in rosso per riferimento). Le colonne rappresentano i singoli geni. Le categorie di tipo sono state determinate sulla base dell’analisi filogenetica dei geni del nucleo. Le varianti INDEL, delineate da ombreggiature grigie e bianche, sono state determinate sulla base del cladogramma. Il plasmide trasportato da LMG3616 è racchiuso da linee tratteggiate. Le lettere minuscole e i numeri (a1, a2, b, b, c1, e c2) elencati lungo la destra, mettono in relazione gli isolati e i loro plasmidi con i potenziali schemi di trasmissione indicati nella Figura 2.Le linee tratteggiate mostrano la congruenza nei risultati dei metodi utilizzati per raggruppare i tipi di plasmidi; (a sinistra) i plasmidi sono stati raggruppati in un cladogramma sulla base della presenza/assenza di geni e (a destra) in un albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi di virulenza. I blocchi rossi e verdi delineano rispettivamente i plasmidi di tipo I e II.L’albero filogenetico dell’analisi della sequenza multiloco (MLSA) dei quattro cladi associati alle piante è stato associato all’albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi della virulenza. Le linee tra gli alberi mostrano relazioni tra isolati e plasmidi. Le due coppie di linee solide (nere e rosse) evidenziano esempi di isolati strettamente correlati che hanno tipi di plasmidi divergenti e isolati distanti che hanno tipi di plasmidi simili.
Abbiamo poi caratterizzato la distribuzione dei plasmidi in isolati epidemiologicamente collegati e abbiamo mostrato che i modelli di plasmidi erano incoerenti con quelli previsti di trasmissione tra gli isolati di un genotipo cromosomico (Figure2C-G e 3). Un genotipo associato con ‘a1’ (il nodo più a sinistra della rete; Figura 2C) comprende gli isolati 05-339-1 e 05-339-2, che differiscono per sette SNP a coppie e che portano plasmidi di tipo IA e tipo IB, rispettivamente. Il genotipo associato a ‘b’ include l’isolato 02-815, che trasporta un plasmide di Tipo IA (Figura 2D). Questo isolato è epidemiologicamente legato a sei isolati (0-2 SNPs coppia), ma gli isolati raccolti quattro anni dopo portano un plasmide di tipo IB virulenza. Allo stesso modo, il genotipo associato a ‘c1’ include l’isolato A22b, che porta un plasmide della virulenza di Tipo IA ed è collegato a due isolati che portano plasmidi di Tipo IB (Figura 2F). La deviazione più evidente si trova nel genotipo associato a ‘c2’ (Figura 2G). Isolato 06-1477-1A porta un plasmide di tipo IB ed è epidemiologicamente legato a due isolati che portano un plasmide di tipo IIA. Anche i due plasmidi di Tipo IIA sono dissimili in quanto uno ha un blocco di geni che, insieme ad altri geni INDEL, lo distingue dall’altro.
I diversi genotipi di rodococco isolati da vivai possono portare tipi di plasmidi simili e questi tipi non sono tassonomicamente limitati. Gli isolati 06-412-2B della Clade Ib, 05-340-1 della Clade Ie e 06-156-3c della Clade IIa, tutti del gruppo ‘a1’ rilevato nel vivaio N15, hanno plasmidi di tipo IB che differiscono solo per alcuni geni INDEL (Figura 2C e D). La filogenesi mostra che ci sono casi in cui gli isolati dallo stesso clade, come RS1C4 e 06-1059B-a (linee nere solide), portano diversi tipi di plasmidi(Figura 3-figure supplement 2; linee nere solide). È stato anche osservato che i rodococcus isolati correlati a distanza portano lo stesso tipo di plasmide. Per esempio, 15-649-1-2 del Clade II e LMG 3623 del Clade I portano entrambi plasmidi di tipo II(Figura 3-figure supplement 2; linee rosse solide).
Due isolati patogeni sono stati esclusi dall’analisi perché i loci della virulenza sono presenti nei loro cromosomi (Creasonet al., 2014b). A25f è stato recuperato dal vivaio N12, mentre A21d2 è stato recuperato dal vivaio N15(file supplementare 1A).

Figura 3-figure supplementary 2.L’analisi della variazione dei plasmidi rivela modelli multipli nella distribuzione.Presenza/assenza-cladogramma e filogenesi supportano lo schema di classificazione per il plasmide della virulenza.entrambi i tipi di plasmidi della virulenza sono presenti in entrambi i cladi di rodococco patogeni .Le righe indicano i geni presenti in (blu) o assenti da (bianco) i plasmidi degli isolati (elencati a destra; D188 è etichettato in rosso per riferimento). Le colonne rappresentano i singoli geni. Le categorie di tipo sono state determinate sulla base dell’analisi filogenetica dei geni del nucleo. Le varianti INDEL, delineate da ombreggiature grigie e bianche, sono state determinate sulla base del cladogramma. Il plasmide trasportato da LMG3616 è racchiuso da linee tratteggiate. Le lettere minuscole e i numeri (a1, a2, b, b, c1, e c2) elencati lungo la destra, mettono in relazione gli isolati e i loro plasmidi con i potenziali schemi di trasmissione indicati nella Figura 2.Le linee tratteggiate mostrano la congruenza nei risultati dei metodi utilizzati per raggruppare i tipi di plasmidi; (a sinistra) i plasmidi sono stati raggruppati in un cladogramma sulla base della presenza/assenza di geni e (a destra) in un albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi di virulenza. I blocchi rossi e verdi delineano rispettivamente i plasmidi di tipo I e II.L’albero filogenetico dell’analisi della sequenza multiloco (MLSA) dei quattro cladi associati alle piante è stato associato all’albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi della virulenza. Le linee tra gli alberi mostrano relazioni tra isolati e plasmidi. Le due coppie di linee solide (nere e rosse) evidenziano esempi di isolati strettamente correlati che hanno tipi di plasmidi divergenti e isolati distanti che hanno tipi di plasmidi simili.
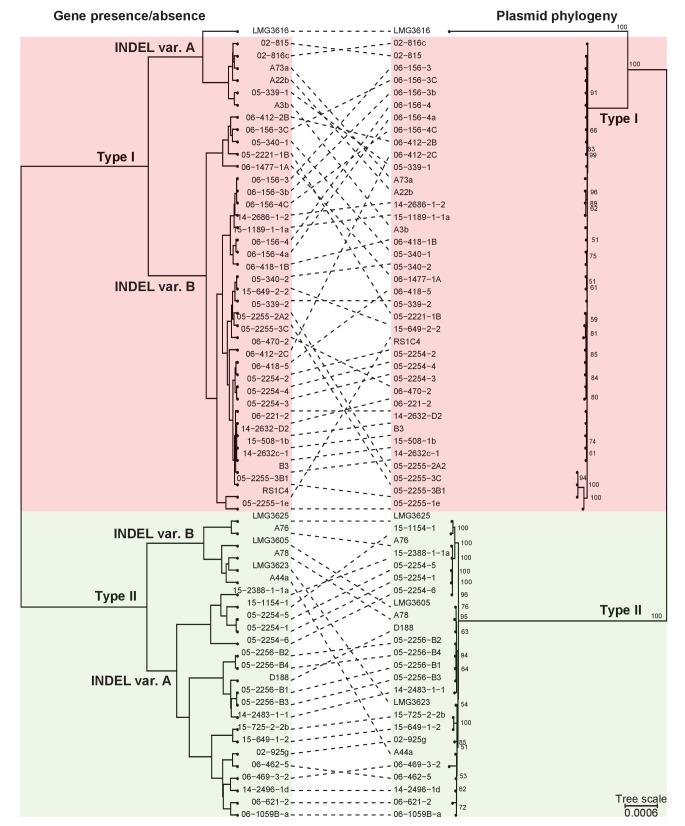
Figura 3-figure supplement 1.Presenza/assenza-cladogramma e filogenesi supportano lo schema di classificazione del plasmide della virulenza.Le linee tratteggiate mostrano la congruenza nei risultati dei metodi utilizzati per raggruppare i tipi di plasmidi; (a sinistra) i plasmidi sono stati raggruppati in un cladogramma sulla base della presenza/assenza di geni e (a destra) in un albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi della virulenza. I blocchi rossi e verdi delineano rispettivamente i plasmidi di tipo I e II.

Figura 3-figure supplement 2.Entrambi i tipi di plasmidi della virulenza sono presenti in entrambi i cladi di rodococco patogeni.L’albero filogenetico di analisi della sequenza multiloco (MLSA) dei quattro clades associati alle piante è stato associato all’albero filogenetico sulla base delle sequenze concatenate di 123 geni che sono presenti in più del 95% delle sequenze di plasmidi della virulenza. Le linee tra gli alberi mostrano relazioni tra isolati e plasmidi. Le due coppie di linee solide (nere e rosse) evidenziano esempi di isolati strettamente correlati che hanno tipi di plasmidi divergenti e isolati distanti che hanno tipi di plasmidi simili.
Gli isolati di rodococco che mancano del gene della virulenza promuovono cambiamenti nell’architettura delle radici
In questo set di dati, abbiamo identificato nove isolati di rodococco chemancano di geni virulenti. Abbiamo anche identificato cinque di questi isolati in uno studio precedente, e spesso non riescono a rilevare i geni della virulenza durante la diagnosi del rodococco coltivato da piante malate (Creason etal., 2014b). Altri hanno sottinteso che si tratta di ceppi che hanno perso il plasmide (Nikolaeva et al., 2009). La diversità genetica degli isolati coesistenti qui descritti suggerisce il contrario(Figura 1; File supplementari 1A-D). Pathogenic 06-1477-1A e virulenza-gene-nolente 06-1477-1B sono stati isolati da una pianta sintomatica Veronica, appartengono a Clades Id e Ia, rispettivamente, e hanno 1521 SNPs coppia. L’isolato patogeno 14-2483-1-1 (Clade Ib) è stato coltivato dalla stessa pianta sintomatica dell’isolato 14-2483-1-2 (Clade Ie), e i due differiscono per 2820 SNPs. È interessante notare che 14-2483-1-2 ha i geni della virulenza attR e attX, così come 228 nucleotidi del gene della virulenza attA, su un contig che è dissimile in sequenza e maggiore in lunghezza rispetto ai plasmidi della virulenza. Le prime due sequenze di codifica e le regioni intergeniche hanno un’identità nucleotidica ≥88% rispetto alle corrispondenti sequenze di D188. Per attA, solo i primi 65 nucleotidi sono identici al suo omologo in D188. I risultati della PCR hanno confermato che la struttura del locus non è il risultato di un assemblaggio errato. Due isolati di virulenza-gene-nerodi, 14-2470-1a e 14-2470-1b, sono stati coltivati da una pianta sintomatica. Questi due isolati sono rispettivamente nelle Clades IIa e IIb e differiscono di 13.858 SNPs. Degli isolati coltivati e testati da questa pianta, non è stato rilevato alcun isolato patogeno.
Una spiegazione alternativa per la presenza di isolati di Rhodococcus geneticamente diversi, virulenti e privi di gene, è che tali isolati sono benefici e arricchiti dalle piante. Abbiamo quindi confrontato il fenotipo di simbiosi di sette isolati di virulenza-plasmide-nolente che rappresentano i quattro cladi contro quattro isolati portatori di virulenza-gene, D188, A44a, A25f e A21d2 (file supplementare1F; Creason et al., 2014b; Desomer et al., 1988; Lundberg et al., 2012; Miteva et al., 2004 ). Gli isolati portatori di virulenza-gene sono stati precedentemente determinati come patogeni e sono stati selezionati in base alle variazioni della struttura e della sequenza dei loro loci di virulenza (Creason etal., 2014b). Sono stati inclusi anche i PBTS1 e PBTS2, implicati come ceppi epidemici e coltivati dal comparto endofitico fogliare del pistacchio(Stamler et al., 2015b).
I sette isolati di virulenza-gene-lacking, così come PBTS1 e PBTS2, non sono riusciti a causare la malattia quando sono stati inoculati sui meristemi di Nicotiana benthamiana matura. I quattro isolati patogeni hanno causato le ghiandole fogliari(Figura 4A). Questi quattro sono stati anche gli unici testati che hanno causato una significativa inibizione dell’allungamento delle radici, l’ispessimento del fusto e l’arresto terminale allo stadio di cotiledone (nessuna crescita primaria o sviluppo delle radici laterali), quando saggiato su piantine di N. ben thamiana(Figura 4B e C). Abbiamo esaminato le piante fino a due mesi dopo l’inoculazione, e le piantine sono rimaste in arresto terminale. La maggior parte degli isolati ha ridotto la crescita verticale delle radici, rispetto a quella delle piante mock-inoculate, ma l’inibizione da parte di virulenza-gene-nolente isolati era più variabile e non così grave come quella misurata nelle radici di piantine inoculate da agenti patogeni (p-valori erano <0,0001 tranne che per il trattamento con PBTS2 [p-valore = 0,1153]). È importante notare che le piantine inoculate con isolati di virulenza-gene-nolente non hanno mostrato ispessimento dello stelo o arresto terminale allo stadio di cotiledone, cambiamenti morfologici associati alla malattia(Figura 4).
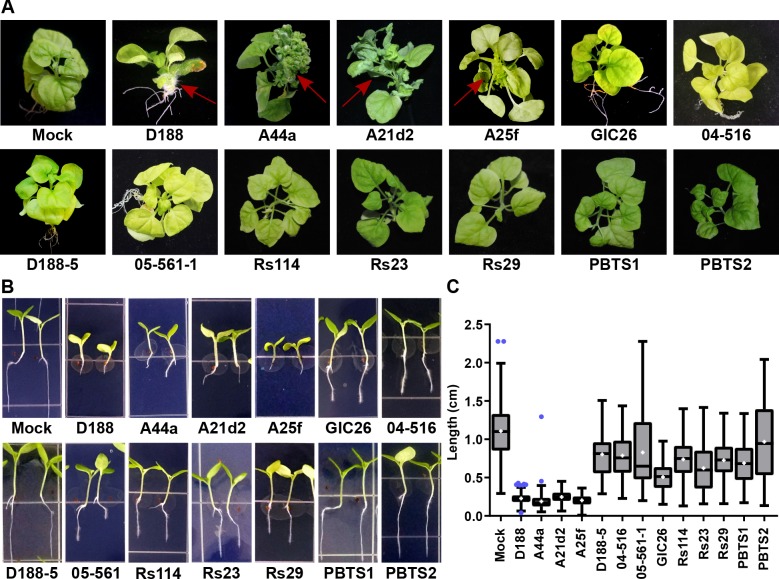
Figura 4 dati fonte 1.Gli att, fasR e fas loci sono necessari per la patogenicità del rodococco .2.Lunghezzedelle radicidellepiantine diN.benthamiana7 giorni dopo l’inoculazione con rodococcusisolati selvatici di tipo Rhodococcusisolati.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Isolati di Rodococcusisolati sono stati inoculati al meristema apicale. Le frecce rosse indicano le palle a foglia. Rs114, Rs23, e Rs29 sono abbreviazioni per gli isolati 114MFTsu3.1, UNC23MFCrub1.1, e 29MFTsu3.1, rispettivamente.(B) Immagini rappresentative della lunghezza delle radici delle piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di Rhodococcus o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante. Gli isolati D188, A44a, A21d2 e A25f sono gli unici isolati con geni di virulenza.(C) Quantificazione della lunghezza delle radici delle piantine. Tutti i trattamenti, ad eccezione di PBTS2, sono stati significativi rispetto al finto trattamento.10.7554/eLife.30925.010Figure 4-source data 1.Lunghezze delle radici dellepiantinedi N. benthamiana 7 giorni dopo l’inoculazione con Rhodococcusisolati di tipo selvatico.
Invece, abbiamo notato che tutti gli isolati di virulenza-genio-nolente hanno causato cambiamenti nell’architettura delle radici (Figura 4B). Rispetto alle piante mock-inoculate, c’erano proliferazioni nei peli delle radici e le piante avevano più radici laterali o un precedente sviluppo delle radici laterali. Il primo cambiamento è stato quantificato in piantine che sono stati inoculati con i membri di un sottoinsieme di virulenza-gene-nolente isolati. Ci sono stati aumenti significativi del numero di peli delle radici (medie che vanno da 166,1 a 217,3; i valori p erano tutti ≤ 0,0045), rispetto alle piante inoculate in modo beffardo (media di 89,8; Figura 5A e B). Gli isolati variavano nel loro effetto, con PBTS2 che ha il più forte effetto misurabile (217,3; p-valore<0,0001). GIC26 ha provocato la proliferazione dei peli delle radici più sorprendente dal punto di vista visivo e il suo effetto estremo ha messo in discussione la nostra capacità di contare e misurare accuratamente i peli delle radici (Figura 5A; p-valore=0,0003). La lunghezza media dei peli delle radici era significativamente più lunga dopo l’inoculazione con i quattro isolati di rodococco (le medie variavano da 0,02286 a 0,03604 mm contro 0,01186 mm nelle piante mock-inoculate; p-valori erano tutti <0,0001; Figura 5C ). L’isolato patogeno D188 è stato incluso come controllo, ma i peli delle radici delle piante che sono stati inoculati con questo isolato erano troppo scarsi in numero per giustificare la quantificazione (Figura 5A). Cinque isolati aggiuntivi, tra cui 14-2483-1-2 che ha parte del locus att, sono stati testati e hanno dimostrato di causare cambiamenti nell’architettura delle radici delle piante (Figura 5-figure supplement 1).
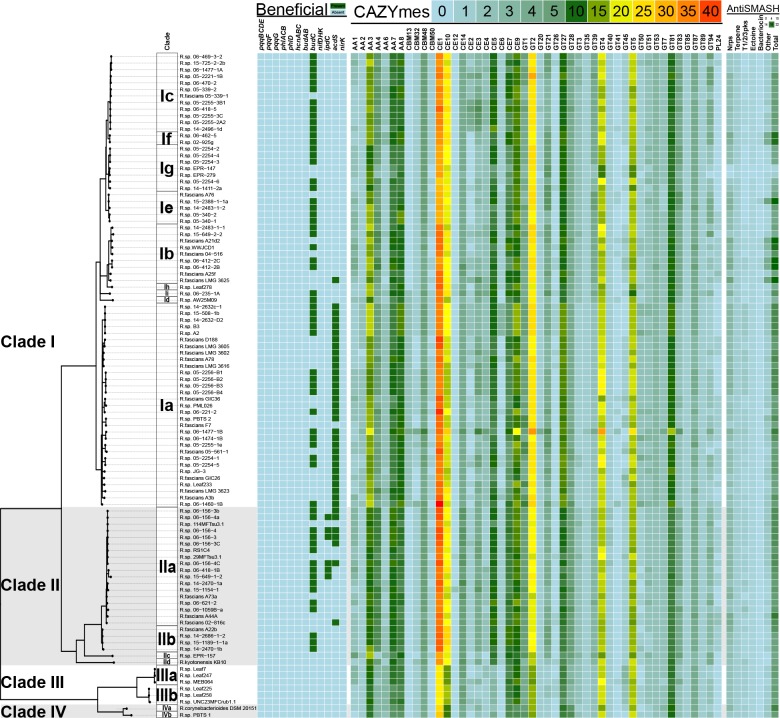
Figura 5-figure supplement 2.Figura 5—figura supplemento 2. I batteri Rhodococcus associati alle piante causano cambiamenti all’architettura delle radici delle piantine.Numeri di N.peli delle radici delle piantinedi N. benthamiana 25 giorni dopo l’inoculazione con isolati di Rhodococcus di tipo selvatico.Numeri di N. peli delleradicidi N. benthamiana25 giornidopo l’inoculazione con isolati di Rhodococcus di tipo selvatico.Numeri di N. (A) Immagini rappresentative dei peli delle radici di N. benthamiana che sono stati inoculati con isolati di rodococco . Le immagini sono state scattate 25 giorni dopo l’inoculazione (dpi). La freccia bianca indica lo stelo più spesso indotto solo dall’isolato D188. Le barre di scala sono di 0,5 mm.(B) Quantificazione del numero medio di capelli radicali a 25 dpi. Tutti i peli delle radici sono stati contati manualmente per almeno cinque piantine per trattamento.(C) Quantificazione della lunghezza dei peli delle radici a 25 dpi. Tutti i peli delle radici sono stati misurati manualmente per almeno cinque piantine per trattamento. Per B e C, i dati sono stati ripetuti in due repliche biologiche indipendenti. * indica una differenza significativa rispetto al finto trattamento.10.7554/eLife.30925.012Figure 5-source data 1.Numeri di N. benthamiana peli delleradici delle piantine25 giorni dopo l’inoculazione con isolati di rodococco di tipo selvatico.10.7554/eLife.30925.013Cifre dati a 5 fonti 2.Lunghezza dei peli delle radici di N. benthamiana25 giorni dopo l’inoculazione con isolati di rodococco di tipo selvatico.Immagini rappresentative di piantine inoculate con gli isolati indicati di Rhodococcuso acqua (mock). Le lunghezze delle radici delle piantine inoculate con isolati privi di geni virulenti erano diverse da quelle delle piantine inoculate con l’isolato D188.I geni associati alle funzioni vegetali benefici sono stati utilizzati come query nelle ricerche di TBLASTN per gli omologhi in Rodococco (verde = presenza [>40% identità, >70% lunghezza]; ciano = assenza). Per i CAZYmes vengono mostrate solo le 48 classi più rappresentate. La chiave mostra il numero di omologhi rilevati. I presunti cluster di metaboliti secondari biosintetici sono stati identificati utilizzando l’antiSMASH. La chiave mostra il numero di loci previsti per ogni categoria.
Abbiamo analizzato le sequenze del genoma per identificare i geni che sono potenzialmente coinvolti nel fornire tratti benefici (Figura 5-figuresupplement 2; Bruto et al., 2014; Glick, 2012; Sparacino-Watkins et al., 2014). Pochi omologhi o percorsi sono stati associati a tratti benefici o a uno stile di vita endofitico. Non c’era inoltre una correlazione evidente con il fenotipo della simbiosi. Alcuni isolati hanno un omologo previsto per codificare la deaminasi 1-amminociclopropan-1-carbossilato (ACC), ma questa sequenza ha avuto un forte segnale filogenetico, ed è prevalentemente nei membri di Clade Ia (Glick, 2014). Nessun percorso completo triptofano dipendente auxina biosintetico è stato identificato(Spaepen et al., 2007). Le classi più rappresentate di enzimi attivi dei carboidrati (CAZYmes) sono i membri dei gruppi di carboidrati esterasi CE1 e CE10. CE1 comprende le xilanasi, che degradano l’emicellulosa(Lombard et al., 2014). CE10 è costituito da colinesterasi, un gruppo di enzimi che agiscono su fonti non carboidrate. Inoltre, abbiamo identificato le cutinasi (C5) e le liasi pectate (PL22). Queste classi di enzimi possono contribuire allo stile di vita endofitico del rodococco. Ci sono 108 geni che si arricchiscono nei genomi degli isolati dei quattro cladi associati alle piante(file supplementare 1G). Quaranta geni sono annotati come ipotetici e molti altri mancano di informazioni sufficienti nelle loro annotazioni. Analisi AntiSMASH identificato ovunque da 1 a ~ 20 loci che possono essere coinvolti nella produzione di metaboliti secondari (Figura 5-figuresupplement 2; Blin et al., 2017). Pochi avevano sufficienti somiglianze con loci precedentemente caratterizzati per consentire l’inferenza dell’identità del metabolita. Non si sa se e come le funzioni di questi geni contribuiscano allo stile di vita associato alle piante.

Figura 4 dati fonte 1.I loci att, fasR e fas loci sono necessari per la patogenicità del rodococco .2. Lunghezze delle radici dellepiantine diN.benthamiana7 giorni dopo l’inoculazione con rodococcusisolati selvatici di tipo Rhodococcusisolates.lunghezze delleradicidelle piantine di N. benthamiana7 giornidopo l’inoculazione con rodococcusolates selvatici di tipo Rhodococcusolates.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Isolati di Rodococcusisolati sono stati inoculati al meristema apicale. Le frecce rosse indicano le palle a foglia. Rs114, Rs23, e Rs29 sono abbreviazioni per gli isolati 114MFTsu3.1, UNC23MFCrub1.1, e 29MFTsu3.1, rispettivamente.(B) Immagini rappresentative della lunghezza delle radici delle piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di Rhodococcus o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante. Gli isolati D188, A44a, A21d2 e A25f sono gli unici isolati con geni di virulenza.(C) Quantificazione della lunghezza delle radici delle piantine. Tutti i trattamenti, ad eccezione di PBTS2, sono stati significativi rispetto al finto trattamento.10.7554/eLife.30925.010Figure 4-source data 1.Lunghezze delle radici dellepiantinedi N. benthamiana 7 giorni dopo l’inoculazione con Rhodococcusisolati di tipo selvatico.

Figura 5-figure supplemento 2.Figura 5—supplemento figura 2. I batteri associati alle piante causano cambiamenti nell’architettura delle radici delle piantine.Numeri di N.peli delle radici delle piantine di N. benthamiana 25 giorni dopo l’inoculazione con isolati di rodococco di tipo selvatico.Numeri di N. peli delle radici di N. benthamiana25 giornidopo l’inoculazione con isolati di rodococco di tipo selvatico.Numeri di N. (A) Immagini rappresentative dei peli delle radici di N. benthamiana che sono stati inoculati con isolati di rodococco . Le immagini sono state scattate 25 giorni dopo l’inoculazione (dpi). La freccia bianca indica lo stelo più spesso indotto solo dall’isolato D188. Le barre di scala sono di 0,5 mm.(B) Quantificazione del numero medio di capelli radicali a 25 dpi. Tutti i peli delle radici sono stati contati manualmente per almeno cinque piantine per trattamento.(C) Quantificazione della lunghezza dei peli delle radici a 25 dpi. Tutti i peli delle radici sono stati misurati manualmente per almeno cinque piantine per trattamento. Per B e C, i dati sono stati ripetuti in due repliche biologiche indipendenti. * indica una differenza significativa rispetto al finto trattamento.10.7554/eLife.30925.012Figure 5-source data 1.Numeri di N. benthamiana peli delleradici delle piantine25 giorni dopo l’inoculazione con isolati di rodococco di tipo selvatico.10.7554/eLife.30925.013Figure 5-source data 2.Lunghezze dei peli delle radici di N. benthamiana25 giorni dopo l’inoculazione con isolati di rodococco di tipo selvatico.Immagini rappresentative di piantine inoculate con gli isolati indicati di Rhodococcuso acqua (mock). Le lunghezze delle radici delle piantine inoculate con isolati privi di geni virulenti erano diverse da quelle delle piantine inoculate con l’isolato D188.I geni associati alle funzioni vegetali benefici sono stati utilizzati come query nelle ricerche di TBLASTN per gli omologhi in Rodococco (verde = presenza [>40% identità, >70% lunghezza]; ciano = assenza). Per i CAZYmes vengono mostrate solo le 48 classi più rappresentate. La chiave mostra il numero di omologhi rilevati. I presunti cluster di metaboliti secondari biosintetici sono stati identificati utilizzando l’antiSMASH. La chiave mostra il numero di loci previsti per ogni categoria.

Figura 5-figure supplemento 1.Cinque isolati aggiuntivi di virulenza-genio-nolente di Rhodococcus causano cambiamenti nell’architettura delle radici.2. Immagini rappresentative di piantine inoculate con gli isolati indicati di rodococcoo acqua (mock). Le lunghezze delle radici delle piantine inoculate con isolati privi di geni virulenti erano diverse da quelle delle piantine inoculate con l’isolato D188.

Figura 5-figure supplemento 2.Heatmap di geni e funzioni arricchito in clades di Rhodococcus associati alle piante.I geni associati a funzioni vegetali-beneficiarie sono stati utilizzati come query in TBLASTN ricerche di omologhi in rodococco (verde = presenza [>40% identità, >70% lunghezza]; ciano = assenza). Per i CAZYmes vengono mostrate solo le 48 classi più rappresentate. La chiave mostra il numero di omologhi rilevati. I presunti cluster di metaboliti secondari biosintetici sono stati identificati utilizzando l’antiSMASH. La chiave mostra il numero di loci previsti per ogni categoria.
Il plasmide virulenza è sufficiente per transizione Rhodococcus Rhodococcus da essere potenzialmente benefico ad essere patogeno
Il plasmidedella virulenza pFiD188Δ att è stato coniugato con successo in un sottoinsieme di isolati di rodococco che originariamente mancavano di geni della virulenza. Questa variante del plasmide codifica fasR e fasA-F, ma ha un gene di resistenza alla kanamicina che disturba attR, attX e attA-G(Maes et al., 2001). Indipendentemente da ciò, il plasmide pFiD188Δ attè sufficiente per isolare D188 per causare malattie in piante mature e piantine, e le piante che sono state trattate con un ceppo contenente questo plasmide non erano diversi da quelli infettati con D188 (p-valore>0,9999; Figura 6-figure supplement 1; Maes et al., 2001). Nonostante i ripetuti tentativi, non siamo stati in grado di coniugare il plasmide in isolati al di fuori del Clade I con successo. Ognuno degli isolati pFiD188Δ att-carrying ha causato delle gallie fogliari sulle piante (Figura 6A). Inoltre, questi isolati non erano più in grado di causare aumenti nella crescita dei peli delle radici, a differenza dei loro corrispondenti genotipi quasi isogenici (Figura 6B). Invece, gli isolati portatori di pFiD188Δ att, se confrontati con i loro genotipi quasi isogenici che rallentano il plasmide, causavano sintomi di malattia e inibivano significativamente la crescita delle piantine (Figura 6B-C; i valori perano tutti <0,0001).
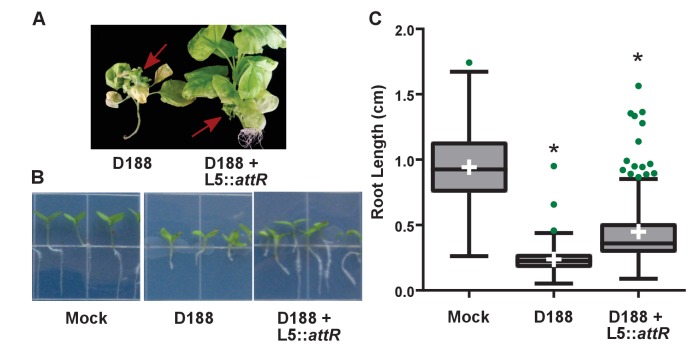
Figura 6-figure supplemento 3-figure dati fonte 1.Figura 6—figura supplemento 3—dati di fonte 1. Plasmide pFiD188 con fasR funzionale e fas è sufficiente per la transizione Rhodococcus isolati a fitopatogeni.lunghezze di N. radicipiantina benthamiana7 giorni dopo l’inoculazione con Rhodococcusisolates+ / – pFiD188Δatt. Lunghezze di N. radici piantinabenthamiana7 giorni dopo l’inoculazione con Rodococcusolates + / –pFiD188Δatt. Ilrodococco isolato D188 con plasmide pFiD188Δ attè patogeno.lo sfratto del plasmide virulento ritorna ad isolare D188 in un batterio benefico.valori di densità ottica di batteri in coltura.valori di densità ottica di batteri in coltura.valori di densità ottica di batteri in coltura.Rodococco isolato D188 portare L5::attR è interessato in virulenza.lunghezze di N. radicipiantina benthamiana 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.lunghezzedi N.piantina benthamianaradici 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Le frecce rosse indicano le palle a foglia. Le immagini per GIC26, 04-516, e PBTS2 sono ripetute dalla Figura 1.(B) Immagini rappresentative delle lunghezze delle radici delle piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di Rhodococcus o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. benthamiana. In tutti i pannelli, -/+ indica l’assenza o la presenza di pFiD188Δ att. * indica una differenza significativa rispetto alle piante trattate con il corrispondente genotipo privo di plasmide.10.7554/eLife.30925.022Figure 6-source data 1.Lunghezze delle radici dellepiantinedi N. benthamiana 7 giorni dopo l’inoculazione con Rodococcusolates+/- pFiD188Δatt.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Le immagini sono state scattate 56 giorni dopo l’inoculazione (dpi). La freccia rossa indica la cistifellea a foglia.(B) Immagini rappresentative della lunghezza delle radici delle piantine. Tre giorni, le piantine di N. ben thamiana sono state inoculate con l’isolato indicato di rodococco o acqua (finto) e cresciute verticalmente per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. I dati per D188 +pFiD188Δ att sonogli stessi di quelli mostrati in Figura 6.(A) Il rodococco D188ΔpFID188 causa cambiamenti nell’architettura delle radici. Le frecce bianche indicano le radici laterali e i peli delle radici. L’immagine è stata scattata 42 giorni dopo l’inoculazione (dpi); gli effetti sono stati visti in precedenza.(B) Il rodococco D188ΔpFID188 non è patogeno. Il ceppo è stato inoculato su N. bentamiana. L’immagine è stata scattata a 56 dpi.(C) Il rodococco isolato D188-5 è compromesso nella crescita in vitro. Rodococco isolati D188, D188-5, e D188ΔpFiD188 sono stati coltivati separatamente in media LB. La crescita, determinata utilizzando un lettore di piastre Tecan, è stata quantificata sulla base delle letture di OD600 per un periodo di 16 ore. È stato mostrato un esperimento rappresentativo di tre; tutti hanno avuto risultati simili. Le barre di errore indicano SEM.10.7554/eLife.30925.019Figure 6 cifre supplemento a 6 cifre 2 dati fonte 1.Valori di densità ottica di batteri cresciuti in coltura.(A) Immagini rappresentative di galli fogliacei su N. bentamiana. Le immagini sono state prese 56 giorni dopo l’inoculazione (dpi). Le frecce rosse indicano una bile a foglia.(B) Immagini rappresentative delle lunghezze delle radici di piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di rodococco o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. * indica una differenza significativa rispetto al trattamento con D188.10.7554/eLife.30925.021Figure 6-figure supplement 3-figure data 3-source 1.Lengths of N. benthamiana radicipiantina 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.
È stata dimostrata anche la transizione inversa. Abbiamo isolato una variante di D188, D188ΔpFiD188, che manca il plasmide virulenza. Quando inoculato sulle radici delle piantine di N. benthamiana, D188ΔpFiD188 ha causato cambiamenti nell’architettura dei sistemi radicali e non ha più causato malattie alle piante mature (Figura 6B; Figura 6-figure supplement 2). Si è dovuto generare un nuovo ceppo che ha perso il plasmide perché il D188-5 generato in precedenza è compromesso nella crescita in vitro(Figura 1; Figura 6C; Figura 6—supplemento 6-figure supplement 2; Desomer et al., 1988). L’analisi della sua sequenza genomica ha rivelato una significativa delezione di 25,4 kb dal cromosoma(file supplementare 1H). La maggior parte dei 25 geni interessati hanno funzioni annotate implicate nelle funzioni di Housekeeping. Il sequenziamento del D188ΔpFiD188 ha confermato che mancava solo il plasmide della virulenza. Anche questo isolato è cresciuto in modo simile al D188 e non aveva difetti di fitness misurabili(Figura 6C; Figura 6-figure supplement 2).
Solo tre loci su pFiD188 sono stati implicati nella virulenza. Non siamo stati in grado di ripetere i risultati che mostrano che la delezione mutante di att è attenuata nella virulenza (Figura 6-figure supplement 1; Crespi et al., 1992; Maes et al., 2001). Ma quando esprime costitutivamente attR, un omologo del regolatore trascrizionale di LysR necessario per l’espressione del gene di att, D188 ha causato insolite ghiandole fogliose su N. benthamiana(Figura 6-figure supplement 3). A differenza delle normali ghiandole che terminano la crescita primaria, quelle causate dal ceppo attR-overexpressing hanno riacquistato l’attività meristematica. Quando inoculate sulle radici, i sintomi erano più variabili, ma comunque simili a quelli causati dal D188. Gli effetti sono stati significativamente diversi rispetto a quelli osservati nelle piantine inoculate in mock-inoculate (p-valore<0,0001) o in quelle inoculate con D188 (p-valore<0,0001). Si prevede che il fas locus sia necessario per il rodococco per produrre e secernere una miscela di citochinine(Pertry et al., 2009). Circa 0,1 μM della citochinina sintetica 6-benzilamminopurina (BA) è stato equivalente ad un inoculo iniziale di soli ~ 2,5×103 unità di formazione di colonia (UFCU) di D188 (Figura 7A). Tuttavia, indipendentemente dalla quantità di BA nel mezzo, le citochinine applicate esogeneamente inibiscono solo l’allungamento delle radici e non provocano l’ispessimento degli steli o arrestano la crescita delle piante.
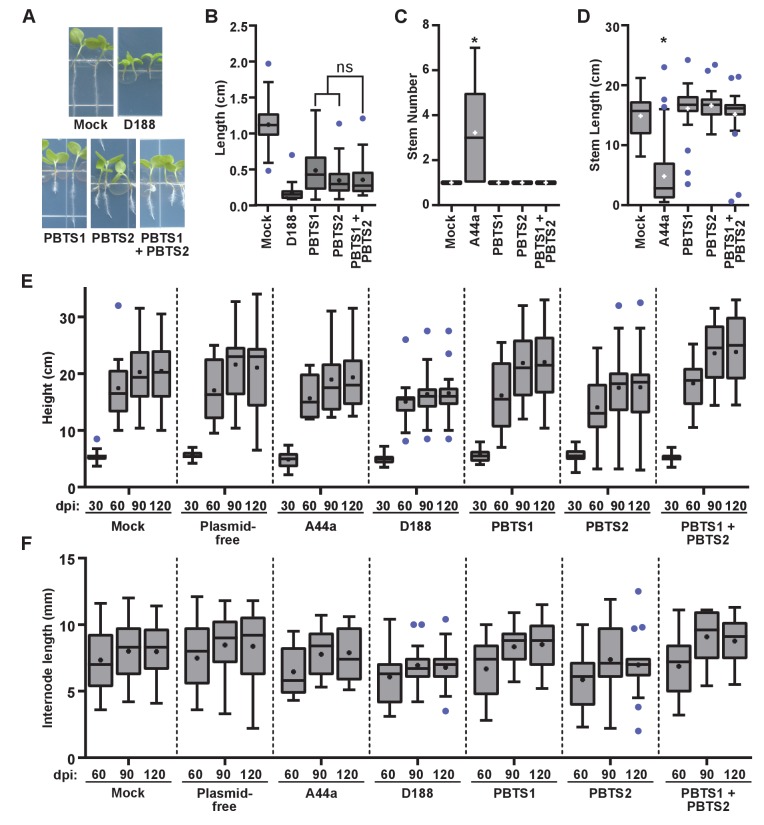
Figura 7-figure supplement 1-source data 5.Ilrodococco ha un effetto dose-dipendente sull’allungamento radicale delle piantine di N. benthamiana. 6. Gli isolati dirodococco PBTS1 e PBTS2 non causano sintomi di malattia nelle specie di piante testate. 7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di stelidipiselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.Lunghezze di radicidi piantine diN.benthamiana7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.(A) Quantificazione delle lunghezze delle radici delle piantine di piante coltivate in citochinina applicata esogeneamente (6-benizlaminopurina; BA). Le piantine di tre giorni di N. benthamiana sono state trasferite su supporti integrati con BA (0,01, 0,1, 1,0 e 10 µM) o dimetilsolfossido di zolfo (DMSO).(B) Quantificazione delle lunghezze delle radici delle piantine inoculate con dosi crescenti di rodococco . Le piantine di tre giorni di N. benthamiana sono state inoculate con isolati D188, PBTS2, o PBTS2 + pFiD188Δ att, con dosi che vanno da 2,5 × 102 a 1,0 × 1012 unità di formazione della colonia(ufc ). Il campione sfumato in grigio evidenzia l’inoculo di OD600 = 0,5 (1x = 2,5 × 1010 ufc) utilizzato in tutti gli altri saggi. Inocula al di sotto di questa diminuzione a intervalli di 100 volte. Inocula sopra aumentano a 2x, 4x, 10x e 20x. Tutti i trattamenti sono significativamente diversi dal mock se non diversamente indicato con ‘ns’.(C) Immagine rappresentativa dei cambiamenti morfologici nelle piantine. Piantine inoculate con Rhodococcus D188 o PBTS2 (5 × 1011 cfu; 10x quantità tipica) o acqua (mock) sono stati fotografati. Le frecce rosse indicano le foglie vere. Le frecce bianche indicano le radici laterali e la proliferazione dei peli delle radici.10.7554/eLife.30925.024Figure 7-source data 1.(A) Immagini rappresentative dei sintomi della malattia o effetti benefici sulle piantine di N. bentamiana. Piantine di tre giorni sono stati inoculati con rodococco o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(B) Quantificazione della lunghezza delle radici delle piantine di N. benthamiana. Tutti erano significativi rispetto a mock. Non ci sono state differenze significative (ns) tra i trattamenti con PBTS1, PBTS2, o la loro miscela.(C, D) Quantificazione del numero di steli (C)e della lunghezza dello stelo (D) di piselli inoculati con rodococco o mock. * indica una differenza significativa rispetto al trattamento mock.(E, F) Quantificazione dell’altezza (E)e della lunghezza degli internodi (F) del portainnesto di pistacchio UCB1 inoculato con rodococco o mock-inoculato. Nessun effetto del trattamento è stato significativamente diverso dal trattamento mock in tempo. Viene mostrato un esperimento rappresentativo di tre.10.7554/eLife.30925.026Figure 7-figure supplemento 7-figure 1-source dati 1.Lengths di N.radici di piantine dibentamiana7giorni dopo la crescita in BA o inoculazione con isolati D188 o PBTS2.10.7554/eLife.30925.027Figure 7-figure supplement 1-source data 2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.028Cifra 7-figure supplemento 7-figure 1-source data 3.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.029Cifra 7-figure supplemento 7-figure 1-source data 4.Altezze di pistacchio UCB1 210 giorni dopo l’inoculazione con isolati di rodococco.10.7554/eLife.30925.030Cifra 7 cifre supplemento 1-figure dati fonte 5.Internode lunghezze di pistacchio UCB1 210 giorni dopo inoculazioni con Rhodococcus isolati.

Figura 6-figure supplement 3-source data 1.Figura 6—figura supplemento 3—dati di fonte 1. Plasmide pFiD188 con fasR funzionale e fas è sufficiente per la transizione Rhodococcus isolati a fitopatogeni.lunghezze di N. radici dipiantina benthamiana7 giorni dopo l’inoculazione con Rhodococcusisolates+ / – pFiD188Δatt. Lunghezze di N. radici piantinabenthamiana7 giorni dopo l’inoculazione con Rodococcusolates + / –pFiD188Δatt. Ilrodococco isolato D188 con plasmide pFiD188Δ attè patogeno.lo sfratto del plasmide virulento ritorna ad isolare D188 in un batterio benefico.valori di densità ottica di batteri in coltura.valori di densità ottica di batteri in coltura.valori di densità ottica di batteri in coltura.Rodococco isolato D188 portare L5::attR è interessato in virulenza.lunghezze di N. radicipiantina benthamiana 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.lunghezzedi N.piantina benthamianaradici 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Le frecce rosse indicano le palle a foglia. Le immagini per GIC26, 04-516, e PBTS2 sono ripetute dalla Figura 1.(B) Immagini rappresentative delle lunghezze delle radici delle piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di Rhodococcus o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. benthamiana. In tutti i pannelli, -/+ indica l’assenza o la presenza di pFiD188Δ att. * indica una differenza significativa rispetto alle piante trattate con il corrispondente genotipo privo di plasmide.10.7554/eLife.30925.022Figure 6-source data 1.Lunghezze delle radici dellepiantinedi N. benthamiana 7 giorni dopo l’inoculazione con Rodococcusolates+/- pFiD188Δatt.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Le immagini sono state scattate 56 giorni dopo l’inoculazione (dpi). La freccia rossa indica la cistifellea a foglia.(B) Immagini rappresentative della lunghezza delle radici delle piantine. Tre giorni, le piantine di N. ben thamiana sono state inoculate con l’isolato indicato di rodococco o acqua (finto) e cresciute verticalmente per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. I dati per D188 +pFiD188Δ att sonogli stessi di quelli mostrati in Figura 6.(A) Il rodococco D188ΔpFID188 causa cambiamenti nell’architettura delle radici. Le frecce bianche indicano le radici laterali e i peli delle radici. L’immagine è stata scattata 42 giorni dopo l’inoculazione (dpi); gli effetti sono stati visti in precedenza.(B) Il rodococco D188ΔpFID188 non è patogeno. Il ceppo è stato inoculato su N. bentamiana. L’immagine è stata scattata a 56 dpi.(C) Il rodococco isolato D188-5 è compromesso nella crescita in vitro. Rodococco isolati D188, D188-5, e D188ΔpFiD188 sono stati coltivati separatamente in media LB. La crescita, determinata utilizzando un lettore di piastre Tecan, è stata quantificata sulla base delle letture di OD600 per un periodo di 16 ore. È stato mostrato un esperimento rappresentativo di tre; tutti hanno avuto risultati simili. Le barre di errore indicano SEM.10.7554/eLife.30925.019Figure 6 cifre supplemento a 6 cifre 2 dati fonte 1.Valori di densità ottica di batteri cresciuti in coltura.(A) Immagini rappresentative di galli fogliacei su N. bentamiana. Le immagini sono state prese 56 giorni dopo l’inoculazione (dpi). Le frecce rosse indicano una bile a foglia.(B) Immagini rappresentative delle lunghezze delle radici di piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di rodococco o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. * indica una differenza significativa rispetto al trattamento con D188.10.7554/eLife.30925.021Figure 6-figure supplement 3-figure data 3-source 1.Lengths of N. benthamiana radicipiantina 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.
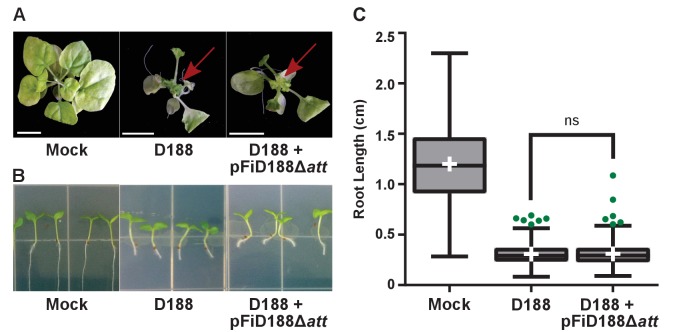
Figura 6-figure supplemento 1.Ilrodococco isolato D188 con plasmide pFiD188Δ attè patogeno.(A) Immagini rappresentative di galli fogliacei su N. bentamiana. Le immagini sono state scattate 56 giorni dopo l’inoculazione (dpi). La freccia rossa indica la cistifellea a foglia.(B) Immagini rappresentative della lunghezza delle radici delle piantine. Tre giorni, le piantine di N. ben thamiana sono state inoculate con l’isolato indicato di rodococco o acqua (finto) e cresciute verticalmente per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. I dati per D188 +pFiD188Δ att sonogli stessi di quelli mostrati in Figura 6.
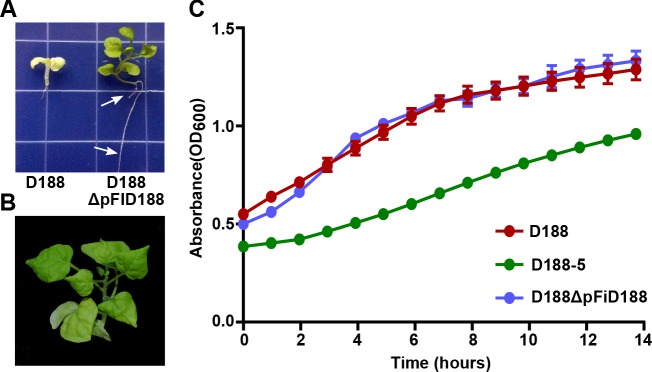
Figura 6-figure supplemento 2-figure dati fonte 1.Lo sfratto del plasmide della virulenza ritorna ad isolare D188 in un batterio benefico.valori di densità ottica dei batteri in coltura.valori di densità ottica dei batteri in coltura.(A) Il rodococco D188ΔpFID188 causa cambiamenti nell’architettura delle radici. Le frecce bianche indicano le radici laterali e i peli delle radici. L’immagine è stata scattata 42 giorni dopo l’inoculazione (dpi); gli effetti sono stati visti in precedenza.(B) Il rodococco D188ΔpFID188 non è patogeno. Il ceppo è stato inoculato su N. bentamiana. L’immagine è stata scattata a 56 dpi.(C) Il rodococco isolato D188-5 è compromesso nella crescita in vitro. Rodococco isolati D188, D188-5, e D188ΔpFiD188 sono stati coltivati separatamente in media LB. La crescita, determinata utilizzando un lettore di piastre Tecan, è stata quantificata sulla base delle letture di OD600 per un periodo di 16 ore. È stato mostrato un esperimento rappresentativo di tre; tutti hanno avuto risultati simili. Le barre di errore indicano SEM.10.7554/eLife.30925.019Figure 6 cifre supplemento a 6 cifre 2 dati fonte 1.Valori di densità ottica di batteri cresciuti in coltura.

Figura 6-figure supplement 3-source data 1.Figura 6—figura supplemento 3—dati di fonte 1.Rodococco isolato D188 che porta L5::attR è influenzato in virulenza.lunghezze di N. radicipiantina bentamiana 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.lunghezzedi N.piantina bentamiana 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.(A) Immagini rappresentative di galle a foglia su N. benthamiana. Le immagini sono state scattate 56 giorni dopo l’inoculazione (dpi). Le frecce rosse indicano una bile a foglia.(B) Immagini rappresentative delle lunghezze delle radici di piantine. Tre giorni, le piantine di N. benthamiana sono stati inoculati con l’isolato indicato di rodococco o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(C) Quantificazione delle lunghezze delle radici delle piantine di N. ben thamiana. * indica una differenza significativa rispetto al trattamento con D188.10.7554/eLife.30925.021Figure 6-figure supplement 3-figure data 3-source 1.Lengths of N. benthamiana radicipiantina 7 giorni dopo l’inoculazione con isolato D188 + / – L5::attR.

Figura 7-figure supplement 1-source data 5.Ilrodococco ha un effetto dose-dipendente sull’allungamento delle radici delle piantine di N. benthamiana. 6. Gli isolati dirodococco PBTS1 e PBTS2 non causano sintomi di malattia nelle specie di piante testate. 7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di stelidipiselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.Lunghezze delle radici dellepiantinedi N.benthamiana7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.(A) Quantificazione delle lunghezze delle radici delle piantine di piante coltivate in citochinina applicata esogeneamente (6-benizlaminopurina; BA). Le piantine di tre giorni di N. benthamiana sono state trasferite su supporti integrati con BA (0,01, 0,1, 1,0 e 10 µM) o dimetilsolfossido di zolfo (DMSO).(B) Quantificazione delle lunghezze delle radici delle piantine inoculate con dosi crescenti di rodococco . Le piantine di tre giorni di N. benthamiana sono state inoculate con isolati D188, PBTS2, o PBTS2 + pFiD188Δ att, con dosi che vanno da 2,5 × 102 a 1,0 × 1012 unità di formazione della colonia(ufc ). Il campione sfumato in grigio evidenzia l’inoculo di OD600 = 0,5 (1x = 2,5 × 1010 ufc) utilizzato in tutti gli altri saggi. Inocula al di sotto di questa diminuzione a intervalli di 100 volte. Inocula sopra aumentano a 2x, 4x, 10x e 20x. Tutti i trattamenti sono significativamente diversi dal mock se non diversamente indicato con ‘ns’.(C) Immagine rappresentativa dei cambiamenti morfologici nelle piantine. Piantine inoculate con Rhodococcus D188 o PBTS2 (5 × 1011 cfu; 10x quantità tipica) o acqua (mock) sono stati fotografati. Le frecce rosse indicano le foglie vere. Le frecce bianche indicano le radici laterali e la proliferazione dei peli delle radici.10.7554/eLife.30925.024Figure 7-source data 1.(A) Immagini rappresentative dei sintomi della malattia o effetti benefici sulle piantine di N. bentamiana. Piantine di tre giorni sono stati inoculati con rodococco o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(B) Quantificazione della lunghezza delle radici delle piantine di N. benthamiana. Tutti erano significativi rispetto a mock. Non ci sono state differenze significative (ns) tra i trattamenti con PBTS1, PBTS2, o la loro miscela.(C, D) Quantificazione del numero di steli (C)e della lunghezza dello stelo (D) di piselli inoculati con rodococco o mock. * indica una differenza significativa rispetto al trattamento mock.(E, F) Quantificazione dell’altezza (E)e della lunghezza degli internodi (F) del portainnesto di pistacchio UCB1 inoculato con rodococco o mock-inoculato. Nessun effetto del trattamento è stato significativamente diverso dal trattamento mock in tempo. Viene mostrato un esperimento rappresentativo di tre.10.7554/eLife.30925.026Figure 7-figure supplemento 7-figure 1-source dati 1.Lengths di N.radici di piantine dibentamiana7giorni dopo la crescita in BA o inoculazione con isolati D188 o PBTS2.10.7554/eLife.30925.027Figure 7-figure supplement 1-source data 2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.028Cifra 7-figure supplemento 7-figure 1-source data 3.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.029Cifra 7-figure supplemento 7-figure 1-source data 4.Altezze di pistacchio UCB1 210 giorni dopo l’inoculazione con isolati di rodococco.10.7554/eLife.30925.030Cifra 7 cifre supplemento 1-figure dati fonte 5.Internode lunghezze di pistacchio UCB1 210 giorni dopo inoculazioni con Rhodococcus isolati.

Figura 7-figure supplement 1-source data 5.Gli isolati dirodococco PBTS1 e PBTS2 non causano sintomi di malattia nelle specie di piante testate. 7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di stelidipiselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.Lunghezze di radicidi piantine diN.benthamiana7 giorni dopo la crescita in BA o l’inoculazione con gli isolati D188 o PBTS2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1 o PBTS2.Altezza del pistacchio UCB1 210 giorni dopo l’inoculazione congli isolatidi rodococco.Lunghezza internade del pistacchio UCB1 210 giorni dopo l’inoculazione con gli isolati di rodococco.(A) Immagini rappresentative dei sintomi della malattia o effetti benefici sulle piantine di N. bentamiana. Piantine di tre giorni sono stati inoculati con Rhodococcus o acqua (finto) e cresciuto in verticale per sette giorni sotto una luce costante.(B) Quantificazione della lunghezza delle radici delle piantine di N. benthamiana. Tutti erano significativi rispetto a mock. Non ci sono state differenze significative (ns) tra i trattamenti con PBTS1, PBTS2, o la loro miscela.(C, D) Quantificazione del numero di steli (C)e della lunghezza dello stelo (D) di piselli inoculati con rodococco o mock. * indica una differenza significativa rispetto al trattamento mock.(E, F) Quantificazione dell’altezza (E)e della lunghezza degli internodi (F) del portainnesto di pistacchio UCB1 inoculato con rodococco o mock-inoculato. Nessun effetto del trattamento è stato significativamente diverso dal trattamento mock in tempo. Viene mostrato un esperimento rappresentativo di tre.10.7554/eLife.30925.026Figure 7-figure supplemento 7-figure 1-source dati 1.Lengths di N.radici di piantine dibentamiana7giorni dopo la crescita in BA o inoculazione con isolati D188 o PBTS2.10.7554/eLife.30925.027Figure 7-figure supplement 1-source data 2.Numero di steli di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.028Cifra 7-figure supplemento 7-figure 1-source data 3.Lunghezza dei gambi di piselli 14 giorni dopo l’inoculazione con gli isolati D188, PBTS1, o PBTS2.10.7554/eLife.30925.029Cifra 7-figure supplemento 7-figure 1-source data 4.Altezze di pistacchio UCB1 210 giorni dopo l’inoculazione con isolati di rodococco.10.7554/eLife.30925.030Cifra 7 cifre supplemento 1-figure dati fonte 5.Internode lunghezze di pistacchio UCB1 210 giorni dopo inoculazioni con Rhodococcus isolati.
PBTS1 e PBTS2 non sono ceppi epidemici
I nostri risultati mostrano che PBTS1 e PBTS2 non sono patogeni su N. bentamiana(Figure 4 e 5). Abbiamo poi testato se l’alterazione della dose influenza l’esito dell’interazione tra N. benthamiana e PBTS2. Come livelli inoculo di PBTS2 sono stati aumentati, vi è stata una maggiore riduzione della lunghezza delle radici(Figura 7B), ma l’effetto non è mai stata la stessa robustezza e il grado di quello misurato in piantine infettate con D188. Inoltre, PBTS2 non ha causato l’ispessimento degli steli o l’arresto terminale nella crescita della pianta. A 28 giorni dopo l’inoculazione (dpi), le foglie delle piantine inoculate con i più alti livelli testati di PBTS2 si erano sviluppate allo stesso stadio di quelle delle piantine inoculate in modo simulato, mentre le piantine inoculate con D188 sono rimaste arrestate nella crescita(Figura 7C). Le radici delle piantine inoculate con PBTS2 hanno formato anche radici laterali. Come livelli di inoculo di PBTS2 sono stati diminuiti, vi è stata una minore riduzione della lunghezza delle radici, e alla più bassa dose testata, le radici erano significativamente più lunghe (1,161 cm; p-valore = 0,0043) rispetto a quelle delle piante trattate in modo simulato (1,007 cm; Figura 7B) . Un ceppo patogeno PBTS2 che trasportava pFiD188Δ att ha mostrato un effetto didose simile a quello visto per D188 (Figura 7B; i valori perano tutti>0,3584 per tutti i confronti all’interno della dose).
Per escludere la possibilità che questi risultati siano dovuti all’incompatibilità tra PBTS1 e PBTS2 e N. benthamiana, sono state testate altre specie di piante. Abbiamo usato il pisello, una specie indicatore per confermare il rodococco patogeno, e il pistacchio UCB-1, che secondo quanto riferito è l’ospite dell’epidemia. Entrambe le specie di piante non hanno mostrato sintomi di malattia, indipendentemente dal fatto che gli isolati siano stati testati singolarmente o in combinazione (Figura 7-figure supplement 1). Nove isolati supplementari di pistacchio che mancano di geni virulenti non sono riusciti a causare la malattia. Quattro isolati, 14-687, 14-688, 14-694 e 14-700, sono stati coltivati da piante di pistacchio asintomatiche, mentre cinque, SR18, AGD2M, AGD3B, AGD6D e AGD6H, sono stati coltivati da piante di pistacchio sintomatiche. Anche gli isolati patogeni D188 e A44a non sono riusciti a causare sintomi di malattia nel pistacchio UCB-1.
Un altro aspetto della diagnosi della sindrome del cespuglio di pistacchio è stato l’uso di vicA per confermare il rodococco patogeno(Stamler et al., 2015a, 2015b). I primer progettati per fasA e fasD hanno specificamente amplificato un prodotto di dimensioni previste dagli isolati patogeni D188, A44a e A25f e non sono riusciti ad amplificare un prodotto da uno qualsiasi dei ceppi benefici testati(Figura 8A; File supplementare 1I; Nikolaeva et al., 2012; Serdani et al., 2013). I primer per fasA, e fasD non sono riusciti a produrre un prodotto da A21d2 perché questo isolato porta un analogo del locus fas(Creason et al., 2014b). Il rilevamento molecolare di fasR mediante un saggio basato sull’amplificazione isotermica mediata da loop (LAMP) ha distinto in modo specifico tutti gli isolati patogeni testati dagli isolati benefici.
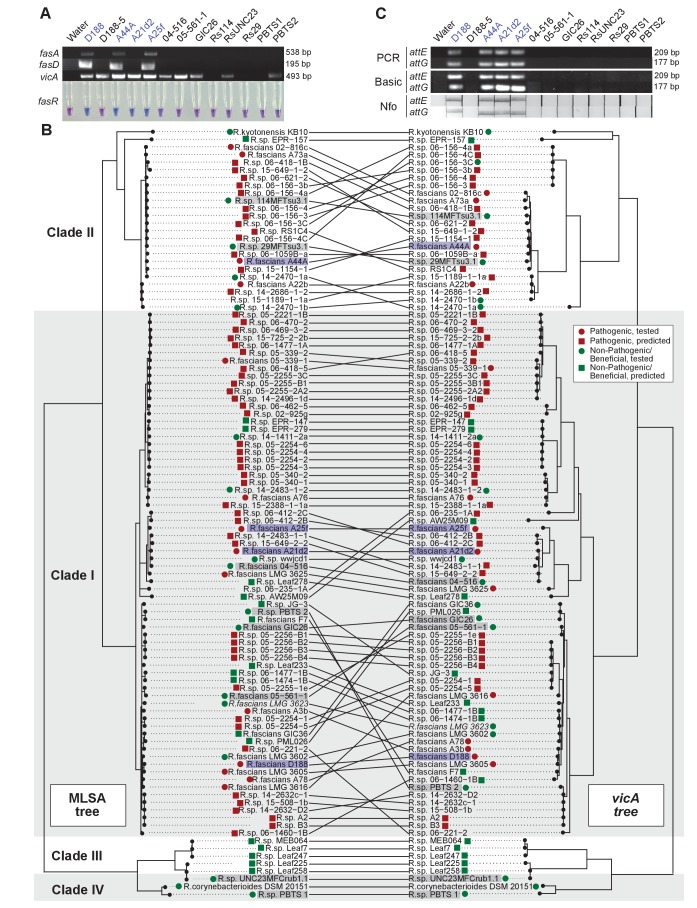
Figura 8.L’uso del gene vicA non discrimina i rodococcus patogeni.(A) (in alto) Utilizzando primer specifici del locus e DNA dagli isolati elencati, i frammenti di fasA, fasD e vicA sono stati amplificati con PCR. Isolati patogeni sono etichettati in blu; virulenza-gene-nolente isolati sono etichettati in nero. I prodotti sono stati risolti su un gel di agarosio all’1% TAE. Le dimensioni degli ampliconi sono elencate a destra delle immagini del gel. (In basso) LAMP test di rilevamento del fasR dagli stessi campioni di DNA. Un risultato positivo è visualizzato da un colore blu. I risultati negativi sono viola chiaro.(B) Congruenza tra le specie (a sinistra) e gli alberi vicA (a destra). Le clave diverse da I-IV non sono mostrate. Gli isolati evidenziati sono gli stessi di quelli in A, gli isolati patogeni sono evidenziati in blu, gli isolati di virulenza-nerogeni sono evidenziati in grigio.(C) Gli endpoint standard PCR, RPA Basic e RPA nfo sono stati utilizzati per rilevare attE o attG da DNA estratto da isolati di rodococco. Gli isolati patogeni sono etichettati in blu. Per PCR e RPA basic, le dimensioni del prodotto sono elencate a destra della figura. Per RPA nfo, la presenza della banda del test è indicativa di una reazione positiva; le bande di controllo per tutte le strisce sono state confermate (non mostrate).
Il rilevamento di vicA non ha seguito uno schema coerente con il fenotipo di patogenicità(Figura 8A). Ha un alto tasso di falsi positivi e ha rilevato diversi, ma non tutti, gli isolati benefici. I nostri ripetuti tentativi di amplificare vicA da PBTS1 non hanno avuto successo; ci sono quattro e sei disallineamenti tra i due primer utilizzati per la PCR e la sequenza vicA da PBTS1. Si prevede che gli omologhi di vicA siano presenti in quasi tutti i membri degli Actinobatteri, compresi tutti i 407 isolati di rodococco per i quali sono disponibili sequenze genomiche. Le topologie del genere Rhodococcus e degli alberi vicA sono in gran parte congruenti, indicando che questo locus è per lo più ereditato verticalmente, ma con qualche evidenza di ricombinazione (Figura 8B).
Per rispondere alla necessità di strumenti molecolari in loco per distinguere i genotipi patogeni da quelli benefici, abbiamo utilizzato un nuovo metodo di rilevamento molecolare che è rapido, robusto e sensibile(Piepenburg et al., 2006). Abbiamo puntato su attE e attG perché sono le più uniche relative a tutte le sequenze presenti nei database e sono conservate tra gli isolati patogeni che abbiamo esaminato (Creasonet al., 2014b). L’uso dei primer per attE e attG nella PCR standard e nell’amplificazione della polimerasi ricombinasi (RPA) ha permesso di amplificare con successo prodotti di base di dimensioni attese dal DNA di ceppi patogeni, tra cui A21d2 e A25f(Figura 8C). Non sono stati rilevati prodotti quando i DNA di ceppi benefici sono stati utilizzati come modelli. Una sonda oligonucleotidica aggiuntiva che anneals all’interno del frammento amplificato è stata progettata per RPA nfo, e quando accoppiata con primer di amplificazione modificati, questa sonda è riuscita a rilevare un prodotto attraverso il flusso laterale. Questo metodo è stato specifico e discriminato tra Rhodococcus patogeno e benefico. Inoltre, RPA nfo può essere completato in assenza di apparecchiature specializzate, e può dare risultati in soli 30 min.

Figura 8.L’uso del gene vicA non discrimina il rodococco patogeno.(A) (in alto) Utilizzando primer specifici del locus e il DNA dagli isolati elencati, frammenti di fasA, fasD e vicA sono stati amplificati PCR. Isolati patogeni sono etichettati in blu; virulenza-gene-nolente isolati sono etichettati in nero. I prodotti sono stati risolti su un gel di agarosio all’1% TAE. Le dimensioni degli ampliconi sono elencate a destra delle immagini del gel. (In basso) LAMP test di rilevamento del fasR dagli stessi campioni di DNA. Un risultato positivo è visualizzato da un colore blu. I risultati negativi sono viola chiaro.(B) Congruenza tra le specie (a sinistra) e gli alberi vicA (a destra). Le clave diverse da I-IV non sono mostrate. Gli isolati evidenziati sono gli stessi di quelli in A, gli isolati patogeni sono evidenziati in blu, gli isolati di virulenza-nerogeni sono evidenziati in grigio.(C) Gli endpoint standard PCR, RPA Basic e RPA nfo sono stati utilizzati per rilevare attE o attG da DNA estratto da isolati di rodococco. Gli isolati patogeni sono etichettati in blu. Per PCR e RPA basic, le dimensioni del prodotto sono elencate a destra della figura. Per RPA nfo, la presenza della banda del test è indicativa di una reazione positiva; le bande di controllo per tutte le strisce sono state confermate (non mostrate).
Discussione
Gli studi epidemiologici basati sul genoma intero hanno rivelato modelli locali, globali e storici per la trasmissione di agenti patogeni umani e hanno fornito informazioni sull’assistenza sanitaria (Comaset al., 2013; Croucher et al., 2011; Harris et al.,2010, 2013; Mutreja et al., 2011; Parkhill e Wren, 2011; Walker et al., 2013 ). Il nostro è un caso di studio per l’utilizzo dell’epidemiologia genomica per scoprire e spiegare i modelli di trasmissione dei fitopatogeni nei sistemi agricoli. L’indagine sulla distribuzione dei plasmidi ha evidenziato il ruolo significativo della HGT nel plasmare la struttura della popolazione di batteri patogeni e ha rivelato sfide nella modellazione della loro trasmissione. La nostra analisi dei SNP cromosomici ha suggerito che i vivai sperimentano introduzioni multiple e indipendenti di rodococco patogeno , esemplificate da isolati ‘a’ osservati nei vivai N15 e N8 (Figura 2). Il legame tra gli isolati raccolti nel tempo (‘b’) è indicativo di una popolazione serbatoio che ha un genotipo patogeno. I legami epidemiologici (‘c1’ e ‘c2’) di isolati provenienti da diversi vivai supportano la possibilità di focolai di origine puntuale e suggeriscono che le fonti hanno popolazioni serbatoio.
Tuttavia, la distribuzione dei plasmidi indica anche che possono verificarsi processi alternativi (Figure 3e 9). In primo luogo, due diversi tipi di plasmidi sono associati a ‘c2’. Questo non è previsto da un’epidemia ed è più coerente con i diversi membri di un lignaggio che acquisiscono i plasmidi separatamente. In secondo luogo, le diverse varianti di plasmidi sono trasportate da isolati che sono definiti da genotipi associati con ‘a1’, ‘b’ e ‘c1’ (Figura 2). Questi non sono modelli previsti e probabilmente riflettono acquisizioni separate di plasmidi da parte di diversi membri di un lignaggio o l’evoluzione rapida e indipendente dei plasmidi. In terzo luogo, i lignaggi geneticamente distinti di Rhodococcus al vivaio N15 portano la stessa variante di plasmidi. Questo si spiega meglio con lignaggi multipli che acquisiscono plasmidi da una popolazione di donatori comuni. I vivai spesso producono una grande varietà di piante perenni e di piante clonate che vengono spesso manipolate e gestite intensamente in diversi contesti di produzione, e che si trovano spesso in regioni che producono molti prodotti agricoli diversi. Questi sono luoghi ideali per l’interazione di diversi genotipi di rodococco e per il trasferimento, lo scambio e l’evoluzione dei plasmidi.
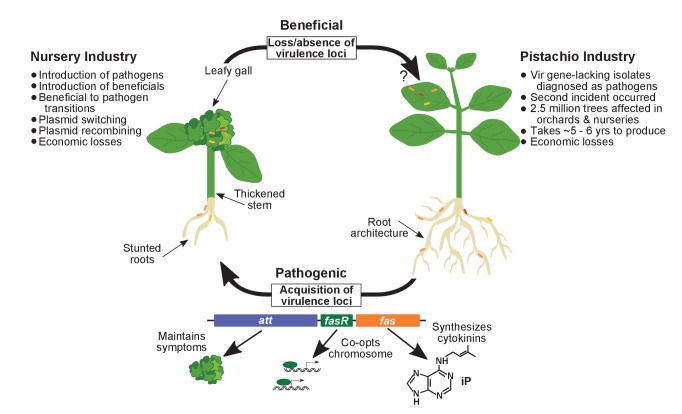
Figura 9.Modello della transizione evolutiva del rodococco e degli effetti sui settori agricoli.La presenza o l’assenza del plasmide virulenza in otto specie di Rhodococcusdeterminase il batterio è benefico (a destra), e promuove la crescita nell’architettura delle radici, o patogena (a sinistra), e causa la comparsa di ghiandole fogliari e inibisce la crescita primaria. Il plasmide della virulenza porta tre loci identificati come necessari per la patogenicità e le loro funzioni previste sono descritte. Il trasferimento genico orizzontale e le transizioni evolutive influenzano il rodococco e hanno un impatto sull’industria del vivaismo (come indicato a sinistra della figura). Gli isolati di Rhodococcus che perdono il gene della virulenza sono stati diagnosticati come ceppi epidemici e la probabile diagnosi errata potrebbe aver avuto un impatto negativo sull’industria del pistacchio (come indicato a destra della figura).
Abbiamo anche dimostrato che le piante nei sistemi agricoli sono ospiti di isolati di rodococco che sono probabilmente simbiotici associativi benefici (file supplementare1A; Vacheron et al., 2013). I peli delle radici sono estensioni che aumentano la superficie delle radici, formando un’estesa interfaccia tra la pianta e il suolo. Gli isolati di rodococco, che perdono la virulenza, hanno causato un aumento significativo del numero e della lunghezza dei peli delle radici, che può consentire alle piante di essere più efficienti nell’assorbimento dell’acqua e dei nutrienti disciolti(Figura 5). Questo fenotipo potenzialmente benefico per la crescita è coerente con la scoperta di una serie di rapporti che identificano il rodococco all’interno dei compartimenti endofitici e della rizosfera delle piante, e con i suggerimenti che i batteri sono arricchiti dalle piante a causa dei loro tratti benefici (Baiet al., 2015; Bodenhausen et al., 2013; Bulgarelli et al., 2012;Francis et al., 2016; Hong et al., 2015, 2016; Lundberg et al., 2012; Qin et al., 2009, 2011; Salam et al., 2017). I simbiotici associativi sono geneticamente diversi e rappresentano 14 specie diverse, circoscritte da quattro cladi gemelli(Figura 1; File supplementare 1B). Il meccanismo con cui gli isolati di rodococco causano cambiamenti nella crescita delle piante è sconosciuto, in quanto gli sforzi di estrazione del genoma suggeriscono che questi tratti sono potenzialmente nuovi (Figura 5-figure supplement 2).
L’acquisizione di un plasmide della virulenza da parte di isolati che rappresentano otto specie di Clades I e II è sufficiente a guidare una transizione evolutiva(Figure 1 e 6). La perdita del plasmide ha riportato il rodococco ad essere benefico, coerentemente con l’ipotesi che i geni della virulenza funzionino indipendentemente dal background genomico(Figura 6-figure supplement 2). Inoltre, non siamo stati in grado di identificare alcun gene cromosomico che sia arricchito in isolati patogeni, rispetto agli isolati non patogeni, che potrebbero essere potenziali geni candidati alla virulenza. Transizioni evolutive, come il passaggio da un lignaggio libero e non patogeno ad un lignaggio patogeno, sono state rilevate frequentemente (per esempio, da Bruto et al., 2017). La transizione descritta per il rodococco è senza soluzione di continuità e la transizione da mutualista a patogeno è stata descritta solo raramente (Sachs et al.,2011). È possibile che l’attenzione alla caratterizzazione dei risultati binari nelle simbiosi abbia oscurato la vera fluidità delle simbiosi. Transizioni simili possono essersi verificate in Agrobacterium/Rhizobium, un gruppo di batteri che esprimono tratti plasmido-mediatati di importanza per l’agricoltura vegetale(de Lajudie et al., 1999; Glaeser et al., 2016; Hao et al., 2012; Lacroix e Citovsky, 2016; Wang et al., 2006).
Le transizioni e la rapida generazione di nuovi lignaggi di agenti patogeni potrebbero verificarsi nei sistemi agricoli, dove le piante ospitano spesso isolati multipli di rodococco di diversi genotipi. L’isolato patogeno 05-2254-6, che è associato con ‘a2’, è più simile all’isolato di virulenza-gene-nolente, 14-1411-2a, anch’esso raccolto da N8. I 220 SNPs a coppie che differiscono tra questi due isolati hanno superato la soglia utilizzata per definire i genotipi, ma i due isolati sono comunque strettamente correlati(Figure 1 e 2; File supplementare 1C). Questo non è un caso unico, in quanto diversi genotipi patogeni sono correlati a genotipi di virulenza-genio-nolente, come indicato dalla loro commistione nella filogenesi e dalla loro connettività nella rete. I modelli che coinvolgono genotipi distinti o tipi di plasmidi, come ‘a1’ e ‘c2’, rispettivamente, sono anche coerenti con le transizioni evolutive (Figure 2 e 3). Un esempio simile ad ‘a1’ è il genotipo di D188 e LMG3605, entrambi con diverse varianti di plasmidi.
La limitata diversità genetica dei plasmidi della virulenza all’interno del nostro dataset è stata inaspettata e suggerisce che il loro antenato comune si è evoluto di recente (Figura 3-figure supplement 1). Questo è in netto contrasto con i plasmidi della virulenza delle specie di batteri Agrobacterium, dove i plasmidi Ti e Ri formano diversi tipi che possono essere facilmente distinti sulla base della filogenesi di un singolo gene della virulenza conservato (Fuller et al.,2017). È inoltre degno di nota il fatto che il plasmide della virulenza, che porta solo tre loci di virulenza, è sufficiente per la patogenicità di otto specie geneticamente diverse di rodococco (Figure1 e 9; Creason et al., 2014b; Francis et al., 2012; Letek et al., 2010). FasR è un regolatore trascrizionale previsto, ed è probabilmente la chiave per la riprogrammazione del genoma ai batteri di transizione verso gli agenti patogeni. I ruoli degli altri due loci non sono ancora chiari. I risultati qui presentati suggeriscono che att contribuisce al mantenimento dei sintomi della malattia, ma il meccanismo è sconosciuto (Figura 6-figuresupplement 3). Si prevede che la miscela di citochinine prodotta da Fas sia secreta nelle piante e necessaria per causare i sintomi della malattia, ma i dati esistenti non sono coerenti con questa ipotesi (Pertry et al., 2009). Quantità eccessivamente basse di citochinine sono rilevate nei batteri coltivati in coltura e le piante hanno una varietà di meccanismi di tamponamento della citochinina(Creason et al., 2014b; Kieber e Schaller, 2014; Pertry et al., 2009, 2010). Una minuscola quantità di inoculo batterico iniziale è sufficiente a provocare i sintomi della malattia, e le applicazioni esogene delle citochinine non riescono a fenocopiare gli effetti del rodococco patogeno (Figura 7). Infine, il modello di miscela di citochinine è messo in discussione dalla rivelazione che l’idoneità di D188-5, un isolato chiave utilizzato per sviluppare il modello, è gravemente compromessa da una delezione di 25kb(Figura 6-figure supplement 2; Supplementary file 1H; Desomer et al., 1988; Maes et al., 2001; Pertry et al., 2009, 2010; Stes et al., 2011; Temmerman et al., 2001 ).
Questo studio evidenzia l’importanza di comprendere le caratteristiche genetiche e fenotipiche di un organismo e le conseguenze di trarre prematuramente conclusioni da dati incompleti. Non ci sono prove che suggeriscano che l’HGT o le transizioni evolutive confondano la diagnosi del pistacchio, e non siamo riusciti a rilevare il rodococco patogeno da tessuti sintomatici del pistacchio. Non siamo riusciti a ripetere i risultati che mostrano che PBTS1 e PBTS2, o altri isolati coltivati da pistacchio, causano sintomi di malattia sulle piante (Figura 4; Figura 7-figure supplemento 1). Non abbiamo potuto amplificare i geni della virulenza da PBTS1 o PBTS2, ma quando un plasmide della virulenza è stato introdotto in PBTS2, è stato sufficiente per passare PBTS2 ad un agente patogeno di una specie vegetale che è dimostrabilmente suscettibile al rodococco (Figure 6e 7B). Se il pistacchio è anche un ospite per il Rhodococcus patogeno è irrisolto(Figura 7-figure supplement 1). Tuttavia, riconosciamo l’insormontabile sfida nel mostrare che non c’è alcuna possibilità che il rodococco patogeno provochi la sindrome della cima cespugliosa del pistacchio.
I risultati di questo lavoro ci hanno spinto ad esaminare retrospettivamente gli studi precedenti (Figura 9; [Stamler et al., 2015a, 2015b]). C’è stata una ricerca mirata per il rodococco, la cui giustificazione è infondata perché i sintomi sul pistacchio sono diversi da quelli prodotti dal rodococco patogeno in uno qualsiasi degli oltre 100 ospiti conosciuti (Putname Miller, 2007). I batteri sono stati inspiegabilmente coltivati da foglie asintomatiche distali a tessuti staminali sintomatici. Lo studio chiave non ha incluso i ceppi di controllo o riprodursi cistifellea e fallimento dell’innesto, i sintomi di malattia più significativi osservati in ambienti di campo. Un alto inoculo di rodococco è stato utilizzato e alte dosi di batteri anche benefici possono avere costi(Figura 7). Ad esempio, alcune malattie umane sono causate da disbiosi in cui uno squilibrio della microbiozia intestinale causa la malattia (Bloom et al.,2011). Inoltre, gli ospiti spesso impiegano meccanismi per regolare o garantire interazioni non persistenti con batteri benefici (Gutjahre Parniske, 2013; Magori et al., 2009; Nyholm e McFall-Ngai, 2004; Reid et al., 2011; Wopereis et al., 2000).
I risultati basati sulla rilevazione molecolare sono stati altrettanto tenui. L’uso del locus vicA è stato fuorviante e ha portato a conclusioni sulla patogenicità del rodococco(Figura 8). L’amplificazione riportata di frammenti di fasA e fasD (numeri di adesione GenBank KP274062 e KP274064) da PBTS1 e fasD (KP274067) da PBTS2 non può essere riconciliata con l’assenza dei geni dalle sequenze genomiche(Stamler et al., 2015b, 2016). Abbiamo utilizzato una tecnologia diversa per ri-sequenziare il DNA preparato in modo indipendente da PBTS2 (file supplementare1F). L’assemblaggio è in linea con la sequenza pubblicamente disponibile e manca dei geni del plasmide della virulenza e della virulenza(Stamler et al., 2016). La possibilità che i plasmidi della virulenza siano instabili in popolazioni cresciute al di fuori degli ambienti vegetali non è supportata dai dati. Il DNA estratto da batteri coltivati in coltura è stato utilizzato come modello sia nella PCR che nel sequenziamento del genoma intero (Stamleret al., 2015b, 2016) . Abbiamo sequenziato con successo plasmidi da 64 isolati patogeni coltivati in coltura, che erano stati sottoposti a trasferimenti multipli, e abbiamo rilevato ripetutamente e con successo isolati patogeni coltivati da tessuti sintomatici(Creason et al., 2014b).
Alcuni dei risultati della rilevazione molecolare erano chiaramente artefatti. I due frammenti riportati per corrispondere a fasD sono identici in sequenza, ciascuno composto da due brevi frammenti di 147 e 194 nucleotidi lunghi che sono uniti artificialmente da 280 ‘Ns’. Nel nostro dataset, A21d2 è l’unico isolato patogeno in sequenza che manca di fasA, ma manca anche un omologo di fasD. Non ci si aspetta quindi che PBTS2 abbia solo fasD ma non fasA(Stamler et al., 2015b). L’artefatto più eclatante è stata l’amplificazione riportata di vicA da PBTS1 (numero di adesione GenBank KP274063), che non siamo riusciti a riprodurre(Figura 8). Quando la sequenza del frammento di amplificazione riportato è stata utilizzata come query per cercare direttamente la sequenza del genoma PBTS1, non siamo riusciti a identificare una regione omologa. Quando è stato usato nelle ricerche contro database disponibili al pubblico, i risultati migliori, oltre al KP274063, sono stati NM-J PBTS (KR153287; 100% identità), DMS3-9 (KJ677035; 97% identità), D188 genoma completo (CP015235; 96% identità), e PBTS2 genoma completo (CP015220; 96% identità).
Un altro studio ha ipotizzato che i plasmidi della virulenza sono instabili nelle popolazioni che persistono all’interno degli ambienti vegetali (Nikolaevaet al., 2009). Le sequenze del genoma hanno invece mostrato che gli isolati di rodococco provengono da lignaggi geneticamente distinti. Questa osservazione sottolinea ulteriormente la necessità di utilizzare adeguati strumenti di diagnostica molecolare che discriminano i batteri patogeni da quelli non patogeni. LAMP per rilevare fasR o RPA nfo per rilevare attE o attG sono entrambi metodi sensibili che hanno l’ulteriore vantaggio di essere rapidi e meno dipendenti da apparecchiature specializzate(Figura 8; Serdani et al., 2013).
I dati sono coerenti con la possibilità che le conclusioni precedenti si basino su una diagnosi errata della sindrome del cespuglio di pistacchio(Stamler et al., 2015a, 2015b). In tal caso, il rodococco è stato implicato come patogeno, indipendentemente dal genotipo e senza tener conto della diversità genetica e fenotipica del genere. Questa mentalità si scontra con tutti i batteri come agenti causali della malattia e dimostra l’importanza dei batteri nel promuovere la salute dei loro ospiti(Figura 9). Questa potenziale diagnosi errata della sindrome del cespuglio di pistacchio potrebbe essere responsabile di effetti catastrofici. Si stima che siano stati distrutti circa 2,5 milioni di alberi, con conseguente enorme perdita economica. Gli sforzi per identificare la vera causa sono stati decelerati. Le accuse riguardanti la fonte del rodococco hanno introdotto il conflitto in un’industria alle prese con un problema considerevole e sconosciuto. Considerando i dati qui descritti, le conclusioni precedenti dovrebbero, come minimo, essere temperate e le raccomandazioni per la gestione degli impianti in cui sono stati rilevati i batteri del rodococco, dovrebbero essere riesaminate. Idealmente, questi risultati rinnoveranno gli sforzi per identificare la vera natura della sindrome che affligge i portainnesti di pistacchio UCB-1.
Gli actinobatteri sono membri di spicco delle comunità associate alle piante, ma i meccanismi e l’evoluzione dei tratti che sono importanti per i batteri Gram-positivi per risiedere nelle comunità microbiche e influenzare la salute delle piante non sono ben compresi (Figura 9). I membri del genere Rhodococcus sono modelli eccellenti per colmare questa lacuna di conoscenza. Una ricchezza di risorse associate, come un’ampia e diversificata raccolta di genotipi, sequenze genomiche associate e strumenti genetici sono stati sviluppati per i Rodococcus. I membri di questo taxon possono anche interagire con le specie di piante geneticamente tracciabili e trarne beneficio. Infine, i membri del Rhodococcus sono organismi modello per la caratterizzazione delle transizioni evolutive tra stati simbiotici alternativi.

Figura 9.Modello della transizione evolutiva in Rhodococcus ed effetti sui settori agricoli.La presenza o l’assenza del plasmide della virulenza in otto specie di Rhodococcusdeterminase il batterio è benefico (a destra), e promuove la crescita nell’architettura delle radici, o patogena (a sinistra), e causa la comparsa di ghiandole fogliari e inibisce la crescita primaria. Il plasmide della virulenza porta tre loci identificati come necessari per la patogenicità e le loro funzioni previste sono descritte. Il trasferimento genico orizzontale e le transizioni evolutive influenzano il rodococco e hanno un impatto sull’industria del vivaismo (come indicato a sinistra della figura). Gli isolati di Rhodococcus che perdono il gene della virulenza sono stati diagnosticati come ceppi epidemici e la probabile diagnosi errata potrebbe aver avuto un impatto negativo sull’industria del pistacchio (come indicato a destra della figura).
Materiali e metodi
Isolati batterici e condizioni di crescita
Gli isolati dirodococco utilizzati in questo studio sono elencati nei file supplementari 1A e F. I batteri sono stati mantenuti su terreno solido LB a 28oC o sono cresciuti durante la notte in terreno LB a 28oC con scuotimento. Prima della coniugazione, i batteri Rhodococcus resistenti alla streptomicina sono stati selezionati per ciascuno dei genotipi riceventi. Le coniugazioni sono state fatte come descritto in precedenza(Desomer et al., 1988). I ceppi donatori e riceventi sono stati coltivati in tampone di estratto di lievito (YEB) e agitati a 28°C. Ogni genotipo è stato miscelato ad un rapporto di 1:1 e filtrato attraverso un filtro alla nitrocellulosa (dimensione dei pori, 0,45 μm; diametro, 25 mm; MilliporeSigma, Temecula, CA, USA). I filtri sono stati incubati su piastre di agar YEB per 24-28 ore a 28°C. Le cellule sono state lavate dal filtro con 5 ml di un tampone contenente 10 mM Tris-HCl pH 7,5 e 10 mM MgSO4 e poi diluito e placcato su terreno YEB contenente l’antibiotico appropriato. Escherichia coli è stato coltivato su terreno LB a 37oC. Quando opportuno, il terreno è stato modificato con 50 µg/ml di kanamicina antibiotica per Rhodococcus o E. coli. Per le curve di crescita, le colture di Rhodococcus cresciuto durante la notte sono state pellettate, lavate e risospese a OD600 = 0,5 in un volume finale di 200 µl di terreno LB in piastre a fondo piatto a 96 pozzetti. I batteri sono stati coltivati per un periodo di 14 ore a 28oC con agitazione in un lettore di piastre Tecan Spark 10 m. Le misurazioni della densità ottica (OD600) sono state effettuate ogni ora. Tre repliche tecniche sono state incluse per ogni isolato, e l’esperimento è stato ripetuto almeno tre volte con risultati simili.
Sequenziamento del genoma, assemblaggio e annotazione
Il kit di preparazione genomica Wizard (Promega, Fitchburg, WI, USA) è stato utilizzato per estrarre il DNA genomico dal rodococco. Sono state seguite le indicazioni per i batteri Gram-positivi. Il DNA è stato quantificato con uno spettrofotometro Nanodrop e regolato a 50 ng/µl. DNA genomico totale è stato utilizzato per preparare le biblioteche Nextera XT, e le librerie multiplexate risultanti sono stati sequenziati su un Illumina HiSeq 3000 per generare 250mer accoppiato fine sequenziamento legge (Center for Genome Research and Biocomputing [CGRB], Oregon State University). Le letture sono state elaborate come segue. FastQC è stato utilizzato per valutare la qualità delle letture di sequenziamento(Andrews, 2014). BBduk v.35.82, con i parametri ‘ktrim = r k = 23 mink = 9 hdist = 1 minlength = 100 tpe tbo’, è stato utilizzato per rimuovere le sequenze di adattamento (Bushnell, 2014). SPAdes v. 3.1.1.1, con i parametri ‘–careful -k 21,33,55,55,77,99’ è stato utilizzato per correggere gli errori e per assemblare de novo le letture in contigs (Bankevich etal., 2012). Blobtools è stato utilizzato per valutare gli assemblaggi e guidare l’eliminazione dei contig probabilmente derivati da batteri contaminanti (sulla base del contenuto di GC combinato, della copertura e dell’annotazione dei contig) (Kumar etal., 2013). Prokka è stato utilizzato per annotare le sequenze genomiche assemblate(Seemann, 2014).
Analisi filogenetiche
Le sequenze per l’albero di analisi delle sequenze multiloco di massima probabilità (MLSA) sono state acquisite utilizzando lo strumento autoMLSA(Davis Ii et al., 2016). Le sequenze per i geni, ftsY (ABG98302.1), infH (ABG98417.1), rpoB (ABG93773.1), rsmA (ABG97450.1), secY (ABG97930.1), tsaD (ABG97962.1) e ychF (ABG97656.1) dalla sequenza genomica del Rhodococcus jostii RHA1 sono stati tradotti e utilizzati come query in TBLASTN v. 2.2.31 ricerche contro le sequenze genomiche assemblate e il database NCBI nt, mascherato a Rhodococcus(Adékambi et al., 2011; accesso 12/2016). Di quelli provenienti da NCBI nt, otto ceppi privi di tutte e sette le sequenze e/o duplicati dei risultati sono stati rimossi dall’analisi. Le sequenze sono state allineate utilizzando MAFFT v. 6.864b con impostazioni predefinite(Katoh e Standley, 2013). Uno script accessorio RAxML è stato utilizzato per determinare il modello proteico più adatto per ogni allineamento della sequenza proteica. Gli alberi filogenetici (100 ricerche ML, repliche di bootstrap con criterio ‘autoMRE’) sono stati generati utilizzando RAxML v. 8.1.17 con un allineamento partizionato delle sequenze proteiche MLSA (Stamatakis, 2014).
Un’analisi simile utilizzando R. fascians D188 vicA (AMY55488.1) come query è stata utilizzata per acquisire e assemblare una filogenesi di 162 sequenze di gene malato sintasi dal database NCBI nr mascherato a Rhodococcus (accesso 04/2017). È stata generata una trama cophylo degli alberi MLSA e vicA utilizzando i fitoli del pacchetto R(Revell, 2012).
Per i geni presenti nel 95% dei plasmidi della virulenza, le sequenze sono state concatenate utilizzando la funzione SuperMatrix del pacchetto R EvobiR prima della costruzione delle filogenie(Blackmon e Adams, 2015).
Sono mostrati solo i valori di bootstrap superiori a 50.
Analisi del genoma e strumenti bioinformatici
Bowtie2 v. 2.2.3, con l’opzione ‘–local’, è stata utilizzata per allineare le letture alle sequenze dei riferimenti cromosomici di D188 (CP015235.1) o A44a (GCF_000760735.1), in base all’assegnazione del clade dei corrispondenti isolati (Langmeadet al., 2009). Gli allineamenti sono stati convertiti in formato bam utilizzando samtools v. 0.1.18 e sono stati aggiunti gruppi di lettura utilizzando Picard tools v. 2.0.1(Li et al., 2009; PicardTools, 2015). GATK v. 3.7 HaplotypeCaller e le opzioni ‘-ERC GVCF -ploidy 1’ sono state utilizzate per richiamare varianti per ogni isolato, e i dati sono stati poi combinati utilizzando GenotypeGVCFs (McKennaet al., 2010). Le varianti sono state filtrate utilizzando il pacchetto R vcfR con filtraggio in profondità utilizzando probabilità quantilitiche di 0,25 e 0,75 come cutoff e un minimo di quattro letture, oltre a un cutoff di dati mancanti del 20%(Knaus e Grünwald, 2017). Le varianti sono state convertite in un allineamento fasta utilizzando bcftools v. 1.3-14-ge0890a1 vcf-to-tab e lo script perl vcftab-to-fasta (Liet al., 2009; Chen , 2012). I genotipi sono stati chiamati sulla base di una soglia di 25 SNPs, e le distanze bitwise, utilizzando il pacchetto R poppr, sono stati utilizzati per assemblare le reti di estensione minima(Kamvar et al., 2014).
L’identità nucleotidica media di coppia (ANI) tra gli isolati di rodococco è stata calcolata utilizzando autoANI(Davis Ii et al., 2016).
Get_homologues v. 20170418 con clustering MCL è stato utilizzato per raggruppare i geni di 206 genomi di rodococco in gruppi ortologi(Contreras-Moreira e Vinuesa, 2013). Lo script parse_pangenome_matrix.pl di get_homologues è stato utilizzato per identificare i geni arricchiti (con una soglia del 95%) nei genomi di rodococco nei quattro cladi associati alle piante.
Per identificare i loci di patogenicità negli isolati sequenziati, fasR, fas e att sono stati utilizzati come query nelle ricerche TBLASTN contro gli assemblaggi genomici. CONTIGuator è stato utilizzato per mappare gli assemblati contigui al ceppo di riferimento D188 per cercare sequenze corrispondenti a pFiD188 o plasmidi simili a pFID188(Galardini et al., 2011). Get_homologues v. 20170418 è stato utilizzato per raggruppare i geni di ciascuno dei plasmidi della virulenza e degli altri plasmidi lineari di Rhodococcus(Francis et al., 2012). I plasmidi sono stati raggruppati sulla base della presenza/assenza del gene utilizzando le distanze binarie e il metodo di Ward per il clustering (ward.D2). dbCAN HMMs 5.0 è stato scaricato e utilizzato con script ad hoc per identificare i CAZYmes da sequenze genomiche tradotte(Yin et al., 2012). Il database antiSMASH è stato scaricato il 06/2017 e analizzato con antiSMASH ver. 4.0(Blin et al., 2017). Le query utilizzate nelle ricerche di TBLASTN erano sequenze geniche accertate dalla ricerca in letteratura(Bruto et al., 2014; Glick, 2012; Sparacino-Watkins et al., 2014).
HISAT2 è stato utilizzato per allineare le letture di sequenziamento dal ceppo D188-5 alla sequenza genomica di riferimento D188. Le varianti (SNPs) sono state chiamate utilizzando freebayes, filtrate a quelle con punteggio di qualità superiore a 20 utilizzando vcffilter, e annotate utilizzando SNPdat v. 1.0.5(Doran e Creevey, 2013).
Condizioni di crescita delle piante, saggi e analisi dei dati
I saggi di inibizione delle radici delle piantine sono stati eseguiti come descritto in precedenza, con l’eccezione che dopo che i batteri sono stati regolati a OD600 = 0,5, a volte sono stati diluiti o concentrati (Creason etal., 2014b). Sono state analizzate almeno 100 piantine per trattamento. Le immagini sono state scattate a 7 giorni dall’inoculazione (dpi) e i dati sono stati analizzati. Per la quantificazione dei peli delle radici, è stato utilizzato un microscopio a dissezione, dotato di una macchina fotografica, per catturare immagini a 10 e 25 dpi. I peli delle radici all’interno di un segmento di 1 cm, 1 cm sotto lo stelo sono stati quantificati utilizzando ImageJ(Schneider et al., 2012). Per i test di inibizione della citochinina, piantine germinate di tre giorni sono state trapiantate in un mezzo contenente DMSO (controllo) o 0,01-10,0 µM 6-benzilamminopurina (BA) e poi coltivate e quantificate allo stesso modo.
Galle fogliose sono state indotte con il metodo della decapitazione su piante di N. benthamiana di quattro settimane(Creason et al., 2014b). Le immagini sono state scattate a 28 dpi.
I semi diPisum sativum ‘Alaska’ sono stati sterilizzati in superficie in etanolo al 70% per 1 min, candeggina al 10% per 10 min, e lavati tre volte con acqua sterile. I semi sono stati immersi in acqua sterile per 60 minuti e poi placcati su agar d’acqua (15 g di agar/L). Le piastre sono state incubate a 23°C fino a quando i radicali erano circa 5 mm. I semi germinati sono stati immersi in sospensione di isolati di rodococco (OD600 = 0,2) o 10 mM MgCl2 tampone MgCl2 per 45 min. Dieci semi per trattamento sono stati inclusi in ogni esperimento. I semi inoculati sono stati messi in provette sterili contenenti 5 ml di agar nutriente Hoagland. I campioni sono stati incubati per 14 giorni a 23°C con un ciclo di 16/8 luce / buio. Il numero e la lunghezza dello stelo sono stati quantificati a 14 dpi.
L’infezione di UCB-1 pistacchio è stato fatto, con piccole modifiche, secondo i protocolli precedentemente descritti(Stamler et al., 2015b). In breve, gli isolati di controllo, PBTS1, PBTS2, e una miscela 1:1 di PBTS1 e PBTS2 sono stati sospesi in 10 mM MgCl2 (finale OD600 = 0,7). I gruppi di trattamento costituiti da 15 piantine sono stati inoculati con 200 ml di sospensione batterica. Un gruppo di controllo mock-inoculato è stato spruzzato con 200 ml di 10 mM MgCl2. Le piantine inoculate sono state poste in camere di umidità per 14 giorni e le piante sono state mantenute in serra per sette mesi. L’altezza degli alberi e la lunghezza degli internodi sono stati misurati a intervalli di 30 giorni, e le misurazioni finali sono state registrate a 210 dpi.
Se non indicato, tutti gli esperimenti sono stati ripetuti almeno tre volte con risultati simili. Per tutte le serie di dati, i valori anomali sono stati identificati con il metodo ROUT (Q = 1%) e rimossi. I dati sono stati analizzati utilizzando ANOVA a senso unico o bidirezionale seguito dal test di confronto multiplo di Tukey (GraphPad Prism v.7, GraphPad Software, La Jolla, CA, USA). I grafici di box e baffi sono stati generati utilizzando il metodo Tukey; i punti colorati indicano i valori erratici. I mezzi sono indicati con +.
Manipolazioni dell’acido nucleico
Il gene attR è stato amplificato mediante PCR dal DNA genomico D188 e subclonato a valle del promotore del batteriofago L5 nel vettore pJDC165 (Jeff Cirillo, Texas A e M). Il costrutto L5::attR è stato verificato tramite il sequenziamento Sanger. Le cellule competenti Rhodococcus sono state preparate da colture da 3 ml coltivate durante la notte. Le cellule sono state pellettate e lavate due volte con dH2O sterile, freddo, seguito da un lavaggio con glicerolo sterile, freddo al 10%. Le cellule sono state risospese in 50 µl di glicerolo al 10%. Plasmide DNA (0,5-1 µg) è stato aggiunto alle cellule. Dopo 30 minuti di incubazione su ghiaccio, le cellule sono state elettroporate in cuvette di 1 mm di distanza a 2,2 kV. Le cellule sono state risospese in 250 µl di terreno SOC e incubate a 28 ° C con agitazione per 16 ore prima della placcatura su terreno LB con antibiotici appropriati.
Per la PCR sono stati utilizzati i seguenti metodi: 1x ThermoPol tampone di reazione ThermoPol (New England Biolab, Ipswich, MA, USA), 200 μM dNTPs, 0,2 μM di ogni primer, 50 ng modello di DNA genomico, 0,625 unità Taq DNA polimerasi (New England Biolab, Ipswich, MA, USA), in un volume finale di 25 μl. Le condizioni di PCR erano 95°C, 3 min; 30 cicli di 95°C per 30 s, 55°C per 30 s, 72°C per 1 min; 72°C per 10 min; tenuta a 16°C. Le reazioni con l’acqua, invece di un modello di DNA, sono state usate come controllo negativo.
Per LAMP, la miscela di reazione è stata la seguente: 0,5 ng DNA template, 1x tampone di reazione ThermoPol (New England Biolab, Ipswich, MA, USA), 5 mM MgSO4, 140 µM dNTPs, 146 µM idoxinaftolo blu (HNB), 1,6 µM ciascuno 16FIP e 16BIP primer, 0,2 µM ciascuno 16F3 e 16B3 primer, e 12 U Bst polimerasi in un volume finale di 25 µl. Le reazioni sono state incubate a 64°C per 60 minuti e poi raffreddate a 4°C. I tubi sono stati centrifugati brevemente a 8000 rpm.
RPA è stato fatto secondo le istruzioni del produttore (TwistAmp Basic, TwistDx Limited, Cambridge, Regno Unito). Le reazioni sono costituite da 0,48 μM per primer, 29,5 μl tampone di reidratazione, 12,2 μl di acqua e 1,0 μl di DNA genomico. Un volume di 2,5 microlitri 280 mM di acetato di magnesio (MgAc) è stato aggiunto per avviare la reazione. La reazione è stata incubata a 37 ° C per 30 minuti (Fuller et al., 2017). I prodotti sono stati purificati utilizzando il kit di purificazione QIAquick PCR (Qiagen, Germania), eseguito su un gel di agarosio al 2,0%, colorato con bromuro di etidio, e visualizzato sotto la luce UV. I prodotti sono stati verificati tramite il sequenziamento Sanger.
Reazioni RPA accoppiato al rilevamento del flusso laterale sono stati composti da 0,42 μM primer in avanti primer 0,42 μM, 0,42 μM biotina marcato primer inversa, sonda 0,12 micron, 29,5 micron tampone di reidratazione, 12,2 micron di acqua, e 1,0 micron di 25 ng/μl di DNA genomico. Le reazioni sono state aggiunte ad un pellet liofilizzato fornito dal produttore (TwistAmp nfo, TwistDx Limited, Cambridge, UK) con la successiva aggiunta di 2,5 μl di 280 mM MgAc per avviare la reazione. Dopo la successiva incubazione a 37°C per 30 minuti, l’amplicone a doppia etichettatura è stato visualizzato utilizzando un astina di flusso laterale (Milenia Biotec GMbH, Germania). Un microlitro del prodotto RPA è stato diluito in 49 μl 1.0x PBST e 10 μl della diluizione sono stati applicati alla base dell’asta di livello, che è stata successivamente immersa in 100 μl 1.0 PBST a temperatura ambiente fino alla visualizzazione della banda di controllo positivo, tipicamente della durata di due minuti.
Le sequenze di primer e sonde, e le loro modifiche, sono descritte nel file supplementare 1I.
References
- Adékambi T, Butler RW, Hanrahan F, Delcher AL, Drancourt M, Shinnick TM. Core gene set as the basis of multilocus sequence analysis of the subclass Actinobacteridae. PLoS ONE. 2011; 6DOI | PubMed
- Andrews S. 2014. Publisher Full Text
- Bai Y, Müller DB, Srinivas G, Garrido-Oter R, Potthoff E, Rott M, Dombrowski N, Münch PC, Spaepen S, Remus-Emsermann M, Hüttel B, McHardy AC, Vorholt JA, Schulze-Lefert P. Functional overlap of the Arabidopsis leaf and root microbiota. Nature. 2015; 528:364-369. DOI | PubMed
- Bankevich A, Nurk S, Antipov D, Gurevich AA, Dvorkin M, Kulikov AS, Lesin VM, Nikolenko SI, Pham S, Prjibelski AD, Pyshkin AV, Sirotkin AV, Vyahhi N, Tesler G, Alekseyev MA, Pevzner PA. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology. 2012; 19:455-477. DOI | PubMed
- Barea JM, Pozo MJ, Azcón R, Azcón-Aguilar C. Microbial co-operation in the rhizosphere. Journal of Experimental Botany. 2005; 56:1761-1778. DOI | PubMed
- Blackmon H, Adams R. 2015. Publisher Full Text | DOI
- Blin K, Wolf T, Chevrette MG, Lu X, Schwalen CJ, Kautsar SA, Suarez Duran HG, de los Santos ELC, Kim HU, Nave M, Dickschat JS, Mitchell DA, Shelest E, Breitling R, Takano E, Lee SY, Weber T, Medema MH. antiSMASH 4.0—improvements in chemistry prediction and gene cluster boundary identification. Nucleic Acids Research. 2017; 45:W36-W41. DOI | PubMed
- Bloom SM, Bijanki VN, Nava GM, Sun L, Malvin NP, Donermeyer DL, Dunne WM, Allen PM, Stappenbeck TS. Commensal Bacteroides species induce colitis in host-genotype-specific fashion in a mouse model of inflammatory bowel disease. Cell Host & Microbe. 2011; 9:390-403. DOI | PubMed
- Bodenhausen N, Horton MW, Bergelson J. Bacterial communities associated with the leaves and the roots of Arabidopsis thaliana. PLoS One. 2013; 8DOI | PubMed
- Bruto M, James A, Petton B, Labreuche Y, Chenivesse S, Alunno-Bruscia M, Polz MF, Le Roux F. Vibrio crassostreae, a benign oyster colonizer turned into a pathogen after plasmid acquisition. The ISME Journal. 2017; 11:1043-1052. DOI | PubMed
- Bruto M, Prigent-Combaret C, Muller D, Moënne-Loccoz Y. Analysis of genes contributing to plant-beneficial functions in plant growth-promoting rhizobacteria and related proteobacteria. Scientific Reports. 2014; 4DOI | PubMed
- Bulgarelli D, Rott M, Schlaeppi K, Ver Loren van Themaat E, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E, Peplies J, Gloeckner FO, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota. Nature. 2012; 488:91-95. DOI | PubMed
- Bushnell B. 2014. Publisher Full Text
- Chen J. 2012. Publisher Full Text
- Comas I, Coscolla M, Luo T, Borrell S, Holt KE, Kato-Maeda M, Parkhill J, Malla B, Berg S, Thwaites G, Yeboah-Manu D, Bothamley G, Mei J, Wei L, Bentley S, Harris SR, Niemann S, Diel R, Aseffa A, Gao Q, Young D, Gagneux S. Out-of-Africa migration and Neolithic coexpansion of Mycobacterium tuberculosis with modern humans. Nature Genetics. 2013; 45:1176-1182. DOI | PubMed
- Contreras-Moreira B, Vinuesa P. GET_HOMOLOGUES, a versatile software package for scalable and robust microbial pangenome analysis. Applied and Environmental Microbiology. 2013; 79:7696-7701. DOI | PubMed
- Creason AL, Davis EW, Putnam ML, Vandeputte OM, Chang JH. Use of whole genome sequences to develop a molecular phylogenetic framework for Rhodococcus fascians and the Rhodococcus genus. Frontiers in Plant Science. 2014a; 5DOI | PubMed
- Creason AL, Vandeputte OM, Savory EA, Davis EW, Putnam ML, Hu E, Swader-Hines D, Mol A, Baucher M, Prinsen E, Zdanowska M, Givan SA, El Jaziri M, Loper JE, Mahmud T, Chang JH. Analysis of genome sequences from plant pathogenic Rhodococcus reveals genetic novelties in virulence loci. PLoS One. 2014b; 9DOI | PubMed
- Crespi M, Messens E, Caplan AB, van Montagu M, Desomer J. Fasciation induction by the phytopathogen Rhodococcus fascians depends upon a linear plasmid encoding a cytokinin synthase gene. The EMBO journal. 1992; 11:795-804. PubMed
- Crespi M, Vereecke D, Temmerman W, Van Montagu M, Desomer J. The fas operon of Rhodococcus fascians encodes new genes required for efficient fasciation of host plants. Journal of Bacteriology. 1994; 176:2492-2501. DOI | PubMed
- Croucher NJ, Harris SR, Fraser C, Quail MA, Burton J, van der Linden M, McGee L, von Gottberg A, Song JH, Ko KS, Pichon B, Baker S, Parry CM, Lambertsen LM, Shahinas D, Pillai DR, Mitchell TJ, Dougan G, Tomasz A, Klugman KP, Parkhill J, Hanage WP, Bentley SD. Rapid pneumococcal evolution in response to clinical interventions. Science. 2011; 331:430-434. DOI | PubMed
- Davis Ii EW, Weisberg AJ, Tabima JF, Grunwald NJ, Chang JH. Gall-ID: tools for genotyping gall-causing phytopathogenic bacteria. PeerJ. 2016; 4DOI | PubMed
- de Carvalho CC, Costa SS, Fernandes P, Couto I, Viveiros M. Membrane transport systems and the biodegradation potential and pathogenicity of genus Rhodococcus. Frontiers in Physiology. 2014; 5DOI | PubMed
- de Lajudie P, Willems A, Nick G, Mohamed SH, Torck U, Coopman R, Filali-Maltouf A, Kersters K, Dreyfus B, Lindström K, Gillis M. Agrobacterium bv. 1 strains isolated from nodules of tropical legumes. Systematic and Applied Microbiology. 1999; 22:119-132. DOI
- Desomer J, Dhaese P, Van Montagu M. Conjugative transfer of cadmium resistance plasmids in Rhodococcus fascians strains. Journal of Bacteriology. 1988; 170:2401-2405. DOI | PubMed
- Doran AG, Creevey CJ. Snpdat: easy and rapid annotation of results from de novo snp discovery projects for model and non-model organisms. BMC Bioinformatics. 2013; 14DOI | PubMed
- Drogue B, Doré H, Borland S, Wisniewski-Dyé F, Prigent-Combaret C. Which specificity in cooperation between phytostimulating rhizobacteria and plants?. Research in Microbiology. 2012; 163:500-510. DOI | PubMed
- Francis I, De Keyser A, De Backer P, Simón-Mateo C, Kalkus J, Pertry I, Ardiles-Diaz W, De Rycke R, Vandeputte OM, El Jaziri M, Holsters M, Vereecke D. pFiD188, the linear virulence plasmid of Rhodococcus fascians D188. Molecular Plant-Microbe Interactions. 2012; 25:637-647. DOI | PubMed
- Francis IM, Stes E, Zhang Y, Rangel D, Audenaert K, Vereecke D. Mining the genome of Rhodococcus fascians, a plant growth-promoting bacterium gone astray. New Biotechnology. 2016; 33:706-717. DOI | PubMed
- Fuller SL, Savory EA, Weisberg AJ, Buser JZ, Gordon MI, Putnam ML, Chang JH. Isothermal amplification and lateral-flow assay for detecting crown-gall-causing agrobacterium spp. Phytopathology. 2017; 107:1062-1068. DOI | PubMed
- Galardini M, Biondi EG, Bazzicalupo M, Mengoni A. CONTIGuator: a bacterial genomes finishing tool for structural insights on draft genomes. Source Code for Biology and Medicine. 2011; 6DOI | PubMed
- Glaeser SP, Imani J, Alabid I, Guo H, Kumar N, Kämpfer P, Hardt M, Blom J, Goesmann A, Rothballer M, Hartmann A, Kogel KH. Non-pathogenic Rhizobium radiobacter F4 deploys plant beneficial activity independent of its host Piriformospora indica. The ISME Journal. 2016; 10:871-884. DOI | PubMed
- Glick BR. Plant growth-promoting bacteria: mechanisms and applications. Scientifica. 2012; 2012:15. DOI | PubMed
- Glick BR. Bacteria with ACC deaminase can promote plant growth and help to feed the world. Microbiological Research. 2014; 169:30-39. DOI | PubMed
- Gutjahr C, Parniske M. Cell and developmental biology of arbuscular mycorrhiza symbiosis. Annual Review of Cell and Developmental Biology. 2013; 29:593-617. DOI | PubMed
- Hao X, Xie P, Johnstone L, Miller SJ, Rensing C, Wei G. Genome sequence and mutational analysis of plant-growth-promoting bacterium Agrobacterium tumefaciens CCNWGS0286 Isolated from a zinc-lead mine tailing. Applied and Environmental Microbiology. 2012; 78:5384-5394. DOI | PubMed
- Harris SR, Cartwright EJ, Török ME, Holden MT, Brown NM, Ogilvy-Stuart AL, Ellington MJ, Quail MA, Bentley SD, Parkhill J, Peacock SJ. Whole-genome sequencing for analysis of an outbreak of meticillin-resistant Staphylococcus aureus: a descriptive study. The Lancet Infectious Diseases. 2013; 13:130-136. DOI | PubMed
- Harris SR, Feil EJ, Holden MT, Quail MA, Nickerson EK, Chantratita N, Gardete S, Tavares A, Day N, Lindsay JA, Edgeworth JD, de Lencastre H, Parkhill J, Peacock SJ, Bentley SD. Evolution of MRSA during hospital transmission and intercontinental spread. Science. 2010; 327:469-474. DOI | PubMed
- Hong CE, Jeong H, Jo SH, Jeong JC, Kwon SY, An D, Park JM. A leaf-inhabiting endophytic bacterium, rhodococcus sp. kb6, enhances sweet potato resistance to black rot disease caused by ceratocystis fimbriata. Journal of Microbiology and Biotechnology. 2016; 26:488-492. DOI | PubMed
- Hong CE, Jo SH, Moon JY, Lee J-S, Kwon S-Y, Park JM. Isolation of novel leaf-inhabiting endophytic bacteria in Arabidopsis thaliana and their antagonistic effects on phytophathogens. Plant Biotechnology Reports. 2015; 9:451-458. DOI
- Kamvar ZN, Tabima JF, Grünwald NJ. Poppr: an R package for genetic analysis of populations with clonal, partially clonal, and/or sexual reproduction. PeerJ. 2014; 2DOI | PubMed
- Katoh K, Standley DM. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Molecular Biology and Evolution. 2013; 30:772-780. DOI | PubMed
- Kieber JJ, Schaller GE. Cytokinins. The Arabidopsis Book. 2014; 12:e0168. DOI | PubMed
- Knaus BJ, Grünwald NJ. vcfr: a package to manipulate and visualize variant call format data in R. Molecular Ecology Resources. 2017; 17:44-53. DOI | PubMed
- Kumar S, Jones M, Koutsovoulos G, Clarke M, Blaxter M. Blobology: exploring raw genome data for contaminants, symbionts and parasites using taxon-annotated GC-coverage plots. Frontiers in Genetics. 2013; 4DOI | PubMed
- Lacroix B, Citovsky V. A functional bacterium-to-plant dna transfer machinery of rhizobium etli. PLOS Pathogens. 2016; 12DOI | PubMed
- Langmead B, Trapnell C, Pop M, Salzberg SL. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 2009; 10DOI | PubMed
- Larkin MJ, Kulakov LA, Allen CC. Biodegradation and Rhodococcus–masters of catabolic versatility. Current Opinion in Biotechnology. 2005; 16:282-290. DOI | PubMed
- Lebeis SL, Paredes SH, Lundberg DS, Breakfield N, Gehring J, McDonald M, Malfatti S, Glavina del Rio T, Jones CD, Tringe SG, Dangl JL. Plant microbiome. salicylic acid modulates colonization of the root microbiome by specific bacterial taxa. Science. 2015; 349:860-864. DOI | PubMed
- Letek M, González P, Macarthur I, Rodríguez H, Freeman TC, Valero-Rello A, Blanco M, Buckley T, Cherevach I, Fahey R, Hapeshi A, Holdstock J, Leadon D, Navas J, Ocampo A, Quail MA, Sanders M, Scortti MM, Prescott JF, Fogarty U, Meijer WG, Parkhill J, Bentley SD, Vázquez-Boland JA. The genome of a pathogenic rhodococcus: cooptive virulence underpinned by key gene acquisitions. PLoS Genetics. 2010; 6DOI | PubMed
- Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R, 1000 Genome Project Data Processing Subgroup. The sequence alignment/map format and SAMtools. Bioinformatics. 2009; 25:2078-2079. DOI | PubMed
- Lin D, Koskella B. Friend and foe: factors influencing the movement of the bacterium Helicobacter pylori along the parasitism-mutualism continuum. Evolutionary Applications. 2015; 8:9-22. DOI | PubMed
- Lombard V, Golaconda Ramulu H, Drula E, Coutinho PM, Henrissat B. The carbohydrate-active enzymes database (CAZy) in 2013. Nucleic Acids Research. 2014; 42:D490-D495. DOI | PubMed
- Lundberg DS, Lebeis SL, Paredes SH, Yourstone S, Gehring J, Malfatti S, Tremblay J, Engelbrektson A, Kunin V, Del Rio TG, Edgar RC, Eickhorst T, Ley RE, Hugenholtz P, Tringe SG, Dangl JL. Defining the core Arabidopsis thaliana root microbiome. Nature. 2012; 488:86-90. DOI | PubMed
- Maes T, Vereecke D, Ritsema T, Cornelis K, Thu HN, Van Montagu M, Holsters M, Goethals K. The att locus of Rhodococcus fascians strain D188 is essential for full virulence on tobacco through the production of an autoregulatory compound. Molecular Microbiology. 2001; 42:13-28. DOI | PubMed
- Magori S, Oka-Kira E, Shibata S, Umehara Y, Kouchi H, Hase Y, Tanaka A, Sato S, Tabata S, Kawaguchi M. Too much love, a root regulator associated with the long-distance control of nodulation in Lotus japonicus. Molecular Plant-Microbe Interactions. 2009; 22:259-268. DOI | PubMed
- McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo MA. The genome analysis toolkit: a mapreduce framework for analyzing next-generation dna sequencing data. Genome Research. 2010; 20:1297-1303. DOI | PubMed
- Miteva VI, Sheridan PP, Brenchley JE. Phylogenetic and physiological diversity of microorganisms isolated from a deep greenland glacier ice core. Applied and Environmental Microbiology. 2004; 70:202-213. DOI | PubMed
- Mutreja A, Kim DW, Thomson NR, Connor TR, Lee JH, Kariuki S, Croucher NJ, Choi SY, Harris SR, Lebens M, Niyogi SK, Kim EJ, Ramamurthy T, Chun J, Wood JL, Clemens JD, Czerkinsky C, Nair GB, Holmgren J, Parkhill J, Dougan G. Evidence for several waves of global transmission in the seventh cholera pandemic. Nature. 2011; 477:462-465. DOI | PubMed
- Nikolaeva EV, Kang S, Olson TN, Kim S. Real-time PCR detection of rhodococcus fascians and discovery of new plants associated with r. fascians in pennsylvania. Plant Health Progress. 2012. DOI
- Nikolaeva EV, Park S-Y, Kang S, Olson TN, Kim SH. Ratios of cells with and without virulence genes in rhodococcus fascians populations correlate with degrees of symptom development. Plant Disease. 2009; 93:499-506. DOI | PubMed
- Nyholm SV, McFall-Ngai MJ. The winnowing: establishing the squid-vibrio symbiosis. Nature Reviews Microbiology. 2004; 2:632-642. DOI | PubMed
- Parkhill J, Wren BW. Bacterial epidemiology and biology–lessons from genome sequencing. Genome Biology. 2011; 12DOI | PubMed
- Pertry I, Václavíková K, Depuydt S, Galuszka P, Spíchal L, Temmerman W, Stes E, Schmülling T, Kakimoto T, Van Montagu MC, Strnad M, Holsters M, Tarkowski P, Vereecke D. Identification of Rhodococcus fascians cytokinins and their modus operandi to reshape the plant. PNAS. 2009; 106:929-934. DOI | PubMed
- Pertry I, Václavíková K, Gemrotová M, Spíchal L, Galuszka P, Depuydt S, Temmerman W, Stes E, De Keyser A, Riefler M, Biondi S, Novák O, Schmülling T, Strnad M, Tarkowski P, Holsters M, Vereecke D. Rhodococcus fascians impacts plant development through the dynamic fas-mediated production of a cytokinin mix. Molecular Plant-Microbe Interactions. 2010; 23:1164-1174. DOI | PubMed
- Picard Tools. 2015. Publisher Full Text
- Piepenburg O, Williams CH, Stemple DL, Armes NA. DNA detection using recombination proteins. PLoS Biology. 2006; 4DOI | PubMed
- Pieterse CM, Zamioudis C, Berendsen RL, Weller DM, Van Wees SC, Bakker PA. Induced systemic resistance by beneficial microbes. Annual Review of Phytopathology. 2014; 52:347-375. DOI | PubMed
- Putnam ML, Miller ML. Rhodococcus fascians in Herbaceous Perennials. Plant Disease. 2007; 91:1064-1076. DOI | PubMed
- Qin S, Li J, Chen HH, Zhao GZ, Zhu WY, Jiang CL, Xu LH, Li WJ. Isolation, diversity, and antimicrobial activity of rare actinobacteria from medicinal plants of tropical rain forests in Xishuangbanna, China. Applied and Environmental Microbiology. 2009; 75:6176-6186. DOI | PubMed
- Qin S, Xing K, Jiang JH, Xu LH, Li WJ. Biodiversity, bioactive natural products and biotechnological potential of plant-associated endophytic actinobacteria. Applied Microbiology and Biotechnology. 2011; 89:457-473. DOI | PubMed
- Reid DE, Ferguson BJ, Hayashi S, Lin YH, Gresshoff PM. Molecular mechanisms controlling legume autoregulation of nodulation. Annals of Botany. 2011; 108:789-795. DOI | PubMed
- Revell LJ. Phytools: an R package for phylogenetic comparative biology (and other things). Methods in Ecology and Evolution. 2012; 3:217-223. DOI
- Sachs JL, Skophammer RG, Regus JU. Evolutionary transitions in bacterial symbiosis. PNAS. 2011; 108:10800-10807. DOI | PubMed
- Salam N, Khieu TN, Liu MJ, Vu TT, Chu-Ky S, Quach NT, Phi QT, Narsing Rao MP, Fontana A, Sarter S, Li WJ. Endophytic actinobacteria associated with dracaena cochinchinensis lour.: Isolation, diversity, and their cytotoxic activities. BioMed Research International. 2017; 2017DOI | PubMed
- Schneider CA, Rasband WS, Eliceiri KW. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 2012; 9:671-675. DOI | PubMed
- Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 2014; 30:2068-2069. DOI | PubMed
- Serdani M, Curtis M, Miller ML, Kraus J, Putnam ML. Loop-mediated isothermal amplification and polymerase chain reaction methods for specific and rapid detection of Rhodococcus fascians. Plant Disease. 2013; 97:517-529. DOI | PubMed
- Soucy SM, Huang J, Gogarten JP. Horizontal gene transfer: building the web of life. Nature Reviews Genetics. 2015; 16:472-482. DOI | PubMed
- Spaepen S, Vanderleyden J, Remans R. Indole-3-acetic acid in microbial and microorganism-plant signaling. FEMS Microbiology Reviews. 2007; 31:425-448. DOI | PubMed
- Sparacino-Watkins C, Stolz JF, Basu P. Nitrate and periplasmic nitrate reductases. Chem. Soc. Rev.. 2014; 43:676-706. DOI | PubMed
- Stamatakis A. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies. Bioinformatics. 2014; 30:1312-1313. DOI | PubMed
- Stamler RA, Heerema R, Randall JJ. First Report of Phytopathogenic Rhodococcus Isolates on Pistachio Bushy Top Syndrome ‘UCB-1’ Rootstock in New Mexico. Plant Disease. 2015a; 99:1854. DOI
- Stamler RA, Kilcrease J, Kallsen C, Fichtner EJ, Cooke P, Heerema RJ, Randall JJ. First Report of Rhodococcus Isolates Causing Pistachio Bushy Top Syndrome on ‘UCB-1’ Rootstock in California and Arizona. Plant Disease. 2015b; 99:1468-1476. DOI | PubMed
- Stamler RA, Vereecke D, Zhang Y, Schilkey F, Devitt N, Randall JJ. Complete genome and plasmid sequences for Rhodococcus fascians D188 and draft sequences for Rhodococcus Isolates PBTS 1 and PBTS 2. Genome Announcements. 2016; 4DOI | PubMed
- Stes E, Vandeputte OM, El Jaziri M, Holsters M, Vereecke D. A successful bacterial coup d’état: how Rhodococcus fascians redirects plant development. Annual Review of Phytopathology. 2011; 49:69-86. DOI | PubMed
- Temmerman W, Ritsema T, Simón-Mateo C, Van Montagu M, Mironov V, Inzé D, Goethals K, Holsters M. The fas locus of the phytopathogen Rhodococcus fascians affects mitosis of tobacco BY-2 cells. FEBS Letters. 2001; 492:127-132. DOI | PubMed
- Temmerman W, Vereecke D, Dreesen R, Van Montagu M, Holsters M, Goethals K. Leafy gall formation is controlled by fasR, an AraC-type regulatory gene in Rhodococcus fascians. Journal of Bacteriology. 2000; 182:5832-5840. DOI | PubMed
- Vacheron J, Desbrosses G, Bouffaud ML, Touraine B, Moënne-Loccoz Y, Muller D, Legendre L, Wisniewski-Dyé F, Prigent-Combaret C. Plant growth-promoting rhizobacteria and root system functioning. Frontiers in Plant Science. 2013; 4DOI | PubMed
- Verbon EH, Liberman LM. Beneficial microbes affect endogenous mechanisms controlling root development. Trends in Plant Science. 2016; 21:218-229. DOI | PubMed
- Vereecke D, Cornelis K, Temmerman W, Jaziri M, Van Montagu M, Holsters M, Goethals K. Chromosomal locus that affects pathogenicity of Rhodococcus fascians. Journal of Bacteriology. 2002; 184:1112-1120. DOI | PubMed
- Walker TM, Ip CL, Harrell RH, Evans JT, Kapatai G, Dedicoat MJ, Eyre DW, Wilson DJ, Hawkey PM, Crook DW, Parkhill J, Harris D, Walker AS, Bowden R, Monk P, Smith EG, Peto TE. Whole-genome sequencing to delineate Mycobacterium tuberculosis outbreaks: a retrospective observational study. The Lancet Infectious Diseases. 2013; 13:137-146. DOI | PubMed
- Wang LL, Wang ET, Liu J, Li Y, Chen WX. Endophytic occupation of root nodules and roots of Melilotus dentatus by Agrobacterium tumefaciens. Microbial Ecology. 2006; 52:436-443. DOI | PubMed
- Wopereis J, Pajuelo E, Dazzo FB, Jiang Q, Gresshoff PM, De Bruijn FJ, Stougaard J, Szczyglowski K. Short root mutant of Lotus japonicus with a dramatically altered symbiotic phenotype. The Plant Journal. 2000; 23:97-114. DOI | PubMed
- Yin Y, Mao X, Yang J, Chen X, Mao F, Xu Y. dbCAN: a web resource for automated carbohydrate-active enzyme annotation. Nucleic Acids Research. 2012; 40:W445-W451. DOI | PubMed
Fonte
Savory EA, Fuller SL, Weisberg AJ, Thomas WJ, Gordon MI, et al. () Evolutionary transitions between beneficial and phytopathogenic . eLife 6e30925. https://doi.org/10.7554/eLife.30925





